一种基于神经归纳矩阵补充的图卷积网络疾病相关lncRNA基因预测方法与流程
一种基于神经归纳矩阵补充的图卷积网络疾病相关lncrna基因预测方法
技术领域
1.本发明所涉及到的生物信息领域,更具体地说,涉及一种基于神经归纳矩阵补充的图卷积网络疾病相关lncrna基因预测方法。
背景技术:
2.由于lncrna是各种生物过程的关键调控因子,并且lncrna的表达或功能异常与人类疾病的发生密切相关,它不仅承担遗传信息中间载体的辅助性角色,而且在各种疾病的转移和进展中发挥着至关重要的作用,随着各类研究的推进和各种lncrnas的大量发现,lncrnas的研究将是rna基因组研究非常吸引人的一个方向。而且本方法可以节省实验室成本和时间成本。因此,识别lncrnas与疾病之间的关联对于了解lncrnas在疾病过程中起的作用非常重要,是生物信息方向的一个重要研究部分。
技术实现要素:
3.根据现有的疾病相关lncrna基因预测方法中的不足,本发明提出了一种基于神经归纳矩阵补充的图卷积网络疾病相关lncrna基因预测方法,该方法不仅节省实验室成本和时间成本,而且精确性高,其中本方法使用图卷积网络和神经归纳矩阵补全方法,对lncrnas、diseases包含的特征进行学习,使之得到相应的特征矩阵,然后将学习的特征嵌入到神经归纳矩阵完成模型中,使用非线性神经评级模型来捕捉lncrna和疾病特征之间复杂和微妙的相互作用,最后通过整合完成预测,而且该方法的预测性能要优于其他方法,更符合生物信息领域的特点。
4.通过已知数据库中的lncrna
‑
disease关联利用两种疾病的有向无环图(dag)和高斯交互剖面核相似度来计算疾病的语义相似性,利用lncrnas的功能相似性评分和高斯相互作用谱核相似性评分来计算lncrnas相似性,得到疾病的语义相似性和lncrnas的功能相似性之后利用图卷积网络来监督学习疾病和lncrnas的潜在特征,之后利用神经归纳矩阵补全方法,使用非线性神经评级模型来捕捉lncrna和疾病特征之间复杂和微妙的相互作用。
5.该方法中所提到的数据来源于mesh数据库和dincrna数据库。
6.本发明中计算疾病之间的语义相似度值和高斯相互作用轮廓核函数是计算疾病语义相似性的主要方法,用其表示疾病之间的语义相似度,有相似性取值为1,否则为0。
7.给定一种疾病d,可以描述为,dag
d =(d,n
d
,e
d
)其中n
d
是包括自身的祖先节点集,e
d
是连接这些疾病的相应边集,如果疾病e在dag
d
中,其对疾病d的贡献计算如下。
8.其中ε是连接疾病d及e的子疾病e’的语义贡献因子,在d的dag中,疾病d对其自身
语义值的贡献被定义为1,因此,我们可以通过下面的公式来计算疾病d的语义值。
9.我们通过在疾病dag中d(i),d(j)的相对位置来计算它们之间的第一语义相似度值ds1(d(i),d(j)),公式如下:由于该计算ds1的模型没有考虑在dags中发生的疾病的数量,忽略了不同疾病的重要性,即如果疾病a只出现在dags(i)中,而疾病b既出现在dag(i)中,也出现在其他疾病的dag中的话,那么对疾病i来说a有比b更高的语义贡献值。相应公式如下:其中num(dags(e))代表包括疾病e在内的dags数量,num(diseases)代表所有疾病的数量,由此,我们可以得到疾病的第二个语义相似性模型,对于疾病d(i)和疾病d(j),它们之间的语义相似性值ds2可以计算如下:由于在mesh数据库中只能找到部分疾病的dag,因此,为了使疾病信息更加全面,我们引入高斯相互作用轮廓核函数计算其他疾病之间的相似程度,疾病的核函数值是从每一个已知lncrna
‑
disease之间的关系中导出来的:disease之间的关系中导出来的:其中θ
d
表示内核带宽参数,n表示矩阵中的疾病数量,矩阵a代表已知的lncrnas和疾病之间是否有联系,如果lncrnas和疾病之间有关系,则a
ij
=1,如果a
ij
=0,则说明关联是未知的或未观察到的。
10.由此得到疾病的语义相似度矩阵为:如果disease d
i
和disease d
j
之间有相似性,则d
ij
=1,否则为0,表示关联是未知的或未观察到的。
11.然后计算lncrnas之间的功能相似性评分和高斯相互作用谱核相似性评分是计算lncrnas相似性的主要方法,该方法通过lncrna的功能相似性评分和高斯相互作用谱核相似性评分来测量lncrna相似性,过程应该满足公式:
其中,fs(i,j)是已知的来自dincrna数据库的lncrna之间的功能相似性评分,lncgs(i,j)是高斯相互作用轮廓核相似性分数,用于补充缺失的条目。lncgs的具体计算方法如下。法如下。
12.其中θ
l
表示内核带宽参数,m表示矩阵中的疾病数量,矩阵l代表已知的lncrnas和疾病之间是否有联系,如果lncrnas和疾病之间有关系,则l
ij
=1,如果l
ij
=0,则说明关联是未知的或未观察到的。
13.得到疾病的语义相似性和lncrnas的功能相似性之后利用图卷积网络来监督学习疾病和lncrnas的潜在特征,之后利用神经归纳矩阵补全方法,使用非线性神经评级模型来捕捉lncrna和疾病特征之间复杂和微妙的相互作用,相似为1,否则为0,表明关联是未知的或未观察到的。
附图说明
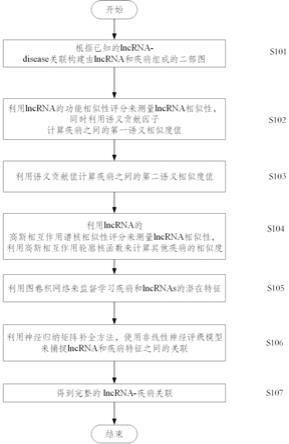
图1为根据本发明示例的一种基于神经归纳矩阵补充的图卷积网络疾病相关lncrna基因预测方法的流程图。
具体实施方式
14.如图1所示,本发明提出一种基于神经归纳矩阵补充的图卷积网络疾病相关lncrna基因预测方法。
15.s101: 根据已知的lncrna
‑
disease关联构建由lncrna和疾病组成的二部图。
16.s102: 计算疾病之间的第一语义相似度值,给定一种疾病d,可以描述为,dag
d =(d,n
d
,e
d
)其中n
d
是包括自身的祖先节点集,e
d
是连接这些疾病的相应边集。如果疾病e在dag
d
中,其对疾病d的贡献计算如下:其中ε是连接疾病d及e的子疾病e’的语义贡献因子,在d的dag中,疾病d对其自身语义值的贡献被定义为1,因此,我们可以通过下面的公式来计算疾病d的语义值:我们通过在疾病dag中d(i),d(j)的相对位置来计算它们之间的第一语义相似度值ds1(d(i),d(j)),公式如下:
然后,通过lncrna的功能相似性评分来测量lncrna相似性。
17.s103: 计算疾病之间的第二语义相似度值由于该计算ds1的模型没有考虑在dags中发生的疾病的数量,忽略了不同疾病的重要性,即如果疾病a只出现在dags(i)中,而疾病b既出现在dag(i)中,也出现在其他疾病的dag中的话,那么对疾病i来说a有比b更高的语义贡献值。相应公式如下:其中num(dags(e))代表包括疾病e在内的dags数量,num(diseases)代表所有疾病的数量,由此,我们可以得到疾病的第二个语义相似性模型,对于疾病d(i)和疾病d(j),它们之间的语义相似性值ds2可以计算如下。
18.s104:为了使疾病信息更加全面,引入高斯相互作用轮廓核函数来计算其他疾病的相似度,疾病的高斯相互作用轮廓核函数值是从每一个已知的lncrna
‑
disease之间的关系中导出来的:系中导出来的:其中θ
d
表示内核带宽参数,n表示矩阵中的疾病数量,矩阵a代表已知的lncrnas和疾病之间是否有联系,如果lncrnas和疾病之间有关系,则a
ij
=1,如果a
ij
=0,则说明关联是未知的或未观察到的,由此得到疾病的语义相似度矩阵为:如果disease d
i
和disease d
j
之间有相似性,则d
ij
=1,否则为0,表示关联是未知的或未观察到的,然后通过高斯相互作用谱核相似性评分来测量lncrna相似性,过程应该满足公式:其中,fs(i,j)是已知的来自dincrna数据库的lncrna之间的功能相似性评分,lncgs(i,j)是高斯相互作用轮廓核相似性分数,用于补充缺失的条目。lncgs的具体计算方法如下:
其中θ
l
表示内核带宽参数,m表示矩阵中的疾病数量,矩阵l代表已知的lncrnas和疾病之间是否有联系,如果lncrnas和疾病之间有关系,则l
ij
=1,如果l
ij
=0,则说明关联是未知的或未观察到的。
19.s105:利用图卷积网络来监督学习疾病和lncrnas的潜在特征。
20.s106: 利用神经归纳矩阵补全方法,使用非线性神经评级模型来捕捉lncrna和疾病特征之间复杂和微妙的相互作用,最后得到完整的 lncrna
‑
疾病关联。
21.s107:最终得到完整的lncrna
‑
disease关联。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1