一种基于lncRNA对的结肠癌预后预测模型及其构建方法与流程
一种基于lncrna对的结肠癌预后预测模型及其构建方法
技术领域
1.本发明属于生物信息学领域,使用机器学习方法根据已有的数据建立模型,具体涉及一种结肠癌预后预测模型及其构建方法。
2.背景
3.结肠癌(colon cancer)是发生于结肠部位的消化道恶性肿瘤,是最常见的消化道恶性肿瘤之一。目前,传统的治疗手段包括手术、化疗和放疗,但对于结肠癌患者生存期没有明显改善。近年来,随着分子靶向药物的出现,例如针对表皮生长因子受体(egfr)的单克隆抗体对肠癌晚期患者有明显疗效,使其中位生存期达到2年,但由于kras等的突变导致抗-egrf单克隆抗体耐药的发生使得该靶向药物治疗效果大大降低。因此,迫切需要理解结肠癌的分子机制并发现新的治疗靶标和治疗方法。
4.长链非编码rna(lncrnrna)是一种缺乏编码蛋白潜力的转录本,具有超过200个核苷酸的长非编码rna(lncrna),可以通过mountainous mechanisms修饰与多种基因和蛋白质相互作用来发挥功能。通过参与基本的发病机制,许多lncrna作为致癌基因或肿瘤抑制基因被鉴定为与癌变有关,包括消化道癌、造血癌、泌尿系癌、肺癌和乳腺癌。它能显示出关键位置,如抗原暴露、识别和免疫渗透。因此,免疫相关的lncrnas对预测肿瘤进展和预后的潜力引起了越来越多的关注。
5.生物信息学是生物学与计算机科学以及应用数学等学科相互交叉而形成的一门新兴学科。它综合运用生物学、计算机科学和数学等多方面知识与方法,可以进行有效的数据挖掘,对不同的生物学数据(基因表达、dna、rna等)都有很好的分析和解释能力。
6.综上,本发明在生物信息学的基础上,对数据进行挖掘分析,使用机器学习手段进行优化建模,建立出一个基于lncrna对的结肠癌预后预测模型。
技术实现要素:
7.本发明的目的是克服现有技术和方法的不足,提供一种基于lncrna对的结肠癌预后预测模型及其构建方法,从分子和免疫相关水平实现结肠癌患者预后的风险分层。
8.为实现上述目的,本发明采用如下技术方案:
9.一种基于lncrna对的结肠癌预后预测模型构建方法,包括以下步骤:
10.数据预处理,收集结肠癌原始基因表达数据和相对应临床数据,结合人类基因注释文件进行id转换,提取其中的lncrna,再结合人免疫基因进行相关性分析,筛选出免疫相关的lncrna;
11.对数据划分训练集和验证集,划分训练集正常样本和肿瘤样本,进行差异性分析,提出具有差异的lncrna,建立lncrna对;
12.使用单因素cox和lasso回归,多因素cox筛选预后预测模型参数,建立模型方程,并计算风险分数(riskscore),得到结肠癌预后预测模型;
13.验证模型的优劣性。
14.优选地,所述结肠癌原始基因表达数据和相应临床数据是在tcga数据库下载得
到;人类基因注释文件是在gencode下载得到;人免疫基因是在immport下载得到。
15.优选地,所述提取lncrna是使用strawberry perl编写程序完成,所述筛选lncrna是使用r语言编写程序完成。
16.优选地,所述对数据划分训练集和验证集使用随机划分的方法。
17.优选地,所述相关性分析使用皮尔逊相关系数(pearson correlation coefficient),参数设置范围为0.4<|pccs|<1,p
‑
value<0.001。
18.优选地,所述差异性分析使用威尔科克森符号秩检验(wilcoxon signed ranktest),参数设置范围为fdr<0.05,|log2fc|>1。
19.优选地,所述单因素cox和多因素cox参数设置范围为p
‑
value<0.001。
20.优选地,所述模型方程使用cox风险回归模型,具体方程为x
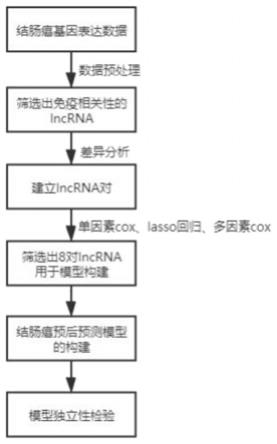
i
是模型中lncrna对的系数,β
i
是模型中lncrna对。
21.优选地,所述验证模型的优劣性,使用指标包括受试者工作曲线(roc)、auc、生存曲线以及临床特征比较;
22.所述roc曲线横坐标是false positive rate(fpr),纵坐标是true positive rate(tpr),划分高低风险的值(cutoff)使用约登指数(youden index=tpr
‑
fpr);
23.所述auc为roc曲线下面积,并且绘制连续三年的roc;
24.所述生存曲线使用kaplan
‑
meier法,绘制各临床特征的roc并结合模型使用单因素cox和多因素cox进行模型独立预后分析,验证模型优劣性。
25.本发明还包括一种结肠癌预后预测模型,模型采用本发明提供的构建方法构建而成。
26.本发明具有以下有益效果:
27.本发明使用lncrna对,目的在于不受数据的影响,仅仅是患者lncrna量之间的比较,避免对于临床其它数据(芯片,pcr)需要进行模型批次矫正。构建了可作为独立预后的结肠癌预测模型,在分子和免疫相关水平实现了结肠癌患者预后的风险分层,可以显著地将高低风险的患者分开,进而可以预测结肠癌的临床结果,具有较高的临床应用价值。
28.附图(6)
29.图1是结肠癌预后预测模型的流程图;
30.图2为差异性分析后选择的lncrna图,a是差异基因热图;b是火山图,其中log2fc>0区域的灰色部分表示上调基因,log2fc<0区域的灰色部分表示下调基因;
31.图3为单因素cox,lasso,多因素cox选择建立模型的lncrna对图,a是单因素cox筛选的预后lncrna对;b是多因素cox筛选的预后lncrna对;c是lasso的λ选择图;d是lncrna对的lasso系数图;
32.图4是roc曲线和auc值,a是训练集的预测结果和截止时间;b是1、2、3年内训练集的预测结果;c是验证集预测结果;d是结肠癌患者的预后风险模型及临床相关病理信息的预后预测结果;
33.图5是生存曲线,a是训练集;b是验证集;
34.图6是独立于后分析图,a是单变量cox;b是多变量cox。
具体实施方式
35.下面结合实施例及附图对本发明作进一步详细描述,但本发明的实施方式不限于此。
36.本发明,一种基于lncrna对的结肠癌预后预测模型的构建方法包括以下步骤:
37.1)对结肠癌数据进行预处理:从tcga下载385个病人共437个样本,其中39个正常组织和398个肿瘤组织。将这些样本结合人类基因注释文件进行基因id转换,提取其中的lncrna,再结合人免疫基因进行相关性分析,筛选出免疫相关的lncrna,共1229个。
38.2)使用随机划分方法将病人划分为265个病人的训练集和120个病人的验证集,将训练集病人的正常和肿瘤组织分开,进行差异分析,提取共226个具有差异的lncrna;
39.3)对步骤2中最后得到的差异lncrna两两配对,前一个含量比后一个含量高,值为1,反之值为0。将这些lncrna对先进行单因素cox筛选,得到11个预后相关lncrna对,再进行lasso筛选,得到9个预后相关lncrna对,最后进行多因素cox筛选,得到8个lncrna对,基于这8个lncrna对建立cox风险回归模型。
40.4)绘制训练集和验证集的roc曲线,对于训练集,另外计算最优风险得分、连续三年的roc曲线和各临床特征的roc曲线。基于最优风险得分分类出病人的高低风险预测结果,绘制训练集和验证集的生存曲线。最后,结合临床特征通过单因素cox和多因素cox验证模型的独立预后性。
41.进一步地,上述步骤1中,所述相关性分析使用皮尔逊相关系数,参数设置为0.4<|pccs|<1,p
‑
value<0.001。
42.进一步地,上述步骤2中,所述差异性分析使用威尔科克森符号秩检验,参数设置为fdr<0.05,|log2fc|>1。
43.进一步地,上述步骤3中,所述的单因素cox和多因素cox参数设置为p
‑
value<0.001。
44.进一步地,上述步骤3中,所述模型方程使用cox风险回归模型,具体方程为x
i
是模型中lncrna对的系数,β
i
是模型中lncrna对。
45.进一步地,所述roc曲线横坐标是false positive rate(fpr),纵坐标是true positive rate(tpr),划分高低风险的值(cutoff)使用约登指数(youden index=tpr
‑
fpr);auc为roc曲线下面积,并且绘制连续三年的roc;生存曲线使用kaplan
‑
meier法。
46.以下结合附图对本发明作进一步的描述。
47.按照图1开展建模,首先在tcga数据库中下载结肠癌基因表达数据和对应患者临床文件,共385个病人437个样本,结合从gencode下载的人类基因注释文件进行基因id转换,转换成标准的基因名称,随后分开mrna和lncrna,并提取lncrna,结合immport下载的人免疫基因进行相关性分析,筛选出免疫相关的lncrna,共1229个。
48.然后通过随机分割数据的方法分割出训练集和验证集,比例为265:120,将训练集病人的正常和肿瘤样本分开,使用皮尔逊相关系数进行差异分析,最后得到226个具有差异的免疫相关lncrna,并进行两两配对,如果前一个含量高于后一个则值为1,反之为0。
49.图2展示了差异分析的结果,a是差异热图,b是火山图,其中log2fc>0区域的灰色部分表示上调的lncrna,log2fc<0区域的灰色部分表示下调的lncrna。
50.得到了lncrna对后,进行筛选,依次使用单因素cox、lasso回归和多因素cox进行
筛选,依次得到的结果为11个、9个和8个。其中设置cox回归p值小于0.001,最终8个lncrna对为cdkn2b
‑
as1|al442125.2,linc00525|ac104823.1,ac008735.2|ac021218.1,linc02038|ac007128.1,pik3ip1
‑
as1|ac073283.1,ac073283.1|linc01357,al136115.2|arhgef38
‑
it1和ac104964.1|linc02474,并建立了cox风险回归模型。
51.图3展示了筛选的结果,其中c图为lasso中λ的选择图,使用最小标准和最小标准误差(1
‑
se标准)找到最优的λ,值为0.018,d图展示了11个lncrna对的系数情况。
52.最后进行模型的验证以及独立性分析,分别绘制了训练集roc曲线(1
‑
3年)和验证集roc曲线,通过训练集找到的最优风险评分划分训练集和验证集的高低风险情况,并分别绘制了生存曲线,其中训练集去除了16个没有生存状态和时间的病人,验证集去除了6个病人,训练集p值小于0.001,验证集p值为0.022。最后结合临床特征绘制roc曲线和单变量cox、多变量cox分析独立性,发现模型评分小于0.001,故可作为独立预后的方法。
53.图4、5、6分别展示了上述内容,其中训练集3年auc分别为0.805、0.831和0.863,验证集的auc为0.745,最优的中间风险评分为0.979。通过模型和各个临床特征相比,模型准确率高于临床特征的准确率。以上结果再次证实了基于lncrna对的结肠癌预后预测模型效果良好。
54.以上所述,仅是本发明的较佳实施例,并非对本发明的技术范围作任何限制。本行业的技术人员,在本技术方案的启迪下,可以做出一些变形与修改。凡是依据本发明的技术实质对以上的实施例所作的任何修改、等同变化与修饰,均仍属于本发明技术方案的范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1