单样本等位基因拷贝数变异检测方法、探针组和试剂盒与流程
1.本发明涉及基因检测领域,特别是涉及等位基因拷贝数变异检测,更具体地说,是涉及同源重组修复基因dna外显子水平以上的拷贝数纯合缺失、杂合缺失、拷贝数扩增和拷贝数扩增杂合性缺失、拷贝数中性杂合性缺失突变检测。
背景技术:
2.近年来,伴随着二代测序技术(ngs,又称高通量测序技术)的发展,测序成本和时间不断下降,二代测序的应用领域逐渐扩展,促进了生命科学领域的飞速发展。测序技术从诞生之初就是为了进行癌症领域方面的研究,二代测序技术在癌症领域的研究也越来越广泛。基于二代测序技术,识别各种癌症相关的生物信号,能够辅助癌症的诊断、治疗和预后。
3.拷贝数变异(copy number vairant,cnv)是指会引起1kb以上dna片段拷贝数变化的结构变异,包括基因扩增(拷贝数增加)和基因缺失(拷贝数减少)。拷贝数变异按照大小可以分为显微水平和亚显微水平,显微水平指显微镜下可见的染色体畸变;亚显微水平指1kb~3mb的dna片段改变。拷贝数变异大部分发生在蛋白编码区域,是肿瘤细胞生长常见的生物学现象,与肿瘤细胞的生长、耐药性和敏感性相关。赫赛汀是最早的靶向精准抗癌药物之一,用于治疗肿瘤中存在erbb2(her2)基因扩增的乳腺癌患者。
4.同源重组修复(homologous recombination repair,hrr)是一种利用相同或者相近的序列为模板,精确地进行dna双链修复的dna修复机制。除了比较有名的brca1和brca2基因外,同源重组修复通路还包含上下游调控的基因,一般认为同源重组修复通路大致包含如下15个基因:brca1、brca2、atm、bard1、brip1、cdk12、chek1、chek2、fancl、palb2、rad51b、rad51c、rad51d、rad54l以及fanca。同源重组缺陷正是由于这些基因的失活而导致的一系列基因组改变。一般存在这些基因的失活的患者,对于parp抑制剂或者dna损伤试剂敏感。截止2021年5月4日,cosmic数据库中,收录的brca1和brca2突变中,乳腺癌患者brca1发生拷贝数缺失的频率为0.13%(1492个测试样本),brca2发生拷贝数缺失频率为0.4%(1492个测试样本)。
5.拷贝数变异的检测方法大致有三种:荧光原位杂交(fluorescence in situ hybridization,fish)、微阵列比较基因组杂交(array comparative genomic hybridization,acgh)和二代测序方法(next
‑
generation sequencing,ngs)。fish利用荧光分子基团与dna片段结合,然后与待测样本进行杂交,最后在荧光显微镜下观察基因序列的改变。微阵列比较基因组杂交技术是一种分子细胞遗传技术,能在基因组范围内高分辨率地检测染色体的拷贝数变化(可以检测5
‑
10kb水平的拷贝数变化)。fish和acgh技术受限于拷贝数变异区域大小和肿瘤样本纯度,识别分辨率和准确性均有待提高。二代测序技术,相比荧光原位杂交和微阵列比较基因组杂交技术,具有以下优势:高覆盖度与高分辨率、拷贝数检测更加精确、断点识别更加准确,并且能够识别新的拷贝数变异。
6.专利号为zl201510744574.4的中国发明专利披露了一种检测基因组拷贝数变异的方法,该方法采用滑动窗口进行拷贝数检测,并通过gc校正,降低gc含量对于覆盖度的影
响,通过确定拷贝数正常的阈值,来确定拷贝数变异的具体区间。但是该方法未利用对照组构建背景库,来排查不同的建库方法、测序过程中引入的系统偏差,因而检测结果可能存在一定的系统偏差,无法准确识别拷贝数缺失突变是纯合还是杂合。
7.申请号为cn202010771902.0的中国发明专利申请披露了一种检测her2基因扩增的试剂盒及方法,该方法采用数字微滴pcr技术检测her2基因拷贝数变异,但该方法的缺点是仅能检测her2基因扩增,无法涵盖多个基因的拷贝数变异检测。
8.申请号为cn202010880479.8的中国发明专利申请披露了一种检测特定区域的等位基因拷贝数的方法,该方法采用机器学习方法利用肿瘤样本和对照样本完成体细胞等位基因拷贝数变异检测,但该方法的缺点是必须要用对照样本完成检测,无法针对单样本进行检测。
技术实现要素:
9.本发明要解决的技术问题之一是提供一组探针,该探针组覆盖的基因组区域包括表1中的15个基因的305个外显子区域。
10.上述探针组优选为包括seq id no:1~214所示序列的探针。
11.本发明要解决的技术问题之二是提供一种包含有上述探针组的试剂盒。
12.本发明要解决的技术问题之三是提供上述探针组和试剂盒的用途。所述探针组和试剂盒可用于等位基因的拷贝数变异检测。所述探针组和试剂盒也可应用于制备等位基因拷贝数变异检测试剂。所述的等位基因优选为同源重组修复基因,特别是优选为与前列腺癌相关的同源重组修复基因。
13.本发明要解决的技术问题之四是提供一种单样本等位基因拷贝数变异检测系统,该系统主要包括:
14.比对数据获取模块,用于权利要求1或2所述探针组捕获的基因组区域的测序以及测序数据与参考基因组的比对去重,获取样本的细胞比对数据;
15.存储模块,用于存储baf潜在snp位点集,背景库,以及背景集样本、阳性样本、阴性样本的测序数据和比对数据;
16.计算模块,用于根据bin参数划分目标区域和非目标区域,计算每个区域的覆盖度、拷贝数比值、覆盖度的log2值、拷贝数比值的log2值,筛选次等位基因频率信息,计算肿瘤总拷贝数和次等位基因拷贝数;
17.注释模块,用于拷贝数变异注释。
18.本发明要解决的技术问题之五是提供一种单样本等位基因拷贝数变异检测系统的构建方法,该方法主要包括baf潜在snp位点筛选步骤和背景集样本筛选步骤;
19.所述baf潜在snp位点筛选步骤包括:对snp筛选样本进行胚系变异检测和硬过滤,筛选出突变频率为0.4~0.6和0.9以上且出现次数在2次以上的snp位点;在snp公共数据集(例如dbsnp)中筛选出上述探针组覆盖的基因组区域对应的snp位点;将筛选出的所有snp位点合并去重,获得baf潜在snp位点;
20.所述背景集样本筛选步骤包括:提取背景集候选样本的dna,扩增,建库,用上述探针组捕获目标基因,测序并将测序数据与参考基因组进行比对去重,获得背景集候选样本的细胞比对数据;计算探针覆盖度并归一化;线性回归聚类,选择样本数目最大的子集作为
背景集样本。
21.进一步的,所述构建方法还可以包括阳性样本集筛选步骤和bin参数、片段化方法、拷贝数比值阈值筛选步骤。
22.所述阳性样本集筛选步骤包括:对潜在阳性样本,分别用不同的软件进行拷贝数变异检测,有2款以上软件检测出拷贝数变异的样本为阳性样本。
23.所述bin参数、片段化方法、拷贝数比值阈值筛选步骤包括:
24.1)按照不同的bin参数划分目标区域和非目标区域,分别计算每个样本目标区域和非目标区域bin的覆盖度和覆盖度的log2值;
25.2)校正背景集样本的覆盖度和覆盖度的log2值,以及目标区域和非目标区域bin的gc含量和重复性,构建背景库;
26.3)利用所述背景库,针对阴性样本和阳性样本的覆盖度数据,进行gc偏好性、重复性和探针偏好性校正,获得目标区域和非目标区域bin的拷贝数比值的log2值,利用默认的片段化方法延长每个bin至拷贝数状态相同的区域,计算每个区域的拷贝数比值,按照默认的拷贝数比值阈值筛选阴性样本和阳性样本的拷贝数缺失变异和拷贝数扩增变异;
27.4)重复上述步骤1)~3),利用阴性样本和阳性样本的拷贝数筛选结果,统计分析二分类评价指标,确定bin参数;
28.5)用不同的片段化方法和阈值,重复上述步骤3),利用阴性样本和阳性样本的拷贝数筛选结果,统计分析二分类评价指标,确定优化后的片段化方法和拷贝数比值阈值。
29.所述bin参数的优选值为目标区域bin参数199bp,非目标区域bin参数500000bp。
30.所述片段化方法优选采用环状二元分割方法。
31.所述拷贝数比值阈值优选为:拷贝数缺失突变判断阈值0.6,拷贝数扩增突变判断阈值1.5。
32.本发明要解决的技术问题之六是提供一种单样本等位基因拷贝数变异检测方法,该方法的步骤包括:
33.1)提取待测样本dna,扩增,建库,用上述探针组捕获目标基因,富集,测序,测序数据比对到参考基因组上,去重,获得待测样本的细胞比对数据;
34.2)划分目标区域和非目标区域,计算每个区域的覆盖度、覆盖度的log2值和拷贝数比值的log2值;
35.3)利用片段化方法计算每个区域的拷贝数比值,获得拷贝数比值变异区域;
36.4)对baf潜在snp位点进行深度检测和变异检测,并筛选得到次等位基因频率信息;
37.5)针对拷贝数状态相同的区域,结合次等位基因频率信息,计算每个区域的肿瘤总拷贝数和次等位基因拷贝数;
38.6)对每个基因进行拷贝数变异区域的外显子注释,获得每个基因的注释拷贝数结果;
39.7)按照检测基因列表和拷贝数比值阈值筛选拷贝数变异区域,并根据肿瘤总拷贝数和次等位基因拷贝数进行纯合缺失、杂合缺失、拷贝数扩增、拷贝数扩增杂合性缺失、拷贝数中性杂合性缺失的注释。
40.上述步骤5),肿瘤总拷贝数tcn、主等位基因拷贝数mcn、次等位基因拷贝数bcn的
计算公式分别为:
41.tcn=ref_copy
×2log2ratio
42.mcn=tcn
×
(|tumor
baf
‑
0.5|+0.5)
43.bcn=tcn
‑
mcn
44.其中,肿瘤次等位基因频率tumor
baf
等于snp频率信息obs
baf
。
45.当肿瘤纯度大于40%时,可以将肿瘤纯度信息纳入肿瘤总拷贝数和次等位基因拷贝数的计算,肿瘤总拷贝数tcn的计算公式和肿瘤次等位基因频率tumor
baf
的校正公式分别为:
[0046][0047][0048]
本发明利用杂交捕获探针组合获取目标基因序列,利用高通量测序平台,进行单样本同源重组修复通路上关键基因的dna外显子水平以上的拷贝数纯合、杂合缺失、拷贝数扩增、拷贝数扩增杂合性缺失、拷贝数中性杂合性缺失等突变的检测。与现有拷贝数变异检测技术相比,本发明的拷贝数变异检测方法,具有以下优点和有益效果:
[0049]
1.本发明采用杂交捕获和ngs测序方式,可一次检测多个基因的外显子水平的拷贝数变异,且检测速度快,效率高,成本低;
[0050]
2.本发明采用线性回归聚类的方法筛选背景集样本,并利用mad方法综合多个样本的探针覆盖度,完成背景库勾选,如此降低了背景噪音,可实现对单样本特定区域等位基因的拷贝数分析;
[0051]
3.本发明利用投票的方式构建阳性样本集,并通过构建训练集,优化了拷贝数变异bin参数、片段化方法和阈值;
[0052]
4.本发明利用特有的人群snp(单核苷酸多态性)位点,可准确识别拷贝数缺失突变为纯合还是杂合。
[0053]
5.本发明对拷贝数变异检测的灵敏度、准确性和特异性高,灵敏度可达94%,特异性可达100%。
附图说明
[0054]
图1是本发明实施例3的单样本等位基因拷贝数变异检测系统构建流程示意图;
[0055]
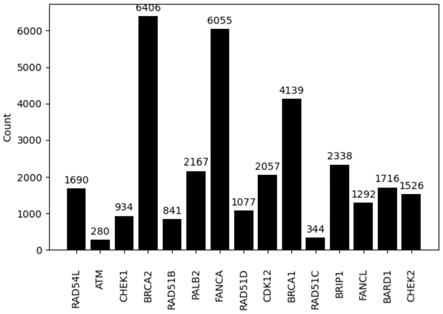
图2是从1000例胚系变异检测样本的胚系突变数据集中筛选出的与表1的外显子区域对应的snp位点的数目分布图;
[0056]
图3是从dbsnp b151版本的公共snp位点数据中筛选出的与表1的外显子区域对应的公共snp位点,在与图2的snp位点合并去重后,获得的snp位点的数目分布图;
[0057]
图4是本发明实施例4的单样本等位基因拷贝数变异检测方法流程示意图。
具体实施方式
[0058]
为对本发明的技术内容、特点与功效有更具体的了解,现结合附图及具体实施例,对本发明的技术方案做进一步详细的说明。
[0059]
实施例1探针制备
[0060]
根据表1中的15个hrr基因的305个外显子区域,设计捕获探针的序列,针对brca1和brca2重点关注基因,采用叠瓦式设计,增加这些区域的覆盖度。然后由美国探针合成公司integrated dna technologies,inc.(idt)按照单条dna探针合成并质控的probes系列产品,合成hrr基因dna序列捕获探针(序列如seq id no:1~214所示),并配套相应的杂交捕获试剂。
[0061]
表1捕获探针组覆盖的基因组区域
[0062]
[0063]
[0064]
[0065]
[0066]
[0067]
[0068]
[0069]
[0070][0071]
实施例2比对数据获取
[0072]
1.dna样本制备
[0073]
组织样本的切片经病理检测后,如肿瘤细胞含量大于20%,则切片可直接用于dna抽提;如果肿瘤细胞含量小于20%,则需要经过富集后再进行dna抽提。
[0074]
ffpe样本先经病理检测确认肿瘤细胞含量在20%以上,然后使用qiagen qiaamp dna ffpe tissue kit进行抽提,将ffpe样本进行脱蜡、蛋白酶k消化、高温逆转交联,最后通过硅胶膜离心柱纯化出dna。
[0075]
白细胞对照样本使用blood
‑
genomic
‑
dna
‑
mini
‑
kit(cw2087m)进行抽提,蛋白酶k消化、裂解缓冲液裂解、无水乙醇沉降,最后通过硅胶膜离心柱纯化出dna。
[0076]
提取的dna使用qubit定量,总量在500ng以上的样本继续实验,并用毛细管电泳确认降解程度。
[0077]
2.dna扩增和建库
[0078]
1)对提取的样本dna进行酶切打断,主峰在200bp左右。
[0079]
2)打断结束后,加入10μl末端修复体系,末端修复条件为65℃30min。
[0080]
3)末端修复结束后,进行连接反应。连接反应体系包括:末端修复产物60μl,无核酸酶水5μl,连接缓冲液30μl,dna连接酶10μl,mgi udi接头5μl。连接反应条件为20℃15min。
[0081]
4)加入0.8x纯化磁珠进行纯化。
[0082]
5)配制表2所示pcr反应体系,涡旋混匀并短暂离心后,进行pcr反应,pcr反应程序如表3所示。
[0083]
表2 pcr反应体系
[0084]
组分体积(μl)kapa hifi hotstart readymix(2x)25mgi双端标签引物5接头连接纯化产物20总体积50
[0085]
表3 pcr反应程序
[0086][0087]
6)使用1x纯化磁珠纯化pcr反应产物。
[0088]
3.文库杂交捕获及目标基因富集
[0089]
1)在0.2ml低吸附离心管中加入表4所示试剂,47℃抽干,抽干后的样品可以继续杂交或者室温放置过夜。
[0090]
表4
[0091]
组分量混合文库500ng/文库cot human dna封闭液5μguniversal blocker通用接头封闭序列2μl
[0092]
2)将表5所示试剂加入到抽干的离心管中,室温放置5~10min,置于pcr仪上95℃30s,65℃杂交孵育4~16小时,热盖温度设为100℃。
[0093]
表5杂交反应试剂
[0094][0095][0096]
3)杂交捕获产物清洗后,进行pcr富集。pcr反应体系如表6所示。pcr反应程序如表7所示。pcr反应产物用1.5x纯化磁珠进行纯化。
[0097]
表6捕获后扩增反应体系
[0098]
组分体积(μl)2
×
kapa hifi hotstart readymix25dual barcode pcr primer mix2.5含有捕获dna的磁珠22.5总体积50
[0099]
表7捕获后扩增反应程序
[0100][0101]
4.文库测序和数据拆分
[0102]
捕获文库定量稀释并混合后,在mgiseq
‑
2000高通量测序仪上进行双端测序,测序结束后根据样本的barcode信息,用splitbarcode v2.0.0软件提取对应的测序数据。提取的原始fastq数据,q30≥85%时,定义为合格下机数据。
[0103]
5.获取比对数据
[0104]
对于质控合格的下机数据,使用fastp去除建库过程中引入的接头序列、引物以及低质量碱基片段。然后使用bwa将质控后数据与hg19人类参考基因组进行比对,使用sambamba对比对后的bam文件进行去重,分别获得待测样本细胞比对数据和正常白细胞比对数据。
[0105]
实施例3单样本等位基因拷贝数变异检测系统构建
[0106]
对166例阴性样本(即正常白细胞样本)、100例背景集候选样本、100例潜在阳性样本,用实施例2的方法,获得细胞比对数据,按照如下步骤构建本发明的单样本等位基因拷贝数变异检测系统(参见图1):
[0107]
1.snp位点筛选
[0108]
针对166例阴性样本、100例背景集候选样本以及其他正常白细胞样本,共计1000例的胚系变异检测样本(即snp筛选样本),利用haplotypecaller进行胚系变异检测,按照haplotypecaller的硬指标:qd≥2、fs≤60、sor≤3、mq≥40、mqranksum≥
‑
12.5和readposranksum≥
‑
8,完成胚系变异硬过滤,并筛选突变频率在0.4~0.6之间以及0.9以上并且在这些样本中的出现次数≥2的snp位点,得到样本的snp筛选位点。统计筛选出的snp位点在15个hrr基因中的分布(见图2)。
[0109]
由于上述snp位点数目相对于部分基因来说较少,因而利用bedtools软件的intersect对dbsnp b151版本的公共数据集进行筛选,获得表1的探针捕获区域对应的公共数据snp位点,与图2的snp筛选位点进行合并去重,获得baf(次等位基因频率)潜在snp位点的集合,其数目分布如图3所示。
[0110]
由图2、3可知,brca2和fanca两个基因对应的snp位点最多,补充之后atm的snp位点数目有了很大提高。
[0111]
2.背景集样本筛选
[0112]
利用100例背景集候选样本的比对数据,利用picard计算每个探针区域的覆盖度。
利用每个样本的平均覆盖度对探针覆盖度进行归一化,即,用探针覆盖度除以样本平均覆盖度,得到探针归一化覆盖度。
[0113]
然后进行线性回归聚类,分别以每个样本为中心,通过探针归一化覆盖度计算该样本与其他样本的线性相关性。具体方法如下:假设y=αx+β,x和y分别对应每个探针的一对样本覆盖度,当截距项β估计值小于0.1、斜率项α估计值在0.94和1.05之间、校正后线性相关性r2大于0.85时,认为两个样本相关,为同类样本。
[0114]
选择样本数目最大的子集作为拷贝数变异检测的背景集样本,共计19例。
[0115]
3.阳性样本集构建
[0116]
针对100例潜在阳性样本,分别利用cnvkit、gatk cnv和facets三款软件进行优化分析,通过投票的方式筛选阳性样本,即,有2款以上软件检测出拷贝数变异的样本即为阳性样本。共计筛选出74例阳性样本,构成阳性样本集。
[0117]
4.bin划分及覆盖度计算
[0118]
探针捕获区域对应的基因组坐标称为目标区域,除探针捕获区域以外的基因组区域称为非目标区域,分别按照不同的bin参数划分目标区域和非目标区域。
[0119]
分别计算每个样本目标区域bin的覆盖度depth和非目标区域bin的覆盖度depth,同时计算覆盖度log2值,即log2=log2depth。
[0120]
5.背景库构建
[0121]
利用步骤4计算得到的每个背景集样本的覆盖度统计文件,用绝对中位差方法(mad)计算目标区域bin和非目标区域bin的校正后的覆盖度depth和覆盖度log2值,以及覆盖度log2值的偏差,同时计算目标区域bin和非目标区域bin的gc含量以及重复性,构建检测拷贝数所需的背景库。
[0122]
6.片段化及拷贝数变异筛选
[0123]
利用步骤5构建的背景库,针对步骤4获得的166例阴性样本和74例阳性样本的覆盖度统计文件,完成gc偏好性、重复性以及探针偏好性校正,获得目标区域bin和非目标区域bin的拷贝数比值log2。利用片段化方法延长每个bin至拷贝数状态相同的区域。计算每个区域的拷贝数比值,即ratio=2log2,这里的log2是拷贝数比值的log2。按照默认的阈值筛选阴性样本和阳性样本的拷贝数缺失和拷贝数扩增变异。
[0124]
7.参数确定
[0125]
针对bin参数,重复步骤4到步骤6,针对片段化方法和拷贝数比值阈值,重复步骤6,利用阴性样本和阳性样本的拷贝数筛选结果,统计分析二分类评价指标完成参数确定。
[0126]
所述bin参数的确定方法如下:
[0127]
利用74例阳性样本集,对比目标区域划分参数266bp、非目标区域划分参数150000bp,以及目标区域划分参数199bp、非目标区域划分参数500000bp,利用背景集评估样本的拷贝数变化,统计灵敏度和阳性预测值,得到前者灵敏度为0.83,后者灵敏度为0.91,因此,确定目标区域划分参数199bp、非目标区域划分参数500000bp为最优。
[0128]
所述片段化方法,通过如下方法确定:
[0129]
对比不同的片段化方法,环状二元分割方法(cbs)、基于小波方法(haar)和3阶隐马尔科夫模型(hmm),利用74例阳性样本集,统计灵敏度和阳性预测值,得到cbs方法的灵敏度是0.90,haar方法的灵敏度是0.78,hmm方法的灵敏度是0.84,因此cbs方法最优,确定片
段化方法采用cbs方法。
[0130]
所述拷贝数比值阈值的确定方法如下:
[0131]
利用74例阳性样本集和166例阴性样本构建训练集,根据灵敏度和特异性确定拷贝数比值阈值,灵敏度和阳性预测值统计见表8,特异性统计见表9,综合表8和表9来看,按照拷贝数比值≤0.6(拷贝数缺失)和拷贝数比值≥1.5(拷贝数扩增)进行筛选最优。因此,拷贝数比值的阈值采用0.6(拷贝数缺失突变判断阈值)和1.5(拷贝数扩增突变判断阈值)。
[0132]
表8 hrr基因的阳性样本集分析结果统计
[0133][0134]
表9 hrr基因的阴性样本集分析结果统计
[0135]
拷贝数比值阴性个数(n)检测真阴性个数(tn)特异性(tnr)0.8|1.2237223330.980.8|1.5237223330.980.8|2.0237223330.980.6|1.22372237210.6|1.52372237210.6|2.02372237210.4|1.22372237210.4|1.52372237210.4|2.0237223721
[0136]
实施例4单样本等位基因拷贝数变异检测
[0137]
提取待测样本dna,用实施例2的方法,获得该待测样本的比对数据,然后基于实施例3构建的单样本等位基因拷贝数变异检测系统,按照以下步骤进行单样本拷贝数变异检测(参见图4)。
[0138]
1.统计目标区域和非目标区域bin覆盖度
[0139]
根据实施例3确定的bin参数(目标区域划分参数199bp、非目标区域划分参数500000bp)划分目标区域和非目标区域,将参考基因组划分为不同的bin区间,利用待测样本的比对数据,用cnvkit v9.8.0软件的coverage统计每个bin区域的覆盖度depth以及覆盖度log2值。
[0140]
2.计算bin拷贝数比值
[0141]
利用实施例3构建的背景库,针对每个bin区域,采用cnvkit的fix方法校正目标区域bin的gc含量、重复性以及探针偏好性,比对背景库,计算得到每个bin区域的拷贝数比值log2。
[0142]
3.获取拷贝数变异区域
[0143]
利用环状二元分割方法,延长每个bin至拷贝数状态相同的区域,计算每个区域的拷贝数比值ratio=2
log2
,获得拷贝数比值变异片段区域。
[0144]
4.snp位点变异检测
[0145]
利用待测样本的比对数据,用bcftools v1.11软件的mpileup进行snp位点的深度检测,获取每个baf潜在snp位点样本的pileup深度数据,然后利用bcftools软件的call进行snp位点的变异检测,利用bcftools软件的view进行snp位点变异的筛选,按照碱基质量≥10,read比对质量≥5,最小read支持数为5,最小覆盖度为5,突变频率为0.4到0.6,筛选得到次等位基因频率(baf)信息。
[0146]
5.肿瘤总拷贝数和次等位基因拷贝数注释
[0147]
针对拷贝数状态相同的区域(即步骤3利用环状二元分割方法分析得到的cns文件),综合步骤4获取的次等位基因频率信息,获得每个区域的肿瘤总拷贝数(total copy number,tcn)和次等位基因拷贝数(b
‑
allele copy number,bcn)。如果通过病理切片镜检确定了肿瘤纯度信息,并且肿瘤纯度大于40%,则将肿瘤纯度信息也纳入肿瘤拷贝数注释程序进行计算。
[0148]
次等位基因拷贝数的具体计算方法如下:
[0149]
获得每个区域的参考拷贝数ref_copy(常染色体拷贝数为2,如果是男性,x染色体是2,y染色体是1;如果是女性,x染色体是2),根据检测到的拷贝数比值log2(log2ratio),按照以下计算公式获得肿瘤总拷贝数tcn:
[0150]
tcn=ref_copy
×2log2ratio
。
[0151]
肿瘤次等位基因频率tumor
baf
默认等于检测到的snp频率信息obs
baf
。
[0152]
如果提供了肿瘤纯度(purity)信息,并且肿瘤纯度大于0.4,则肿瘤总拷贝数tcn计算方法如下:
[0153][0154]
并且对snp频率信息obs
baf
进行校正,校正公式如下:
[0155][0156]
主等位基因拷贝数(mcn)计算公式如下:
[0157]
mcn=tcn
×
(|tumor
baf
‑
0.5|+0.5):
[0158]
mcn的范围为0到tcn。
[0159]
次等位基因拷贝数(bcn)计算公式如下:
[0160]
bcn=tcn
‑
mcn。
[0161]
利用cnvkit软件的scatter综合snp位点变异信息和拷贝数变异信息,可视化展示
拷贝数变异片段,该图形包含每个划分区域的拷贝数比值及次等位基因频率信息散点信息。
[0162]
6.拷贝数变异结果及注释
[0163]
利用ncbi assembly的grch37.p13得到基因的外显子区域,利用bedtools软件的intersect进行拷贝数区域基因注释,完成变异区域的外显子注释。如果cnv变异区域覆盖基因的80%以上的外显子区域,则定义为是整个基因区域的覆盖,注释为整个基因;否则按照外显子水平进行注释,获得每个基因的注释拷贝数结果(包括tcn、bcn、拷贝数比值、baf等信息)。
[0164]
7.筛选拷贝数变异基因并注释其等位基因拷贝数
[0165]
按照检测基因列表和拷贝数比值阈值筛选拷贝数变异区域(拷贝数比值≤0.6为拷贝数缺失突变,拷贝数比值≥1.5为拷贝数扩增突变),根据tcn和bcn的数值,分别注释纯合缺失、杂合缺失、拷贝数扩增和拷贝数扩增杂合性缺失等。
[0166]
注释方法如下:针对常染色体的变异区域,如果tcn和bcn均为0,则表示该突变为纯合缺失(dell);如果tcn为1(常染色体和女性x染色体),并且bcn为0,则认为该突变为杂合缺失(hemizyg);如果tcn大于2,并且bcn≥1,则认为该突变为拷贝数扩增(dup);如果tcn大于2,并且bcn等于0,则认为该突变为拷贝数扩增杂合性缺失(dup
‑
loh);如果tcn为2(常染色体和女性x染色体是2,男性性染色体是1),并且bcn为0,则认为该突变为拷贝数中性杂合性缺失(copy neutral loh)。
[0167]
实施例5 rad51b纯合缺失标准品的等位基因拷贝数变异检测
[0168]
本实施例用rad51b纯合缺失标准品来验证实施例4的等位基因拷贝数变异检测方法的准确性。
[0169]
提取rad51b纯合缺失标准品的dna,利用实施例1的探针,按照实施例4的等位基因拷贝数变异检测方法,对该rad51b纯合缺失标准品的基因拷贝数变异情况进行检测。
[0170]
获得的cns文件如表10所示。
[0171]
表10 rad51b纯合缺失标准品的cns文件结果
[0172][0173]
变异区域筛选及纯合、杂合注释最终结果如表11所示。表中,baf是归一化后的次等位基因频率。表11显示的注释结果与rad51b纯合缺失标准品的实际拷贝数变异情况相符,验证了本发明实施例4的拷贝数变异检测方法的准确性。
[0174]
表11 rad51b纯合缺失标准品的拷贝数变异检测结果
[0175]
拷贝数比值基因范围转录本编号baftcnbcn拷贝数注释纯合杂合注释0.001827rad51bex2
‑
ex10nm_133509.4 00缺失纯合
[0176]
实施例6 brca2杂合缺失模拟样本的拷贝数变异检测
[0177]
利用室内软件模拟brca2杂合缺失,用实施例4的方法,获得brca2杂合缺失模拟样本的拷贝数变异检测结果,参见表12所示,tcn为1,bcn为0,该突变为杂合缺失(hemizyg),
与实际一致,验证了本发明实施例4的拷贝数变异检测方法的准确性。
[0178]
表12 brca2杂合缺失检测结果
[0179]
拷贝数比值基因范围转录本编号baftcnbcn拷贝数注释纯合杂合注释0.498046brca2全基因nm_000059.30.16743510缺失杂合
[0180]
实施例7 fancl和fanca拷贝数变异检测
[0181]
利用实施例1的捕获探针,使用实施例4的拷贝数变异检测方法,对fancl和fanca基因的拷贝数变异进行检测。这两个基因的cns文件结果如表13所示,拷贝数变异检测结果如表14所示。
[0182]
表13 fancl和fanca的cns文件结果
[0183][0184]
表14 fancl和fanca的拷贝数变异检测结果
[0185][0186]
实施例8不同肿瘤纯度下拷贝数变异检测的准确性
[0187]
利用室内软件模拟brca2纯合和杂合缺失,肿瘤纯度从0.1逐渐增加到0.5,参见表15,可以看出在纯度为0.1时,brca2纯合缺失检测结果和真实值较为接近,在肿瘤纯度为0.2时,brca2杂合缺失检测值和真实值较为接近。综合来说,在极低肿瘤纯度10%时,本发明检测的拷贝数比值仍比较精确。
[0188]
表15模拟brca2缺失突变检测结果
[0189][0190]
上述实施例仅为本发明的可行或较佳实施例而已,是用来说明本发明的,并非用以限制本发明申请专利的范围,因此,凡依本发明申请专利范围所作的均等变化与修饰,均应属于本发明专利涵盖的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1