一种通过序列相似性排查测序样本交叉污染的方法与流程
1.本发明属于生信分析领域,具体涉及一种通过序列相似性排查测序样本交叉污染物种的方法。
背景技术:
2.基因组组装是通过读取片段(reads)间的连接关系(overlap)构建出更长的连续性片段(contig)的方法,组装的目标是得到一个没有gap的、单倍体精度的组装结果,但是,即使是研究最完善的人类基因组,仍存在gaps。不过随着测序技术的发展,尤其是三代技术nanopore测序的产生,超长的reads弥补了二代短reads在基因组组装中的局限性,基因组组装又迎来了新的契机。
3.不过三代nanopore测序质量比较低,即使拼接前进行了纠错,组装结果中仍会存在错误。在高通量微生物检测中,面临的一个常见并且严峻的问题就是样本间的交叉污染,在整个样品处理过程中,交叉污染可能由多种原因导致,如:样本dna可能在初始样本处理和置入试管时发生意外转移;由于错误的barcode导致的样本之间串扰;同时测序的多个样本中其中一个样本含有高峰度的强阳物种,造成的其他样本被强阳污染等。这些原因导致的交叉污染,最终都会影响样本病原报出,进一步影响后续的临床治疗。而面对这一问题,目前并没有公认的排查三代样本之间交叉污染的方法。
4.有鉴于此,提出本发明。
技术实现要素:
5.本发明基于项目:基于纳米孔测序的耐药菌感染实时诊断、监测和干预的关键技术研发,项目编号:2018yfe0102100,进行的开发研究。
6.本发明的目的是开发一种全新思路的排查三代测序样本之间交叉污染的方法。
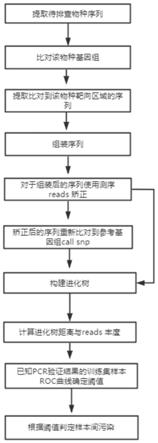
7.为实现上述目的,本发明首先提供一种根据序列相似性,通过比对组装矫正的方法,或者组装矫正后进行单核苷酸多态性snp分析的方法,构建进化树排查三代样本交叉污染。
8.本发明具体提供如下技术方案。
9.本发明首先提供一种基因测序数据交叉污染的判断方法,包括如下步骤:
10.1)进化树构建:对测序序列进行进化树构建;
11.2)样本距离计算:根据进化树结果,计算待排查样本与同run待排查物种检出reads最高的样本之间的待排查物种的进化距离;
12.3)reads丰度计算:对以上每组样本(即待排查样本与同run待排查物种检出reads最高的样本)计算样本之间待排查物种检出reads的比值,得出reads丰度;
13.4)交叉污染判断:对于以上每组样本待排查物种,基于reads丰度和进化距离进行交叉污染判断。
14.进一步的,所述测序数据来自包括但不限于第二代测序数据,或第三代测序数据;
优选的,所述测序数据来自nanopore测序数据;更优选的,所述测序数据来自nanopore宏基因组测序数据。
15.进一步的,所述步骤1)中测序序列包括但不限于组装矫正后的测序序列、分析单核苷酸多态性后的结果序列。
16.进一步的,所述测序序列可通过如下方法制备:
17.a)物种序列提取:提取待分析物种的测序序列,比对到该物种基因组库;
18.b)靶向区域序列提取:待分析样本比对到基因组的靶向区域序列;
19.c)序列组装:对待分析靶向区域序列进行组装;
20.d)序列矫正:对组装后序列进行序列矫正,获得组装矫正后的测序序列;
21.优选的,进一步包括
22.e)单核苷酸多态性分析:将矫正后的序列比对到该物种参考基因上,得到排序后的bam文件,使用bcftools来分析单核苷酸多态性(即call snp),得到分析单核苷酸多态性后的结果序列。
23.更进一步的,所述测序序列制备步骤中:
24.所述步骤a)可采用minimap2进行比对;
25.所述步骤b)可采用canu进行组装;优选的,所述canu组装关键参数为genomesize=5m-nanopore-raw;
26.所述步骤d)可采用medaka进行矫正;
27.所述步骤e)中,分析单核苷酸多态性后过滤质量值小于20的位点。
28.进一步的,上述所述步骤4)中,交叉污染判断具体为:分别确定reads丰度和进化距离的cutoff阈值,基于cutoff阈值进行物种样本间交叉污染判定;
29.优选的,所述cutoff阈值确立为:以物种已知样本作为训练集,构建roc曲线,获得物种判断为交叉污染的进化距离以及reads丰度的cutoff阈值。
30.更优选的,当针对cmv疱疹病毒时,所述reads丰度小于0.003,进化距离小于0.089,为潜在的由于交叉污染导致的假阳检出。
31.进一步的,上述测序数据来自包括但不限于第二代测序数据,或第三代测序数据;优选的,所述测序数据来自nanopore测序数据;更优选的,所述测序数据来自nanopore宏基因组测序数据。
32.本发明还提供一种基因测序数据交叉污染的判断系统,包括如下模块:
33.1)进化树构建模块:用于对测序序列进行进化树构建;
34.2)样本距离计算模块:用于根据进化树结果,计算待排查样本与同run待排查物种检出reads最高的样本之间的待排查物种的进化距离;
35.3)reads丰度计算模块:用于对以上每组样本(即待排查样本与同run待排查物种检出reads最高的样本)计算样本之间待排查物种检出reads的比值,得出reads丰度;
36.4)交叉污染判断模块:用于对于以上每组样本待排查物种,基于reads丰度和进化距离进行交叉污染判断。
37.进一步的,该系统中的不同模块具体执行与上述相同各个细分步骤。
38.本发明还提供一种基于reads丰度和进化距离在测序数据判断交叉污染的应用,所述reads丰度为待排查样本与同run待排查物种检出reads最高的样本检出待排查物种
reads的比值;所述进化距离为待排查物种与同一批次测序即同run待排查物种检出reads最高的样本之间的待排查物种的进化距离。
39.进一步的,所述reads丰度和进化距离是基于对测序矫正后序列构建进化树获得。
40.进一步的,所述交叉污染判断具体为:分别确定reads丰度和进化距离的cutoff阈值,基于cutoff阈值进行样本间物种交叉污染判定;
41.优选的,所述cutoff阈值确立为:以物种已知样本作为训练集,构建roc曲线,获得物种判断为交叉污染的进化距离以及reads丰度的cutoff阈值。
42.进一步的,所述测序数据来自包括但不限于第二代测序数据,或第三代测序数据;优选的,所述测序数据来自nanopore测序数据;更优选的,所述测序数据来自nanopore宏基因组测序数据。
43.本发明还提供一种电子设备,包括:处理器和存储器;所述处理器和存储器相连,其中,所述存储器用于存储计算机程序,所述处理器用于调用所述计算机程序,以执行上述的方法。
44.本发明还提供一种计算机存储介质,所述计算机存储介质存储有计算机程序,所述计算机程序包括程序指令,所述程序指令当被处理器执行时,执行上述的方法。
45.与现有技术相比,本发明至少具有如下优势:
46.1、本发明所述方法能够有效排查由于样本之间交叉污染导致的样本间假阳问题,降低物种报出的不准确性。
47.2、本发明所述方法适用于三代nanopore宏基因组测序,区别于illumina二代测序,可以独特的应用于长读长质量相对较低的nanopore序列。
48.3、本发明所述方法提出了进化距离以及reads丰度作为参考指标。
附图说明
49.为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。
50.图1本发明一个特定的实施例方案;
51.图2本发明一个特定的实施例用来确定污染阈值的训练集数据;
52.图3本发明一个特定的实施例训练集确定污染阈值的roc曲线结果;
具体实施方式
53.下面将结合附图对本发明的技术方案进行清楚、完整地描述,显然,所描述的实施例是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
54.以下术语或定义仅仅是为了帮助理解本发明而提供。这些定义不应被理解为具有小于本领域技术人员所理解的范围。
55.除非在下文中另有定义,本发明具体实施方式中所用的所有技术术语和科学术语的含义意图与本领域技术人员通常所理解的相同。虽然相信以下术语对于本领域技术人员
很好理解,但仍然阐述以下定义以更好地解释本发明。
56.如本发明中所使用,术语“包括”、“包含”、“具有”、“含有”或“涉及”为包含性的(inclusive)或开放式的,且不排除其它未列举的元素或方法步骤。术语“由
…
组成”被认为是术语“包含”的优选实施方案。如果在下文中某一组被定义为包含至少一定数目的实施方案,这也应被理解为揭示了一个优选地仅由这些实施方案组成的组。
57.在提及单数形式名词时使用的不定冠词或定冠词例如“一个”或“一种”,“所述”,包括该名词的复数形式。
58.本发明中的术语“大约”、“大体”表示本领域技术人员能够理解的仍可保证论及特征的技术效果的准确度区间。该术语通常表示偏离指示数值的
±
10%,优选
±
5%。
59.此外,说明书和权利要求书中的术语第一、第二、第三、(a)、(b)、(c)以及诸如此类,是用于区分相似的元素,不是描述顺序或时间次序必须的。应理解,如此应用的术语在适当的环境下可互换,并且本发明描述的实施方案能以不同于本发明描述或举例说明的其它顺序实施。
60.本发明术语“序列组装”,是通过读取片段(reads)间的连接关系(overlap)构建出更长的连续性片段(contig)的方法,组装的终极目标是得到一个没有gap的、单倍体精度的组装结果。目前的组装策略一般分为两种:纠错后组装,以及不纠错直接组装。
61.本发明术语“call snp”,即通过高通量测序以及序列比对的方式检测单核苷酸的多态性。
62.本发明术语“进化树”,可以利用树状分支图形来表示各物种或基因间的亲缘关系。目前常用的构建进化树的软件有phylip,mega等。
63.本发明术语“靶向区域序列”,是指样本序列比对到待排查物种基因组特定区域的序列,在前期测序实验中,特异的使用了引物靶向该基因组这一区域,起到序列富集的作用,该区域测序深度更高。
64.本发明所述“reads丰度”为待排查样本与同run待排查物种检出reads最高的样本检出reads的比值。
65.本发明所述“进化距离”为待排查物种与同一批次测序即同run该待排查物种检出reads最高的样本之间的进化距离。
66.本发明大体提供了一种基因测序数据交叉污染的判断方法,具体包括如下步骤:
67.1)进化树构建;2)样本距离计算;3)reads丰度计算;4)交叉污染判断。
68.进一步的,所述1)进化树构建是对测序序列进行进化树构建;
69.更进一步的,所述步骤1)中测序序列包括但不限于组装矫正后的测序序列、分析单核苷酸多态性后的结果序列。
70.在一些实施方式中,所述测序序列通过如下方法制备:
71.1)物种序列提取:提取待分析物种的测序序列,比对到该物种基因组库;
72.2)靶向区域序列提取:待分析样本比对到基因组的靶向区域序列;
73.3)序列组装:对待分析靶向区域序列进行组装;
74.4)序列矫正:对组装后序列进行序列矫正,获得组装矫正后的测序序列;
75.优选的,当以分析单核苷酸多态性后的结果序列为构建进化树基础时,进一步包括:
76.5)单核苷酸多态性分析:将矫正后的序列比对到该物种参考基因上,得到排序后的bam文件,使用bcftools来分析单核苷酸多态性(即call snp),得到分析单核苷酸多态性后的结果序列。
77.在一些具体实施方式中,
78.所述步骤1)采用minimap2进行比对;
79.所述步骤2)采用canu进行组装;优选的,所述canu组装关键参数为genomesize=5m-nanopore-raw;
80.所述步骤4)采用medaka进行矫正;
81.所述步骤5)分析单核苷酸多态性后过滤质量值小于20的位点。
82.上述具体采用的软件等并不限制本发明。
83.进一步的,所述2)样本距离计算是根据进化树结果,计算待排查样本与同run待排查物种检出reads最高的样本之间的待排查物种的进化距离;所述3)reads丰度计算:是对以上每组样本(即待排查样本与同run待排查物种检出reads最高的样本)计算样本之间待排查物种检出reads的比值,得出reads丰度;
84.进一步的,所述4)交叉污染判断:是对于以上每组样本(即待排查样本与同run待排查物种检出reads最高的样本),基于reads丰度和进化距离进行交叉污染判断。
85.在一些实施方式中,所述步骤4)中,交叉污染判断具体为:分别确定reads丰度和进化距离的cutoff阈值,基于cutoff阈值进行物种样本间交叉污染判定;
86.优选的,所述cutoff阈值确立为:以物种已知样本作为训练集,构建roc曲线,获得物种判断为交叉污染的进化距离以及reads丰度的cutoff阈值。
87.更优选的,当针对cmv疱疹病毒时,所述reads丰度小于0.003,进化距离小于0.089,为潜在的由于交叉污染导致的假阳检出样本。
88.进一步的,本发明所述测序数据来自包括但不限于第二代测序数据,或第三代测序数据;优选的,所述测序数据来自nanopore测序数据;更优选的,所述测序数据来自nanopore宏基因组测序数据。
89.在此,可以理解,本发明所述的测序数据交叉污染的判断方法,除了适用于本发明特定实施例中的纳米孔测序数据外,也可以应用于ngs等二代测序数据,理由是本方法本质上是基于组装矫正的方法或组装校正后分析单核苷酸多态性的方法构建进化树,以进化树距离以及reads丰度进行交叉污染的判断,二代测序数据只需要替换比对以及组装软件即可,因此该方法能够适用于二代测序数据的交叉污染分析。
90.同时,可以理解,本发明实施例虽然只针对cmv疱疹病毒进行例举,但该方法适用范围不限于疱疹病毒,可适用于任意已知参考基因组的可测序的物种;理由是物种的种类对该方法所述步骤,如进化距离计算以及reads丰度计算,以及最终的roc曲线确定阈值,不存在本质上影响。
91.基于上述的发明构思,可以理解本发明还涉及了reads丰度和进化距离在测序数据交叉污染判断中的应用,其特征在于,所述reads丰度为待排查样本与同run待排查物种检出reads最高的样本检出待排查物种的reads的比值;所述进化距离为待排查样本与同一批次测序即同run待排查物种检出reads最高的样本之间的待排查物种的进化距离。
92.在一些实施方式中,所述reads丰度和进化距离是基于对测序矫正后序列构建进
化树获得。
93.下面为具体的实施例。
94.本发明所涉及的软件和技术如下:
95.1).oxford nanopore技术:minion,gridion;
96.2).samtools;
97.3).bcftools;
98.4).bedtools;
99.5).minimap2;
100.6).caun;
101.7).medaka;
102.8).mega
103.9).vcf2phylip。
104.实施例1进化树构建
105.本实例使用上述发明内容介绍的构建进化树的方式,以cmv疱疹病毒为例:
106.1.提取样本中被注释为cmv的序列,minimap2比对疱疹基因组;
107.2.根据minimap2比对结果,提取比对到靶向区域的序列;
108.3.使用canu分别组装每个样本的序列,组装关键参数参加官方以及文献。设定为
109.genomesize=5m-nanopore-raw;
110.4.组装之后的contigs序列,使用medaka再次矫正;
111.5.矫正后得到的一致性序列,minimap2比对到cmv的参考基因组上,排序得到bam文件;
112.6.bcftools mpileup对比对后的序列call snp,过滤质量值小于20的位点;
113.7.vcf2phylip转换vcf文件为phy文件;
114.8.mega根据phy文件检索最佳模型,选取最佳模型,bootstrap=1000,maximumlikelihood算法构建进化树。
115.实施例2训练集确认交叉污染阈值
116.1.根据进化树的结果,计算出该样本与同run该物种检出reads最高样本之间的距离(检出最高样本为其本身则为0);
117.2.对以上每组数据计算样本之间物种检出reads的比值,即丰度。(检出最高样本为其本身则为1);
118.3.对以上每个样本进行pcr验证。上述距离,丰度以及pcr验证结果,如图2。
119.4.上述结果作为训练集,做roc曲线。如图3。
120.5.根据roc曲线获得指标cut-off值:read_ratio(丰度)=0.003,distance(进化距离)=0.089。
121.6.
122.实施例3交叉污染检测
123.1.根据训练集交叉污染阈值已确定,以临床其他检出cmv样本结果为例:
124.sample_iddistancesample1_reads/sample2_reads440120700.000276125
700986501b00099990.152870.019675022332506012103_08x360.097950.3320349672103_07x1201
125.2.对于每个样本,reads丰度小于0.003,进化距离小于0.089,认为是潜在的由于交叉污染导致的假阳检出样本,因此样本4401207推论为由于交叉污染导致的假阳。
126.3.为了进一步验证本方法的准确性,对以上cmv样本进行pcr试剂盒验证。
127.sample_idpcr4401207阴性7009865阳性b0009999阳性2332506阳性2103_08x36阳性2103_07x12阳性
128.可以明显的看出,本发明结合进化树和reads丰度,判断的假阳样本和pcr的验证结果一致,很好的区分出了由于样本交叉污染,导致的假阳检出,证明了本发明技术方案的有效性和准确性。
129.前述对本发明的具体示例性实施方案的描述是为了说明和例证的目的。这些描述并非想将本发明限定为所公开的精确形式,并且很显然,根据上述教导,可以进行很多改变和变化。对示例性实施例进行选择和描述的目的在于解释本发明的特定原理及其实际应用,从而使得本领域的技术人员能够实现并利用本发明的各种不同的示例性实施方案以及各种不同的选择和改变。本发明的范围意在由权利要求书及其等同形式所限定。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1