基于集成学习的分子识别特征功能预测方法
基于集成学习的分子识别特征功能预测方法
1.本发明属于生物信息学技术领域,特别涉及一种分子识别特征的功能预测方法,可用于预测蛋白质序列中分子识别特征的功能,为药物靶点作用位置提供参考。
背景技术:
2.分子识别特征,是指蛋白质中包含10到70个残基之间的固有无序区,在与其伙伴结合之后从无序区转变成有序区的区域。所述伙伴,包括指碳水化合物、离子、脂质、核酸、蛋白质和小分子。分子识别特征的功能包括分子识别组装器(molecular recognition assembler)、分子识别清除剂(molecular recognition scavenger)、分子识别效应器(molecular recognition effector)、分子识别显示位点(molecular recognition display sites)和分子识别伴侣(molecular recognition chaperone)。其中分子识别组装器,用于组装复合物和目标活动;分子识别清除剂,用于储存或中和小的配体;分子识别效应器,用于调节其伙伴分子的活性;分子识别显示位点,用于使得翻译后修饰不表达;分子识别伴侣,用于协助核酸折叠。
3.在药物发现领域,传统的药物发现模式是针对蛋白质的结构活性位点区域进行实验,近些年研究人员发现,将药物作用位点设置为分子识别特征,对于某些疾病的治疗有着很好的效果,在对分子识别特征的研究中有研究人员认为,蛋白质的某些功能发挥作用与分子识别特征的功能密切相关,因此,分子识别特征的功能对药物与靶标的作用方式同样有很大的作用,分子识别特征的功能研究有很大的潜在生物学意义。
4.现有对于分子识别特征功能的预测主要集中在生物实验方面,例如通过x射线衍射或者通过质子核磁共振来确定分子识别特征的功能,这些方式对于新的蛋白质进行分子识别特征功能预测不但实验流程复杂,资源耗费大,而且成本高。
技术实现要素:
5.本发明的目的在于提供一种基于集成学习的蛋白质分子识别特征功能预测方法,以解决现有技术实验流程复杂,资源耗费大,成本高的不足,。
6.本发明的技术思路是:将蛋白质分子识别特征的功能预测看成一个多标记学习任务,使用二元关联策略,充分利用现有的数据集,采取不同的机器学习方法构建单个模型,利用集成学习中的权重法,权重值由遗传算法得到,融合单个模型获得最终的预测模型,通过预测模型对蛋白质分子识别特征的功能进行预测。
7.根据上述思路,本发明的技术方案包括如下:
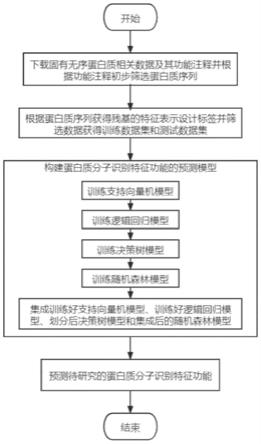
8.(1)下载固有无序蛋白质及其功能注释数据,根据固有无序蛋白质功能注释筛选蛋白质序列,并基于序列相似度将固有无序蛋白质数据划分训练蛋白质序列集h1和测试蛋白质序列集h2;
9.(2)将训练蛋白质序列集h1和测试蛋白质序列集h2中的每个蛋白质序列表示为位置特异性频率矩阵psfm,该矩阵的每一行代表蛋白质序列对应位置残基的特征表示;
10.(3)根据psfm矩阵,得到训练位置特异性频率矩阵集m1和测试位置特异性矩阵集
m2,即由训练蛋白质序列集h1所有蛋白质序列的位置性特异矩阵组成训练位置特异性频率矩阵集m1,由测试蛋白质序列集h2所有蛋白质序列的位置性特异矩阵组成测试位置特异性频率矩阵集m2;
11.(4)将训练位置特异性频率矩阵集m1和测试位置特异性矩阵集m2中的数据分别表示为训练残基数据集和测试残基数据集,再根据功能注释对训练残基数据集和测试残基数据集中每个残基设计标签,并根据每个残基标签对训练残基特征数据集和测试残基数据集进行筛选,得到训练数据集s1和测试数据集s2;
12.(5)构建蛋白质分子识别特征功能的预测模型:
13.5a)选取支持向量机模型,将训练集数据集s1输入到模型中,采用最大化间隔距离法对支持向量机模型进行训练,得到训练好的支持向量机模型;
14.5b)选取逻辑回归模型,将训练集数据集s1输入到模型中,采用反向传播梯度下降法对逻辑回归模型进行训练,得到训练好的逻辑回归模型;
15.5c)选取决策树模型,将训练集数据集s1输入到模型中,采用基尼系数作为划分节点的依据对训练集数据集进行划分,得到划分后的决策树模型;
16.5d)选取随机森林模型,并将训练集数据集s1输入到模型中,采用自助采样法对随机森林模型中的多个决策树进行集成,得到集成后的随机森林模型;
17.5e)使用集成学习策略将训练好的支持向量机模型、训练好的逻辑回归模型和划分后的决策树模型、集成后的随机森林模型进行并联,组成的分子识别特征功能的预测模型;
18.(6)将测试数据集s2的残基特征输入到分子识别特征功能预测模型中,输出五维向量,该五维向量为残基的分子识别特征功能的预测结果。
19.本发明与现有的分子识别特征的功能技术相比,具有以下创新点:
20.1)本发明在构建蛋白质分子识别特征功能的预测模型时,对于每个功能训练一个结果,然后将五个训练结果并联得到训练后的的模型,可以多次使用训练数据集正样本,有助于提高模型预测的准确性;
21.2)本发明在构建蛋白质分子识别特征功能的预测模型时,使用集成学习策略让预测模型融合了四种机器学习模型,使预测模型具有不同模型的优点,提升预测模型准确性;
22.3)本发明使用预测模型来预测蛋白质的分子识别特征功能,相对于现有实验流程简单,实验周期短,有利于对大规模的蛋白质分子识别特征功能进行预测,资源耗费小,成本低。
附图说明
23.图1是本发明的实现流程图;
24.图2是本发明和随机猜想实验在测试数据集s2下的宏准确度及五种分子识别特征功能准确度的比较图。
具体实施方式
25.以下结合附图对本发明的具体实施例和效果作进一步详细描述。
26.参照图1,本实例的实现步骤如下:
27.步骤1,获取固有无序蛋白质相关数据及其功能注释,并根据功能注释初步筛选蛋白质序列。
28.1.1)从公开网站下载disprot数据库中2020_12版本的数据集,其中包括1590条固有无序蛋白质序列和对应的7种功能注释,7种功能注释分别为熵链、生物浓缩、分子识别组装器、分子识别伴侣、分子识别显示位点、分子识别效应器和分子识别清除剂,其中分子识别组装器、分子识别伴侣、分子识别显示位点、分子识别效应器和分子识别清除剂为5种分子识别特征功能;
29.1.2)删除1.1)中不包含分子识别组装器功能、分子识别伴侣功能、分子识别显示位点功能、分子识别效应器功能、分子识别清除剂功能和出现错误表示的蛋白质序列,得到585条蛋白质序列及其功能注释。
30.步骤2,根据蛋白质序列获得残基的特征表示设计标签,并筛选数据获得训练数据集和测试数据集。
31.2.1)基于序列相似度将固有无序蛋白质数据划分训练蛋白质序列集h1和测试蛋白质序列集h2:
32.2.1.1)使用blastclust软件包,对原始的蛋白质序列进行聚类,在长度覆盖率大于70%,序列相似度25%下对收集到的585条固有无序蛋白质进行聚类,共得到508类;
33.2.1.2)将2.1.1)的聚类结果按类分成两份,第一份共有258类,第二份共有250类,将第一份中所有序列作为训练蛋白质序列集h1,将第二份所有序列作为测试蛋白质序列集h2,其中h1中包含蛋白质序列数283,h2中包含蛋白质序列数282;
34.2.2)将训练蛋白质序列集h1和测试蛋白质序列集h2中的每个蛋白质序列表示为位置特异性频率矩阵:
35.2.2.1)将长度为l的蛋白质序列p表示为:p=r1r2…rt
…rl
,其中r
t
代表第t位残基,1≤t≤l;
36.2.2.2)使用psi-blast软件包,对蛋白质序列p与nrdb90数据库进行序列比对,得到蛋白质序列p的位置特异性频率矩阵psfm:
[0037][0038]
其中f
t,j
代表蛋白质序列p中第t个残基出现第j个标准残基的频率,1≤t≤l,1≤j≤20,20代表标准残基的数量,该矩阵的每一行代表蛋白质序列对应位置残基的特征;
[0039]
2.3)将训练蛋白质序列集h1和测试蛋白质序列集h2中的每个蛋白质序列均用2.2.2)方法得到psfm矩阵,获得训练位置特异性频率矩阵集m1和测试位置特异性矩阵集m2;
[0040]
2.4)由训练位置特异性频率矩阵集m1和测试位置特异性矩阵集m2获得训练数据集s1和测试数据集s2:
[0041]
2.4.1)分别将训练位置特异性频率矩阵集m1和测试位置特异性矩阵集m2中psfm矩阵每一行的残基特征提取出来,获得训练残基数据集和测试残基数据集;
[0042]
2.4.2)将训练残基数据集和测试残基数据集中没有固有无序蛋白质功能的残基删除,并将有熵链和生物浓缩两种功能之一,且有5种分子识别特征功能之一的残基删除;
[0043]
2.4.3)将训练残基数据集和测试残基数据集中仅包含熵链和生物浓缩至少一种
功能的残基作为负样本,将该负样本的标签设计为[0,0,0,0,0];
[0044]
2.4.4)将训练残基数据集和测试残基数据集中仅包含5种分子识别特征功能至少一种的残基作为正样本,该正样本的标签使用1个五维向量[y
*1
,y
*2
,y
*3
,y
*4
,y
*5
]表示,其中y
*1
代表该残基是否具有分子识别组装器功能,y
*2
代表该残基是否具有分子识别伴侣功能,y
*3
代表该残基是否具有分子识别显示位点功能,y
*4
代表该残基是否具有分子识别效应器功能,y
*5
代表该残基是否具有分子识别清除剂功能,
[0045]
至此对于训练残基数据集和测试残基数据集中每个残基得到由2.4.1)获得的残基特征和由2.4.3)或2.4.4)获得的标签,将训练残基数据集残基的特征和标签作为训练数据集s1,将测试残基数据集残基的特征和标签作为测试数据集s2;
[0046]
步骤3,选取支持向量机模型,将训练集数据集s1输入到模型中,采用最大化间隔距离法对支持向量机模型进行训练,得到训练好的支持向量机模型。
[0047]
本步骤是对训练数据集s1中的5种分子识别特征功能分别训练,计算支持向量机模型超平面方向参数和超平面位置参数得到5个训练结果,将这5个训练结果并联得到训练后的支持向量机模型,实现如下:
[0048]
3.1)基于残基特征对训练数据集s1中的分子识别组装器功能y1进行训练:
[0049]
3.1.1)构建约束条件为:y
i1
(w
1t
xi+b1)≥1的最大化间隔距离的数学模型:其中w1为预测分子识别组装器功能支持向量机模型的超平面方向参数,b1为预测分子识别组装器功能支持向量机模型的超平面位置参数,xi代表第i个残基的特征,y
i1
第i个残基的分子识别组装器功能,i=1,2,
…
m,m为训练数据集s1的残基个数;
[0050]
3.1.2)利用最小序列优化算法对3.1.1)构建的数学模型进行求解,求得预测分子识别组装器功能支持向量机模型的超平面方向参数w1和超平面位置参数b1,得到分子识别组装器功能的训练结果,即第一个训练结果;
[0051]
3.2)基于残基特征对训练数据集s1中的分子识别伴侣功能y2进行训练:
[0052]
3.2.1)构建约束条件为:y
i2
(w
2t
xi+b2)≥1的最大化间隔距离的数学模型:其中w2为预测分子识别伴侣功能支持向量机模型的超平面方向参数,b2为预测分子识别伴侣功能支持向量机模型的超平面位置参数,xi代表第i个残基的特征,y
i2
第i个残基的分子识别伴侣功能;
[0053]
3.2.2)利用最小序列优化算法对3.2.1)构建的数学模型进行求解,求得预测分子识别伴侣功能支持向量机模型的超平面方向参数w2和超平面位置参数b2,得到分子识别伴侣功能的训练结果,即第二个训练结果;
[0054]
3.3)基于残基特征对训练数据集s1中的分子识别显示位点功能y3进行训练:
[0055]
3.3.1)构建约束条件为:y
i3
(w
3t
xi+b3)≥1的最大化间隔距离的数学模型:其中w3为预测分子识别显示位点功能支持向量机模型的超平面方向参数,b3为预测分子识别显示位点功能支持向量机模型的超平面位置参数,xi代表第i个残基的特征,y
i3
第i个残基的分子识别显示位点功能;
[0056]
3.3.2)利用最小序列优化算法对3.3.1)构建的数学模型进行求解,求得预测分子
识别显示位点功能支持向量机模型的超平面方向参数w3和超平面位置参数b3,得到分子识别显示位点功能的训练结果,即第三个训练结果;
[0057]
3.4)基于残基特征对训练数据集s1中的分子识别效应器功能y4进行训练:
[0058]
3.4.1)构建约束条件为:y
i4
(w
4t
xi+b4)≥1的最大化间隔距离的数学模型:其中w4为预测分子识别效应器功能支持向量机模型的超平面方向参数,b4为预测分子识别效应器功能支持向量机模型的超平面位置参数,xi代表第i个残基的特征,y
i4
第i个残基的分子识别效应器功能;
[0059]
3.4.2)利用最小序列优化算法对3.4.1)构建的数学模型进行求解,求得预测分子识别效应器功能支持向量机模型的超平面方向参数w4和超平面位置参数b4,得到分子识别效应器功能的训练结果,即第四个训练结果;
[0060]
3.5)基于残基特征对训练数据集s1中的分子识别清除剂功能y5进行训练:
[0061]
3.5.1)构建约束条件为:y
i5
(w
5t
xi+b5)≥1的最大化间隔距离的数学模型:其中w5为预测分子识别清除剂功能支持向量机模型的超平面方向参数,b5为预测分子识别清除剂功能支持向量机模型的超平面位置参数,xi代表第i个残基的特征,y
i5
第i个残基的分子识别清除剂功能;
[0062]
3.5.2)利用最小序列优化算法对3.5.1)构建的数学模型进行求解,求得预测分子识别清除剂功能支持向量机模型的超平面方向参数w5和超平面位置参数b5,得到分子识别清除剂功能的训练结果,即第五个训练结果;
[0063]
3.6)将3.1.2)至3.5.2)获得的5个训练结果并联,得到训练好的支持向量机模型。
[0064]
步骤4,选取逻辑回归模型,将训练集数据集s1输入到模型中,采用反向传播梯度下降法对逻辑回归模型进行训练,得到训练好的逻辑回归模型。
[0065]
本步骤是对训练数据集s1中的5种分子识别特征功能分别训练,更新逻辑回归模型参数得到5个训练结果,将这5个训练结果并联得到训练好的逻辑回归模型,实现如下:
[0066]
4.1)基于残基特征对训练数据集s1中的分子识别组装器功能y1进行训练:
[0067]
4.1.1)初始化预测分子识别组装器功能的逻辑回归模型参数θ1;
[0068]
4.1.2)将训练数据集s1的残基特征输入到预测分子识别组装器功能的逻辑回归模型,得到概率值输出y1',计算该概率值输出y1'与分子识别组装器功能y1之间的交叉熵损失loss:
[0069]
loss=-[y
1 ln y1'+(1-y1)ln(1-y1')];
[0070]
4.1.3)使用反向传播梯度下降算法,更新预测分子识别组装器功能的逻辑回归模型参数θ1,直到训练数据集s1的loss不再降低,得到分子识别组装器功能的训练结果;
[0071]
4.2)基于残基特征对训练数据集s1中的分子识别伴侣功能y2进行训练:
[0072]
4.2.1)初始化预测分子识别伴侣功能的逻辑回归模型参数θ2;
[0073]
4.2.2)将训练数据集s1的残基特征输入到预测分子识别伴侣功能的逻辑回归模型,得到概率值输出y2',计算该概率值输出y2'与分子识别伴侣功能y2之间的交叉熵损失loss:
[0074]
loss=-[y
2 ln y2'+(1-y2)ln(1-y2')];
[0075]
4.2.3)使用反向传播梯度下降算法,更新预测分子识别伴侣功能的逻辑回归模型参数θ2,直到训练数据集s1的loss不再降低,得到分子识别伴侣功能的训练结果;
[0076]
4.3)基于残基特征对训练数据集s1中的分子识别显示位点功能y3进行训练:
[0077]
4.3.1)初始化预测分子识别显示位点功能的逻辑回归模型参数θ3;
[0078]
4.3.2)将训练数据集s1的残基特征输入到预测分子识别显示位点功能的逻辑回归模型,得到概率值输出y3',计算该概率值输出y3'与分子识别显示位点功能y3之间的交叉熵损失loss:
[0079]
loss=-[y
3 ln y3'+(1-y3)ln(1-y3')];
[0080]
4.3.3)使用反向传播梯度下降算法,更新预测分子识别显示位点功能的逻辑回归模型参数θ3,直到训练数据集s1的loss不再降低,得到分子识别显示位点功能的训练结果;
[0081]
4.4)基于残基特征对训练数据集s1中的分子识别效应器功能y4进行训练:
[0082]
4.4.1)初始化预测分子识别效应器功能的逻辑回归模型参数θ4;
[0083]
4.4.2)将训练数据集s1的残基特征输入到预测分子识别效应器功能的逻辑回归模型,得到概率值输出y4',计算该概率值输出y4'与分子识别效应器功能y4之间的交叉熵损失loss:
[0084]
loss=-[y
4 ln y4'+(1-y4)ln(1-y4')];
[0085]
4.4.3)使用反向传播梯度下降算法,更新预测分子识别效应器功能的逻辑回归模型参数θ4,直到训练数据集s1的loss不再降低,得到分子识别效应器功能的训练结果;
[0086]
4.5)基于残基特征对训练数据集s1中的分子识别清除剂功能y5进行训练:
[0087]
4.5.1)初始化预测分子识别清除剂功能的逻辑回归模型参数θ5;
[0088]
4.5.2)将训练数据集s1的残基特征输入到预测分子识别清除剂功能的逻辑回归模型,得到概率值输出y5',计算该概率值输出y5'与分子识别清除剂功能y5之间的交叉熵损失loss:
[0089]
loss=-[y
5 ln y5'+(1-y5)ln(1-y5')];
[0090]
4.5.3)使用反向传播梯度下降算法,更新预测分子识别清除剂功能的逻辑回归模型参数θ5,直到训练数据集s1的loss不再降低,得到分子识别清除剂的训练结果;
[0091]
4.6)将4.1.3)至4.5.3)获得的5个训练结果并联,得到训练好的逻辑回归模型。
[0092]
步骤5,选取决策树模型,将训练集数据集s1输入到模型中,采用基尼系数作为划分节点的依据对训练集数据集进行划分,得到划分后的决策树模型。
[0093]
本步骤是对训练数据集s1中的5种分子识别特征功能分别训练,划分决策树模型得到5个训练结果,将这5个训练结果并联得到划分后的决策树模型,实现如下:
[0094]
5.1)基于残基特征对训练数据集s1中的分子识别组装器功能y1进行训练:
[0095]
5.1.1)设计分子识别组装器数据集d1,分子识别组装器数据集d1的特征为训练数据集s1的残基特征,分子识别组装器数据集为d1的类别分为有分子识别组装器功能和没有分子识别组装器功能两类,分子识别组装器数据集d1的属性集为a={a1,a2…aj
…a20
},其中aj代表残基特征的第j项;
[0096]
5.1.2)对分子识别组装器数据集d1进行划分,计算其属性集a中每个属性对于分子识别组装器数据集d1的基尼系数,选择基尼系数最小的属性a
*
作为划分节点,将分子识别组装器数据集d1中a
*
≥0.5的作为第一待划分数据集d
′1,将分子识别组装器数据集d1中a
*
<
0.5的作为第二待划分数据集d1″
,并将a
*
从属性集a中删除;
[0097]
5.1.3)将第一待划分数据集d
′1和第二待划分数据集d1″
分别递归执行5.1.2)操作继续进行划分,直到属性集a为空或待划分数据集中样本均为同一类数据,得到分子识别组装器功能的训练结果;
[0098]
5.2)基于残基特征对训练数据集s1中的分子识别伴侣功能y2进行训练:
[0099]
5.2.1)设计分子识别伴侣数据集d2,分子识别伴侣数据集d2的特征为训练数据集s1的残基特征,分子识别伴侣数据集为d2的类别分为有分子识别伴侣功能和没有分子识别伴侣功能两类;
[0100]
5.2.2)对分子识别伴侣数据集d2进行划分,计算其属性集a中每个属性对于分子识别伴侣数据集d2的基尼系数,选择基尼系数最小的属性a
*
作为划分节点,将分子识别伴侣数据集d2中a
*
≥0.5的作为第一待划分数据集d
′2,将分子识别伴侣数据集d2中a
*
<0.5的作为第二待划分数据集d
″2,并将a
*
从属性集a中删除;
[0101]
5.2.3)将第一待划分数据集d
′2和第二待划分数据集d
″2分别递归执行5.2.2)操作继续进行划分,直到属性集a为空或待划分数据集中样本均为同一类数据,得到分子识别伴侣功能的训练结果;
[0102]
5.3)基于残基特征对训练数据集s1中的分子识别显示位点功能y3进行训练:
[0103]
5.3.1)设计分子识别显示位点数据集d3,分子识别显示位点数据集d3的特征为训练数据集s1的残基特征,分子识别显示位点数据集为d3的类别分为有分子识别显示位点功能和没有分子识别显示位点功能两类;
[0104]
5.3.2)对分子识别显示位点数据集d3进行划分,计算其属性集a中每个属性对于分子识别显示位点数据集d3的基尼系数,选择基尼系数最小的属性a
*
作为划分节点,将分子识别显示位点数据集d3中a
*
≥0.5的作为第一待划分数据集d
′3,将分子识别显示位点数据集d3中a
*
<0.5的作为第二待划分数据集d
″3,并将a
*
从属性集a中删除;
[0105]
5.3.3)将第一待划分数据集d
′3和第二待划分数据集d
″3分别递归执行5.3.2)操作继续进行划分,直到属性集a为空或待划分数据集中样本均为同一类数据,得到分子识别显示位点功能的训练结果;
[0106]
5.4)基于残基特征对训练数据集s1中的分子识别效应器功能y4进行训练:
[0107]
5.4.1)设计分子识别效应器数据集d4,分子识别效应器数据集d4的特征为训练数据集s1的残基特征,分子识别效应器数据集为d4的类别分为有分子识别效应器功能和没有分子识别效应器功能两类;
[0108]
5.4.2)对分子识别效应器数据集d4进行划分,计算其属性集a中每个属性对于分子识别效应器数据集d4的基尼系数,选择基尼系数最小的属性a
*
作为划分节点,将分子识别效应器数据集d4中a
*
≥0.5的作为第一待划分数据集d
′4,将分子识别效应器数据集d4中a
*
<0.5的作为第二待划分数据集d
″4,并将a
*
从属性集a中删除;
[0109]
5.4.3)将第一待划分数据集d
′4和第二待划分数据集d
″4分别递归执行5.4.2)操作继续进行划分,直到属性集a为空或待划分数据集中样本均为同一类数据,得到分子识别效应器功能的训练结果;
[0110]
5.5)基于残基特征对训练数据集s1中的分子识别清除剂功能y5进行训练:
[0111]
5.5.1)设计分子识别清除剂数据集d5,分子识别清除剂数据集d5的特征为训练数
据集s1的残基特征,分子识别清除剂数据集为d5的类别分为有分子识别清除剂功能和没有分子识别清除剂功能两类;
[0112]
5.5.2)对分子识别清除剂数据集d5进行划分,计算其属性集a中每个属性对于分子识别清除剂数据集d5的基尼系数,选择基尼系数最小的属性a
*
作为划分节点,将分子识别清除剂数据集d5中a
*
≥0.5的作为第一待划分数据集d
′5,将分子识别清除剂数据集d5中a
*
<0.5的作为第二待划分数据集d
″5,并将a
*
从属性集a中删除;
[0113]
5.5.3)将第一待划分数据集d
′5和第二待划分数据集d
″5分别递归执行5.5.2)操作继续进行划分,直到属性集a为空或待划分数据集中样本均为同一类数据,得到分子识别清除剂功能的训练结果;
[0114]
5.6)将5.1.3)至5.5.3)获得的5个训练结果并联,得到划分后的决策树模型。
[0115]
步骤6,选取随机森林模型,并将训练集数据集s1输入到模型中,采用自助采样法对随机森林模型中的多个决策树进行集成,得到集成后的随机森林模型。
[0116]
本步骤是对训练数据集s1中的5种分子识别特征功能分别训练,集成随机森林模型中的多个决策树得到5个训练结果,将这5个训练结果并联得到集成后的随机森林模型,实现如下:
[0117]
6.1)基于残基特征对训练数据集s1中的分子识别组装器功能y1进行训练:
[0118]
6.1.1)对训练数据集s1采用自助采样法进行采样10次,得到其中表示第k次采样得到的数据,1≤k≤10,使用每次采样得到的数据分别划分决策树模型,得到10个划分后的分子识别组装器功能基决策树模型;
[0119]
6.1.2)将10个划分后的分子识别组装器功能基决策树模型通过简单投票法融合,得到分子识别组装器功能的训练结果;
[0120]
6.2)基于残基特征对训练数据集s1中的分子识别伴侣功能y2进行训练:
[0121]
6.2.1)对训练数据集s1采用自助采样法进行采样10次,得到其中表示第k次采样得到的数据,使用每次采样得到的数据分别划分决策树模型,得到10个划分后的分子识别伴侣功能基决策树模型;
[0122]
6.2.2)将10个划分后的分子识别伴侣功能基决策树模型通过简单投票法融合,得到分子识别伴侣功能的训练结果;
[0123]
6.3)基于残基特征对训练数据集s1中的分子识别显示位点功能y3进行训练:
[0124]
6.3.1)对训练数据集s1采用自助采样法进行采样10次,得到其中表示第k次采样得到的数据,使用每次采样得到的数据分别划分决策树模型,得到10个划分后的分子识别显示位点功能基决策树模型;
[0125]
6.3.2)将10个划分后的分子识别显示位点功能基决策树模型通过简单投票法融合,得到分子识别显示位点功能的训练结果;
[0126]
6.4)基于残基特征对训练数据集s1中的分子识别效应器功能y4进行训练:
[0127]
6.4.1)对训练数据集s1采用自助采样法进行采样10次,得到
其中表示第k次采样得到的数据,使用每次采样得到的数据分别划分决策树模型,得到10个划分后的分子识别效应器功能基决策树模型;
[0128]
6.4.2)将10个划分后的分子识别效应器功能基决策树模型通过简单投票法融合,得到分子识别效应器功能的训练结果;
[0129]
6.5)基于残基特征对训练数据集s1中的分子识别清除剂功能y5进行训练:
[0130]
6.5.1)对训练数据集s1采用自助采样法进行采样10次,得到其中表示第k次采样得到的数据,使用每次采样得到的数据分别划分决策树模型,得到10个划分后的分子识别效应器功能基决策树模型;
[0131]
6.5.2)将10个划分后的分子识别清除剂功能基决策树模型通过简单投票法融合,得到分子识别清除剂功能的训练结果;
[0132]
6.6)将6.1.2)至6.5.2)获得的5个训练结果并联,得到集成后的随机森林模型。
[0133]
步骤7,对步骤3到步骤6的结果进行并联,即使用集成学习策略构建蛋白质分子识别特征功能的预测模型。
[0134]
7.1)随机初始化训练好的支持向量机、训练好的逻辑回归模型、划分后的决策树模型和集成后的随机森林模型的权重,分别表示为w1、w2、w3、w4,其中w1、w2、w3、w4均在0至1之间,且满足w1+w2+w3+w4=1;
[0135]
7.2)重复7.1)操作50次,每次得到的一组w1、w2、w3、w4个体,将这50个个体集合成种群w0;
[0136]
7.3)将训练数据集s1中所有的残基特征分别输入到训练好的4个模型中,得到4个不同的矩阵:
[0137]
将训练数据集s1中所有的残基特征输入到训练好的支持向量机模型,得到维度为m
×
5的第一矩阵x1,其中x1中每一行代表训练好的支持向量机模型对训练数据集s1对应位置残基的分子识别特征功能预测;
[0138]
将训练数据集s1中所有的残基特征输入到训练好的逻辑回归模型,得到维度为m
×
5的第二矩阵x2,x2中每一行代表训练好的逻辑回归模型对训练数据集s1对应位置残基的分子识别特征功能预测;
[0139]
将训练数据集s1中所有的残基特征输入到划分后的决策树模型,得到维度为m
×
5的第三矩阵x3,x3中每一行代表划分后的决策树模型对训练数据集s1对应位置残基的分子识别特征功能预测;
[0140]
将训练数据集s1中所有的残基特征输入到集成后的随机森林模型,得到维度为m
×
5的第四矩阵x4,x4中每一行代表集成后的随机森林模型对训练数据集s1对应位置残基的分子识别特征功能预测;
[0141]
7.4)根据训练好的支持向量机、训练好的逻辑回归模型、划分后的决策树模型、集成后的随机森林模型的权重w1、w2、w3、w4和7.3)得到的4个矩阵x1、x2、x3、x4,计算待优化的分子识别特征预测结果y
′
:
[0142]y′
=w1x1+w2x2+w3x3+w4x4;
[0143]
7.5)计算预测结果y
′
与残基标签y之间的宏准确度a:
[0144][0145]
其中acc1为分子识别组装器功能准确度,acc2分子识别伴侣功能准确度,acc3分子识别显示位点功能准确度,acc4分子识别效应器功能准确度,acc5分子识别清除剂功能准确度;
[0146]
7.6)将宏准确度a作为评价个体优劣的适应度g,计算种群w0中每个个体的适应度g,淘汰种群w0中适应度的后20个个体,对于剩余个体做突变率为0.001的变异操作,将新的变异个体加入种群w0中,使得种群的个体数为50,得到当前更新后的种群w1;
[0147]
7.7)对当前更新后的种群w1,迭代执行7.6)共799次,得到更新后的种群w
800
,从该种群w
800
中选择适应度最高的个体,记为w1′
、w2′
、w3′
、w4′
,其中w1′
、w2′
、w3′
、w4′
分别代表训练好的支持向量机模型、训练好的逻辑回归模型和划分后的决策树模型、集成后的随机森林模型的权重;
[0148]
7.8)根据7.7)得到的个体w1′
、w2′
、w3′
、w4′
对训练好的支持向量机模型、训练好的逻辑回归模型和划分后的决策树模型、集成后的随机森林模型按权重并联,得到蛋白质分子识别特征功能的预测模型e:
[0149]
e=w1′
*svm+w2′
*lr+w3′
*dt+w4′
*rf,
[0150]
其中svm代表训练好的支持向量机模型、lr代表训练好的逻辑回归模型、dt代表划分后的决策树模型、rf代表集成后的随机森林模型。
[0151]
步骤8,将测试数据集s2的残基特征输入到分子识别特征功能预测模型中,输出五维向量,该五维向量为残基的分子识别特征功能的预测结果。
[0152]
8.1)将测试数据集s2的残基特征输入分子识别特征功能预测模型中,训练好的支持向量机模型、训练好的逻辑回归模型、划分后的决策树模型和集成后的随机森林模型分别会输出1个五维向量,总共得到4个五维向量表示为x1、x2、x3、x4;
[0153]
8.2)将8.1)得到的4个五维向量x1、x2、x3、x4和步骤7得到的个体w1′
、w2′
、w3′
、w4′
通过带权相加,融合成一个五维向量x:
[0154]
x=w1′
*x1+w2′
*x2+w3′
*x3+w4′
*x4,其中*代表叉乘;
[0155]
该五维向量x为残基的分子识别特征功能的预测结果。
[0156]
本发明的效果可以通过以下仿真实验说明:
[0157]
1.仿真条件
[0158]
仿真实验在intel(r)xeon(r)gold 5115 cpu(20核心)、主频2.40ghz,内存48g,tesla p40显卡,red hat 4.8.5-11平台上的python 2.7结合scikit-learn 0.17进行。
[0159]
2.仿真内容:
[0160]
仿真1,在测试数据集s2上,分别采用支持向量机模型、逻辑回归模型、决策树模型和随机森林模型来对分子识别特征功能进行预测,并且计算宏准确度和每个分子识别特征功能的准确度,并将宏准确度作为衡量模型性能的总体指标,将每个分子识别特征功能的准确度作为衡量模型每个功能性能的指标,结果如表1所示。
[0161]
表1 不同机器学习模型在宏准确度和每个分子识别特征功能的准确度对比
[0162][0163][0164]
从表1可见,支持向量机、逻辑回归、决策树和随机森林四个模型在六个指标上各有不同,由此可以看出四个模型的可集成性。
[0165]
仿真2,在测试数据集s2上,将集成学习得到的蛋白质分子识别特征功能的预测模型e与支持向量机模型、逻辑回归模型、决策树模型和随机森林模型四个基模型在宏准确度和每个功能的准确度上对比,对比结果如表2所示。
[0166]
表2 预测模型e与四个基模型在宏准确度和5个分子特征识别功能的准确度对比
[0167][0168]
从表2可见,蛋白质分子识别特征功能的预测模型e可以在总体评价指标宏准确度上有最好的效果,且在分子识别组装器功能准确度、分子识别效应器功能准确度和分子识
别清除剂功能准确度这三个方面有最好的效果,在其余两个功能上与最好的基模型差距不大,由此可以证明本发明采用集成学习的具有预测能力强的效果。
[0169]
仿真3,在测试数据集s2上设计随机猜想实验,设置每个残基对于每个功能的预测为正样本的概率为0.5,对测试集中的所有残基进行预测,并对预测结果计算宏准确度和每个功能的准确度,同时使用蛋白质分子识别特征功能的预测模型e与随机猜想实验进行对比实验,结果如图2所示。
[0170]
从图2可见,蛋白质分子识别特征功能的预测模型e在宏准确度和每个分子识别特征功能上均远优于随机猜想实验,表明本发明对蛋白质分子识别特征功能预测的可靠性更强。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1