基于机器学习预测抗生素抗性表型的方法、设备及应用与流程
1.本发明属于生物信息学领域,具体涉及一种基于机器学习预测抗生素抗性表型的方法。
背景技术:
2.自20世纪40年代以来,抗生素在卫生保健和农业领域的使用显著增加,导致耐药菌株出现的频率大幅增加,抗生素抗性感染目前已经对公共健康构成了全球威胁。目前哪些抗生素对细菌病原体有效的金标准就是通过抗生素药物敏感性测试确定,而这需要在一组抗生素存在下培养微生物,由于培养过程可能很慢,因此往往需要临床医生依赖经验判断抗生素的使用。而当抗生素使用不正确或者不当时就会增加死亡率以及加剧抗生素耐药性的传播。
3.因此开发能够实时确定细菌病原体抗生素抗性表型的诊断方法,对降低患者的发病率和死亡率,以及抗生素耐药性的流行水平起到至关重要的作用。现有技术中,已经有一些研究利用计算机预测测试微生物对抗生素抗性表型的方法。比如cn201780042446.2用于抗微生物剂敏感性预测的流式细胞术数据处理,就是将计算机分为学习阶段和预测阶段从而实现预测测试微生物的敏感性表型,通过将包含易感、中间和抗性表型微生物的一组微生物和抗微生物剂等混合经过流式细胞仪测试并分析的方法获得特征向量,在此基础上利用被测微生物对应的值预测其特征向量,虽然是基于计算机学习的预测却没有特征选择和参数调优的过程,但其还是需要两次培养为前提,另外这种预测并不能进行多组抗性同时预测,效率较低。
4.cn201880057328.3细菌菌株对治疗组合物的敏感性谱的确定方法公开了一种不依赖于细菌培养物生长的用于预测细菌对特定噬菌体株的敏感性的快速筛选方法,其通过使用多种细菌菌株的基因组序列数据来训练机器学习模型,治疗组合物机器学习模型被配置成接收查询细菌基因组并且选择基于受过训练的机器学习模型被估计为对细菌基因组具有敏感性的治疗组合物,能够达到筛选有效药物的目的,但是其机器学习怎么评估敏感性并未明确论述,而且这种选择往往只能针对被学习模型确认的有限治疗组合物,而且缺乏反向验证评估其可靠性存在问题。
技术实现要素:
5.针对上述内容中所记载的技术问题中的一种,本发明提供了一种基于机器学习预测抗生素抗性表型的方法、设备及应用,整合快速发展的基因组测序技术积累的大量菌株的多组学数据和全球多实验室公开的抗生素抗性数据,本发明通过机器学习算法预测细菌的耐药表型,从而在无需药敏试验的情况下利用机器学习方法根据基因组数据来预测细菌的抗生素抗性表型,提示临床个体化用药,并有效遏制因不合理使用抗生素导致新耐药表型的产生和传播。
6.根据本发明实施例的第一个方面,本发明提供了一种基于机器学习预测抗生素抗
性表型的方法,包括:
7.获得基因组数据集和抗生素的抗性表型并将其处理成矩阵格式;
8.将菌株数据集合随机分为训练集和测试集;
9.筛选对抗性表型预测起到重要作用的特征;
10.基于机器学习方法,以重要特征作为输入,以抗性表型作为输出,采用交叉验证的方法来对每种抗生素分别构建模型、评估模型并调整得到最优参数;
11.基于最优参数重新构建训练集模型,并应用于测试集得到最终的性能评估。
12.具体的,本发明提供的方法为:
13.(1)获得目标菌种的全部菌株的基因组数据集,并获得每个菌株对每种抗生素的抗性表型;
14.(2)将菌株的基因组数据集处理为特征矩阵,菌株的抗生素抗性表型处理成表型矩阵;按照特定比例将菌株集合随机分为训练集和测试集;
15.(3)基于训练集对每种抗生素进行特征选择,筛选对抗性表型预测起到重要作用的特征;
16.(4)基于机器学习方法,以重要特征作为输入,以抗性表型作为输出,采用交叉验证的方法来对每种抗生素分别构建模型、评估模型并调整得到最优参数;
17.(5)基于最优参数重新构建训练集模型,并应用于测试集得到最终的性能评估。
18.更进一步的,本发明所述基因组数据集包括但不限于snp/indel、结构变异、dna甲基化、基因组序列、基因表达中的一组或者多组。
19.所述snp/indel可通过二代测序数据与参考基因组比对获得;所述结构变异可通过三代测序数据与参考基因组比对获得;所述dna甲基化可通过nanopore测序的电信号获得;所述基因组序列可通过测序数据组装或者公共数据库(如genbank、ncbi)下载获得;所述基因表达可通过转录组测序获得。
20.所述抗生素抗性表型通过药敏实验测定,或者通过patric数据库或者从相应的文献中下载。
21.所述抗生素的抗性表型包含mic值和sir分类两种类型;所述mic值为抑制培养基内细菌生长的最低的抗菌药物浓度,抗菌药物的浓度通常通过倍比稀释(log2);所述sir分类中s为敏感、i为中度、r为抗性,通常抗生素抗性表型以mic值发表,通过clsi或者eucast指导标准转化为sir分类。
22.进一步的,本发明特征矩阵包含但不限于snp/indel矩阵、结构变异矩阵、dna甲基化矩阵、基因组kmer矩阵、基因表达矩阵中的一种或者多种。
23.所述基因组kmer矩阵是利用kmc软件将每个基因组序列拆分成非冗余的k-bp长度字符串的集合,所述kmer选择10bp或者15bp,其中10bp的kmer相比于15bp的kmer是更冗余的,而计算内存却更小;所述kmer矩阵以kmer长度的字符串作为行,以所述目标菌种的菌株作为列,以kmer的计数或者kmer是否存在(即0:不存在;1:存在)作为值。
24.优选地,本发明选择15bp的kmer作为非冗余字符串,选择kmer计数作为kmer矩阵的值。
25.本发明中,表型矩阵以所述抗生素作为行,以所述目标菌种的菌株作为列,对于sir分类表型,定义s、i、r分别为-1、0、1或者0、1、2,作为抗生素的表型值;对于mic值,则采
用log2(mic)值作为表型值;
26.所述sir分类如果只存在两个类别如sr或ir或si则可定义为0、1。
27.本发明中所述特定比例是训练集样品数量大于等于测试集样品数量的比例。
28.优选地,本发明中选择训练集样品数量比测试集样品数量为2:1;所述训练集的抗生素表型比例应与所述测试集菌株相同或相近。
29.进一步的,本发明所述特征选择采用xgboost或者随机森林方法进行特征选择,或者本发明选择取全部特征作为模型输入特征。
30.本发明所述特征选择结合杂交验证来进行。
31.优选地,本发明采用随机森林进行特征选择。
32.进一步的,本发明所述机器学习方法包含adaboost,bagging,xgboost,随机森林,随机树,支持向量机,线性回归中的一种或者多种。
33.本发明所述预测抗生素抗性是一个回归或者多分类的问题,如果抗性表型是mic值则属于回归模型,sir分类则属于分类模型。
34.本发明中所述交叉验证将所有训练集样品进一步随机分成若干个互斥的集合,每个集合具有相同的抗生素和表型的组合;其中1个集合用于验证,其余集合用于训练;训练集合被用来训练模型,验证集合被用来防止模型过度拟合进行参数调整;所述交叉验证选择3、5或10倍交叉验证,即将样品随机拆分为3份、5份或者10份。
35.优选地,交叉验证选择采用10倍交叉验证。
36.本发明所述调整最优参数时,不同机器学习方法需要调整的参数不同,如所述xgboost可调整maximum tree depth、column subsampling、row subsampling以及learning rate等,所述随机森林可调整max_features、max_depth、n_estimators、criterion等。
37.优选地,本发明选择xgboost方法构建模型。
38.进一步的,本发明所述模型性能评估包括的指标有acc、mcc、vme、me、f1、灵敏度、特异度、auc中的一个或者多个;
39.所述acc是准确性,即正确预测的样品数除以总样品数;
40.所述mcc是马修斯相关系数,当两类别的样品含量相差较大时使用,取值范围从-1到1,其中-1代表预测与实际分类完全不一致,0代表预测结果并不比随机预测好,1代表完美预测,mcc计算公式如下:
[0041][0042]
其中,tp、tn、fp、fn分别是真阳性数、真阴性数、假阳性数、假阴性数;
[0043]
所述vme是非常主要错误率,是指抗性的菌株被预测为敏感的比例;
[0044]
所述me是主要错误率,是指敏感的菌株被预测为抗性的比例;
[0045]
所述f1是综合衡量精确率与召回率的指标,其中精确率是真阳性数除以真阳性数和假阳性数之和,召回率是真阳性数除以真阳性数与假阴性数之和;
[0046]
所述灵敏度是真阳性数除以真阳性数和假阴性数之和,与召回率相同;
[0047]
所述特异度是真阴性数除以真阴性数和假阳性数之和;
[0048]
所述auc是受试者工作特征曲线的曲线下面积,取值范围在0.5-1之间越接近1则
真实性越高,越接近0.5则真实性越低无应用价值;
[0049]
所述回归模型的acc和mcc计算,可以用
±
1两倍稀释法进行计算,即预测的log2(mic)加1或者减1与真实情况相同则认为预测结果正确。
[0050]
优选地,本发明选择acc或者mcc作为评估指标。
[0051]
进一步的,本发明所述方法还包括随机抽样过程;所述随机抽样过程为:当所述目标菌种的目标菌株的某种抗生素的mic值或者sir分类样品偏倚严重时,随机从较多数量的分类中选择与较少数量分类相同的样品数,重复这个过程100次或者1000次。例如s样品有10个,而r样品有1000个,则随机选择r样品中的10个样品,进行模型构建,重复这个过程100次。
[0052]
本发明所述方法还包括重要特征功能的分析;
[0053]
所述重要特征根据xgboost或者随机森林特征重要性值进行挑选;
[0054]
所述特征功能分析包含该特征snp/indel、结构变异、dna甲基化位点、kmer中一种或者多种所在的基因及基因的注释和富集分析;
[0055]
所述kmer的特征功能分析还包含不同分类中kmer上显著的snp分析。
[0056]
第二方面,本发明采用上面任一技术方案所述的方法构建抗生素抗性表型预测模型。
[0057]
第三方面,本发明采用上面任一技术方案所述的方法构建抗生素抗性表型预测模型获取的模型预测中的重要特征。
[0058]
第四方面,本发明提供了一种基于机器学习预测抗生素抗性表型所述的方法在评估待测菌株的抗生素抗性表型方面的应用。
[0059]
第五方面,本发明提供了一种基于机器学习预测抗生素抗性表型所述的方法在评估制备用于评估待测菌株的抗生素抗性表型方面的应用。
[0060]
第六方面,本发明还提供了一种基于机器学习预测抗生素抗性表型的系统,包括:
[0061]
数据存储模块,用于获得基因组数据集和抗生素的抗性表型并将其处理成矩阵格式;
[0062]
数据分析模块,用于将菌株数据集合随机分为训练集和测试集;并筛选对抗性表型预测起到重要作用的特征;
[0063]
数据处理模块,用于进行机器学习和交叉验证,通过以重要特征作为输入,以抗性表型作为输出,采用交叉验证的方法来对每种抗生素分别构建模型、评估模型并调整得到最优参数;
[0064]
结果反馈模块,用于利用训练集构建抗生素抗性预测模型和/或利用测试集评估抗生素抗性模型效能。
[0065]
第七方面,本发明还提供了一种电子设备,包括:
[0066]
至少一个处理器;以及
[0067]
与所述至少一个处理器通信连接的存储器;其中
[0068]
所述存储器存储有可被所述至少一个处理器执行的计算机指令,所述计算机指令被所述至少一个处理器执行,以使所述至少一个处理器执行本发明上述的方法。
[0069]
第八方面,本发明还提供了一种计算机可读存储介质,所述计算机可读存储介质中存储有多条计算机指令,所述多条计算机指令用于使计算机执行本发明上述的方法。
[0070]
通过实施本发明的技术方案,可以达到以下有益效果:
[0071]
本发明提供的一种基于机器学习预测抗生素抗性表型的方法,可以在不需要药敏实验的情况下基于既往数据所构建的模型对抗生素抗性表型进行预测,涉及的数据基数大,抗生素类别多,预测准确率高,具有重要的应用价值。
附图说明
[0072]
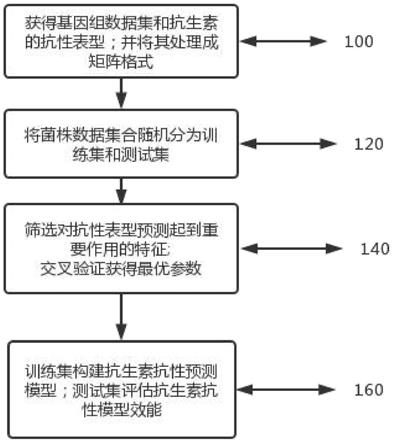
图1为本发明中基于机器学习预测抗生素抗性表型的技术路线图。
[0073]
图2为实施例1中不同菌株的抗生素表型分布图。
[0074]
图3为实施例1中抗生素预测模型的roc曲线图。
具体实施方式
[0075]
下面将结合说明书附图对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
[0076]
下述实施例中的实验方法,如无特殊说明,均为常规方法,按照本领域内的文献所描述的技术或条件或者按照产品说明书进行。下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
[0077]
如图1所示,本发明提供了一种基于机器学习预测抗生素抗性表型的方法,包括:
[0078]
s100,获得基因组数据集和抗生素的抗性表型并将其处理成矩阵格式;
[0079]
s120,将菌株数据集合随机分为训练集和测试集;
[0080]
s140,筛选对抗性表型预测起到重要作用的特征;采用交叉验证的方法得到最优参数;
[0081]
s160,基于最优参数重新构建训练集模型,并应用于测试集得到最终的性能评估。
[0082]
本发明还提供基于上述方法的应用、系统、电子设备和计算机可读存储介质。
[0083]
实施例1利用xgboost算法基于基因组序列特征预测鲍曼不动杆菌的抗生素抗性表型
[0084]
本实施例中,对74个鲍曼不动杆菌的菌株进行nanopore测序,并对这些菌株在8种抗生素中进行药敏实验。基于nanopore测序得到的数据进行质控进而对每个菌株进行组装,对组装得到的基因组序列文件采用kmc软件进行10bp的kmer拆分,获得全部菌株的kmer矩阵;并采用8种抗生素的sir分类作为表型特征,如图2所示。
[0085]
本实施例基因组数据集为基因组序列,基因组序列通过测序数据组装获得。
[0086]
本实施例抗生素抗性表型通过药敏实验测定。
[0087]
本实施例抗性表型为sir分类,sir分类中s为敏感、i为中度、r为抗性,通常抗生素抗性表型以mic值发表,通过clsi或者eucast指导标准转化为sir分类。
[0088]
本实施例特征矩阵是基因组kmer矩阵,基因组kmer矩阵是利用kmc软件将每个基因组序列拆分成非冗余的k-bp长度字符串的集合,kmer选择10bp,其中10bp的kmer相比于15bp的kmer是更冗余的,而计算内存却更小;所述kmer矩阵以kmer长度的字符串作为行,以所述目标菌种的菌株作为列,以kmer的计数作为值。
[0089]
本实施例表型矩阵以抗生素作为行,以目标菌种的菌株作为列,对于sir分类表型,定义s、i、r分别为-1、0、1,作为抗生素的表型值,sir分类如果只存在两个类别如sr或ir或si则可定义为0、1。
[0090]
首先将74个菌株在每种抗生素下的表型按照7:3的比例进行随机拆分成训练集和测试集。其次,采用随机森林算法在训练集中筛选对抗生素表型具有重要区分效能的kmer,每种抗生素选择500个kmer。
[0091]
本实施例特定比例是训练集样品数量大于等于测试集样品数量的比例,选择训练集样品数量比测试集样品数量为7:3;训练集的抗生素表型比例应与测试集菌株相同或相近。
[0092]
本实施例特征选择采用随机森林方法进行特征选择。
[0093]
最后,基于这500个kmer,构建xgboost分类模型(xgboost.xgbclassifier),利用5倍交叉验证的方法确定learning_rate、min_child_weight这两个参数的最优值,其他参数采用默认值。其中learning_rate代表学习率,候选值从0.1到1以0.1为梯度变化,较小的learning_rate意味着更多弱分学习器;min_child_weight代表叶子上最小的样品数,候选值为1,3,5,7。
[0094]
本实施例机器学习方法是adaboost。
[0095]
本实施例预测抗生素抗性是一个多分类的问题。
[0096]
本实施例交叉验证将所有训练集样品进一步随机分成若干个互斥的集合,每个集合具有相同的抗生素和表型的组合;其中1个集合用于验证,其余集合用于训练;训练集合被用来训练模型,验证集合被用来防止模型过度拟合进行参数调整;交叉验证选择5倍交叉验证,即将样品随机拆分为5份。
[0097]
本实施例交叉验证选择采用5倍交叉验证。
[0098]
调整最优参数时,不同机器学习方法需要调整的参数不同,本实施例xgboost调整min_child_weight以及learning rate。
[0099]
每种抗生素训练集的最优参数,如下表1:
[0100]
表1
[0101]
antibioticlearning_ratemin_child_weightdrug30.11drug70.13drug60.11drug40.23drug50.11drug10.13drug80.63drug20.33
[0102]
利用最优参数的最优模型于测试集,测试集的准确可达78%-96%,模型的效能如下表2:
[0103]
表2
[0104]
antibiotictrain_acctest_acctrain_mcctest_mcc
drug70.940.910.8828978120.818181818drug210.9510.885614886drug80.980.780.9645236670.59426092drug410.9610.887151079drug50.980.950.9613049170.91146543drug610.8310.724139246drug310.9610.887151079drug110.8210.674074074
[0105]
其中,drug7的受试者工作特征曲线(roc曲线)的曲线下面积auc为0.91,如图3所示。
[0106]
本实施例模型性能评估包括的指标有acc、mcc和auc;
[0107]
acc是准确性,即正确预测的样品数除以总样品数;
[0108]
mcc是马修斯相关系数,当两类别的样品含量相差较大时使用,取值范围从-1到1,其中-1代表预测与实际分类完全不一致,0代表预测结果并不比随机预测好,1代表完美预测,mcc计算公式如下:
[0109][0110]
其中,tp、tn、fp、fn分别是真阳性数、真阴性数、假阳性数、假阴性数;
[0111]
auc是受试者工作特征曲线的曲线下面积,取值范围在0.5-1之间越接近1则真实性越高,越接近0.5则真实性越低无应用价值;
[0112]
由此可见,基于机器学习方法可以利用基因组序列的特征来预测鲍曼不动杆菌对于抗生素的抗性表型,无需药敏实验可直接得到抗生素的抗性表型,测试集准确率高达96%,具有重要的应用价值。
[0113]
实施例2:
[0114]
本实施例基因组数据集分别采用snp/indel、结构变异、dna甲基化、基因表达中的数据集。
[0115]
snp/indel通过二代测序数据与参考基因组比对获得;结构变异通过三代测序数据与参考基因组比对获得;dna甲基化通过nanopore测序的电信号获得;基因表达通过转录组测序获得。
[0116]
抗生素的抗性表型为mic值;mic值为抑制培养基内细菌生长的最低的抗菌药物浓度,抗菌药物的浓度通常通过倍比稀释(log2)。
[0117]
和基因组数据集对应的,本实施例特征矩阵分别采用snp/indel矩阵、结构变异矩阵、dna甲基化矩阵、基因表达矩阵。
[0118]
本实施例表型矩阵以所述抗生素作为行,以所述目标菌种的菌株作为列,对于mic值,则采用log2(mic)值作为表型值。
[0119]
本实施例特征选择采用xgboost方法进行特征选择,或者选择取全部特征作为模型输入特征。
[0120]
本实施例特征选择结合杂交验证来进行。
[0121]
本实施例机器学习方法包含adaboost,bagging,随机森林,随机树,支持向量机和
线性回归。
[0122]
本实施例预测抗生素抗性是一个回归或者多分类的问题,如果抗性表型是mic值则属于回归模型。
[0123]
本实施例调整最优参数时,不同机器学习方法需要调整的参数不同,如所述xgboost通常调整maximum tree depth、column subsampling、row subsampling以及learning rate,所述随机森林通常调整max_features、max_depth、n_estimators、criterion。
[0124]
本实施例模型性能评估包括的指标有acc、mcc、vme、me、f1、灵敏度、特异度;
[0125]
vme是非常主要错误率,是指抗性的菌株被预测为敏感的比例;
[0126]
me是主要错误率,是指敏感的菌株被预测为抗性的比例;
[0127]
f1是综合衡量精确率与召回率的指标,其中精确率是真阳性数除以真阳性数和假阳性数之和,召回率是真阳性数除以真阳性数与假阴性数之和;
[0128]
灵敏度是真阳性数除以真阳性数和假阴性数之和,与召回率相同;
[0129]
特异度是真阴性数除以真阴性数和假阳性数之和;
[0130]
回归模型的acc和mcc计算,可以用
±
1两倍稀释法进行计算,即预测的log2(mic)加1或者减1与真实情况相同则认为预测结果正确。
[0131]
实施例3
[0132]
和实施例1不同的是本实施例采用了随机抽样过程;随机抽样过程为:当所述目标菌种的目标菌株的某种抗生素的mic值或者sir分类样品偏倚严重时,随机从较多数量的分类中选择与较少数量分类相同的样品数,重复这个过程100次或者1000次。例如s样品有10个,而r样品有1000个,则随机选择r样品中的10个样品,进行模型构建,重复这个过程100次。
[0133]
本实施例还采用了重要特征功能的分析;
[0134]
重要特征根据xgboost或者随机森林特征重要性值进行挑选;
[0135]
特征功能分析包含该特征snp/indel、结构变异、dna甲基化位点、kmer所在的基因及基因的注释和富集分析;
[0136]
kmer的特征功能分析还包含不同分类中kmer上显著的snp分析。
[0137]
实施例4本发明基于机器学习预测抗生素抗性表型的系统
[0138]
本实施的系统包括:
[0139]
数据存储模块,用于获得基因组数据集和抗生素的抗性表型并将其处理成矩阵格式。
[0140]
数据分析模块,用于将菌株数据集合随机分为训练集和测试集;并筛选对抗性表型预测起到重要作用的特征。
[0141]
数据处理模块,用于进行机器学习和交叉验证,通过以重要特征作为输入,以抗性表型作为输出,采用交叉验证的方法来对每种抗生素分别构建模型、评估模型并调整得到最优参数。
[0142]
结果反馈模块,用于利用训练集构建抗生素抗性预测模型和/或利用测试集评估抗生素抗性模型效能。
[0143]
实施例5本发明提供的一种电子设备
[0144]
本发明电子设备包括:
[0145]
至少一个处理器;以及与所述至少一个处理器通信连接的存储器;其中所述存储器存储有可被所述至少一个处理器执行的计算机指令,所述计算机指令被所述至少一个处理器执行,以使所述至少一个处理器执行本发明实施例1的方法。
[0146]
实施例6本发明提供的一种计算机可读存储介质
[0147]
本发明所述计算机可读存储介质中存储有多条计算机指令,所述多条计算机指令用于使计算机执行本发明实施例1的方法。
[0148]
以上对本发明进行了详述。对于本领域技术人员来说,在不脱离本发明的宗旨和范围,以及无需进行不必要的实验情况下,可在等同参数、浓度和条件下,在较宽范围内实施本发明。虽然本发明给出了特殊的实施例,应该理解为,可以对本发明作进一步的改进。总之,按本发明的原理,本技术欲包括任何变更、用途或对本发明的改进,包括脱离了本技术中已公开范围,而用本领域已知的常规技术进行的改变。按以下附带的权利要求的范围,可以进行一些基本特征的应用。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1