一种马尾松高产脂功能SNP标记的筛选方法及其应用与流程
一种马尾松高产脂功能snp标记的筛选方法及其应用
技术领域
1.本发明属于林业技术领域,特别涉及一种马尾松高产脂功能snp标记的筛选方法及其应用。
背景技术:
2.研发目标性状功能标记是植物分子辅助育种的常用方法,也是实现优良个体早期选择的重要手段。现在常用的snp开发技术包括:基于酶切的简化基因组测序(slaf-seq、gbs、rad等)、全基因组重测序(genome re-sequencing)等。这些方法通常需要先收集大量的群体遗传资源,在提取基因组dna后,采用酶切或随机打断的方式对群体全部个体dna进行测序,并通过序列比较的方式开发群体snp分子标记。随后,利用gwas方法开发目标性状关联标记是目前常用的手段。
3.由于snp开发技术要对群体内的每个个体开展测序,通常需要消耗大量的研发费用。而基于酶切的简化基因组测序所开发的snp位点通常位于非编码区,不利于挖掘功能基因。全基因组重测序要根据基因组大小和测序深度计费,在面对如马尾松等具有较大基因组(通常在20gb以上)的物种时所消耗研发费用过高,不具有广泛的适用性。此外,利用gwas方法所得到的关联标记通常需要群体pcr方法验证,并且所得到的关联标记假阳性率较高。
技术实现要素:
4.本发明的首要目的在于克服现有技术的缺点与不足,提供一种马尾松高产脂功能snp标记的筛选方法。
5.本发明的另一目的在于提供所述马尾松高产脂功能snp标记的筛选方法的应用。
6.本发明的目的通过下述技术方案实现:
7.一种马尾松高产脂功能snp标记的筛选方法,包括如下步骤:
8.(1)依据表型选择材料
9.采集m个无性系马尾松的松脂,每个无性系马尾松选择n个植株,然后根据产脂力计算公式分别计算其产脂力,并计算每个植株产脂力的平均值;接着分别以每个无性系马尾松的产脂力最大的植株作为高产脂力植株,以产脂力最小的植株作为低产脂力植株,以产脂力等于或接近产脂力平均值的植株作为中产脂力植株;再分别采集每个无性系马尾松的高、中、低产脂力植株的次生木质部组织材料;其中,m≥3,n≥3;产脂力ryc的计算公式如下:
[0010][0011]
式中:wt表示松脂总重量;d表示每棵树切割树脂的次数;wd表示割面宽度;c表示树皮被切割处的树干周长;
[0012]
(2)基于多组学数据联合分析筛选候选基因
[0013]
对步骤(1)中采集的每个无性系马尾松的高、中、低产脂力植株的组织材料分别进
行转录组和代谢组测序,同时将所有组织材料混合后进行pacbio全长转录组测序;然后利用多组学数据进行差异表达分析,并根据转录组与代谢组联合分析初步筛选得到候选基因;
[0014]
(3)qrt-pcr验证基因表达量
[0015]
分别提取每个无性系马尾松的高、中、低产脂力植株的组织材料的rna,并将其反转录为cdna,然后采用qrt-pcr方法验证步骤(2)中筛选得到的候选基因的表达量,选择差异表达的基因用于snp位点开发;
[0016]
(4)开发snp位点
[0017]
分别提取每个无性系马尾松的高、中、低产脂力植株的组织材料的dna,然后进行pcr全长扩增(基因全长扩增),再直接进行高、中、低产脂力植株的序列相互比较开发snp分子标记,筛选得到snp位点(snp/indel等位变异位点);
[0018]
(5)开发产脂力性状功能snp位点
[0019]
根据步骤(4)中筛选得到的snp位点,对每个无性系马尾松的高、中、低产脂力植株的基因型进行关联分析,得到与产脂力性状显著相关的snp标记,进而筛选得到马尾松高产脂功能snp标记。
[0020]
步骤(1)中所述的无性系马尾松的个数(m的取值)可以根据实际需要进行选择;优选为m≥20;进一步优选为m≥50;再进一步优选为m≥150。
[0021]
步骤(1)中所述的每个无性系马尾松的植株数(n的取值)可以根据实际需要进行选择;优选为n≥5;更优选为:5≤n≤20。
[0022]
步骤(1)中所述的高、中、低产脂力植株可根据实际需要选择生物学重复的次数,优选为3次以上(即各个无性系马尾松各3个以上单株作为生物学重复)。
[0023]
步骤(1)中所述的松脂的采集时间优选为每年的7~9月。
[0024]
步骤(1)中所述的组织材料的采集部位高度与割脂部位相同(每个单株需收集约15克),采集时间以中午12点~1点晴朗天气为宜。
[0025]
步骤(1)中所述的无性系马尾松的树龄10年生以上或胸径达到16cm以上的无性系马尾松;优选为29年生无性系马尾松。
[0026]
步骤(1)中所述的中产脂力植株优选为选择产脂力为中位数对应的植株作为中产脂力植株。
[0027]
步骤(1)中所述的组织材料包括次生木质部、成熟针叶、成熟枝条或未成熟枝条;优选为次生木质部的组织材料。
[0028]
步骤(2)中所述的转录组筛选差异表达基因的条件为:log
2ratio
≥1.0和p-value《0.05。
[0029]
步骤(2)中所述的代谢组筛选差异代谢物的条件为:log
2(fold change)
》0.5和q-value《0.01。
[0030]
步骤(2)中所述的蛋白组筛选差异蛋白的条件为:fold change≥1.2和p《0.5。
[0031]
步骤(2)中所述的联合分析筛选差异基因的条件为不同组学数据的差异倍数相关性r2》0.9。
[0032]
步骤(2)中所述的候选基因可以从转录因子及其他基因中进行选择,优选为选择3个以上的候选基因;进一步优先为选择5个以上的候选基因;更优选为mg7、mg25、mg26、
mg27、mg36基因,其核苷酸序列如seq id no.1~5所示。
[0033]
所述的马尾松高产脂功能snp标记的筛选方法,在步骤(2)之后、步骤(3)之前还包括如下至少一个步骤:
[0034]
a、利用go、kegg等数据库对差异基因进行功能和代谢通路注释,并选择功能基因并使用候选基因fpkm值进行表达分析;
[0035]
b、采集的每个无性系马尾松的高、中、低产脂力植株的其他组织材料,分析候选基因的表达量,即对候选基因进行表达验证;
[0036]
c、根据pacbio全长转录组测序数据库获得候选基因的全长序列,利用ncbi保守结构域搜索软件和pfam保守结构域数据库分析确定差异基因的所属基因家族。
[0037]
步骤(3)中所述的qrt-pcr方法中所用的引物序列优选为如seq id no.19~28所示。
[0038]
步骤(3)中所述的qrt-pcr方法中的扩增反应程序为:变性温度95℃持续10秒,退火温度57℃~62℃持续30秒,延伸温度72℃持续20秒。
[0039]
步骤(4)中所述的pcr全长扩增(基因全长扩增)的反应程序为:预变性温度94℃持续5分钟,变性温度94℃持续1分钟,退火温度55℃~63℃持续30秒,延伸温度72℃持续时间(约1kb/分钟)1~3分钟。
[0040]
步骤(4)中所述的pcr全长扩增所用的引物序列优选为如seq id no.29~38所示。
[0041]
步骤(4)中所述的开发snp分子标记优选为在转录因子基因序列上进行开发。
[0042]
所述的马尾松高产脂功能snp标记的筛选方法,在步骤(4)之后、步骤(5)之前还包括snp位点在群体的多态性验证的步骤,具体如下:在snp标记所在位点的两端特意序列设计引物,然后提取马尾松种质资源的基因组dna,进行pcr扩增与序列比对,验证snp标记与产脂力性状的相关性。
[0043]
所述的马尾松高产脂功能snp标记的筛选方法在筛选马尾松高产脂功能snp标记或马尾松育种方面的应用,该方法获得的马尾松高产脂功能snp标记有助于实现马尾松产脂力性状的早期选择,(如筛选出高产脂马尾松),可用在分子辅助选择育种领域。
[0044]
一种马尾松高产脂功能snp标记,所述的snp标记为如下任一种:
[0045]
(1)位于seq id no.6所示的核苷酸序列,其中自5’端起第76位碱基中的等位基因为t/c;
[0046]
(2)位于seq id no.7所示的核苷酸序列,其中自5’端起第78位碱基和第1073位碱基中的等位基因均为t/c;
[0047]
(3)位于seq id no.8所示的核苷酸序列,其中自5’端起第169位碱基中的等位基因为c/t;
[0048]
(4)位于seq id no.9所示的核苷酸序列,其中自5’端起第128位碱基中的等位基因为t/g;
[0049]
(5)位于seq id no.10所示的核苷酸序列,其中自5’端起第103位碱基中的等位基因为g/t,或第194位碱基中的等位基因a/g;
[0050]
(6)位于seq id no.11所示的核苷酸序列,其中自5’端起第130位碱基中的等位基因为g/t;
[0051]
(7)位于seq id no.12所示的核苷酸序列,其中自5’端起第29位碱基中的等位基
因为c/t;
[0052]
(8)位于seq id no.13所示的核苷酸序列,其中自5’端起第73位碱基中的等位基因为t/a,或第486位碱基中的等位基因为g/c;
[0053]
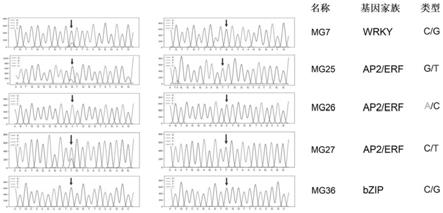
(9)位于seq id no.14所示的核苷酸序列,其中自5’端起第109位碱基中的等位基因为a/g;
[0054]
(10)位于seq id no.15所示的核苷酸序列,其中自5’端起第143位碱基中的等位基因为c/t;
[0055]
(11)位于seq id no.16所示的核苷酸序列,其中自5’端起第125位碱基中的等位基因为g/t,第527位碱基中的等位基因为g/t,第895位碱基中的等位基因为g/c,或第1087位碱基中的等位基因为t/g;
[0056]
(12)位于seq id no.17所示的核苷酸序列,其中自5’端起第138位碱基中的等位基因为a/g;
[0057]
(13)位于seq id no.18所示的核苷酸序列,其中自5’端起第97位碱基中的等位基因为a/c。
[0058]
所述的snp标记中,(2)、(3)、(6)、(7)以及(5)中第194位碱基中的等位基因为a/g的马尾松无性系植株的产脂力相对高于其他snp标记中的马尾松无性系植株。
[0059]
本发明相对于现有技术具有如下的优点及效果:
[0060]
(1)本发明提供了一种开发目标性状功能snp位点的技术,以马尾松高产脂性状为例,研发与目标性状关联的功能标记,实现了多组学测序技术与极端性状表型的有效结合,大幅降低研发成本,且材料选择上只需选择具有最高、中等、最低产脂力的马尾松品系收集组织材料,提取rna后等量混合用来进行pacbio全长转录组测序,同时开展二代转录组、代谢组、蛋白组测序并进行差异表达分析,测序成本较低,该方法适用于不同物种不同性状,具有较高的适用性,尤其在面对较大基因组物种时具有明显优势。
[0061]
(2)本发明简化了功能snp研发技术流程,现有技术常采用:表型测定+全部样本重测序+群体开发snp位点+全基因组关联分析开发snp功能标记的方法,而本发明采用:表型测定+少量样本多组学测序+筛选候选基因+开发snp功能标记的技术流程。
[0062]
(3)本发明针对目标性状差异表型,利用多组学精准定位候选基因,并通过qrt-pcr验证差异表达,接着利用pcr针对特异片段开展群体验证,即先选择差异基因,再进行序列扩增比对,具有较高的snp位点开发效率。
[0063]
(4)本发明对新选择基因开发snp时,仅需选择高、中、低产脂力马尾松各3个无性系就可开发与目标性状相关snp位点,降低snp研发成本,本技术只需针对少数样品进行转录组等多组学测序,成本较低,研发的snp标记有助于实现马尾松产脂力性状早期选择。
[0064]
(5)本发明适用性更广,研发费用更低,现有技术常采用全基因组重测序开发群体snp标记,测序个体数量大,所需研发费用高,并且该技术不适于拥有较大基因组的物种,而大基因组物种常用的简化基因组测序技术不利于挖掘功能基因,而本发明仅需对少量拥有极端性状表型的个体进行多组学测序,直接定位候选功能基因,所需研发费用较低,且适于多种性状和不同大小基因组的物种,尤其具有较大基因组的物种。
seq.scientific reports.2019.(9):13157.doi:10.1038/s41598-019-49737-2.)中公开),各选3个单株采集次生木质部组织材料(高、中、低产脂力马尾松各3个单株,相当于3次生物学重复),材料采集部位高度与割脂部位相同,采集材料时需先将树皮剥去,待露出次生木质部组织时用小刀刮取组织材料,每个单株共需收集约15克,采集时间以中午12点-1点晴朗天气为宜。
[0081]
随后,开展多组学测序,分别对上述采集的3个无性系高、中、低产脂力共9个单株的次生木质部组织材料进行测序,包括:转录组、代谢组、蛋白组测序,每个无性系3个单株作为生物学重复独立测序;同时,将3个无性系共9个单株rna样品等量混合开展pacbio全长转录组测序。本发明委托北京诺禾致源生物科技有限公司进行转录组、代谢组、蛋白组测序。
[0082]
(3)基于多组学数据筛选精准定位候选基因
[0083]
利用多组学数据开展差异表达分析与联合分析筛选候选基因,其中:转录组筛选差异表达基因的条件为log
2ratio
≥1.0和p-value《0.05;代谢组筛选差异代谢物的条件为log
2(fold change)
》0.5和q-value《0.01;蛋白组筛选差异蛋白的条件为fold change≥1.2和p《0.5;联合分析筛选差异基因的条件为不同组学数据的差异倍数相关性r2》0.9。由于结合了转录组、代谢组、蛋白组所定位的候选基因数量大幅降低(图2),只依靠转录组可得到28075个差异表达基因,转录组与蛋白组联合分析则将差异基因数量缩减至2821个,转录组与代谢组联合分析的差异基因数量为226个。
[0084]
利用go、kegg等数据库对差异基因进行功能和代谢通路注释,选择转录因子等功能基因mg7、mg25、mg26、mg27、mg36(利用这5个基因主要是想说明在群体间存在较高的多样性)并使用转录组得到的候选基因fpkm值进行表达分析(图3)。在分析表达量时也从上述3个无性系高、中、低产脂力共9个单株中选用无病虫害的已发育成熟的针叶、未成熟的枝条和成熟的枝条等其他组织用于候选基因的表达验证,mg7、mg25、mg26、mg27、mg36等候选基因在高、中、低产脂力马尾松不同组织差异表达。其中,候选基因序列如下所示:
[0085]
mg7(seq id no.1):
[0086]
atgcagaagattgtggatcaaactgatgcaactataagaaaagcccgagtttcagtccgagctagaaccgagtcacccatgataagtgatggttgccaatggaggaaatatggacaaaagatggccaagggcaatccatgccccagagcttactacagatgcaccatgtcaccgtgctgtccagtgcgcaagcaggtgcaacgcctggccgaagacagatcgattctgataacaacatacgagggcagccataaccacatgcttcctccagcagccacagccatggcatccactacagcagcagctgcctcaatgcttctgtccggttcttccacatcagctgataacatcgcactaaatgcaagcttcatggcaggtgccctcatgcagcatccttgcaacacatcaactgctagcatctcggcctcagctccattccccactatcacactggatctcacacacaatcccaaccaaatggccaacgctccaggtcacatggcagcctcaaacccaagagcactagcaggactcccagctcatgccatgccctttgtaggcatgccacaccaattccccaccaacacaccccagggggcattattccatgggcaatcaatttacaatgctccttccatgtttgcacccttagcaggccagctgcaacgcccccaacaacccatgatgcccacaccaccaaagctcaatattcaagctggccaaccaccaacaccaccacaacaacaaccatcattcatagacacagtcagtgcagctacagctgccataacttctgatcccaatttcacagcagcccttgcagcagccatcacatcccttatgaacaacaacaacaatgctgccaatgccatttctaaacccaactcctctcaggcactcaatcctagtccccacgttgcagctcatgtcaagacagatcacacccaa;
[0087]
mg25(seq id no.2):
[0088]
atggaaaacctccccaatcagcaacctgaccttgaaattgctcaaacacacgaggatcccgggtgccgccgatttaagggaattcgactgcgaaaatggggaaggtgggtatcggaaatccggatgccaaaatctcgagagaaaatatggctgggctcttatacgactcccgagcaggctgcccgtgcttacgacgccgcagtgtattgtctgagagggcccaacgccaaatttaactttccggaatccgtgcacgacattccgtctgtgacttctgtttcccgtcaggaaattcagcacgccgccttcaaatatgccttgggccagccccctccgagtttgcagtctctggaagggcacgccgccctcaaatatgccttgggccagccccctccgagtttgcagtctctggaagaagggcatgccgccctcaaatatgccttgggccagcctcctccgagtttgcagtctctggaagggcacgcgtcgccgtcacagtcgtctacggtttcggaaacggagttatcgggagaacagctgaagatatcggaagagtgccccgacttagcactgtgttggtcgctgtttgcggcagacgacactgggattcccaattcggaaaaagtcccgtcgattgacgaatatttcagtgcgactttgcaggagcagcgggaggagggttacattttcacagatttgtggaatttccaagatcaagatgtt;
[0089]
mg26(seq id no.3):
[0090]
atggagaaatcctcccaacaggaggatgaccatgcccatactccagaagaagaagttcgcgggcaaaagtgccgtcaatttaagggaacccgattgcgaaagtgggggagatgggtagcagaaattcgaatgcccaaatctcgagagaagttatggctggggtcatacaaaaagcccgagcaggccgcccgcgcctacgacgccgcagtgtattgtctgagagggccgaacgccaaattcaatttacccaattctctacctgacattccgtctgcgtcttctctttcccgccggcagattcaactcgctgctgccaaatgtgcgttggatcaattcccttcgagtgcgccccctctgcagaattttaataataaggccatggacgaggccgcatcgccgtcaagactggatccggtatcagaaactgagttgtcgagcgatggtcatcaaatatcagaggaaggggagttggatttgtgggaaagtccgtttgaggtatcaggcggcaattatgaagggcgcatgaacctgaatttagagagaatgccatcgattgaggagttctcggccttggaaattatttacagtatttgtcagcagcatgaggaggaggaacacataaaccttttcctcgaccccacagagttgtggaacttt;
[0091]
mg27(seq id no.4):
[0092]
atggataatggatcctctctcgtgcccatcgccatgcccaattccttgacagacattgaagcaataagcaattctccttttgcggataagggtggaaacaagagaattcgaacgcaagacgaagctgcctcttcgccttcacagcaaagcaggctgaacctgcaaaatccagcatacagaggcgtgcgccgtcgtagctggggcaaatgggtgtctgaaattcgggaaccaaaaaagaaaaaccgaatctggctcggctcctacgatacaccagagatggccgctcgagctcacgatgtcgctgcattctacttgaaaggaaagaaacattcgttgctcaattttccagagctcattgatcaacttccagaaccaatttcttcggctccgccccacattcaagctgccgcggcagcagcagccgtcgctttcaattctgcatcccgtagtgcacagaactctggaatgtcaagcgatatcaataaacaagacagcggaaggccaagaaatactccagtaagcaatgagcacgcaggtatctcctcttcgaatcaactatcggcagtaattagctcagagttaaacttggagacggctaatgtcgagttattagggaaaccgaattatactaattcatcatcaatggagatcgtaacagaggaggacttgttcgaatccaccaacttctatacgaatttggcagaaggccttatgcttcctccacctctgttcagtattcctgaattagacatagaggagcagagattggaggaaggatttctttggtctggtttt;
[0093]
mg36(seq id no.5):
[0094]
atggctggcatggatgacggagacattaattttagcagcaatatagtagatgattttggcaatgggtcctccatggaaagctttttcgaggagattttgagggatactactcatgcctgcactcatacacacacctgcaaccctcccgggccagataacacacatacacacacatgcttccacacgcacacaaaaatccttgctgctcccgatgacgagaagtctgcagacactgctgagtctccacaaaacagctcttccaagccaaaaaaacgaccagtaggtaatcgagaggcagttagaaaatacagggaaaaaaaaaaggcccggacggcctccctggaggagcaggttgttcaactgaccactgttaatcagcaattgcataggagattacagggtcaagcagctttagaggctgagattgcaagattgaagtgtctgctgg
ctgactttaggggccggatcgatggggaattggggtcctatccttaccaaaagtcaattagaatggataaaacttgcaatgatgcaccattccggcaaccgatgcctgggggatatgtcctggatccctgcaatatctggtgcaatgcagatgcagcttgccgtgaaccgactctggcatccaacagtgagggtggtgtgcagcacgaacgtgatagtgctgcacgttggaatggcgattgtggccagatcgcaggccattgtcaaggtttgaagggtgatatggcagtaacctctagtggactttctggatgctcagagggtacagcaacaaaaacagtgcctgctgccatggcttcttctggaaaagagagaaaaggtgcatttggtgtg。
[0095]
除了go和kegg注释外,根据pacbio全长转录组测序数据库获得候选基因的全长序列,利用ncbi保守结构域搜索软件和pfam保守结构域数据库分析确定差异基因的所属基因家族(图4),为后续研究其生物功能提供基础。
[0096]
(4)qrt-pcr验证基因表达量
[0097]
利用trizol法分别提取高、中、低产脂力马尾松不同组织(成熟针叶、成熟枝条、未成熟枝条、次生木质部)的rna,用反转录试剂盒获得cdna,采用qrt-pcr方法验证初筛差异基因的表达量。根据获得的候选基因全长序列并利用primer 3设计引物(表1)。选择actin作为内参基因,扩增反应程序设置如下:变性温度95℃持续10秒,退火温度57℃~62℃持续30秒,延伸温度72℃持续20秒,每个基因每个样品均为三次重复。
[0098]
表1候选基因qrt-pcr引物
[0099][0100]
反应结束后,利用2-δδct
方法对候选基因进行相对表达量分析,以次生木质部等不同组织高产脂力马尾松作为对照,分析mg7、mg25、mg26、mg27、mg36在中、低产脂力马尾松的表达量(图5),选择差异表达的基因用于snp位点开发。
[0101]
(5)开发snp位点
[0102]
对经qrt-pcr验证的差异表达基因mg7、mg25、mg26、mg27、mg36开发snp分子标记(图6)。收集不同无性系马尾松的成熟针叶,提取基因组dna(高、中、低各选一个单株分别提取dna),利用转录组数据获得候选基因的5’端序列、编码序列、3’端序列并进行pcr全长扩增,首先设计扩增引物(表2),pcr反应程序包括:预变性温度94℃持续5分钟,变性温度94℃持续1分钟,退火温度根据引物特性一般在55℃~63℃之间持续30秒,延伸温度72℃持续时间(约1kb/分钟)根据序列长度而定一般在1~3分钟。根据不同序列特征,一些序列需经过多次pcr扩增加拼接的到完整序列。初步分析显示,在wrky、ap2/erf、bzip等转录因子基因序列开发得到snp分子标记,等位基因类型包括c/g、g/t、a/c、c/t等。
[0103]
表2候选基因扩增引物
[0104][0105]
(6)snp位点在群体的多态性验证
[0106]
根据snp标记所在位点两端特意序列设计引物(表2),在150份马尾松种质资源的基因组dna开展pcr扩增与序列比对,分析表明,在mg7、mg25、mg26、mg27、mg36等5个基因的snp位点具有较高的扩增效率与成功率。以上5个基因均含有纯合型与杂合型等位基因(图7)。其中,mg7基因的基因型包括c/g、c/c、g/g;mg25基因的基因型包括g/t、g/g、t/t;mg26基因的基因型包括a/c、a/a、c/c;mg27基因的基因型包括c/t、c/c、t/t;mg36基因的基因型包括g/c、c/c、g/g。各基因的基因型频率分析表明,mg7和mg36两个基因的杂合度较高,其c/g基因型在群体中的占比分别为48%和57%;mg25和mg26两个基因的纯合度较高,其纯合基因型(g/g、t/t)和(a/a、c/c)在群体中的占比和分别为83%和94%。因此,在马尾松重要差异基因开发snp位点及研发重要性状关联标记具有极高的可行性。
[0107]
(7)产脂力性状功能snp位点研发
[0108]
基于多组学测序数据在高、中、低产脂力马尾松共筛选得到1014个含有单核苷酸多态性(snp)/插入缺失(indel)(简称snp/indel)等位变异位点,其中10个为indel变异位点。利用序列信息设计引物并对其开展pcr扩增与序列比对验证,结果在根据差异表达信息和序列比较信息筛选出来的13个基因中共开发得到19个snp位点(表3),其等位变异类型包括c/t、t/g、c/g、a/g、a/c、t/a等。其中,cluster-35640.0的snp位点最多为4个,其次为cluster-19799.0、cluster-24038.0与cluster-32834.3均含有2个snp位点,剩余9个基因仅含1个snp位点。
[0109]
在高、中、低产脂力马尾松各选3个株系,利用pcr与序列比对方法对snp分子标记基因型进行验证(表3)。结果表明,除了cluster-19799.0(c/t)、cluster-31423.0(t/c)两个基因在高产脂马尾松中为杂合基因型,其余基因在高产脂马尾松的基因型均为纯合型;15个snp位点在低产脂马尾松中为杂合基因型。在高、中、低产脂力马尾松的基因型关联分析显示,本技术所开发的snp位点均与产脂力表现出较高的相关性。位于cluster-19799.0(2个)、cluster-24038.0(2个)、cluster-32834.3(2个)、cluster-35640.0(4个)等4个基因的多个变异位点为连锁遗传的snp标记;其中,cluster-24038.0、cluster-29744.0、cluster-32834.3、cluster-35640.0在高产脂马尾松中为纯合基因型,在低产脂马尾松中为杂合基因型。因此,本研究得到的与产脂力性状紧密相关的snp功能标记在分子辅助选择育种领域均具有较高的应用价值。
[0110]
表3与产脂力性状紧密关联的19个功能snp位点
[0111][0112]
cluster-19758.0
[0113]
该snp位点在cluster-19758.0所示核苷酸序列,其中自5’端起第76位碱基中的等位基因为t/c,核苷酸如下所示(seq id no.6):
[0114]
gttttggacatatccagcaatcatgacagtccaggacacaacactcctctcagacattttgtcaaacaattgtct(t/c)gcaatttctatatatccacatctagcatacatgtcaataagagcacccctaacatagatatcggactcaaaaccagttttgattacataatcatgaacttctttgcccttttccagggacttgaagtaagcacatgctgagag;
[0115]
cluster-19799.0:
[0116]
该snp位点在cluster-19799.0所示核苷酸序列,其中自5’端起第78位碱基和第1073位碱基中的等位基因均为t/c,核苷酸如下所示(seq id no.7):
[0117]
gtttctgctctttctaacagtttccaagcttcatcaacattgcatgccttacagaagccaccaataagtgtattaaa(t/c)aaagaagcattaggggggatgcctttctggtacatctcatttaaaagggtatgagcttcatatactctgcctccttcacaaagacagttcattagcagactatagctaaaggcattaggcacacaacccttcttctgcatgtaattgagtaatttataggccttatctattttgcctccaatactaaggcctcgcatcagtaaattgtaggttccagttgtaggagcacactcccttgtttgcatctcacttaagagtttgaaagccattgacaattctccatgcttacaaaagccatctattagagtactataagagatagcatctgggtgaaggcctttctccaacatctgaataaaaaacctctttgcttcatccaatctgccctctttgcagaggccatcaatgactgttgtatatgtgacaacatcaggaaagcagttgtcatgtttaactgaatttaccagccccaaagaagcatcgtcagtctcatagaaatcaccaatagcagcactgccatgaagccatatgcgatccatcattttaatcgctttgtttactttgccatttttacaaaggccttctatcataatattacaagttatcgtatctggagtgagacctttttccctcattttgtgtagaagatgttctgcttctgaaatcctgccctccttgcagacactttggaccaaaatgttgtaagtaaccgtattagcaaaacagcctttgctcaacatttcaccaagcaattgattagctcgttcaacattacctctcttgcaatacccatgaagaagcgtgctgtatgttatgctgtctggaacaactccatttcgaagcatcgcattcaggatcctctctgcatcagatagcataccctccctacacatgccgt
cgactaatacattgtaggagatcacatcgggagtgatgttagctgtcatcatcgcttccaggagcttgcgagcctcagaaattctccctagcttgg(t/c)cagtccacttaaaagaatgttgtaggacaccacattgcaggaatgacaattatccttcatgtgctccataaccttgaaagcttcgtcaagtttgccttc;
[0118]
cluster-20708.0:
[0119]
该snp位点在cluster-20708.0所示核苷酸序列,其中自5’端起第169位碱基中的等位基因为c/t,核苷酸如下所示(seq id no.8):
[0120]
ttaaggcctacataacctccgaagctctcttaagcgttctatgtcgaaattccttttcttcatcagtctgctctgcctggcactgaagcgagtattgaagaagctcttgaatgaaggatttgtggttgtgttaaggaagaatagataccccgccatgaagtatataga(c/t)gcgagaaagtagcatacgggttccatcacatcccatgacagctcccagaatgtcagcctcattaaacccgcagtttgaagcccgaaaaatcccaagcccaaccgtagttcagtcatggcgtcactcttagctttctga;
[0121]
cluster-23480.0:
[0122]
该snp位点在cluster-23480.0所示核苷酸序列,其中自5’端起第128位碱基中的等位基因为t/g,核苷酸如下所示(seq id no.9):
[0123]
tgcccatgtagagcatagcctacaatcatcgcagtccacgaaaccacatctgtcttcggtatcattttaaagacttcacatgcatcacctatggaactacatttagcatacattgtaataagagcag(t/g)ccccacagaggcatcctcctgaaatccatttttgatggtctggcaatgaaactgcttgccttgatcaataattgccaggtccgcacacacg;
[0124]
cluster-24038.0:
[0125]
该snp位点在cluster-24038.0所示核苷酸序列,其中自5’端起第103位和第194位碱基中的等位基因为g/t和a/g,核苷酸如下所示(seq id no.10):
[0126]
atggtgccttctctaattcaaattgattcatcacaggtgatggagtcagaaacagcagcagtaacagggtctctactttttggaacagataaaatgactatg(g/t)gaccaggatctgaaaatgtttgccctgttttttcctctttaacaggcctgcaatcaaaccagatgcttatttctcattccaaccaaatgg(a/g)cttccacaaaagttctgtagtcccaatccatgcagcagaattaagcagagaaatatatgatatggaaagtacccaatacaacaatattcactttt;
[0127]
cluster-29744.0:
[0128]
该snp位点在cluster-29744.0所示核苷酸序列,其中自5’端起第130位碱基中的等位基因为g/t,核苷酸如下所示(seq id no.11):
[0129]
ggttgttccgcaaatccagttggattactaaacccttgccatttttaagcaccctgcttaaattctcgtcatattcttcaagaggggaccattcctctgattcttcttctatttcacactcgtccaata(g/t)ccaaaatcaatctcctatgctcacccctgcgcgcgtgcacaaattaattgctaagcagacagatgcattgttagcaatggaaatttttg;
[0130]
cluster-31423.0:
[0131]
该snp位点在cluster-31423.0所示核苷酸序列,其中自5’端起第29位碱基中的等位基因为c/t,核苷酸如下所示(seq id no.12):
[0132]
ttatatgcgagaccttggattaccattg(c/t)ttggtgatgcacggtaggagtagacgcccttcccactcttccgaccaaggtaccctgcatcaacatattgcacaagcagtgggcatggggaatatttgctttctccaagcccgtgatggagaatcttcataattgagaggcaaacatctaatccaatgaagtctgcaagttccaggggtcccatgg;
[0133]
cluster-32834.3
[0134]
该snp位点在cluster-32834.3所示核苷酸序列,其中自5’端起第73位和第486位碱基中的等位基因为t/a和g/c,核苷酸如下所示(seq id no.13):
[0135]
caacaacatacccaaaccagtctcagttcccaaggagaatctaaccgactaacgaggtcgtcatcctccaga(t/a)ccaaagcaaacacaatacctgcaagtgctgctttcatagatggggtggccaccaaagacatcaacattgatcctagtacactggtttcccttcgaatttttctgccagaaccagaattagcagtagcagccctcccacagggccagcagagagattcaagggatgtggatctcactctcagagcttggggtttggccaagagagaaaccatacatgaagtggattcagctcaaagttccaggaaattgaacaggaatgccagtactaatgcaaaaggggtcagttacgaatcaagttcaacacacaggaggaaaagttatggtggtattgtagaaaacatgagaagcagctatggtggtattggggacaagcagcagcgagtttgtcaggattcgttgctcagacaaggaat(g/c)gacgaagacgtgaatccatcagagaaggatagtttgctcaggtgtacggataagacatcaggatcaggccaaataaatgat;
[0136]
cluster-32938.2
[0137]
该snp位点在cluster-32938.2所示核苷酸序列,其中自5’端起第109位碱基中的等位基因为a/g,核苷酸如下所示(seq id no.14):
[0138]
agacaaaacaccaataaaggtgacatggtttggtttcacatgggagttcctcatatgttcaaacagcttgagagccttctttccttgaccatgtatggcatatcctac(a/g)atcatggcagtccatgaaacaacatttcgcctaggcattttgtcaaatactccgcatgcatcctctaggcacccacatttggcatacatatctataagggcattccccac;
[0139]
cluster-34813.0
[0140]
该snp位点在cluster-34813.0所示核苷酸序列,其中自5’端起第143位碱基中的等位基因为c/t,核苷酸如下所示(seq id no.15):
[0141]
ggtatctaactctgtattgtcaacaatttttgcctgctgtttggatgtcaaaagacctgcatcagaagattcattgcaagtttgtgcagctgtgaaatcctcatttctgaccaccatctctgaatcccttttgcaatgatat(c/t)catgctttccagtatgggatacgccctccagtctgttaaattttggttccagaacaacagttgtttcccctttatctg;
[0142]
cluster-35640.0
[0143]
该snp位点在cluster-35640.0所示核苷酸序列,其中自5’端起第125位、第527位、第895位、第1087位碱基中的等位基因为g/t、g/t、g/c、t/g,核苷酸如下所示(seq id no.16):
[0144]
ggaatgtggtgtcatggaatgcaataattgcggcatctatacagcatggttgggttaagcaggcattggaattatttgctgaaatgcaattagcaggcataaaggcgagccacgttacttttgg(g/t)ctcgttctcaacgcgtttgcgattctaggaaacctcgaacagggtaagcaactccatgcctgtgtgtttagaaatggatttggatcggatttgcttgtgggtagctctgttattctaatgtatataaagtgtggaaacatagatggtgcccgccaagtgtttgacaaaatgcctatggtagacctggggttattcaatgctacaattcaaggatatagcagcgagggccataataatgaggcaatggaactatttggtcaactactacgaacgggtttgaaaccaaatgatattaccttcaccattgttcttagagcctgtgccagctttgaaactgcccttgaacagggcaggcaactccatgtacagataatgaaatctgtgtttcagcacaatgtttc(g/t)gtgattagttctctcatcactgtgtatgctaaaagtggttgcatagctgatgcacaaaaagtgtttgacaagatgactgtacaaaacaatgtagttttatggactgcattgatagctggttacacccagaaagggtatacagacgaagccttgaaactctttctcttaatgcaacgtgcaggtgttaaacctaatcaattcacctacccaactgttc
tccgtgcttgtgcaaacttagcttctatagaagaaggaaaacaagtccattgtcatattatcaaagctggggctgcgtcggatactttcgttgccagcgccattgttgacatgtatattaagtgcggtagtctagaggatggccaacgag(g/c)gtttgatagaattcctagacgagacattgtatcgtggaatacaatgattgcaggatatgctcaacatgggtatgttgacaaggcacttttaatctttgaagaaatgcaacagtatggcatgaaacccaaccacgtaacttttgttgcagttctctctgcatgcagccatggaggacttgttcgtttagggc(t/g)ccgatactttagttccttgagccgaactcatggcattattccaagaatggagcattatgcttgcatagttgacctccttggccgtgttgggcacttgtatgaggcagaagactttatcaacaatatgcctttcgagcca;
[0145]
cluster-39957.3
[0146]
该snp位点在cluster-39957.3所示核苷酸序列,其中自5’端起第138位碱基中的等位基因为a/g,核苷酸如下所示(seq id no.17):
[0147]
ttgcagcgcatgccttaagaacacagggaaatgtgaaattatccggtcgcaaacccatcccttgcatctgatcatataatgcaagcgcctctctgcagccaccctgcctagcatacgctccaatcatggcattccac(a/g)taaacttattcctttgagccattttgtcaaacacttcgcgtgcatattccaagtttccacactttgcatacatggtgatg;
[0148]
cluster-40412.10076
[0149]
该snp位点在cluster-40412.10076所示核苷酸序列,其中自5’端起第97位碱基中的等位基因为a/c,核苷酸如下所示(seq id no.18):
[0150]
aaattggttttttgttttagaaaagacataatagaagaactgagaatcattgaaatacataaagagcagttttgtggaagactgatgtctgctcgt(a/c)tagttactctaataacacagatcatggtaatggcggtggtgatatcagttattttgcttttccttggaattggaattctggtttcaattcatctgtgtattgtgggtagagcactcagga。
[0151]
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1