一种融合多组学数据的网络标志物识别方法
1.本发明涉及生物信息领域,具体涉及网络标志物识别方法。
背景技术:
2.现代医学表明,很多疾病都是多基因疾病(由多个基因控制的疾病)。一组基因突变会导致多个生物功能发生病变,这些生物功能相互交织在一起,共同调控着某一生理活动。通过分析不同患者组的高通量组学数据,传统方法可以较容易地发现不同疾病表型背后潜在的差异基因。例如对疾病各个阶段患者的基因表达数据进行统计学意义上的倍数分析或t检验能够找出疾病背后的差异表达基因。但是,这些方法的缺点也很明显,即找出的差异基因数量很大,且无法保证这些差异基因都与疾病有关。此外,这些差异基因大部分都是孤立的,不能有效的解释疾病背后复杂的致病机理。
3.随着生物学的不断发展以及计算机运算速度的不断提高,基于生物分子网络的新方法开始被应用于分析复杂疾病背后的致病机理。例如,通过分析患者组与对照组的生物分子网络,可以得到差异子网,这极大地减少了差异基因的数目,同时精确地缩小了致病基因的寻找范围。然而,尽管基于生物分子网络的分析方法能够找到疾病相关的基因子网络,但是仍然无法准确全面地从子网络中找出致病基因。目前,许多数据挖掘方法只关注了基因间的静态调控关系,忽略了动态变化的信息。实际上,复杂疾病背后的基因调控网络往往会随着生物体内外环境的改变而动态变化。因此,为了更加准确地挖掘出疾病的致病基因,人们开始研究疾病相关分子网络的动态变化。另一方面,通过融合各种组学数据和生物分子网络数据对复杂疾病进行系统的分析,也逐渐成为了一个研究热点。因为,融合不同组学的数据能够增加数据的维度,降低数据中噪音的干扰,使数据包含更多的有效信息,从而极大地提高致病基因发现的准确率。但值得注意的是,整合使用这些异构数据,给实现这一目标的方法开发带来了巨大的挑战。
技术实现要素:
4.传统的方法虽然能够检测出疾病表型背后的差异基因,但检测出的基因数量往往较多,且基因间关联性未知,不能解释疾病的致病机理。本发明旨在通过分析正常组与患病组之间的基因差异网络,找出导致疾病的潜在基因。本发明的方法结合基因表达数据、基因突变数据、蛋白质相互作用网络数据,考虑了基因间的关联信息,因此得到基因可解释性更好。
5.本发明采用以下技术方案:
6.(1)处理基因突变数据,将样本分为三组,即cn(对照组)、mci(早期认知障碍)、ad(阿尔茨海默病患病组);使用vep工具中的sift分数评估全基因组测序文件中的每个snp,确定哪些遗传变异对于患者是有害的。基于氨基酸的序列同源性和物理性质,sift评估每个氨基酸被替代对蛋白质功能的影响。基因突变数据的表示形式如下:
[0007][0008]
其中,yk表示第k个样本的标签,k=c+m+a,c、m和a分别表示cn、mci和ad阶段的样本数量,n表示基因个数。当样本i中基因j上的snp对该基因编码的蛋白质影响程度高时,设置s
ij
=2,影响程度中等时,设置s
ij
=1表示,其他情况设置为0。
[0009]
(2)处理人类ppin(蛋白质相互作用网络)数据,将网络中的蛋白质名称转换为对应的基因名称。蛋白质相互作用数据的表示形式如下:
[0010][0011]
其中,w
n*n
为蛋白质相互作用网络的邻接矩阵的表示形式,该矩阵为对称矩阵,值为0或1,n表示基因的个数。当两个蛋白质间存在相互作用时,设置w
ij
=1,否则设置w
ij
=0。
[0012]
(3)由于使用vep工具打分后的基因突变数据过于稀疏,因此需要进一步将基因突变数据映射到蛋白质互作网络上应用网络传播算法以平滑每个基因的突变得分。网络传播算法是一种在网络上模拟随机游走的算法,其公式定义如下:
[0013]ft+1
=αf
ta′
+(1-α)f0ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(1)
[0014]a′
=d-1aꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(2)
[0015]
其中,f0是患者的原始基因突变矩阵,a是蛋白质互作网络的邻接矩阵,d是对角矩阵,d(i,i)为矩阵a第i行行和,α是一个调节参数,控制变异信号在网络传播过程中扩散的距离,α的最佳值取决于网络结构。传播函数f
t
以t=[0,1,2,
…
]迭代运行,直到f
t+1
收敛(||f
t+1-f
t
||2《1
×
10-6
,其中,||*||2为矩阵范数)。在经过网络传播之后,将所得到矩阵f
t
按行使用分位数归一化,以确保每个患者的平滑突变曲线大致符合相同的分布。
[0016]
(4)处理基因表达数据,对基因突变数据集、基因表达数据集、蛋白质互作网络数据集的基因取交集,确保在接下来的分析中所有基因都存在于每个样本中。基因表达数据的表示形式如下:
[0017][0018]
其中,yk表示第k个样本的标签,k=c+m+a,c、m和a分别表示cn、mci和ad阶段的样本数量,n表示基因个数,p
ij
表示样本i中基因j的表达量值。
[0019]
(5)将基因表达数据与基因突变数据映射到蛋白质互作网络上,然后分组分析基因间的相互作用和协同调控,并推断潜在的基因调控网络机制。通过测量疾病阶段与对照组之间的基因调控变化,研究基因调控网络和表型变化的生物学通路。定义疾病阶段a与健康的对照组b中基因a与基因b之间的差异表达调控如下:
[0020]
p(ab)=|ρa(ab)-ρb(ab)|
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(3)
[0021]
其中ρ为两个变量的皮尔森相关系数,定义如下:
[0022][0023]
当两组中的基因a与基因b的差异表达调控p(ab)≥0.7时(阈值0.7不固定,可根据实验做适当调整),认定基因a与基因b在疾病阶段和对照组中的表达调控方式发生了显著变化。
[0024]
(6)对患病组与对照组进行差异基因分析,使用t检验方法计算两个基因表达的统计量tg,通过查表得到p值,定义p值小于0.0005的基因为差异基因。假设患病组与疾病组的基因突变得分和是相等的,则零假设为:
[0025][0026]
对应的备选假设为:
[0027][0028]
t检验的计算公式是:
[0029][0030]
其中,
[0031]
[0032][0033]
这里,ni是组i(即患病组或对照组)中的样本数,表示为基因g在组i中第j个样本的基因突变的得分值。通过计算tg的值,就可以得到p值。
[0034]
本发明的有益效果:与现有的传统方法相比,本发明在考虑了基因表达数据之外,还结合了蛋白质相互作用网络数据与基因突变数据。引入蛋白质相互作用网络数据,考虑了基因间的关联信息;引入基因突变数据,考虑了基因突变对疾病进展的影响。因此,通过融合这些不同组学数据,本发明有效地解决了传统方法找出的生物标志物之间关联性较差的问题。
附图说明
[0035]
图1为本发明基于差异网络分析的阿尔茨海默病网络标志物标识方法流程图。
[0036]
图2为本发明方法找出的候选基因与随机选择的基因对cn和mci分类的roc曲线。
[0037]
图3为本发明方法找出的候选基因与随机选择的基因对cn和ad分类的roc曲线。
[0038]
图4为本发明方法找出的候选基因与随机选择的基因对mci和ad分类的roc曲线。
[0039]
图5为候选基因的go富集bp(biological process)分析结果。
[0040]
图6为候选基因的go富集mf(molecular function)分析结果。
具体实施方式
[0041]
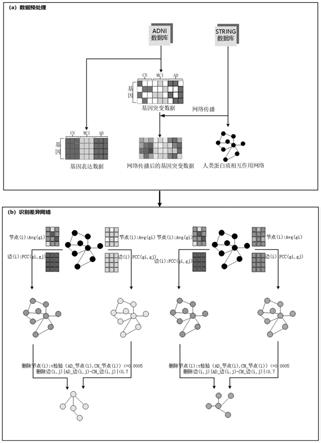
在本发明中,首先,根据全基因组测序数据对每个样本全基因组范围内所有单核苷酸多态性位点(snp)进行基因功能损害程度评估,接着对基因突变数据使用网络传播算法以保证基因突变评分的波动趋于平滑。然后,使用人类ppin(蛋白质相互作用网络)数据构建疾病不同阶段的分子相互网络,网络上的一个节点对应一个基因,节点值为该阶段所有样本上该基因突变打分的平均值;网络中的边代表基因间的相互作用,边的权重为该阶段所有患者基因表达数据中两个基因间的皮尔森相关系数,可得到不同疾病阶段的基因网络表达数据。最后,对这些网络进行差异网络分析,其中,两个网络上的差异节点(即差异基因)由t检验确定;两个网络中的差异边由皮尔逊相关系数之差的绝对值确定。
[0042]
为了使本发明的目的、技术方案及优点更加清楚明白,下面将结合附图及实施例,进一步详细说明本发明的目的以及技术方案。需注意,此处所描述的具体实施例仅仅用以解释本发明,并不对本发明的保护范围构成任何限定。
[0043]
如图1所示,先对三种数据集进行处理,并构建基因表达网络用于后续分析。
[0044]
1.处理基因突变数据:从adni数据库下载了全基因组测序vcf文件,该数据包含809个样本,平均每个样本持有超过3.88亿个snp。将样本分为三组,分别是cn(对照组)、mci(早期认知障碍)、ad(阿尔茨海默病患病组)。由于基因突变数据存储的是所有基因的snp位点信息,因此使用vep工具中的sift分数(设置sift《0.05)评估每个突变基因对患者的影响程度。处理后的基因突变数据集包含809个样本和16163个基因。使用矩阵来存储处理后的结果。矩阵的形式如下:
[0045][0046]
其中,当s
ij
=2,表示样本i中基因j上的snp对该基因编码的蛋白质影响程度高,s
ij
=1表示影响程度中等,其他情况为0;
[0047]
2.处理蛋白质相互作用网络数据:从string数据库中下载人类ppin数据,为保证后续结果的可靠性,从中筛选出具有实验佐证的蛋白质相互作用对,接着将筛选后的数据中的蛋白质名称转化为对应的基因名称。蛋白质相互相作用网络使用邻接矩阵的形式表示,当两个蛋白质间存在相互作用时,设置w
ij
=1,否则设置w
ij
=0。
[0048][0049]
3.由于使用vep工具打分后的基因突变数据过于稀疏,因此需要进一步将基因突变数据映射到蛋白质互作网络上应用网络传播算法以平滑每个基因的突变得分。网络传播算法是一种在网络上模拟随机游走的算法,其公式定义如下:
[0050]ft+1
=αf
ta′
+(1-α)f0ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(1)
[0051]a′
=d-1aꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(2)
[0052]
其中,f0是患者的原始基因突变矩阵,a是蛋白质互作网络的邻接矩阵,d是对角矩阵,d(i,i)为矩阵a第i行行和,α是一个调节参数,控制变异信号在网络传播过程中扩散的距离,α的最佳值取决于网络结构。传播函数f
t
以t=[0,1,2,
…
]迭代运行,直到f
t+1
收敛(||f
t+1-f
t
||2《1
×
10-6
,其中,||*||2为矩阵范数)。在经过网络传播之后,将所得到矩阵f
t
按行使用分位数归一化,以确保每个患者的平滑突变曲线大致符合相同的分布。
[0053]
4.处理基因表达数据:从adni数据库中下载基因表达数据集,该基因表达数据集总共包含746个样本和10061个基因,由于这些基因表达数据已经经过了标准化处理,所以可以直接使用。接着对基因突变数据集、基因表达数据集、蛋白质互作网络数据集的基因取交集,确保在接下来的分析中所有基因都存在于每个样本中。最后得到了715个共同样本(cn:247例,mci:428例,ad:40例)和8007个共同基因。基因表达数据采用矩阵存储,形式如
下:
[0054][0055]
5.将步骤1、4处理得到的基因突变数据和基因表达数据映射到蛋白质互作网络上,然后分组分析基因间的相互作用和协同调控,并推断潜在的基因调控网络机制。通过测量疾病与正常条件之间的基因相关性变化,研究基因调控网络和表型变化的生物学通路。定义疾病阶段a与健康的对照组b中基因a与基因b之间的差异表达调控如下:
[0056]
p(ab)=|ρa(ab)-ρb(ab)|
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(3)
[0057]
其中ρ为两个变量的皮尔森相关系数,定义如下:
[0058][0059]
当p(ab)≥0.70时(阈值0.7不是固定,可根据实验做适当调整),认为基因a与基因b在疾病阶段和对照组中的表达调控方式发生了显著变化。输出调控方式发生显著变化的基因对。
[0060]
6.对患病组与疾病组进行差异基因分析,使用t检验方法来寻找差异基因。
[0061]
假设患病组与疾病组的基因突变得分和是相等的,则零假设为:
[0062][0063]
对应的备选假设为:
[0064][0065]
t检验的计算公式是:
[0066][0067]
其中,
[0068][0069][0070]
这里,ni是组i(即患病组或对照组)中的样本数,表示为基因g在组i中第j个样
本的基因突变的得分值。通过计算tg的值,就可以得到p值。定义p值小于0.0005的基因为差异基因。按p值排列,输出符合条件的基因作为候选基因。
[0071]
实验结果:分析cn与mci的基因网络,选择score大于0.45的调控基因对以及p《0.0005的差异基因,共找到4个差异调控基因对和25个差异基因,使用roc曲线进行评估,结果如图2所示,本发明方法找出的候选基因曲面下面积达到了0.72,远大于随机选择基因的曲面下面积;分析cn与ad的基因网络,选择score大于0.6的调控基因对,共找到11个差异调控基因对。使用roc曲线进行评估,结果如图3所示,候选基因的曲面下面积远大于随机选择基因;分析mci与ad的基因网络,选择score大于0.7的调控基因对,共找到6个差异调控基因对。使用roc曲线进行评估,结果如图4所示,候选基因的曲面下面积依然远大于随机选择基因。
[0072]
为进一步验证候选基因的有效性,对候选基因进行go富集bp(生物过程)、mf(分子功能)分析,结果如图5、图6所示,候选基因的生物过程主要集中在神经元的发育和死亡、血管内皮细胞的增殖和迁移、肽基苏氨酸磷酸化、蛋白质结合和生长因子活性等;分子功能主要集中在蛋白磷酸酶结合、细胞因子受体结合、生长因子结合等。这表明候选基因参与了疾病相关的重要信号通路。因此本发明找出的候选基因具有较高的可靠性。
[0073]
cn与mci中差异调控基因对
[0074]
gene1gene2scorecol1a2col4a10.56cdc20chek10.48cdc45chek10.47mki67shcbp10.48
[0075]
cn与ad中差异调控基因对
[0076]
gene1gene2scoreakt1pax30.60maptpdhb0.62ctsbcst10.65cacna1hkcnc10.62nrp2vegfc0.73camk2bngf0.70stat3mgst10.65acvr1inha0.60ribc2gfap0.62ube2catg70.70fkbp3calb10.64
[0077]
mci与ad中差异调控基因对
[0078]
gene1gene2scorecol1a2col4a10.79sirt6xpc0.78
cdk18eml20.74hnrnpdlmcat0.70fmn2efcab20.71xpcnpm10.70
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1