一种用于筛选抗癌活性小分子的组合化学分子数据库CCSMD及应用
一种用于筛选抗癌活性小分子的组合化学分子数据库ccsmd及应用
技术领域
1.本发明属于医药领域,具体涉及一种用于筛选抗癌活性小分子的组合化学分子数据库ccsmd及应用。
背景技术:
2.目前,每年获批上市的新药大多是各种小分子抑制剂化合物。小分子抗癌药物在其中占有重要地位。利用小分子数据库和计算机辅助医学(cadd)是一项强大而有前景的技术,可以用于更快、更便宜,更有效的药物设计。使用公共数据库和cadd筛选活性化合物已成为一种常用方法。但是,在公共分子数据库中筛选的化合物往往面临合成困难或无法购买的结果。并且公共数据库中有大量的小分子片段(如单环芳烃、多环芳烃、短链脂肪烃等),通常很难使用。因此需要找到更好的方式来利用公共数据库中的相关资源,解决上述问题。
3.组合化学是一种更好地利用公共数据库并解决上述问题的方法。筛选数据库的建立可分为实际的组合化学数据库和虚拟组合化学数据库。然而,这些方法各有利弊,实际的组合化学数据库的筛选是建设使用化学反应罐生成大量化合物,然后使用真正的筛选方法,例如高通量筛选(hts)进行筛选,这种方法价格昂贵、速度更慢且没有靶向性。而虚拟组合化学数据库往往是以现有的框架分子为核心构建数据库。虚拟分子库建立后,虚拟筛选方法用于预测化合物如何与给定的靶蛋白相互作用。但这种方法只能与固定骨架相结合,不能广泛通用。因此,有必要建立一个能够利用现有小分子片段、易于合成、应用范围广、可供公众通过cadd发现新的活性小分子的虚拟小分子数据库。
4.本发明便是通过利用现实存在且可购买的分子砌块利用python进行组合,进而建立了一个数量较大的组合分子数据库。并且由于智能组合的条件是成熟且简易的反应条件,因此该数据库的小分子拥有易合成的特点,更加具有现实意义。可以发现各类抗癌的活性小分子。
技术实现要素:
5.针对上述技术问题,本发明的目的在于提供一种用于筛选抗癌活性小分子的组合化学分子数据库ccsmd及应用,具体包括以下内容:
6.第一方面,本发明提供了一种用于筛选抗癌活性小分子的组合化学分子数据库ccsmd,所述组合化学分子数据库ccsmd的构建方法:
7.(1)分别搜集以胺或乙酸为主的酸和胺类化合物,将收集的分子由结构式转化为smile格式;
8.(2)利用智能反应模块,将上述smile格式的酸和胺类化合物进行化学拼接,输出合成分子的smile格式,将其转化为分子结构式,并储存为sdf格式,初步构建小分子数据库;
9.(3)使用配体分子能量最小化模块最小化小分子数据库所有小分子的能量,删除最小化能量高于10000的分子;分别计算剩余的组合小分子的属性;将所有的数据进行汇总,组建组合化学分子数据库ccsmd;所述属性包括分子量、可旋转键数、氢键接受体、氢键供体、拓扑极性表面积和辛醇/水分配系数。
10.优选地,所述步骤(1)和步骤(2)中进行分子结构式与smile格式的转化软件为openbabel。
11.优选地,所述步骤(2)中智能反应模块源于python中的rdkit程序中的智能反应模块;所述步骤(3)中利用python中的rdkit程序计算组合小分子的属性。
12.优选地,所述步骤(3)中配体分子能量最小化模块为autodock的配体分子能量最小化模块。
13.第二方面,本发明提供了上述第一方面所述的组合化学分子数据库ccsmd或在筛选抗癌活性小分子中的应用。
14.第三方面,本发明提供了一种用于筛选抗癌活性小分子检索系统,其所述系统包括:
15.构建上述第一方面所述的组合化学分子数据库ccsmd,所述组合化学分子数据库ccsmd用于存储化合物的属性数据,所述化合物属性包括分子量、可旋转键数、氢键接受体、氢键供体、拓扑极性表面积和辛醇/水分配系数;
16.根据输入的化合物属性生成检索模型;
17.基于检索模型在组合化学分子数据库ccsmd中对用户输入的待检索化合物进行检索,若检索到与用户输入化合物属性对应一致的化合物,则反馈检索出的化合物组合分子结果至用户;若未检索到与用户输入化合物属性对应一致的化合物,则反馈未检索到匹配化合物的结论至用户。
18.第四方面,本发明提供了上述第三方面所述的检索系统在筛选抗癌活性小分子中的应用。
19.第五方面,本发明提供了一种筛选抗癌活性小分子的方法,所述方法包括以下步骤:
20.(1)构建上述第一方面所述的组合化学分子数据库ccsmd或上述第三方面所述的检索系统;
21.(2)选择分子量、可旋转键的数量、氢键受体、氢键供体、拓扑极性表面积、辛醇/水分配系数中的任一项或几项为检索对象,生成检索模型;通过组合化学分子数据库ccsmd检索获得目标化合物;
22.(3)通过分子对接软件将检索的目标化合物与人环蛋白依赖性激酶6蛋白结构进行对接,筛选获得高分组合分子集合;
23.(4)根据获得的高分组合分子集合合成对应的小分子化合物;
24.(5)通过体外mtt活性评价,筛选获得抗癌活性小分子。
25.优选地,所述步骤(3)中的分子对接软件选自开源分子对接软件autodock vina。
26.第六方面,本发明提供了根据上述第五方面所述方法筛选获得的化合物在制备抗肿瘤药物中的应用。
27.第七方面,本发明提供了一种根据上述第五方面所述方法筛选获得的具有抗肿瘤
活性的化合物,所述化合物的结构式如下式(ⅰ)-(ⅱ)所示:
[0028][0029]
第八方面,本发明提供了上述第七方面所述的化合物或其药学上可接受的盐在制备抗肿瘤药物中的应用。
[0030]
优选地,所述肿瘤为胃癌。
[0031]
第九方面,本发明提供了上述第七方面所述的化合物或其药学上可接受的盐加入药学上可接受的载体和/或辅料,制成片剂、喷雾剂、颗粒剂、胶囊剂、口服液、针剂、混悬剂中的任一种剂型。
[0032]
第十方面,本发明提供了上述第七方面所述化合物的制备方法,所述方法为:将原料酸在氯化亚砜中加热至75℃搅拌2h;之后将氯化亚砜浓缩蒸干获得白色固体的酰氯中间体,将酰氯加入冰浴的thf中搅拌5min之后,再加入原料胺,搅拌3h后减压浓缩体系,残余物通过硅胶柱色谱法提纯即得。
[0033]
本发明的有益效果是:本发明首先利用智能虚拟组合化学的方法可以构建一个数量巨大的组合化学分子数据库ccsmd;所述数据库采用市售的价廉易得的分子砌块作为基础,采用简便的化合反应作为条件进行合成拼接,不但具有较大的数量级,并且可以简易的自我合成,也从而具有性价比高的特点;利用本发明所述数据库进行虚拟筛选从而得到的分子结构,可以低价且简便的获得;并且利用本发明诉述数据库能够筛选获得对于癌症具有低μm活性的化合物,所述化合物安全性较好,对正常胃黏膜细胞的基本无毒,具有显著的治疗胃癌的效果;同时所述化合物具有易合成的特点,更加具有现实意义,因此具有良好的应用前景
[0034]
说明书附图
[0035]
为了更清楚地说明本发明实施例的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,应当理解,以下附图仅示出了本发明的某些实施例,因此不应被看作是对范围的限定,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他相关的附图。
[0036]
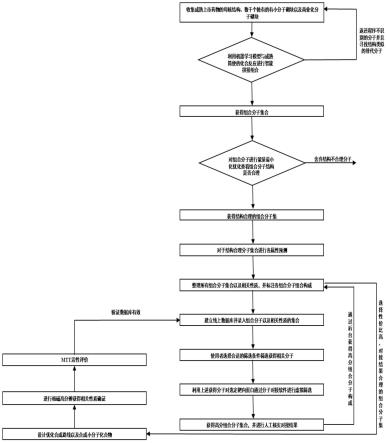
图1数据库的构建及化合物筛选流程图;
[0037]
图2数据库ccsmd相关网页信息;其中a为在ccsmd的搜索页面,可以使用图标搜索栏来限制clogp和分子量的范围,从而筛选出分子;b为搜索到的分子显示其相关的计算化学特性;c为显示了每个化学性质在不同数量级上的比例;
[0038]
图3建立数据库到使用数据库的具体过程,其中,a为建立数据库的主要过程;b为进行虚拟筛选的步骤;
[0039]
图4使用ccsmd对选定的蛋白质进行虚拟筛选的相关结果;其中,a为以cdk6(pdb:5l2i)为目标蛋白,选择一个能完全包裹蛋白自身配体分子的方框(center x=12.46,center y=29.63,center z=13.28,size x=17.34,size y=14.67,size z=25.11)作为定位框;b为用热图直观地显示ccsmd的对接结果,将删除的不合适分子的分数定义为0,将合成的组合分子的分数定义为14,可以直观地观察合成分子的位置,其余的分子作为分数结果输出(图表的限定范围为-14至14);c为了增加所有评分结果的对比度,将所有化合物作为评分结果输出,未对接的评分结果为0(图表的限定范围为-14到-7);
[0040]
图5化合物(ⅰ)和(ⅱ)与目标蛋白结合的对接结果;其中,a为cdk6的疏水性表面,红色为疏水性,蓝色为亲水性;b和c为筛选的化合物(ⅰ)和(ⅱ)cdk6的对接结果显示,配体分子范围内的所有蛋白质的氨基酸残基都标为黄色;d中深蓝色的线是cdk6和筛选的化合物(ⅰ)的氨基酸残基之间的力;
[0041]
图6化合物(ⅰ)和(ⅱ)的细胞存活率;其中,a为化合物(ⅰ)对ags的48小时抑制活性。b为(ⅰ)对ges-1的48h抑制活性;c为化合物(ⅱ)对ags的48h抑制活性;d为化合物(ⅱ)对ges-1的48小时抑制活性;e为顺铂对ags的48小时抑制活性;f为顺铂对ges-1的48小时抑制活性;g为三种化合物对ags存活率的比较;h为三个化合物对ges-1存活率的比较。
具体实施方式
[0042]
为使本发明实施例的目的、技术方案和优点更加清楚,下面将对本发明实施例中的技术方案进行清楚、完整地描述。实施例中未注明具体条件,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市售购买获得的常规产品。
[0043]
实施例1组合化学分子数据库ccsmd的构建及筛选
[0044]
数据库的构建及化合物筛选流程图如图1所示;数据库检索流程如图2所示;
[0045]
收集数千种独特的小分子构建块和商业化的分子构建块。包含各种商业化构建单元分子和临床药物片段的cas号、化学式、分子结构式等信息。
[0046]
通过间接生成mysql查询的web界面访问集成数据。接口支持“分子量范围”、“clogp范围”等查询功能。此外,根据个人需求进行基本统计和可视化。为感兴趣的作者提供了用于验证或进一步研究的文章链接。
[0047]
ccsmd系统包括smile格式的所有分子及其各自的物理化学性质。数据库是由rdkirt的智能模块构建的,通过简单的化学反应将两种不同的分子构建块组合成一个新的小分子。这种化学反应可以通过简单的条件完成。将所有收集到的小分子积木组合起来,初步构建了一个小分子数据库,共有12992个。但是这个数据库中有很多不合理的分子结构,因此使用autodock的配体分子能量最小化模块,将所有小分子的能量最小化。将最小化能量高于10,000的分子全部被删除,剩余分子总数为12,908,可用molecules占所有分子的
99.35%。剩余的组合小分子使用rdkit程序计算以下性质:分子量(mw)、可旋转键数(rb)、氢键受体(hba)、氢键供体(hbd)、拓扑极性表面积(tpsa),和辛醇/水分配系数(clogp)。所有上述过程都可以在图2中a中表示。
[0048]
以人cyclin-dependent kinase 6(cdk6)和palbociclib(pdb:5l2i)的晶体结构为靶蛋白结构,选择一个完全包裹配体的盒子作为对接位点(如图4所示)。配体数据库采用数据库ccsmd,并选择广泛认可的开源分子对接软件autodock vina进行对接。对接结果通过chimera 1.14可视化工具对评分结果中排名前100的分子进行可视化和检查。提出了可能与蛋白质口袋结合的化合物并准备合成。
[0049]
实施例2筛选获得的化合物的合成及活性验证实验
[0050]
根据数据库筛选的化合物的结构式如下式(ⅰ)-(ⅱ)所示:
[0051][0052][0053]
1.化合物(ⅰ)和(ⅱ)的合成
[0054]
化合物(ⅰ):(9h-芴-9-基)甲基(s)-(1-((4-氯-2,5-二甲基苯基)氨基)-1-氧代-4-炔-2-基)氨基甲酸酯。
[0055]
合成方法:将原料(s)-2-((((9h-芴-9-基)甲氧基)羰基)氨基)戊-4-炔酸(50.0毫克,0.149毫摩尔)加入5毫升氯化亚砜中,在75℃搅拌2小时。然后减压浓缩体系,直到产生白色固体,得到中间体(9h-芴-9-基)甲基(s)-(1-氯-1-氧代-4-炔-2-基)氨基甲酸酯,将其加入到thf中,然后将原料4-氯-2,5-二甲基苯胺(23.1毫克,0.148毫摩尔)和dipea(77.0毫克,0.596毫摩尔)加入到thf,在冰浴条件下搅拌3小时。之后,减压浓缩体系,残余物通过硅胶柱色谱法提纯,产品用石油醚/乙酸乙酯(4:1)提纯,得到白色固体化合物(ⅰ)(46.5毫克,产率66%)。化合物(ⅰ);产量,66%;白色固体;1h nmr(400mhz,dmso-d6)δ9.47(s,1h),7.85(d,j=7.5hz,2h),7.76(d,j=8.0hz,1h),7.70(d,j=7.4hz,2h),7.37(t,j=7.4hz,2h),7.31(s,1h),7.30
–
7.24(m,2h),7.23(s,1h),4.36(q,j=7.4hz,1h),4.28(t,j=7.1hz,2h),4.20(t,j=6.9hz,1h),2.90(s,1h),2.58(qd,j=17.6,16.4,7.0hz,2h),2.22(s,3h),2.10(s,3h)。
[0056]
化合物(ⅱ):(9h-芴-9-基)甲基(s)-(1-((6-溴萘-2-基)氨基)-1-氧代-4-炔-2-基)氨基甲酸酯。
[0057]
合成方法:将原料(s)-2-((((9h-芴-9-基)甲氧基)羰基)氨基)戊-4-炔酸(50.0毫克,0.149毫摩尔)加入5毫升氯化亚砜中,在75℃搅拌2小时。然后减压浓缩体系,直到产生白色固体,得到中间体(9h-芴-9-基)甲基(s)-(1-氯-1-氧代-4-炔-2-基)氨基甲酸酯,将其加入thf中,然后将原料6-溴萘-2-胺(32.8毫克,0.148毫摩尔)和dipea(77.0毫克,0.596毫摩尔)加入thf中,在冰浴条件下搅拌3小时。之后,将体系减压浓缩,残余物在硅胶上柱层析提纯,用石油醚/乙酸乙酯(3:1)提纯产品,得到白色固体化合物(ⅱ)(58.7毫克,产率73%)。化合物(ⅱ);产率,73%;白色固体;1h nmr(400mhz,dmso-d6)δ10.40(s,1h),8.30(s,1h),8.08(s,1h),7.91
–
7.73(m,5h),7.70(d,j=7.4hz,2h),7.62(dd,j=8.9,2.1hz,1h),7.54(dd,j=8.8,2.1hz,1h),7.37(t,j=6.4hz,2h),7.28(td,j=7.5,3.6hz,2h),4.36(q,j=8.0hz,1h),4.33
–
4.16(m,3h),2.89(t,j=2.6hz,1h),2.70
–
2.60(m,1h),2.55(ddd,j=16.7,8.4,2.6hz,1h)。
[0058]
2.化合物(ⅰ)和(ⅱ)与目标蛋白结合的对接结果
[0059]
方法:使用chimera 1.14先打开cdk6蛋白结构文件(pdb:5l2i),去除文件中的蛋白配体,将之前对接好的对接结果文件在此基础上打开,发现目标分子在靶蛋白对接位点中出现,将分子和靶蛋白之间的氢键显现出,观察分子和蛋白之间的结合关系,为了更好的观察,将对接分子以内的所有蛋白质分子都显现出来进行观察。
[0060]
结果:化合物(ⅰ)与目标蛋白结合的对接结果如图5中c和d所示,可以看出,氢键作用和疏水作用对(ⅰ)的结合最为关键。37tyr 24残基的骨架上的羟基可以分别与(ⅰ)的氧原子和氢原子形成氢键。gln 149和asn 150的肽键与(ⅰ)的氢原子形成氢键。(ⅰ)的三环结构被一些疏水残基所掩盖,如phe 98、val 77、val 101leu 152、ala 162、val 27和ile 17形成一个疏水口袋,使(ⅰ)和cdk6形成强烈的疏水作用,进一步稳定了(ⅰ)的结合。化合物(ⅱ)与目标蛋白结合的对接结果如图5b所示。和(ⅰ)是类似的,(ⅱ)的一侧也有一个fmoc基团。三环结构的fmoc基团是一个疏水基团,所以它可以很好地与cdk6的疏水口袋结合,其它的结合位点与(ⅰ)相似。
[0061]
3.化合物(ⅰ)和(ⅱ)的抗肿瘤活性检测
[0062]
方法:mtt方法用于通过线粒体脱氢酶活性来测量活细胞(sigma)。细胞被安置在96孔板中,10,000个细胞/100微升/孔。24小时后,用新鲜培养基中的化合物处理细胞浓度分别为。48小时后,每孔加入10μl mtt溶液(5mg/ml),除去培养基,加入100μl dmso作为裂解液,然后在摇晃孔后在490nm处测量吸收率。
[0063]
结果:化合物(ⅰ)和(ⅱ)以及顺铂的抗肿瘤活性检测结果如图6所示,结果表明,化合物(ⅰ)和(ⅱ)对于ags的抑制率都随着浓度的增加而增加,而对于ges-1则无明显的梯度抑制作用。本发明所述的化合物(ⅰ)和(ⅱ)能显著抑制胃癌细胞的增殖,效果优于顺铂,且对正常细胞基本无影响。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1