鲍曼不动杆菌多糖抗原检索和注释分析系统及其构建方法与流程
1.本发明涉及多糖抗原研究领域,主要涉及鲍曼不动杆菌各分型多糖抗原结构、多糖抗原合成及单糖合成信息快速检索和基于基因组的糖基转移酶注释系统。
背景技术:
2.抗生素是治疗细菌性的传统有效手段,但是,抗生素滥用加剧了多重耐药的“超级细菌”出现。多重耐药细菌病体在全球范围内流行和传播,尽管有感染防护程序在运转,这些病原体在临床环境中仍然持续存在。多重耐药的鲍曼不动杆菌,如鲍曼不动杆菌的碳青霉烯类耐药菌株已被世界卫生组织(world health organization)确定为治疗药物开发的关键事项,因为目前几乎已达到无抗生素可用的境地。针对鲍曼不动杆菌感染,多糖结合疫苗的研发,备受全球市场的瞩目。细菌表面多糖可作为疫苗、诊断试剂组分和免疫相关药物,深入了解鲍曼不动杆菌表面多糖结构及合成机制,是多糖生物学合成和疫苗研发的重要基础。
3.鲍曼不动杆菌表面多糖,主要包括k-抗原(k-antigen )/荚膜多糖(capsular polysaccharide,cps),和脂低聚糖外核心(lipooligosaccharide outer core,oc)。荚膜多糖是细菌重要的保护性抗原和主要毒力决定因素,在噬菌体疗法和疫苗的关键靶点,通常由寡糖亚单位(k units)重复紧密连接组成。荚膜多糖因其良好的免疫原性,已被广泛应用于多糖疫苗的研发。荚膜多糖的结构特征、合成机制和生物学功能,近年来逐渐成为研究热点。通常,鲍曼不动杆菌k-unit由4-6个糖基组成,第一的单糖由初始转移酶(itr)催化,然后各特异糖基转移酶(glycosyl transferase,gtr)逐个催化糖基供体和受体间生成糖苷键,可形成完整的k-unit,最后经wzx转位酶和wzy聚合酶形成多糖结构。糖基转移酶是多糖合成中的关键酶分子,具有高度的特异性、底物专一性和多样性,且可以活性形式体外表达,是生物法制备多糖疫苗中最为关键的要素。糖基转移酶基因位于多糖合成基因簇中,可编码特异性的糖基转移酶,对应不同的糖基受体、糖基供体和糖苷键。
4.荚膜多糖合成基因簇(k locus),位于染色体的fkpa和lldp基因间序列,包括荚膜转运、荚膜多糖生成、单糖(simple sugar)合成三个基因编码区域。部分的简单糖(simple sugar,如glc)合成基因位于染色体其他位置,而复杂糖(complex sugar,如pse5ac7ac、aci5ac7ac)的合成和修饰基因通常均位于合成基因簇内。目前主流的生物学合成多糖疫苗方法,包括通过克隆多糖合成基因簇,构建表达体系后在工程菌中进行表达,或者在多糖合成基因簇基础上改造以实现目标多糖的表达。所以,明确多糖合成基因簇中的基因和功能对应关系,包括糖基转移酶先关基因和单糖合成相关基因,对多糖疫苗研发中多糖的高效表达体系设计和建立,至关重要。依据荚膜抗原,鲍曼不动杆菌可分为多种分型,随着高通量测序技术的发展,可直接基于荚膜多糖合成基因簇序列进行分型,各分型间的多糖抗原结构(k-unit结构)、糖基转移酶、合成基因簇序列等存在差异。鲍曼不动杆菌脂低聚糖外核由两个区域组成,合成基因簇位于染色体asps和ilve基因之间,每种基因簇对应一个分型,如ocl1、ocl2等。鲍曼不动杆菌已报道的荚膜多糖分型(k loci serotype,kl)分型超过128
种,已报道的脂低聚糖外核分型(oc loci, ocl)分型14种。
5.细菌表面多糖合成基因簇序列、多糖结构、单糖合成通路、糖基转移酶及其特异糖基供体、受体和糖苷键信息,对多糖合成机制研究及多糖结合疫苗研发是重要研究基础。近年来高通量测序技术的快速发展,推动了细菌表面多糖研究,基于基因组序列可进行菌株分型,如已报道的kaptive软件,可实现基于肺炎克雷伯氏菌和鲍曼不动杆菌基因组的菌株分型。近年来发表的细菌表面多糖信息整合和搜索平台,如大肠杆菌表面多糖数据库ecodab(https://nevyn.organ.su.se/ecodab/index.php)和ek3d(https://www.iith.ac.in/ek3d/),分别展示了大肠杆菌o抗原和k抗原的分型列表和多糖抗原结构图;肺炎克雷伯氏菌在线数据平台k-pam(https://iith.ac.in/k-pam/),展示了分型列表、多糖结构图和在线分型预测工具。
6.然而,这些已公开发表的大肠杆菌和肺炎克雷伯氏菌表面多糖信息平台,在实际应用中存在着以下几点不足:1)缺失多糖合成生物学中重要信息,如多糖合成中的糖基转移酶(及其对应的基因、糖基供体、糖基受体和糖苷键)信息、单糖合成通路、合成基因簇序列和功能结构信息等,无法进行快速的分析、检索和浏览,而这些信息可作为多糖抗原改造合成和定制合成的重要基础;2)缺乏糖基转移酶注释分析工具,糖基转移酶是多糖疫苗生物学合成中的关键要素,探索新菌种或新菌株基因组中糖基转移酶基因位置、序列和功能,可为生物学合成中酶的功能集成或改造提供方向;3)缺失各分型对应的基因组序列和菌株信息,无法快速检索公共数据库数千个基因组对应对应的分型、糖基转移酶和多糖抗原结构信息,这些信息可作为临床研究或疫苗多糖合成生物学研究的基础;4)多糖结构图中缺失糖基转移酶位置信息,用户无法直观获得全部糖苷键对应的糖基转移酶信息,而基于文献资料的该类信息搜集比较耗费时间资源;5)仅涉及大肠杆菌和肺炎克雷伯氏菌,而鲍曼不动杆菌作为重要的重耐药感染菌,其表面多糖信息多见于发表的文献中,目前缺乏可快速、准确搜索鲍曼不动杆菌多糖信息的数据库或平台。
技术实现要素:
7.为了克服现有技术的不足,本发明的目的是提供一种鲍曼不动杆菌多糖抗原检索和注释分析系统及其构建方法。
8.一种鲍曼不动杆菌多糖抗原检索和注释分析系统的构建方法,步骤如下:s1 abausp-db数据库构建从已发表文献资料和公共数据库,下载鲍曼不动杆菌多糖抗原和基因组序列数据,分析数据、绘制结构图,构建abausp-db;s1-1 多糖抗原结构及合成、单糖合成通路信息整理从已发表文献资料和公共数据库ncbi、gtdb中,下载和整理鲍曼不动杆菌的荚膜多糖(capsular polysaccharide/k antigen)和脂低聚糖外核(outer core of lipooligosaccharide)分型信息、结构信息、糖基转移酶及对应糖苷键类型、糖基供体和糖
基受体信息、合成基因簇序列和功能信息,同时根据已发表文献整理单糖合成通路信息,基于整理多糖抗原信息,绘制鲍曼不动杆菌多糖抗原结构图和合成基因簇功能结构图;s1-2 基因组数据分析从公共数据库下载已发表鲍曼不动杆菌基因组序列,过滤低质量、高污染或命名错误的基因组序列。对基因组采用自主撰写流程进行分型和糖基转移酶注释,结果关联菌株号、基因组和多糖抗原信息;s2 分型和糖基转移酶数据库(gtrdb)构建从公共数据库ncbi、gtdb和metacyc和文献资料,下载所有细菌物种的糖基转移酶序列信息和糖基供体、受体信息,结合文献资料中下载的鲍曼不动杆菌糖基转移酶信息,去除冗余信息后构建糖基转移酶注释用参考数据;从公共数据库(ncbi)和文献资料,下载用于各多糖抗原分型的基因序列信息,构建分型参考数据,整合以上数据构建可用于分型和糖基转移酶数据库(gtrdb);s3 多糖抗原浏览模块创建创建鲍曼不动杆菌多糖抗原分型列表,选择任一血清型名称,可查看该血清型对应的多糖抗原结构、基因簇结构和菌株信息;s4 多糖抗原检索模块创建创建检索输入模块,可输入分型,快速检索多糖抗原信息;可输入菌株号,检索相应多糖抗原信息;可输入单糖名称、糖基转移酶名称或糖苷键类型,检索其关联的糖基供体、糖基受体、糖苷键类型、糖基转移酶和血清分型等信息;输入糖基转移酶名称,检索其相应的糖基供体、糖基受体、糖苷键、血清型信息;s5 单糖合成通路浏览和查询模块根据已发表文献资料,绘制各单糖合成通路图和所有单糖合成总通路图,并创建单糖合成通路列表,用户可检索获得单糖对应的合成通路详细信息;s6 用户数据上传模块上传提交细菌基因组序列,或鲍曼不动杆菌基因组序列,以进行细菌糖基转移酶注释或鲍曼不动杆菌分型和注释;s7 注释分析模块:对上传的细菌基因组序列,进行糖基转移酶注释分析;s7-1 通过blastx程序将基因组序列比对至gtrdb数据库,得到比对的匹配数据;s7-2 基于相似性和比对期望值,筛选获得最佳匹配基因信息;s7-3 根据最佳匹配基因信息,得到糖基转移酶注释结果,并邮件反馈用户;s8 构建鲍曼不动杆菌分型和注释模块:s8-1 通过公共数据库和文献资料,下载鲍曼不动杆菌各分型荚膜多糖合成基因簇和脂低聚糖外核合成基因簇序列,转换成可识别的dna参考序列;s8-2 用户上传的鲍曼杆菌不动杆菌基因组序列,采用tblastn程序比对至dna参考序列;s8-3 根据覆盖度、相似性阈值,对比对结果进行筛选,并以列表形式展示分型结果;s8-4 分型完成后,进行糖基转移酶注释,结果同分型结果共同反馈给用户。
9.所述abausp数据库用于整合鲍曼不动杆菌抗原多糖数据,包括各分型抗原多糖结构式和结构图、合成基因簇功能结构图、菌株名称、多糖合成信息和单糖合成通路信息。
10.所示多糖抗原浏览模块,含多糖抗原109种,可浏览多糖抗原信息表,含血清分型对应的多糖抗原名称和结构、基因簇功能结构、菌株名称和糖基转移酶信息。
11.所述多糖抗原检索模块可输入菌株名称或基因组序列号,可返回用户多糖抗原信息表和糖基转移酶注释表格,含物种名称、菌株号、基因名称、基因描述信息、基因位置、是否为糖基转移酶、糖苷键、糖苷键类型、糖基供体、糖基受体;输入血清型名称,检索匹配后可返回用户多糖抗原信息表;输入单糖、糖基转移酶或糖苷键信息,检索匹配后并返回用户糖基转移酶信息列表,含糖基转移酶基因名称、基因编号、糖苷键、糖基供体、糖基受体、糖苷键类型、物种、菌株号、血清型和参考文献信息。
12.所述单糖合成通路模块,可浏览鲍曼不动杆菌合成通路信息,用户也可输入单糖名称,检索匹配后可获得单糖合成通路信息,含对应的血清分型、菌株号、合成基因名称、终产物单糖名称、反应前体、反应产物、合成步骤信息、单糖合成分类信息、单糖合成通路图。
13.所述分析和糖基转移酶注释模块,可上传鲍曼不动杆菌基因组序列,返回用户的分析结果含血清分型结果和糖基转移酶注释结果,注释结果包括基因序号、糖基转移酶名称、位置、糖基供体、糖基受体、糖基转移酶简称、糖基转移酶家族分类信息;或可上传任一细菌基因组序列进行糖基转移酶注释,返回用户结果列表含基因序号、糖基转移酶名称、位置、糖基供体、糖基受体、糖基转移酶简称、糖基转移酶家族分类信息。
14.所述分型和糖基转移酶数据库gtrdb,去除冗余后共计细菌糖基转移酶是24807条,可对细菌全基因组范围的糖基转移酶进行注释,不限于荚膜多糖。
15.所述的s1-1中采用csdb/snfg structure editor工具绘制的结构图,结构图中的糖苷键位置标注了对应的糖基转移酶信息。
16.所述的s1-2中下载鲍曼不动杆菌基因组后,采用checkm软件、kmerfinder软件、分型软件等工具进行基因组评估、菌种鉴定和血清型分型(k抗原分型和ocl分型),过滤掉基因组片段数大于500、完整度小于85%、污染率大于5%、鉴定结果非鲍曼不动杆菌、多糖抗原基因簇不完整的低质量基因组序列。
17.一种鲍曼不动杆菌多糖抗原检索和注释分析系统,包括多糖抗原浏览模块、多糖抗原检索模块、单糖合成通路查询模块,用于糖基转移酶注释的分析和用户数据提交模块;所示多糖抗原浏览模块,含多糖抗原109种,可浏览多糖抗原信息表,含血清分型对应的多糖抗原名称和结构、基因簇功能结构、菌株名称和糖基转移酶信息;所述多糖抗原检索模块可输入菌株名称或基因组序列号,可返回用户多糖抗原信息表和糖基转移酶注释表格,含物种名称、菌株号、基因名称、基因描述信息、基因位置、是否为糖基转移酶、糖苷键、糖苷键类型、糖基供体、糖基受体;输入血清型名称,检索匹配后可返回用户多糖抗原信息表;输入单糖、糖基转移酶或糖苷键信息,检索匹配后并返回用户糖基转移酶信息列表,含糖基转移酶基因名称、基因编号、糖苷键、糖基供体、糖基受体、糖苷键类型、物种、菌株号、血清型和参考文献信息;所述单糖合成通路模块,可浏览鲍曼不动杆菌合成通路信息,用户也可输入单糖名称,检索匹配后可获得单糖合成通路信息,含对应的血清分型、菌株号、合成基因名称、终产物单糖名称、反应前体、反应产物、合成步骤信息、单糖合成分类信息、单糖合成通路图;
所述分析和糖基转移酶注释模块,可上传鲍曼不动杆菌基因组序列,返回用户的分析结果含血清分型结果和糖基转移酶注释结果,注释结果包括基因序号、糖基转移酶名称、位置、糖基供体、糖基受体、糖基转移酶简称、糖基转移酶家族分类信息;或可上传任一细菌基因组序列进行糖基转移酶注释,返回用户结果列表含基因序号、糖基转移酶名称、位置、糖基供体、糖基受体、糖基转移酶简称、糖基转移酶家族分类信息。
18.本发明的有益效果本发明建立鲍曼不动杆菌表面多糖信息快速检索、基因组糖基转移酶注释方法及系统,适用于建立一个整合合成生物学、基因组学的多糖抗原信息检索及糖基转移酶注释分析平台。
19.1、与已发布的大肠杆菌或肺炎克雷伯氏菌多糖信息平台相比,呈现更丰富多糖合成生物学相关信息,包括多糖合成信息(关键酶即糖基转移酶及对应的糖苷键、供体糖、受体糖、合成基因簇结构等信息)和单糖合成信息(合成反应步骤信息和合成通路图),用户可快速浏览或检索,为疫苗尤其是鲍曼不动杆菌多糖疫苗研发等方向的合成生物学研究奠定重要基础。
20.2、本发明不仅可用于多糖抗原信息检索,还可用于糖基转移酶注释,在线平台可对任一细菌基因组序列直接进行糖基转移酶注释,为如疫苗研发中的多糖合成提供快速、便捷的技术工具,利于用户分析新发现菌株或人工合成基因簇中的糖基转移酶位置和功能信息。
21.3、本发明对已发表鲍曼不动杆菌(质控后为7012个)基因组信息,全部进行分型、糖基转移酶注释,用户可直接输入基因组序列名称或菌株名称,检索该菌株对应的分型信息、表面多糖抗原信息、合成基因簇结构和序列信息、糖基转移酶注释信息,为鲍曼不动杆菌表面多糖基础研究提供可靠的信息平台。
22.4、填补了鲍曼不动杆菌表面多糖抗原信息检索平台空白,可浏览或检索各分型对应的多糖结构图(含糖基转移酶和糖苷键对应信息)、合成基因簇功能结构图(含基因功能分类)、发表文献信息,可输入单糖、糖苷键、糖基转移酶等任一名称进行相关信息检索,用户可在数秒内获得检索结果。
附图说明
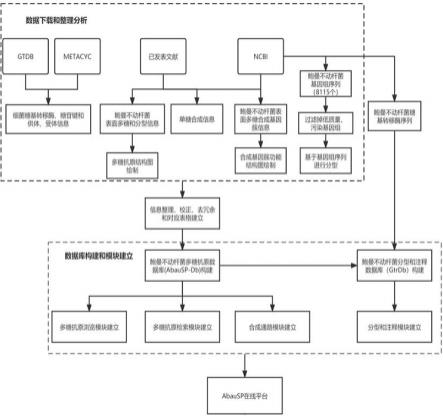
23.图1为本发明数据库平台abausp构建流程图。
24.图2为实施例abausp平台-鲍曼不动杆菌多糖抗原分型列表界面。
25.图3为实施例abausp平台-鲍曼不动杆菌多糖抗原分型信息浏览页面。
26.图4为实施例abausp平台-鲍曼不动杆菌多糖抗原信息检索界面。
27.图5为实施例abausp平台-鲍曼不动杆菌单糖合成通路浏览检索界面。
28.图6为实施例abausp分型和注释模块。
29.图7为实施例鲍曼不动杆菌基因组序列提交abausp分型和糖基转移酶注释结果图。
30.图8为实施例大肠杆菌k-12菌株基因组糖基转移酶注释分析结果图(糖基转移酶注释结果信息行数较多,仅示意性截取部分内容)。
具体实施方式
31.为了便于本领域研究人员理解和实施本发明,下面结合附图及具体实施方式对本发明做进一步的详细描述。
32.实施例1如图1所示,abausp数据库和gtrdb数据库构建步骤如下:1)从公共数据库(ncbi、metacyc、gtdb)和已发表文献资料中,下载和整理鲍曼不动杆菌的荚膜多糖(k antigen/capsular polysaccharide)和脂低聚糖外核(outer core of lipooligosaccharide)分型信息、结构信息、糖苷键类型及对应糖基转移酶、合成基因簇序列(genbank格式)、单糖合成通路信息。共整理荚膜多糖抗原分型(kl分型)94个、脂低聚糖外核抗原分型(ocl分型)15个,及各分型对应的多糖结构式、合成基因簇序列、糖基转移酶和糖苷键对应信息,建立对应表格。
33.2)基于整理多糖结构、糖基转移酶和合成基因簇信息,采用在线工具csdb/snfg structure eidtor绘制鲍曼不动杆菌多糖抗原结构图,并添加在糖苷键位置添加对应的糖基转移酶名称;根据文献中基因功能分类,包括简单糖合成(simple sugar synthesis)、核糖合成(nucletide-sugar biosynthesis)、重复单元加工(reapt unit processing)、糖基转移酶(glycosyltransferase)、起始转移酶(initial transferase)、其他(other)等功能,绘制合成基因簇功能结构图,与各抗原分型建立对应表格。
34.3)从公共数据库(ncbi)下载已发表鲍曼不动杆菌基因组序列(共计8115个),采用checkm软件和自主撰写perl程序看流程,过滤掉contigs/scaffolds数目大于500个、命名错误、完整度小于85%,污染率大于5%的基因组,筛选后鲍曼不动杆菌基因组序列为7012个。采用自主撰写的perl程序流程,对筛选后基因组进行血清型分型和糖基转移酶注释,对应菌株号、多糖抗原分型、基因组序列号和多糖抗原结构信息。
35.4)基于所述1)、2)、3)步骤结果,构建abausp数据库,含鲍曼不动杆菌多糖抗原分型(kl、ocl),及各分型对应的菌株号、基因组序列号、多糖抗原结构图、合成基因簇功能结构图。
36.5)基于公共数据库(ncbi、matacyc、gtdb)和文献资料,下载和整理糖基转移酶序列和功能信息共计是89959条序列,去冗余后,共计是24807条;6)从已发表文献和公共数据库(ncbi)下载已发表的15个ocl分型和94个kl分型位点序列(genbank格式),从中提取dna序列和蛋白序列(fasta格式)作为分型参考数据;7)基于abausp数据库和5)、6)信息,对应糖基转移酶名称、基因序列、糖苷键、糖基受体、糖基沟通、多糖抗原分型表格,构建分型和注释数据库(gtrdb),可用于鲍曼不动杆菌基因组抗原分型和糖基转移酶注释。
37.实施例2如图1所示,本实施例提供了一种鲍曼不动杆菌多糖抗原检索和注释分析系统(abausp)。该系统使用方便,功能丰富,可浏览各个鲍曼不动杆菌多糖抗原分型列表和信息,可快速检索鲍曼不动杆菌多糖抗原结构和合成基因簇结构信息、单糖合成通路,用户可上传基因组序列进行鲍曼不动杆菌分型和细菌糖基转移酶功能注释。可为多糖抗原生物学合成和疫苗研发及鲍曼不动杆菌流行病学研究,提供快速的知识查询和分型、注释分析工具。
38.本实施例采用的数据库整合了已发表鲍曼不动杆菌的分型信息和多糖信息资源,建立了一种鲍曼不动杆菌表面多糖快速检索与基因组糖基转移酶注释系统:该系统旨在构建网络数据库以提供鲍曼不动杆菌分型与注释结果,可根据分型结果对鲍曼不动杆菌流行状况进行溯源追踪,同时可对全基因组进行糖基转移酶功能注释。abausp-db数据库收录的多糖抗原分型和多糖结构、合成基因簇数据来源于公共数据库和已发表文献资料,分型和注释数据库收录已发表文献资料和公共数据库(ncbi、gtdb)数据,结合文本挖掘和手动检索资料并予以整合。在数据库数据资源的基础上,设计数据库系统并提供在线网页检索服务,兼容用户可上传基因组序列进行自动化分型和糖基转移酶注释的分析工具。
39.1)数据库系统架构本实施例采用的数据库系统架构包括:linux操作系统、mysql数据库管理系统、php和html程序设计语言。用django框架构搭建系统分析网站,采用html5+js+css构建前端页面,同时采用自主编写的python后台嵌入分析流程及提供检索入口和可视化入口,并采用d3.js和echarts.js框架等实现可视化。本发明,搭建了一个鲍曼不动杆菌表面多糖抗原信息检索和基因组糖基转移酶注释分析系统。
40.2)数据库构建abausp-db包含的信息有:鲍曼不动杆菌多糖抗原分型列表,各分型对应的多糖抗原结构(snfg格式)、代表性菌株、合成基因簇功能结构图、参考文献信息;鲍曼不动杆菌菌株或基因组对应的kl分型和ocl分型、多糖抗原结构、合成基因簇结构;多糖合成信息,包括糖基转移酶及对应的基因名称、蛋白序列(fasta格式)、糖苷键、供体糖、受体糖、菌株名称、抗原分型和参考文献资料;单糖合成通路信息,及对应的抗原分型、菌株名称列表、合成关键酶基因、合成步骤和单糖合成分类、通路图。
41.gtrdb分型和注释数据库包含的信息有:分型参考数据(kl分型参考序列)、糖基转移酶序列(fsata格式)、供体糖、受体糖和糖苷键。
42.3)数据整合和分析鲍曼不动杆菌多糖抗原检索数据库缺失,目前已发表的鲍曼不动杆菌的各个抗原分型信息需要逐个文献查找,缺乏可供快速检索的统一整合资源。abausp-db数据库将公共数据库(ncbi、gtdb、metacyc)和已发表文献中鲍曼不动杆菌多糖抗原分型信息、多糖抗原结构、合成基因簇、糖苷键、糖基转移酶等信息进行整合,绘制多糖结构图(标注糖基转移酶和对应糖苷键位置)和合成基因簇功能结构图,并进行人工核对,保存到数据库中。abausp-db同时整合已发表鲍曼不动杆菌菌株基因组序列的分型信息和糖基转移酶注释信息。ncbi公共数据库是用户提交基因组序列常用的数据库网站,但是该网站数据存在基因组污染和低质量问题。本数据库鲍曼不动杆菌基因组序列由ncbi下载,经过checkm软件检测基因组完整性、污染率,过滤掉低完整性和高污染的基因组序列。基于基因组序列,采用自主编写的python程序进行分型和糖基转移酶注释,后续将菌株信息与抗原分型、多糖抗原结构、合成基因簇、多糖合成信息整合,保存至abausp-db数据库中。
43.本系统的gtrdb分型和注释数据库,将由公共数据库(ncbi、gtdb)和已发表文献中下载的分型数据(基因和蛋白序列, fasta格式)、糖基转移酶序列(fasta格式),保存至数据库中。
44.4)在线数据浏览、检索和分析功能模块构建
采用html5+js+css构建前端页面,并调用后台python程序及提供检索入口和可视化入口。本实施例的在线工具包括:4-1)鲍曼不动杆菌多糖抗原分型浏览和查询如图2和图3,该工具为用户提供快速的鲍曼不动杆菌多糖抗原分型(kl和ocl分型)和多糖信息查询,用户在工具页面选择具体分型名称,即可反馈该分型对应的多糖抗原结构图(含糖基转移酶位置信息)、合成基因簇功能图、代表性菌株名称、参考文献信息。用户也可通过输入分型名称,直接查询。
45.4-2)鲍曼不动杆菌多糖抗原信息检索如图4,检索页面为用户提供快速鲍曼不动杆菌多糖抗原信息检索功能,分为菌株名称检索、糖基转移酶信息检索和多糖抗原分型检索、选择菌株检索模式,输入鲍曼不动杆菌菌株名称或菌株基因组序列id(ncbi),可快速反馈两个信息表格,其中多糖抗原信息表含抗原分型名称、多糖抗原结构图、合成基因簇功能结构图信息,糖基转移酶注释信息表包含该菌株全基因组序列上的糖基转移酶位置、对应糖苷键、供体糖和受体糖等多糖合成信息;选择多糖抗原分型检索模式,输入分型名称进行检索,可快速返回该分型的多糖抗原信息表;选择单糖、糖基转移酶或糖苷键检索模式,输入单糖、糖基转移酶或糖苷键名称,可快速返回用户信息表,含糖基转移酶名称、糖苷键、供体糖、受体糖、对应菌株列表、抗原分型、参考文献等信息。
46.4-3)合成通路浏览和查询如图5,用户可直接浏览鲍曼不动杆菌长得单糖合成通路列表,每种单糖的合成通路按照步骤呈现,包含对应的多糖抗原分型、菌株列表、合成步骤关键酶和反应信息、合成通路图(含关键步骤合成酶)。或输入单糖名称,可返回该单糖的一条或多条合成通路信息和相关步骤信息。
47.4-4)分型和注释分析如图6中的工作页面,用户可直接上传鲍曼不动杆菌基因组序列(fasta文件),数分钟内返回用户抗原分型结果表格和糖基转移酶注释表格信息,含全基因组序列中所有预测到的糖基转移酶位置、糖基转移酶名称、供体糖、受体糖、糖基转移酶家族。
48.用户也可上传任一细菌基因组序列,进行糖基转移酶注释,数分钟后返回用户糖基转移酶注释表格信息。
49.实施例3本发明的鲍曼不动杆菌基因组分型和糖基转移酶注释分析如下:1)获取公开发表的鲍曼不动杆菌已知k19分型对应的菌株基因组序列,gca_001675265.1(a. baumannii strain 28);2)提交到abausp分析平台的分析(analysis)页面(图6),通过基于python语言自主撰写的分型和注释流程(sergly-pipe)进行鲍曼不动杆菌表面多糖抗原分型分析和糖基转移酶注释分析;sergly-pipe分析流程如下:首先将基因组序列比对至参考分型数据库;第二步,根据比对结果中的覆盖度、相似性阈值和期望值(1e-5)进行结果过滤,确定分型结果为完美匹配(perfect)、高度匹配(high)或低度匹配(low);最后,比对到各分型多糖抗原蛋白序列数据,以找到相似度最高的匹配结果,返回分型结果为kl19(图7);对全基因组进行基因
预测,采用工具为prodigal v2.6.3软件,得到基因注释结果和基因序列;采用blastx将基因序列比对到分型和注释数据库中的糖基转移酶序列,进行同源序列比对,相似性阈值设置为80,期望值为1e-5,进行结果过滤;最终注释结果输出为表格形式,含注释基因名称、注释的糖基转移酶名称、基因组起始位置、对应糖苷键类型、受体糖、供体糖、注释(糖基转移酶、起始转移酶等)、糖基转移酶公共数据库序列号、糖基转移酶家族分类信息(图7)。
50.3)同时将获取的基因组序列,采用已发表的bautype工具(http://bautype.net/acinetobacter_baumannii/home/)和kaptive(https://kaptive-web.erc.monash.edu/)进行分型,与abausp分型结果一致,分型结果均为为kl19,目前无在线预测鲍曼不动杆菌糖基转移酶工具;实施例4本发明的细菌糖基转移酶注释分析如下:1)分别获取公开发表的大肠杆菌基因组序列和注释文件(escherichia coli str. k-12 substr. mg1655, gcf_000005845.2)。
51.2)提交数据至abausp分析平台的分析(analysis)页面(图6),通过基于python语言自主编写的分型和注释流程(sergly-pipe)的注释模块进行全基因组糖基转移酶数据注释;sergly-pipe注释流程如下:对全基因组进行基因预测,采用工具为prodigal (v2.6.3)软件,得到基因注释结果和基因序列;采用blastx将基因序列比对到分型和注释数据库中的糖基转移酶序列,进行同源序列比对,相似性阈值设置为80,期望值为1e-5,进行结果过滤;最终注释结果输出为为表格形式,含注释基因名称、注释的糖基转移酶名称、基因组起始位置、对应糖苷键类型、受体糖、供体糖、注释(糖基转移酶、起始转移酶基因结构和名称预测)、糖基转移酶公共数据库序列号、糖基转移酶家族分类信息。
52.3)比对abausp系统注释结果(图8)和ncbi下载的注释结果(genbank格式),abausp系统在共注释了51个糖基转移酶基因,其中48个基因注释结果一致,同时abausp注释的3个糖基转移酶基因在ncbi注释结果中缺失,这3个糖基转移酶数据为gtrdb数据库中收集自gtdb数据库(2021年发表)的数据。
53.以上所述实施例仅表达了本发明的几种实施方式,其描述较为具体和详细,但并不能因此而理解为对发明范围的限制。应当指出的是,对于本领域的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干变形和改进,这些都属于本发明的保护范围。因此,本发明的保护范围应以所附权利要求为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1