一种基于单分子自适应采样测序的病原微生物和耐药基因的检测方法及系统
1.本发明属于微生物检测技术领域,具体涉及一种基于单分子自适应采样测序的病原微生物和耐药基因的检测方法及系统。
背景技术:
2.感染性疾病为人类常见病,包括细菌、病毒、真菌、寄生虫等多类病原体,可引起包括中枢、血流、呼吸道、局灶感染等多种临床感染类型,严重威胁人类健康。据世界卫生组织(world health organization,who)统计,下呼吸道感染仍是世界上最致命的感染性疾病。感染性疾病发病率和死亡率的高低取决于感染部位,病原体和宿主等因素,尽早确定致病菌,使用抗生素可显著提高生存率,降低死亡率。近几年来,病原菌的耐药率明显升高,多重耐药菌和产广谱β-内酰胺酶(esbls)细菌不断增多,给临床治疗带来极大困难。因此,明确的病原学诊断和病原耐药性信息,不仅可以指导下呼吸道感染的治疗还可以减少病原体耐药现象和药物不良反应、缩短病程、降低医疗费用、合理分配医疗资源。
3.快速准确的病原微生物诊断可以实现量身定制的治疗,减少广谱抗生素的过度使用。临床微生物检验室使用的金标准的培养和药敏试验太慢,典型的周转时间为48-72h,并且临床敏感性低。分子方法可能有助于克服培养的局限性,通过在几个小时内识别病原体及其抗生素耐药性特征,实现早期靶向治疗并支持抗生素管理。尽管核酸扩增试验(包括pcr)快速且高度特异性/敏感性,但多重扩增存在局限性,并且还需要不断更新基于pcr的方法,以包括新出现的耐药基因和突变。
4.基于宏基因组测序的方法有潜力克服培养和pcr的缺点,方法是将速度与对临床样本所有存在微生物的全面覆盖相结合。但是呼吸道标本对宏基因组测序来说是一个困难的挑战,因为病原体负载量可变,存在共生呼吸道菌群,并且存在高比例的宿主与病原体核酸(痰中高达105:1)。相关技术曾采用nanopore测序检测两名细菌性肺炎患者的感染性疾病病原体及其携带耐药基因,没有宿主细胞/dna缺失,导致绝大多数测序 reads是人类来源的,只有一个和两个测序reads分别与感染病原体铜绿假单胞菌和金黄色葡萄球菌对齐。但是通过引入宿主dna缺失后,需要对特定样本进行调整,通常需要许多额外的步骤,会增加测序的时长和成本且在复杂的高宿主临床样本中仍表现不佳,需要更好的方法。
技术实现要素:
5.本发明旨在至少解决上述现有技术中存在的技术问题之一。为此,本发明提出一种基于单分子自适应采样测序的病原微生物检测方法,能够在4.5小时内识别病原体和抗生素耐药基因,并且极大的降低检测成本。
6.本发明还提出一种基于单分子自适应采样测序的数据分析系统。
7.在本发明的第一方面,提出了一种基于单分子自适应采样测序的病原微生物检测方法,所述方法包括如下步骤:
8.s1、使用单分子自适应采样测序方法获得样本原始序列集,将样本原始序列集进行质控得到高质量序列集;
9.s2、使用argpore2分析软件对所述高质量序列集进行分析,即得样本的病原微生物和耐药基因检测结果。
10.所述单分子自适应采样测序方法还包括将宿主序列进行过滤的步骤,所述步骤为以人类基因组为参考,以“delete”的方式来耗竭样本中的宿主dna,可以实时消除宿主 dna序列,检测微生物dna序列。
11.在本发明的一些实施方式中,所述宿主dna序列为人源序列。
12.在本发明的一些实施方式中,所述人源序列为人类参考基因组gca_000001405.28_grch38.p13。
13.在本发明的一些实施方式中,所述样本原始序列集为去除宿主序列的原始序列集。
14.在本发明的一些实施方式中,所述质控为过滤小于150bp的短序列,和剔除碱基质量分数低于7的序列。
15.在本发明的一些实施方式中,所述病原微生物选自细菌、真菌或者病毒中的至少一种。
16.在本发明的一些实施方式中,argpore2分析软件为自主构建用于临床样本单分子测序数据的分析软件。
17.在本发明的一些实施方式中,所述自主构建的argpore2分析软件是将argpore2 分析软件与病原微生物数据库和耐药基因数据库进行有机结合得到的。
18.在本发明的一些实施方式中,所述病原微生物数据库包括病原微生物参考序列数据库和病原微生物注释数据库。
19.在本发明的一些实施方式中,所述病原微生物数据库包括细菌数据库、真菌数据库和/或病毒数据库。
20.在本发明的一些实施方式中,所述病原微生物检测结果包括物种信息、物种reads、 reads占该物种比例、覆盖度、深度、耐药基因。
21.在本发明的一些实施方式中,所述耐药基因检测结果包括耐药基因信息、耐药基因的归属物种信息或携带耐药基因的质粒信息。
22.在本发明的一些实施方式中,所述分析包括使用argpore2分析软件将所述高质量序列集比对到病原微生物数据库系统参考基因上生成第一数据集(微生物物种鉴定结果);使用argpore2分析软件将所述高质量序列集比对到耐药基因数据库系统参考基因上生成第二数据集(耐药基因检测结果);将第一序列集和第二序列集进行关联生成第三序列集(携带耐药基因的物种及携带耐药基因)。
23.在本发明的一些实施方式中,所述方法还包括在测序前进行文库的构建步骤。
24.在本发明的第二方面,提供了一种基于单分子自适应采样测序的数据分析系统,所述系统包括:
25.数据库存储模块,用于存储病原微生物数据库和耐药基因数据库;
26.病原微生物鉴定模块,分别与数据输入模块和数据库存储模块连接,用于将所述序列集与所述病原微生物数据库进行比对,得到相应的病原微生物信息作为病原微生物鉴
定结果;
27.耐药基因鉴定模块,分别与数据输入模块和数据库存储模块连接,将所述序列集与耐药基因数据库的核酸序列信息进行比对,获得耐药基因的鉴定结果;
28.结果输出模块,与所述病原微生物鉴定模块和所述耐药基因鉴定模块相连接,用于输出病原微生物鉴定结果和耐药基因检测结果。
29.在本发明的一些实施方式中,所述数据分析系统为自主构建的argpore2分析软件。
30.根据本发明的一些实施方式,至少具有以下有益效果:本发明方案基于单分子自适应采样测序的病原微生物检测方法,通过本发明方案的方法,无需人工对样本进行复杂前处理除去样本中高含量的宿主dna,不会对样本中的微生物组造成破坏,可以4.5小时完成样本病原体的识别和抗生素耐药基因的检测,方法简单,可以使用一张芯片同时检测≥5个样本,节约了检测时间,节省了检测成本,可以广泛使用。
附图说明
31.下面结合附图和实施例对本发明做进一步的说明,其中:
32.图1为本发明实施例1中的技术流程图;
33.图2为本发明实施例1中的b1样本物种鉴定结果图;
34.图3为本发明实施例1中的b1样本耐药基因预测信息结果图;
35.图4为本发明实施例2中的b2-b11样本病原菌检出和耐药基因预测信息结果图。
具体实施方式
36.以下将结合实施例对本发明的构思及产生的技术效果进行清楚、完整地描述,以充分地理解本发明的目的、特征和效果。显然,所描述的实施例只是本发明的一部分实施例,而不是全部实施例,基于本发明的实施例,本领域的技术人员在不付出创造性劳动的前提下所获得的其他实施例,均属于本发明保护的范围。
37.实施例1基于单分子自适应采样测序的病原微生物检测方法,对支气管肺泡灌洗液微生物样本进行检测鉴定
38.样本的收集:从医院收集11例呼吸道感染患者肺泡灌洗液标本,命名为b1-b11,临床金标准培养验证得到b1-b11标本的病原微生物如表1所示。技术流程图如图1所示。
39.表1
40.样本编号病原菌培养鉴定结果b1鲍曼不动杆菌、肺炎克雷伯菌b2肺炎链球菌b3铜绿假单胞菌b4金黄色葡萄球菌b5铜绿假单胞菌b6流感嗜血杆菌b7肺炎链球菌b8大肠杆菌
b9大肠杆菌b10铜绿假单胞菌、大肠杆菌b11铜绿假单胞菌、金黄色葡萄球菌
41.1、单样本的检测
42.(1)样本dna提取
43.使用zymobiomics(zymoresearch)quick-dnahmwmagbeadkit,按照试剂盒的使用说明书,提取收集的b1肺泡灌洗液标本的dna。将提取得到的dna分别采用quabit4.0荧光计(thermofisherscientific,us)结合高灵敏度双链dna分析试剂盒(thermofisher,q32851)进行浓度检测。使用tapestation4150(agilenttechnologiesinc,usa)自动电泳平台评估提取dna质量和片段大小。
44.(2)测序文库的构建
45.b1样本提取后的核酸样本按照规范流程进行建库。建库方案选取自牛津纳米孔公司提供的1drapidsequencinggdnagprotocol,取1.5μgdna,依据nanopore提供的建库说明书,使用rapidsequencingkit(sqk-rad004)制备测序文库。在文库构建的cleanup步骤,选择使用长片段缓冲液(longfragmentbuffer,lfb)来富集长的dna片段。
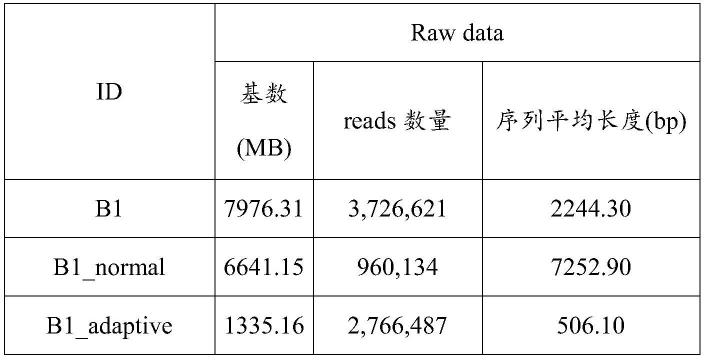
46.(3)测序
47.使用ontgridion测序平台进行单分子自适应采样测序。取建库后样本加载到r9.4flowcell测序芯片。为检测单分子自适应采样测序的病原微生物检测方法的优势,采用单分子自适应采样测序(adaptivesampling)作为实验组(b1_adaptive),常规单分子纳米孔测序对照组(b1_normal),通过设定测序通道1-256为自适应采样通道,257-512通道用于单分子纳米孔测序,在同一测序芯片上同时进行单样本b1的自适应测序和标准测序。对于设定为自适应采样的1-256通道,提供人类参考基因组gca_000001405.28_grch38.p13用作参考序列,通过选择“deplete”启用宿主序列耗竭,从而可以在测序进行时实时动态地终止对人类基因片段的测序,耗竭人类序列。测序时间2小时后收集数据后。在2小时测序的基础上,再延长adaptivesampling测序和正常nanopore测序至72h。
48.(4)生物信息学分析
49.使用gridion测序平台自带guppybasecallingsoftware(oxfordnanoporetechnologies)在测序进行中实时将电信号转换为碱基信号,测序与实时碱基识别,检测运行状态,得到fastq格式的序列信息,生成数据集1,即为原始宏基因组序列集。利用qc_for_nanopore对数据集1质控,生成数据集2:过滤小于150bp的短序列,以及碱基质量分数低于7的序列将被剔除,质控后的数据集为数据集2,即高质量序列集;
50.质控后的数据集2使用argpore2分析软件进行和自建病原数据库、耐药基因数据库的分析流程,argpore2软件快速将dna序列比对到病原微生物数据库系统参考基因组上,生成数据集3;argpore2软件快速将dna序列比对到耐药基因数据库系统参考基因上,生成数据集4;argpore2软件将生成数据集3和生成数据集4中匹配到的序列比对结果提取出来,形成数据集5,即关联耐药基因和病原微生物,获得鉴定出的病原微生物的耐药性。
51.argpore2数据分析软件的具体信息和参数,使用方法,自建病原数据库、耐药基因数据库等,可从github(https://github.com/sustc-xylab/argpore2上浏览argpore2的完整手册。
52.b1标本的基本测序数据如表2-表4所示;样本物种鉴定结果如图2所示;耐药基因预测信息如表5和图3所示。
53.表2
[0054][0055]
表3
[0056]
表4
[0057][0058]
表5
[0059]
[0060][0061]
检测结果如表2-5和图2-3所示,从表2中可以看出,正常测序产生的微生物组数 据只占测序数据的0.39%,而nas测序产生的微生物组数据占测序数据的16.44%,微 生物的测序量提高40多倍,而nas测序数据总量只有正常测序总量的五分之一,从表 3、表4和表5中可以看出,nas测序2h的可以检测出病原菌上的耐药基因,而正常 nanopore测序72h产生的微生物组数据检测不到病原菌上的耐药基因;从图2中可以看 出,鲍曼不动杆菌和肺炎克雷伯菌在两种测序方式中均有检出,且都属于优势菌株。但 nas测序2h检出匹配鲍曼不动杆菌和肺炎克雷伯菌的reads数分别为498和137,远高 于正常nanopore测序72h检出的48和22。从图3中可以看出,nas测序2h可以准确 测定得到病原菌,同时可以检测出病原菌上的耐药基因,而正常nanopore测序2h收集 得到的数据极少无法用于分析。结果表明,本发明方案基于单分子自适应采样测序,通 过提供人类基因组为参考,以“delete”的方式来耗竭样本中的宿主dna,增强对微生 物序列的富集产生的数据,测序长度较长,集中平均长度在2k以上,且nas测序在1h 就可以准确检测到2种病原体的reads数,与金标准得到的数值一致,所以,2小时的 nas测序结果就可用于随后的数据分析。本发明方法的敏感性和特异性为100%。
[0062]
实施例2基于单分子自适应采样测序的病原微生物检测方法,对10例支气管肺泡灌洗液微生物样本进行检测鉴定
[0063]
(1)样本dna提取
[0064]
使用zymobiomics(zymo research)quick-dna hmw magbead kit,按照试剂盒的
使用说明书,提取同实施例1一致的收集的b2-b11肺泡灌洗液标本的dna。将提取得到的dna分别采用quabit4.0荧光计(thermofisherscientific,us)结合高灵敏度双链dna分析试剂盒(thermofisher,q32851)进行浓度检测。使用tapestation4150(agilenttechnologiesinc,usa)自动电泳平台评估提取dna质量和片段大小。
[0065]
(2)测序文库的构建
[0066]
建库方案选取自牛津纳米孔公司提供的1drapidsequencinggdna-barcodinggprotocol,采用添加测序条码的方式,以五个样本为一组混合建库(b2-b6为第一组,b7-b11为第二组)。每样本取1.5μgdna,依据nanopore提供的混合建库说明书,使用rapidbarcodingsequencingkit(sqkrbk004)制备测序文库。在文库构建的cleanup步骤,选择使用长片段缓冲液(longfragmentbuffer,lfb)来富集长的dna片段。
[0067]
(3)测序
[0068]
取50fmol建库后第一组样本和第二组分别加载到两张r9.4flowcell测序芯片,使用ontgridion测序平台进行测序。adaptivesampling测序是使用minknow软件应用,提供人类参考基因组gca_000001405.28_grch38.p13用作参考序列,通过选择“deplete”启用宿主序列耗竭,所有测序通道均设置为自适应采样通道,测序时间2小时。
[0069]
(4)生物信息学分析
[0070]
使用gridion测序平台自带guppybasecallingsoftware(oxfordnanoporetechnologies)在测序进行中实时将电信号转换为碱基信号,测序与实时碱基识别,检测运行状态,得到fastq格式的序列信息,生成数据集1,即为原始宏基因组序列集。利用qc_for_nanopore对数据集1质控,生成数据集2:过滤小于150bp的短序列,以及碱基质量分数低于7的序列将被剔除,质控后的数据集为数据集2,即高质量序列集;
[0071]
质控后的数据集2使用argpore2分析软件进行和自建病原数据库、耐药基因数据库的分析流程,argpore2软件快速将dna序列比对到病原微生物数据库系统参考基因组上,生成数据集3;argpore2软件快速将dna序列比对到耐药基因数据库系统参考基因上,生成数据集4;argpore2软件将生成数据集3和生成数据集4中匹配到的序列比对结果提取出来,形成数据集5,即关联耐药基因和病原微生物,获得鉴定出的病原微生物的耐药性。
[0072]
argpore2数据分析软件的具体信息和参数,使用方法,自建病原数据库、耐药基因数据库等,可从github(https://github.com/sustc-xylab/argpore2上浏览argpore2的完整手册。
[0073]
b2-b11样本病原菌检出和耐药基因预测信息如图4和表6所示。
[0074]
表6
[0075]
[0076][0077]
检测结果如表6和图4所示,从表6和图4中可以看出,本发明方案基于单分子自适应采样测序,去除了宿主(人)序列,造成芯片检测能力的空置,可以进行多样本的同一检测,一张芯片上进行五个样本的检测,在测序2h就可以准确检测到病原体的reads 数与金标准得到的数值一致。
[0078]
三代单分子长读长测序的自适应采样(adaptive sampling)概念(也可以称为选择性测序,selective sequencing),是一种替代实验室耗竭或富集的方法,它代表了软件控制富集的一种形式。自适应采样通过先进的生物信息学管道进行操作,在测序过程中,每0.4秒将单个分子(约200-400个碱基对)的核苷酸与用户指定的参考文件进行比较。这种动态方法有广泛的应用,可以选择性地丰富感兴趣的目标序列,并在测序过程中拒绝不需要的序列。这对于临床样本可能具有重要意义,尤其是在预期人类dna水平较高的情况下(例如,低丰度病毒和/或细菌病原体与》99%的宿主分子混合)。首先,通过提供宿主参考基因组(例如,人类)并拒绝宿主序列,可以实时完成宿主基因组dna 序列的消耗。由此产生的序
列数据对整个微生物群体进行了丰富,然后对其进行挖掘,以便对低丰度病原体进行监测。主要优点是,不会延长整个测序运行时间,不会增加额外的测序成本。同时,还可以结合湿实验耗竭或富集的方法使用。本发明方案通过基于单分子自适应采样测序的病原微生物检测方法,只需4.5h即可实现整个病原微生物的检测流程,包括临床样本dna提取(1小时),测序文库自备(1小时),测序(2小时) 和实时数据分析(0.5小时)。即实现了在4.5小时内识别病原体和抗生素耐药基因。在复杂的(病原体负载量可变,存在共生菌群,存在高比例的宿主dna等)临床样本中有良好的表现。
[0079]
综上所述,本发明方案基于单分子自适应采样测序的病原微生物检测系统,与常规单分子nanopore测序相比,存在以下优势:
[0080]
(1)单分子自适应采样测序识别病原体和耐药基因的时间大幅度减少:利用 nanopore测序快速鉴定感染性疾病病原体及其携带耐药基因,需要24h或者更长的时间才可以收集到足够数目的reads用于鉴定病原体,并且很可能检测不到病原菌上的耐药基因。由于nas测序对微生物序列的富集,本发明在4.5h内就可以测到病原菌和耐药基因。
[0081]
(2)如本发明实施例1所示,单分子自适应采样测序在2h监测到2种病原体的reads 数,高于进行nanopore测序72h监测到的reads数。所以,2小时的nas测序结果就可用于随后的数据分析。因此,整个流程(建技术框架图)可以在4.5小时得到结果,比金标准实验室传统培养36-72h,二代测序技术检测的24h,以及相关技术的nanopore 测序的24h,有极大的进步,特别适用于急危重症和疑难感染的诊断。
[0082]
(3)本发明下游生物信息学分析流程采用argpore2,操作简便友好:argpore2 pipeline集合主流比对分类软件,物种数据库,耐药基因数据库,用户只需要提供下机数据,不用做其他额外处理,半个小时内就可以得到标本的物种鉴定信息和耐药基因预测信息。
[0083]
(4)与nanopore测序检测成本相比,本发明检测成本的大幅度降低:nanopore测序芯片单价8000-9000元左右,加上提取,建库等试剂,单个样本的检测成本在9500 元。因为单分子自适应采样测序在测序过程中去除宿主基因序列,造成芯片检测能力的空置,可以进行多样本的同一检测,在本专利中,本发明在一张芯片上进行五个balf 样本的检测,获得良好的检测结果。至此,本发明将每个样本的检测成本降低到2000 元左右,另外多样本的同时检测极大增加临床样本的检测效率。我们推测,对于低宿主背景(如粪便,尿液,脓液)的临床样本,可以在一张芯片上进行更多样本的检测,从而再次降低检测成本。
[0084]
上面结合附图对本发明实施例作了详细说明,但是本发明不限于上述实施例,在所属技术领域普通技术人员所具备的知识范围内,还可以在不脱离本发明宗旨的前提下作出各种变化。此外,在不冲突的情况下,本发明的实施例及实施例中的特征可以相互组合。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1