一种基于人体全基因组基因型预测个体表型的方法和设备与流程
1.本发明属于表型预测技术领域,具体涉及一种基于人体全基因组基因型预测个体表型的方法和设备。
背景技术:
2.人类的许多性状和疾病都受到大量遗传位点的控制,从理论上讲,利用一个个体全基因组的基因型数据可以较为准确地估计该个体某种遗传性状的表现或者某个遗传疾病的发病风险。目前,高通量测序与全基因组芯片的快速发展使得个体的全基因组数据的获取成本越来越低,相应的基因组数据处理软件和算法的发展也使得数据分析速度越来越快。然而,现有的算法仍无法很好地完成依据基因型预测表型或疾病风险这一任务,具体如下:
3.现有的算法直接以基因型作为输入,以形状或疾病作为输出,并建立基因变异与性状或疾病的关联,但这些关联结果仍然缺乏生物学机制上的解释,也难以预测疾病风险。造成这一现象的原因是现有的算法均采用线性模型进行关联分析,而基因间存在复杂的非线性相互作用。此外,基因位点间存在连锁不平衡,人群之间也存在显著的基因差异,从而导致在这一人群中获取的结果或预测模型难以在另一人群中得到应用。因此,虽然目前积累了大量的全基因组基因分型数据,但仍缺乏能够有效利用这些数据的方法。
技术实现要素:
4.本发明的目的是提供一种基于人体全基因组基因型预测个体表型的方法和设备,用以解决现有技术中存在的缺乏能够有效利用全基因组基因分型数据的方法的技术问题。
5.为了实现上述目的,本发明采用以下技术方案:
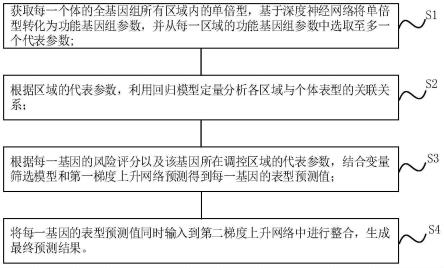
6.第一方面,本发明提供一种基于人体全基因组基因型预测个体表型的方法,包括:
7.获取每一个体的全基因组所有区域内的单倍型,基于深度神经网络将单倍型转化为功能基因组参数,并从每一区域的功能基因组参数中选取至多一个代表参数;
8.根据区域的代表参数,利用回归模型定量分析各区域与个体表型的关联关系;
9.根据每一基因的风险评分以及该基因所在调控区域的代表参数,结合变量筛选模型和第一梯度上升网络预测得到每一基因的表型预测值;
10.将每一基因的表型预测值同时输入到第二梯度上升网络中进行整合,生成最终预测结果。
11.基于上述公开的内容,本发明不直接利用基因型数据,而通过深度神经网络将基因型(单倍型)转化为功能基因组参数,由于考虑到基因间存在复杂的相互作用关系,通过从功能基因组参数选取代表参数,并利用区域的代表参数分析各区域与个体表型的关联以及单基因的预测值,从而在单基因水平上整合来自各功能基因组改变的信息,避免了孤立分析每一基因位点的线性关联的局限性,最后通过梯度上升网络对整合所有基因的预测值,以充分反映基因间的非线性关系,从而提高预测的准确度,对于各种个体表型的预测具
有重要意义。
12.在一种可能的设计中,获取每一个体的全基因组所有区域内的单倍型,基于深度神经网络将单倍型转化为功能基因组参数,并从每一区域的功能基因组参数中选取至多一个代表参数,包括:
13.对每一个体的全基因组进行区域分割,获取每一区域内的单倍型以及对应的碱基序列;
14.基于深度神经网络将每条碱基序列转化为对应的多个功能基因组参数;
15.基于精细定位算法从每一区域的多个功能基因组参数中选取至多一个代表参数。
16.基于上述公开的内容,本发明通过对全基因组进行区域分割,以使得分割后得到的碱基序列满足神经网络数据输入长度的要求,通过将基因型数据转化为单倍型数据,能够节约计算资源,通过深度神经网络将每条碱基序列转化为多个功能基因组参数,以便将基因变异造成的功能基因组改变定量体现,通过选取代表参数,能够提高模型预测的准确度。
17.在一种可能的设计中,对每一个体的全基因组进行区域分割,获取每一区域内的单倍型以及对应的碱基序列,包括:
18.按照预设长度将每一个体的全基因组分割为多个区域,并保证每一区域尽可能完整覆盖全基因组的功能元件;
19.根据非等位基因频率对基因型进行筛选,将筛选后的基因型转化为单倍型,并获取每一单倍型对应的碱基序列。
20.在一种可能的设计中,基于精细定位算法从每一区域的多个功能基因组参数中选取至多一个代表参数之前,所述方法包括:
21.对多个功能基因组参数进行整合,得到每条碱基序列的多个类别分数,并对多个类别分数进行处理;
22.对处理后的每一类别分数按照加性遗传模型、隐形遗传模型和显性遗传模型进行转化,生成对应的中介参数,以便精细定位算法从每一区域的中介参数中选取至多一个代表参数。
23.基于上述公开的内容,本发明通过将功能基因组参数进行整合,得到多个类别分数,从而可以在降低数据量的同时提高数据信息量。
24.在一种可能的设计中,根据区域的代表参数,利用回归模型定量分析各区域与个体表型的关联关系,包括:
25.在每一代表参数的区域内,基于线性回归模型定量分析该区域与个体表型的关联关系;
26.利用精细定位算法从内部存在连锁不平衡的区域中选出起效区域;
27.获取每一区域的生物学意义注释,基于广义线性回归模型定量分析影响个体表型的起效区域是否更多地与某一生物学意义注释相关。
28.基于上述公开的内容,能够确定每一区域与个体表型的关联关系以及影响个体表型的起效区域是否更多地与某一生物学意义注释相关,从而能够重点关注关联关系大的区域或生物学意义注释,提高数据分析效率。
29.在一种可能的设计中,根据每一基因的风险评分以及该基因所在调控区域的代表
参数,结合变量筛选模型和第一梯度上升网络预测得到每一基因的表型预测值,包括:
30.对每一基因预处理后输入到变量筛选模型中进行计算,根据输出的基因位点权重,计算该基因在的外显子多基因风险评分;
31.将外显子多基因风险评分与该基因所在调控区域内的所有代表参数输入到第一梯度上升网络中,得到该基因的表型预测值。
32.在一种可能的设计中,对每一基因预处理后输入到变量筛选模型中进行计算,包括:
33.对每一基因进行预处理,将预处理后的多个基因位点输入到snpnet算法模型中,进行lasso批量筛选迭代回归分析;
34.在回归分析中加入协变量,且不对协变量的参数进行lasso缩放,以输出去除连锁不平衡的基因位点权重。
35.在一种可能的设计中,将外显子多基因风险评分与该基因所在调控区域内的所有代表参数输入到第一梯度上升网络中,得到该基因的表型预测值,包括:
36.根据功能基因组注释,预先设定每一基因的调控区域;
37.将外显子多基因风险评分与该基因在调控区域内的所有代表参数作为模型输入,并将待预测形状的回归模型的残差作为模型预测标签,利用第一梯度上升网络预测得到该基因的表型预测值。
38.在一种可能的设计中,将每一基因的表型预测值同时输入到第二梯度上升网络中进行整合,生成最终预测结果,包括:
39.将每一基因的表型预测值作为模型输入,并将待预测形状的真实值作为预测标签,利用第二梯度上升网络得到个体表型的最终预测结果。
40.第二方面,本发明提供一种基于人体全基因组基因型预测个体表型的装置,包括:
41.参数转化模块,用于获取每一个体的全基因组所有区域内的单倍型,基于深度神经网络将单倍型转化为功能基因组参数,并从每一区域的功能基因组参数中选取至多一个代表参数;
42.关联分析模块,用于根据区域的代表参数,利用回归模型定量分析各区域与个体表型的关联关系;
43.第一预测模块,用于根据每一基因的风险评分以及该基因所在调控区域的代表参数,结合变量筛选模型和第一梯度上升网络预测得到每一基因的表型预测值;
44.第二预测模块,用于将每一基因的表型预测值同时输入到第二梯度上升网络中进行整合,生成最终预测结果。
45.第三方面,本发明提供一种计算机设备,包括依次通信相连的存储器、处理器和收发器,其中,所述存储器用于存储计算机程序,所述收发器用于收发消息,所述处理器用于读取所述计算机程序,执行如第一方面任意一种可能的设计中所述的方法。
46.第四方面,本发明提供一种计算机可读存储介质,所述计算机可读存储介质上存储有指令,当所述指令在计算机上运行时,执行如第一方面任意一种可能的设计中所述的方法。
47.第五方面,本发明提供一种包含指令的计算机程序产品,当所述指令在计算机上运行时,使所述计算机执行如第一方面任意一种可能的设计中所述的方法。
附图说明
48.图1为本发明提供的基于人体全基因组基因型预测个体表型的方法的流程图;
49.图2为本发明提供的步骤s1的流程图;
50.图3为本发明提供的步骤s2的流程图;
51.图4为本发明提供的步骤s3的流程图。
具体实施方式
52.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将结合附图和实施例或现有技术的描述对本发明作简单地介绍,显而易见地,下面关于附图结构的描述仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。在此需要说明的是,对于这些实施例方式的说明用于帮助理解本发明,但并不构成对本发明的限定。
53.实施例
54.为了克服现有技术中存在的缺乏能够有效利用全基因组基因分型数据的方法的技术问题,本实施例提供一种基于人体全基因组基因型预测个体表型的方法,该方法在单基因水平上整合来自各功能基因组改变的信息,避免了孤立分析每一基因位点的线性关联的局限性,最后通过梯度上升网络对整合所有基因的预测值,以充分反映基因间的非线性关系,从而提高预测的准确度,对于各种个体表型的预测具有重要意义。
55.其中,需要说明的是,本技术实施例提供的方法可应用于各种人体表型的预测场景中,包括但不限于身高、体重等,所采用的基因型数据的采样人群也可以多种多样,为了便于说明,本实施例以使用欧洲人群全基因组芯片分型数据构建预测个人身高水平的模型为例,对本实施例进行详细说明。当然,可以理解的是,本领域技术人员基于下文描述,完全有动机将本实施例应用于其他人群上的数据,构建预测其它性状的模型,因此其他实施例也在本发明的保护范围内。为便于理解,本实施例具体说明了可使用的一些软件工具、算法模型及算法参数,当然可以理解的是,将这些软件工具、算法模型及算法参数替换为其他类似的工具等,并不代表对本实施例作出的创造性改变,均在发明的保护范围内。
56.下面将对本技术实施例提供的基于人体全基因组基因型预测个体表型的方法进行详细说明。
57.如图1-图4所示,是本技术实施例提供的方法的流程图,所述方法包括但不限于由步骤s1~s4实现:
58.步骤s1.获取每一个体的全基因组所有区域内的单倍型,基于深度神经网络将单倍型转化为功能基因组参数,并从每一区域的功能基因组参数中选取至多一个代表参数;
59.其中,优选的,本实施例中的深度神经网络采用sei网络模型。
60.在步骤s1中,获取每一个体的全基因组所有区域内的单倍型,基于深度神经网络将单倍型转化为功能基因组参数,并从每一区域的功能基因组参数中选取至多一个代表参数,包括:
61.步骤s11.对每一个体的全基因组进行区域分割,获取每一区域内的单倍型以及对应的碱基序列,具体包括:
62.步骤s111.按照预设长度将每一个体的全基因组分割为多个区域,并保证每一区
域尽可能完整覆盖全基因组的功能元件;
63.其中,需要说明的是,当采用sei网络模型时,由于sei模型指定的输入长度为4096bp,因此本实施例将全基因组分割为若干个4096bp长度的区域,并保证这些区域尽量完整覆盖所有基因组功能元件。具体的,首先,基于epimap中222个人类组织的染色质状态注释,对每一个组织,将所有至少在一半样本中被注释为“转录起始位点(tss)转录区域(tx),增强子(enhancer),启动子 (promoter)”的染色体区域标注为活性区域,将所有组织的活性区域取并集,去除hg38基因组中被注释为genomic gap的区域。然后,对这一系列活性区域,若长度小于4096bp,则以活性区域为中心将该区域定义为一个4096bp的区域,若长度大于4096bp,则从区域中点开始向外逐步划定一4096bp长度的区域。最后,使用不重复的4096bp区块覆盖剩余基因组区域。
64.步骤s112.根据非等位基因频率对基因型进行筛选,将筛选后的基因型转化为单倍型,并获取每一单倍型对应的碱基序列。
65.其中,需要说明的是,本实施例假定预先已获取了所有个体的.bgen格式基因型数据,且已进行单倍型分型。其中,根据非等位基因频率对基因型进行筛选,具体包括:对于每一个区域,首先使用bgenix工具从.bgen文件中获取所有位于该区域的变异,去掉质控参数info《0.8,hardy-wenberg检验p值小于10-6
,变异基因型总数小于10的变异,再去掉在该区域内丢失超过50%变异的个体,当然,可以理解的是,上述取值的前提是本实施例采用的人群样本和拟预测的形状类型,若应用其他样本或其他形状时,则可根据需求对上述取值进行调整,此处不做限定。
66.优选的,在对基因型进行筛选后,还可使用plink将上述筛选后的.bgen数据转化为.haps数据,并通过awk及datamash获取所有单倍型的.vcf数据以及所有个体的单倍型基因型。通过awk及bcftools获取所有单倍型对应的碱基序列,并以一个fasta文件储存。
67.步骤s12.基于深度神经网络将每条碱基序列转化为对应的多个功能基因组参数;
68.在步骤s12中,通过将fasta文件储存输入sei算法网络中,其中数据读入步骤使用selene工具包提供的fasta格式处理函数。其中,sei算法网络在进行数据转化时,包含三个步骤:1)具有线性和非线性双重路径的卷积网络用以编码序列信息,2)残差扩张卷积层,3)空间基函数变换和输出层。通过这三个步骤,sei网络可计算出每条碱基序列对应的21906个功能基因组参数。
69.优选的,在获得功能基因组参数之后,所述方法包括:
70.1)对多个功能基因组参数进行整合,得到每条碱基序列的多个类别分数,并对多个类别分数进行处理;
71.具体的,sei计算出每条碱基序列对应的21906个功能基因组参数,得到一个功能基因组参数矩阵fn×
21906
。随后,基于该矩阵按如下公式计算40个序列类别分数矩阵score1,计算公式如下:
72.score1=f
×
p
21906
×
40
;
ꢀꢀꢀꢀ
(1)
73.其中,p是一个基于umap降维及聚类分析得到的投射矩阵,它根据每一个碱基序列的功能基因组参数计算出该碱基序列属于某一个序列类别的可能性(共 40种序列类别),以score1表示。最终输出的矩阵score1为n行40列,即:每一条碱基序列均对应40个序列类别分数。那么,这个分数越大,说明碱基序列属于这个序列类别的可能性越大。
74.优选的,在对一个区域内的所有碱基序列以及这个序列的hg38参考序列均完成sei预测后,所述方法还包括,使用如下公式完成分数的标准化:
[0075][0076]
其中,score
histone
表示组蛋白标志参数,ref表示每一区域参考序列,表示每一区域内参考序列对应的所有组蛋白标志参数之和;
[0077]
优选的,对标准化的类别分数,所述方法还包括:
[0078]
剔除序列类别分数矩阵score2中不符合预设条件的分数,例如类别为无功能基因组信号(low signal),异染色质(heterochromatin)及染色质抑制区域(polycomb)的类别分数,并分别计算剩余的每一序列类别分数的变异系数cv,计算公式如下:
[0079][0080]
其中,se表示每一序列类别分数的标准差,mean表示每一序列类别分数的标准差的平均数。
[0081]
2)对处理后的每一类别分数按照加性遗传模型、隐性遗传模型和显性遗传模型进行转化,生成对应的中介参数,以便精细定位算法从每一区域的中介参数中选取至多一个代表参数。
[0082]
具体的,步骤2)包括:
[0083]
选取变异系数cv最大的前10个序列类别分数,分别按照加性遗传模型、隐性遗传模型和显性遗传模型进行转化,得到30个中介参数,当然,可以的理解的是,上述取值可根据具体需求进行调整,此处不做限定。
[0084]
具体的,按照加性遗传模型进行转化包括:取两个单倍型对应类别分数的平均值;按照隐性遗传模型进行转化包括:取两个单倍型对应类别分数的最小值;按照显性遗传模型进行转化包括:取两个单倍型对应类别分数的最大值。
[0085]
步骤s13.基于精细定位算法从每一区域的多个功能基因组参数中选取至多一个代表参数。
[0086]
优选的,本实施例中的精细定位算法采用susie算法,利用susie算法每一区域的多个功能基因组参数中选取至多一个代表参数,包括:
[0087]
使用susie算法对30个中介参数进行变量筛选,参数设定均选择默认值。
[0088]
其中,结果变量选为如下回归模型的残差:
[0089]
height~age+sex+center+pc
1-10
+covriate;
ꢀꢀꢀꢀꢀ
(4)
[0090]
其中,height,age,sex,center,pc
1-10
,covriate分别表示身高,年龄,性别,样本来源,基因主成分及其他用户指定的协变量。susie对于一个包含大量线性相关自变量的回归模型,将自变量分为若干集合,并估算每一个集合至少包含一个真正起效的自变量的后验概率。在每一个集合内,susie 再次估算每一个自变量是真正起效自变量的后验概率。该过程的数学表达如下:
[0091]
y=xb+ε,ε~nn(0,δ2in),b=bγ,γ=mult(1,π),b~n1(0,δ2)
[0092]
;(5)
[0093]
其中,b表示由数据估算的各个自变量的系数,b表示这些自变量真实起效的系数,γ表示一个0/1变量,susie的任务即估计γ的每一项为1的后验概率。
[0094]
从susie结果中,选择包含概率最大的一组中介参数,再选择其中后验概率最大的一个中介参数。假如所有组的包含概率均小于0.5,则跳过这一区域,不进行分析。将选出的一个中介参数作为该区域的代表参数用于后续分析。
[0095]
步骤s2.根据区域的代表参数,利用回归模型定量分析各区域与个体表型的关联关系;
[0096]
在步骤s2中,根据区域的代表参数,利用回归模型定量分析各区域与个体表型的关联关系,包括:
[0097]
步骤s21.在每一代表参数的区域内,基于线性回归模型定量分析该区域与个体表型的关联关系,该线性回归模型表达式如下:
[0098]
height~age+sex+center+pc
1-10
+covriate;
ꢀꢀꢀꢀꢀꢀ
(6)
[0099]
其中,height,age,sex,center,pc
1-10
,covriate分别表示身高,年龄,性别,样本来源,基因主成分及其他用户指定的协变量,该回归模型中代表参数对应的回归系数及p值作为该区域与表型的关联结果。
[0100]
步骤s22.利用精细定位算法从内部存在连锁不平衡的区域中选出起效区域;
[0101]
具体的,使用欧洲人hg38版本千人基因组数据进行ldetect分析,获取全基因组连锁不平衡区块。这些区块内部的变异存在连锁不平衡,区块与区块之间则基本不存在连锁不平衡。在每一个区块内,将所有区域的代表参数全部输入 susie,分析方法与第一步中的susie相同。以susie输出的后验概率》0.8为阈值,确定起效区域。
[0102]
步骤s23.获取每一区域的生物学意义注释,基于广义线性回归模型定量分析影响个体表型的起效区域是否更多地与某一生物学意义注释相关。
[0103]
具体的,首先收集三类生物学意义注释,并将每一个体的所有区域均进行以下三类注释:
[0104]
1)组织特异性活性:从epimap获取222个人类组织的染色质状态注释,按照前述方法获取每种组织中基因组有活性的位置。
[0105]
2)细胞特异性活性:获取人单细胞染色质可及性测序峰值数据,获取每种细胞中基因组有活性的位置。
[0106]
3)生物学功能:从msigdb数据库下载所有生物学通路注释数据,排除包含少于10个基因或多于500个基因的通路,构建基因x通路注释矩阵(每个元素均为0或1),基于此矩阵进行层次聚类并对所有同路排序。从排序的一端开始,向另一端逐次将通路纳入候选集合。每当相邻两个通路有超过80%的重叠时,删除掉较小的通路,将较大的通路纳入候选集合,直至所有通路均被删除或纳入。对每个区域,若其存在于每条通路内基因的100000bp范围内,则将其注释为影响该通路。
[0107]
基于上述公开的内容,对于每种注释,所有区域对其均有1和0两种状态。对于每种注释,构造如下的广义回归分析模型以分析如下问题:影响表性的区域是否更多地与这一注释有关;其中,广义回归分析模型的表达式如下:
[0108]
status~pip+n+n+ref
1-40
+rec+asmc+con+(1|block) ;(7)
[0109]
其中,status示区域是否符合该注释(1/0),pip表示susie计算的该区域是起效区域的后验概率,n表示该区域内单倍型总数,n表示该区域纳入分析的总样本数,ref
1-40
表示该区域参考序列对应的序列类别分数,rec表示该区域内的染色体重组频率,asmc表示该区域在人类历史上的背景选择强度,con表示该区域的保守度,(1|block)表示作为随机效应项的block编号。其中,回归结果中,pip项的回归系数与p值用于衡量性状遗传度与某一注释的关联关系。
[0110]
步骤s3.根据每一基因的风险评分以及该基因所在调控区域的代表参数,结合变量筛选模型和第一梯度上升网络预测得到每一基因的表型预测值;
[0111]
在步骤s3中,根据每一基因的风险评分以及该基因所在调控区域的代表参数,结合变量筛选模型和第一梯度上升网络预测得到每一基因的表型预测值,包括:
[0112]
其中,优选的,变量筛选模型采用snpnet算法模型,第一梯度上升网络采用xgboost一梯度上升网络。
[0113]
步骤s31.对每一基因预处理后输入到变量筛选模型中进行计算,根据输出的基因位点权重,计算该基因在的外显子多基因风险评分,包括:
[0114]
对每一基因进行预处理,将预处理后的多个基因位点输入到snpnet算法模型中,进行lasso批量筛选迭代回归分析;
[0115]
在回归分析中加入协变量,且不对协变量的参数进行lasso缩放,以输出去除连锁不平衡的基因位点权重。
[0116]
具体的,获取genecodev29所有编码蛋白质基因的外显子范围的bed文件,对于每一个基因,使用bgenix及plink筛选处于其外显子区域的变异,去掉info参数《0.8,hardy-wenberg检验p值小于10-6
,变异基因型总数小于10的变异,再去掉在该区域内丢失超过50%变异的个体。将上述筛选后的基因位点输入snpnet算法,进行batchscreeningiterativelasso回归分析。其中,batchscreeningiterativelasso是一个多次迭代算法,起始值为一个空集作为候选变量集,在多次迭代中重复以下步骤:
[0117]
1)计算每个变量与残差的内积,逐步寻找解释残差最多的变量(即与残差内积最大的变量)并加入候选变量集。
[0118]
2)在其上求解lasso,即使得如下损失项最小的参数λ与回归系数β:
[0119][0120]
其中,y及x表示待预测表型及用于预测的参数,β表示回归系数,λ表示缩放系数,
[0121]
3)在所有解中找出符合如下边界条件的最小的参数λ,及其对应的一组回归系数β:
[0122][0123]
其中,rk(λ)|表示在第k次迭代中,λ对应的残差,x表示用于预测的参数,n表示参数总数。
[0124]
其中,在lasso回归模型中加入性别、年龄、样本来源、基因型主成分及其他用户提供的协变量,并指定这些协变量的回归参数不进行lasso缩放。依据输出的基因位点权重,
即可计算所有个体在该基因外显子上的多基因风险评分。
[0125]
步骤s32.将外显子多基因风险评分与该基因所在调控区域内的所有代表参数输入到第一梯度上升网络中,得到该基因的表型预测值,包括:
[0126]
1)根据功能基因组注释,预先设定每一基因的调控区域;
[0127]
具体的,获取epimap提供的人类222个组织的增强子-基因关系注释,对每个组织,取至少在一半样本中被标记为有关联的增强子并取并集,对每个基因,将所有组织中与其有关联的增强子取并集,再加上该基因上下游100,000bp范围作为该基因的全部调控区域。则对每个基因,取其调控区域内的所有代表参数进入下一步骤。
[0128]
2)将外显子多基因风险评分与该基因在调控区域内的所有代表参数作为模型输入,并将待预测形状的回归模型的残差作为模型预测标签,利用第一梯度上升网络预测得到该基因的表型预测值。
[0129]
具体的,对每一个基因,将上述外显子多基因风险评分及调控区域内所有代表参数共同输入xgboost建立梯度提升模型(r语言xgboost函数)。xgboost 利用自变量构建树形预测器,并在迭代中组合已有的树及其预测残差,达到梯度上升的效果。在迭代中,第m次迭代生成的树遵循如下规则:
[0130]fm
(x)=f
m-1
(x)+a
mhm
(x,τ
m-1
);
ꢀꢀꢀ
(10)
[0131]
其中,am表示第m次迭代的正则系数,τ
m-1
表示第m-1次迭代的残差,hm表示训练得到的用以预测第m-1次迭代残差的函数。在本实施例中,结果变量选为如下回归模型的残差:
[0132]
height~age+sex+center+pc
1-10
+covriate;
ꢀꢀꢀꢀꢀ
(11)
[0133]
其中,height,age,sex,center,pc
1-10
,covriate分别表示身高,年龄,性别,样本来源,基因主成分及其他用户指定的协变量。
[0134]
优选的,xgboost选择regression:squarederror模式,eta值设为1,gamma 值设为1,最大深度设为3。xgboost在每一个体上计算出每一个基因的身高预测值。删除所有输出模型为空的基因后,将每个基因的预测值分别与作为结果变量的待预测性状残差做相关分析,优选的,选出p值最小的前5000个基因对应的预测值进入下一步分析。
[0135]
步骤s4.将每一基因的表型预测值同时输入到第二梯度上升网络中进行整合,生成最终预测结果。
[0136]
在步骤s4计中,将每一基因的表型预测值同时输入到第二梯度上升网络中进行整合,生成最终预测结果,包括:
[0137]
将每一基因的表型预测值作为模型输入,并将待预测形状的真实值作为预测标签,利用第二梯度上升网络得到个体表型的最终预测结果。
[0138]
具体的,将上述步骤选出的5000个基因对应的身高预测值输入一个新的 xgboost模型中,标签为待预测表型的原始值。假如用户在之前的步骤提供了需要矫正的协变量covriate,且这些协变量经专家知识判定为可用于表型预测,则同样纳入xgboost中。由于身高为连续变量,xgboost选择 regression:squarederror模式,eta值设为1,gamma值设为1,最大深度设为 5。由于这一步变量数较多,计算需求较大,在使用者计算资源不足的情况下,可将最大深度调整为3。
[0139]
优选的,在利用模型进行预测之后,所述方法还包括对模型验证及应用,具体如下:
[0140]
在独立的验证人群中,重复上述步骤s1-s3得到基因水平预测量,并输入最后一步得到的xgboost模型中。假如在步骤s4训练时未进行待预测表型的标准化,则xgboost输出的值即为与真实表型单位相同的预测值,可能受到不同人群间的系统性偏移的影响。假如训练时将待预测表型进行了标准化,则输出值为z 值,需根据目标人群的身高的一般分布转化为真实值,即
[0141]
height=mean+z
×
se;
ꢀꢀꢀ
(12)
[0142]
基于上述公开的内容,本实施例不直接利用基因型数据,而通过深度神经网络将基因型(单倍型)转化为功能基因组参数,解决了现有技术中中直接分析基因型与疾病的关联时遇到的无法定位起效位点、无法根据起效位点准确预测疾病的缺陷,由于考虑到基因间存在复杂的相互作用关系,通过从功能基因组参数选取代表参数,并利用区域的代表参数分析各区域与个体表型的关联以及单基因的预测值,从而在单基因水平上整合来自各功能基因组改变的信息,避免了孤立分析每一基因位点的线性关联的局限性,最后通过梯度上升网络对整合所有基因的预测值,以充分反映基因间的非线性关系,从而提高预测的准确度,对于各种个体表型的预测具有重要意义。
[0143]
第二方面,本发明提供一种基于人体全基因组基因型预测个体表型的装置,包括:
[0144]
参数转化模块,用于获取每一个体的全基因组所有区域内的单倍型,基于深度神经网络将单倍型转化为功能基因组参数,并从每一区域的功能基因组参数中选取至多一个代表参数;
[0145]
关联分析模块,用于根据区域的代表参数,利用回归模型定量分析各区域与个体表型的关联关系;
[0146]
第一预测模块,用于根据每一基因的风险评分以及该基因所在调控区域的代表参数,结合变量筛选模型和第一梯度上升网络预测得到每一基因的表型预测值;
[0147]
第二预测模块,用于将每一基因的表型预测值同时输入到第二梯度上升网络中进行整合,生成最终预测结果。
[0148]
本实施例第二方面提供的前述装置的工作过程、工作细节和技术效果,可以参见如上第一方面或第一方面中任意一种可能设计所述的方法,于此不再赘述。
[0149]
第三方面,本发明提供一种计算机设备,包括依次通信相连的存储器、处理器和收发器,其中,所述存储器用于存储计算机程序,所述收发器用于收发消息,所述处理器用于读取所述计算机程序,执行如第一方面任意一种可能的设计中所述的方法。
[0150]
具体举例的,所述存储器可以但不限于包括随机存取存储器(random-accessmemory,ram)、只读存储器(read-only memory,rom)、闪存(flash memory)、先进先出存储器(first input first output,fifo)和/或先进后出存储器(firstinput last output,filo)等等;所述处理器可以不限于采用型号为stm32f105 系列的微处理器;所述收发器可以但不限于为wifi(无线保真)无线收发器、蓝牙无线收发器、gprs(general packet radio service,通用分组无线服务技术)无线收发器和/或zigbee(紫蜂协议,基于ieee802.15.4标准的低功耗局域网协议)无线收发器等。此外,所述计算机设备还可以但不限于包括有电源模块、显示屏和其他必要的部件。
[0151]
本实施例第三方面提供的前述计算机设备的工作过程、工作细节和技术效果,可以参见如上第一方面或第一方面中任意一种可能设计所述的方法,于此不再赘述。
[0152]
第四方面,本发明提供一种计算机可读存储介质,所述计算机可读存储介质上存储有指令,当所述指令在计算机上运行时,执行如第一方面任意一种可能的设计中所述的方法。
[0153]
其中,所述计算机可读存储介质是指存储数据的载体,可以但不限于包括软盘、光盘、硬盘、闪存、优盘和/或记忆棒(memory stick)等,所述计算机可以是通用计算机、专用计算机、计算机网络、或者其他可编程装置。
[0154]
本实施例第四方面提供的前述计算机可读存储介质的工作过程、工作细节和技术效果,可以参见如上第一方面或第一方面中任意一种可能设计所述的方法,于此不再赘述。
[0155]
第五方面,本发明提供一种包含指令的计算机程序产品,当所述指令在计算机上运行时,使所述计算机执行如第一方面任意一种可能的设计中所述的方法。
[0156]
本实施例第五方面提供的前述包含指令的计算机程序产品的工作过程、工作细节和技术效果,可以参见如上第一方面或第一方面中任意一种可能设计所述的方法,于此不再赘述。
[0157]
最后应说明的是:以上所述仅为本发明的优选实施例而已,并不用于限制本发明的保护范围。凡在本发明的精神和原则之内,所作的任何修改、等同替换、改进等,均应包含在本发明的保护范围之内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1