一种抗癌药物筛选方法及系统
1.本发明涉及药物筛选技术领域,更具体的说是涉及一种抗癌药物筛选方法及系统。
背景技术:
2.恶性肿瘤发病和死亡数逐年上升,恶性肿瘤负担严重,是城乡居民的第一位死亡原因。由于发病人数多,致死性高,因此癌症不仅仅是个简单的疾病问题,更是公共卫生问题,甚至社会民生问题。
3.为了广大民众能够获得具备较好疗效、毒副作用又小的药品,寻找新药是一个投资巨大、时间漫长的过程。传统的新药研发模式包括生物萃取、化合物合成等,耗时长,成本高。因此,如何提供一种成本低、耗时短的抗癌药物快捷筛选方法,是本领域技术人员亟需解决的问题。
技术实现要素:
4.有鉴于此,本发明提供了一种抗癌药物筛选方法及系统,以克服上述技术缺陷。
5.为了实现上述目的,本发明提供如下技术方案:
6.一种抗癌药物筛选方法,包括以下步骤:
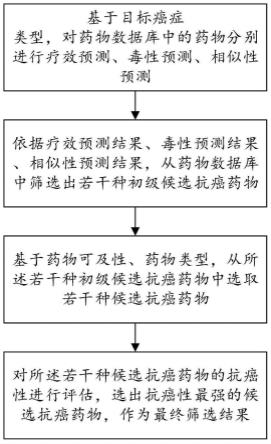
7.步骤1、基于目标癌症类型,对药物数据库中的药物分别进行疗效预测、毒性预测、相似性预测;
8.步骤2、依据疗效预测结果、毒性预测结果、相似性预测结果,从药物数据库中筛选出若干种初级候选抗癌药物;
9.步骤3、基于药物可及性、药物类型,从所述若干种初级候选抗癌药物中选取若干种候选抗癌药物;
10.步骤4、对所述若干种候选抗癌药物的抗癌性进行评估,选出抗癌性最强的候选抗癌药物,作为最终筛选结果。
11.可选的,所述药物数据库为drug repurposing hub数据库。
12.可选的,所述步骤1中,对药物进行疗效预测的方法为:
13.收集癌症药物敏感性基因组学gdsc数据库中每种化合物对目标癌细胞的半数杀灭浓度ic50;
14.确定癌症药物敏感性基因组学gdsc数据库中每种化合物的smiles表达式;
15.基于每种化合物对目标癌细胞的半数杀灭浓度ic50和每种化合物的smiles表达式,共同构建第一训练数据集;
16.以导向性信息传递网络为基础,加入全连接层,构建第一深度神经网络模型;
17.以第一训练数据集中的每种化合物对目标癌细胞的半数杀灭浓度ic50作为标签,以每种化合物的smiles表达式作为输入特征,训练第一深度神经网络模型,得到疗效预测模型;
18.将目标药物输入所述疗效预测模型中进行疗效预测。
19.可选的,所述步骤1中,对药物进行毒性预测的方法为:
20.收集clintox数据集中每种化合物的毒性数据和fda批准数据;
21.确定clintox数据集中每种化合物的smiles表达式;
22.基于每种化合物的毒性数据和每种化合物的smiles表达式,共同构建第二训练数据集;
23.以导向性信息传递网络为基础,加入全连接层,构建第二深度神经网络模型;
24.以第二训练数据集中的每种化合物的毒性数据作为标签,以每种化合物的smiles表达式作为输入特征,训练第二深度神经网络模型,得到毒性预测模型;
25.将目标药物输入所述毒性预测模型中进行毒性预测。
26.可选的,所述步骤1中,对药物进行相似性预测的方法为:
27.从fda官网和开源的药物数据平台收集fda已批准的现有抗癌药物,确定每种现有抗癌药物的摩根指纹向量;
28.基于目标药物的摩根指纹向量与现有抗癌药物的摩根指纹向量,计算目标药物与现有抗癌药物的tanimoto系数,从而评估目标药物与现有抗癌药物的结构相似性。
29.可选的,所述步骤4中,通过cck-8细胞活性实验对所述若干种候选抗癌药物的抗癌性进行评估。
30.可选的,所述第一深度神经网络模型和第二深度神经网络模型,均是以导向性信息传递为基础,使用relu激活,使用贝叶斯优化法确定最优超参数,使用rdkit进行特征增强,每次训练时采用scaffold-based法随机分割训练集和验证集,训练结果以20个不同的随机化权重进行集成,所构建的模型c-index达到0.954。
31.一种抗癌药物筛选系统,包括:
32.预测模块,用于基于目标癌症类型,对药物数据库中的药物分别进行疗效预测、毒性预测、相似性预测;
33.初级筛选模块,用于依据疗效预测结果、毒性预测结果、相似性预测结果,从药物数据库中筛选出若干种初级候选抗癌药物;
34.二级筛选模块,用于基于药物可及性、药物类型,从所述若干种初级候选抗癌药物中选取若干种候选抗癌药物;
35.最终筛选模块,用于对所述若干种候选抗癌药物的抗癌性进行评估,选出抗癌性最强的候选抗癌药物,作为最终筛选结果。
36.经由上述的技术方案可知,本发明提供了一种抗癌药物筛选方法及系统,与现有技术相比,具有以下有益效果:
37.给定一个目标癌症类型时,本发明能够通过多级筛选的方式,综合抗癌性、毒性、相似性等因素,结合深度神经网络模型,普适性地选出最优的抗癌药物,筛选方法简单、成本低、耗时短,优于现有的抗癌新药筛选方法。
附图说明
38.为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本
发明的实施例,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据提供的附图获得其他的附图。
39.图1为本发明的方法步骤示意图;
40.图2为本发明的系统模块示意图。
具体实施方式
41.下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
42.乳腺癌是全球发病率第一的恶性肿瘤,也是造成癌症相关死亡的主要原因之一。严重危害人类,特别是女性的生命健康和生活质量。乳腺癌是起源于乳腺上皮细胞的恶性肿瘤,具有高度异质性,三阴性乳腺癌是其中恶性程度最高的亚型,占全部乳腺癌人群的10-20%。三阴性乳腺癌的激素受体、her-2受体表达均为阴性,因此患者无法从内分泌治疗和抗her-2靶向治疗中获益。
43.目前标准的三阴性乳腺癌治疗药物为化疗,包括蒽环类、紫杉类、铂类等,但这些药物毒性较大。并且,三阴性乳腺癌的异质性使患者容易发生耐药,导致无药可用。这一现状催生了三阴性乳腺癌药物的大力研发,包括免疫治疗、parp抑制剂、抗血管生成治疗和抗体偶联药物等。然而,这些药物的适应症局限,疗效往往未得到大型临床试验验证。
44.本发明实施例以三阴性乳腺癌为例,提供一种抗三阴性乳腺癌药物的筛选方法,参见图1,包括以下步骤:
45.步骤1、对drug repurposing hub数据库中的6797种药物分别进行疗效预测、毒性预测、相似性预测。
46.(1)药物的疗效预测
47.癌症药物敏感性基因组学gdsc数据库中包含tnbc细胞系mda-mb-231对363种化合物的实验数据,收集癌症药物敏感性基因组学gdsc数据库中每种化合物对mda-mb-231(人乳腺癌细胞)的半数杀灭浓度ic50;
48.确定癌症药物敏感性基因组学gdsc数据库中每种化合物的smiles表达式;
49.基于每种化合物对mda-mb-231细胞的半数杀灭浓度ic50和每种化合物的smiles表达式,共同构建第一训练数据集;
50.以导向性信息传递网络为基础,加入全连接层,构建第一深度神经网络模型;
51.以第一训练数据集中的每种化合物对mda-mb-231细胞的半数杀灭浓度ic50作为标签,以每种化合物的smiles表达式作为输入特征,训练第一深度神经网络模型,在模型中对化合物结构进行解构、提取潜在特征信息,学习其与ic50之间的非线性关系,得到疗效预测模型;
52.将目标药物输入所述疗效预测模型中进行疗效预测。
53.(2)药物的毒性预测
54.clintox数据集中包含1478种化合物的毒性数据和fda批准数据,收集clintox数据集中每种化合物的毒性数据和fda批准数据;
55.确定clintox数据集中每种化合物的smiles表达式;
56.基于每种化合物的毒性数据和每种化合物的smiles表达式,共同构建第二训练数据集;
57.以导向性信息传递网络为基础,加入全连接层,构建第二深度神经网络模型;
58.以第二训练数据集中的每种化合物的毒性数据作为标签,以每种化合物的smiles表达式作为输入特征,训练第二深度神经网络模型,得到毒性预测模型;
59.将目标药物输入所述毒性预测模型中进行毒性预测。
60.(3)药物的相似性预测
61.从fda官网和开源的药物数据平台收集fda已批准的531种现有抗癌药物,确定每种现有抗癌药物的摩根指纹向量;
62.基于目标药物的摩根指纹向量与现有抗癌药物的摩根指纹向量,计算目标药物与现有抗癌药物的tanimoto系数,从而评估目标药物与现有抗癌药物的结构相似性。
63.所述第一深度神经网络模型和第二深度神经网络模型,均是以导向性信息传递为基础,使用relu激活,使用贝叶斯优化法确定最优超参数,使用rdkit进行特征增强,每次训练时采用scaffold-based法随机分割训练集和验证集,训练结果以20个不同的随机化权重进行集成,所构建的模型c-index达到0.954。
64.步骤2、依据疗效预测结果、毒性预测结果、相似性预测结果,从drug repurposing hub数据库中进行筛选,筛选标准为;ic50《0.01,可能被fda批准,毒性低,与既往抗癌药物结构不相似,共获得307种初级候选药物。
65.步骤3、基于药物可及性、药物类型,从所述307种初级候选药物中选取4种候选抗癌药物。
66.在这307种药物中,第一类是已经成熟的临床药物,包括:抗生素类、肌松药类、凝血酶抑制剂和其他药物,第二类是一些在非临床领域使用的化学药物,如羟丙基-β-环糊精等,第三类是在研的临床试验药物,第四类为临床前药物,第五类是生物提取物。筛选标准:1、查阅pubmed,以“药物+cancer”作为关键词进行检索,相关检索结果小于2条。2、同类型药物只选择一种。3、国内可及。
67.步骤4、通过cck-8细胞活性实验对4种候选抗癌药物的抗癌性进行评估,选出抗癌性最强的候选抗癌药物,作为最终筛选结果。具体的,cck-8细胞活性实验的步骤为:
68.步骤4.1、取对数生长期的细胞,消化并计数,稀释成5
×
104个细胞/ml的细胞悬液;
69.步骤4.2、取96孔板,在预备进行实验的孔腔周围的一圈孔腔内各加入100μl pbs溶液,以防止液体蒸发影响实验结果;
70.步骤4.3、在96孔板的第二列加入100μl完全培养基,将细胞悬液吹打混匀,从第三列起,在96孔板的每个孔内加入100μl细胞悬液,每次加液前重新吹打,确保每个孔内加入的细胞数量相同。为保证细胞均匀分布,加液时动作轻柔,从一侧缓缓加入,不要使96孔板产生无谓的晃动;
71.步骤4.4、24小时后,在显微镜下观察细胞,保证细胞贴壁;
72.步骤4.5、吸弃96孔板中的旧培养基,分别在含有细胞的孔腔内加入含浓度为0、5、10、25、50、100、250、500μm/l药物的完全培养基,制作3个复孔;
73.步骤4.6、在药物处理12、24、36和48小时后,吸弃原培养基,用pbs溶液洗涤1次,加入含10%cck-8试剂的完全培养基100μl,放入37℃细胞培养箱;
74.步骤4.7、2小时后,使用酶标仪测定450nm处的吸光度(od值),空白孔的吸光度为ab,对照孔的吸光度为ac,实验孔的吸光度为as;细胞存活率=[(as-ab)/(ac-ab)]
×
100%;绘制细胞生长曲线,计算12、24、36和48小时的ic50值。
[0075]
在另一种实施例中还公开一种抗癌药物筛选系统,参见图2,包括:
[0076]
预测模块,用于基于目标癌症类型,例如三阴性乳腺癌等,对药物数据库中的药物分别进行疗效预测、毒性预测、相似性预测;
[0077]
初级筛选模块,用于依据疗效预测结果、毒性预测结果、相似性预测结果,从药物数据库中筛选出若干种初级候选抗癌药物;
[0078]
二级筛选模块,用于基于药物可及性、药物类型,从所述若干种初级候选抗癌药物中选取若干种候选抗癌药物;
[0079]
最终筛选模块,用于对所述若干种候选抗癌药物的抗癌性进行评估,选出抗癌性最强的候选抗癌药物,作为最终筛选结果。
[0080]
对于实施例公开的系统模块而言,由于其与实施例公开的方法相对应,所以描述的比较简单,相关之处参见方法部分说明即可。
[0081]
本说明书中各个实施例采用递进的方式描述,每个实施例重点说明的都是与其他实施例的不同之处,各个实施例之间相同相似部分互相参见即可。
[0082]
对所公开的实施例的上述说明,使本领域专业技术人员能够实现或使用本发明。对这些实施例的多种修改对本领域的专业技术人员来说将是显而易见的,本文中所定义的一般原理可以在不脱离本发明的精神或范围的情况下,在其它实施例中实现。因此,本发明将不会被限制于本文所示的这些实施例,而是要符合与本文所公开的原理和新颖特点相一致的最宽的范围。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1