一种用于化合物性质预测的图结构优化方法、系统及设备
1.本发明属于计算机科学、生物化学和机器学习技术领域,涉及一种图结构优化方法、系统及设备,具体涉及一种用于化合物性质预测的图结构优化方法、系统及设备。
背景技术:
2.在生物医学领域,通常需要对一个未知的生物分子结构进行性质归类,使用传统的生物学分析方法确定分子的性质十分耗时和费力,需要引入神经网络模型去高效准确地对分子性质进行预测。在化合物性质预测模型的研究上,max welling等人在2016年通过定义图上的傅里叶变换提出了gcn(图卷积神经网络),以聚合策略和组合函数的主体,得到各个节点的表征,再通过一些常见的图池化函数,例如sum、max等方式,得到图级别的表征,根据这一表征对原有化合物进行性质归类;而后续在2017年,petar velickovic等人将注意力机制初步引入gcn当中,提出gat(图注意力网络),使其能获得更好的节点表征;keyulu xu等人则在2018年通过理论分析,将一种新的聚合策略引入gcn,提出gin(图同构网络),其能够兼顾节点表征和图级别的表征,在化合物性质归类上的准确度更高;同样是改变聚合策略,will hamilton等人考虑到大规模图数据的情况下,模型难以训练的问题,将图采样方法作用于图神经网络的聚合策略上,提出了graphsage(使用图采样的图神经网络),在保证准确度的同时对大型图网络数据也有很好的实用性;另一方面,组合函数也是人们的关注点之一,hao zhu等人在2020年由图上的随机游走方式,定义了一种新的组合函数,使图神经网络能够考虑不同跳数之间的节点,由此提出ssgc(基于马尔科夫图扩散核的图神经网络),在许多方面都有不错的效果。
3.除了上述的一些方法,另一些方法则设计了专门的图池化函数来获取图级别的表征,以得到更好的分类、预测结果。早在2015年,oriol vinyals就试图将图上的节点对进行池化处理,提出了set2set(基于递归神经网络对所有节点进行编码的图池化方法);而最近的2020年,ekagra ranjan等人借助节点舍弃机制,通过使用评分函数删除分数较低的节点(或集群)来进行池化,提出了asap(基于一种节点丢弃机制的图池化方法)并取得不错的效果。
4.综上所述,从最早的简单图池化方法set2set,到图神经网络gcn与一般图池化方法的组合,再到一些针对不同问题进行改进的图神经网络的变体gat、gin等方法,最终到设计专门的图池化方法asap。观察这一发展历程可以发现,模型所使用的方法或技巧更多去考虑模型内部的机制,例如图神经网络中的聚合策略、组合函数,又例如图池化函数中的不同池化机制。它们都试图挖掘图中的信息,而没有很好地去利用原本就在图中的信息。而生物医学领域的生物分子结构,比其他类型的图结构数据具有性质繁复、结构性强等特点,所以对于生物分子结构的性质预测,需要考虑更能突出图结构、使不同类生物分子间差异更大的方法。
技术实现要素:
5.为了解决上述技术问题,本发明提出了一种基于图上通勤时间距离,预先对化合物的图结构数据进行优化,使其具备更良好的拓扑表示,进而使模型具有更好性能的化合物性质预测方法、系统及设备,即让多个模型在结构性强、种类繁复的预测情形下,也有良好表现。
6.本发明的方法所采用的技术方案是:一种用于化合物性质预测的图结构优化方法,包括以下步骤:
7.步骤1:获取化合物数据,包括化合物总数、化合物类别数、化合物中的平均小分子数和平均连边数,对化合物数据进行数据预处理;
8.步骤2:使用预处理后的数据进行图上的通勤时间距离的计算,再经过邻域选取函数得到优化后的化合物图结构;
9.步骤3:将步骤2中获得的优化后的化合物图结构输入gnn分类器,在化合物图数据上进行性质预测;
10.所述gnn分类器,整体由图卷积模块和图池化模块构成,包括第一input_dim
×
hidden卷积块、第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块、图池化函数和hidden
×
output_dim多层线性感知机组成;
11.所述第一input_dim
×
hidden卷积块、第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块顺序连接,共同构成图卷积模块,输入优化后的化合物图结构,经过图卷积模块,聚合并更新图上节点特征,提取节点级表征;
12.所述图池化函数和所述hidden
×
output_dim多层线性感知机顺序连接,共同构成图池化模块,输入上述的节点级表征,经过图池化模块,提取出图级别表征,作为所述hidden
×
output_dim多层线性感知机的输入;这个过程共同构成图池化模块,输入为图上节点级表征,经过图池化函数处理与多层线性感知机更新后,输出化合物性质预测结果;
13.所述第一input_dim
×
hidden卷积块由一个input_dim
×
hidden卷积层和一个relu激活函数构成;所述第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块由一个hidden
×
hidden卷积层和一个relu激活函数构成;所述图池化函数由各种图池化层组成,一般为mean、add或sum方法;所述hidden
×
output_dim多层线性感知机由一个hidden
×
hidden线性层、一个relu激活函数、一个dropout层和一个hidden
×
output_dim线性层和一个log_softmax函数组成。
14.本发明的系统所采用的技术方案是:一种用于化合物性质预测的图结构优化系统,包括以下模块:
15.模块1,用于获取化合物数据,包括化合物总数、化合物类别数、化合物中的平均小分子数和平均连边数,对化合物数据进行数据预处理;
16.模块2,用于使用预处理后的数据进行图上的通勤时间距离的计算,再经过邻域选取函数得到优化后的化合物图结构;
17.模块3,用于将模块2中获得的优化后的化合物图结构输入gnn分类器,在化合物图数据上进行性质预测;
18.所述gnn分类器,整体由图卷积模块和图池化模块构成,包括第一input_dim
×
hidden卷积块、第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块、图池化函数和
hidden
×
output_dim多层线性感知机组成;
19.所述第一input_dim
×
hidden卷积块、第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块顺序连接,共同构成图卷积模块,输入优化后的化合物图结构,经过图卷积模块,聚合并更新图上节点特征,提取节点级表征;
20.所述图池化函数和所述hidden
×
output_dim多层线性感知机顺序连接,共同构成图池化模块,输入上述的节点级表征,经过图池化模块,提取出图级别表征,作为所述hidden
×
output_dim多层线性感知机的输入;这个过程共同构成图池化模块,输入为图上节点级表征,经过图池化函数处理与多层线性感知机更新后,输出化合物性质预测结果;
21.所述第一input_dim
×
hidden卷积块由一个input_dim
×
hidden卷积层和一个relu激活函数构成;所述第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块由一个hidden
×
hidden卷积层和一个relu激活函数构成;所述图池化函数由各种图池化层组成,一般为mean、add或sum方法;所述hidden
×
output_dim多层线性感知机由一个hidden
×
hidden线性层、一个relu激活函数、一个dropout层和一个hidden
×
output_dim线性层和一个log_softmax函数组成。
22.本发明的设备所采用的技术方案是:一种用于化合物性质预测的图结构优化设备,包括:
23.一个或多个处理器;
24.存储装置,用于存储一个或多个程序,当所述一个或多个程序被所述一个或多个处理器执行时,使得所述一个或多个处理器实现所述的用于化合物性质预测的图结构优化方法。
25.相对于现有技术,本发明的有益效果是:由于蛋白质与生物分子等化合物具有强结构性、种类繁复、性质多样等特点,现有的技术虽然在化合物性质预测上取得了一定的突破,但在应对这种强结构的数据时,预测结果还不够好,往往难以捕获高阶结构信息,通勤时间距离通过综合考虑图中长距离节点与短距离节点,对数据进行预处理,得到更好的图结构,使模型具有更好的准确率,并且在种类繁多、性质复杂的情况下也能有良好表现。
附图说明
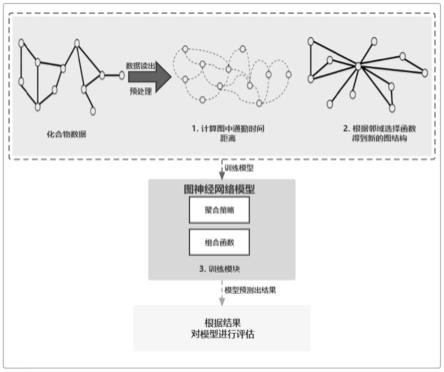
26.图1为本发明实例的方法原理框图;
27.图2为本发明实例的gnn分类器结构图;
28.图3为本发明实施例的参数选择时在mutag数据集上使用不同topk值的结果准确度示意图;
29.图4为本发明实施例的参数选择时在ptc-mr数据集上使用不同topk值的结果准确度示意图;
30.图5为本发明实施例的不同算法在mutag数据集上的accuracy对比示意图;
31.图6为本发明实施例的不同算法在d&d数据集上的accuracy对比示意图;
32.图7为本发明实施例的不同算法在proteins数据集上的accuracy对比示意图;
33.图8为本发明实施例的不同算法在ptc-mr数据集上的accuracy对比示意图;
34.图9为本发明实施例的不同算法在enzymes数据集上的accuracy对比示意图。
具体实施方式
35.为了便于本领域普通技术人员理解和实施本发明,下面结合附图及实施例对本发明作进一步的详细描述,应当理解,此处所描述的实施示例仅用于说明和解释本发明,并不用于限定本发明。
36.请见图1,本发明提供的一种用于化合物性质预测的图结构优化方法,包括以下步骤:
37.步骤1:获取化合物数据,包括化合物总数、化合物类别数、化合物中的平均小分子数和平均连边数,对化合物数据进行数据预处理;
38.本实施例中对化合物数据进行数据预处理,包括变量衍生、数据清洗转换与特征标准化处理;
39.本实施例进行变量衍生原因在于化合物结构图中的拓扑信息,如一阶连接信息、二阶信息等,对于其性质预测有非常重要的作用。仅仅依靠单一的节点特征信息,可能难以预测出化合物的性质,因此既要针对分子节点之间的连接关系来进行,更需要加工整合形成新的变量。在分子和蛋白质数据中,某些结构模式与功能属性有着必然联系,因此对于原本没有节点特征的数据集,我们可以把这些结构模式间接地建模成它的节点特征,比如说根据图中节点的连通情况,把每个节点的度进行onehot编码后构成它的特征,这个衍生出来的新变量可以输入到模型当中,有效地提升模型对化合物性质预测的准确度。再者,对于节点度较大的图而言,onehot编码难以全面描述它的度信息,可以对每个节点度进行正交变换处理,以此作为它们的特征。除此之外,还可以根据图中一些比较重要的属性,加入一系列反映这些属性的权重,对变量进行相加、连接、计算笛卡尔积等各种计算方式,将多个属性整合到一起的方式,得到新的、更能反映出化合物性质的特征变量。
40.本实施例数据清洗转换主要包括以下工作。孤立节点的清理,在部分化合物的图数据中,会存在一些孤立节点,它们虽然也包含了一部分信息,但它们的存在给模型带来的噪声影响更大,因此往往直接剔除,得到一个最大连通子图。数据清洗技术上较为简单,处理起来比较繁杂,除了使用工具或者人工处理以外,也可以编写一些代码帮助清洗。
41.数据转换包括将原本不同数据类型的图数据,包括scipy,sparse.coo_matrix,numpy.ndarray等数据类型,统一处理成edge_index+edge_weight的形式,一方面有利于接下来的模型训练,另一方面处理成这样的形式可以显著降低内存开销。对于某些模型而言,稀疏矩阵形式并不能满足其输入要求,我们还需要针对该方法把图数据转换成密集矩阵的形式,再进行下一步模型的训练。
42.特征标准化处理的具体操作是,对原有图数据中的节点特征进行线性变换,使得结果值映射到[0,1]之间,将每个属性的值都映射到比较接近的数值大小和波动幅度,从而可以提高梯度下降时的收敛速度和模型的精度。
[0043]
步骤2:使用预处理后的数据进行图上的通勤时间距离的计算,再经过邻域选取函数得到优化后的化合物图结构;
[0044]
对于一个具有n个节点和m条连边的图结构数据,本实施例用g=(v,e)来表示它,其中v为g的初始节点集合,e为g的初始连边集合,此外本实施例用a来表示g的邻接矩阵,d表示g的度信息对角矩阵。对于其中的每个节点,都会拥有相应的特征,本实施例用来表示,其中q0为g中节点的特征维度。
[0045]
图上的通勤时间距离(记作ectd)可以通过计算图拉普拉斯矩阵的广义逆l+来实现,节点i与节点j之间的通勤时间距离的具体计算公式如(1)所示:
[0046]
ectd
i,j
=volg(e
i-ej)
t
l
+
(e
i-ej)
ꢀꢀꢀꢀ
(1)
[0047]
其中,volg表示整个图g的总度数,可以视作常数;ei表示单位矩阵的第i行。
[0048]
显然,用这种方式计算图上通勤时间距离的时间开销是非常大的,为了减少时间开销同时不影响其效果,本实施例采取了以下这些措施:
[0049]
首先,本实施例使用归一化的拉普拉斯矩阵l
sym
。图上的拉普拉斯矩阵定义为l,其计算方式如(2)式:
[0050]
l=d-a
ꢀꢀꢀꢀ
(2)
[0051]
显然,l中包含了图上的度信息和连接信息。但在图中,直接使用l可能会面临一些问题。例如,不同节点间的度信息可能相差很大,节点m可能只跟2个节点相连,而节点n却和100个节点相连,这样一来,在模型训练过程中的收敛速度将大大减缓,模型训练的耗时也就更长;再者,例如有蛋白质或者生物分子的图结构在连边权重上差异非常大,节点a与其他节点的连边权重范围在0~1之间,而节点b与其他节点的连边权重却在1~100之间,这样模型会着重学习这些具有大权重值的节点,而忽视了连边权重小的节点。为了应对这些问题,本实施例采用对称归一化的拉普拉斯矩阵l
sym
,因为对于不同节点而言,连边权重与连边数量的绝对值并不重要,它们的相对大小才重要。对称归一化的拉普拉斯矩阵l
sym
的计算如(3)式,其中i表示单位矩阵:
[0052][0053]
接着,本实施例采用低秩近似来计算归一化拉普拉斯矩阵的广义逆由l
sym
计算一般是通过svd操作,这个过程中往往需要计算多个n
×
n的矩阵。为了简化计算复杂度,本实施例采用低秩近似的方式,即丢弃部分结构信息来换取计算效率,其计算方式如(4)式:
[0054][0055]
rank为低秩近似的阈值,本实施例并没有对这个参数进行调优,而是使用它的默认值1e-15。
[0056]
最后,本实施例通过计算平均首次到达时间(记作aft),来间接计算通勤时间距离。由(1)式本实施例知道ectd矩阵的单个元素计算方式,但在计算全图通勤时间距离的情况下,这种方式无疑是效率低下的。因此,本实施例通过先计算aft的方式来更高效地计算ectd。根据定义,节点i和节点j间的通勤时间距离等同于节点i到节点j的平均首次到达时间,加上节点j到节点i的平均首次到达时间,即如(5)式所示:
[0057]
ectd
i,j
=aft
i,j
+aft
j,i
ꢀꢀꢀꢀ
(5)
[0058]
而在计算aft的过程中,为了简化其计算复杂度,fouss等人通过将原图中的拉普拉斯矩阵和移除一个节点后子图的拉普拉斯矩阵相联系,推导出了一个十分巧妙的aft计算方式,其流程如(6)-(8)式所示:
[0059][0060]
[0061]
aft_norm=aft/volgꢀꢀ
(8)
[0062]
diag()为取矩阵的主对角线元素,由(7)式本实施例就已经计算出了aft,不过同样考虑到模型收敛与节点特征学习的问题,本实施例用volg来对aft做标准化。计算出aft后,通过(5)式本实施例便可得出全图的ectd。
[0063]
在计算得到全图的ectd之后,本实施例通过一个邻域选取函数来确定每个节点的新邻域,其过程如(9)式所示:
[0064]nectd
(i)=selection(ectd)
ꢀꢀ
(9)
[0065]
这里n
ectd
(i)代表节点i基于ectd的邻域,而selection函数基于节点间的ectd值来为其选取合适的邻域,在此处本实施例简单地选取topk个拥有最小ectd值的节点作为中心节点的邻域,也即每个节点最多拥有topk个邻居。这样,邻域选择函数就为每个节点带来了更合理的邻域分布,以及更合适的邻域信息。因此,本实施例努力将每个节点连接到一个固定数量的topk个邻居,这些邻居可能有一个更强的联系。
[0066]
步骤3:将步骤2中获得的优化后的化合物图结构输入gnn分类器,在化合物图数据上进行性质预测;
[0067]
请见图2,本实施例的gnn分类器,整体由图卷积模块和图池化模块构成,包括第一input_dim
×
hidden卷积块、第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块、图池化函数和hidden
×
output_dim多层线性感知机组成;
[0068]
第一input_dim
×
hidden卷积块、第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块顺序连接,共同构成图卷积模块,输入优化后的化合物图结构,经过图卷积模块,聚合并更新图上节点特征,提取节点级表征;
[0069]
图池化函数和hidden
×
output_dim多层线性感知机顺序连接,共同构成图池化模块,输入上述的节点级表征,经过图池化模块,提取出图级别表征,作为hidden
×
output_dim多层线性感知机的输入;这个过程共同构成图池化模块,输入为图上节点级表征,经过图池化函数处理与多层线性感知机更新后,输出化合物性质预测结果。
[0070]
第一input_dim
×
hidden卷积块由一个input_dim
×
hidden卷积层和一个relu激活函数构成;第一hidden
×
hidden卷积块、第二hidden
×
hidden卷积块由一个hidden
×
hidden卷积层和一个relu激活函数构成;图池化函数由各种图池化层组成,一般为mean、add或sum方法;hidden
×
output_dim多层线性感知机由一个hidden
×
hidden线性层、一个relu激活函数、一个dropout层和一个hidden
×
output_dim线性层和一个log_softmax函数组成;
[0071]
本实施例gnn分类器,为训练好的gnn分类器;其训练过程是:
[0072]
(1)获取化合物数据集中若干相关信息/数据,包括化合物总数、化合物类别数、化合物中的平均小分子数和平均连边数,对化合物数据进行数据预处理;并将数据划分为训练集、测试集和验证集;
[0073]
(2)使用预处理后的数据进行图上的通勤时间距离的计算,再经过邻域选取函数得到优化后的化合物图结构;为邻域选取函数设定合适的阈值;
[0074]
通过对比一个参数在选择不同值时gnn分类器的表现来确定,如图2所示,在mutag数据集中,邻域选取函数的topk值在10左右时gnn分类器预测的性能达到最优,因此在mutag数据集中使用gnn分类器的情况下,邻域选取函数的topk值选取为10比较合适。其他
情况下,参数的选择同理。
[0075]
(3)使用优化后的化合物结构数据进行半监督学习,训练出一个gnn分类器,并将模型序列化永久储存到硬盘中,其具体实施方式为:
[0076]
在化合物性质预测中,gnn分类器包括:图池化模块和分类模块。图池化模块用于对图结构执行池化操作,得到图结构的表征;分类模块基于图结构的表征,得到该图结构的分类结果。图池化模块的输出与分类模块的输入连接,分类模块的输出作为gnn分类器的输出。
[0077]
图池化模块可以采用图神经网络,分类模块可以是至少一层全连接层,图池化模块和分类模块组成分类网络,分类网络每进行一次迭代训练,图池化模块对图像执行一次图池化方法,得到当前迭代训练下的图像的表征。
[0078]
接着,将从训练数据集中选取的样本图像的图结构输入分类网络,计算得到分类网络的损失值。在每次迭代训练中,基于图池化模块的损失值和分类模块的损失值,计算得到分类网络的损失值,而图池化模块的损失值和分类模块的损失值均可以通过选取的样本图像的图结构和两者各自的损失函数计算得到。
[0079]
在进行多次迭代训练之后,通过分类网络的损失值检测gnn分类器是否满足训练完成条件,若gnn分类器满足训练完成条件,则将gnn分类器作为训练好的gnn分类器。
[0080]
训练完成条件包括以下至少一项:训练迭代次数达到预定迭代阈值(通过gnn分类器的损失值预先估计得到的阈值),gnn分类器的损失值小于预定损失值阈值。例如,训练迭代达到5千次。损失值小于0.05。设置训练完成条件可以加快模型收敛速度。
[0081]
若gnn分类器不满足训练完成条件,则调整gnn分类器中的相关参数使得gnn分类器的损失值收敛,基于调整后的gnn分类器和调整后的gnn分类器的损失值。
[0082]
整个gnn分类器训练完成,随后可将模型序列化永久储存到硬盘中。
[0083]
使用gnn分类器在化合物图数据上进行性质预测,其具体步骤为从硬盘反序列化到内存中,输入新的化合物结构数据,便可对其性质进行预测。
[0084]
若经过了一段时间出现了新数据需要重新训练,则返回步骤1。
[0085]
本实施例针对现有的技术在预测种类繁多、具有强结构性的化合物性质时,预测准确度不高的情况,提出了一种用于化合物性质预测的图结构优化方法以解决这些问题,为验证基于通勤时间距离的化合物性质预测方法的有效性,使用以下5个数据集:
[0086]
(1)mutag是一组硝化化合物的数据集,目的是预测它们在鼠伤寒沙门氏菌上的诱变。输入图用于表示化合物,其中节点代表原子,并由原子类型标记,而节点之间的连边表示相应的原子之间的键。它包括188个具有7个离散节点标签的化合物样品,2类化合物,每个图平均节点数为17.93,平均连边数为19.79。
[0087]
(2)d&d是一个包含1,178个蛋白质结构的数据集。每个蛋白质用一个图表示,其中的节点是氨基酸,如果两个节点相距小于6埃,则用一条边连接。预测目标是分辨这些蛋白质中的酶和非酶。它包含1,178张图,分为2类蛋白质,每个图平均节点数为284.32,平均连边数为715.66。
[0088]
(3)proteins是一个包含1,113个蛋白质的数据集,它们被分为酶或非酶。节点代表氨基酸,如果两个节点相距小于6埃,则通过一条边连接。它包含1,113张图,分为两类蛋白质,每个图平均节点数为39.06,平均连边数为72.82。
[0089]
(4)ptc-mr是一组包含344种化合物的数据集,报告了它们对大鼠的致癌性。每个节点表示其中的化合物分子,拥有19个节点标签,连边代表它们之间的键,预测任务是分辨它们对大鼠是否有致癌性。它包含344张图,分为2类化合物,每个图平均节点数为14.30,平均连边数为14.69。
[0090]
(5)enzymes是从brenda酶数据库中提取的600个蛋白质三级结构的数据集。它包含6种酶,预测任务是对这6中酶进行分类。它包含600张图,分为六类酶,每个图平均节点数为32.63,平均连边数为124.20。
[0091]
将用于化合物性质预测的图结构优化方法与七种图神经网络方法进行比较:
[0092]
(1)gcn(图卷积神经网络);
[0093]
(2)gat(图注意力神经网络);
[0094]
(3)ssgc(基于马尔科夫图扩散核的图神经网络);
[0095]
(4)sage(使用图采样的图神经网络);
[0096]
(5)gin(图同构网络);
[0097]
(6)set2set(基于递归神经网络对所有节点进行编码的图池化方法);
[0098]
(7)asap(基于一种节点丢弃机制的图池化方法)。
[0099]
本实施例为了探究用于化合物性质预测的图结构优化方法的参数设置,设计实验对比同一参数在选择不同值时模型的表现,得到最好的参数设置;
[0100]
本实施例为了验证通勤时间距离用于化合物性质预测的可行性,设计实验将通勤时间距离应用于传统方法里的图同构网络,与它原本的性能进行比较,对比在多个数据集上的准确度;
[0101]
本实施例为了为了验证通勤时间距离对图神经网络的提升作用,设计实验将通勤时间距离作用于传统方法中的图卷积神经网络、图注意力神经网络、基于马尔科夫图扩散核的图神经网络、使用图采样的图神经网络、使用图采样的图神经网络、图同构网络、基于递归神经网络对所有节点进行编码的图池化方法、基于一种节点丢弃机制的图池化方法,对比它们在不同数据集上的准确度。
[0102]
用于化合物性质预测的图结构优化方法涉及的相关参数主要是邻域选择函数的topk值,它对算法的性能表现有较大影响,本实施例进行实验探究参数的最优设置。
[0103]
如图3所示,在mutag数据集中gcn、gin、asap三个模型的分类准确度随着topk值的改变而发生变化,对gin模型而言,topk值选取为10左右性能提升最大,而对gcn、asap模型而言,topk值选取为13左右性能提升最大。因此在同一个数据集上,对不同的模型选取的topk值可以是不一样的,以达到最大的提升效果。
[0104]
如图4所示,在ptc-mr数据集中gcn、gin、asap三个模型的分类准确度随着topk值的改变而发生变化,对gin模型而言,此时topk值依旧选取为10左右性能提升最大,而对gcn、asap模型而言,topk值选取为10左右性能提升最大。因此在不同的数据集上,同一个模型选取的topk值可以是不一样的,以达到最大的提升效果。
[0105]
其他情况下,参数的选择同理。
[0106]
从图5到图9可以观察到,gin模型与使用图结构优化方法在五个数据集上运行的准确度对比,准确度直接体现了模型的正确率,也是最能直观反应模型效果的一个指标。
[0107]
从这些图上可以看到,对gin模型而言,在五个生物化学数据集上,使用图结构优
化方法的准确度均比原模型的高。与原模型相比,使用图结构优化方法在mutag数据集上的准确度要高出1.11%;在d&d数据集上高出3.19%;在proteins数据集上高出3.67%;在ptc-mr数据集上高出1.77%;在enzymes数据集上高出1.45%。综上所述,图结构优化方法可以适配神经网络方法,且在化合物性质预测方面是有效的,同时从proteins、d&d、enzymes数据集上的结果反映了,图结构优化方法在化合物种类繁多、性质多样的情况下同样适用。
[0108]
从图5到图9可以观察到,七个神经网络模型与其使用通勤时间距离方法在五个数据集上运行的准确度对比,准确度直接体现了模型的正确率,也是最能直观反应模型效果的一个指标。
[0109]
从这些图上可以看到,在五个生物化学数据集上,使用图结构优化方法的准确度在大多数情况下可以比原模型高,尤其在mutag数据集上取得了比较大的性能提升,而在proteins和ptc-mr数据集上取得了比较有竞争力的结果。同时可以观察到,使用图结构优化方法的gcn模型仅在一半的数据集上比原模型有性能提升,这可能是图池化方法的选择影响了模型的准确度,未来可以探究不同的图池化方法与图结构优化方法的组合策略。综上所述,图结构优化方法对多数神经网络方法有性能提升,证明该方法的实用性很高,且在多个生物化学数据集中均在准确度这一指标上有突破,进一步证实了图结构优化方法的优化确实提升了模型的预测能力。
[0110]
应当理解的是,本说明书未详细阐述的部分均属于现有技术。
[0111]
应当理解的是,上述针对较佳实施例的描述较为详细,并不能因此而认为是对本发明专利保护范围的限制,本领域的普通技术人员在本发明的启示下,在不脱离本发明权利要求所保护的范围情况下,还可以做出替换或变形,均落入本发明的保护范围之内,本发明的请求保护范围应以所附权利要求为准。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1