一种分子靶点快速预测及关联机理分析方法
1.本发明涉及靶向治疗和基本蛋白质的识别技术领域,特别是涉及一种分子靶点快速预测及关联机理分析方法。
背景技术:
2.随着社会的发展,人类疾病的治疗观念正在发生根本的变化,既由经验科学向循证医学,由细胞攻击模式向靶向性治疗模式转变。靶向性治疗是在细胞水平上,针对已经明确的致癌位点进行专向性攻克,细胞内部的某些致癌蛋白分子就是靶向位点,致癌蛋白质分子就是细胞病变过程中的关键蛋白质。关键蛋白质的缺失将会导致蛋白质复合物的生物功能丧失和生物体死亡。因此关键蛋白质的鉴定研究,不仅有助于我们了解生物体活动的本质,也有助于发现人类疾病基因。蛋白质的鉴定及作用机理的可解析性研究已经成为生物信息界,医学界的热点,在当今的脑胶质瘤、肝癌、肺癌、胰腺癌等疾病的靶向治疗中得到了广泛应用。
3.随着可用医学信息量的增加,关键蛋白质的识别已成功引起了学者的关注。传统的生物实验方法也被相继提出,例如:基因敲除、rna干扰、条件敲除等,这些经典的生物实验方法对预测关键蛋白质做出了一定的贡献,但是实验过程耗时久,成本高,已不能满足生物医药快速发展的需要。随着高通量技术的进步,后基因组时代蛋白质-蛋白质相互作用数据快速积累,利用大量蛋白质相互作用数据构建蛋白质相互作用网络为理解蛋白质复合物、生物途径和细胞组织功能提供了新的视角。基于蛋白质相互作用网络的特点,研究人员提出了许多基于生物网络拓扑中心性的方法来识别关键蛋白质。
4.后基因组时代的到来,使得多数据预测关键蛋白质成为了可能。为了进一步提高关键蛋白质识别率,一些研究人员提出将网络拓扑特征与生物信息(基因表达数据、亚细胞位置数据、正畸数据、基因本体)相结合,构建预测模型,提高预测精度。这些方法在结合了蛋白质的生物信息和拓扑结构特性后,提高了关键蛋白质的识别精度。然而,通过融合生物特征信息和拓扑信息,构建完全特征数字优化模型预测关键蛋白质的方法属于组合优化算法,这是一个np困难问题。
5.启发式算法通过近似最优解能够快速求解np困难问题。元启发式算法具有高鲁棒性、低复杂性和优化性能好的特点,为解决组合优化问题、聚类和其他领域提供了新的视角。化学优化反应算法是一种新型演化计算技术,通过模拟化学反应中分子间的变化和能量迁移方式快速求解np困难问题。
6.关键蛋白质的鉴定对理解细胞的生命活动具有重要意义。随着后基因组时代的到来,生物医学领域里的数据量呈几何级数增长,研究者们更加关注这些多样化数据之间的复杂集成关系,分析蛋白质之间的关联机理,并快速识别生物体的大部分关键蛋白质靶点是接下来的一大挑战。
技术实现要素:
7.本发明通过提供一种分子靶点快速预测及关联机理分析方法,引入朴素贝叶斯概率模型分析蛋白质间的同质性,并建立蛋白质与蛋白质关联概率模型。随后,提出化学优化反应算法对网络模型进行搜索优化,同时依据蛋白质同质率对分子结构进行变异选择,平衡局部和全局优化策略,最终达到提高关键蛋白质识别精度和识别速度的目的。
8.为实现上述目的,本发明提供了如下方案:
9.一种分子靶点快速预测及关联机理分析方法,包括:
10.建立蛋白质与蛋白质关联概率模型,所述蛋白质与蛋白质关联概率模型用于分析蛋白质之间的关联机理;
11.将蛋白质与蛋白质交叠网络拓扑特征、基因表达数据、直系同源数据组合,获得分子靶点候选种群关键性评分依据,基于所述分子靶点候选种群关键性评分依据,构建分子靶点快速预测的化学反应优化模型;
12.基于所述化学反应优化模型与所述蛋白质之间的关联机理,平衡化学反应,优化局部与全局搜索策略,进行分子靶点快速预测及关联机理分析。
13.优选地,建立所述蛋白质与蛋白质关联概率模型,包括:
14.结合蛋白质与蛋白质交叠网络拓扑特征和蛋白质生物特征,通过朴素贝叶斯模型求解蛋白质节点间的同质率epri,分析蛋白质之间的关联机理,构建所述蛋白质与蛋白质关联概率模型。
15.优选地,分析所述蛋白质之间的关联机理,包括:
16.获取蛋白质与蛋白质之间的特征集,通过关键蛋白质节点与邻域蛋白质节点相互作用的局部性,以及关键蛋白质节点之间的独立性,基于所述蛋白质与蛋白质关联概率模型,分析蛋白质之间的关联机理,其中,所述同质率epr
i,j
越大,表示两个蛋白质节点之间的同质性越高,所述关联机理即所述同质性。
17.优选地,所述蛋白质与蛋白质之间的特征集为w={w1,w2,w3};
18.其中,w1表示蛋白质与蛋白质所构成的边聚集系数ecc,w2表示蛋白质的基因表达系数pcc,3表示蛋白质的直系同源相关性nos。
19.优选地,获得所述分子靶点候选种群关键性评分依据,包括:
20.使用蛋白质的基因表达系数pcc来捕获蛋白质及蛋白质邻域的共表达特性,通过边聚集系数ecc捕获蛋白质的高连通性和集聚类特征,并使用同源相关性nos来捕获蛋白质进化过程中的保守性,获得蛋白质之间的相关性,基于所述蛋白质之间的相关性,得到所述分子靶点候选种群关键性评分依据。
21.优选地,所述分子靶点候选种群关键性评分依据为:
[0022][0023]
其中,ow表示当ecc(vi)=0条件下的关键性评分依据,vi与vj分别表示第i个和第j个蛋白质节点,n表示蛋白质数量,ecc(vi)为捕获蛋白质聚类特征的参数,nos(vj)表示捕获蛋白质同源相关性参数,pcc(vi,vj)表示蛋白质与其邻域的共表达特性参数。
[0024]
优选地,若所述捕获蛋白质聚类特征的参数时,所述关键性评分依据ow为:
[0025][0026]
其中,vi与vj分别表示第i个和第j个蛋白质节点,n表示蛋白质数量。
[0027]
优选地,构建分子靶点快速预测的化学反应优化模型,包括:
[0028]
通过模拟化学反应优化算法中分子参与四种初等反应的过程,构建所述分子靶点快速预测的化学反应优化模型,从局部和全局搜索关键节点,进而达到分子靶点快速预测的目的;其中,所述四种初等反应包括:单分子无效碰撞反应,单分子分解反应,多分子间无效碰撞反应,多分子间结合反应。
[0029]
优选地,基于所述化学反应优化模型与所述蛋白质之间的关联机理,将朴素贝叶斯模型所得到的蛋白质同质率作为分子变异的选择条件,平衡化学反应,优化局部与全局搜索策略。
[0030]
本发明的有益效果为:
[0031]
本发明将朴素贝叶斯概率模型及化学优化反应算法引入到生物信息学的关键蛋白质分析和预测过程中,针对现有的关键蛋白质识别技术对生物意义及生物功能关联关系挖掘不够深这一缺陷引入朴素贝叶斯概率模型分析蛋白质分子之间的关联机理,为更深入开展分子靶点功能应用研究提供了新的思路。同时,与具有高假阳性和假阴性的识别技术不同的是,本技术通过引入化学反应优化算法从局部和全局进行搜索提高了分子靶点的识别速度和精度,降低了假阴性和假阳性。
附图说明
[0032]
为了更清楚地说明本发明实施例或现有技术中的技术方案,下面将对实施例中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图仅仅是本发明的一些实施例,对于本领域普通技术人员来讲,在不付出创造性劳动性的前提下,还可以根据这些附图获得其他的附图。
[0033]
图1为本发明实施例中蛋白质与蛋白质关联概率模型示意图;
[0034]
图2为本发明实施例中分子靶点快速预测的化学反应优化流程图。
具体实施方式
[0035]
下面将结合本发明实施例中的附图,对本发明实施例中的技术方案进行清楚、完整地描述,显然,所描述的实施例仅仅是本发明一部分实施例,而不是全部的实施例。基于本发明中的实施例,本领域普通技术人员在没有做出创造性劳动前提下所获得的所有其他实施例,都属于本发明保护的范围。
[0036]
为使本发明的上述目的、特征和优点能够更加明显易懂,下面结合附图和具体实施方式对本发明作进一步详细的说明。
[0037]
本发明提供一种分子靶点快速预测及关联机理分析方法,具体包括:
[0038]
s1、将蛋白质与蛋白质交叠(ppi)网络拓扑特征与蛋白质生物特征(基因表达、直
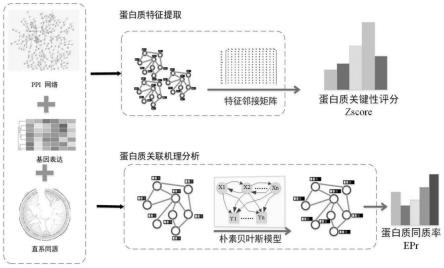
系同源)相结合,引入朴素贝叶斯模型分析蛋白质间的同质性,构建蛋白质与蛋白质关联概率模型,从而分析蛋白质之间的关联机理,如图1。
[0039]
s2、将蛋白质与蛋白质交叠网络拓扑特征、基因表达数据、直系同源数据组合形成分子靶点候选种群关键性评分,并作为化学反应优化模型中分子的势能,构建分子靶点快速预测的化学反应优化模型。
[0040]
s3、基于化学优化反应算法的基础上,将朴素贝叶斯模型所得到的蛋白质同质率作为分子变异的选择条件,去平衡化学优化反应局部与全局搜索策略,如图2。
[0041]
进一步,所述步骤s1包括,
[0042]
s101、具有n个蛋白质相互作用的ppi网络表示为无向图g=(v,e,w),其中顶点集v={v1,
……
,vn}表示某生物蛋白质的集合,w表示蛋白质的特征集。边集e表示蛋白质与蛋白质之间的关联。若无向图g=(v,e,w),vi∈v(g),vj∈v(g),(i,j=1,
……
,n)存在,则
[0043][0044]
其中,表示蛋白质节点vi与vj之间存在相互作用。
[0045]
s102、令w={w1,w2,w3}为蛋白质与蛋白质之间的特征集,其中w1表示蛋白质与蛋白质所构成的边聚集系数ecc,w2表示蛋白质的基因表达系数pcc,w3表示蛋白质的直系同源相关性nos。进一步可分解为:
[0046]
s1021、边聚集系数ecc能够描述一条边的两个连接节点与其周围其他节点之间的紧密程度,可表达为:
[0047][0048]
其中,是指相互作用的边在图g中形成的实际的三角形的数量,表示取蛋白质节点vi与vj度d的最小值。可见,当ecc值越大,该蛋白质与邻域蛋白质在同一簇的概率越大。
[0049]
s1022、令exp(vi)={exp(vi,1),exp(vi,2),
…
,exp(vi,n)}表示蛋白质节点vi在不同时刻的表达,其中exp(vi,n)表示蛋白质vi在第n时刻的基因表达值。采用皮尔逊系数来描述蛋白质节点vi和vj的基因关联关系则
[0050][0051]
其中,表示蛋白质节点vi在所有时刻的基因表达均值,σ(vi)表示vi在各个时刻基因表达量的标准方差。当为正值时,则蛋白质节点vi与vj正相关;如果值为负,则蛋白质节点vi与vj呈负相关。
[0052]
s1023、令os(vi)表示蛋白质节点vi在参考物种集s中具有同源物的数量。则对于蛋白质节点vi,其直系同源相关性nos(vi)被定义为:
[0053][0054]
max
os
表示g中所有蛋白质节点的os值的最大值。如果蛋白质节点vi与s物种集中的
所有生物都存在同源关系,则同源得分为1。相反,如果蛋白质vi没有和任何生物存在同源关系,同源得分为0。
[0055]
s103、针对关键蛋白质节点与邻域蛋白质节点相互作用的局部性,及部分关键蛋白质节点之间的独立性,引入朴素贝叶斯模型分析蛋白质节点之间的同质性,从而求解蛋白质同质率。
[0056]
给定蛋白质训练集(x,y)其中每个样本x和y都包括特征w={w1,w2,w3}。每个样本的先验概率通过训练样本特征w可以统计计算出来,p(y=vi)表示第i个蛋白质的先验概率。p(y=vi)可表示为:
[0057][0058]
p(x=vj|y=vi)表示第i个蛋白质的后验概率,同时代表蛋白质节点vi与vj的同质率。p(x=vj|y=vi)由下式给出:
[0059][0060]
由(5)式(6)式结合可得:
[0061][0062]
其中,当假设特征条件独立时,样本联合概率由各自的局部条件概率分布相乘求,得:
[0063][0064]
由(5)式到(8)可得蛋白质的同质率公式为:
[0065][0066]
进一步,所述步骤s2包括,
[0067]
s201、化学反应优化是多代理算法,操作代理是分子,即描述所求问题的解。每个蛋白质候选集表示一个分子结构ω,即每个候选集包含m个蛋白质(m≤n),每个蛋白质为潜在的基本蛋白质。分子结构ω表达为:
[0068]
ω={v1,v2,
…
,vm}
ꢀꢀ
(10)
[0069]
初始种群pop可表示为
[0070]
pop∈{ω
γ
|γ=1,2,3,
…
,popsize}
ꢀꢀ
(11)
[0071]
s202:化学反应是一个自然的不稳定物质转化到稳定的过程。它的转化以最优的能量来支持它们的存在,以动能ke和势能pe转化为主。其中分子的动能ke为分子的分解和化合提供能量,动能越高,越容易产生分解和化合反应,从而避免算法陷入局部最优,设每个分子的初始动能为0。势能pe代表关键蛋白质的关键性评分,即各个分子中蛋白质之间的关键性。
[0072]
第γ个化学分子在反应过程中势能表达式为:
[0073]
[0074]zscore
(vi)表示第γ个化学分子结构中m个蛋白质关键性评分依据和。
[0075]
s203、使用皮尔逊系数pcc来捕获蛋白质及其邻域的共表达特性,使用边距集系数ecc来捕获蛋白质的高连通性和集聚类特征,并使用同源相似性评分nos来捕获蛋白质进化过程中的保守性,蛋白质之间的关键性评分依据和可表示为:
[0076][0077]
其中,ow表示其他情况下关键性评分依据,其值由s204给出。
[0078]
s204、在ppi网络中与邻居蛋白质关联越频繁的蛋白质成为关键蛋白质的可能性越大,但是独立存在网络中的蛋白质也有可能是关键蛋白质。ecc是捕获蛋白质聚类特征的重要参数,当其值为0时,表示该蛋白质在网络中的连通性较低甚至没有。为了减小该参数缺失带来的影响,的条件下,关键性评分依据ow为:
[0079][0080]
进一步,所述步骤s3包括,
[0081]
s301:单分子无效碰撞反应
[0082]
单分子无效碰撞指单个分子在独立空间内进行碰撞并得到一个邻域分子的过程。根据蛋白质同质率,从原始蛋白质集里选择k(k∈[1,m])个蛋白质替换反应前分子结构ω
γ
中的k个蛋白质,从而得到一个新的分子结构ω
′
γ
,替换过程如下式:
[0083]
ω
′
γ
=ek(ω
γ
)
ꢀꢀꢀ
(15)
[0084]
其中,替换过程应满足epr
′i≥epri(i=1,2,
…
,n),epri和epr
′i分别表示替换前和替换后的蛋白质同质率,rk(*)表示选择替换算子。
[0085]
第γ个分子发生碰撞应满足的条件为:
[0086][0087]
其中,ω
′
γ
表示反应后分子结构,和分别为ω
γ
的势能和
[0088]
动能,pe
ω
′
和ke
ω
′
分别为ω
γ
的势能和动能。
[0089]
根据能量守恒定律,可以得到反应后分子的动能ke计算公式为:
[0090][0091]
式中,α为动能损失率,α∈[0,1]。损失的部分动能被存储在系统中央缓冲区中,为其他分子的分解或者合成提供能量。
[0092]
s302、单分子分解反应
[0093]
分解是指单个分子在独立空间内进行碰撞并分解多个分子的过程(假设分解成两个分子)。在ω
γ
的基础上,从初始蛋白质候选集里选择k个蛋白质代替反应前分子结构ω
γ
中的k个蛋白质,获得ω
′1结构。同时选择m-k个蛋白质替换ω
γ
中未被选择蛋白质,获得ω
′2。替换过程如下式:
[0094]
ω
′1=rk(ω
γ
)
ꢀꢀ
(18)
[0095]
ω
′2=r
m-k
(ω
γ
)
ꢀꢀꢀꢀ
(19)
[0096]
其中,替换过程应满足epr
′i≥epri(i=1,2,
…
,n),r
m-k
(
·
)表示选择替换算子将选择m-k个蛋白质。
[0097]
第γ个分子发生分解应满足的条件为:
[0098][0099]
其中,ω
′1和ω
′2是分解反应后的两个新分子结构。
[0100]
根据能量守恒定律,可以计算新分子的动能和假设则:
[0101][0102][0103]
其中,q是指属于区间[0,1]的随机数。
[0104]
由于分解过程需要非常大的动能支持,当反应过程中分子自身产生的动能不足以支持反应发生时,那么它的分解能量来源于中央缓冲区buffer。反应满足:
[0105][0106]
当满足公式(23)时,两个新分子的动能可以被表示为:
[0107][0108][0109]
其中,m1、m2、m3、m4是[0,1]的随机数。
[0110]
s303、多分子间无效碰撞反应
[0111]
分子间无效碰撞指多个分子(假设两个分子)相互碰撞,生成相同数量新分子的过程。即ω1+ω2→
ω
′1+ω
′2。生成新分子的过程如下:
[0112]
ω
′1=rk(ω1)
ꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(26)
[0113]
ω
′2=rk(ω2)
ꢀꢀꢀꢀꢀꢀꢀꢀꢀ
(27)
[0114]
其中,上述替换过程满足
[0115]
多分子反应满足的条件为:
[0116][0117]
其中,ω
′1和ω
′2是碰撞反应后的分子结构。
[0118]
根据能量守恒定律,可以计算新分子的动能和和则:
[0119][0120][0121]
其中,q为区间[0,1]的随机数。
[0122]
s304、多分子间合成反应
[0123]
合成指多个分子(假设两个分子ω1和ω2)相互碰撞,并融合在一起的过程。新分子生成过程如下:
[0124]
ω
′
γ
=rk(ω1)+rk(ω2)
ꢀꢀꢀꢀ
(31)
[0125]
其中,替换替换过程满足epr
′i≥epri(i=1,2,
…
,n)。
[0126]
合成反应发生的条件为:
[0127][0128]
其中,ω
′
γ
是碰撞反应后的分子结构。
[0129]
新分子的动能可计算为:
[0130][0131]
本发明将朴素贝叶斯概率模型及化学优化反应算法引入到生物信息学的关键蛋白质分析和预测过程中,提出了一种融合化学反应优化模型及朴素贝叶斯模型的分子靶点快速预测及关联机理分析方法。通过朴素贝叶斯概率模型分析蛋白质之间的关联机理,通过化学优化反应算法加快了关键蛋白质识别速度。引入dip、gavin生物信息数据集进行验证。本发明的实验结果显示,能够平衡启发式算法的局部和全局搜索能力,快速获取全局优解;也能够解析关键蛋白质之间的作用机理,在预测精度方面也优于现有的经典预测方法。
[0132]
以上所述的实施例仅是对本发明优选方式进行的描述,并非对本发明的范围进行限定,在不脱离本发明设计精神的前提下,本领域普通技术人员对本发明的技术方案做出的各种变形和改进,均应落入本发明权利要求书确定的保护范围内。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1