miRNA靶基因预测及其模型训练的方法、系统和存储介质与流程
本技术涉及生物信息学,更具体地,涉及一种mirna靶基因预测及其模型训练的方法、系统和存储介质。
背景技术:
1、microrna(以下简称“mirna”)是一类由内源基因编码的非编码单链rna分子,其长度一般约为18-25nt。在生物发展进程中,mirna具有重要的调节作用。mirna主要通过参与基因的转录后调控实现对靶基因表达的负调节,具体作用方式主要为翻译抑制(常见于动物)和降解靶基因(常见于植物)。mirna在肿瘤发生发展、生物发育、器官形成、病毒防御、表观调控以及代谢等方面起着极其重要的调控作用。了解mirna调控的靶基因对于肿瘤防治以及其他疾病诊疗具有重要意义。
2、然而,mirna具有非常复杂的调控网络,往往一个mirna可以调控多种靶基因,而同一个靶基因也可以有多个mirna来进行调节。目前业内广泛使用miranda、rnahybrid、pita、targetscan等软件预测mirna的靶基因。这类软件的思路是计算mirna和靶基因的互补配对情况,并进一步根据mirna和靶基因结合的热力学稳定性来判断该靶基因是否是与mirna互作的基因。虽然这类软件在预测过程中考虑了mirna特定序列和靶基因的碱基互补情况、靶基因非翻译区的跨物种保守性以及mirna和靶基因二聚体热力学稳定性,并且这类方法可以用于任何物种,计算量相对较小,然而,这类软件的算法本身并未真正反映出mirna和靶基因的互作生物学机制。因此,这类软件对于mirna靶基因预测的准确率往往不足40%。这大幅增加了后期验证的工作量,因此时间成本和经济成本并不低廉。此外,现有的mirna靶基因预测软件(例如miranda、pita、rnahybrid等)通常需要用户在进行靶基因预测时输入热力学能量阈值、得分阈值、折叠utr需要考虑的目标上下游位置等信息,这些信息一方面对于预测结果具有一定的偏好干扰(尤其是在当前没有真正理解mirna与靶基因作用机制的情况下),另一方面也带来了一些使用上的不便利。因此,市场上需要一种相对高效、准确、便捷的mirna靶基因预测方法。
技术实现思路
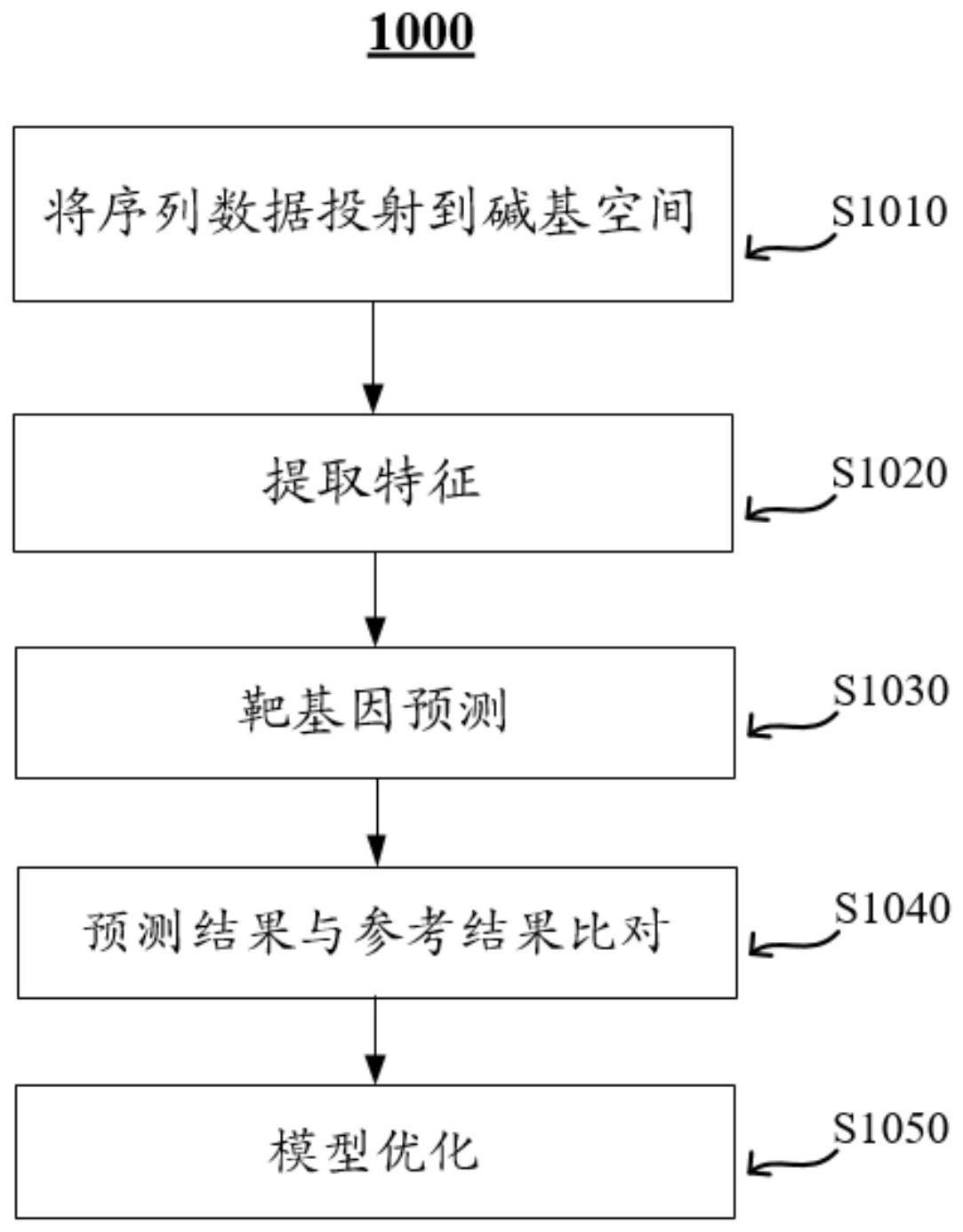
1、本技术提供了一种mirna靶基因预测模型的训练方法,包括:将由mirna的序列和mrna的序列拼接而成的输入数据投射到a碱基空间、u碱基空间、g碱基空间和c碱基空间,从而获得a碱基向量、u碱基向量、g碱基向量和c碱基向量;利用mirna靶基因预测模型的四级依次连接的卷积层、激活层和池化层从所述a碱基向量、所述u碱基向量、所述g碱基向量和所述c碱基向量提取特征张量;将所提取的特征张量输入全连接层以获得预测结果;将所获得的预测结果与参考结果进行比对;以及基于比对的结果优化所述mirna靶基因预测模型。
2、根据本技术实施方式,所述mirna的序列和所述mrna的序列包括阳性训练集中彼此互作的mirna的序列和mrna的序列以及阴性训练集中随机生成的mirna的序列和mrna的序列,其中:所述阳性训练集中彼此互作的mirna的序列和mrna的序列是从encori、mirdb、mirtarbase、mirnet、mirwalk中至少一个数据库中提取的经低通量实验验证的、彼此互作的mirna的序列及对应的mrna的序列;所述阴性训练集中随机生成的mirna的序列和mrna的序列是随机生成的序列,并且所述随机生成的序列排除了所述阳性训练集中彼此互作的mirna的序列和mrna的序列以及经miranda、rnahybrid和pita预测互作的mirna的序列和mrna的序列。
3、根据本技术实施方式,所述阳性训练集中的彼此互作的mirna的序列和mrna的序列的对数与所述阴性训练集中随机生成的mirna的序列和mrna的序列的对数相同。
4、根据本技术实施方式,所述四级依次连接的卷积层、激活层和池化层包括:具有16个4通道卷积核、relu激活函数和窗口为2的最大池化层的第一级;具有32个16通道卷积核、relu激活函数和窗口为2的最大池化层的第二级;具有64个32通道卷积核、relu激活函数和窗口为2的最大池化层的第三级;具有128个64通道卷积核、relu激活函数和窗口为2的最大池化层的第四级。
5、根据本技术实施方式,将所提取的特征张量输入全连接层以获得预测结果包括:将所提取的特征张量降维成一维的第一向量;将所述第一向量随机剪除一半的元素,形成第二向量;将所述第二向量输入到以relu作为激活函数的第一全连接层以获得第三向量;将所述第三向量随机剪除一半的元素,形成第四向量;将所述第四向量输入到以sigmoid作为激活函数的第二全连接层以获得所述预测结果。
6、根据本技术实施方式,所述基于比对的结果优化所述mirna靶基因预测模型包括:以二分类交叉熵为损失函数,使用adam优化函数来修正所述mirna靶基因预测模型各层的参数。
7、本技术还提供了一种mirna靶基因预测模型的训练系统,包括:存储器,所述存储器存储可执行指令;以及一个或多个处理器,所述一个或多个处理器与所述存储器通信以执行所述可执行指令从而完成以下操作:将由mirna的序列和mrna的序列拼接而成的输入数据投射到a碱基空间、u碱基空间、g碱基空间和c碱基空间,从而获得a碱基向量、u碱基向量、g碱基向量和c碱基向量;利用mirna靶基因预测模型的四级依次连接的卷积层、激活层和池化层从所述a碱基向量、所述u碱基向量、所述g碱基向量和所述c碱基向量提取特征张量;将所提取的特征张量输入全连接层以获得预测结果;将所获得的预测结果与参考结果进行比对;以及基于比对的结果优化所述mirna靶基因预测模型。
8、本技术还提供了一种用于mirna靶基因预测模型的训练的计算机可读存储介质,其特征在于,所述计算机可读存储介质存储可执行指令,所述可执行指令能够被一个或多个处理器执行以完成以下操作:将由mirna的序列和mrna的序列拼接而成的输入数据投射到a碱基空间、u碱基空间、g碱基空间和c碱基空间,从而获得a碱基向量、u碱基向量、g碱基向量和c碱基向量;利用mirna靶基因预测模型的四级依次连接的卷积层、激活层和池化层从所述a碱基向量、所述u碱基向量、所述g碱基向量和所述c碱基向量提取特征张量;将所提取的特征张量输入全连接层以获得预测结果;将所获得的预测结果与参考结果进行比对;以及基于比对的结果优化所述mirna靶基因预测模型。
9、本技术还提供了一种mirna靶基因的预测方法,包括:将目标mirna序列和目标mrna序列输入到基于现有数据库整合的mirna靶基因数据库,以查询所述目标mirna序列与所述目标mrna序列是否互作;响应于在所述mirna靶基因数据库中未查询到所述目标mirna序列与所述目标mrna序列互作,将所述目标mirna序列与所述目标mrna序列输入到根据本技术提供的训练方法训练完成的mirna靶基因预测模型,以预测所述目标mirna序列与所述目标mrna序列是否互作。
10、根据本技术实施方式,基于现有数据库整合的mirna靶基因数据库包括:从encori、mirdb、mirtarbase、mirnet、mirwalk中至少一个数据库中提取的经低通量实验验证的、彼此互作的mirna的序列及对应的mrna的序列;以及通过预定阈值筛选后的经高通量测序验证的mirna的序列及对应的mrna的序列。
11、本技术提供的mirna靶基因预测模型的训练方法创新性地将mirna序列和mrna序列的互作判断转化成类图像处理的计算任务,从而可以利用卷积神经网络(cnn)来进行mirna靶基因预测的任务,从而提供了一种更高效、准确、便捷的mirna靶基因预测方案。
- 还没有人留言评论。精彩留言会获得点赞!