基于蛋白三维结构和图神经网络的免疫原预测系统及方法
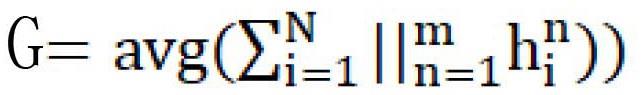
本发明公开了一种基于蛋白三维结构和图神经网络的免疫原预测系统和方法,属于生物医药和生物信息学。
背景技术:
1、免疫原决定了疫苗所诱导免疫应答攻击的靶向性,是新型疫苗研发的决定性因素。传统的疫苗免疫原鉴定方法流程长、效率低,已经不能满足新型疫苗的快速研制需求。反向疫苗学(reverse vaccinology)为寻找复杂病原体新型免疫原提供了全新手段,主要方法是基于组学研究发现,对大量的数据进行计算分析和预测,筛选靶标抗原并验证保护性反应(rappuoli r. (2000) curr. opin. microbiol., 3, 445–450.)。目前该方法已在脑膜炎奈瑟菌、金黄色葡萄球菌等复杂病原体疫苗研制中得到成功应用(pizza m. (2000)science (80-), 287, 1816–1820.)。相应的多种反向疫苗学免疫原预测方法及软件系统陆续被开发出来,以第一个本地化免疫原发现系统nerve和第一个在线免疫原发现系统vaxign为代表,主要采用基于规则的过滤方法,依照预定的步骤对蛋白特性(如蛋白亚细胞定位、分子量大小、粘附性、毒力概率等)进行分析,符合规则的蛋白通过过滤进入下一阶段,直至筛选出目的免疫原(vivona s. (2006) bmc biotechnol., 6;35.;he y. (2010)j. biomed. biotechnol., 1;15.)。
2、随着人工智能的迅速发展,基于机器学习的分类模型已逐步应用于反向疫苗学免疫原预测发现领域。代表性的如darren等人基于一维氨基酸序列注释获得的45个理化参数特征,采用偏最小二乘法(da-pls)算法建立了vaxijen方法(doytchinova i.a., (2007)bmc bioinformatics, 8;4.);zai等人基于一维氨基酸序列注释获得的6个核心生物学特征,采用投票算法等建立了mppa-ml方法(zai x, (2021)vet res 52;75.);he等人基于一维氨基酸序列注释获得的509个生物学特征和理化参数特征,采用支持向量机(svm)等算法建立了vaxign-ml方法(he y, (2020) bioinformatics,36;10:3185–3191.);以上方法相比于传统基于规则的过滤方法,在免疫原预测的准确性上得到较大改善。近期,基于transformer的无监督的蛋白质语言模型esm-2相比于传统算法显示出较好的提取一维氨基酸序列特征的能力(rives a, (2021) proc natl acad sci u s a.118(15):e2016239118.)。
3、但是,目前以上所有方法均需要从一维氨基酸序列出发,采用大量生物信息学软件进行蛋白质生物学特征和理化参数特征的注释,过程极其繁琐复杂(dalsass m,(2019).front immunol,14;10:113)。同时,针对不同类型病原体如细菌、病毒、寄生虫等,蛋白质的生物学特征注释方法和软件各异,模型广泛适用性较差。此外,目前方法预测免疫原范围仍然较广,免疫评价验证较为困难,有待进一步提升方法预测准确性。
4、蛋白质的三维结构最终决定了其生物学特征和理化参数特征,具有远超一维氨基酸序列的信息丰度。本发明的目的是建立一种基于三维结构特征的免疫原预测新方法,实现普适(适用于细菌、病毒、寄生虫等)、高精度的免疫原预测。
技术实现思路
1、本发明旨在于克服现有反向疫苗学仅基于一维氨基酸序列特征开展免疫原预测技术的缺陷,提供一种全新的基于蛋白三维结构和图神经网络的免疫原高精度预测系统和方法。
2、蛋白质的三维结构最终决定了其生物学特征和理化参数特征,具有远超一维氨基酸序列的信息丰度。基于蛋白质三维结构特征建立免疫原的机器学习分类预测模型,有望突破已有仅基于一维氨基酸序列模型的局限。
3、基于蛋白质三维结构特征建立免疫原预测模型,首先需要解决如何从蛋白结构中提取有效的特征向量。通过实验测定以及alphafold2等软件预测获得的蛋白质三维结构通常以pdb文件格式存储。pdb文件本质是一种ascii码文本文件,其中包含构成蛋白质的氨基酸各个原子的坐标等信息。这种文本文件格式的结构数据难以直接进行特征提取和机器学习模型建立。
4、蛋白质的三维结构由氨基酸组成,氨基酸之间由化学键形成连边。因此,可以将蛋白质三维结构看作一个“图”(graph),其中氨基酸是节点,化学键是边。目前捕获蛋白质三维结构的方法有很多,包括将蛋白质的三维结构抽象成3d栅格图,或者将蛋白质在三维空间的结构抽象成一维或者二维结构之后再做分析。然而,蛋白质的三维结构是由原子组成,原子之间又由化学键形成连边,本质上是一个“图”(graph),因此,本发明选择使用图神经网络(gnn,graphic nuaral network)作为处理蛋白质三维结构特征的工具,建立了一种基于蛋白三维结构和图神经网络的免疫原预测系统。
5、所述系统具体包括以下模块:
6、(1)三维结构特征提取模块:使用一种针对免疫原结构特点设计的图神经网络模型表征并提取蛋白质三维结构特征,通过对蛋白质三维结构pdb数据库进行预训练学习,获得蛋白质中氨基酸在三维空间中相互作用的特征表示;
7、(2)免疫原结构数据集处理模块:收集病原体已知免疫原作为正样本集,病原体蛋白数据库中随机抽取与已知免疫原非同源蛋白的非免疫原作为负样本集,通过结构预测软件获得蛋白质三维结构pdb文件,形成首个免疫原结构数据集和非免疫原结构数据集,将结构信息输入上述预训练好的图神经网络模型,提取得到三维结构对应的特征向量;
8、(3)机器学习分类器模块:对特征向量进行降维后,联合一维氨基酸序列特征,采用机器学习算法进行分类模型训练,得到训练好的免疫原分类器,基于测试集对模型预测准确性进行评估;
9、(4)自动预测输出模块:输入待预测病原体全部注释蛋白三维结构信息和一维氨基酸序列,应用上述预训练好的图神经网络模型与免疫原分类器,自动输出候选免疫原列表。
10、在一个优选的实施方案中,在模块(1)中所述蛋白结构数据库包括但不限于pdb(protein data bank)、alphafold protein structure database数据库。
11、在另一个优选的实施方案中,在模块(1)中所述优化的改进的图神经网络模型具体为改进型邻域增强神经网络模型(negcn,neighbor enhanced graph convolutionalnetwork)。基于蛋白质中氨基酸的三维空间分布,negcn分别依据氨基酸之间的空间距离、氨基酸的链式连接、氨基酸之间的最邻近距离等关系构建邻接图。然后,将氨基酸邻接图转换为基于氨基酸之间作用力的边图,同时根据不同作用力边在空间的向量方向进行方向敏感的消息传递。在本发明的一个具体实施方案中,本发明首次将边的类型按照空间位置分为类,更为优选的,实验中为8,即把空间按照x,y,z轴划分成8个象限,更好的区分了三维空间中不同邻边所起到的不同作用。最后,基于对比学习机制,获得蛋白质中氨基酸在三维空间中相互作用的特征表示,包括但不限于氨基酸的空间位置、不同氨基酸之间的相互作用、氨基酸理化性质。更为优选的,获得4层神经网络并分别输出[512,256,128,64]维度向量,共计960维度。该模型针对免疫原蛋白质的结构特点进行了上述的具有独特技术特征的设计,相比于已有的negcn邻域增强神经网络模型具有增强的蛋白质三维结构表征能力。
12、在另一个优选的实施方案中,在模块(2)中所述病原体是指可对人和/或动物宿主致病的细菌类、病毒类或寄生虫类微生物。
13、在另一个优选的实施方案中,在模块(2)中所述已知免疫原指经实验验证可在宿主体内激发有效免疫保护的病原体蛋白质成分,已知免疫原的收集方式包括整合数据库筛选、文献调研以及实验发现,数据库包括但不限于iedb databse、antigen databse、protegen databse;所述非免疫原指尚没有实验证据证实可在宿主体内激发有效免疫保护的病原体蛋白质成分,收集方式为,从uniprot蛋白数据库中下载所有病原体全部蛋白质序列,采用随机抽取方式从中获得候选蛋白质,通过基于局部比对算法的搜索工具(例如blast,basic local alignment search tool)排除与已知免疫原同源序列后,建立非免疫原数据集,即负样本集。
14、在另一个优选的实施方案中,在模块(2)中所述结构预测软件包括但不限于alphafold2、rosettafold、esmfold。
15、在另一个优选的实施方案中,在模块(3)中所述一维氨基酸序列特征的提取由包括但不限于蛋白质语言模型esm-2进行,本领域技术人员应用技术常识使用现有技术其它的一维氨基酸序列特征提取工具也可以完成一维氨基酸序列特征的提取。
16、在另一个优选的实施方案中,在模块(3)中所述降维方法包括但不限于主成分分析(pca),过大的三维结构特征维数会导致过拟合,还会导致模型参数和复杂度的增加,更为优选的,将图神经网络提取的960维特征向量降维至20至100维,可获得最优的机器学习模型性能。
17、在另一个优选的实施方案中,在模块(3)中所述分类模型算法包括但不限于极端梯度提升(xgboost)进行模型训练,得到训练好的分类器。
18、其次,本发明还提供了基于上述的一种基于蛋白三维结构和图神经网络的免疫原预测系统的免疫原预测方法,所述方法具体包括以下步骤:
19、(1)三维结构特征提取步骤:使用一种针对免疫原结构特点设计的图神经网络模型表征并提取蛋白质三维结构特征,通过对蛋白质三维结构pdb数据库进行预训练学习,获得蛋白质中氨基酸在三维空间中相互作用的特征表示;
20、(2)免疫原结构数据集处理步骤:收集病原体已知免疫原作为正样本集,病原体蛋白数据库中随机抽取与已知免疫原非同源蛋白的非免疫原作为负样本集,通过结构预测软件获得蛋白质三维结构pdb文件,形成首个免疫原结构数据集和非免疫原结构数据集,将结构信息输入上述预训练好的图神经网络模型,提取得到三维结构对应的特征向量;
21、(3)机器学习分类器步骤:对特征向量进行降维后,联合深度学习模型esm-2提取得到的一维氨基酸序列特征,采用机器学习算法进行分类模型训练,得到训练好的免疫原分类器,基于测试集对模型预测准确性进行评估;
22、(4)自动预测输出步骤:输入待预测病原体全部注释蛋白三维结构信息,应用上述预训练好的图神经网络模型与免疫原分类器,自动输出候选免疫原列表。
23、在一个优选的实施方案中,在步骤(1)中所述蛋白结构数据库包括但不限于pdb(protein data bank)、alphafold protein structure database数据库。
24、在另一个优选的实施方案中,在步骤(1)中所述优化的改进的图神经网络模型具体为改进型邻域增强神经网络模型(negcn,neighbor enhanced graph convolutionalnetwork)。基于蛋白质中氨基酸的三维空间分布,negcn分别依据氨基酸之间的空间距离、氨基酸的链式连接、氨基酸之间的最邻近距离等关系构建邻接图。然后,将氨基酸邻接图转换为基于氨基酸之间作用力的边图,同时根据不同作用力边在空间的向量方向进行方向敏感的消息传递。在本发明的一个具体实施方案中,本发明首次将边的类型按照空间位置分为类,更为优选的,实验中为8,即把空间按照x,y,z轴划分成8个象限,更好的区分了三维空间中不同邻边所起到的不同作用。最后,基于对比学习机制,获得蛋白质中氨基酸在三维空间中相互作用的特征表示,包括但不限于氨基酸的空间位置、不同氨基酸之间的相互作用、氨基酸理化性质。更为优选的,获得4层神经网络并分别输出[512,256,128,64]维度向量,共计960维度。该模型针对免疫原蛋白质的结构特点进行了上述的具有独特技术特征的设计,相比于已有的negcn邻域增强神经网络模型具有增强的蛋白质三维结构表征能力。
25、在另一个优选的实施方案中,在步骤(2)中所述病原体是指可对人和/或动物宿主致病的细菌类、病毒类或寄生虫类微生物。
26、在另一个优选的实施方案中,在步骤(2)中所述已知免疫原指经实验验证可在宿主体内激发有效免疫保护的病原体蛋白质成分,已知免疫原的收集方式包括整合数据库筛选、文献调研以及实验发现,数据库包括但不限于iedbdatabse、antigen databse、protegendatabse;所述非免疫原指尚没有实验证据证实可在宿主体内激发有效免疫保护的病原体蛋白质成分,收集方式为,从uniprot蛋白数据库中下载所有病原体全部蛋白质序列,采用随机抽取方式从中获得候选蛋白质,通过基于局部比对算法的搜索工具(blast,basic local alignment search tool)排除与已知免疫原同源序列后,建立非免疫原数据集,即负样本集。
27、在另一个优选的实施方案中,在步骤(2)中所述结构预测软件包括但不限于alphafold2、rosettafold、esmfold。
28、在另一个优选的实施方案中,在步骤(3)中所述一维氨基酸序列特征的提取由包括但不限于蛋白质语言模型esm-2进行,本领域技术人员应用技术常识使用现有技术其它的一维氨基酸序列特征提取工具也可以完成一维氨基酸序列特征的提取。
29、在另一个优选的实施方案中,在步骤(3)中所述降维方法包括但不限于主成分分析(pca),过大的三维结构特征维数会导致过拟合,还会导致模型参数和复杂度的增加,更为优选的,将图神经网络提取的960维特征向量降维至20至100维,可获得最优的机器学习模型性能。
30、在另一个优选的实施方案中,在步骤(3)中所述分类模型算法包括但不限于极端梯度提升(xgboost)进行模型训练,得到训练好的分类器。
31、本发明是人工智能算法在新型疫苗免疫原发现领域的创新应用,其核心思想是提出一种基于蛋白三维结构和图神经网络的免疫原高精度预测方法,克服了传统仅基于一维氨基酸序列特征开展机器学习分类导致的局限,实现普适(适用于细菌、病毒、寄生虫等)、高精度的免疫原预测。该方法将有助于新型疫苗的快速研发,在生物医药领域具有重要应用价值。
- 还没有人留言评论。精彩留言会获得点赞!