一种基于图卷积神经网络的RNA分层嵌入聚类方法
本发明涉及生物信息学,具体涉及一种基于图卷积神经网络的rna分层嵌入聚类方法。
背景技术:
1、单细胞成千上万的基因种类造成了rna测序数据的维度灾难,由于rna获取率低产生dropout噪声,导致数据中存在大量虚假零值。单细胞rna测序数据中同时存在高维和强噪声的问题。
技术实现思路
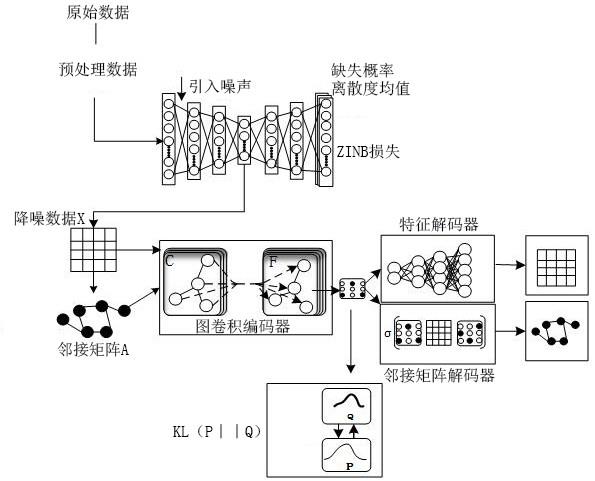
1、针对现有技术的不足,本发明提供一种基于图卷积神经网络的rna分层嵌入聚类方法,通过融合的数据降噪自编码器和数据降维图卷积自编码器解决了数据同时存在的dropout噪声和高维灾难问题,采用深度嵌入聚类优化了聚类结果,提升了聚类精度。
2、本发明是通过如下技术方案实现的:
3、提供一种基于图卷积神经网络的rna分层嵌入聚类方法,包括以下步骤:
4、s1、数据预处理:将单细胞rna测序的读取计数数据处理得到数据矩阵x,矩阵中行代表细胞,列代表基因,每个细胞具有相同种类的基因;
5、s2、数据降噪:
6、将预处理后的数据矩阵x输入到降噪自编码器,在降噪自编码器的编码层的每层引入随机的高斯噪声e,添加噪声后的数据为:
7、x(count )=x+e#(1);
8、编码器函数z=fw=(xcorrupt),解码器函数x’=gw’(z),降噪自编码器采用zinb的负对数似然函数作为损失函数,对数据中的dropout值计算并插补实现数据降噪;
9、s3、数据降维:
10、选择具有双解码器的图卷积自动编码器,将降噪后的数据利用k近邻算法生成knn图,用邻接矩阵a表示图结构作为图卷积自动编码器的一个输入,用表示图节点基因特征的降噪后数据矩阵x作为图卷积自动编码器的另一个输入,通过两个解码器分别对输入knn图数据的邻接矩阵和节点特征矩阵进行重构,使得图卷积自编码器的潜在空间生成学习到的低维的数据特征和数据之间的结构特征;
11、将编码层学习的数据特征向量保存为潜在空间的潜在变量 h=e(x,a),通过两个解码器将变量 h解码为重构数据节点矩阵xr=dx( h)和重构边矩阵ar=da( h),产生邻接矩阵a的损失和数据节点x的损失,整个学习过程是使总的损失函数逐渐减小的过程,直到达到一个最小值,图卷积自编码器的总损失计算公式如下:
12、;
13、其中λ是超参数,实验将参数λ设置为0.6,通过不断训练优化损失函数,使得图卷积自编码器实现对单细胞rna测序数据降维的同时保留高维数据之间的拓扑结构特征;
14、s4、数据聚类:
15、利用图卷积自编码器的初始化生成目标分布p,对图卷积自编码的潜在空间数据使用kl散度函数作为聚类的损失函数,通过不断的迭代训练使图卷积自编码器不断更新聚类参数直到编码层输出的聚类分布q与目标分布p拟合,生成整个聚类更新过程中的最优聚类结果。
16、进一步的,在步骤s1中,采用python包中的scanpy对单细胞rna测序的读取计数数据进行预处理,先过滤掉在任何细胞中都没有表达计数值的基因,再根据库大小对计数矩阵进行归一化并计算大小因子,后读取计数进行对数变换和缩放,使计数值遵循零均值和单位方差。
17、进一步的,在步骤s2中,zinb的表达式为下面的公式:
18、;
19、其中: μ为平均值, θ为离散度, π为dropout概率;
20、相应的优化目标为:
21、;
22、 m,θ,п分别表示均值 μ、离散度 θ和丢失概率 π的矩阵形式,nllzinb表示zinb分布的负对数似然函数,利用此损失函数优化均值、离散度、dropout概率,通过计算并插补dropout值实现数据降噪。
23、进一步的,在步骤s2中,降噪自编码器的结构为编码层、瓶颈层、解码层,编码层和解码层神经网络结构关于瓶颈层对称,且都是全连接的神经网络层,采用relu作为每层神经网络的激活函数。
24、进一步的,在步骤s2中,模型的编码层采用的每层神经元结构为d-256-54-32,其中d为输入数据的维度,32为瓶颈层的神经元个数,解码层结构从左到右的结构为32-54-256-d,d为输出数据的维度,与编码层结构对称,降噪自编码器训练过程中选择的批大小为256,在编码层引入的高斯噪声强度为2.5,利用adam优化算法对模型优化。
25、进一步的,在步骤s4中,训练图卷积自编码器使公式(4)的损失函数lr最小化,交替训练使训练得到的神经网络参数作为第二阶段参数优化的初始化,训练阶段的损失函数定义为l,该损失同时支配图自编码器的损失lr和聚类损失lc;
26、;
27、其中:表示聚类质心,γ是聚类模型实验的超参数并且设置值为2.5,在训练过程中给定了一组初始聚类质心,通过最小化l更新编码网络层的计算数据嵌入到潜在空间,通过对嵌入数据进行聚类迭代的更新聚类质心,将上述步骤交替进行直到损失函数收敛;聚类损失lc采用的是kl散度损失:
28、;
29、通过迭代训练不断更新聚类结果,直到损失函数达到最小值,生成对潜在空间数据的最优聚类结果。
30、本发明的有益效果:
31、本发明针对单细胞rna测序数据构建的聚类方法中融合降噪、降维方法有效解决了数据中同时存在的高维性和强噪声问题,提升了聚类精度。采用分层的方式实现了具有插补dropout事件功能的深度降噪自编码器、同时抓捕数据拓扑结构特征和数据自身特征的双解码图卷积降维自编码器、利用kl散度函数作为聚类的损失函数进行深度嵌入聚类。
32、降噪中采用zinb分布拟合单细胞rna测序数据分布,利用该分布的负对数似然函数作为损失函数对均值 μ、离散度 θ、dropout概率 π三个目标参数进行优化,因此对数据中的dropout值进行了有效的插补。
33、基于图卷积网络的图自编码器对数据进行降维,图卷积自编码器保留了数据之间的拓扑结构,并利用细胞之间邻域信息有效地提高了聚类的效率。实验结果证明,使用融合的数据降噪自编码器和数据降维图卷积自编码器解决了数据同时存在的dropout噪声和高维灾难问题,采用深度嵌入聚类优化了聚类结果。通过分层实现了数据的降噪、降维、聚类,每一层都促进聚类精度的提升,通过在9个真实的高维度和高噪声的数据集上进行实验,与其它传统聚类方法相比,具有更好的聚类效果。
- 还没有人留言评论。精彩留言会获得点赞!