基于鲁棒性残差图卷积网络的单细胞RNA序列数据聚类方法
本发明涉及基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类方法,特别涉及一种基于深度学习去噪的单细胞rna序列数据聚类方法,属于生物信息学和人工智能领域。
背景技术:
1、单细胞rna测序(scrna-seq)技术在过去几年中得到了迅速发展,并在细胞类型、状态和功能的鉴定和表征方面做出了重要贡献。scrna-seq技术可以在一次实验中同时检测多个细胞的转录状态,因此在生物学研究中具有重要意。scrna-seq技术能够测量单个细胞转录组内的基因表达,对于鉴定细胞簇类型、描述复杂疾病的细胞异质性、研究转录动力学以及研究组织组成或基因网络之间的关系具有重要意义。
2、一般情况下,scrna-seq数据聚类分为两个步骤:特征建模(如主成分分析,低秩表示,深度神经网络,图卷积网络)和细胞聚类(例如k-means,分层聚类,谱聚类,和鲁汶算法)。scrna-seq数据是聚类的关键,所以特征建模是目前研究的重点。它的本质是特征降维或表征学习。然而,高维、稀疏和噪声特性通常限制了单细胞测序数据在实际应用中的性能。
3、近年来,由于深度学习具有强大的表征学习能力,它被广泛应用于细胞测序数据的聚类学习。深度学习可以分为三大类:监督学习、无监督学习、半监督学习。深度自动个编码器通常学习数据的紧凑表示,同时试图最大限度地保留输入数据信息。自动编码器包含两个组件:编码器和解码器。深度自动编码器在瓶颈层将数据压缩到低维特征空间中,解码器以编码器的镜像结构,从编码器到处的低维表示重构原始空间中的数据。自动编码器的参数可以通过反向传播最小化这种重构误差来学习。学习到的样本的低维表示也称为嵌入。与传统方法相比,自动编码器能够找到数据所在非线性流行。
4、但是,自动编码器及其变体在学习细胞的特征时只考虑到细胞本身,忽略了细胞之间的相关性。于是,图神经网络被应用于学习细胞特征表示的同时学习细胞之间的相似性关系。通常,在每个图卷积网络层中,每个节点从图中的本地邻居处聚集信息以更新其表示。但是,单细胞测序数据有一个固有缺陷,即根据测序过程中使用的化学物质的版本和每个细胞有限的测序深度,只有少部分的转录本被捕获。于是就存在所谓的“辍学”或所有转录本丢失的给定基因,这是没有生物学意义。因此,在输入的单细胞测序数据中含有缺失值时,需要区分“真”零和“假”零。识别“真”零和“假”零的这一过程叫做去噪。
5、基于此背景,为了实现对单细胞测序数据的准确聚类,本发明提出了一种简单有效的单细胞测序数据去噪以及聚类方法。
技术实现思路
1、本发明提出了一种基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类方法,针对单细胞rna序列数据存在的噪声问题,将图神经网络和残差神经网络结合,并且引入了图结构优化等相关约束,有效学习细胞特征表示的同时学习细胞间的结构关系,实现更加准确的单细胞rna序列数据聚类以及去噪。
2、本发明的技术方案是:基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类方法,所述方法的具体步骤如下:
3、step1、准备若干通用公开的单细胞rna序列数据集用于网络训练;
4、step2、对单细胞rna序列数据进行预处理,筛选出在每个细胞中都表达的细胞以及至少有一个基因表达的细胞,并提取前若干个高表达量基因用于网络训练;
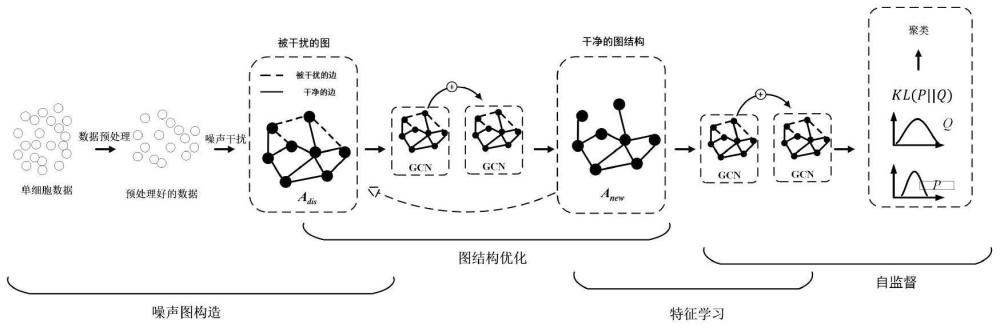
5、step3、构建基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类网络,整个网络由一个噪声图构造模块、一个图结构优化模块、一个特征学习模块和一个自监督模块构成;噪声图构造模块模拟现实中的噪声对图结构进行干扰,图结构优化模块对噪声图结构进行优化,学习一个干净的图结构,然后特征学习模块对干净的图结构进行表征学习;
6、step4、用单细胞rna序列数据集对基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类网络进行训练,并在每次训练完成后用四个聚类评价指标对训练后的网络进行验证;
7、step5、将单细胞rna序列数据输入训练好的基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类网络,得到单细胞rna序列数据中每个细胞的聚类标签,完成单细胞rna序列数据聚类。
8、作为本发明的进一步方案,所述step3的具体步骤如下:
9、step3.1、搭建由图卷积网络、relu激活函数层串联构成的噪声图构造模块;
10、step3.2、搭建由残差图卷积网络和若干项约束构成的图结构优化模块;
11、step3.3、搭建由图卷积网络和残差神经网络构成的特征学习模块;
12、step3.4、搭建由预测分布和目标分布构成的自监督模块。
13、作为本发明的进一步方案,所述step3.1的具体步骤如下:
14、step3.1.1、设计包含三种噪声干扰方式的算法,分别为去除边,增加边,随意翻转边;
15、step3.1.2、设置干扰比例,选择干扰方式;
16、step3.1.3、输入预处理后的数据,计算邻接矩阵,通过矩阵计算得到干扰后的图结构。
17、作为本发明的进一步方案,所述step3.2中,图结构优化模块由一个包含7层图神经网络的残差图卷积网络和5项相关约束组成;
18、所述残差图卷积网络为图神经网络和残差神经网络的组合,图神经网络每一次计算后,都加上第一层原始输入的特征值作为残差块;
19、所述5项相关约束为:约束学习到的干净邻接矩阵与高噪声矩阵的f范数约束,低秩与稀疏约束的一范数与和范数,保证特征平滑的图正则化约束以及提高聚类精度的谱聚类约束;
20、每次图结构优化后都进行图结构的更新以及特征的更新。
21、作为本发明的进一步方案,所述step3.4中,自监督模块通过计算目标分布与预测分布之间的kl散度实现;
22、所述预测分布是step3.3学习到的特征潜在分布;其公式表示为:
23、
24、其中o是t分布的自由度,hi是图卷积网络的输出,uj是聚类簇心,qij看做是预测分布;
25、所述目标分布是由预测分布计算而来的一种分布,其公式表示为:
26、
27、kl散度计算公式为:
28、
29、作为本发明的进一步方案,所述step4中,通过acc值,nmi值,ari值,fmi值评估聚类性能,这四个值的计算公式如下:
30、
31、其中y是真实标签,y是预测标签,n表示细胞数量;
32、
33、其中mi(·)表示互信息,h(·)表示信息熵;
34、
35、其中:
36、
37、tp和tn表示真正例和假正例,fp和fn分别表示假正例和假负例;
38、
39、其中a表示在真实标签和预测标签中都属于同一个簇的细胞数量,b表示在真实标签中属于同一个簇但在预测标签中属于不同簇的细胞数量,c表示在真实标签中属于不同的簇但在预测标签中属于同一个簇的细胞数量;
40、以上四个评价指标最大值为1,值越接近1,表明聚类效果越好。
41、所述基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类网络由一个噪声图构造模块、一个图结构优化模块、一个特征学习模块和一个自监督模块构成,图结构优化模块和特征学习模块都包含了残差图卷积网络。残差图卷积网络有图神经网络和残差神经网络构成,图神经网络学习细胞的相关关系以及特征表示,残差神经网络能够解决图神经网络的特征过平滑问题。首先将单细胞测序数据输入到噪声图构造模块得到一个被噪声干扰的图结构,然后将噪声图输入图结构优化模块得到一个最优图结构,最后通过特征学习模块和自监督模块实现最后聚类。
42、本发明的有益效果是:
43、1、本发明构建的一种基于鲁棒性残差图卷积网络的单细胞rna序列数据聚类网络,设计了一个多层的残差图卷积网络来获取细胞间的高维关系,这能够更好的学习单细胞测序数据的特征表示。
44、2、本发明设计了一个噪声鲁棒性网络,该网络通过不同的约束迭代产生更优的图结构,这能够很好的解决单细胞测序数据聚类面临的噪声问题,对单细胞rna序列数据做出高准确率的聚类,可用于单细胞类型的注释与识别。
- 还没有人留言评论。精彩留言会获得点赞!