一种基于U-Net架构的RNA序列二级结构分析方法
本发明属于生物信息,具体涉及一种rna序列二级结构分析方法。
背景技术:
1、线性rna二级结构预测方法可以分为两类:传统方法和机器学习方法。
2、传统方法包括湿氏实验室实验法、基于多序列的比较序列分析法,基于单序列的计算预测法。湿氏实验法包括x射线晶体学、nmr、低温电子显微镜等方法。基于多序列的比较序列分析法的代表模型有rnaalifold、dcfold、centroidfold,该方法的基本思想是通过已知的同源rna序列获取它们的共同特征,建立协方差模型,然后利用该模型对新的同源rna序列进行二级结构预测。基于单序列的计算预测法的代表模型有rnafold、mfold、rnastructure。该方法基于rna序列的二级结构能量最低这一假设,寻找最低自由能态,这种思想将rna二级结构预测问题视为寻找能量最低状态的问题。
3、机器学习方法的代表模型有e2efold、spot-rna、ufold、mxfold2等,模型e2efold使用了transformer的编码器部分对rna序列进行特征提取,通过卷积层后得到评分矩阵。然后使用拉格朗日乘子法使得评分矩阵满足rna二级结构自身的碱基配对约束,最终得到预测的二级结构。模型spot-rna采用了迁移学习方法,首先在大型数据集bprna-1m上进行预训练,然后在小样本数据集pdb上进一步训练和测试,并达到了很好的预测效果。模型ufold提出了将序列转化为图像输入的思想,将不同位置碱基的依赖关系视为像素点进行求解。不同于上述的机器学习模型,mxfold2是一种机器学习方法与热力学方法的混合模型,该模型首先利用了lstm和cnn等模块获取待测rna序列中任意两个碱基的四种相互关系,然后将该张量作为评分矩阵,使用zuker动态规划法寻找最优评分对应的二级结构作为最终的预测结果。
4、实验室方法普遍具有成本高、耗时耗力、吞吐量低的特点。比较序列分析法需要大量的同源rna序列作为基础,并且同源rna序列的数量和相似程度对二级结构预测的准确率具有很大的影响。计算预测法通常只能处理不含伪结结构的序列,但是该结构对三级结构的形成至关重要。
5、基于机器学习的方法已经成为rna二级结构预测问题的主流方法,但是当前基于机器学习的rna二级结构预测模型在小样本rna家族的二级结构预测准确率较差。
技术实现思路
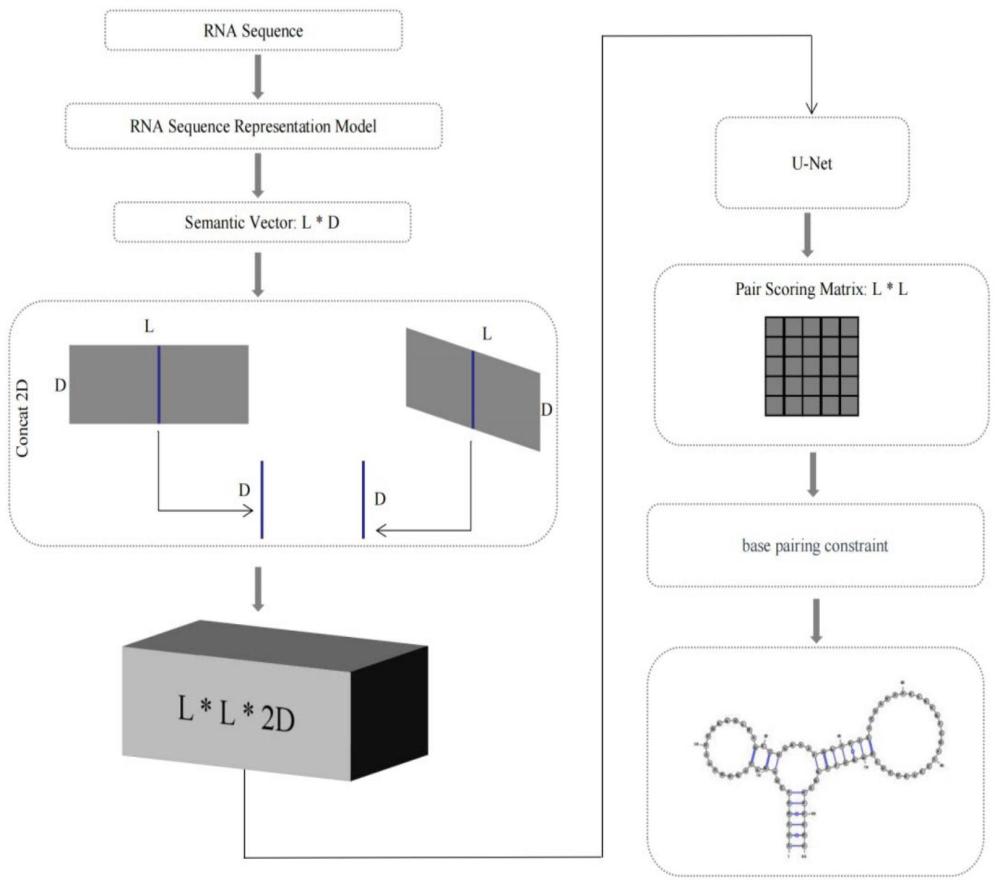
1、为了克服现有技术的不足,本发明提供了一种基于u-net架构的rna序列二级结构分析方法,首先基于序列上下文的rna序列表征模型,完成线性rna序列的可靠表征;然后将表征向量进行全拼接,得到类图像数据,然后将将类图像输入u-net网络转化为概率配对矩阵;最后经过碱基配对约束模块,由rna二级结构的约束条件将概率配对矩阵转化为最终预测的二级结构。本发明方法能够很好地对rna序列进行二级结构预测,在公共数据集上能够取得较好的结果。
2、本发明解决其技术问题所采用的技术方案包括如下步骤:
3、步骤1:构建基于序列上下文的rna序列表征模型;
4、模型从碱基一致性和子序列一致性两个视角学习rna序列的上下文特征表示,保留rna分子碱基序列的二级结构信息,将长度为l的线性rna序列将转化为(l,d)的表征向量,其中d为特征维度,即一个碱基所对应的向量长度;
5、将rna序列输入到表征模型进行无监督训练,训练完成的表征模型对与训练集长度相同的rna序列进行表征;
6、步骤2:将(l,d)的表征向量进行全拼接,拼接成(l,l,2d)的表征向量,然后输入到u-net网络中,得到(l,l)的概率预测矩阵y;u-net网络的训练过程使用像素级别作为监督信号,每一个像素点对应着碱基对的匹配概率;
7、步骤3:构建碱基配对约束模块;
8、步骤3-1:约束一:碱基配对过滤;
9、只有a和u、c和g、u和g三种碱基配对被考虑,其它碱基对即使配对概率大于设定的阈值,也不允许配对,即碱基配对过滤;该约束表示为:
10、mij=1,如果ij∈{au、c、u},否则mij=0
11、其中,mij表示约束矩阵m中的元素;i、j分别表示不同的碱基;
12、步骤3-2:约束二:距离约束;
13、在约束一的基础上,只有两个碱基的距离大于等于4时,才考虑配对,否则不允许配对,即距离约束;该约束表示为:
14、mij=0,如果|i-j|<4
15、在约束矩阵m满足约束一和约束二的前提下,使用约束矩阵m对概率预测矩阵进行约束,约束结果t表示为:
16、t=y°m
17、上式中的约束矩阵m为{0,1}矩阵,y为概率预测矩阵,°指矩阵的元素级乘法,即矩阵y与矩阵m对应位置的元素相乘;
18、将概率预测矩y视为对称矩阵,最终的约束结果t表示为:
19、
20、步骤3-3:约束三:唯一配对约束;
21、rna序列中每个碱基最多只与其他一个碱基形成配对,即唯一配对约束;该约束条件表示为:
22、
23、其中,后面的约束条件指矩阵a每一行的和必须小于等于1,即该行所对应的碱基最多只能与其它一个碱基形成配对;a*为输出矩阵,a为待求解矩阵;公式第一部分中的t(y)使得概率预测矩阵y满足了约束一和约束二;“<>”指矩阵的元素级运算符,即对应位置的元素相乘再求和;ρ为超参数,用于控制矩阵a的稀疏程度;
24、步骤4:对输出矩阵a*进行调整,调整规则如下;
25、步骤4-1:设定一个配对阈值,将a*中大于等于该阈值的元素设定为1,小于该阈值的元素设定为0,将a*转化为一个0-1矩阵;
26、步骤4-2:将碱基序列号越小的碱基与碱基序列号越大的碱基配对;
27、步骤5:基于步骤4的规则,对输出矩阵a*进行最优化搜索;
28、对a*中的每个元素进行迭代搜索,同时考虑其周围的碱基;根据碱基配对的特点,将当前元素与其周围的碱基进行比较,按照碱基序号越小与其匹配的碱基序号越大的原则,选择与其最匹配的碱基作为最终配对;
29、通过最优化搜索,最终得到的二维矩阵满足约束条件,即每个碱基至多只能与一个碱基形成配对,并且满足rna二级结构的特点,根据最终得到的二维矩阵,绘制出对应rna序列的二级结构。
30、优选地,所述rna序列二级结构分析方法的训练过程包括预训练过程和正式训练过程;
31、所述预训练过程使用交叉熵损失函数作为优化目标;在训练过程中,通过最小化交叉熵损失函数优化参数;交叉熵损失函数的公式表示为:
32、lpre=y·-log(sigmoid(x))+(1-y)·-log(1-sigmoid(x))
33、式中,x为u-net网络的输出,y为真实的标签矩阵;
34、所述正式训练使用的损失函数是f1损失函数,f1损失函数表示为:
35、
36、式中p指的是精确率,r指的是召回率;p和r的计算公式如下:
37、
38、
39、其中tp指真实形成配对且被正确预测出配对的碱基对数量,fp指预测的碱基对形成了配对但实际未形成配对的碱基对数量,fn指预测的碱基对未形成配对但实际形成配对的碱基对数量。
40、本发明的有益效果如下:
41、本发明方法构建的基于u-net架构的rna二级结构预测模型能够很好地对rna序列进行二级结构预测。验证结果表明:基于u-net架构的rna序列二级结构高效分析技术在公共数据集上能够取得较好的结果。
- 还没有人留言评论。精彩留言会获得点赞!