用于预测肾乳头状癌患者术后进展风险的多模态模型的制作方法
本发明涉及医学诊断,特别是涉及肿瘤患者的预后预测,具体涉及一种结合分子生物学、人工智能和临床数据分析,用于精准预测肾乳头状癌患者术后进展的多模态模型系统。
背景技术:
1、肾癌是泌尿系统最常见的肿瘤之一,每年新发病例占所有新发癌症病例的4.2%,且发病率逐年上升[1]。肾癌包括具有不同组织学特征、预后和药物敏感性的各种亚型[2-4]。肾乳头状癌是肾癌第二常见的组织学亚型,占所有病例的20%,仅次于肾透明细胞癌[5,6]。近期的临床研究显示,包括肾乳头状癌在内的部分高危肾癌患者可以从辅助治疗中受益,而低危肾癌患者无法从辅助治疗中获益[7]。然而现有的风险分层完全是根据分期和组织学分级系统,我们迫切需要一个更全面的预后模型来准确预测术后肿瘤进展。
2、分子模型已经被发现在多种肿瘤中可以比临床病理因素更准确地预测疾病进程,并帮助临床医生制定更为明智、个体化的治疗决策和随访计划。在肾癌领域目前已有几种经验证的预后基因标志物,如16-gene[8]、clearcode34[9]和基于六个单核苷酸多态性(snp)的分子模型[10];然而,这些分子模型基本都是基于肾透明细胞癌构建的,不适用于肾乳头状癌患者。最近,有研究使用tcga数据库开发了几种肾乳头状癌的分子模型[6,11]。然而,这些分子模型在肾乳头状癌中的预测准确性仍然不令人满意,并且缺乏多中心队列进行验证,因而限制了它们的临床应用。
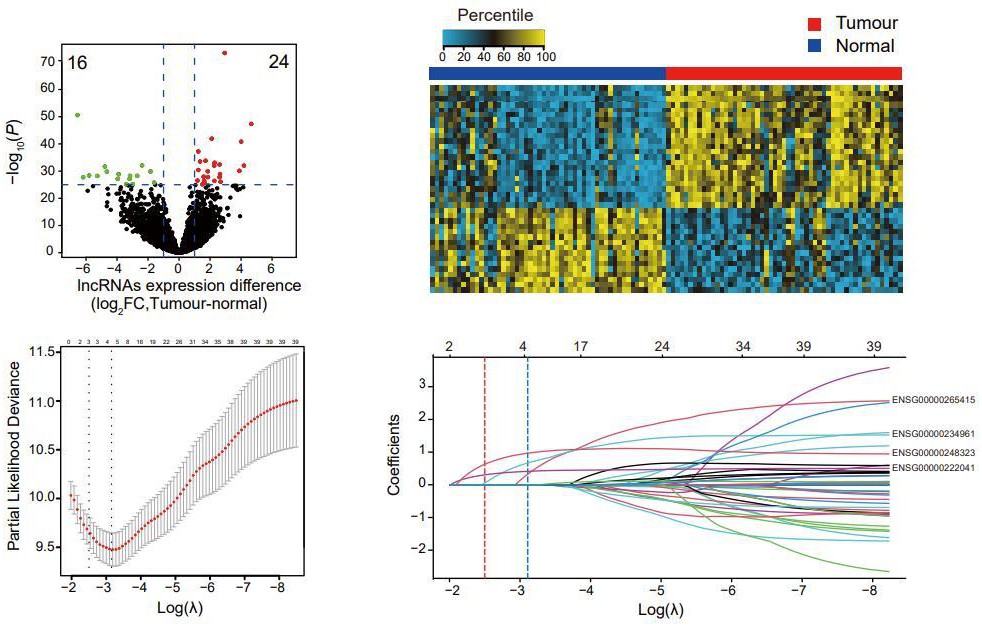
3、长链非编码rna(lncrna)是长度超过200个核苷酸的rna,它们没有编码蛋白质的潜力[12],但涉及多种生物学行为的关键调控[13-15]。许多lncrna被认为是可以有效预测许多肿瘤进展的新型生物标志物[16-19]。近年来,以机器学习和深度学习为代表的人工智能已经在许多肿瘤的诊断、分型乃至预后中展现出了重要价值,一些研究者成功地应用深度学习来识别h&e染色的肿瘤切片上的组织病理学和分子特征[20-23]。已有研究证明基于全切片图像(wsi)的深度学习模型能够准确预测结直肠癌和软组织肉瘤患者的结果[20,24]。
4、因此,我们旨在发明一个将基于lncrna的分子模型和基于wsi的深度学习模型整合的多模态模型,来改进对肾乳头状癌患者术后进展的预测,并为术后辅助治疗策略的制订提供帮助,进而改善肾乳头状癌患者的生存时间和生存质量。
5、参考文献:
6、1. siegel, r.l., miller, k.d., wagle, n.s.&jemal, a. cancerstatistics, 2023. ca cancer j clin73, 17-48 (2023).
7、2. ljungberg, b. , et al.the epidemiology of renal cell carcinoma. eur urol60, 615-621 (2011).
8、3. leibovich, b.c. , et al.histological subtype is an independentpredictor of outcome for patients with renal cell carcinoma. j urol183, 1309-1315 (2010).
9、4. martínez chanzá, n. , et al.cabozantinib in advanced non-clear-cellrenal cell carcinoma: a multicentre, retrospective, cohort study. lancet oncol20, 581-590 (2019).
10、5. leibovich, b.c. , et al.predicting oncologic outcomes in renal cellcarcinoma after surgery. eur urol73, 772-780 (2018).
11、6. linehan, w.m. , et al.comprehensive molecular characterization ofpapillary renal-cell carcinoma. n engl j med374, 135-145 (2016).
12、7. ryan, c.w. , et al.adjuvant everolimus after surgery for renal cellcarcinoma (everest): a double-blind, placebo-controlled, randomised, phase 3trial. lancet402, 1043-1051 (2023).
13、8. rini, b. , et al.a 16-gene assay to predict recurrence aftersurgery in localised renal cell carcinoma: development and validationstudies. lancet oncol16, 676-685 (2015).
14、9. brooks, s.a. , et al.clearcode34: a prognostic risk predictor forlocalized clear cell renal cell carcinoma. eur urol66, 77-84 (2014).
15、10. wei, j.h. , et al.predictive value of single-nucleotidepolymorphism signature for recurrence in localised renal cell carcinoma: aretrospective analysis and multicentre validation study. lancet oncol20, 591-600 (2019).
16、11. ricketts, c.j. , et al.the cancer genome atlas comprehensivemolecular characterization of renal cell carcinoma. cell rep23, 313-326.e315(2018).
17、12. kapranov, p. , et al.rna maps reveal new rna classes and apossible function for pervasive transcription. science316, 1484-1488 (2007).
18、13. prensner, j.r.&chinnaiyan, a.m. the emergence of lncrnas incancer biology. cancer discov1, 391-407 (2011).
19、14. de goede, o.m. , et al.population-scale tissue transcriptomicsmaps long non-coding rnas to complex disease. cell184, 2633-2648.e2619 (2021).
20、15. mendell, j.t. targeting a long noncoding rna in breast cancer. n engl j med374, 2287-2289 (2016).
21、16. prensner, j.r. , et al.nomination and validation of the longnoncoding rna schlap1 as a risk factor for metastatic prostate cancerprogression: a multi-institutional high-throughput analysis. lancet oncol15,1469-1480 (2014).
22、17. li, j. , et al.lncrna profile study reveals a three-lncrnasignature associated with the survival of patients with oesophageal squamouscell carcinoma. gut63, 1700-1710 (2014).
23、18. white, n.m. , et al.multi-institutional analysis shows that lowpcat-14 expression associates with poor outcomes in prostate cancer. eur urol71, 257-266 (2017).
24、19. farrar, j.e. , et al.long noncoding rna expression independentlypredicts outcome in pediatric acute myeloid leukemia. j clin oncol41, 2949-2962 (2023).
25、20. skrede, o.j. , et al.deep learning for prediction of colorectalcancer outcome: a discovery and validation study. lancet395, 350-360 (2020).
26、21. woerl, a.c. , et al.deep learning predicts molecular subtype ofmuscle-invasive bladder cancer from conventional histopathological slides. eur urol78, 256-264 (2020).
27、22. van der laak, j., litjens, g.&ciompi, f. deep learning inhistopathology: the path to the clinic. nat med27, 775-784 (2021).
28、23. acosta, p.h. , et al.intratumoral resolution of driver genemutation heterogeneity in renal cancer using deep learning. cancer res82,2792-2806 (2022).
29、24. foersch, s. , et al.deep learning for diagnosis and survivalprediction in soft tissue sarcoma. ann oncol32, 1178-1187 (2021).
技术实现思路
1、本发明旨在解决现有方法评估肾乳头状癌术后风险分级不够精准的问题。为此,本发明提供了一种革新的多模态预后模型,该系统整合了基于lncrna的分子模型、基于wsi的深度学习模型和关键临床病理指标,用于提升对肾乳头状癌患者风险分层的精确度。该多模态模型利用一套综合性预后模型,通过精确分析lncrna表达数据、病理学图像以及临床病理信息,以实现对肾乳头状癌患者术后病情走向的精准预测。本发明特别关注改进与肾乳头状癌相关的风险评估指标,力图为患者提供更为个性化的治疗选择和随访计划。与当前技术相比,本发明提供了一种新型的风险分层方法,该方法不仅具有在中国多中心队列以及美国多中心队列集中验证的预测准确性和重复性,而且能够解决传统预后模型仅仅基于临床病理特征或分子模型的限制。通过本发明的应用,我们能够显著提高对于肾乳头状癌患者术后风险评估的精准度,从而避免了不必要的治疗和医疗资源浪费,对肾癌精准医疗和个体化治疗的发展具有重大的临床意义。
- 还没有人留言评论。精彩留言会获得点赞!