用于肥胖症相关疾病的生物标记物的制作方法
相关申请的交叉引用无本发明涉及用于预测与微生物相关的疾病、特别是肥胖症或相关疾病的风险的生物标记物和方法。
背景技术:
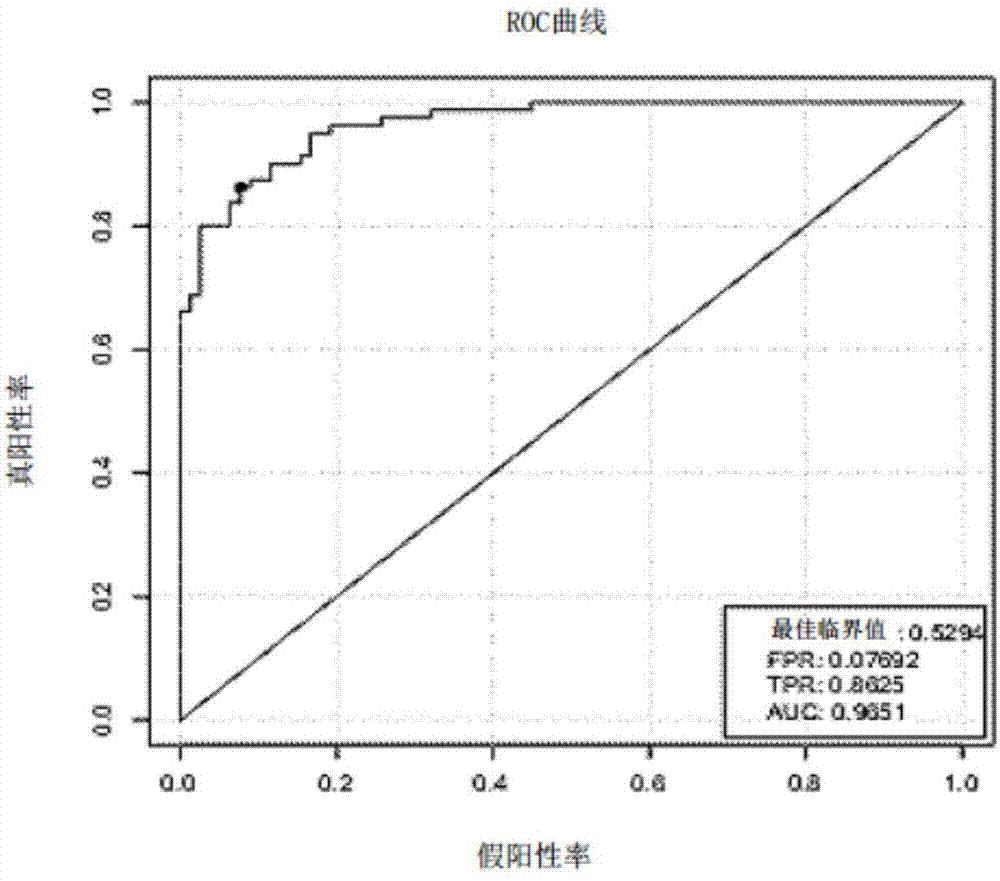
:肥胖症在发达国家很普遍,在全世界范围内显著增加(decarvalhopereira等人,2013)。据报道,在1980年至2013年期间,世界上总共的超重和肥胖症的患病率中,成人增长了27.5%,儿童增长了47.1%。超重的人口从1980年的8.57亿增加到2013年的21亿,其中,6.71亿人口受到了肥胖症的影响。这其中,超过50%的肥胖症患者生活在十个国家中,且美国拥有最大数量的肥胖人群,其次是中国(ng等人,2014)。越来越多的证据表明,由医生诊断为超重的患者相对于未被诊断为超重的患者更有可能减轻体重。然而,医生的低诊断率与对和肥胖症有关的行为的健康风险因素的建议有关(bleich等人,2011)。在儿童中,肥胖症的诊断是基于年龄与性别特异性的身体质量指数(bmi)的切入点。这与成人相反,在成人中,肥胖症的诊断是基于不考虑年龄或性别的bmi而作出的。与成年人不同,对于成年人来说肥胖症的诊断标准更加简单,少数肥胖儿童以更复杂的诊断标准被准确地诊断且对儿童肥胖症的术语发生了改变(walsh等人,2013年)。此外,应考虑bmi在不同人群同一性方面的局限性(nevill等人,2006)。因此,可以认为腰围(wc)是用于评估腹壁多脂症的流行病学研究的可靠而有用的工具,但是这种测量似乎更加难以执行(miguel-etayo等人,2014)。此外,采用国际疾病分类(第九次修订(icd-9))、国家门诊医疗护理调查(namcs)和国家住院医疗护理调查(nhamcs)对儿童肥胖症诊断的区域研究显示出临床诊断的相对低的敏感性(walsh等人,2013)。最近的观察表明,人类肠道微生物群在肥胖症中可以发挥重要作用。基于扩增的16srrna基因测序的早期报告表明,来自12名肥胖人类的粪便样品中的厚壁菌(firmicutes)与拟杆菌(bacteroidetes)之比远高于两个瘦的对照(ley等人,2006)。在人类肥胖症中采用宏基因组测序的最近观察性研究中已经证明细菌多样性降低、拟杆菌(bacteroidetes)的相对缺乏和涉及碳水化合物和脂质代谢的基因的富集(allin和pedersen,2014)。这些相关的发现表明,肠道微生物群的改变是肥胖症的发病机理中的致病因素。这表明,也许我们可以利用用肠道微生物群的该特点作为肥胖症诊断的标准。总之,对肥胖症的诊断有相当多的被忽略的机会和低灵敏度。需要开发更有效的(偏差较小的)对超重和/或肥胖症的评估。技术实现要素:本公开的实施方案试图至少在一定程度上解决现有技术中存在的至少一个问题。本发明基于本发明人的以下发现:对肠道微生物群的评估和表征已经成为对包括肥胖症的人类疾病中的主要研究领域。为了对肥胖症患者的肠道微生物成分进行分析,本发明人基于来自158名个体的肠道微生物dna的深度鸟枪测序实施了宏基因组关联分析(mgwas)方案(qin,j.等人,ametagenome-wideassociationstudyofgutmicrobiotaintype2diabetes.nature490,55-60(2012),通过引用并入本文)。本发明人鉴别并验证了54种肥胖症相关的肠道微生物。为了利用通过肠道微生物群进行肥胖症分类器的潜在能力,发明人通过基于54种肥胖症相关的肠道微生物的随机森林模型计算了疾病概率。本发明人的数据对与肥胖症风险相关的肠道宏基因组的特征进行了深入研究,提供了肠道宏基因组在其它相关疾病中的病理生理作用的未来研究的范例以及用于基于肠道微生物群对此类疾病风险的个体进行评估的潜在应用。人们相信,54种肥胖症相关的肠道微生物对于提高早期阶段肥胖症的检测性是有价值的,这是由于以下原因。首先,本发明的标记物与常规标记物相比更具特异性且更敏感。第二,粪便分析确保了准确性、安全性、可负担性和患者依从性。并且粪便样品是可运输的。因此,本发明涉及一种舒适且无创的体外方法,使得人们更容易地参与给定的筛选程序。第三,本发明的标记物还可以用作肥胖症患者的治疗监测工具,以检测其对治疗的响应。本公开一方面提供了用于预测受试者与微生物群相关的疾病的生物标记物集,其由肠道生物标记物或微生物组成:所述肠道生物标记物包括肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、alistipesshahiiwal8301、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6),卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、规则粪球菌atcc27759(coprococcuseutactusatcc27759)、肺炎克雷伯菌342(klebsiellapneumonia342)、韦荣氏球菌属口腔分类群158菌株f0412(veillonellasp.oraltaxon158str.f0412)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、dialisterinvisusdsm15470、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、拟杆菌属3_1_33faa(bacteroidessp.3_1_33faa)、普拉梭菌kle1255(faecalibacteriumcf.prausnitziikle1255)、产酸克雷伯菌kctc1686(klebsiellaoxytocakctc1686)、多形拟杆菌vpi-5482(bacteroidesthetaiotaomicronvpi-5482)、卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、瘤胃球菌属5_1_39bfaa(ruminococcussp.5_1_39bfaa)、伴生粪球菌atcc27758(coprococcuscomesatcc27758)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、doreaformicigeneransatcc27755、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、dorealongicatenadsm13814、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、扭链瘤胃球菌l2-14(ruminococcustorquesl2-14)、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353),所述微生物具有包括seqidno:1至48497的基因组dna序列。可选地,该生物标记物集由表3中所列种类中的至少一种物种组成,优选由表3中所列的至少10%、至少20%、至少30%、至少40%、至少50%、至少60%、至少70%、至少80%、至少90%、至少100%的物种组成。本公开另一方面提供了用于预测受试者与微生物群相关的疾病的生物标记物集,其由以下组成:包括如表4中所述seqidno:1至48497的至少部分序列的肠道生物标记物。根据本公开的实施方案,该疾病为肥胖症或相关疾病。本公开另一方面提供了用于确定上述基因标记物集的试剂盒,其包括用于pcr扩增并根据seqidno:1至48497的至少部分序列所述的dna序列而设计的引物。本公开另一方面提供了用于确定上述基因标记物集的试剂盒,其包括一个以上根据seqidno:1至48497所述的基因而设计的探针。本公开的另一方面提供了上述基因标记物集用于预测待测试的受试者肥胖症或相关疾病的风险中的用途,包括:(1)从所述待测试的受试者收集样品;(2)确定步骤(1)得到的样品中的根据权利要求1至3中任一项的生物标记物集的每一个生物标记物的相对丰度信息;(3)使用多元统计模型,通过将待测试的受试者的每个生物标记物的相对丰度信息与训练数据集进行比较来获得肥胖症概率,其中,肥胖症概率大于临界值表示待测试的受试者具有肥胖症或相关疾病或处于发展肥胖症或相关疾病的风险中。根据本公开的实施方案,训练数据集是基于多个具有肥胖症的受试者和多个正常受试者的每一个生物标记物的相对丰度信息使用多元统计模型来构建的,可选地,多元统计模型是随机森林模型。根据本公开的实施方案,训练数据集是矩阵,其中各行代表根据权利要求1至3中任一项所述的生物标记物集的各个生物标记物,各列代表样品,各个单元代表样品中生物标记物的相对丰度谱,样品疾病状态是矢量,其中1为肥胖症,0为对照。根据本公开的实施方案,肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、alistipesshahiiwal8301、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6),卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、规则粪球菌atcc27759(coprococcuseutactusatcc27759)、肺炎克雷伯菌342(klebsiellapneumonia342)、韦荣氏球菌属口腔分类群158菌株f0412(veillonellasp.oraltaxon158str.f0412)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、dialisterinvisusdsm15470、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、拟杆菌属3_1_33faa(bacteroidessp.3_1_33faa)、普拉梭菌kle1255(faecalibacteriumcf.prausnitziikle1255)、产酸克雷伯菌kctc1686(klebsiellaoxytocakctc1686)、多形拟杆菌vpi-5482(bacteroidesthetaiotaomicronvpi-5482)、卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、瘤胃球菌属5_1_39bfaa(ruminococcussp.5_1_39bfaa)、伴生粪球菌atcc27758(coprococcuscomesatcc27758)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、doreaformicigeneransatcc27755、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、dorealongicatenadsm13814、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、扭链瘤胃球菌l2-14(ruminococcustorquesl2-14)、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)和霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)中的每一个的相对丰度信息都是根据seqidno:1至48497的相对丰度信息获得的。根据本公开的实施方案,训练数据集是表5-1、5-2、5-3和5-4中的至少一个,并且肥胖症概率为至少0.5说明待测试的受试者具有肥胖症或相关疾病或具有发展肥胖症或相关疾病的风险。本公开另一方面提供了上述基因标记物集在制备用于预测待测试的受试者肥胖症或相关疾病的风险的试剂盒中的用途,其包括:(1)从所述待测试的受试者收集样品;(2)确定步骤(1)得到的样品中根据权利要求1至3中任一项的生物标记物集的每一个生物标记物的相对丰度信息;(3)使用多元统计模型,通过将待测试的受试者的每个生物标记物的相对丰度信息与训练数据集进行比较来获得肥胖症概率,其中,肥胖症的概率大于临界值表示待测试的受试者具有肥胖症或相关疾病或处于发展肥胖症或相关疾病的风险中。根据本公开的实施方案,训练数据集是基于多个具有肥胖症的受试者和多个正常受试者的每一个生物标记物的相对丰度信息使用多元统计模型来构建的,可选地,多元统计模型是随机森林模型。根据本公开的实施方案,训练数据集是矩阵,其中各行代表根据权利要求1至3中任一项所述的生物标记物集的各个生物标记物,各列代表样品,各个单元代表样品中生物标记物的相对丰度谱,样品疾病状态是矢量,其中1为肥胖症,0为对照。根据本公开的实施方案,肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、alistipesshahiiwal8301、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6),卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、规则粪球菌atcc27759(coprococcuseutactusatcc27759)、肺炎克雷伯菌342(klebsiellapneumonia342)、韦荣氏球菌属口腔分类群158菌株f0412(veillonellasp.oraltaxon158str.f0412)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、dialisterinvisusdsm15470、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、拟杆菌属3_1_33faa(bacteroidessp.3_1_33faa)、普拉梭菌kle1255(faecalibacteriumcf.prausnitziikle1255)、产酸克雷伯菌kctc1686(klebsiellaoxytocakctc1686)、多形拟杆菌vpi-5482(bacteroidesthetaiotaomicronvpi-5482)、卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、瘤胃球菌属5_1_39bfaa(ruminococcussp.5_1_39bfaa)、伴生粪球菌atcc27758(coprococcuscomesatcc27758)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、doreaformicigeneransatcc27755、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、dorealongicatenadsm13814、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、扭链瘤胃球菌l2-14(ruminococcustorquesl2-14)、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)和霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)中的每一个的相对丰度信息都是根据seqidno:1至48497的相对丰度信息获得的。根据本公开的实施方案,训练数据集是表5-1、5-2、5-3和5-4中的至少一个,并且肥胖症概率为至少0.5说明待测试的受试者具有肥胖症或相关疾病或具有发展肥胖症或相关疾病的风险。本公开另一方面提供了诊断受试者是否具有与微生物群相关的异常状况或处于发展与微生物群相关的异常状况的风险中的方法,其包括:确定来自受试者的样品中的上述生物标记物的相对丰度,和基于所述相对丰度确定受试者是否具有与微生物群相关的异常状况或者处于发展与微生物群相关的异常状况的风险中。根据本公开的实施方案,该方法包括:(1)从待测受试者收集样品;(2)确定步骤(1)得到的样品中根据权利要求1至3中任一项的生物标记物集的每一个生物标记物的相对丰度信息;(3)使用多元统计模型,通过将待测试的受试者的每个生物标记物的相对丰度信息与训练数据集进行比较来获得肥胖症概率,其中,肥胖症的概率大于临界值表示待测试的受试者具有肥胖症或相关疾病或处于发展肥胖症或相关疾病的风险中。根据本公开的实施方案,训练数据集是基于多个具有肥胖症的受试者和多个正常受试者的每一个生物标记物的相对丰度信息使用多元统计模型来构建的,可选地,多元统计模型是随机森林模型。根据本公开的实施方案,训练数据集是矩阵,其中各行代表根据权利要求1至3中任一项所述的生物标记物集的各个生物标记物,各列代表样品,各个单元代表样品中生物标记物的相对丰度谱,样品疾病状态是矢量,其中1为肥胖症,0为对照。根据本公开的实施方案,肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、alistipesshahiiwal8301、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6),卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、规则粪球菌atcc27759(coprococcuseutactusatcc27759)、肺炎克雷伯菌342(klebsiellapneumonia342)、韦荣氏球菌属口腔分类群158菌株f0412(veillonellasp.oraltaxon158str.f0412)、拟杆菌属1_1_30(bacteroidessp.1_1_30)、dialisterinvisusdsm15470、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、拟杆菌属3_1_33faa(bacteroidessp.3_1_33faa)、普拉梭菌kle1255(faecalibacteriumcf.prausnitziikle1255)、产酸克雷伯菌kctc1686(klebsiellaoxytocakctc1686)、多形拟杆菌vpi-5482(bacteroidesthetaiotaomicronvpi-5482)、卵形拟杆菌3_8_47faa(bacteroidesovatus3_8_47faa)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、普拉梭菌l2-6(faecalibacteriumprausnitziil2-6)、肠道拟杆菌dsm17393(bacteroidesintestinalisdsm17393)、副流感嗜血杆菌t3t1(haemophilusparainfluenzaet3t1)、瘤胃球菌属5_1_39bfaa(ruminococcussp.5_1_39bfaa)、伴生粪球菌atcc27758(coprococcuscomesatcc27758)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、doreaformicigeneransatcc27755、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、dorealongicatenadsm13814、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)、霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)、扭链瘤胃球菌l2-14(ruminococcustorquesl2-14)、dorealongicatenadsm13814、产气柯林斯菌atcc25986(collinsellaaerofaciensatcc25986)、卵瘤胃球菌a2-162(ruminococcusobeuma2-162)和霍氏真杆菌dsm3353(eubacteriumhalliidsm3353)中的每一个的相对丰度信息都是根据seqidno:1至48497的相对丰度信息获得的。根据本公开的实施方案,训练数据集是表5-1、5-2、5-3和5-4中的至少一个,并且肥胖症概率为至少0.5说明待测受试者具有肥胖症或相关疾病或具有发展肥胖症或相关疾病的风险。附图说明本公开的这些和其它的方面和优点从以下结合附图的描述中将变得明显和更容易理解,其中:图1肥胖p值分布的关联分析确定出强相关标记物在较低p值时不成比例的过度表示。图2roc由训练集中的疾病概率绘制,auc=0.9651。图3为了验证54个mlg标记物,本发明人采用随机森林设置模型,然后预测42个样品,并计算每个样品的疾病概率。预测错误率为8/42=19.05%。图4测试集(42个样品)的roc由测试集的肥胖症指数绘制,auc=0.9188。图5计算手术之前和之后的样品的疾病概率。图6示出了每个样品中疾病概率的变化。图7示出了三组中疾病概率。手术后样本的概率显著低于手术之前。具体实施方式示例本文使用的术语具有与本发明相关领域的普通技术人员通常理解的含义。诸如“一”、“一个”和“该”的术语不是旨在仅指单数实体,而是包括可用于说明特定示例的一般类别。除非在权利要求中有所说明,本文中的术语用于描述本发明的具体实施方案,但是它们的用法不限制本发明。本发明在以下非限制性示例通过中进一步举例说明。除非另有说明,份数和百分比都以重量计,度数为摄氏度。对于本领域普通技术人员显而易见的是,这些示例虽然代表了本发明的优选实施方案,但仅以说明的方式给出,并且所有试剂都是可商购的。示例1.鉴别评估肥胖症风险的生物标记物1.1样品收集来自158名中国受试者(包括78名肥胖症患者和80名对照受试者(训练集))的粪便样品由上海交通大学医学院瑞金医院于2012年收集。肥胖症患者年龄从18至30岁,bmi高于25。要求受试者在医院收集新鲜粪便样品。将收集的样品置于无菌管中,立即储存于-80℃直至进行进一步分析。取得了完整的伦理批准,且所有患者都给予了书面知情同意。该研究获得了上海交通大学医学院瑞金医院伦理审查委员会的批准。1.2dna提取将粪便样品在冰上解冻,并使用qiagenqiaampdnastoolmini试剂盒(qiagen),根据制造商的说明进行dna提取。采用无dna酶的rna酶处理提取物以消除rna污染。使用nanodrop分光光度计、qubit荧光计(具有quant-ittmdsdnabr测定试剂盒)和凝胶电泳测定dna量。1.3粪便样品的dna文库构建和测序根据制造商的说明进行dna文库构建(illumina,插入尺寸大小350bp,读段长度100bp)。本发明人使用与前述相同的工作流程进行簇生成、模板杂交、等温扩增、线性化、封闭和变性以及测序引物的杂交。本发明人对于每个样品都构建了一个具有插入尺寸大小为350bp的末端配对的(pe)文库,随后进行高通量测序获得了长度为2x100bp的约3千万个pe读段。通过从illumina原始读段中过滤出具有不确定的“n”碱基、接头污染和人源dna污染的低质量读段并通过同时剪切读段的低质量末端碱基来获得高质量读段。本发明人在illuminahiseq2000平台上从158个样品(78个病例和80个对照)中总共输出每个样品约5.9gb的粪便微生物群测序数据(高质量干净数据)(表1)。表1宏基因组数据汇总。第四列报告来自wilcoxon秩和检验的结果。1.4宏基因组数据处理和分析1.4.1读段比对本发明人使用了li,j.等人,anintegratedcatalogofreferencegenesinthehumangutmicrobiome.nat.biotechnol.(2014)(通过引用并入本文)建立的更新的人类肠道基因目录,并且以比对标准同一性≥90来将高质量读段比对到该更新的人类肠道基因目录。平均的读段比对率示于表1中。该比对率接近li,j.等人,2014,同上中的样品,这说明了该比对率足以进行进一步研究。在读段比对之后,本发明人使用与li,j.等人,2014,同上相同的方法从比对结果中导出基因谱(9.9mb基因)。基因的分类学分配。采用在已公开的论文(li,j.等人,2014,同上)中所描述的内部开发的流程(pipeline)进行预测基因的分类学分配。1.4.2数据文件构建基因谱。基于读段比对的结果,本发明人使用在公开的t2d论文(qin等人,2012,同上)中所描述的相同方法来计算相对基因丰度。1.4.3影响肠道微生物群基因谱的因素分析。基于基因谱,本发明人使用非参数多元方差分析(permanova)来评估6个临床参数(包括年龄、性别、身高、体重、bmi和肥胖)的影响。发明人采用在r中的“vegan”包中实施的方法进行分析,并且通过10,000次置换(permutation)获得置换的(permuted)p值。本发明人还利用benjamini-hochberg方法在r中采用“p.adjust”来校正多重测试,以获得每个测试的q值。permanoa确定了与肠道微生物相关的三个重要因素(基于基因谱)(q<0.05,表2)。分析表明,体重、bmi和肥胖状态是强关联标记,证明了疾病(肥胖)状态是影响肠道微生物群组成的主要决定性因素。表2基于基因谱的欧几里德距离分析的permanova。在q值<0.05下进行分析以测试临床参数和肥胖状况是否对肠道微生物群具有显著影响。表型dfsqs汇总平均sqsf.模型r2pr(>f)年龄10.3170347380.3170347381.0041125790.0063954540.4094性别10.3773294970.3773294971.1965429030.0076117630.1727身高10.3314096670.3314096671.0499472840.0066854350.3291体重10.9695365150.9695365153.1119418570.0195581921.00e-04bmi10.9541868930.9541868933.06170690.0192485481.00e-04肥胖10.9721853520.9721853523.1206139590.0196116262.00e-041.4.4肥胖症相关标记物的确定肥胖症相关基因的确定。为了确定宏基因组谱和肥胖症之间的关联,在9,879,897个高发生率基因(移除在所有158个样品中存在于少于10个样品中的基因)谱中采用双尾wilcoxon秩和检验。得到在病例和对照中都富集的396,100个基因标记物,p值<0.01、fdr=3.8%(图1)。错误发现率估计(fdr)。本发明人应用在先前研究中提出的“q值”法而不是连续p值排除法(sequentialp-valuerejectionmethod)来估计fdr(storey,jdadirectapproachtofalsediscoveryrates.journaloftheroyalstatisticalsociety64,479-498(2002),通过引用并入本文)。受试者工作特征(roc)分析。本发明人应用roc分析来评估基于宏基因组标记物的肥胖症分类的表现。然后,本发明人使用r中的“proc”包来绘制roc曲线。1.4.5mlg的构建和与肥胖症相关的mlg物种标记物的鉴别基于与396,100肥胖症相关的标记物基因谱的237个mlg物种。本发明人使用396,100个基因标记物,采用在公开的t2d论文(qin等人,2012,同上)中所描述的相同方法构建宏基因组连锁群(mlg)。通过在imgv400中将这些基因比对至4,653个参考基因组来注释所有396,100个基因。如果超过50%的组成基因被注释到该基因组,则将mlg指定至该基因组,否则将其称为未分类。选择基因数>100的总共237个mlg基因组(p值<0.01)。为了估计mlg物种的相对丰度,本发明人在去除5%的最低和5%的最高丰度基因后,估计了mlg物种的基因的平均丰度(qin等人2012,同上)。1.5基于mlg的分类器采用训练队列(158个样品)的mlg丰度谱对随机森林模型(r.2.14,随机森林4.6-7包)(liaw,andy&wiener,matthew.classificationandregressionbyrandomforest,rnews(2002),第2/3卷,第18页,通过引用并入本文)进行训练,以选择mlg标记物的最佳集。在一个以上测试集上测试模型,并计算预测误差。关于随机森林模型,利用r版本2.14中“随机森林4.6-7包”,输入的是训练数据集(即所选择的训练样品中mlg的相对丰度谱)、样品疾病状态(训练样品的样品疾病状态是向量,1为肥胖症,0为对照)和测试集(仅为所选择的mlg在测试集中的的相对丰度谱)。然后,发明人使用来自r软件中的随机森林包的随机森林函数来构建分类,并且使用预测函数来预测测试集。输出的是预测结果(疾病概率;临界值为0.5,如果疾病概率≥0.5,则受试者处于肥胖症的风险)。54个mlg物种标记物鉴别。为了鉴别237个mlg物种标记物,发明人基于237个肥胖症相关的mlg物种采用r版本2.14中的“随机森林4.6-7包”。首先,本发明人通过“随机森林”方法给出的重要性(liaw,andy&wiener,matthew,classificationandregressionbyrandomforest,rnews(2002),第2/3卷,第18页,通过引用并入本文)将所有237个mlg物种进行排序。通过创建从1个mlg物种开始并到包含所有237个mlg物种结束的排名靠前的mlg物种的增量子集来构建mlg标记物集。对于每个mlg标记物集,发明人计算了158个样本中的假预测比。最后,选择出具有最低假预测比的54个mlg物种集作为mlg物种标记物(表3和表4)。此外,发明人基于所选择的mlg物种标记物使用来自随机森林模型的oob(outofbag)预测疾病概率来绘制roc曲线(表5-0、5-1、5-2、5-3、5-4),并且在158个样品中,roc曲线下的面积(auc)为0.9651(图2)。在最佳临界值0.5294处,真阳性率(tpr)为0.8625,假阳性率(fpr)为0.07692,表明这54个mlg标记物可用于准确地分类肥胖症个体。表354个与肥胖症相关的最能判别的mlg(物种标记物)表4.54个mlg物种的seqid表5-0在158个样品中54个mlg的预测结果示例2.验证42个样品(测试集)中的54个生物标记物本发明人采用另一个新的独立研究组(包括在上海交通大学医学院瑞金医院收集的17名肥胖症患者和25名非肥胖症对照)验证了肥胖症分类器的辨别能力。提取每个样品的dna并构建dna文库,然后如示例1所述进行高通量测序。本发明人使用与qin等人,2012,同上中所述相同的方法计算这些样品的基因丰度谱。然后确定如seqidno:1-48497所示的每个标记物的基因相对丰度。本发明人通过使用来自该mlg的基因的相对丰度值(qin等人,2012,同上)估计了所有样品中mlg的相对丰度。为了验证54个mlg标记物,本发明人使用随机森林来设置模型。关于随机森林模型,采用r版本2.14中的“随机森林4.6-7包”,输入的是训练数据集(即所选择的训练样品中的mlg的相对丰度谱,表5-1、5-2、5-3、5-4)、样品疾病状态(训练样品的样品疾病状态是向量,1为肥胖症,0为对照)和测试集(仅为所选择的测试集中的mlg的相对丰度谱)。然后,发明人使用来自r软件中的随机森林包的随机森林函数来构建分类,并且使用预测函数来预测测试集。输出的是预测结果(疾病概率;临界值为0.5,如果疾病概率≥0.5,则受试者处于肥胖症的风险)。然后本发明人预测了42个样品,并计算每个样品的疾病概率(表6)。预测错误率为8/42=19.05%(图3)。并且大多数肥胖症患者(16/17)被正确诊断为肥胖症。此外,测试集的roc由测试集的疾病概率绘制,auc=0.9188(图4),其证实54个mlg标记物可用于准确地分类肥胖症个体。在最佳临界值0.592处,真阳性率(tpr)为0.8824,假阳性率(fpr)为0.16。表642个样品的54个mlg的预测结果示例3.验证22个样品(测试集)中的54个基因生物标记物发明人使用另一个测试集样品验证了肥胖症分类器的辨别能力(表7),其中包括9个病例样品和13个对照样品(手术1个月后的5个样品和手术3个月后的8个样品),样品也在上海交通大学医学院瑞金医院收集。病例表示手术前的样本,对照表示手术后1个月和3个月。表722个样品的信息*之前:手术前;1-m:一个月后手术;3-m:三个月后手术。提取每个样品的dna并构建dna文库,然后如示例1所述进行高通量测序。本发明人使用与qin等人,2012,同上中所述相同的方法计算这些样品的基因丰度谱。然后确定如seqidno:1-48497所示的每个标记物的基因相对丰度。本发明人通过使用来自该mlg的基因的相对丰度值(qin等人,2012,同上)估计了所有样品中mlg的相对丰度。为了验证54个mlg标记物,本发明人使用随机森林来设置模型。关于随机森林模型,采用r版本2.14中的“随机森林4.6-7包”,输入的是训练数据集(即所选择的训练样品中的mlg的相对丰度谱,表5-1、5-2、5-3、5-4)、样品疾病状态(训练样品的样品疾病状态是向量,1为肥胖症,0为对照)和测试集(仅为所选择的测试集中的mlg的相对丰度谱)。然后,发明人使用来自r软件中的随机森林包的随机森林函数来构建分类,并且使用预测函数来预测测试集。输出的是预测结果(疾病概率;临界值为0.5,如果疾病概率≥0.5,则受试者处于肥胖症的风险)。在手术样品之前和之后计算疾病概率。这表明,手术后疾病概率降低(图5,表8)。错误率为9%(2/22)。为了得知每个样本的变化,疾病概率如图6所示。在三组中,手术样品后的疾病概率显著低于手术以前(图7,表9),证实该54个mlg标记物可用于准确地分类肥胖症个体。表822个样品中54个mlg的预测结果表9三组中患病概率的p值之前1-m3-m之前0.00074374.114e-051-m0.8584因此,本发明人通过基于肥胖症相关的基因标记物的随机森林模型鉴别出并验证了54个肥胖症相关的肠道微生物。并且本发明人基于这54个肠道微生物构建了评估了肥胖症的风险的方法。尽管已经示出和描述了说明性实施方案,但是本领域技术人员应当理解的是,上述实施方案不能被解释为限制本公开,并且可以对实施方案进行改变、替换和修改而不脱离本发明的精神、原理和范围。当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1