用于抑制高表达FAK基因的肝细胞癌细胞的siRNA及其应用的制作方法
技术领域:
本发明涉及一种siRNA及其应用,特别是涉及一种用于抑制高表达FAK基因的肝细胞癌细胞的siRNA及其应用。
背景技术:
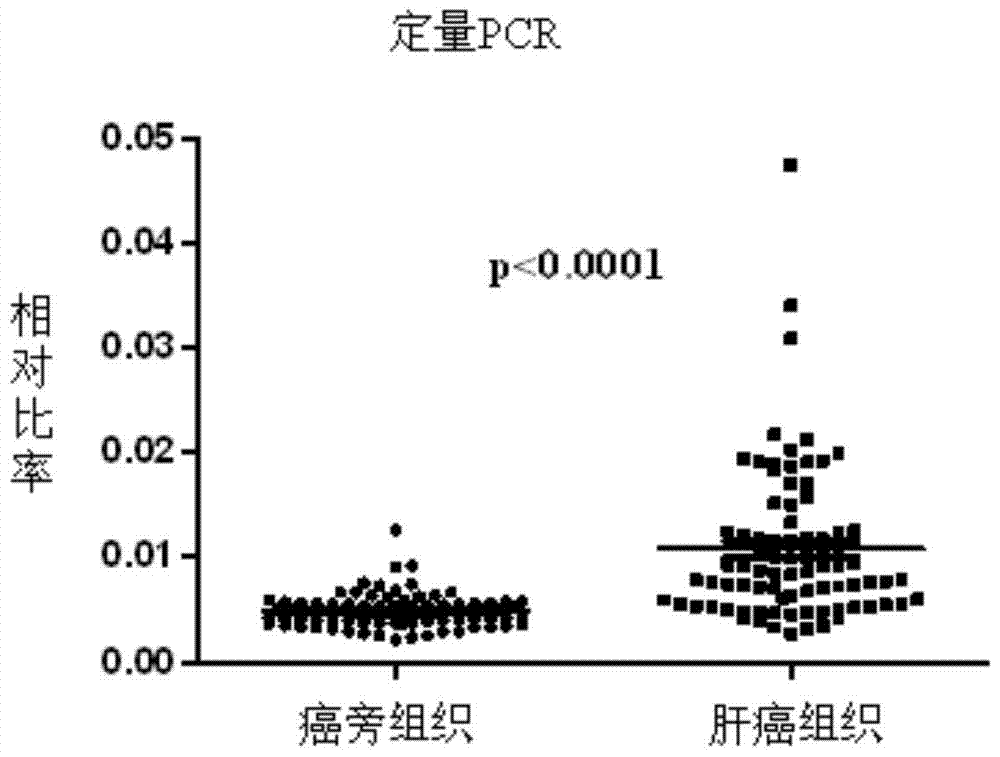
:粘附斑激酶(focaladhesionkinase,FAK)是一类胞质非受体蛋白酪氨酸激酶,它在细胞信号转导中处于十分重要的位置,介导多条信号通路,可以调节细胞生长、并与胚胎发育、肿瘤发生与迁移有关。肿瘤细胞的侵袭性生长是一个多步骤的复杂过程,有多种生物化学因子参与其中。肿瘤细胞必须黏附于细胞外基质,通过促进依赖于PTK激酶活性的细胞外基质信号转导,进而影响细胞的黏附、运动与迁移。其中,FAK介导的信号转导系统是其中重要的细胞信号转导途径之一。RNA干扰(RNAi)是指在进化过程中高度保守的、由双链RNA(dsRNA)诱发的、同源mRNA高效特异性降解的现象。由于使用RNAi技术可以特异性剔除或关闭特定基因的表达,所以该技术已被广泛用于探索基因功能和传染性疾病及恶性肿瘤的基因治疗领域。因而,利用RNA干扰技术,并结合FAK的功能,为肿瘤的治疗提供良好的指导意义。技术实现要素:本发明要解决的技术问题是提供一种用于抑制高表达FAK基因的肝细胞癌细胞的siRNA及其应用。通过利用RNA干扰技术,能有效抑制FAK基因的表达,因而,能更加有效地进行肿瘤治疗。为解决上述技术问题,本发明提供一种用于抑制高表达FAK基因的SMMC7721、QGY7701肝细胞癌细胞的siRNA,所述siRNA为如SEQIDNO.9-10所示的RNA序列。另外,本发明还提供一种编码如上所述的siRNA的DNA序列。本发明还提供一种表达载体,所述表达载体包含有如上所述的siRNA的DNA序列。本发明又提供一种宿主细胞,所述宿主细胞是一种可表达如上所述的siRNA序列的细胞,如可包含有如上所述的表达载体。此外,本发明还提供一种如上所述的siRNA在制备抑制高表达FAK基因的SMMC7721、QGY7701肝细胞癌细胞的药物中的应用。此外,本发明还提供一种抑制高表达FAK基因的SMMC7721、QGY7701肝细胞癌细胞的药物,含有有效量的如上所述的siRNA和药学上可接受的载体。该药物可以采用常规方法制备成相应制剂,如可被制成针剂形式,例如用生理盐水或含有葡萄糖和其他辅剂的水溶液通过常规方法进行制备。针剂、溶液等宜在无菌条件下制造。该药物可以方便的方式给药,如通过局部、静脉内、肌肉内、皮下、皮内、鼻内等给药途径。本发明药物的用量和疗程可根据剂型、患者的年龄、疾病的轻重程度作适当调整,即具体施用剂量取决于多种因素,如给药方式、用药者健康状况等因素,这些都是在熟练医师技能范围内。再者,本发明还提供一种药盒,含有如上所述的siRNA或含有如上所述的药物。该药盒可根据制造、使用或销售药品或生物制品的政府管理机构所给出的指示性提示,该提示反映出生产、使用或销售的政府管理机构许可其在人体上施用。本发明实验证明FAK基因在肝癌组织中的表达明显高于癌旁组织,因此,FAK基因可作为诊断肝癌的特异标志基因,使肝癌诊断更加准确、快速。同时,本发明的siRNA能抑制人FAK基因表达,减少肝癌细胞侵袭,因此,本发明的siRNA可应用于在制备抑制肝癌的药物中,为治疗或防治肝癌新药的开发提供新的途径。附图说明下面结合附图与具体实施方式对本发明作进一步详细的说明:图1是RT-PCR验证FAK基因在10例肝癌病人癌组织和癌旁组织中的表达情况示意图,其中,“N”指癌旁组织,“C”指肝癌组织;图2是荧光定量PCR检测FAK基因在95例肝癌病人中的表达情况图;图3是Panomics分析FAK基因在146例肝癌病人中的表达情况图;图4是siRNA转染SMMC7721细胞后的FAK表达图;图5是siRNA转染WRL68细胞后的FAK表达图;图6是siRNA转染QGY7701细胞后的FAK表达图;图7是siRNA转染SMMC7721细胞后的相对荧光值图;图8是siRNA转染WRL68细胞后的相对荧光值图;图9是siRNA转染QGY7701细胞后的相对荧光值图;图10是siRNA转染SMMC7721后的细胞划痕图。具体实施方式以下实施例仅用于说明本发明而不用于限制本发明的范围。实施例中未注明具体条件的实验方法,通常按照常规条件,例如Sambrook等人,分子克隆:实验室手册(NewYork:ColdSpringHarborLaboratoryPress,1989)中所述的条件,或按照制造厂商所建议的条件。实施例1FAK基因在肝癌组织中的表达1、组织分离实验用组织来源于原发性肝癌的手术病人。手术切除的肝脏一经离体,迅速切取病灶及周围5公分外癌旁组织,立即分装在冻存管内,放入液氮中(-80℃)保存。癌和癌旁的诊断均以病理诊断为最终依据。2、总RNA提取采用Trizol(Invitrogen公司)试剂,按照其说明书进行组织的总RNA提取。具体步骤如下:1)Homogenize匀浆:组织匀浆:每100mg组织加入1mlTRIzol试剂,并用匀浆器匀浆。2)加入约1/5体积的氯仿,上下颠倒充分混匀1min左右,室温静置5min。3)4℃,12,000rpm离心15min。4)小心取出上清液,避免触及中间层,将上清夜转入新的1.5ml离心管,加入等体积的异丙醇,轻轻颠倒混匀,室温静置5min。5)4℃,12000rpm离心15min。6)吸去上清,保留沉淀,并向沉淀中加入1ml75%乙醇,4℃、12000rpm离心15min洗涤沉淀。7)吸去上清,沉淀室温自然晾干后加入适量RNase-free的水,用Tip头吹吸,充分溶解沉淀。8)取1-2μL,测定RNA浓度以及A260/A280值。一般A260/A280比值在1.8-2.1。RNA存放在-80℃冰箱待用。3、逆转录合成cDNA用PrimeScriptRTMasterMixPerfectRealTime(TAKARA公司)逆转录cDNA,反应体系(总量为10μl)如表1所示。表1反应体系反应条件如下:1)37℃15分钟;2)85℃5秒钟;3)4℃保存(-20℃长期保存)。4、RT-PCR采用相对定量法检测FAK基因在肝癌样本中的表达差。使用仪器:ThermalCyclerDiceTMRealTimeSystemTP800,Takara。模板:上述反转录产物按1:10比例稀释后作为PCR反应的模板。引物如表2所示:表2引物序列PCR反应的体系:PCR反应采用TAKARA公司的耐热性TaqDNA聚合酶(DR001),以人基因组为模板时,能很好地扩增3.0kbp(p53基因)的DNA片段。其中,PCR反应的试剂及其用量如表3所示。表3PCR反应的试剂及其用量试剂使用量TakaraTaq(5U/μl)0.1μl10×PCR缓冲液(Mg2+Plus)2μldNTP混合物(每种2.5mM)1.6μl引物混合物(每个5μM)1.6μl模板0.5-4μlddH2O补至20μlPCR反应条件:目的基因:结果如图1所示,FAK基因在肝癌组织中的表达水平明显高于癌旁组织中的表达水平。5、荧光定量PCR采用荧光定量PCR检测FAK基因在肝癌样本中的表达差异。使用仪器:7300型荧光定量PCR仪,ABI公司。模板:上述反转录产物按1:5比例稀释后作为PCR反应的模板。引物序列如表4所示:表4引物序列PCR反应的体系:PCR反应采用TAKARA公司的SYBRPremixExTaq。其中,PCR反应的试剂及其用量如表5所示。表5PCR反应的试剂及其用量成分用量SYBRPremixEXTaq10.0μl上游引物(10μM)0.5μl下游引物(10μM)0.5μlROXReferenceDye(50*)0.4μlcDNA1.0μlddH2O7.6μlPCR反应条件:预变性:95℃30秒PCR反应:熔解曲线测定:95℃5秒60℃60秒95℃5秒荧光定量PCR检测表明,FAK基因在肝癌组织中的表达水平明显高于癌旁组织中的表达水平,并经采用GraphpadPrism5统计软件包(GraphPadSoftware,SanDiego,CA,USA)进行分析,定量资料的比较用t检验进行分析,当P<0.05认为统计学有差异,结果显示P<0.0001(见图2)。6、Panomics分析1)主要仪器Bio-plex悬液芯片系统:Bio-plex100system(Biorad公司)Bio-plex洗板机:Bio-plexproIIwashstation(Biorad公司)恒温振荡摇床:Vortemp56shakingincubator(LabnetInternational)2)操作步骤人的30核酸因子panel的悬浮液体芯片检测试剂盒为Quantigeneplex2.0Kit(Panomics,Affymetrixcorp)。上样量为400ng。按照试剂盒说明书要求进行如下操作:①将组织提取的RNA样本浓度调整至20ng/μl。②根据试剂盒说明书要求配制微珠探针混悬液,取80μl加入杂交板的各孔中,再将空白样本或RNA样本20μl加入孔中。贴膜封闭后,恒温振荡摇床中600rpm,54℃避光孵育18-22h。③将样本探针微珠孵育液转移至磁珠分离板的孔中,利用Bio-plex洗板机进行洗涤,每孔100ul,洗涤三次。④加入预放大液100ul,50℃,600rpm孵育1h。⑤洗板机洗涤三次后,加入100μl放大液,50℃,600rpm孵育1h。⑥洗涤后,加入标记探针液100μl,50℃,600rpm孵育1h。⑦洗涤后,加入SAPE工作液100μl,室温600rpm孵育30min。⑧SAPE洗涤液洗去多余的SAPE后,加入130μlSAPE洗涤液,送入Bio-plex悬液芯片系统进行读值,并经采用GraphpadPrism5统计软件包(GraphPadSoftware,SanDiego,CA,USA)进行分析,定量资料的比较用t检验进行分析,当P<0.05认为统计学有差异。结果如图3所示,FAK基因在肝癌组织中的表达水平明显高于癌旁组织中的表达水平,P<0.0001(见图3)。实施例2利用RNAi技术沉默FAK基因的表达1、细胞培养采用常见的肝癌细胞株,即SMMC7721、WRL68、QGY7701(实验室保存)进行FAK基因的表达研究。其中,细胞株的培养条件为含有10%(体积百分数)胎牛血清(LifeTechnologies)的DMEM(dulbecco'smodifiedeaglemedium)培养液,在37℃、含5%CO2培养箱中培养。2、siRNA的设计根据网络上一些公司提供的免费服务器,如Clontech公司的界面http://bioinfo2.clontech.com/rnaidesigner/sirnaSequenceDesign.do、Invitrogen公司的界面https://rnaidesigner.invitrogen.com/sirna/以及根据InvivoGene服务器(网址http://www.sirnawizard.com)来设计相应的siRNA。其中,设计的针对FAK基因的siRNA序列分别如下:FAK-siRNA1,正义链:5’-CAGGUGAAGAGCGAUUAUAtt-3’(SEQIDNO.7)FAK-siRNA1,反义链:5’-UAUAAUCGCUCUUCACCUGtt-3’(SEQIDNO.8)FAK-siRNA2,正义链:5’-CUCCAGUCUACAGAUUUGAtt-3’(SEQIDNO.9)FAK-siRNA2,反义链:5’-UCAAAUCUGUAGACUGGAGtt-3’(SEQIDNO.10)FAK-siRNA3,正义链:5’-CCCAGGUUUACUGAACUUAtt-3’(SEQIDNO.11)FAK-siRNA3,反义链:5’-UAAGUUCAGUAAACCUGGGtt-3’(SEQIDNO.12)阴性对照siRNA,正义链:5’-UUCUCCGAACGUGUCACGUtt-3’(SEQIDNO.13)阴性对照siRNA,反义链:5’-ACGUGACACGUUCGGAGAAtt-3’(SEQIDNO.14)FAK-siRNA4,正义链:5’-GCAUCUUCCAGUUACAAAUtt-3’(SEQIDNO.15)FAK-siRNA4,反义链:5’-AUUUGUAACUGGAAGAUGCtt-3’(SEQIDNO.16)FAK-siRNA5,正义链:5’-GACCCCAACUUGAAUCACAtt-3’(SEQIDNO.17)FAK-siRNA5,反义链:5’-UGUGAUUCAAGUUGGGGUCtt-3’(SEQIDNO.18)FAK-siRNA6,正义链:5’-GUGCAAUGGAGCGAGUAUUtt-3’(SEQIDNO.19)FAK-siRNA6,反义链:5’-AAUACUCGCUCCAUUGCACtt-3’(SEQIDNO.20)FAK-siRNA7,正义链:5’-GCAAUGGAGCGAGUAUUAAtt-3’(SEQIDNO.21)FAK-siRNA7,反义链:5’-UUAAUACUCGCUCCAUUGCtt-3’(SEQIDNO.22)FAK-siRNA8,正义链:5’-GAGCGAGUAUUAAAGGUCUtt-3’(SEQIDNO.23)FAK-siRNA8,反义链:5’-AGACCUUUAAUACUCGCUCtt-3’(SEQIDNO.24)FAK-siRNA9,正义链:5’-GGAGAUGCUACUGAUGUCAtt-3’(SEQIDNO.25)FAK-siRNA9,反义链:5’-UGACAUCAGUAGCAUCUCCtt-3’(SEQIDNO.26)FAK-siRNA10,正义链:5’-GGGCAUCAUUCAGAAGAUAtt-3’(SEQIDNO.27)FAK-siRNA10,反义链:5’-UAUCUUCUGAAUGAUGCCCtt-3’(SEQIDNO.28)FAK-siRNA11,正义链:5’-GCAUCAUUCAGAAGAUAGUtt-3’(SEQIDNO.29)FAK-siRNA11,反义链:5’-ACUAUCUUCUGAAUGAUGCtt-3’(SEQIDNO.30)FAK-siRNA12,正义链:5’-GUGGACAGUCACAAAGUAAtt-3’(SEQIDNO.31)FAK-siRNA12,反义链:5’-UUACUUUGUGACUGUCCACtt-3’(SEQIDNO.32)FAK-siRNA13,正义链:5’-GUGUGAGGGAGAAGUAUGAtt-3’(SEQIDNO.33)FAK-siRNA13,反义链:5’-UCAUACUUCUCCCUCACACtt-3’(SEQIDNO.34)FAK-siRNA14,正义链:5’-GAGGAGUGGAAAUAUGAAUtt-3’(SEQIDNO.35)FAK-siRNA14,反义链:5’-AUUCAUAUUUCCACUCCUCtt-3’(SEQIDNO.36)FAK-siRNA15,正义链:5’-GUGGAAAUAUGAAUUGAGAtt-3’(SEQIDNO.37)FAK-siRNA15,反义链:5’-UCUCAAUUCAUAUUUCCACtt-3’(SEQIDNO.38)FAK-siRNA16,正义链:5’-GUGAAGAGCGAUUAUAUGUtt-3’(SEQIDNO.39)FAK-siRNA16,反义链:5’-ACAUAUAAUCGCUCUUCACtt-3’(SEQIDNO.40)FAK-siRNA17,正义链:5’-GCGAUUAUAUGUUAGAGAUtt-3’(SEQIDNO.41)FAK-siRNA17,反义链:5’-AUCUCUAACAUAUAAUCGCtt-3’(SEQIDNO.42)FAK-siRNA18,正义链:5’-GAAAUACGGCGAUCAUACUtt-3’(SEQIDNO.43)FAK-siRNA18,反义链:5’-AGUAUGAUCGCCGUAUUUCtt-3’(SEQIDNO.44)FAK-siRNA19,正义链:5’-GAAGUCUAACUAUGAAGUAtt-3’(SEQIDNO.45)FAK-siRNA19,反义链:5’-UACUUCAUAGUUAGACUUCtt-3’(SEQIDNO.46)FAK-siRNA20,正义链:5’-GAUCCAACAAACAUUUAGAtt-3’(SEQIDNO.47)FAK-siRNA20,反义链:5’-UCUAAAUGUUUGUUGGAUCtt-3’(SEQIDNO.48)FAK-siRNA21,正义链:5’-GGUUCAAGCUGGAUUAUUUtt-3’(SEQIDNO.49)FAK-siRNA21,反义链:5’-AAAUAAUCCAGCUUGAACCtt-3’(SEQIDNO.50)FAK-siRNA22,正义链:5’-GCUGGAUUAUUUCAGUGGAtt-3’(SEQIDNO.51)FAK-siRNA22,反义链:5’-UCCACUGAAAUAAUCCAGCtt-3’(SEQIDNO.52)FAK-siRNA23,正义链:5’-GAAGGAAUCAGUUACCUAAtt-3’(SEQIDNO.53)FAK-siRNA23,反义链:5’-UUAGGUAACUGAUUCCUUCtt-3’(SEQIDNO.54)FAK-siRNA24,正义链:5’-GUGCAAACCAUUCAGUAUUtt-3’(SEQIDNO.55)FAK-siRNA24,反义链:5’-AAUACUGAAUGGUUUGCACtt-3’(SEQIDNO.56)FAK-siRNA25,正义链:5’-GCAAACCAUUCAGUAUUCAtt-3’(SEQIDNO.57)FAK-siRNA25,反义链:5’-UGAAUACUGAAUGGUUUGCtt-3’(SEQIDNO.58)FAK-siRNA26,正义链:5’-GGAGAAUAUGGCUGACCUAtt-3’(SEQIDNO.59)FAK-siRNA26,反义链:5’-UAGGUCAGCCAUAUUCUCCtt-3’(SEQIDNO.60)FAK-siRNA27,正义链:5’-GAAUAUGGCUGACCUAAUAtt-3’(SEQIDNO.61)FAK-siRNA27,反义链:5’-UAUUAGGUCAGCCAUAUUCtt-3’(SEQIDNO.62)FAK-siRNA28,正义链:5’-GAAUGGAACCUCGCAGUCAtt-3’(SEQIDNO.63)FAK-siRNA28,反义链:5’-UGACUGCGAGGUUCCAUUCtt-3’(SEQIDNO.64)FAK-siRNA29,正义链:5’-GGAACCUCGCAGUCAUUUAtt-3’(SEQIDNO.65)FAK-siRNA29,反义链:5’-UAAAUGACUGCGAGGUUCCtt-3’(SEQIDNO.66)FAK-siRNA30,正义链:5’-GAACCUCGCAGUCAUUUAUtt-3’(SEQIDNO.67)FAK-siRNA30,反义链:5’-AUAAAUGACUGCGAGGUUCtt-3’(SEQIDNO.68)FAK-siRNA31,正义链:5’-GCAGUCAUUUAUCAUCAGAtt-3’(SEQIDNO.69)FAK-siRNA31,反义链:5’-UCUGAUGAUAAAUGACUGCtt-3’(SEQIDNO.70)FAK-siRNA32,正义链:5’-GAUGAUUAUGCUGAGAUUAtt-3’(SEQIDNO.71)FAK-siRNA32,反义链:5’-UAAUCUCAGCAUAAUCAUCtt-3’(SEQIDNO.72)FAK-siRNA33,正义链:5’-GGAUUAUGAGAUUCAAAGAtt-3’(SEQIDNO.73)FAK-siRNA33,反义链:5’-UCUUUGAAUCUCAUAAUCCtt-3’(SEQIDNO.74)FAK-siRNA34,正义链:5’-GUACAUCAAGGCAUUUAUAtt-3’(SEQIDNO.75)FAK-siRNA34,反义链:5’-UAUAAAUGCCUUGAUGUACtt-3’(SEQIDNO.76)FAK-siRNA35,正义链:5’-GUGAAGCUGAUUGGAGUCAtt-3’(SEQIDNO.77)FAK-siRNA35,反义链:5’-UGACUCCAAUCAGCUUCACtt-3’(SEQIDNO.78)FAK-siRNA36,正义链:5’-GCAAGUAAGGAAAUACAGUtt-3’(SEQIDNO.79)FAK-siRNA36,反义链:5’-ACUGUAUUUCCUUACUUGCtt-3’(SEQIDNO.80)FAK-siRNA37,正义链:5’-GGUGUCCUCAAAUGAUUGUtt-3’(SEQIDNO.81)FAK-siRNA37,反义链:5’-ACAAUCAUUUGAGGACACCtt-3’(SEQIDNO.82)FAK-siRNA38,正义链:5’-GGCUCCAGAGUCAAUCAAUtt-3’(SEQIDNO.83)FAK-siRNA38,反义链:5’-AUUGAUUGACUCUGGAGCCtt-3’(SEQIDNO.84)FAK-siRNA39,正义链:5’-GCUCCAGAGUCAAUCAAUUtt-3’(SEQIDNO.85)FAK-siRNA39,反义链:5’-AAUUGAUUGACUCUGGAGCtt-3’(SEQIDNO.86)FAK-siRNA40,正义链:5’-GAGUGAAGAACAAUGAUGUtt-3’(SEQIDNO.87)FAK-siRNA40,反义链:5’-ACAUCAUUGUUCUUCACUCtt-3’(SEQIDNO.88)FAK-siRNA41,正义链:5’-GUGAAGAACAAUGAUGUAAtt-3’(SEQIDNO.89)FAK-siRNA41,反义链:5’-UUACAUCAUUGUUCUUCACtt-3’(SEQIDNO.90)FAK-siRNA42,正义链:5’-GCCCAGGUUUACUGAACUUtt-3’(SEQIDNO.91)FAK-siRNA42,反义链:5’-AAGUUCAGUAAACCUGGGCtt-3’(SEQIDNO.92)FAK-siRNA43,正义链:5’-GCAGCAUCUAUCCAGGUCAtt-3’(SEQIDNO.93)FAK-siRNA43,反义链:5’-UGACCUGGAUAGAUGCUGCtt-3’(SEQIDNO.94)FAK-siRNA44,正义链:5’-GUGGAGGACUCUACAGUAUtt-3’(SEQIDNO.95)FAK-siRNA44,反义链:5’-AUACUGUAGAGUCCUCCACtt-3’(SEQIDNO.96)FAK-siRNA45,正义链:5’-GAGGGAUUGGGCAAGUGUUtt-3’(SEQIDNO.97)FAK-siRNA45,反义链:5’-AACACUUGCCCAAUCCCUCtt-3’(SEQIDNO.98)FAK-siRNA46,正义链:5’-GACUCUCUCGAGGCAGUAUtt-3’(SEQIDNO.99)FAK-siRNA46,反义链:5’-AUACUGCCUCGAGAGAGUCtt-3’(SEQIDNO.100)FAK-siRNA47,正义链:5’-GGUCGAAUGAUAAGGUGUAtt-3’(SEQIDNO.101)FAK-siRNA47,反义链:5’-UACACCUUAUCAUUCGACCtt-3’(SEQIDNO.102)FAK-siRNA48,正义链:5’-GCCUGGUGAAAGCUGUCAUtt-3’(SEQIDNO.103)FAK-siRNA48,反义链:5’-AUGACAGCUUUCACCAGGCtt-3’(SEQIDNO.104)FAK-siRNA49,正义链:5’-GGACAUUAUUGGCCACUGUtt-3’(SEQIDNO.105)FAK-siRNA49,反义链:5’-ACAGUGGCCAAUAAUGUCCtt-3’(SEQIDNO.106)FAK-siRNA50,正义链:5’-GCACAGAAGCUAUUGAACUtt-3’(SEQIDNO.107)FAK-siRNA50,反义链:5’-AGUUCAAUAGCUUCUGUGCtt-3’(SEQIDNO.108)FAK-siRNA51,正义链:5’-GCCCAGCAGUAUGUCAUGAtt-3’(SEQIDNO.109)FAK-siRNA51,反义链:5’-UCAUGACAUACUGCUGGGCtt-3’(SEQIDNO.110)FAK-siRNA52,正义链:5’-GCCUCCAGCAAGAGUACAAtt-3’(SEQIDNO.111)FAK-siRNA52,反义链:5’-UUGUACUCUUGCUGGAGGCtt-3’(SEQIDNO.112)FAK-siRNA53,正义链:5’-GCAUCAUGAAGAACAAUUUtt-3’(SEQIDNO.113)FAK-siRNA53,反义链:5’-AAAUUGUUCUUCAUGAUGCtt-3’(SEQIDNO.114)FAK-siRNA54,正义链:5’-GCUGCAUAGUGGAAGAGGAtt-3’(SEQIDNO.115)FAK-siRNA54,反义链:5’-UCCUCUUCCACUAUGCAGCtt-3’(SEQIDNO.116)FAK-siRNA55,正义链:5’-GAGCAUGAAGCAAAGAAUUtt-3’(SEQIDNO.117)FAK-siRNA55,反义链:5’-AAUUCUUUGCUUCAUGCUCtt-3’(SEQIDNO.118)FAK-siRNA56,正义链:5’-GCUAAUCCCACUUUACAAAtt-3’(SEQIDNO.119)FAK-siRNA56,反义链:5’-UUUGUAAAGUGGGAUUAGCtt-3’(SEQIDNO.120)FAK-siRNA57,正义链:5’-GUCGGGAACUAGCUGUAGAtt-3’(SEQIDNO.121)FAK-siRNA57,反义链:5’-UCUACAGCUAGUUCCCGACtt-3’(SEQIDNO.122)FAK-siRNA58,正义链:5’-GGGAACUAGCUGUAGAACAtt-3’(SEQIDNO.123)FAK-siRNA58,反义链:5’-UGUUCUACAGCUAGUUCCCtt-3’(SEQIDNO.124)FAK-siRNA59,正义链:5’-GUAGCAAUGUUAUUUCUCUtt-3’(SEQIDNO.125)FAK-siRNA59,反义链:5’-AGAGAAAUAACAUUGCUACtt-3’(SEQIDNO.126)3、siRNA瞬间转染选择内源性表达较高FAK基因的SMMC7721、WRL68、QGY7701细胞株。将合成好的每种siRNA(分别为FAK-siRNA1、FAK-siRNA2、FAK-siRNA3和阴性对照siRNA)用DEPC水溶解,得到20μM浓度的溶液。用1μlLipofectamineTM2000(Invitrogen)和1μl浓度为20μM的每种siRNA分别混合后,将混合液转到提前一天传代细胞密度在30-40%左右的SMMC7721、WRL68和QGY7701细胞中(24孔细胞培养皿),4小时后换完全培养液,继续培养。瞬转48h后收集细胞,然后进行后续的总RNA提取。4、细胞总RNA的提取采用Trizol(TAKARA公司)试剂,按照其说明书进行提取细胞的总RNA,步骤如下:1)培养的贴壁细胞(24孔培养皿),采用上述“3、siRNA瞬间转染”方法进行转染后,培养48h,吸去培养液,加入500μlTRIzol至培养皿中,用枪头吹打几次;2)将裂解液转移至1.5ml的离心管中,室温放置5min;3)按0.2ml氯仿/mlTRIzol的量加入氯仿,盖好管盖,剧烈振摇15s,室温放置5min,4℃12000g离心15min;4)将上层液相转移至一干净的离心管中,加入异丙醇(0.5ml/mlTRIzol),轻轻颠倒混匀数次,室温放置10min,4℃12000g离心15min;5)弃上清,加入75%的乙醇(1ml/mlTRIzol)充分振荡混匀,4℃12000g离心10min;6)去掉上清,沉淀在室温放置5~10min,使其自然凉干(不要完全干燥);加入30-50μl的DEPC水溶解RNA;用紫外分光光度计测定260/280比值并定量;7)去除DNA:采用DNaseI(RNaseFree,TaKaRa公司)去除总RNA中可能的DNA污染,操作步骤如下:①在微量离心管中配制如表6所示的反应液(总量50μl),然后,37℃反应20min。表6反应液成分用量总RNA20~50μg10×DNaseIBuffer5μlDNaseI(RNase-free,5U/μl)2μlRNaseInhibitor(40U/μl)0.5μlDEPC处理的H2O直到反应液总量为50μl②加入50μl的DEPC处理的H2O。③加入100μl(等量)的苯酚/氯仿/异戊醇(体积比分别为25:24:1),充分混匀。④离心12000rpm,15min,取上层(水层)移至另一微量离心管中。⑤加入100μl(等量)的氯仿/异戊醇(体积比为24:1),充分混匀。⑥离心12000rpm,15min,取上层(水层)移至另一微量离心管中。⑦加入10μl(1/10量)的3M乙酸钠(pH5.2)。⑧加入250μl(2.5倍量)的冷无水乙醇,-20℃放置30-60分钟。⑨离心回收沉淀12000rpm,10min,用70%的冷乙醇清洗沉淀,干燥。⑩用适量的DEPC处理的H2O溶解,定量。5、逆转录合成cDNA用PrimeScriptRTMasterMixPerfectRealTime(TAKARA公司)逆转录cDNA,反应体系(总量为10μl)如表7所示。表7反应体系成分用量5*PrimeScriptRTMasterMix(forRealTime)2μl总RNA500ng去除RNase的dH2O补足至10μl反应条件如下:1)37℃15分钟;2)85℃5秒钟;3)4℃保存(-20℃长期保存)。6、荧光定量PCR使用ABI7300实时荧光定量PCR仪和SYBRPremixExTaq试剂盒进行,以β-actin(ACTB)作为内参照,分析转染siRNA对肝癌细胞株FAK表达的影响,所用引物如表4所示,反应体系(总量为20μl)如表8所示。表8PCR反应体系PCR反应条件为:94℃预变性30s,之后94℃10s,60℃30s,共进行40个循环后,进行溶解曲线的检测。其中,荧光本底信号和阈值一般采用仪器默认数值,每次运行结束以后自动生成,每个反应管内的荧光信号达到设定的阈值时所经历的循环数定义为Ct值;数据分析按照如下方法进行:每对引物(基因)在每个模板中做3个重复管,得到的Ct值取平均值;每个目的基因的Ct平均值减去对应模板的内参基因(ACTB)的Ct平均值,得到ΔCt。实验组的ΔCt减去对照组的ΔCt,得到ΔΔCt值,对照组和实验组中的待测基因的倍数关系用2-ΔΔCt。实验结果表明,FAK-siRNA1、FAK-siRNA2和FAK-siRNA3这三个siRNA片段均能有效地沉默FAK基因的表达,提示可以用于下一步的研究所用。其中,SMMC7721、WRL68、QGY7701细胞中的实验结果如图4-6所示。7、细胞生长曲线的测定1)转染前一天接种适量细胞,使转染时细胞密度控制在30%~50%之间;2)采用脂质体LipofectamineTM2000(Invitrogen)转染siRNA配制siRNA与LipofectamineTM2000的混合液,混合液的配方为用1μlLipofectamineTM2000和1μl浓度为20μM的每种siRNA(FAK-siRNA1、FAK-siRNA2、FAK-siRNA3和阴性对照siRNA分别用DEPC水溶解)分别混合后,室温放置约20分钟,形成siRNA-LipofectamineTM2000复合物;然后,将siRNA-LipofectamineTM2000复合物(2μl)加入细胞中,混匀;4~6小时后换正常培养液;3)24小时后,消化转染siRNAs后的SMMC7721、WRL68、QGY7701细胞,根据其生长特性按1×103/100μl/孔计算细胞总量,并充分稀释后,种入96孔板内。每组每天六复孔,一般按7-8天接种细胞;4)待半小时左右,细胞基本贴壁后观察细胞状态和数目。用显色剂显色反应,以确定细胞实际的起始密度,作为生长相对零点。显色剂Cellcountingkit-8(CCK-8,Dojindo,Japan)的用法:每100μl培液加10μlCCK-8,37℃、5%CO2放置1h,在酶标仪上测定450nm处的吸光度;5)光镜下观察细胞形态。按要求定时测量,记录生长状况;6)一般需要测4至7天。等数据收集后,再进行处理,用Excel绘出图表。结果发现,FAK-siRNA1、FAK-siRNA2和FAK-siRNA3这三个siRNA片段均能有效地抑制肝癌细胞的生长。其中,SMMC7721、WRL68、QGY7701细胞中的实验结果如图7-9所示。8、细胞划痕实验将SMMC7721细胞以80%-90%的密度铺入24孔细胞培养皿中。用1μlLipofectamineTM2000和1μl浓度为20μM的每种siRNA(FAK-siRNA1、FAK-siRNA2、FAK-siRNA3和阴性对照siRNA分别用DEPC水溶解)分别混合后,分别转染细胞,来沉默FAK。在24小时后,使用塑料枪头划过细胞表面,37℃、5%CO2的细胞培养箱中培养4天,分别在0、1、2、3天时采集相同位置的图像。实验重复三次。用阴性对照转染的细胞或者未经任何处理的细胞作为对照。结果发现,FAK-siRNA1、FAK-siRNA2和FAK-siRNA3这三个siRNA片段能抑制SMMC7721细胞的生长。其中,FAK-siRNA1和FAK-siRNA3的实验结果如图10所示。9、细胞侵袭实验接种适量细胞(SMMC7721、SK-hep1),使转染时细胞密度控制在30%~50%之间;24小时内采用脂质体LipofectamineTM2000(Invitrogen)转染siRNA:配制siRNA与LipofectamineTM2000的混合液,即用1μlLipofectamineTM2000和1μl浓度为20μM的每种siRNA(FAK-siRNA1和阴性对照siRNA分别用DEPC水溶解)分别混合后,室温放置约20分钟,形成siRNA-LipofectamineTM2000复合物;将siRNA-LipofectamineTM2000复合物(每种2μl)加入细胞中,混匀;4~6小时后换正常培养液。细胞侵袭实验均采用24孔Transwell小室(8-μm孔径,BDBiosciences公司),涂有人工基底膜(Falcon354480;BDBiosciences公司)。在无血清培养基中肝癌细胞(SMMC7721、SK-hep1)饥饿过夜,用胰蛋白酶消化处理,并在含1%FBS的DMEM培养液中洗涤3次。将含有1×105个细胞的500μl含1%FBS的DMEM培养液中,加入到上部腔室。同时将750μl含10%FBS的DMEM培养基和10μg/ml的纤维连接蛋白(目录编号356008,BDBiosciences公司)置于在下部腔室。作为对照,将含1%FBS的培养基中加入下部腔室。经过48个小时的潜伏期,上腔室中剩余的人工基底膜和细胞用棉签去除。在膜下表面上的细胞用4%多聚甲醛固定,并用0.5%结晶紫染色。细胞在至少6个随机显微镜视野(倍率×100)中计数并拍照。所有实验均在重复进行,并重复3次,并采用GraphpadPrism5统计软件包(GraphPadSoftware,SanDiego,CA,USA)进行分析,定量资料的比较用t检验进行分析,当P<0.05认为统计学有差异。结果发现,FAK-siRNA1能明显抑制肝癌细胞SMMC7721、SK-hep1的侵袭。综上所述,本发明利用RNAi技术沉默FAK的表达可抑制肝癌的细胞生长、运动和侵袭。因而,本发明的siRNA可应用于制备治疗或防治肿瘤(如肝癌)药物,为肿瘤的治疗,提供新的途径。当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1