一种DNA银纳米簇检测单碱基错配位置的方法及其试剂盒与流程
本发明涉及一种dna银纳米团簇检测单碱基错配位置的方法及其试剂盒,属于生物分析检测技术领域。
背景技术:
基因变异与许多人类遗传疾病有着紧密的关系,其中发生频率最高,包含信息最多的是单碱基多态性(singlenucleotidepolymorphisms,snps)。单碱基基因突变的检测对于癌症病人的早期诊断是非常重要的。目前检测单碱基基因突变的方法主要分为两大类,一类是基于酶促反应的精确基因测序,比如基于聚合酶链式反应(polymerasechainreaction,pcr)的第二代测序技术。这类基于酶促反应的检测方法步骤复杂,耗时长和花费高,更重要的是多数情况下,对于一条特定的dna基因序列,通过精确的基因测序检测该条链上的所有碱基是不必要的,因为我们只是关心某些与疾病相关的特定基因点突变。另外一类是dna生物传感器。最近几年,dna生物传感器引起了人们很大的关注,这类检测方法对于特定基因点突变引起的疾病(如镰刀形红细胞贫血症、鼠类肉瘤病毒癌、地中海贫血等)的检测是十分有效的。但是传统的dna生物传感器只能确定基因序列上是否存在基因突变,并不能准确的检测出突变碱基的位置。
以dna为模板合成的银纳米簇拥有不大于2nm的尺寸、接近于电子的费米波长,展示出了分立的电子能级,并通常伴随着荧光的发射,表现出很多“人工原子”的性质。与传统的荧光团相比,dna银纳米簇具有包括较宽的发射波长范围和较大的斯托克斯位移在内的突出的光学性质。
技术实现要素:
本发明的目的是提供一种dna银纳米团簇检测单碱基错配位置的方法及其试剂盒,该方法相比较与精确的dna测序技术,本发明整个过程不涉及酶促反应,一次杂交反应即可,步骤简单,快速方便,价格低廉;相比较与传统的dna生物传感器,本发明不仅能确定是否存在单碱基错配,而且还能精确检测出单碱基错配的位置。
本发明提供的一种用于检测单碱基错配位置的试剂盒,所述试剂盒为下述(1)或(2):
(1)所述试剂盒包括dna银纳米簇探针ⅰ和dna银纳米簇探针ⅱ;所述dna银纳米簇探针ⅰ和所述dna银纳米簇探针ⅱ是分别以dna探针ⅰ和dna探针ⅱ为模板合成得到的带有银纳米簇的探针;
所述dna探针ⅰ的序列自5’至3’的方向依次为银纳米簇模板序列和杂交序列ⅰ, 所述dna探针ⅱ的序列自5’至3’的方向依次为杂交序列ⅱ和银纳米簇模板序列,所述杂交序列ⅱ和所述杂交序列ⅰ自5’至3’的方向依次连接构成的序列与完全匹配的目标dna的序列反相互补且完全匹配;所述完全匹配的目标dna为未发生任何单碱基错配的dna;
(2)所述试剂盒包括dna探针ⅰ和dna探针ⅱ;所述dna探针ⅰ的序列自5’至3’的方向依次为银纳米簇模板序列和杂交序列ⅰ,所述dna探针ⅱ的序列自5’至3’的方向依次为杂交序列ⅱ和银纳米簇模板序列,所述杂交序列ⅱ和所述杂交序列ⅰ自5’至3’的方向依次连接构成的序列与完全匹配的目标dna的序列反相互补且完全匹配;所述完全匹配的目标dna为未发生任何单碱基错配的dna。
上述的试剂盒中,所述银纳米簇模板序列可为5’-ccctaactcccc-3’。
上述的试剂盒中,所述dna银纳米簇探针ⅰ和dna银纳米簇探针ⅱ的摩尔比可为1:1;所述dna探针ⅰ和所述dna探针ⅱ的摩尔比为1:1。
上述的试剂盒中,所述dna银纳米簇探针ⅰ和所述dna银纳米簇探针ⅱ均以水溶液的形式存在,所述水溶液中,所述dna银纳米簇探针ⅰ或所述dna银纳米簇探针ⅱ的摩尔浓度可为1~20um,具体可为15um,溶剂可为ph为6.0~8.0的磷酸盐缓冲溶液。
上述的试剂盒中,具体地,所述杂交序列ⅰ的长度与所述杂交序列ⅱ的长度相等或相差一个碱基。
上述的试剂盒中,所述试剂盒还包括标准品,所述标准品为所述完全匹配的目标dna自5’至3’的方向或自3’至5’的方向依次发生错配的目标dna,其数目由所述完全匹配的目标dna的碱基数决定。
上述的试剂盒中,所述完全匹配的目标dna的序列具体可为5’-aacttcttgctgtatcttctatgtcgttcttcaa-3’,所述dna银纳米簇探针ⅰ的序列可为5’-ccctaactccccagatacagcaagaagtt-3’;所述dna银纳米簇探针ⅱ的序列可为5’-ttgaagaacgacatagaccctaactcccc-3’。
本发明进一步提供了利用上述试剂盒检测单碱基错配位置的方法,包括如下步骤:
(1)按照所述完全匹配的目标dna设计合成一系列不同错配位置的目标dna;
(2)将所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ与不同错配位置的目标dna杂交,激发测量得到的不同杂交体系的荧光强度,建立错配位置和荧光强度的工作曲线;
(3)将所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ与待测目标dna杂交,激发测量得到的杂交体系的荧光强度,根据所述工作曲线即可得到所述待测目 标dna的错配位置。
上述检测方法,所述错配为所述完全匹配的目标dna中的碱基自5’至3’的方向或自3’至5’的方向依次错配,得到各个错配位置下的目标dna,具体地,当所述完全匹配的目标dna为对称结构时,可将单碱基错配的位置从目标dna的中间位置依次向端侧移动得到一系列不同错配位置的目标dna。
上述的检测方法,当所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ与不同错配位置的目标dna杂交后,两个探针中的银纳米簇会靠近产生荧光增强,当目标dna上有错配碱基时,荧光增强效果减弱,并且错配碱基在目标dna上不同的位置,会导致两个银纳米团簇之间距离的变化,利用表面等离子体共振增强荧光对距离变化的敏感性,产生不同强度的荧光信号。
上述的检测方法,所述杂交时,所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ和所述完全匹配的目标dna的摩尔比为1:1:1;所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ和所述不同错配位置的目标dna的摩尔比为1:1:1。
上述的检测方法中,所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ与不同错配位置的目标dna形成的杂交体系中,所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ和所述不同错配位置的目标dna的摩尔浓度为1um~20um(具体可为7.28um),溶剂为ph为6.0~8.0的磷酸盐缓冲溶液;
所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ与待测目标dna形成的杂交体系中,所述dna银纳米簇探针ⅰ、所述dna银纳米簇探针ⅱ和所述待测目标dna的摩尔浓度为1um~20um(具体可为7.28um),溶剂为ph为6.0~8.0的磷酸盐缓冲溶液。
上述的检测方法中,所述激发波长具体可为555nm。
本发明与现有技术相比具有以下特点:
1、通过荧光光谱检测,操作简单;
2、通过dna模板合成银纳米团簇作为信号单元,不需要荧光团的标记与修饰,步骤简单;
3、设计了三明治结构的杂交方式,将两个银纳米团簇挤压在三明治结构的中间,可通过银纳米团簇之间的空间位阻效应放大不同错配位置的目标dna与探针dna杂交过程中结合能的细微差别,导致从错配碱基位置到银纳米团簇位置的两段dna序列不能正常杂交,两个银纳米团簇之间的距离产生变化;
4、利用表面等离子体共振效应对于距离的变化很敏感,实现对由不同错配碱基位置引起两个银纳米团簇之间距离细微变化的高灵敏度检测;
5、相比较与精确的dna测序技术,本发明整个过程不涉及酶促反应,一次杂交 反应即可,步骤简单,快速方便,价格低廉;相比较与传统的dna生物传感器,本发明不仅能确定是否存在单碱基错配,而且还能精确检测出单碱基错配的位置。
附图说明
图1为本发明dna银纳米团簇检测单碱基错配位置的方法的示意图。
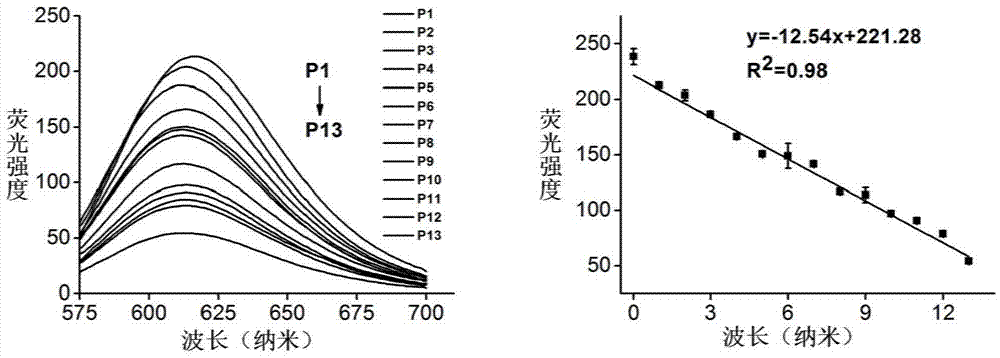
图2为不同单碱基错配位置目标dna与探针杂交后的荧光光谱图(左图)和最大发射处的荧光强度与错配位置的工作曲线(右图)。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
ph=7.410mm的pb缓冲溶液和ph=7.020mmpb缓冲溶液均从北京鼎国昌盛生物技术有限责任公司购买。
dna从生工生物工程上海(股份)有限公司购买。
实施例1、利用本发明方法检测单碱基的错配位置
按照图1所示的示意图,检测单碱基的错配位置,步骤如下:
(1)按照完全匹配的目标dna(pmdna)设计合成dna探针ⅰ(ncprobe-1)和dna探针ⅱ(ncprobe-2),序列分别如下:
pmdna序列:aacttcttgctgtatcttctatgtcgttcttcaa(序列1)
ncprobe-1序列:ccctaactccccagatacagcaagaagtt(序列2)
ncprobe-2序列:ttgaagaacgacatagaccctaactcccc(序列3)
下划线表示用于合成银纳米团簇的模板序列。
(2)分别以dna探针ⅰ和dna探针ⅱ为模板,制备dna银纳米簇探针ⅰ和dna银纳米簇探针ⅱ,具体步骤如下:
将dna探针ⅰ(dna探针ⅱ)溶解在10mmph=7.4的pb缓冲溶液中,浓度为100um;将15ul上述dna探针ⅰ(dna探针ⅱ)溶液加到73ul20mmph=7.0的pb缓冲溶液中,震荡搅拌均匀后,向上述混合液中加入新配置的6ul1.5mm硝酸银水溶液,快速剧烈震荡30秒,避光冰浴保存30分钟;进一步向上述溶液中加入新配置的6ul1.5mm硼氢化钠水溶液,快速剧烈震荡30秒,得dna银纳米簇探针ⅰ和dna银纳米簇探针ⅱ,避光4℃保存6个小时待用。
(3)测量dna银纳米簇探针ⅰ、dna银纳米簇探针ⅱ与完全匹配的目标dna杂交后得到的杂交体系的荧光强度,具体步骤如下:
将49uldna探针1和49uldna探针2的银纳米团簇复合物等量混合,然后加入2.94ul250um(溶剂为10mmph=7.4的pb缓冲溶液)完全匹配的目标dna(即dna银纳米簇探针1、dna探针银纳米簇探针2和完全匹配的目标dna的摩尔比为 1:1:1),震荡混合均匀后,放入37℃水浴30分钟,555纳米激发测量上述增强体系的荧光强度。
(4)建立目标dna中单碱基错配位置与体系荧光强度的工作曲线,具体步骤如下:
将49uldna探针1和49uldna探针2的银纳米团簇复合物等量混合(摩尔比为1:1),然后分别加入2.94ul250um(溶剂为10mmph=7.4的pb缓冲溶液)错配位置在1号至13号位置的含有单碱基错配的13条目标dnap1-p13(1号位置在目标dna的中间位置,2号,3号至13号,依次从中间位置向目标dna端侧移动一个碱基位置)(即dna银纳米簇探针ⅰ、dna银纳米簇探针ⅱ和待测的目标dna的摩尔比为1:1:1),震荡混合均匀后,放入37℃水浴30分钟,555纳米激发测量上述13个体系的荧光强度,建立单碱基错配位置与荧光强度间的工作曲线。
含有单碱基错配的13条目标dnap1-p13的序列分别如下:
p1:aacttcttgctgtatcctctatgtcgttcttcaa(序列4)
p2:aacttcttgctgtattttctatgtcgttcttcaa(序列5)
p3:aacttcttgctgtagcttctatgtcgttcttcaa(序列6)
p4:aacttcttgctgtgtcttctatgtcgttcttcaa(序列7)
p5:aacttcttgctgaatcttctatgtcgttcttcaa(序列8)
p6:aacttcttgctatatcttctatgtcgttcttcaa(序列9)
p7:aacttcttgccgtatcttctatgtcgttcttcaa(序列10)
p8:aacttcttggtgtatcttctatgtcgttcttcaa(序列11)
p9:aacttcttactgtatcttctatgtcgttcttcaa(序列12)
p10:aacttctagctgtatcttctatgtcgttcttcaa(序列13)
p11:aacttcgtgctgtatcttctatgtcgttcttcaa(序列14)
p12:aacttgttgctgtatcttctatgtcgttcttcaa(序列15)
p13:aactgcttgctgtatcttctatgtcgttcttcaa(序列16)
p1-p13中的加粗碱基表示错配的碱基。
如图2中左图所示,随着碱基错配位置向端侧的移动,荧光强度逐渐减小,如图2中右图所示,碱基错配位置与荧光强度呈线性关系,线性拟合方程为y=-12.54x+221.28(r2=0.98)。
(5)检测待测目标dna中的单碱基错配位置
重复上述步骤(4),仅将错配位置在1号至13号位置的含有单碱基错配的13条目标dnap1-p13替换为dna序列为p4的待测目标dna,得到的荧光曲线与图2 左图中的p4相同,根据图2右图中的工作曲线和该待测目标dna的最大发射波长下的荧光强度,得到该待测目标dna的错配位置为4号位置,与预测结果一致。
- 还没有人留言评论。精彩留言会获得点赞!
- 核酸杂交的电化学检测法的制作方法
- 一种评价中草药对dna氧化损伤保护作用的方法
- 卷烟烟气导致dna氧化损伤产生8-羟基脱氧鸟苷的测定评价方法及其应用的制作方法
- 一种新的多肽——人dna错配修复蛋白15和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白8.9和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白9.4和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白8和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复基因蛋白13和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复基因蛋白11和编码这种多肽的多核苷酸的制作方法
- 一种新的多肽——人dna错配修复蛋白12.98和编码这种多肽的多核苷酸的制作方法