一种靶向人STAT6的CRISPR‑Cas9系统及其用于治疗过敏性疾病的应用的制作方法
本发明涉及基因工程领域,更具体地说涉及CRISPR-Cas9特异性敲除人STAT6基因的方法以及用于特异性靶向STAT6基因的sgRNA。
背景技术:
支气管哮喘(简称哮喘)是由嗜酸粒细胞、肥大细胞、T细胞及白细胞等多种细胞及细胞组分参与的慢性气道炎症,在慢性炎症基础上伴随气道的高反应性(airway hyperresponsiveness)和气道重建(airway remodelling),其本质是气道慢性炎症。哮喘是世界公认的医学难题,被世界卫生组织列为疾病中四大顽症之一。近十多年来,世界上许多国家和地区哮喘的发病率和病死率均呈增高趋势,运一现象已引起世界卫生组织(WHO)和各国政府的重视。
哮喘以可恢复的支气管阻塞、炎性细胞浸润和气道高反应性为其特征。哮喘设及嗜酸性粒细胞、肥大细胞和T淋己细胞等多种炎症细胞、炎症介质、细胞因子的相互作用,呈现一种慢性气道炎症过程和气道高反应,从而引起可逆性气道阻塞,即迟发性哮喘反应。有报道指出,哮喘病人气道中肥大细胞数量增加,哮喘发作病人气道肥大细胞的活动性增强,气道活检标本中显示肥大细胞脱颗粒,支气管肺泡灌洗液中肥大细胞的产物增加。
目前认为,哮喘主要是气道慢性非特异性炎症,是一种多基因病,其发病由环境和遗传共同作用所致.STAT6是JAKs-STATs传导通路中的重要因子,哮喘的发病机制中很多因子都涉及STAT6。STAT6基因位于人类12q13-24,此为过敏性疾病如哮喘等的重要候选基因,近年来STAT6及基因研究在哮喘发病中的地位和作用也越来越重要。
有报道指出STAT6基因敲除小鼠的化细胞不能分化为化2细胞,而且B细胞不能产生IgEeEotaxin属于嗜酸性粒细胞趋化因子,其主要可吸引嗜酸粒细胞在肺内的募集,促进其粘附在血管内皮细胞上,进而导致组织损伤。有研究报道AL0X15有助于哮喘的发病机制,并且上皮细胞过表达AL0X15与疾病的严重度相关联。STAT6作为转录因子对Eotaxin和AL0X15的mRNA表达具有增强作用。
规律成族间隔短回文重复系统(clustered regularly interspaced short palindromic repeat;CRISPR-associated,CRISPR_Cas9)是一种具有核酸内切酶活性的复合体,识别特定的DNA序列,进行特定位点切割造成双链DNA断裂(Double-strand breaks,DSB),在没有模板的条件下,发生非同源重组末端连接(Non-homologous end joining,NHEJ),造成移码突变(frameshift mutation),导致基因敲除(图1)。
这一技术由于能快速、简便、高效地靶向基因组任何基因,从而引起了广泛的关注,在2012年开始像爆炸一般流行开来。由于其容易操作、可以同时靶向多个基因,可以高通量制备、造价低等优势,Cas9已经成为一种发展最快的技术(Pennisi,2013)。正是由于其优越性,这一技术在Nature推荐的2013十大进展中位列第一。
Cas9靶向切割DNA是通过两种小RNA--crRNA(CRISPR RNA)和tracrRNA(trans-activating crRNA)和革E序列互补识别的原理实现的。现在已经把两种小RNA融合成一条RNA链,简称sgRNA(single guide RNA)。因此,sgRNA能否做到特异性、精确祀向目标基因是CRISPR-Cas9能否特异性敲除目标基因的先决条件,无论是脱靶还是错误靶向,都会影响CRISPR-Cas9对目标基因的特异性敲除。因此,能够设计、制备出精确性和特异性靶向目标基因的sgRNA成为CRISPR-Cas9基因敲除的关键技术(图1)。
与ZFN相比,CRISPR-Cas9具有更快速、简便、高效、多位点、特异性靶向敲除基因的优势。为高效靶向敲除CCR5,实现艾滋病及其相关疾病的治疗提供了一种可能的选择。本发明的目的就是要验证利用CRISPR-Cas9高效靶向敲除STAT6提供相应的技术方案,以达到特异性敲除STAT6的目的。
技术实现要素:
针对现有使用同源重组或者ZFN靶向STAT6进行过敏性疾病治疗所存在的问题:(1)效率较低,只能少量敲除STAT6;(2)设计与合成一对特异性的ZFN费时、费力、费钱,使得其花费昂贵等。同源重组的方法效率低下的问题。本发明设计、合成了在CRISPR-Cas9特异性敲除人STAT6基因中特异性靶向STAT6基因的sgRNA,并分别将该sgRNA寡核苷酸载体与酶切质粒一起成功转染细胞即可实现STAT6基因的敲除。本申请提供了一种利用Cas9/sgRNA快速、简便、高效、特异性敲除STAT6的策略。有效地解决了利用ZFN治疗存在的问题:(1)效率高,STAT6敲除效率达到80%以上;(2)既可以针对STAT6的单个编码序列也可以针对片段进行同时敲除。sgRNA只需要小量合成多核苷酸片断,就能大批量生产,节约成本提高了效率。
为了解决上述技术问题,本申请的技术方案如下:
一、sgRNA寡核普酸的设计和选择
1.靶向STAT6基因的sgRNA的设计:
因为没有使用体外转录,只是构建普通载体的方式制作。所以如无特殊说明,文中的sgRNA序列指的是sgRNA对应DNA序列。
首先在STAT6基因上选择5’-GGN(19)GG的序列,如果没有5’-GGN(19)GG的序列,5’-GN(20)GG或者5’-N(21)GG也可以。sgRNA在STAT6基因上的靶向位点位于基因的外显子。在UCSC数据库中用BLAT或NCBI数据库中用BLAST,确定sgRNA的靶序列是否唯一。同时根据其他的sgRNA的设计规则,共设计了97个sgRNA,然而通过最后的实验证实,其中只有15个具有靶向修饰的功能,在此,没有功能的序列就不一一列举,仅仅给出3个反例,这也充分说明在现有技术中,sgRNA的设计并不能仅仅根据设计规则不需要实验即可得到有功能的sgRNA,本发明涉及的sgRNA序列如下:
STAT6-sg1:ggaagtgcccgctgagaaagg(SEQ ID NO:2)
STAT6-sg2:gcggcatcttctgggtgactgg(SEQ ID NO:3)
STAT6-sg3:cgacgccttctgctgcaacttgg(SEQ ID NO:4)
STAT6-sg4:gtccagcaccttcaggcctcgg(SEQ ID NO:5)
STAT6-sg5:acccttgagagcatatatcagagg(SEQ ID NO:6)
STAT6-sg6:gaagaactcaagtttaagacagg(SEQ ID NO:7)
STAT6-sg7:gccctggccatgctactgcagg(SEQ ID NO:8)
STAT6-sg8:gtcaccagttgcttcctggtgg(SEQ ID NO:9)
STAT6-sg9:gcgggagctgagtgtgcctcagg(SEQ ID NO:10)
STAT6-sg10:ccaggccctgtctctgcccctgg(SEQ ID NO:11)
STAT6-sg11:ggacaatgccttctctgagatgg(SEQ ID NO:12)
STAT6-sg12:caatgacaacagcctcagtatgg(SEQ ID NO:13)
STAT6-sg13:ggacctcaccaaacgctgtctccgg(SEQ ID NO:14)
STAT6-sg14:cactacaagcctgaacagatgg(SEQ ID NO:15)
STAT6-sg15:ggcccagatatggtgccccagg(SEQ ID NO:16)
STAT6-sg16:gagagcctggccccactccagg(SEQ ID NO:17)
STAT6-sg17:tccgctggtcagggccgacatgg(SEQ ID NO:18)
STAT6-sg18:tcccagagcacttcctcttcctgg(SEQ ID NO:19)
二、构建sgRNA的寡聚核苷酸双链
根据选择的sgRNA,在其5’加上CCGG得到正向寡核苷酸(Forward oligo)(如果序列本身在5’端已经有I或者2个G,那么就对应的省略I或者2个G);根据选择的sgRNA,获得其对应DNA的互补链,并且在其5’加上AAAC得到反向寡核苷酸(Reverse oligo)。分别合成上述正向寡核苷酸和反向寡核苷酸,将合成的sgRNA寡聚核苷酸的forward oligo和reverse oligo成对变性、退火,退火之后形成可以连入U6真核表达载体的双链,如下:
针对STAT6-sg1:ggaagtgcccgctgagaaagg,设计的正向寡核苷酸(Forward oligo)为:CCggaagtgcccgctgagaaagg(SEQ ID NO:20);反向寡核苷酸(Reverse oligo)为aaaccctttctcagcgggcac(SEQ ID NO:21)。
三、sgRNA寡聚核普酸质粒的构建
1.线性化pGL3-U6-sgRNA质粒。
2.将退火的sgRNA寡聚核普酸双链与线性化pGL3-U6-sgRNA质粒连接获得pGL3-U6-stat6-sg1质粒。
3.转化并涂Amp+平板(50微克/ml)。
4.用通用引物U6测序的方法鉴定阳性克隆,所述引物为atggactatcatatgcttaccgta。
5.37度摇床摇菌过夜并用质粒抽提试剂盒抽提pGL3-U6-hPDlsg质粒。
四、转染细胞获得STAT6基因敲除细胞
1、按照LipofectamineTM2000Transfection Reagent(Invitrogen,11668-019)的操作手册,将分别带有对应sgRNA寡聚核苷酸的pGL3-U6-stat6-sg1质粒与pST1374-NLS-flag-Cas9-ZF质粒混匀,共转染细胞。
2、用T7EN1酶切检测和TA克隆测序确认STAT6基因己经被敲除。
附图说明
图1:CRISPR系统的工作原理
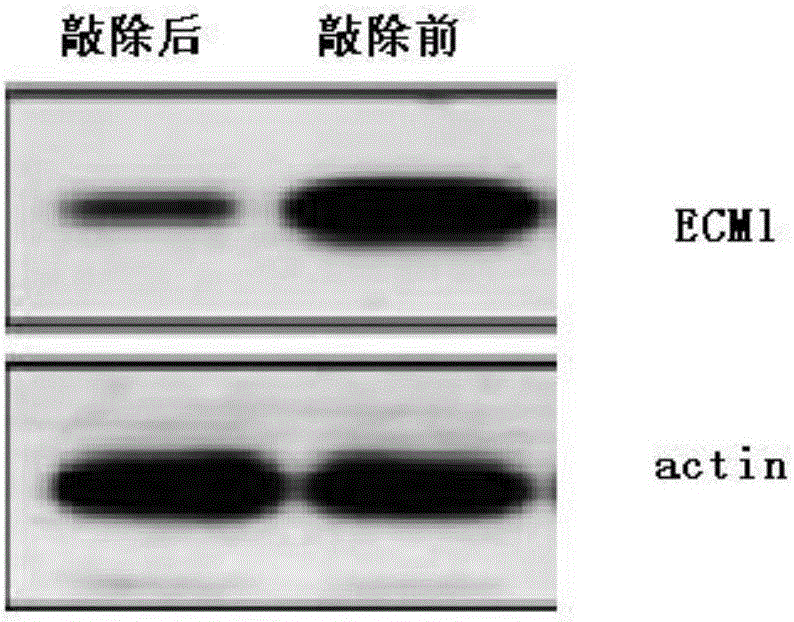
图2:STAT6基因敲除前后细胞内ECMl的蛋白表达量的差异图
具体实施方式
下面结合附图和具体的实施例对本发明的技术方案做进一步介绍。
实施例1sgRNA的设计
首先根据SEQ ID NO:1所示的STAT6基因上选择5’-GGN(19)GG的序列,如果没有5’-GGN(19)GG的序列,5’-GN(20)GG或者5’-N(21)GG也可以。sgRNA在STAT6基因上的靶向位点位于基因的外显子。在UCSC数据库中用BLAT或NCBI数据库中用BLAST,确定sgRNA的靶序列是否唯一。同时根据其他的sgRNA的设计规则,共设计了97个sgRNA,然而通过最后的实验证实,其中只有15个具有靶向修饰的功能,在此,没有功能的序列就不一一列举,仅仅给出3个反例,这也充分说明在现有技术中,sgRNA的设计并不不能仅仅根据设计规则不需要实验即可得到有功能的sgRNA,本发明涉及的sgRNA序列如下:
STAT6-sg1:ggaagtgcccgctgagaaagg(SEQ ID NO:2)
STAT6-sg2:gcggcatcttctgggtgactgg(SEQ ID NO:3)
STAT6-sg3:cgacgccttctgctgcaacttgg(SEQ ID NO:4)
STAT6-sg4:gtccagcaccttcaggcctcgg(SEQ ID NO:5)
STAT6-sg5:acccttgagagcatatatcagagg(SEQ ID NO:6)
STAT6-sg6:gaagaactcaagtttaagacagg(SEQ ID NO:7)
STAT6-sg7:gccctggccatgctactgcagg(SEQ ID NO:8)
STAT6-sg8:gtcaccagttgcttcctggtgg(SEQ ID NO:9)
STAT6-sg9:gcgggagctgagtgtgcctcagg(SEQ ID NO:10)
STAT6-sg10:ccaggccctgtctctgcccctgg(SEQ ID NO:11)
STAT6-sg11:ggacaatgccttctctgagatgg(SEQ ID NO:12)
STAT6-sg12:caatgacaacagcctcagtatgg(SEQ ID NO:13)
STAT6-sg13:ggacctcaccaaacgctgtctccgg(SEQ ID NO:14)
STAT6-sg14:cactacaagcctgaacagatgg(SEQ ID NO:15)
STAT6-sg15:ggcccagatatggtgccccagg(SEQ ID NO:16)
STAT6-sg16:gagagcctggccccactccagg(SEQ ID NO:17)
STAT6-sg17:tccgctggtcagggccgacatgg(SEQ ID NO:18)
STAT6-sg18:tcccagagcacttcctcttcctgg(SEQ ID NO:19)
实施例2、构建sgRNA的寡聚核苷酸双链
根据选择的sgRNA:STAT6-sg1,在其5’加上CCGG得到正向寡核苷酸(Forward oligo)(如果序列本身在5’端已经有1或者2个G,那么就对应的省略1或者2个G);根据选择的sgRNA,获得其对应DNA的互补链,并且在其5’加上AAAC得到反向寡核苷酸(Reverse oligo)。分别合成上述正向寡核苷酸和反向寡核苷酸,针对STAT6-sg1:ggaagtgcccgctgagaaagg,设计的正向寡核苷酸(Forward oligo)为:CCggaagtgcccgctgagaaagg(SEQ ID NO:20);反向寡核苷酸(Reverse oligo)为aaaccctttctcagcgggcac(SEQ ID NO:21)。
将合成的sgRNA寡聚核苷酸的forward oligo和reverse oligo成对变性、退火,退火之后形成可以连入U6真核表达载体的双链,如下:
所述条件为:2.5μl forward Oligo(100μM),2.5μl reverse Oligo(100μM),1u 1NEB buffer,4μl灭菌水。在PCR仪中按照以下touch down程序运行:95度,5min;95-82度at-1.8度/s;85-25度at-0.1度/s;hold at4度。
实施例3、sgRNA寡聚核普酸质粒的构建
1.线性化pGL3-U6-sgRNA质粒。酶切体系和条件如下:2μg pGL3-U6-sgRNA(400ng/u 1);1μ1CutSmart Buffer;1μ1BsaI(NEB,R0535L);补水至50μ1,37度孵育3-4小时,每隔一段时间振荡一下并离心以防液滴蒸发至管盖上。
2.将退火的sgRNA寡聚核苷酸双链与线性化pGL3-U6-sgRNA质粒连接获得pGL3-U6-stat6-sg1质粒。
3.转化并涂Amp+平板(50微克/ml)。
4.用通用引物U6测序的方法鉴定阳性克隆,所述引物为atggactatcatatgcttaccgta。
5.37度摇床摇菌过夜并用质粒抽提试剂盒抽提pGL3-U6-stat6-sg1质粒。
实施例4、转染细胞获得STAT6基因敲除细胞
(1)HEK293T细胞接种培养于DMEM高糖培养液中(HyClone,SH30022.01B),其中含10%FBS,penicillin(100U/ml)和streptomycin(100微克/ml)。(2)在转染前分至12孔板中,待80%密度时进行转染。按照LipofectamineTM2000Transfection Reagent(Invitrogen,11668-019)的操作手册,将分别带有对应sgRNA寡聚核苷酸的pGL3-U6-stat6-sg1质粒1微克与pST1374-NLS-flag-Cas9-ZF质粒2微克混匀,共转染至每孔细胞中,6.5h换液,加入Blasticidin和Puromycin药筛,48小时后收集细胞。
T7EN1酶切检测
将收集的细胞在裂解液(10u M Tris-HCl,0.4M NaCl,2u M EDTA,1%SDS)中用100μg/ml蛋白酶K裂解消化后,酚-氯仿抽提后溶解到50u 1去离子水中。(2)使用引物,上游序列为:agagctccag ggagggacct gg,下游引物为:cagattgtgtacagtagattat(4010bp扩增大小)进行PCR扩增,纯化获得PCR回收产物,取200ng统一稀释到20u 1,在20u 1体系中加入T7EN1 0.3u 1,370C酶切30分钟后,加入2u 1lOXLoading Buffer,用2.5%的琼脂糖胶电泳检测。结果显示,通过琼脂糖胶电泳可以发现:发生断裂末端连接修复的基因组因为与原基因组不完全匹配,而被T7EN1切割。显示出较小的条带。说明基因敲除成功。
将上述步骤获得的PCR回收产物用rTaq进行加A反应。加A反应体系为:800ng PCR回收产物,5u 1lOX Buffer(Mg2+free),3u 1Mg2+,4u 1dNTP,0.5u 1rTaq(TAKARA,ROOlAM),补水至50u 1体系。
37度温育30分钟后,取lul产物与pMD19-T vector连接并转化感受态细胞DH5a。挑取单克隆以通用引物U6序列atggactatcatatgcttaccgta测序,根据测序结果发现:靶基因STAT6缺失了sgRNA靶序列,基因敲除成功。
实施例5
将HEK293T细胞敲除前后的各取1X 106细胞,收获后,用新鲜加入了各种抑制剂(1mM Na orthovanadate,1mMPMSF,10μg/ml Aprotinin,Leupeptin,胃酶抑素)的2Oμl裂解液(5OmMHEPES[pΗ7.0],1%NP-40,5mM EDTA,450mM NaCl,IOmM Na pyrophosphate和50mM NaF)在室温下超声处理后,加入I%β巯基乙醇,100℃中放置5分钟。在SDS-PAGE胶中,每孔上样10μ1样品。电泳后转膜将蛋白样品转移到硝酸纤维素膜上。转膜结束后用TTBS洗膜一次,用5%脱脂奶粉封闭膜1小时,用TTBS洗膜一次,将稀释后的一抗与膜室温杂交2小时或4℃过夜。用TTBS洗涤三次后将稀释后的二抗与膜室温杂交1小时,用TTBS洗涤后加入底物显色,暗室曝光,其中以actin作为内参蛋白对照。
结果显示,敲除了STAT6基因的细胞中的ECMl蛋白的表达相对于原始细胞,蛋白的表达量显著下降86.4%,这也充分的说明,通过敲除STAT6,可以用于治疗过敏性疾病。
实施例6其它sgRNA效果验证
sgRNA选择STAT6-sg2~18,按照实施例2-5相同的实验方法进行相应的基因敲除以及蛋白水平检测,在此,由于步骤基本相同,具体的操作条件就不一一赘述。通过实验发现,STAT6-sg2~15,这14个sgRNA可以实现基因的敲除,其效率达到了85%,而STAT6-sg16、STAT6-sg17、STAT6-sg18都没有实现基因的敲除,这也说明sgRNA的选择并非简单容易的。
另外蛋白表达水平检测的结果如下:
从以上结果可以看出,STAT6-sg1~15都能够实现基因的敲除,并且都能够达到相似的降低ECMl蛋白表达的效果。
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后,本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申请所附权利要求书所限定的范围。
序列表
〈110〉李蒙
〈120〉一种靶向人STAT6的CRISPR-Cas9系统及其用于治疗过敏性疾病的应用
〈160〉19
〈210〉1
〈211〉4031
〈212〉DNA
〈213〉人工序列
〈400〉STAT6
1 agagctccag ggagggacct gggtagaagg agaagccgga aacagcgggc tggggcagcc
61 actgcttaca ctgaagaggg aggacgggag aggagtgtgt gtgtgtgtgt gtgtgtgtgt
121 gtgtatgtat gtgtgtgctt tatcttattt ttctttttgg tggtggtggt ggaagggggg
181 aggtgctagc agggccagcc ttgaactcgc tggacagagc tacagaccta tggggcctgg
241 aagtgcccgc tgagaaaggg agaagacagc agaggggttg ccgaggcaac ctccaagtcc
301 cagatcatgt ctctgtgggg tctggtctcc aagatgcccc cagaaaaagt gcagcggctc
361 tatgtcgact ttccccaaca cctgcggcat cttctgggtg actggctgga gagccagccc
421 tgggagttcc tggtcggctc cgacgccttc tgctgcaact tggctagtgc cctactttca
481 gacactgtcc agcaccttca ggcctcggtg ggagagcagg gggaggggag caccatcttg
541 caacacatca gcacccttga gagcatatat cagagggacc ccctgaagct ggtggccact
601 ttcagacaaa tacttcaagg agagaaaaaa gctgttatgg aacagttccg ccacttgcca
661 atgcctttcc actggaagca ggaagaactc aagtttaaga caggcttgcg gaggctgcag
721 caccgagtag gggagatcca ccttctccga gaagccctgc agaagggggc tgaggctggc
781 caagtgtctc tgcacagctt gatagaaact cctgctaatg ggactgggcc aagtgaggcc
841 ctggccatgc tactgcagga gaccactgga gagctagagg cagccaaagc cctagtgctg
901 aagaggatcc agatttggaa acggcagcag cagctggcag ggaatggcgc accgtttgag
961 gagagcctgg ccccactcca ggagaggtgt gaaagcctgg tggacattta ttcccagcta
1021 cagcaggagg taggggcggc tggtggggag cttgagccca agacccgggc atcgctgact
1081 ggccggctgg atgaagtcct gagaaccctc gtcaccagtt gcttcctggt ggagaagcag
1141 cccccccagg tactgaagac tcagaccaag ttccaggctg gagttcgatt cctgttgggc
1201 ttgaggttcc tgggggcccc agccaagcct ccgctggtca gggccgacat ggtgacagag
1261 aagcaggcgc gggagctgag tgtgcctcag ggtcctgggg ctggagcaga aagcactgga
1321 gaaatcatca acaacactgt gcccttggag aacagcattc ctgggaactg ctgctctgcc
1381 ctgttcaaga acctgcttct caagaagatc aagcggtgtg agcggaaggg cactgagtct
1441 gtcacagagg agaagtgcgc tgtgctcttc tctgccagct tcacacttgg ccccggcaaa
1501 ctccccatcc agctccaggc cctgtctctg cccctggtgg tcatcgtcca tggcaaccaa
1561 gacaacaatg ccaaagccac tatcctgtgg gacaatgcct tctctgagat ggaccgcgtg
1621 ccctttgtgg tggctgagcg ggtgccctgg gagaagatgt gtgaaactct gaacctgaag
1681 ttcatggctg aggtggggac caaccggggg ctgctcccag agcacttcct cttcctggcc
1741 cagaagatct tcaatgacaa cagcctcagt atggaggcct tccagcaccg ttctgtgtcc
1801 tggtcgcagt tcaacaagga gatcctgctg ggccgtggct tcaccttttg gcagtggttt
1861 gatggtgtcc tggacctcac caaacgctgt ctccggagct actggtctga ccggctgatc
1921 attggcttca tcagcaaaca gtacgttact agccttcttc tcaatgagcc cgacggaacc
1981 tttctcctcc gcttcagcga ctcagagatt gggggcatca ccattgccca tgtcatccgg
2041 ggccaggatg gctctccaca gatagagaac atccagccat tctctgccaa agacctgtcc
2101 attcgctcac tgggggaccg aatccgggat cttgctcagc tcaaaaatct ctatcccaag
2161 aagcccaagg atgaggcttt ccggagccac tacaagcctg aacagatggg taaggatggc
2221 aggggttatg tcccagctac catcaagatg accgtggaaa gggaccaacc acttcctacc
2281 ccagagctcc agatgcctac catggtgcct tcttatgacc ttggaatggc ccctgattcc
2341 tccatgagca tgcagcttgg cccagatatg gtgccccagg tgtacccacc acactctcac
2401 tccatccccc cgtatcaagg cctctcccca gaagaatcag tcaacgtgtt gtcagccttc
2461 caggagcctc acctgcagat gccccccagc ctgggccaga tgagcctgcc ctttgaccag
2521 cctcaccccc agggcctgct gccgtgccag cctcaggagc atgctgtgtc cagccctgac
2581 cccctgctct gctcagatgt gaccatggtg gaagacagct gcctgagcca gccagtgaca
2641 gcgtttcctc agggcacttg gattggtgaa gacatattcc ctcctctgct gcctcccact
2701 gaacaggacc tcactaagct tctcctggag gggcaagggg agtcgggggg agggtccttg
2761 ggggcacagc ccctcctgca gccctcccac tatgggcaat ctgggatctc aatgtcccac
2821 atggacctaa gggccaaccc cagttggtga tcccagctgg agggagaacc caaagagaca
2881 gctcttctac tacccccaca gacctgctct ggacacttgc tcatgccctg ccaagcagca
2941 gatggggagg gtgccctcct atccccacct actcctgggt caggaggaaa agactaacag
3001 gagaatgcac agtgggtgga gccaatccac tccttccttt ctatcattcc cctgcccacc
3061 tccttccagc actgactgga agggaagttc aggctctgag acacacccca acatgcctgc
3121 acctgcagcg cgcacacgca cgcacacaca catacagagc tctctgaggg tgatggggct
3181 gagcaggagg ggggctgggt aagagcacag gttagggcat ggaaggcttc tccgcccatt
3241 ctgacccagg gcctaggacg gataggcagg aacatacaga cacatttaca ctagaggcca
3301 gggatagagg atattgggtc tcagccctag gggaatggga agcagctcaa gggaccctgg
3361 gtgggagcat aggaggggtc tggacatgtg gttactagta caggttttgc cctgattaaa
3421 aaatctccca aagccccaaa ttcctgttag ccaggtggag gcttctgata cgtgtatgag
3481 actatgcaaa agtacaaggg ctgagattct tcgtgtatag ctgtgtgaac gtgtatgtac
3541 ctaggatatg ttaaatgtat agctggcacc ttagttgcat gaccacatag aacatgtgtc
3601 tatctgcttt tgcctacgtg acaacacaaa tttgggaggg tgagacactg cacagaagac
3661 agcagcaagt gtgctggcct ctctgacata tgctaacccc caaatactct gaatttggag
3721 tctgactgtg cccaagtggg tccaagtggc tgtgacatct acgtatggct ccacacctcc
3781 aatgctgcct gggagccagg gtgagagtct gggtccaggc ctggccatgt ggccctccag
3841 tgtatgagag ggccctgcct gctgcatctt ttctgttgcc ccatccaccg ccagcttccc
3901 ttcactcccc tatcccattc tccctctcaa ggcaggggtc atagatccta agccataaaa
3961 taaattttat tccaaaataa caaaataaat aatctactgt acacaatctg aaaagaaaaa
4021 aaaaaaaaaa a
〈210〉2
〈211〉21
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg1
ggaagtgcccgctgagaaagg
〈210〉3
〈211〉22
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg2
gcggcatcttctgggtgactgg
〈210〉4
〈211〉23
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg3
cgacgccttctgctgcaacttgg
〈210〉5
〈211〉22
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg4
gtccagcaccttcaggcctcgg
〈210〉6
〈211〉24
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg5
acccttgagagcatatatcagagg
〈210〉7
〈211〉23
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg6
gaagaactcaagtttaagacagg
〈210〉8
〈211〉22
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg7
gccctggccatgctactgcagg
〈210〉9
〈211〉22
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg8
gtcaccagttgcttcctggtgg
〈210〉10
〈211〉24
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg9
gcgggagctgagtgtgcctcaggg
〈210〉11
〈211〉23
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg10
ccaggccctgtctctgcccctgg
〈210〉12
〈211〉23
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg11
ggacaatgccttctctgagatgg
〈210〉13
〈211〉23
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg12
caatgacaacagcctcagtatgg
〈210〉14
〈211〉25
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg13
ggacctcaccaaacgctgtctccgg
〈210〉15
〈211〉22
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg14
cactacaagcctgaacagatgg
〈210〉16
〈211〉22
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg15
ggcccagatatggtgccccagg
〈210〉17
〈211〉22
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg16
gagagcctggccccactccagg
〈210〉18
〈211〉23
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg17
tccgctggtcagggccgacatgg
〈210〉19
〈211〉24
〈212〉DNA
〈213〉人工序列
〈400〉STAT6-sg18
tcccagagcacttcctcttcctgg
- 还没有人留言评论。精彩留言会获得点赞!