长链非编码RNALINC00673的应用的制作方法
本发明属于肿瘤分子生物学领域,具体涉及长链非编码RNA LINC00673的应用方法。
背景技术:
口腔颌面部恶性肿瘤是常见高发的头颈部恶性肿瘤,容易发生颈部淋巴结转移,预后较差。研究表明,这种肿瘤的发生发展是多基因参与、多步骤、多阶段的复杂过程,其中抑癌基因的失活与癌基因的激活发挥了关键的作用。
长链非编码RNAs(long non-coding RNAs,lncRNAs)是一类长度超过200nt、缺少特异完整的开放阅读框、无或很少有蛋白编码功能的RNAs分子。最近的研究表明,lncRNAs通过在表观遗传、转录和转录后水平广泛调节基因的表达,它们参与了生物体生长、发育、衰老及死亡等重要生命活动的调控。越来越多的研究表明lncRNAs的异常表达或功能缺失与肿瘤的发生发展密切相关。一些lncRNAs分子在肿瘤的诊断和治疗方面具有重要的意义,且可以作为肿瘤预后判断新的分子标记。目前,已经克隆的人类lncRNAs基因已超过4万多个,但绝大部分lncRNA的功能未知。
我们检测了LINC00673在临床离体肿瘤样本中的表达水平,并分析了LINC00673表达水平与临床病例资料的相关性。我们从口腔肿瘤及相对应的正常对照组织中抽提RNA后通过逆转录,实时定量PCR,检测了LINC00673的表达情况,结果显示LINC00673在口腔肿瘤组织中表达上调。我们随后又利用原位杂交的方法,进一步在存档石蜡样本验证了LINC00673在口腔肿瘤中表达显著上调,因此针对LINC00673的检测制剂可用于口腔肿瘤的辅助诊断,结合临床回访资料进行生存曲线分析发现LINC00673表达高的患者其生存时间短于该lncRNA表达低的患者,因此针对该lncRNA的检测制剂也可以用于口腔肿瘤的预后判断。
我们还针对该lncRNA基因的序列设计并合成了用于抑制LINC00673表达的小RNA干扰序列(siRNAs),利用siRNAs在口腔肿瘤细胞系中抑制了LINC00673的表达,且发现抑制该lncRNA表达可抑制口腔肿瘤细胞的侵袭与迁移能力。因此针对该lncRNA的抑制制剂也具有口腔肿瘤基因治疗的潜在用途。
技术实现要素:
本发明的目的是提供lncRNALINC00673的应用方法,利用检测LINC00673的试剂可以制备用于口腔肿瘤辅助诊断或者预后预测的制剂。
长链非编码RNA LINC00673的应用,检测长链非编码RNA LINC00673的试剂用于制备口腔肿瘤辅助诊断或者疗效预测的试剂,该长链非编码RNA LINC00673的序列见SEQ NO:1。
所述的口腔肿瘤辅助诊断或者疗效预测的试剂为实时荧光定量检测制剂。用于实时荧光定量检测LINC00673表达的引物序列:
正向引物:5’-TTCTCCTGTAACGTGTGGCC-3’,
反向引物:5’-CTGGTGGGAATGTGGATCAGT-3’。
一种口腔肿瘤辅助诊断或者疗效预测的试剂盒,包括用于实时荧光定量检测LINC00673表达的引物:
正向引物:5’-TTCTCCTGTAACGTGTGGCC-3’,
反向引物:5’-CTGGTGGGAATGTGGATCAGT-3’。
试剂盒中还含有内参基因GAPDH特异性PCR引物:
正向引物5’-ACCACAGTCCATGCCATCAC-3’
反向引物5’-TCCACCACCCTGTTGCTGTA-3’。
试剂盒中还含有:
(1)从口腔肿瘤组织中抽提总RNA所用试剂,包括RNA稳定溶液、Trizol试剂、三氯甲烷、异丙醇、无酶水;
(2)以总RNA为模板将LncRNA LINC00673逆转录为cDNA所用试剂,包括逆转录缓冲液、三磷酸碱基脱氧核苷酸、RNA酶抑制剂、MMLV逆转录酶以及随机引物;
(3)将cDNA实时定量PCR所用试剂,包括实时荧光定量SYBR染料、无酶水。
我们从口腔肿瘤及正常口腔上皮样本中抽提RNA后逆转录,实时荧光定量法检测了LINC00673的表达,结果显示LINC00673在口腔肿瘤组织中表达上调。提示LINC00673可作为口腔肿瘤预后预测的分子标志。本发明为口腔肿瘤的辅助诊断和预后预测提供了强有力的分子生物学工具,具有深远的临床意义和重要的推广应用前景。
附图说明
图1为利用实时荧光定量PCR的方法检测口腔肿瘤离体样本中LINC00673的表达水平;
LINC00673在口腔肿瘤(T)中的表达比正常对照(N)样本中显著上调;
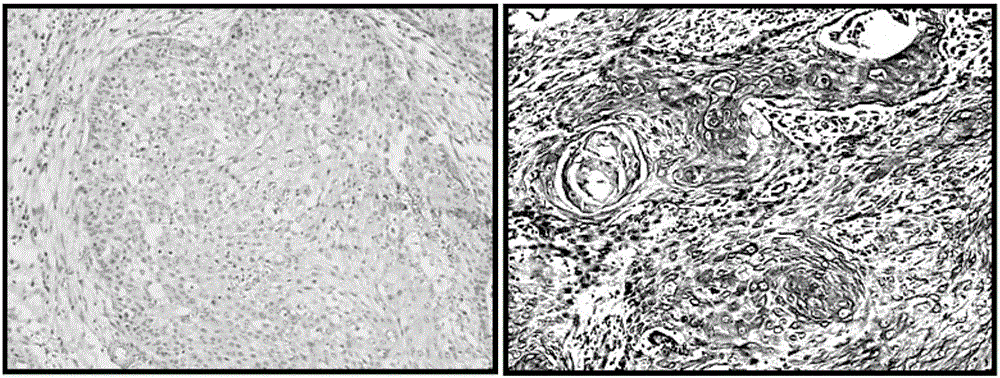
图2原位杂交检测LINC00673在口腔肿瘤及正常口腔粘膜中的表达;
左图为正常口腔粘膜组织,右图为口腔肿瘤组织(放大倍数:200×),LINC00673在口腔肿瘤中表达水平较高;
图3为口腔肿瘤中LINC00673的表达与患者预后的相关性;
LINC00673低表达的患者预后比LINC00673高表达或不表达者好,即LINC00673高表达的患者更快死亡。
图4为在口腔癌细胞中导入LINC00673的干扰siRNA序列,可显著抑制LINC00673在口腔癌细胞中的表达;图中si673为导入LINC00673的干扰siRNA序列的口腔癌细胞,NC为阴性对照;
在口腔癌细胞系Tca8113中导入靶向LINC00673的siRNA混合物后,实时荧光定量PCR方法检测了口腔癌细胞中LINC00673的表达情况,LINC006730的表达均受到明显的抑制。阴性对照(NC)为Scramble干扰siRNA序列。
图5为在口腔癌细胞中导入LINC00673的siRNA抑制LINC00673表达后,细胞迁移能力降低;图中si673为导入LINC00673的干扰siRNA序列的口腔癌细胞,NC为阴性对照;
细胞划痕实验证实,在口腔癌细胞系Tca8113中转入靶向LINC00673的siRNA,抑制LINC00673的表达后,划痕愈合速度减慢,表明细胞向划痕中央迁移的能力降低。
图6为在口腔癌细胞中导入LINC00673的siRNA抑制LINC00673表达后,细胞侵袭能力降低;图中si673为导入LINC00673的干扰siRNA序列的口腔癌细胞,NC为阴性对照;
细胞穿膜(transwell)实验证实,在口腔癌细胞系Tca8113中转入靶向LINC00673的siRNA,抑制LINC00673的表达后,能穿过基质胶膜的口腔癌细胞数目显著减少,表明细胞侵袭能力降低。
具体实施方式
以下结合具体实施方式进一步说明本发明,而非限制本发明。
实施例1,实时荧光定量法检测证实LINC00673在口腔肿瘤中表达上调
1.材料与方法:
15例正常口腔粘膜和15例口腔肿瘤组织抽提总RNA,2μg RNA经逆转录成cDNA后,进行实时荧光定量PCR。
正向引物:5’-TTCTCCTGTAACGTGTGGCC-3’,
反向引物:5’-CTGGTGGGAATGTGGATCAGT-3’。
用于对照的内参看家基因GAPDH特异性PCR引物:
正向引物5’-ACCACAGTCCATGCCATCAC-3’
反向引物5’-TCCACCACCCTGTTGCTGTA-3’。
实时荧光定量PCR反应体系
实时荧光定量PCR反应步骤
反应结束后确认实时荧光定量PCR的扩增曲线和熔解曲线,各基因的表达强度根据CT值(threshold cycle values)、内参基因(GAPDH)标化后,采用group t-test检验计算P值。
2.结果
LncRNA LINC00673在正常对照组织中表达较低,而在口腔肿瘤组织中有较高表达P=0.02(图1)
实施例2,原位杂交检测发现LINC00673在口腔肿瘤中的表达与患者预后相关
1.材料方法
1.1用于口腔肿瘤和正常对照组织原位杂交的寡核苷酸探针
根椐LINC00673基因序列分析设计出杂交参数最佳特异性最好的3条寡核苷酸探针,另外,以看家基因GAPDH为阳性对照。
用于原位杂交检测LINC00673表达的寡核苷酸探针:
LINC00673探针1:
5’-GAAAAACCTCTTGCACCACCTTAGTCTCCAAAGA-3’;
LINC00673探针2:5’-CTTTCCTGTTCTTTCTCCTACCCTTCCTGACTAG-3’;
LINC00673探针3:5’-CATGAAGTAATAATAAAGGTTCCGCTTATCAACC-3’。
阳性对照探针(检测看家基因GAPDH):
GAPDH探针1:5'-CCACTTTACCAGAGTTAAAAGCAGCCCTGG-3';
GAPDH探针2:5'-GTCAGAGGAGACCACCTGGTGCTCAGTGTA-3';
GAPDH探针3:5’-CAGTAGAGGCAGGGATGATGTTCTGGAGAG-3’。
寡核苷酸探针采用化学合成方法合成
1.2寡核苷酸探针标记试剂盒和原位杂交检测试剂
Dig Oligonucleitide Tailing Kit(2nd Generation)试剂盒(Roche公司),抗地高辛-辣根过氧化物酶复合物检测试剂盒(Anti-Digoxigenin-POD,Fab fragments,Roche公司)。增强原位表达检测信号的TSA信号放大系统(TSATM Biotin System,NEL700试剂盒,PerkinElmer公司)。DAB染色试剂盒(北京中山公司)。20×SSC,硫酸葡聚糖(Dextran sulphate),去离子甲酰胺(Deionized Formamide),多聚腺苷酸(polyadenylic acid,Poly A),多聚脱氧腺苷酸(polydeoxyadenylic acid,Poly dA),变性剪切的蛙精DNA(denatured and sheared salmon sperm DNA,ssDNA),酵母转运RNA(yeast t-RNA,tRNA),DTT,50×Denhardts’s solution,PBS buffer,胃蛋白酶K,BSA(牛血清清蛋白),三乙醇胺(TEA),TNB Buffer(0.1M Tris-HCl,pH7.5,0.15M NaCL,0.5%Blocking Reagent),TNT Buffer(0.1M Tris-HCl,pH7.5,0.15M NaCL,0.05%Tween 20)醋酸酐,阻断试剂(Blocking reagent agent,Roche公司)。
1.3其他主要试剂和材料
无水乙醇、90%酒精、70%酒精、50%酒精、松节油、双蒸水、PBS缓冲液(pH7.2~7.4,NaCl 137mmol/L,KCl 2.7mmol/L,Na2HPO4 4.3mmol/L,KH2PO41.4mmol/L);3%甲醇-双氧水溶液(80%甲醇和30%双氧水配置);0.01mol/L柠檬酸盐缓冲液(citrate buffer,CB,pH6.0±0.1,9ml 0.1M柠檬酸溶液和41ml 0.1M柠檬酸钠溶液加入450ml蒸馏水中临时配置后再校正工作液pH值);0.1%胰蛋白酶;苏木素;1%盐酸酒精(1ml浓盐酸+99ml 70%酒精配置);组织微阵列专用封片胶(PTS Cure MountⅡ);专用盖玻片(480×240mm2)定制于郑州玻璃仪器厂。Leica低熔点(58℃)石蜡,国产蜂蜡,无水酒精,二甲苯,10%中性多聚甲醛(0.01mol/L,pH7.4,DEPC双蒸水和PBS缓冲液配制),苏木素,伊红,中性封片树胶,盖玻片,载玻片。
1.4寡核苷酸探针的标记
利用3-tailing DIG Olignucleutide Kit进行寡核苷酸探针标记,反应体系如下。
100pmol oligonucleotide+ddH2O=9μl(control:control oligonucleutide 5μl+ddH2O 4μl)
混匀,稍离心。37℃水浴反应30min,加2ul EDTA(0.2M,pH 8.0)中止反应。
1.5寡核苷酸探针标记后纯化
为了增加标记探针的纯度,需对已标记的探针进行纯化,具体操作如下:
a)探针反应混合物(22μl)+2.5μl 4M LiCL+75μl 100%冷乙醇(-20℃).
b)-70℃沉淀60min,or-20℃2h。
c)13.000×g 4℃离心15min。
d)弃上清,用50μl冰冷的70%(V/V)乙醇洗涤。
e)13.000xg 4℃,离心5min。
f)弃上清,真空4℃干燥。
g)用无菌双蒸水重溶探针。
1.6组织切片杂交前处理
a)4℃保存的组织切片置于58℃烤片30min,熔化表面石蜡。
b)二甲苯依次脱蜡3×5min。
c)梯级酒精洗涤,100%酒精2×2min→95%酒精1×5min→70%酒精1×5min→50%酒精1×5min→DEPC水洗涤2×3min→DEPC-PBS洗涤2×5min。
d)滴加300μl胃蛋白酶K(10μg/ml)于切片上,37℃消化20min。
e)切片入PBS(0.1M PBS+2mg/ml谷氨酸)洗涤1min,中止反应。
f)切片入0.2N HCL,于37℃反应20-30min,增加组织的通透性。
g)切片用4%多聚甲醛(0.1M PBS溶解)后固定10min,室温。
h)为了增加组织阳性杂交强度,对切片进行乙酰化处理。切片入0.25%乙酸酐BufferⅠ(0.1M三乙醇胺),室温10min。
i)1M PBS洗涤2×5min。
1.7组织预杂交和杂交
a)预杂交:-20℃保存的预杂交液,先置于37℃孵育60min,预杂交液的用量为每平方厘米切片面积12.5μl,相应大小的石蜡膜覆盖切片,37℃湿盒中预杂交2小时。(预杂交液成份包括:2XSSC,10%Dextran sulphate,1X Denhardt’s solution,50mM Phosphate Buffer(PH 7.0),50mM DTT,250μl,100μg/ml poly A,5μg/ml poly dA,250μg/ml yeast t-RNA,500μg/ml ssDNA,47%Deionized formamide)。
b)移去石蜡膜,甩掉预杂交液,切片置于2×SSC中5min。
c)杂交反应:37℃杂交过夜(18-20h)。每一切片加入250μl杂交液并用石蜡膜覆盖。预杂交液中加入相应的探针就成为杂交液。杂交液在预杂交时配制,放置37℃孵育,使探针充分溶解于杂交液中,本实验用多条寡核苷酸探针混合,按每一探针500ng/ml浓度配制成探针杂交液。地高辛加尾标记试剂盒标记探针浓度计算依据:每一探针的浓度按其与阳性定量探针时检测反应时显色进行比对和100pmol 30个碱基的裸探针标记反应理论探针产量为900ng两种标准进行综合计算出标记探针的浓度。
d)杂交后洗涤,切片浸入2×SSC,10min,揭去石蜡膜。依次于摇床上摇动洗涤,2×SSC(0.5%SDS),2×15min→0.25×SSC(0.5%SDS),2×15min。
1.8杂交后显色检测反应
a)采用Anti-Digoxigenin-POD检测地高辛探针与目的RNA结合复合物;TSA放大系统增强原位杂交反应显色反应的阳性信号,DAB显色。
b)切片转至TNT缓冲液中,3×5min。
c)滴加TNB阻断缓冲液,300μl/TMAs,室温,30min。
d)不洗,吸去多余阻断剂,1:100稀释的Anti-Digoxigenin-POD(TBS+0.1%Triton X-100+1%阻断剂),室温4小时。
e)TNT Buffer(0.1M Tris-HCl,PH7.5,0.15M NaCL,0.05%Tween 20)洗3×5min。
f)切片上滴加信号放大试剂Biotinyl Tyamid,300μl/TMAs,(Biotinyl Tyramid贮存液:Biotinyl Tyramid溶解于0.2ml DMSO,Biotinyl Tyramid工作液:1×稀释液,1:50稀释Biotinyl Tyramid贮存液),室温10分钟。
g)TNT洗,3×5min。
h)切片滴加SA-HRP(链霉卵白素-辣根过氧化物酶),300μl/TMAs,室温30min。
i)TNT洗,3×5min。
j)蒸溜水洗涤,1×1min。
k)DAB显色,显微镜下控制显色反应。
l)苏木素复染,
m)酒精梯级脱水,切片干燥。
n)滴加组织切片专用封片胶,相应规格的盖玻片盖片,切片胶带转移系统配备的紫外灯下交联切片1min。
1.9结果判断及标准
应用光学显微镜分别在低倍和高倍镜下进行观察,首先观察目标RNA的阳性表达信号在观察目标细胞内的定位:位于细胞核、细胞浆或细胞膜。
再分别以该检测RNA表达部位阳性信号的强度和阳性表达的细胞数两种标准进行综合评分,判断标准为:(1)依据阳性细胞染色强度判断:a.细胞无染色,记0分;b.细胞染成浅棕色为弱阳性,记1分;c.细胞染成棕色且无背景着色,或细胞染成深棕色并有浅棕色背景为中等阳性,记2分;d.细胞染成深棕色且无背景着色为强阳性,记3分。(2)依据阳性细胞表达数计分:a.无阳性细胞表达,记0分;b.阳性表达细胞数≤25%,记1分;c.25%<阳性细胞数<50%,记2分;d.阳性表达细胞数≥50%,记3分。
为了尽量降低评分结果的主观因素,由两位病理学专家分别按上述标准之一各自进行判断和评分,再将两者评分相乘,结果为:①0分者最终计为0分,认为阴性表达;②1分和2分者最终计为1分,认为弱阳性表达;③3分和4分者最终计为2分,认为中等阳性表达;④6分到9分者最终计为3分,认为强阳性表达。
1.10分析和统计软件
应用SPSS13.0统计软件对实验结果进行统计学分析,两两比较用χ2test或Fisher exact test,相关性分析采用Spearmen correlation方法;P<0.05即差异有统计学意义。生存曲线分析采用Kaplan-Meier method及log-rank test;多变量分析采用Cox’s proportional hazards model;P<0.05即差异有统计学意义。
2结果
1)lncRNA LINC00673在口腔肿瘤及正常对照组织中的表达
LINC00673在正常对照组织中均不表达或者表达很低(图2左),而在大部分口腔肿瘤组织中表达较高(图2右),两者之间具有明显的统计学差异(P<0.05)。
2)Kaplan-Meier生存分析
我们对所有202例口腔肿瘤患者进行了电话随访,详细询问了他们的首发时间、治疗情况、有无复发、有无再患其他疾病、复发及死亡时间等,并登记了生存时间和状态,并对口腔肿瘤组织中LINC00673的表达与病人的生存时间和状态进行的生存分析,发现LINC00673高表达的患者生存时间较LINC00673表达低或者不表达的患者要短(图3)。说明LINC00673是一个与口腔肿瘤预后相关的分子标记,该lncRNA表达低,病人预后好。
实施例3,siRNA干扰LINC00673的表达
1.材料方法
1.1试剂及试剂盒
TRIZOLTM Reagent(Invitrogen);
逆转录试剂盒(#A3500,Promega);
抗生素G418(Ameresc)。
1.2shRNA的设计
首先将LINC00673序列输入Invitrogen公司的Block-It RNAi designer软件,寻找该lncRNA的siRNA最佳靶点,挑选最佳的2条相应的靶点序列如下:
siRNA-1::5'-CCAGUUGUCCUUGACUGCAUGGUUU-3',
siRNA-2:5'-AGGGAACCACAGGAUUCCAUGUGAU-3'
阴性对照Scramble序列:5’-GACACGCGACUUGUACCAC-3’。
1.3细胞培养与转染
口腔癌细胞系Tca8113购自中南大学细胞中心,细胞培养所用RPMI 1640培基和胎牛血清,及消化细胞所用的胰蛋白酶均为美国Gibco公司产品。
将生长状态良好的口腔癌细胞系Tca8113按2×105个细胞/孔接种于6孔板中,将6孔板置于37℃,5%CO2培养箱中,待培养细胞生长至50-70%密度即可开始siRNA的转染;转染过程如下:
在无菌EP管中加入3μl的Hipefect于100μl无血清培养基中混匀静置5min;
将2条siRNA混合加入100μl无血清培养基中;然后与上述包含Hipefect的100μl无血清培养基温和混匀,室温静置30分钟,使siRNA与脂质体形成复合体;
用D-Hank's液洗涤细胞3次;
将上述混合物中加入800μl无血清培养基(无抗生素),温和混匀后加入6孔板中的1个孔;
将6孔板置于CO2培养箱中,37℃培养6小时,然后弃上清,加入完全培养基继续培养48小时。
1.5实时定量PCR检测siRNA干扰lncRNA表达的效果:
将siRNA转染后的口腔癌细胞抽提总RNA,2μg RNA经逆转录成cDNA后,进行实时荧光定量PCR。
LINC00673正向引物:5’-TTCTCCTGTAACGTGTGGCC-3’,
LINC00673反向引物:5’-CTGGTGGGAATGTGGATCAGT-3’。
用于对照的GAPDH
正向引物为5’-ACCACAGTCCATGCCATCAC-3’
反向引物5’-TCCACCACCCTGTTGCTGTA-3’。
实时荧光定量PCR反应体系
实时荧光定量PCR反应步骤
反应结束后确认实时荧光定量PCR的扩增曲线和熔解曲线,各基因的表达强度根据CT值(threshold cycle values)、内参基因(GAPDH)标化后,采用group t-test检验计算P值。
2.结果
siRNA转染口腔癌细胞Tca8113后,可显著下调口腔癌细胞中LINC00673的表达水平(图4)。
实施例5,LINC00673 siRNA抑制口腔癌细胞中LINC00673的表达和口腔癌的侵袭迁移
1.材料方法
1.1细胞培养与转染
将生长状态良好的口腔癌细胞Tca8113按2×105个细胞/孔接种于6孔板中,将6孔板置于37℃,5%CO2培养箱中,待培养细胞生长至50-70%密度即可开始LINC00673 siRNA转染。
1.2细胞划痕实验
细胞划痕实验是验证肿瘤细胞迁移能力的实验方法。转染了LINC00673 siRNA干扰序列或对照序列(scramble)的口腔癌细胞接种于6孔板,待细胞密度达到90%时,用200ul pipet在每个6孔板中划一条直线(划痕),随后在0、24、48、60小时等各个时间点(视不同细胞迁移能力而定)在显微镜下观察划痕愈合情况,拍照,并计算各组细胞迁移速度。
1.3细胞穿膜实验
细胞穿膜((Transwell)实验是验证肿瘤细胞侵袭能力的实验方法。Transwell小室(孔径8μm)及基质胶(Matrigel)购自美国BD公司,4%多聚甲醛固定液、结晶紫染液(0.1%g/ml)购自Sigma公司。将Matrigel按1:8稀释,包被在Transwell小室底部膜的上室面,置37℃30分钟使Matrigel聚合成凝胶。使用前按BD公司说明书进行基质胶膜水化。
在每个Transwell小室上层加入无血清培养基和1×105个转染了LINC00673siRNA和阴性对照的口腔癌细胞,在Transwell小室下层加入含20%胎牛血清的培养基。细胞继续培养36小时后,用4%多聚甲醛固定液固定,结晶紫染色,用棉签轻轻擦掉上层未迁移细胞,用PBS洗3遍。在显微镜下观察穿过基质胶膜的口腔癌细胞。
2.结果
2.1在口腔癌细胞中导入LINC00673的干扰序列抑制LINC00673表达后,细胞迁移能力降低
细胞划痕实验证实,在口腔癌细胞系Tca8113中转入靶向LINC00673的干扰序列,抑制LINC00673的表达后,口腔癌细胞从划痕两边往划痕中央迁移的速度减慢,划痕愈合的时间延长,表明细胞运动迁移能力降低(图5)。
2.2在口腔癌细胞中导入LINC00673 siRNA抑制LINC00673后,细胞侵袭能力降低
细胞穿膜(Transwell)实验证实,在口腔癌细胞系Tca8113中转入靶向LINC00673 siRNA,抑制LINC00673的表达后,能穿过基质胶膜的口腔癌细胞数目显著减少,表明细胞侵袭能力降低(图6)。
SEQUENCE LISTING
<110> 中南大学
<120> 长链非编码RNA LINC00673的应用
<130> 无
<160> 14
<170> PatentIn version 3.3
<210> 1
<211> 2275
<212> RNA
<213> LINC00673 RNA序列
<400> 1
agggcgcgca ggcggcgcgg gugcgcggug cggcgcuggu auccagagga cgcggucacc 60
gccucuggca uuugucguuc ugcgcuucuc cgcaaggacc cucuguuagg caggcgccca 120
ccguaagccu cccgggccuu gugaaccugc aaacccaagu cugagagacg auccgccuuc 180
agcgcuuucc agcuuggcag agaggcuuuc ccggcgggga ucuuugguug gcgcuggcga 240
ugcgcgggga agaaaggcga ggagcggcgu ccaggcuggg ugauguccca gcacgaguag 300
gcgggaugcg cucgcuuggu ccuccgggcg cccggucccu gcccgcgucg cgcgcccacc 360
ccuggggacg agaaggcggc cgccugagga cccccgcccg cgaccuccgc gagucuggag 420
cgcagaggac agggucuggc ugcucuuugg ccuuggaugg aaagugggga auuggguggg 480
gggcugcgga ccccuuaacg uggauuacuu gguguguauc agcugggcuc agaagaccca 540
cgaccucuuc uccauccgug gauugauuug uucugcuuaa cagcuggguc gccaagcugg 600
agguauuuuu cccucuccac ccuggucuuc uccuguaacg uguggccgcc uuuuccagca 660
cggccuccug ccuuccuggu gcacuuuuug gagaacgugg uggaaucaga gguuucuggc 720
ugacucggug ggugcuuuga accaggaaag gacaagaaag aggaugggaa ggacugaucc 780
acauucccac caggaaguuu agcagaaccc ccgcgugcca ccuggacccc uuggaaggac 840
cuggcucagg cuggaccacc ucuugagagg caggagcucu ggauuugauc aagaauucuu 900
ugcugagcau ggugccucau gccuauaauc ccaacacuuu gggaggccag ugugggagga 960
ucucuugagc ccaggaguuc aagacuagcc ugggcaacac agagagaccc caucucuaaa 1020
auaauaauaa uaauaaaaua aaaaauuagc agggcauggu ggcaugugcc uguaguccca 1080
gcuacccagg aggcugaggc aagaggaugg cuggagccug ggauguugag gcugcaauga 1140
acugugauua ccccacugca cuccagccug ggcaaaagag cgagagaccc ugucucaaau 1200
aauaauaaua auaauaaucu uauuuuggag aauaaagaga ccucuggauu ugaggugcca 1260
uuuggguaga aagaaaagac guuuacaccg agaaauaguc uguguugccc ugaaggagca 1320
gagggaugca ucgcuggagg ugaccuacag uugaagaaga cucauuauga cagaccuugu 1380
ccuucuuccu uguggaaagu guuuccucug cugcuacugc ucaugagacu cuucccccuc 1440
ccugucccag ggaaccaaag ggcuuucuac cacacccuuu cuugcccccc gccucccaug 1500
ucugcugugc cuuuguacuc agcaauucuu guuugcucca uuaucuucca gccggauaca 1560
gagugaauag uuaaccacac uuaggucaaa uaggaucuaa auuuuuguuc cugcuccgug 1620
uaaagaggcc aguguuugug uguugcaagc agccuuggaa uaguaacucu ucucauuugu 1680
uugggaucug gccaccaagu uccagaauga uacacggauc agugcagaag uucaucaggc 1740
ucucggaccu uagggcuguu ggagaaggcu ucagcagcag aacugauggu gaaggcucgu 1800
guucuccauc cucaacuuuc uuugcuucga ucauacacaa gaauacauuu ggaagggcaa 1860
aaaaugaaca cugucguuca uugcagccgu guuuugugac acagaugcac agucugcugu 1920
gaagaccuuc ucucaagugg cauuugggag uccaugccag aucauggugc uucaugagag 1980
acugacagcu aucagggguu guggcacuua gugaggacuc uccuccccca gugugugcug 2040
augacacaua cacaccugac aauagcuuga gucuucucug uuccuuuuac ucuguagcca 2100
acauacacau gauuuaaaac ccuuucuaaa uaucuaucau gguucauccu uguccaaaug 2160
cagagucaga gcuauuugua cuucauuauu auuuccaagg cgaauaguug gcuuucuuuu 2220
ugcaaaaaua auuaaaguuu uuguauguug caguugcaaa aaaaaaaaaa aaaaa 2275
<210> 2
<211> 20
<212> DNA
<213> 实时荧光定量检测LINC00673表达的正向引物
<400> 2
ttctcctgta acgtgtggcc 20
<210> 3
<211> 21
<212> DNA
<213> 实时荧光定量检测LINC00673表达的反向引物
<400> 3
ctggtgggaa tgtggatcag t 21
<210> 4
<211> 20
<212> DNA
<213> 内参基因GAPDH特异性PCR正向引物
<400> 4
accacagtcc atgccatcac 20
<210> 5
<211> 20
<212> DNA
<213> 内参基因GAPDH特异性PCR反向引物
<400> 5
tccaccaccc tgttgctgta 20
<210> 6
<211> 34
<212> DNA
<213> 原位杂交检测LINC00673表达的寡核苷酸探针1
<400> 6
gaaaaacctc ttgcaccacc ttagtctcca aaga 34
<210> 7
<211> 34
<212> DNA
<213> 原位杂交检测LINC00673表达的寡核苷酸探针2
<400> 7
ctttcctgtt ctttctccta cccttcctga ctag 34
<210> 8
<211> 34
<212> DNA
<213> 原位杂交检测LINC00673表达的寡核苷酸探针3
<400> 8
catgaagtaa taataaaggt tccgcttatc aacc 34
<210> 9
<211> 30
<212> DNA
<213> GAPDH探针1
<400> 9
ccactttacc agagttaaaa gcagccctgg 30
<210> 10
<211> 30
<212> DNA
<213> GAPDH探针2
<400> 10
gtcagaggag accacctggt gctcagtgta 30
<210> 11
<211> 30
<212> DNA
<213> GAPDH探针3
<400> 11
cagtagaggc agggatgatg ttctggagag 30
<210> 12
<211> 25
<212> RNA
<213> siRNA-1
<400> 12
ccaguugucc uugacugcau gguuu 25
<210> 13
<211> 25
<212> RNA
<213> siRNA-2
<400> 13
agggaaccac aggauuccau gugau 25
<210> 14
<211> 19
<212> RNA
<213> 阴性对照Scramble序列
<400> 14
gacacgcgac uuguaccac 19
- 还没有人留言评论。精彩留言会获得点赞!