使用数字PCR的产前诊断方法与流程
本申请要求2015年10月29日和2016年10月14日,提交的韩国专利申请号10-2015-0151313和10-2016-0133529的优先权,其全部内容通过引用纳入本文。
技术领域
本发明涉及使用数字PCR进行产前诊断的方法。
发明背景
由于例如结婚年龄增加和女性生育年龄增加的原因,胎儿遗传疾病发生的概率在最近10年急速升高。如果在妊娠早期检测到包括胎儿遗传疾病在内的疾病,就能够采取早期医疗措施以确保安全,并且能够减少孕妇和胎儿的痛苦。因此,胎儿遗传异常的早期诊断是非常重要的。胎儿体内可发生的遗传异常包括部分染色体易位、缺失、复制、插入和数目异常(例如,三体性)。当存在这种染色体异常时,在整个胎儿体内会产生结构异常和功能异常。尤其地,众所周知基因数目异常在高龄孕妇中发生频率更高。
通常使用羊膜穿刺术来诊断胎儿遗传异常。然而,羊膜穿刺术是侵入性的测试方法,可能引起各种问题,包括注射器对羊膜的细菌感染、注射器引起的伤口、和羊水泄漏,在严重情况下也可能引起流产。由于这个缘故,近年来,引入了通过从孕妇血液中提取胎儿DNA诊断胎儿遗传异常的方法。然而,孕妇血液中所含的胎儿DNA数量比孕妇的DNA数量少得多,因此胎儿遗传信息的分析准确度很低。与此相关,韩国专利10-1387582披露了一种通过实时PCR能够检测孕妇体内无细胞胎儿核酸的方法,所述实时PCR以存在于孕妇血液中的极少量的无细胞胎儿核酸为目标。所述实时PCR是一种非侵入性方法,但其缺点在于,同时进行多种测试是受限制的,为了精确检测胎儿遗传异常需要使用标准曲线,并且需要一定量的或更多的样本。
因此,为了解决现有技术中出现的上述问题而作出本发明,本发明目的在于一种能够提高胎儿遗传信息分析的准确性的方法,所述方法在使用孕妇血液进行产前诊断时通过浓缩胎儿DNA进行。本发明的产前诊断方法对于所有孕妇和胎儿是安全、方便、准确和可靠的,因此预期在产前诊断领域是非常有用的。
技术实现要素:
本发明是为了解决现有技术中存在的上述问题而做出的,涉及使用数字PCR进行产前诊断的方法。
然而,本发明实现的技术目标不限于上述技术目标,本领域技术人员可从以下描述中充分理解上述未提到的其它目标。
在下文中,将参考附图描述本文所述的各种实施例。在以下的描述中,阐述了许多具体细节,例如具体的配置、组成和过程等,以透彻理解本发明。然而,可以在没有一个或多个这些具体细节的情况下,或者与其他已知的方法和配置相组合来实施某些实施例。在其它实例中,没有特别详细地描述已知的工艺和制备方法,以免不必要地混淆本发明。说明书全文中提及的“一个实施例”或“某个实施例”意味着描述的与该特征相关的特定的特征、配置、组成或特性包括在本发明的至少一个实施例中。因此,说明书全文中各处出现的短语“一个实施例”或“某个实施例”不必指本发明的同一个实施例。另外,在一个或多个实施例中,特定的特征、配置、组成或特性可以以任何合适的方式组合。
除非在说明书中另有规定,本说明书中使用的所有科学和技术术语具有与本发明所属技术领域的技术人员通常理解的相同的含义。
在本发明的一个实施例中,“产前诊断”是指在出生前诊断先天性异常,也称为出生前诊断或宫内诊断。使用胎儿的或来自胎儿的蜕膜材料作为样本,通过使用细胞遗传学、生物化学分析和成像的方法进行测试。其目的在于早发现与发育和生育保护相关的先天性异常。技术包括侵入性方法(羊膜穿刺术、绒毛取样术、胎儿脐血取样术、胎儿皮肤活检和胎儿肝脏活检)和非侵入性方法(超声波测试,来自孕妇的胎儿细胞取样等)。
当父母中的任意一人或多人表现出染色体异常或严重的遗传疾病时,或者当孕妇有可能生出畸形儿,或者当孕妇为高龄产妇,或者超声波检查出孕妇具有胎儿毒性(fetal poisoning),或者怀疑其具有染色体异常、单一遗传疾病或遗传性代谢紊乱等,需要进行遗传产前诊断。在核型分析中,从安全性角度讲,应当进行羊膜穿刺术,并且,一般来说,在怀孕第15周和18周之间血液采样。也可进行绒毛取样术用于DNA诊断,其中在怀孕第9周和11周之间通过阴道或腹部绒毛采样。在怀孕16周后通过腹部从脐带处采样胎血。为进行细胞遗传学诊断,通过荧光原位杂交(FISH)进行核型测试以培养胎儿细胞,通过Southern印迹杂交法或聚合酶链反应(PCR)进行直接诊断用于分子基因诊断,或者通过限制性片段长度多态性(RFLP)或PCR-RFLP技术等进行间接诊断。
在本发明的一个实施例中,术语“染色体异常”也称为“染色体异倍体”,并且意味着细胞中、个体中或种群中每个细胞的染色体数目不是基本数目的整倍数,而是比整倍数增加或减少一个或几个数字,也就是说,包括具有不完全结构的基因组的状态。处于这种状态的细胞或个体称为非整倍体(或异倍体)。通常,染色体数目大于基本数目的整数倍的情况被称为超倍体,而染色体数目小于基本数目的整数倍的情况被称为亚倍体。特别地,对于二倍体,缺失一对两条同源染色体的情况被称为缺体性(nullisomy),将缺失一条同源染色体而另一条同源染色体继续存在的情况称为单体性(monosomy),并且将除了一对同源染色体外还存在另外的染色体的情况称为三体性。
通常的性染色体三体性为XXY,是主要由睾丸功能障碍引起的克氏综合征(Klinefelter’s syndrome)。此外,还存在X染色体数目为1(XO)并且卵巢未发育的特纳综合征(Turner’s syndrome),X染色体数为3的XXX女性,以及具有XYY的YY综合征(YY男性)。典型的三体性障碍包括但不限于,21号染色体是三体性的唐氏综合征,18号染色体是三体性的并且在胎儿期引起发育障碍的爱德华氏综合征,以及13号染色体是三体性的并且包括严重畸形的帕陶综合征。猫叫综合征是指具有特征性哭泣声音的症状的病症,并且是由5号染色体短臂的部分缺失引起的。
在本发明的一个实施例中,“实时聚合酶链式反应(实时PCR)”也称为定量实时聚合酶链反应(qPCR),是一种扩增靶DNA分子并且同时测量靶DNA分子的量的分子生物学实验技术。通常使用的RT-PCR是指逆转录聚合酶链反应,而不是实时PCR。然而,本领域技术人员不能清楚地区分它们。实时PCR可以检测某一DNA样本中具有特定序列的一个或多个基因的绝对拷贝数目或相对量。
根据通常的PCR进行实验。一个重要的特征是实时测量扩增的DNA。这与在最终阶段观察DNA的基础PCR不同。在实时PCR中使用两种通用方法:(1)可进入任何DNA双螺旋的非特异性荧光染色;和(2)由寡核苷酸组成的序列特异的DNA探针。用荧光标记报告(基因),并且在结合至互补DNA目标后进行检测。通常,实时PCR与逆转录PCR结合进行,以测量未由细胞或组织的mRNA编码的非编码RNA的量。
在本发明的一个实施例中,“数字聚合酶链式反应(数字PCR)”是用于检测和定量核酸的新方法,并且,与常规的qPCR相比,其能够精确地定量分析和高灵敏度地检测靶核酸分子。分析常规qPCR的结果的方法是模拟方法,其中,所述数字PCR方法,其结果是通过数字方法(因为得到的信号具有“0”或“1”的数值)分析的,具有可分析大体积样本、可同时检测不同样本以及同时进行不同测试的优点。数字PCR技术是一种可使用不具有标准曲线的单分子计数法来绝对定量DNA样本的技术,并且可以通过PCR对每孔单个液滴进行更精确的绝对定量(参见Gudrun Pohl和le-Ming Shih,数字PCR的原理和应用(Principle and applications of digital PCR),Expert Rev.Mol.Diagn.4(1),41-47(2004))。
在数字PCR中,含有经制备从而可用于稀释至平均拷贝数目为0.5-1的样本基因模板、扩增引物和荧光探针的各液滴分配到单个孔中,并进行微乳液PCR。然后,显示荧光信号的孔计数为数值“1”,因为具有基因拷贝数目为1的样本分配到所述孔中并且扩增后显示荧光信号,并且,没有显示信号的孔计数为“0”,因为具有基因拷贝数目为0的样本分配到所述孔中,由于无扩增,不显示荧光信号。采用这种方式,可以实现绝对定量。
在数字PCR中,只有每个孔的基因拷贝数目显示荧光信号为1时,数据的可靠性才能保证。在数字PCR的定量中,如果每孔的信号出现或不出现泊松分布,可认为是数字值1或0,并且可计算正值(1)与负值(0)的比率。如果正值与正值和负值之和的比率大大偏离于泊松分布中的1的基因拷贝数目,可能意味着每孔的基因拷贝数目大于1。因此,在这种情况下,数据的可靠性不能得到保证。由于这个缘故,在数字PCR中,扩增后定量的数值不满足泊松分布。因此,如果每孔的基因拷贝数目远大于1,稀释基因样本以满足泊松分布是重要的。如果每孔的基因拷贝数目接近1,校正该数值则是重要的(例如,可以使用Poisson9程序进行校正)。
然而,在数字PCR方法中,尽管能简单进行定量分析、高通量分析并且能处理大量样本,但迄今为止仍需要校正定量分析的技术以保证数字PCR的数据可靠性,并且,将每孔的基因拷贝数目调整至1存在技术困难。特别地,如上所述,当对来自孕妇的血液或血浆的胎儿基因数量的异常进行基因分析,并且使用靶基因的特异性引物对和探针对染色体数目异常进行数字PCR时,存在的问题是,由于存在大量孕妇基因的背景信号,与存在于孕妇血液或血浆中的少量胎儿靶向基因相关的基因数目的异常无法检测或者检测为不清晰信号。该问题成为难以将胎儿无细胞基因的基因型分析技术与数字PCR技术相结合的障碍。
因此,当通过使用存在于孕妇血液或血浆中的极少量胎儿基因,基于数字PCR技术分析胎儿染色体数目异常时,需要提供一种技术,其通过满足数字PCR技术所需的泊松分布而显示出可靠的定量结果,同时克服了难以分离仅来自胎儿基因的信号的问题,以及由孕妇基因引起的背景信号的问题。
在本发明的一个实施例中,“对照基因”指的是用作测试参考以确定靶基因是否异常的正常基因。例如,如本文所用,所述“对照基因”是孕妇染色体组或来自于孕妇血液或血浆的胎儿染色体组中已知的与染色体异常无关的染色体上的基因,并且可以是1号染色体或2号染色体,但不限于此。
在本发明的一个实施例中,待分析样本的基因中,“靶基因”是具有利于基因型分析的突变的基因。靶基因的例子包括用于遗传分型或个体识别的标记基因、用于诊断特定疾病的标记基因、显示出染色体数值异常或染色体数目异常的基因、具有遗传学显着突变的基因、具有STR(短串联重复)的基因、以及具有单核苷酸多态性的基因。例如,本文所用的靶基因是位于已知与染色体异常有关的染色体上的基因,并且可以是21号染色体(唐氏综合征),18号染色体(爱德华氏综合征)或13号染色体(帕陶综合征),但不限于此。
在本发明的一个实施例中,“诊断”是指确认病理的存在或特征。为了本发明的目的,诊断是指分析来自于孕妇血液的胎儿遗传信息以确定染色体数目是否异常。
在一个实施例中,本发明提供了为诊断胎儿中染色体非整倍性提供信息的方法,所述方法包括步骤:(a)从孕妇血液中提取DNA;(b)根据大小分级DNA,以获得大小为1,000bp或更小的DNA;(c)使用步骤(b)获得的DNA为染色体上与染色体非整倍性无关的对照基因和染色体上与染色体非整倍性相关的靶基因进行数字PCR;(d)计算靶基因的定量数字PCR值与对照基因的定量数字PCR值的比率;以及,(e)当步骤(d)中计算的比例为0.70-1.14时,确定胎儿的染色体数目是正常的。本发明的为诊断胎儿染色体非整倍性提供信息的方法包括步骤:当步骤(d)中计算的比率为0.95-1.10时,确定胎儿的染色体数目是正常的。在本发明的用于提供诊断胎儿染色体非整倍性的信息的方法中,根据大小对DNA进行分级的步骤(b)包括步骤:(a)将第一磁珠(珠A)添加到DNA中,并且分离出未结合至珠A的DNA;和(b)将第二磁珠(珠B)添加到分离的DNA中,并且洗脱出结合至珠B的DNA,其中所述珠A和珠B以0.75-1.25:1的比例(珠A或珠B:DNA)添加至DNA中。在本发明的为诊断胎儿染色体非整倍性提供信息的方法中,珠A或珠B以0.75-1.0:1的比例(珠A或珠B:DNA)加入DNA。
在另一优选例中,本发明提供为诊断胎儿中染色体非整倍性提供信息的方法,所述方法包括步骤:(a)从孕妇血液中提取DNA;(b)根据大小分级DNA,以获得大小为1,000bp或更小的DNA;(c)使用步骤(b)的DNA为染色体上与染色体非整倍性无关的对照基因和染色体上与染色体非整倍性相关的靶基因进行数字PCR;(d)计算靶基因的定量数字PCR值与对照基因的定量数字PCR值的比率;以及,(e)当步骤(d)中计算的比例为0.10-0.69或1.15-1.80时,确定胎儿的染色体数目是异常的。本发明的为诊断胎儿染色体非整倍性提供信息的方法包括步骤:当步骤(d)中计算的比率为0.45-0.69或1.15-1.31时,确定胎儿的染色体数目是异常的。本发明的为诊断胎儿染色体非整倍性提供信息的方法包括步骤:当步骤(d)中计算的比例为0.10-0.69时,确定胎儿的染色体数目是单体性。在本发明的方法中,所述单体性是特纳综合征。本发明的的方法包括步骤:当步骤(d)中计算的比例为1.15-1.80时,确定胎儿的染色体数目是三体性。在本发明的方法中,所述三体性是唐氏综合征、爱德华氏综合征或帕陶综合征。在本发明的方法中,根据大小对DNA进行分级的步骤(b)包括步骤:(a)将第一磁珠(珠A)添加到DNA中,并且分离出未结合至珠A的DNA;和(b)将第二磁珠(珠B)添加到分离的DNA中,并且洗脱出结合至珠B的DNA,其中所述珠A和珠B以0.75-1.25:1的比例(珠A或珠B:DNA)添加至DNA中。本发明的用于提供诊断胎儿染色体非整倍性的信息的方法中,珠A或珠B以0.75-1.0:1的比例(珠A或珠B:DNA)加入DNA。
此外,在上述为诊断胎儿中的染色体非整倍性提供信息的方法中,所述靶基因可以是选自下组的任何一种或多种基因:具有SEQ ID NO:1至4的核苷酸序列的基因。在本发明的方法中,SEQ ID NO:7和8的一对引物作为引物对用于扩增具有SEQ ID NO:1的核苷酸序列的靶基因,并且,SEQ ID NO:9的寡核苷酸作为探针用于检测具有SEQ ID NO:1的核苷酸序列的靶基因的扩增产物。而且,SEQ ID NO:10和11的一对引物作为引物对用于扩增具有SEQ ID NO:2的核苷酸序列的靶基因,并且,SEQ ID NO:12的寡核苷酸作为探针用于检测具有SEQ ID NO:2的核苷酸序列的靶基因的扩增产物。此外,SEQ ID NO:13和14的一对引物作为引物对用于扩增具有SEQ ID NO:3的核苷酸序列的靶基因,并且,SEQ ID NO:15的寡核苷酸作为探针用于检测具有SEQ ID NO:3的核苷酸序列的靶基因的扩增产物。除此之外,SEQ ID NO:16和17的一对引物作为引物对用于扩增具有SEQ ID NO:4的核苷酸序列的靶基因,并且,SEQ ID NO:18的寡核苷酸作为探针用于检测具有SEQ ID NO:4的核苷酸序列的靶基因的扩增产物。
此外,在上述为诊断胎儿中的染色体非整倍性提供信息的方法中,所述对照基因可以是选自下组的任何一种或多种基因:具有SEQ ID NO:5至6的核苷酸序列的基因。在本发明的方法中,SEQ ID NO:19和20的一对引物作为引物对用于扩增具有SEQ ID NO:5的核苷酸序列的对照基因,并且,SEQ ID NO:21的寡核苷酸作为探针用于检测具有SEQ ID NO:5的核苷酸序列的对照基因的扩增产物。而且,SEQ ID NO:22和23的一对引物作为引物对用于扩增具有SEQ ID NO:6的核苷酸序列的对照基因,并且,SEQ ID NO:24的寡核苷酸作为探针用于检测具有SEQ ID NO:6的核苷酸序列的对照基因的扩增产物。
在另一优选例中,本发明提供一种为诊断胎儿染色体非整倍性提供信息的试剂盒,所述试剂盒包括:
(a)从孕妇血液中提取DNA的器件;
(b)根据大小分级DNA,以获得具有1,000bp或更小的DNA的器件;
(c)使用步骤(b)获得的DNA为染色体上与染色体异常无关的对照基因和染色体上与染色体非整倍性相关的靶基因进行数字PCR的器件;
(d)计算靶基因的定量数字PCR值与对照基因的定量数字PCR值的比率的器件;和
(e)当步骤(d)中计算的比例为0.70-1.14时,显示胎儿的染色体数目是正常的器件。
在另一优选例中,本发明所述方法和试剂盒用于非诊断目的。
在下文中,本发明所述方法的每一步骤会详细地描述。
附图说明
图1显示了21号、1号、2号染色体上各自的靶基因或对照基因的基因座。
图2显示了在正常或唐氏综合征细胞上进行数字PCR,并且将靶基因的定量数字PCR值相对于对照基因的定量数字PCR值标准化得到的结果。
图3显示了对怀有正常胎儿或唐氏综合征胎儿的妇女的羊水样本进行数字PCR,并且将靶基因的定量数字PCR值相对于对照基因的定量数字PCR值标准化所得的结果。
图4显示了根据片段化颗粒大小对DNA样本分级所得的结果,所述DNA样本包含1:1的长DNA(LD)和短DNA(SD)的混合物,并且根据加入DNA样本的第一磁珠(珠A)的量分析LD是如何除去的。
图5显示了使用APP基因,通过定量来自孕妇血液样本的胎儿DNA和孕妇DNA的比率所得结果。
图6显示了在gDNA样本上进行数字PCR所得结果,所述样本含有以不同比例混合的正常gDNA和唐氏综合征gDNA,并且计算了唐氏综合征基因与正常基因的比率。
图7显示了从怀有正常或唐氏综合征胎儿(T21)的孕妇血浆中提取的cfDNA的所得结果,根据片段化颗粒大小对提取的DNA进行分级,在DNA上进行数字PCR,并且将靶基因的定量数字PCR值相对于对照基因的定量数字PCR值作标准化。
具体实施方式
在下文中,将进一步详细地描述本发明。本领域技术人员显而易见地知道这些实施例仅仅作为示例性目的,并不意图限制本发明的范围。
实施例1:对照基因和靶基因的选取,以及引物对和探针的设计
为了进行胎儿基因的遗传分析,例如,诸如三体性等基因异常的分析,选取21号染色体(由于三体性引起唐氏综合征)作为靶染色体,选取1号染色体和2号染色体作为对照染色体。然后,为了基因检测,选取21号染色体上的四个区域的基因作为靶基因,选取1号染色体上的一个区域的基因和2号染色体上一个区域的基因作为对照基因。根据这些基因,设计用于扩增各基因的引物对和能够确认扩增反应产物的荧光标记探针。这些引物和探针分别由Bioneer Co.,Ltd.(大田,韩国)和Thermo Fisher Scientific(美国)合成。21号染色体、1号染色体和2号染色体上所选取的区域如图1所示,上述引物对和探针的序列和设计如下表1和表2所示。
表1
表2
实施例2:对于正常或唐氏综合征细胞的数字PCR
正常的或唐氏综合征(T21)的gDNA购自Coriell,以获得正常或唐氏综合征(T21)gDNA的数字PCR结果数值。
使用gDNA作为模板,使用QX200数字PCR系统(Bio-Rad,美国)进行数字PCR。为了建立用于数字PCR的样本的合适浓度,在实验中将gDNA样本逐级稀释1-5000倍。制备20μl的含有0.5-1.0μM引物和0.1-0.25μM探针的反应溶液作为数字PCR的预混液(master mix),调节至2-10ng的样品注射浓度(参见下表3),并分配到各PCR管中。将含有该溶液的PCR管安装在DG8微滴发生器筒中,然后将20μl样品分配到样品孔中,并将70μl微滴发生器油(垫圈(Gasket))分配到油孔中。接下来,将垫圈安装在筒中,然后使用QX200微滴发生器进行微滴产生反应。将所产生的微滴分配到96孔PCR板上,然后在下表4所示的温度条件下进行PCR。
表3
表4
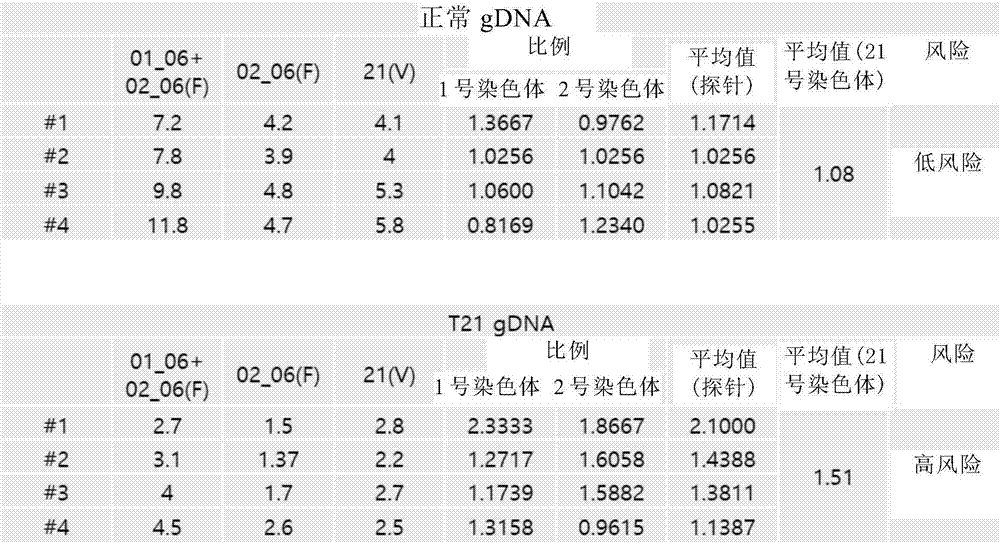
反应完成后,将每个孔安装在QX200微滴分析仪上,并且分析各探针的每μl拷贝数。使用各探针的每μl拷贝数,计算相对于各对照基因(SEQ ID NO:5或6)的定量数字PCR值作标准化的靶基因(SEQ ID NO:1至4)的定量数字PCR值。所得结果示于图2。
实验结果表明,正常体细胞样本(n=4)的计算比例值分别为1.1714、1.0256、1.0821和1.0255,其平均值为1.08,并且唐氏综合征(T21)体细胞样本(n=4)的计算比例值分别为2.1000、1.4388、1.3611和1.1387,其平均值为1.51。因此,可以看出,唐氏综合征样本的计算值约为正常样本的1.5倍。
实施例3:怀有正常或唐氏综合征胎儿的孕妇的羊水样本的数字PCR
从怀有正常或唐氏综合征(T21)胎儿的孕妇的羊水样品中提取胎儿cfDNA。使用胎儿cfDNA作为模板样本,进行数字PCR。
首先,使用QIAamp循环核酸试剂盒(Qiagen目录号55114,凯杰公司,德国),从细胞中提取gDNA。根据制造商的手册进行gDNA提取。具体地,将细胞用蛋白裂解缓冲液裂解,并将100μl蛋白酶K加入到50ml离心管中,以除去蛋白质组分,从而提高样品纯度。然后,向含有蛋白酶K,含有1.0μg载体RNA的ACL缓冲液的50ml离心管中,随后涡旋混合细胞裂解物与ACL缓冲液。将混合物在60℃下培养30分钟,然后将3.6ml ACB缓冲液(裂解物缓冲液)加入离心管,随后涡旋以获得ACB缓冲液混合物,然后将其在冰上温育5分钟。
为提高提取的gDNA的产量,使用真空泵系统以提高QIAamp循环核酸试剂盒的柱上存在的膜的吸附能力。使用真空泵系统,使得ACB缓冲液混合物流入配备有20ml管延伸器的QIAamp Mini柱。在操作真空泵系统以允许ACB缓冲液混合物流动之后,将真空泵系统的值调节至0,并且移除管延伸器。将最大可能量的反应材料通过该过程结合到QIAamp Mini柱的膜上。然后,通过操作真空泵系统,将600μl的ACW1缓冲液流入QIAamp Mini柱,所述ACW1缓冲液是用于洗涤结合至QIAamp Mini柱上的反应材料的第一缓冲液,然后,将真空泵系统的值调整至0,移除管延伸器。结合至QIAamp Mini柱的DNA通过该过程被洗涤。除此之外,进行第二次洗涤操作以得到高纯度的DNA。通过操作真空泵系统,将750μl作为第二缓冲液的ACW2缓冲液流入QIAamp Mini柱,然后将真空泵系统的值调整至0,移除管延伸器。结合至QIAamp Mini柱的DNA通过该过程进一步被洗涤以得到高纯度的DNA。
通过操作真空泵系统,使作为最终洗涤溶液的750μl的乙醇(96%至100%)流至QIAamp Mini柱,然后将真空泵系统的值调整至0,移除管延伸器。此外,通过干燥在最大可能范围内除去乙醇组分,从而提高提取的DNA的纯度,然后关闭QIAamp Mini柱,然后在20,000g和14,000rpm下离心3分钟。打开QIAamp Mini柱,随后在56℃下干燥10分钟。此后,向QIAamp Mini柱中加入20μl的AVE缓冲液,随后在室温下培育3分钟,随后关闭QIAamp Mini柱,之后在20,000g和14,000rpm下离心1分钟。因此,得到了来自于正常体细胞或唐氏综合征(T21)体细胞的各个相应的cfDNA样本。
在实施例2描述的相同条件下,采用相同方法,进行各样本的数字采样。结果示于图3。
实验结果表明,怀有正常胎儿的孕妇羊膜样本(n=4)的计算比例值分别为1.1991、1.0574、1.0006和0.9560,因此其平均值为1.05,并且,怀有唐氏综合征(T21)胎儿的孕妇羊膜样本(n=4)的计算比例值分别为1.6584、1.6954、1.4886和1.5062,因此其平均值为1.59。因此,可以看出,唐氏综合征样品的计算比率约为正常样品的1.5倍,并且该结果值与使用正常或唐氏综合征细胞作为样品进行的实验的结果值相似。
实施例4:以不同比例的SPRIselsect磁珠根据片段化颗粒大小对DNA进行分级的验证
因为胎儿DNA在大小上小于孕妇DNA,选择根据DNA长度提取cfDNA的方法以提高显示短长度(bp)的胎儿cfDNA的信息反映率(reflection rate)。为了构建具有不同大小的DNA,根据制造商手册,使用Ion ShearTM Plus Reagents Kit(目录号4471252,Thermo Fisher Scientific),gDNA在两个条件下片段化(大长度和短长度;5和15分钟)。根据制造商手册,使用SPRIselsect试剂盒(Beckman Coulter,德国)根据片段化颗粒大小进行gDNA分级。现在将详细描述。
首先,将含gDNA的样本和磁珠(称为“珠A”)彼此混合,使得长DNA(LD)可首先吸附到磁珠上。随后,使用磁铁分离珠子,然后将保留在上清液中而未被吸附到珠子上的DNA转移到新管中,并向管中加入新鲜珠子(称为“珠B”),以提取剩余的DNA(短DNA(SD))。在各管中从珠A和珠B中提取吸附的DNA。
对于含有根据上述方法以1:1比例混合的LD和SD的DNA样本,根据加入DNA样本中的珠A的量分析LD是怎样除去的。分析结果示于图4。
实验结果表明,当加入相当于DNA样品的0.5倍的量的珠A时,珠A和珠B都不影响DNA大小的鉴别。然而,可以看出,当加入相当于DNA样品的0.75倍的量的珠A时,珠A吸附具有400bp或更大长度的DNA,珠B吸附具有500bp或更短长度的DNA,当加入相当于DNA样品的1.0倍的量的珠A时,珠A吸附具有200bp或更大长度的DNA,珠B吸附具有300bp或更短长度的DNA,当加入相当于DNA样品的1.25倍的量的珠A时,珠A吸附具有150bp或更大长度的DNA,珠B吸附具有200bp长度的DNA。这表明,随着珠A的添加量的增加,除去的LD的量也增加。考虑到胎儿cfDNA的长度约为150bp或更小,可以确定,加入到DNA样本中用于浓缩胎儿cfDNA的珠A的比率优选0.75-1.25,更优选地为DNA样本的0.75-1.0倍。
实施例5:定量测定来自孕妇血液样本的胎儿DNA和孕妇DNA的比例
众所周知,孕妇的血液或血浆中的胎儿DNA的尺寸小于孕妇的DNA。因此,当具有相对较小尺寸的胎儿DNA和具有较大尺寸的孕妇DNA通过与特定基因有关的实时PCR扩增时,可以定量存在于孕妇的血液或血浆中的与特定基因相关的胎儿DNA和孕妇的DNA,并且可以计算这些基因的数量的比例。
因此,在本实施例中,APP(淀粉样前体蛋白)基因(GenBank登录号NM_000484)和β-肌动蛋白基因是在孕妇血液或血浆中的胎儿DNA和孕妇DNA中通常存在的特异性基因。另外,设计了能够扩增与APP基因和β-肌动蛋白基因相关的胎儿DNA和孕妇DNA的引物对,以及能够确认扩增反应产物的荧光标记探针(参见下表5)。
表5
供参考,在上表5中,为了通过实时PCR定量地比较胎儿DNA与孕妇DNA以评价这些基因数量的比例,鉴于以下事实设计了引物对:胎儿DNA的大小小于孕妇DNA的大小。特别地,如上表5所示,为了准确地评价孕妇基因的数量和胎儿基因的数量,并且在定量评估中标准化分析条件,将用于胎儿DNA和孕妇DNA的一组短扩增子,以及孕妇DNA的一组长扩增子的检测探针和正向引物设计成一样的,,而将反向引物设计成不同的。另外,为了确认实时PCR扩增产物并定量分析PCR扩增产物,用荧光物质和淬灭物质双重标记每个检测探针。在本实施例中,用荧光物质FAM标记每个检测探针的5’端,并用猝灭物质TAMRA标记3’端。
如上表5所示,在APP靶基因的情况下,从SEQ ID NO:25和26所示引物对获得来自胎儿DNA和孕妇DNA的短扩增子(67bp长度),以及从SEQ ID NO:25和27所示引物对获得来自孕妇DNA的长扩增子(180bp长度)。
使用本实施例中采用的短长度DNA浓缩方法,从怀有正常胎儿的孕妇体内获得cfDNA,并进行实时PCR以测量SD与LD的比率。在本实施例中,使用7900HT快速实时PCR系统(Life Technologies,美国)进行实时PCR。
如下表6所示制备包含APP基因的各引物对和检测探针、如上所述制备的DNA模板以及实时PCR预混液的实时PCR组合物,并且,在以下表7所示的实时PCR条件下进行实时PCR。同时,PCR扩增组合物的PCR预混液由,例如,PCR缓冲液、dNTP、DNA聚合酶等组成,并且,在该实施例中,使用TaqMan通用预混液II,无UNG(Life Technologies,目录号4440040)作为实时PCR的预混液。每当重复一次实时PCR扩增反应时,在FAM波长下测量荧光,并使用SDS软件分析每个反应循环的荧光强度。对于每个样品,总结了APP基因的实时PCR定量结果,并且将短扩增子(67bp长度)的量除以长扩增子(180bp长度)的量,并示于图5。
表6
表7
实施例6:对包含正常或唐氏综合征gDNA混合物的样本进行数字PCR
将正常gDNA和唐氏综合征gDNA以不同比例彼此混合以制备gDNA混合物。使用各gDNA混合物作为模板,使用如上表2所示的引物和探针进行数字PCR,计算唐氏综合征基因数量与正常基因数量的比率。计算结果示于图6中。
实验结果表明,随着添加的唐氏综合征gDNA的百分比的下降,唐氏综合征基因数量与正常基因数量之比接近1。特别地,结果表明,当关于唐氏综合征基因的信息反映率低至低于10%时,该比率接近正常值(1.1或更低)。考虑到孕妇血液中只有约3-13%的cfDNA通常是胎儿cfDNA,可以看出,从孕妇血液中提取的cfDNA本身用于进行产前诊断,诊断的准确性非常低。
实施例7:根据片段化颗粒大小和对cfDNA进行的数字PCR,对提取自孕妇血液的cfDNA进行分级
以下列方式,从怀有正常或唐氏综合征(T21)的胎儿的孕妇血浆中提取cfDNA。
首先,使用MagMAXTM无细胞DNA提取试剂盒(MagMAXTM Cell-Free DNA Isolation Kit(目录号A29319,Thermo Fisher Scientific)),从血浆中提取cfDNA。根据制造商的手册使用2mL血浆进行cfDNA提取。
通过实施例4的方法根据片段化颗粒大小对提取的cfDNA进行分级。使用含有所得SD区域的各样本作为模板,进行数字PCR。从各样本的结果值,计算靶基因与对照基因的比例。计算结果示于图7中。
实验结果表明,正常样本(n=3)的计算比例分别为1.04、1.08和0.98,但是唐氏综合征样本(n=3)的计算比例分别为1.31和1.15(对于两个样本)。在使用根据片段化颗粒大小由DNA获得的SD区域进行数字PCR的情况下,可以确定的是,当靶基因与对照基因的计算比例为0.70-1.14时,胎儿的染色体数目是正常的,更优选地,当计算比例为0.95-1.10时,胎儿的染色体数目是正常的。除此之外,可以确定的是,当计算比例为0.10-0.69或1.15-1.80时,胎儿的染色体数目是异常的,更优选地,当计算比例为0.45-0.69或1.15-1.31时,胎儿的染色体数目是异常的。
如上所述,采用本发明的数字PCR进行产前诊断的方法,使用非侵入性的取样方法,因此对孕妇和胎儿都是安全的。另外,本发明的方法使得能够稳定分离少量胎儿基因的信号与孕妇基因的背景信号,因此提高了胎儿基因信息的准确性。因此,可以预期本发明的方法对于胎儿遗传疾病的早期诊断是非常有用的。
尽管本发明结合具体特征进行了详细描述,本领域技术人员显然知晓,这种描述仅仅是其一种优选实施方式,并不限制本发明的范围。因此,本发明的实质范围将由附加的权利要求及其等同范围所限制。
序列表
<110> 博康有限公司
<120> 使用数字PCR的产前诊断方法
<130> P2016-1693
<150> KR 10-2015-0151313
<151> 2015-10-29
<150> KR 10-2016-0133529
<151> 2016-10-14
<160> 36
<170> PatentIn version 3.5
<210> 1
<211> 165
<212> DNA
<213> 人工序列
<220>
<223> 靶基因
<400> 1
ctgcagttct ttgtgaacac tctcatttgt tgtatctgta ggctgtctct ctcagtggta 60
aatgccttcg tgtgtttgta atgctgatgg ttacttgagg taaataagaa tgtaccactt 120
ggctcagtgt gcatgatgta agcttgtctt tgttgtatgt tggct 165
<210> 2
<211> 161
<212> DNA
<213> 人工序列
<220>
<223> 靶基因
<400> 2
tgactatcac atgtctttgg ttgtaaactg ctgtgatagt taccctaagt aatgggacag 60
gagatgaacc cacccattaa ataacacagc aattaagcag ccacttttag aaaaatttaa 120
atgtgtggct tcgagttggg tacttgcatg tacagcttac t 161
<210> 3
<211> 118
<212> DNA
<213> 人工序列
<220>
<223> 靶基因
<400> 3
cccaggctat gctagaaggt atgcttacaa ttggaaaagt gtaggcagat aacattaaat 60
ggcaatagca tgtgtaaact aactgcaaat gaggaaaagg acattctaaa gacaggat 118
<210> 4
<211> 110
<212> DNA
<213> 人工序列
<220>
<223> 靶基因
<400> 4
ggaccagagg tttattggag gtctaaatat ttatggagag caatgatggc taattttaga 60
aaccattagg ttgctatttt taaacgtgtg ctataaggat ttgctaattt 110
<210> 5
<211> 127
<212> DNA
<213> 人工序列
<220>
<223> 对照基因
<400> 5
gaggaagcaa ctagaaaaca atggaaggga cttcagatgg taaggtttct gtttagtact 60
tatttcaatt ttaggcctcc tgaatagtag aggtggtgac aggaggatac ctgaaacctt 120
ggttata 127
<210> 6
<211> 112
<212> DNA
<213> 人工序列
<220>
<223> 对照基因
<400> 6
gggaacatcc caggttcagt aaaaatacag agtatttgcg ttaaactgga cctcagtggg 60
gatgtgatgg gaggtatgag acagattgtg cccttatcct tttctcttct tg 112
<210> 7
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 7
cgtgtgtttg taatgctgat ggt 23
<210> 8
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 8
catcatgcac actgagccaa gt 22
<210> 9
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 9
acttgaggta aataagaatg tac 23
<210> 10
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 10
ccctaagtaa tgggacagga gatg 24
<210> 11
<211> 23
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 11
aagtggctgc ttaattgctg tgt 23
<210> 12
<211> 15
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 12
acccacccat taaat 15
<210> 13
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 13
tgctagaagg tatgcttaca attgga 26
<210> 14
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 14
tcatttgcag ttagtttaca catgct 26
<210> 15
<211> 17
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 15
aagtgtaggc agataac 17
<210> 16
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 16
gaccagaggt ttattggagg tctaaat 27
<210> 17
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 17
cacgtttaaa aatagcaacc taatgg 26
<210> 18
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 18
tttatggaga gcaatgat 18
<210> 19
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 19
caactagaaa acaatggaag ggactt 26
<210> 20
<211> 25
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 20
tcaggaggcc taaaattgaa ataag 25
<210> 21
<211> 22
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 21
agatggtaag gtttctgttt ag 22
<210> 22
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 22
catcccaggt tcagtaaaaa tacaga 26
<210> 23
<211> 24
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 23
ctgtctcata cctcccatca catc 24
<210> 24
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 24
tatttgcgtt aaactggacc 20
<210> 25
<211> 18
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 25
tcaggttgac gccgctgt 18
<210> 26
<211> 19
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 26
ttcgtagccg ttctgctgc 19
<210> 27
<211> 26
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 27
tctataaatg gacaccgatg ggtagt 26
<210> 28
<211> 21
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 28
accccagagg agcgccacct g 21
<210> 29
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 29
tacaggaagt cccttgccat 20
<210> 30
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 30
cctgtgtgga cttgggagag 20
<210> 31
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 31
cacgaaggct catcattcaa 20
<210> 32
<211> 27
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 32
cccacttctc tctaaggaga atggccc 27
<210> 33
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 33
agagctacga gctgcctgac 20
<210> 34
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 34
ccatctcttg ctcgaagtcc 20
<210> 35
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 引物
<400> 35
ggcaggactt agcttccaca 20
<210> 36
<211> 20
<212> DNA
<213> 人工序列
<220>
<223> 探针
<400> 36
ttccgctgcc ctgaggcact 20
- 还没有人留言评论。精彩留言会获得点赞!