一种蚕豆SSR指纹图谱的构建方法与流程
本发明涉及分子生物学和植物品种鉴别
技术领域:
,特别是涉及一种蚕豆SSR指纹图谱的构建方法以及利用构建得到的不同品种蚕豆的指纹图谱鉴别蚕豆品种的方法及应用。
背景技术:
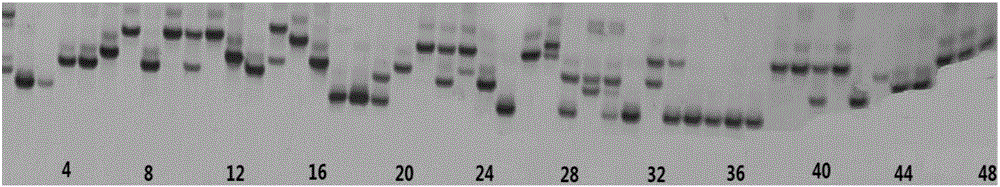
:蚕豆(ViciafabaL.)作为一种粮食、蔬菜和饲料、绿肥兼用作物,在我国农作物中占有重要地位。蚕豆在世界各洲均有生产和分布,全世界有超过43个国家生产蚕豆。蚕豆在我国已有2000余年的栽培历史,全国除山东、海南和东北三省极少种植外,其余各地均有种植。近10年,中国蚕豆的种植面积达140多万hm2,占世界总种植面积50%左右,是世界第一蚕豆生产大国。蚕豆可通过与根瘤菌共生结瘤,固定大气中的氮素,在改善土壤营养结构中起着重要的作用。蚕豆营养丰富,蛋白质平均含量为27.6%,维生素含量均超过大米和小麦。因此开展蚕豆品种的有效甄别显得尤为重要。目前我国对蚕豆品种进行鉴定主要方法是按照NY/T2345-2013《植物新品种特异性、一致性和稳定性测试指南蚕豆》进行田间测定来鉴定的。该方法简便、经济,但由于许多形态学性状鉴定周期长、易受环境影响,不能精确、快捷地为新品种侵权案件提供鉴定依据,同时也常出现不同测试人员对某些性状的认识存在偏差,导致判定结果不一致的问题。随着育种亲本的利用集中化,传统的形态学和细胞学鉴定方法测试性状多、工作量大,很难做到准确而真实的鉴定蚕豆的品种差别,为此需要在分子水平上进行分析鉴定。随着生物技术的发展,基于分子标记技术而建立起来的DNA指纹图谱(fingerprint)在品种、品系鉴别方面发挥着重要作用。品种DNA指纹数据库的构建在蚕豆品种纯度及真实性鉴定、种子质量标准化、品种审定及产权登记保护、真假种辨别、品种纠纷等方面具有重要应用价值,在探究我国蚕豆种质资源的亲缘关系、杂种优势群划分、种质资源创新利用及新品种选育等方面发挥着显著作用。当前用于构建作物DNA指纹图谱的标记技术主要有RFLP、RAPD、AFLP、EST、ISSR、SSR等。简单重复序列(Simplesequencerepeat,SSR),是第二代基于PCR技术的分子标记技术。与其它类型分子标记相比,SSR标记具有多态性丰富、遗传信息多、操作便利、共显性、高度可重复性等优点,已被广泛应用于植物资源遗传研究和分子辅助育种实践中。DNA指纹图谱能够从DNA水平上鉴定出品种间的差异或遗传相似性。微卫星(又称为简单重复序列,SSR)是由1-6个核苷酸组成的短序列串联重复多次而组成的一段DNA。由于微卫星中重复单元的重复次数不同,因而扩增出的微卫星序列的长度呈现多态性。微卫星序列具有多态性较高,重复性和稳定性好等特点,是十分有效的分子标记,被广泛应用于品种鉴定、指纹图谱构建等领域。为了有效保护我国蚕豆的品种知识产权,有必要在我国开展蚕豆开发微卫星标记开发,并应用于蚕豆优良品种“分子身份证”构建的研究。技术实现要素:本发明的目的在于提供一种蚕豆SSR指纹图谱的构建方法,本发明的另一目的在于提供一种高效、准确、低成本的利用基因组SSR指纹图谱鉴别蚕豆品种的方法。本发明的目的还在于提供该方法的应用。为实现上述目的,本发明以48份代表性蚕豆品种基因组DNA为模板,通过分析蚕豆基因组序列和EST序列,合成核心基因组SSR和EST-SSR引物。经过筛选,分别有15对基因组SSR和6对EST-SSR引物组成的分子标记组合可完全鉴别48份蚕豆品种的差异。所述的48份代表性蚕豆品种见表1。表148份蚕豆品种经过多次筛选,选择在连锁群上的均匀分布、多态性较高、PCR扩增较稳定,同时满足常规非变性凝胶电泳检测和毛细管荧光电泳检测两种技术平台,最终筛选出在蚕豆品种间多态性信息含量值最高、等位位点数最多、重复性好、扩增带型清晰度高、染色体分布均匀的SSR引物作为蚕豆品种指纹图谱库构建的核心引物。基于上述筛选方法,本发明提供用于鉴别蚕豆品种的分子标记组合,其特征在于,含有15个SSR分子标记和6个EST-SSR分子标记,分别为SSR11908、EST181、SSR11771、SSR12080、SSR13683、SSR12396、SSR14328、EST1672、EST1280、EST1741、SSR17991、SSR10911、SSR13106、SSR760、EST1142、SSR11052、SSR13771、SSR11885、SSR12536、SSR12868、EST1584;所述分子标记分别通过以下引物对扩增得到:SEQIDNO.1-2、SEQIDNO.3-4、SEQIDNO.5-6、SEQIDNO.7-8、SEQIDNO.9-10、SEQIDNO.11-12、SEQIDNO.13-14、SEQIDNO.15-16、SEQIDNO.17-18、SEQIDNO.19-20、SEQIDNO.21-22、SEQIDNO.23-24、SEQIDNO.25-26、SEQIDNO.27-28、SEQIDNO.29-30、SEQIDNO.31-32、SEQIDNO.33-34、SEQIDNO.35-36、SEQIDNO.37-38、SEQIDNO.39-40、SEQIDNO.41-42所示的引物。本发明提供一种引物组合,含有以下21对特异性引物对,其核苷酸序列分别如SEQIDNO.1-42所示。本发明提供含有上述引物组合的试剂盒。本发明提供了上述分子标记组合或引物组合在鉴定蚕豆品种中的应用。本发明提供了上述分子标记组合或引物组合在蚕豆种质资源改良中的应用。本发明提供了上述分子标记组合或引物组合在构建蚕豆SSR植物图谱中的应用。本发明还提供一种蚕豆品种SSR指纹图谱的构建方法,以本发明所述的含有上述21对引物的引物组合对不同蚕豆品种的DNA进行PCR扩增,对PCR产物进行非变性聚丙烯酰胺凝胶电泳进行检测或对PCR产物进行荧光毛细管电泳检测,纯合位点的等位变异大小数据记录为X/X,其中X为该位点等位变异的大小;杂合位点的等位变异数据记录为X/Y,其中X、Y为该位点上两个不同的等位变异;无效等位变异的大小记录为0/0;上述“/”前为小片段,“/”后为大片段;通过对不同位点的数据整合,形成不同蚕豆品种的SSR指纹图谱。其中,PCR扩增方法为:反应程序为:95℃预变性5min;95℃变性35s,60℃退火40s,72℃延伸45s,共32个循环;72℃延伸5min,10℃保温5min;10μL扩增体系为:2×TaqPCRMasterMix5μL,2μM/μL的上、下游引物各1μL,DNA模板1.5μL,ddH2O2.5μL。其中,当采用非变性聚丙烯酰胺凝胶进行电泳检测时,是采用8%的非变性聚丙烯酰胺凝胶进行电泳,电压300V,电流280mA,功率260W。当采用荧光毛细管电泳检测时,为引物加入荧光标记,详细情况见表2。本发明提供了利用上述构建方法构建得到的蚕豆品种指纹图谱。本发明构建得到的蚕豆品种指纹图谱,其如表5-表16所示。本发明进而提供一种鉴定蚕豆品种的方法,包括如下步骤:(1)提取待检蚕豆品种的DNA,以权利要求2所述的引物组合中的21对引物对分别对检蚕豆品种的DNA进行PCR扩增;(2)扩增产物通过非变性聚丙烯酰胺凝胶电泳进行检测,银染显色,根据扩增产物在电泳凝胶上的相对位置;或对扩增产物进行荧光毛细管电泳检测,根据扩增产物的相对位置,纯合位点的等位变异大小数据记录为X/X,其中X为该位点等位变异的大小;杂合位点的等位变异数据记录为X/Y,其中X、Y为该位点上两个不同的等位变异;无效等位变异的大小记录为0/0;上述“/”前为小片段,“/”后为大片段;(3)结果对照表5-表16的蚕豆品种指纹图谱,品种间差异位点数≥3,则为不同品种;品种间差异位点数<3,则为近似品种。本发明提供了上述方法在蚕豆种质资源改良中的应用。本发明蚕豆DNA指纹图谱的构建方法,利用了SSR引物组合对某一蚕豆品种DNA进行扩增,得到一组特有的DNA指纹,于是本发明方法采用引物组合鉴别的方法对蚕豆品种进行分析和筛选,使每一个品种都找到了它特有的DNA指纹。本发明的SSR和EST-SSR引物的扩增产物带型间大小差异明显,能够很好的区分不同的品种。对同一品种内的个体间的扩增产物,其带型一致。本发明的检测方法在短时间内即可完成蚕豆种子品种鉴定与遗传多样性评价工作,具有高效、准确、低成本、操作简便等优势。同时本发明的检测方法可以有效的对蚕豆种子真伪进行监控,从DNA水平揭示品种的遗传变异与亲缘关系,保护了作物品种,防止假冒伪劣品种进入市场,也为蚕豆品种选育过程中优异种质的合理利用提供了技术参考。附图说明图1为48份代表性蚕豆品种聚类分析结果图。图2为实施例2中48份代表性蚕豆品种的PCR扩增结果电泳图。具体实施方式以下实施例进一步说明本发明的内容,但不应理解为对本发明的限制。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本发明的范围。若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段;若未特别指明,实施例中所用试剂均为市售。本发明品种判定原则遵照以下国家标准进行,NY/T2594-2014植物品种鉴定DNA指纹方法总则;NY/T2345-2013《植物新品种特异性、一致性和稳定性测试指南蚕豆》;GB/T19557.1植物新品种特异性、一致性和稳定性测试指南总则。实施例1用于鉴定蚕豆品种的SSR和EST-EST引物的筛选本实施例以我国蚕豆主产区代表性推广的48个蚕豆(见表1)品种基因组DNA为模板,通过分析蚕豆基因组序列和EST序列,合成核心基因组SSR和EST-SSR引物。经过筛选,分别有15对基因组SSR和6对EST-SSR引物组成的分子标记组合可完全鉴别48份蚕豆品种的差异。最终筛选得到15对基因组SSR分子标记和6对EST-SSR分子标记。表221对SSR核心引物上述表1的引物分别对应序列表中的SEQIDNO.1-42。实施例2蚕豆品种特征指纹图谱的构建与蚕豆品种鉴定方法的建立1、蚕豆品种总DNA提取采用改良CTAB法提取供试品种DNA:取2g左右的新鲜幼嫩叶片,在液氮中研磨成粉末状装入2.0ml离心管中,放入-80℃冰箱保存待用;将预热到65℃的2×CTAB提取液,按占溶液容量的1%的比例加入β-巯基乙醇,充分混匀;取出研磨好的叶片组织,每份样品中加入800μL预热后的CTAB提取液,涡旋1-2min,65℃水浴1h(每15min上下颠倒轻翻一次,充分混匀);水浴加热结束后,加入800μL氯仿/异戊醇(24:1)溶液于离心管中,混合15-20min,之后10000rpm离心15min;吸取600μL左右上清液移至新离心管(量可适当减少,但切不可接触蛋白质层),后加入95%等体积乙醇,摇匀后于-20℃冰箱放置2h或者4℃冰箱过夜,保证充分沉淀;12000rpm离心10min,倒掉上清,保留沉淀;用90%乙醇500μL清洗白色沉淀,10000rpm离心5min,小心倒掉上清;室温风干,加入100μLddH2O充分溶解沉淀。利用Nanodrop2000/2000C仪器检测样品DNA浓度,将DNA浓度稀释到50ng/μL,4℃保存备用。2、以实施例1所述48份蚕豆品种为材料,利用实施例1筛选得到的用于扩增21个分子标记的引物分别对实施例1所述的48份代表性蚕豆品种的DNA进行PCR扩增,反应程序为:95℃预变性5min;95℃变性35s,60℃退火40s,72℃延伸45s,共32个循环;72℃延伸5min,10℃保温5min;10μL扩增体系为:2×TaqPCRMasterMix5μL,2μM/μL的上、下游引物各1μL,DNA模板1.5μL,ddH2O2.5μL。3、PCR扩增产物凝胶电泳及银染显色:扩增产物采用8%非变性聚丙烯酰胺凝胶电泳检测,电泳缓冲液为0.5×TBE,200V稳压电泳2-2.5h,至加样缓冲液移到凝胶底部时电泳结束。银染显色,照相记录。(1)玻璃板清洗用清水和洗涤剂将平、凹玻璃板充分清洗干净,然后用蒸馏水冲洗,置于玻璃板架晾干;为防止灌胶时有气泡产生,灌胶前用酒精擦洗玻璃板,晾干备用;将组装好的玻璃板水平放齐,用夹子固定。(2)灌胶在45ml8%PAGE胶中加入TEMED45μL和10%过硫酸铵450μLTEMED(TEMED和过硫酸铵的使用量应依据环境温度做出相应调整),迅速混匀后灌胶。待胶液充满玻璃板夹层时,在上部轻轻插入梳子(根据上样量自行调整梳子插入深度),使其聚合25min左右(具体聚合时间应随不同环境温度做出相应调整)。在灌胶过程中,应防止气泡的产生(若出现小气泡,可借助塑料薄片将其赶出)。(3)PCR扩增产物预处理在10μLPCR扩增产物中加入2.5μL6×Loadingbuffer,混匀30s待用。(4)电泳用移液器吹吸加样槽,清除杂质和气泡。每加样孔点入1.5μL产物进行检测,电泳1-1.5小时(电压300V,电流280mA,功率260W),电泳时间长短可根据目标片段大小和实际功率适度调节。待上部的指示带(二甲苯青)到达胶板底部,电泳结束,小心剥离聚丙烯酰胺凝胶,待染色。(5)银染漂洗:使用蒸馏水快速漂洗聚丙烯酰胺凝胶1次,时间不超过10s。银染:按以下银染液配比对凝胶进行染色,然后平行震荡染色10min。表3清洗:染色10min后,将染色液倒入废液桶,继续用蒸馏水漂洗2次,每次不超过15s。显色:按以下显色液配比,加入适当的显色液于塑料盒中,轻轻混匀后置于平行震荡仪,使凝胶与显色液充分作用,直至条带清晰。表4洗涤:将显影液倒掉并用蒸馏水清洗2-3次,每次不超过15s。保存:清洗干净后,将凝胶平铺在玻璃板上,用保鲜膜将其密封,读出条带结果并统计记录,然后照相留存。结果记录:纯合位点的等位变异大小数据记录为X/X,其中X为该位点等位变异的大小;杂合位点的等位变异数据记录为X/Y,其中X、Y为该位点上两个不同的等位变异(小片段在前,大片段在后);无效等位变异的大小记录为0/0。从图2可以看出引物的多态性好,能够很好的鉴别品种。构建的48份蚕豆的指纹图谱见表5-表16。4、与上述步骤3平行的检测方法,还可采用对PCR扩增产物进行荧光毛细管电泳检测,具体如下:PCR产物样品准备及电泳分析结果将FAM(蓝色)和HEX(绿色)荧光标记的PCR产物用超纯水稀释25倍,分别取等体积的上述2种稀释后溶液混合形成混合液,吸取1μL混合液、0.1μLLIZ500分子量内标和8.9μL去离子甲酰胺分别加入到DNA分析仪专用深孔板孔中;然后将其在PCR仪上95℃变性5min,取出后立即置于碎冰上,冷却15min左右;瞬时离心10s,进行毛细管电泳检测,用GENEMAPPER进行图像分析和数据采集。以DNAMarkerI为DNA分子量标准,纯合位点的等位变异大小数据记录为X/X,其中X为该位点等位变异的大小;杂合位点的等位变异数据记录为X/Y,其中X、Y为该位点上两个不同的等位变异;无效等位变异的大小记录为0/0(注:小片段在前,大片段在后)。通过对多个位点的数据整合,最终形成不同蚕豆品种的SSR指纹图谱(表5-表16)。除待测样品外,每个SSR位点还应同时包括3~5个参照品种的扩增产物。不同荧光标记的扩增产物的最适稀释倍数最好通过预试验确定。检测结果判定:品种间差异位点数≥3,则为不同品种;品种间差异位点数<3,则为近似品种;比较48份品种的指纹数据,发现任意两个品种间的差异位点数都大于3,说明这21对核心引物可以有效的把这些品种鉴别开;利用PowerMarker和MEGA软件进行品种聚类,分析品种间的遗传关系,从图中可以发现在遗传相似系数小于0.11时可以把全部品种区分开(图1)。表5-表16中的C1-C48分别对应表1中的各蚕豆品种名称。表5表6SSR13771EST1142EST1584SSR11771SSR12868EST1672SSR13683C1163/163213/219183/183149/149144/146181/187147/150C2157/161206/217184/187136/142143/146190/190145/145C3158/158230/236185/185139/146146/146181/184146/150C4164/164206/206185/185149/149144/146187/193144/144C5163/163206/217184/184143/143143/143186/186149/149C6162/162208/218187/187146/146144/147181/181144/144C7162/162217/221188/188146/146144/144190/190150/150C8159/159206/206185/185142/142143/143187/187150/150C9163/167206/206184/184142/142143/143181/193144/144C10160/164206/208188/188141/146144/144181/181145/145C11159/159219/219187/187142/146143/143186/186150/150C12159/159219/237182/182146/149144/147181/181144/147表7SSR760SSR10911SSR13106SSR11052SSR14328SSR12536SSR17991C1168/168166/169167/167187/187177/177192/198181/181C2165/168163/169167/177184/184177/179202/202158/181C3165/174160/176167/167184/184177/177195/195158/158C4165/174166/180167/171184/193177/177198/198159/181C5171/171166/166167/167193/193171/178198/198158/158C6168/168158/158167/169193/193177/177198/198159/159C7168/171157/166169/169184/184171/171198/198158/158C8168/168163/163167/167193/193178/178198/198159/159C9168/167166/166167/167184/193177/182198/198159/180C10159/159163/176169/169193/193177/177201/201158/158C11168/168166/166167/167184/184170/170201/201159/159C12158/168166/169164/164184/187177/177192/198158/158表8表9SSR13771EST1142EST1584SSR11771SSR12868EST1672SSR13683C13161/161210/219186/186136/136143/143181/181150/150C14159/159218/218185/185136/136146/146181/190146/148C15161/167217/217187/187136/136143/143181/181147/147C16162/162217/217182/182136/136143/143181/181144/144C17160/160221/221188/188146/146144/144187/187147/147C18161/161230/230187/187136/142143/146180/192144/150C19163/163226/226187/187136/136146/146181/181147/147C20159/159224/226180/184142/149147/147190/190147/147C21159/159217/219183/187142/146144/144187/187146/146C22161/161206/215184/184136/146144/147181/193144/144C23157/161206/206187/187136/146144/144187/190147/150C24159/159208/218184/184146/146146/146181/187144/144表10SSR760SSR10911SSR13106SSR11052SSR14328SSR12536SSR17991C13168/168163/163169/169187/187177/177199/199160/181C14168/168158/158171/173184/184177/177193/198180/180C15165/165169/169167/167184/184178/178195/198157/157C16165/165169/169167/167178/178178/178192/192159/159C17159/174166/166171/171178/178177/177198/198158/158C18165/165169/169165/165187/187177/177192/198157/157C19174/174166/169167/167187/193178/181193/193159/159C20171/174169/169171/171184/193179/179198/198180/180C21174/174166/166167/169187/187171/178198/198158/158C22168/168166/166171/171193/193178/181201/201158/179C23159/168163/163169/169178/193178/178193/201158/180C24168/168163/169167/167193/193178/178195/198158/181表11表12SSR13771EST1142EST1584SSR11771SSR12868EST1672SSR13683C25157/157217/221185/188146/146144/150181/181146/150C26159/159206/206187/187136/136146/146181/181145/145C27159/159206/206187/187146/146143/143193/193144/144C28158/167213/217187/187146/146146/146181/193145/147C29160/160223/225188/188136/142144/147181/184145/145C30159/159212/227187/187136/146146/146181/181146/146C31161/161221/221187/187146/146144/144187/187145/145C32159/159217/226182/182136/149144/147187/193145/145C33161/163216/216187/187146/146144/147187/187145/145C34163/163218/218187/187146/146144/144187/187145/145C35159/159206/206188/188146/146144/144187/187145/145C36159/159221/221188/188143/143143/143187/187144/144表13SSR760SSR10911SSR13106SSR11052SSR14328SSR12536SSR17991C25159/168166/166165/165184/196171/178195/195160/181C26168/168163/163167/167193/193181/181202/202158/158C27168/168158/166171/171193/193178/178198/198158/158C28171/171166/169167/171184/184177/177198/198180/180C29158/167168/171171/177184/184178/178198/198158/158C30168/171166/169165/165178/184177/177198/198157/180C31168/168163/163171/171136/136178/178198/198158/158C32174/174163/169167/167187/187171/181198/201180/180C33168/168166/166171/171193/193177/177198/198158/158C34174/174163/163169/171193/239177/177198/198181/181C35174/174166/166169/169194/194177/177199/199158/158C36168/168163/163171/171236/236178/178198/198158/158表14表15SSR13771EST1142EST1584SSR11771SSR12868EST1672SSR13683C37164/164221/221186/186146/146144/144187/187145/145C38162/162206/206185/185146/146144/144187/187145/145C39163/163219/219186/188136/136143/143187/187150/150C40162/162219/219188/188146/146144/144181/187147/147C41159/159206/206185/185146/146143/143181/181145/145C42159/159217/217183/183143/143146/146187/187150/150C43159/159211/211185/185146/146147/147181/181146/146C44159/159204/204185/185127/127143/143190/190147/147C45159/159225/230184/187136/136146/146187/187144/149C46160/165221/228185/188146/146144/144187/183144/144C47161/161212/219185/188136/146147/147187/193145/150C48160/160217/217188/188146/146144/144187/187145/145表16SSR760SSR10911SSR13106SSR11052SSR14328SSR12536SSR17991C37174/174158/158171/171237/237178/178203/203158/158C38174/174163/163169/169237/237178/178199/199158/158C39168/168163/163167/167184/184170/178195/202160/160C40168/168163/163167/167184/193170/177196/203157/157C41168/168163/163167/167184/184177/177201/201159/159C42158/158169/169165/167184/184178/178199/199158/158C43165/165169/169167/167187/187181/181198/198158/158C44174/174166/166169/169187/187179/179198/198159/159C45165/174166/169167/167184/184178/178192/198157/157C46168/168163/163167/171193/193178/182196/202158/158C47159/171163/166167/169193/193178/182199/199159/159C48168/168167/167167/167237/237178/178199/199180/180实施例3本发明鉴定蚕豆品种方法的应用提取已知品种名称的6个蚕豆样品的叶片DNA,以本发明实施例1筛选得到的分子标记组合中的21对引物分别对待检蚕豆品种的DNA进行PCR扩增;参照本发明实施例2的PCR反应体系,PCR反应程序,聚丙烯酰胺凝胶电泳和变异位点记录方法(方法1),同时重复实验采用荧光毛细管电泳法和变异位点记录方法(方法2),将得到的指纹代码与本发明得到的蚕豆品种的特征指纹图谱(表5-表16)进行比对,以验证本发明实施例2建立的鉴定方法准确度。结果发现:21对引物组合对6个蚕豆品种进行PCR扩增,上述两种方法测得的6个蚕豆样品变异位点均一致,结果见表17-表19,且与本发明建立的蚕豆指纹图谱目标品种的品种间差异位点数<3,F1-F6为待测6个蚕豆品种,经过对比鉴定得到F1-F6分别对应临蚕3号,启豆5号,青海4号,成胡11号,翼蚕二号,云豆83-63,与已知品种名称一致,准确度100%。表明本发明实施例1筛选得到的21对引物组合可以较好的可以区分不同蚕豆品种。表17DNA编号EST181SSR11885SSR12396SSR11908SSR12080EST1280EST1741F1200/202167/167164/164163/169211/214184/184181/181F2208/208167/167171/171166/166207/207186/186186/186F3206/206170/170170/170163/163210/210184/184189/189F4208/208168/171164/164160/163211/211184/186181/181F5200/207167/167170/170161/161204/211183/183181/181F6208/208167/167170/170166/166204/204198/198180/180表18DNA编号SSR13771EST1142EST1584SSR11771SSR12868EST1672SSR13683F1158/158230/236185/185139/146146/146181/184146/150F2159/159219/219187/187142/146143/143186/186150/150F3162/162217/217182/182136/136143/143181/181144/144F4157/157217/221185/188146/146144/150181/181146/150F5159/159212/227187/187136/146146/146181/181146/146F6163/163218/218187/187146/146144/144187/187145/145表19DNA编号SSR760SSR10911SSR13106SSR11052SSR14328SSR12536SSR17991F1165/174160/176167/167184/184177/177195/195158/158F2168/168166/166167/167184/184170/170201/201159/159F3165/165169/169167/167178/178178/178192/192159/159F4159/168166/166165/165184/196171/178195/195160/181F5168/171166/169165/165178/184177/177198/198157/180F6174/174163/163169/171193/239177/177198/198181/181说明应用本发明实施例2提供的鉴别蚕豆品种的方法准确性较高,适合推广使用。虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。SEQUENCELISTING<110>中国农业科学院作物科学研究所<120>一种蚕豆SSR指纹图谱的构建方法<130>KHP161117235.4<160>42<170>PatentInversion3.5<210>1<211>20<212>DNA<213>人工序列<400>1tccatgggatcttcacaaca20<210>2<211>20<212>DNA<213>人工序列<400>2tcgaagtcccttcatcatca20<210>3<211>20<212>DNA<213>人工序列<400>3gagaaaagcggctgcttaga20<210>4<211>20<212>DNA<213>人工序列<400>4gctgtcaccgagaatgatga20<210>5<211>20<212>DNA<213>人工序列<400>5tcctccaggtccaaaaacac20<210>6<211>19<212>DNA<213>人工序列<400>6aacccgatccgtttcatct19<210>7<211>20<212>DNA<213>人工序列<400>7ccgatttcagcaacctgttt20<210>8<211>20<212>DNA<213>人工序列<400>8gtgaccccatttgcagactc20<210>9<211>20<212>DNA<213>人工序列<400>9aaatatgggttggcgacttg20<210>10<211>23<212>DNA<213>人工序列<400>10gaattgaccattgactctcttca23<210>11<211>20<212>DNA<213>人工序列<400>11gctagtggacctcccattga20<210>12<211>21<212>DNA<213>人工序列<400>12tccaatgcaaactctccaaac21<210>13<211>20<212>DNA<213>人工序列<400>13gcaccgtgctaagatgatga20<210>14<211>20<212>DNA<213>人工序列<400>14gggcccactcatttttgtta20<210>15<211>24<212>DNA<213>人工序列<400>15gctgacatattgtctgcaataacc24<210>16<211>23<212>DNA<213>人工序列<400>16tgcggaaaacagtaatattgaaa23<210>17<211>20<212>DNA<213>人工序列<400>17tgctggaacaacaacctgaa20<210>18<211>20<212>DNA<213>人工序列<400>18tgtgcactctccgcatctac20<210>19<211>19<212>DNA<213>人工序列<400>19cggccacaaacaaggttta19<210>20<211>20<212>DNA<213>人工序列<400>20caatccaagcacaccaatca20<210>21<211>20<212>DNA<213>人工序列<400>21cacctccaccccgtactcta20<210>22<211>20<212>DNA<213>人工序列<400>22aaacaaggtgcttccaccag20<210>23<211>20<212>DNA<213>人工序列<400>23accaccttctgaggaacagc20<210>24<211>27<212>DNA<213>人工序列<400>24ttgtgcaaatatgacattttatttaag27<210>25<211>20<212>DNA<213>人工序列<400>25ctctctagtggcctgggtgt20<210>26<211>20<212>DNA<213>人工序列<400>26cgatggggtgtttctctctc20<210>27<211>23<212>DNA<213>人工序列<400>27gaattttcaaaacatgagtccca23<210>28<211>20<212>DNA<213>人工序列<400>28ccggatctgaaaagacttgc20<210>29<211>20<212>DNA<213>人工序列<400>29ccaaagcaaacccaacaagt20<210>30<211>20<212>DNA<213>人工序列<400>30cctttgcttccagattctcc20<210>31<211>20<212>DNA<213>人工序列<400>31tcctcaaacatgcaggacag20<210>32<211>20<212>DNA<213>人工序列<400>32ttgttccgagcaacagtttg20<210>33<211>21<212>DNA<213>人工序列<400>33cacccttcttgttcccttttt21<210>34<211>20<212>DNA<213>人工序列<400>34ccggcggaactaggtatctt20<210>35<211>20<212>DNA<213>人工序列<400>35ctccgcgagcatcactttat20<210>36<211>20<212>DNA<213>人工序列<400>36ttgcacgatctcaactcacc20<210>37<211>20<212>DNA<213>人工序列<400>37cgttgcctggttcgtactct20<210>38<211>20<212>DNA<213>人工序列<400>38acgacgaatttccaccactc20<210>39<211>20<212>DNA<213>人工序列<400>39aaattggtgggagacaccag20<210>40<211>21<212>DNA<213>人工序列<400>40aagaactaccggaagcagaca21<210>41<211>20<212>DNA<213>人工序列<400>41agccaatttgacccatcatt20<210>42<211>20<212>DNA<213>人工序列<400>42tcctcccttagtggctcttg20当前第1页1 2 3
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1