经遗传修饰的动物及其生成方法与流程
本申请是基于申请日为2012年2月24日,优先权日为2011年2月25日,申请号为201280020439.x,发明名称为:“经遗传修饰的动物及其生成方法”的专利申请的分案申请。
对相关申请的交叉引用
本专利要求2011年2月25日提交的美国流水号61/446,651的优先权,其在此通过提及并入本文用于所有目的。
有关联邦资助研究的声明
本发明在国立健康研究所资助的拨款号1r41hl108440-01“developmentofporcinegeneticmodelsofatherosclerosisawardedbythenationalinstitutesofhealth”下得到政府支持而做出。美国政府具有本发明的某些权利。
发明领域
技术领域涉及创建经遗传修饰的动物,例如具有功能性状的家畜动物。
发明背景
转录激活物样(tal)效应序列可以通过装配重复可变二残基(rvd)序列来装配以特异性结合dna靶物。tal效应物和核酸酶的融合蛋白(talen)可以在细胞dna中产生靶向性双链断裂,其可以用于对细胞产生特定的遗传修饰。已经报告了talen可用于生成稳定修饰的人胚胎干细胞和诱导的多能干细胞克隆,从而生成敲除的秀丽隐杆线虫(c.elegans)和敲除的斑马鱼。
发明概述
转录激活物样(tal)效应物(tale)与核酸酶的融合(talen)已被报告用在了对细胞的遗传修饰中。这些报告一般已经聚焦于植物细胞、经转化的(永生化)动物细胞系、或小鼠胚胎干细胞。图1描绘了基于talen的遗传工程化系统的一些特征。
本文中列出的一个实施方案是一种创建遗传修饰的方法,其包括将体外培养物中的原代细胞或胚胎暴露于编码talen的核酸,其中所述细胞是家畜细胞,且所述胚胎是家畜胚胎。所述方法可以包括将所述原代细胞暴露于所述体外培养物中的所述核酸。所述编码talen的核酸可以包括mrna。所述talen可以是左talen,且所述方法可以进一步包括右talen,该右talen与左talen协作以在dna中产生双链切割。第二载体可以包含所述编码talen的核酸。所述方法可以进一步包括将所述细胞或所述胚胎暴露于编码报告物的载体。所述方法可以包括编码选择标志物的载体。所述报告物可以包含选择标志物。报告物可以需要由要表达的转座酶实施的转座事件。所述方法可以包括选择表达所述报告物的细胞或胚胎,用于进一步处理。所述方法可以包括其中基于消除所述培养物中不表达所述报告物的细胞来选择所述细胞。所述方法可以包括其中所述报告物包含荧光生物分子。所述方法可以包括其中基于报告物的表达选择所述胚胎。所述方法可以包括其中基于报告物的表达选择细胞。所述方法可以包括其中所述载体包含质粒。所述方法可以包括其中载体包含编码所述报告物的转座子,且进一步包括编码识别所述转座子的转座酶的载体。所述方法可以在本文中包括所述转座酶选自下组:sleepingbeauty、frogprince、tol2、minos、hsmar、plaice、和passport。所述方法可以包括选择表达所述报告物的细胞或胚胎,并使用所述细胞或胚胎来创建经遗传修饰的动物,其中所述遗传修饰是在所述talen特异性结合的dna位点处。所述方法可以包括对所述动物育种,并选择表达所述遗传修饰并且没有外源报告物的后代。所述方法可以包括其中所述动物在所述修饰方面是纯合双等位的,或者在修饰方面是杂合的,或者在修饰方面是杂合双等位的。所述方法可以包括从所述培养物的所述细胞克隆经遗传修饰的动物,其中所述细胞在所述talen特异性结合的dna位点处包含遗传修饰。所述方法在本文中可以包括使用所述细胞通过体细胞核转移或染色质转移来克隆所述动物。所述方法可以包括其中所述遗传修饰选自下组:插入、缺失、外源核酸片段的插入、倒位、基因转化成天然等位基因、基因转化成合成等位基因、和基因转化成新等位基因。所述方法可以包括将重组酶投递至所述细胞或胚胎。所述方法可以包括通过直接注射来递送重组酶,或通过编码蛋白质、mrna形式的重组酶的载体来投递重组酶,或通过编码重组酶的载体来投递重组酶。所述方法可以包括其中所述重组酶与核酸组合以形成纤丝,其中所述核酸提供同源性依赖性修复的模板。所述方法在本文中可以包括所述重组酶选自下组:reca、reca803、uvsx、reca突变体、reca样重组酶、cre重组酶、hin重组酶、rad51、tre、flp、ruvc、dst2、kem1、xrn1、stpa/dstl、和hpp-1。所述方法在本文中可以包括所述细胞是受精卵,或者所述胚胎是囊胚。
本文中所列的另一个实施方案是细胞或胚胎,其包含编码talen的核酸片段和talen特异性结合的dna位点处的遗传修饰,其中所述细胞包含自动物组织分离的原代细胞,且其中所述细胞和所述胚胎选自下组:偶蹄动物、猪、牛、鱼、兔、和家畜。例如,可以将所述原代细胞暴露于体外培养物中的所述核酸。所述编码talen的核酸可以包括mrna。细胞或胚胎可以包含编码报告物的载体。细胞或胚胎可以包含编码选择标志物的载体。细胞或胚胎可以包含其中所述报告物包含选择标志物。细胞或胚胎可以包含编码报告物的载体,和编码所述talen的载体。细胞或胚胎可以包含编码转座酶的载体,其中所述报告物和所述talen是由其相应载体编码的转座子。所述细胞或胚胎在所述修饰方面可以是纯合双等位基因的。细胞或胚胎可以包含外源重组酶。
本发明的一个实施方案是经遗传修饰的家畜动物,其包含选自下组的遗传修饰:插入、缺失、外源核酸片段的插入、倒位、基因转化成天然等位基因、基因转化成合成等位基因、和基因转化成新等位基因,其中所述动物没有外源报告物基因。家畜动物可以选自下组:偶蹄动物、牛、猪、绵羊、山羊、兔、家禽、和鱼。所述遗传修饰可以包含缺失,该缺失包括500个碱基对、或兆碱基对、或范围为约10%至约90%的染色体量的缺失;技术人员会立即领会涵盖明确叙述范围内的所有范围和数值。所述遗传修饰可以包含缺失,该缺失包括兆碱基的染色体dna。所述遗传修饰可以包含倒位,该倒位包含500个碱基对。所述遗传修饰可以包含倒位,其中所述倒位编码遗传性状。家畜动物可以包含不是belgianblue牛的动物牛系,其中所述遗传修饰包含belgianblue牛中普遍的肌肉生长抑制素突变缺失。
一个实施方案是一种遗传修饰家畜动物的方法,其包括选择性状,并在用作家畜动物祖先的细胞或胚胎中将编码该性状的dna区倒位。所述遗传修饰可以包括倒位或缺失,所述倒位或缺失包括500个碱基对、或兆碱基对、或范围为约10%至约90%的染色体量;技术人员会立即领会涵盖明确叙述范围内的所有范围和数值。
一个实施方案是一种遗传修饰动物的方法,其包括将祖细胞或胚胎暴露于第一位点处的第一talen和第二位点处的第二talen,其中所述位点之间的区域是缺失的。所述第一talen可以包含第一talen对,且所述第二talen可以包含第二talen对。例如,在使用祖细胞的情况中,可以使用自所述祖细胞的体细胞核转移或染色质转移来创建所述动物。所述方法可以是如下的方法,其包括将所述talen导入所述祖细胞中,而且还导入包含报告物的载体,并仅在表达所述报告物时选择所述祖细胞或胚胎以创建所述动物。
一个实施方案是将等位突变从第一家畜品种转移至第二家畜品种的方法,其包括:在存在编码所述第一家畜品种的所述等位突变的核酸的情况中将talen导入所述第二家畜品种的细胞或胚胎中,其中将所述等位突变复制到所述细胞或所述胚胎中,并从所述细胞或所述胚胎创建所述第二品种的动物。复制过程可以包含hdr模板。等位突变可以包含例如存在于belgianblue牛中的肌肉生长抑制素突变等位基因。talen可以以蛋白质导入或者由mrna或由载体编码。所述方法可以包括不依赖于所述talen导入报告物载体。所述等位基因可以与所述第一家畜品种的性状有联系。所述方法可以进一步包括将重组酶投递至所述细胞或胚胎。以蛋白质、mrna、或通过编码所述重组酶的载体投递所述重组酶。可以使用所述重组酶来与核酸组合以形成纤丝。
一个实施方案是一种经遗传修饰的家畜动物,其包含:家畜动物的第一品种,其包含家畜动物的第二品种的等位基因,其中所述家畜动物的第一品种没有与所述动物的第二品种的减数分裂重组。例如,所述动物可以选自下组:猪、牛、绵羊、山羊、鸡、兔、和鱼。举例而言,所述第一品种是wagyu牛,且所述第二品种是belgianblue牛。所述等位基因可以选自下组:插入、缺失、多态性、和单核苷酸多态性。所述动物可以没有外源标志物基因。动物可以包含多个所述等位基因。一种或多种等位基因可以与所述第二品种的数量性状或质量性状有联系。
在此通过提及将下列专利申请收入本文用于所有目的;在冲突的情况中,以说明书为准:us2010/0146655,us2010/0105140,us2011/0059160,和us2011/0197290。
本发明包括以下内容:
实施方案1.一种创建遗传修饰的方法,其包括将体外培养的原代细胞或将胚胎暴露于编码talen的核酸,其中所述细胞是家畜细胞,且所述胚胎是家畜胚胎。
实施方案2.实施方案1的方法,其中将所述原代细胞暴露于所述体外培养的所述核酸。
实施方案3.实施方案1的方法,其中所述编码talen的核酸包括mrna。
实施方案4.实施方案1的方法,其中所述talen是左talen,且所述方法进一步包括右talen,该右talen与左talen协作以在dna中产生双链切割。
实施方案5.实施方案1的方法,其中有第二载体来包含所述编码talen的核酸。
实施方案6.实施方案1的方法,其进一步包括将所述细胞或所述胚胎暴露于编码报告物的载体。
实施方案7.实施方案6的方法,其进一步包括编码选择标志物的载体。
实施方案8.实施方案6的方法,其中所述报告物包含选择标志物。
实施方案9.实施方案6的方法,其进一步包括选择表达所述报告物的细胞或胚胎以进一步处理。
实施方案10.实施方案9的方法,其中基于从所述培养物中消除不表达所述报告物的细胞来选择所述细胞。
实施方案11.实施方案9的方法,其中所述报告物包含荧光生物分子。
实施方案12.实施方案9的方法,其中基于所述报告物的表达选择所述胚胎。
实施方案13.实施方案9的方法,其中基于所述报告物的表达选择所述细胞。
实施方案14.实施方案6的方法,其中所述载体包含质粒。
实施方案15.实施方案6的方法,其中所述载体包含编码所述报告物的转座子,且进一步包括编码识别所述转座子的转座酶的载体。
实施方案16.实施方案15的方法,其中所述转座酶选自下组:sleepingbeauty、frogprince、tol2、minos、hsmar、plaice、和passport。
实施方案17.实施方案1的方法,其进一步包括选择表达所述报告物的细胞或胚胎,并使用所述细胞或胚胎来创建经遗传修饰的动物,其中所述遗传修饰位于被所述talen特异性结合的dna位点处。
实施方案18.实施方案17的方法,其进一步包括对所述动物育种,并选择表达所述遗传修饰并且没有外源报告物的后代。
实施方案19.实施方案18的方法,其中所述动物在所述修饰方面是纯合双等位的。
实施方案20.实施方案1的方法,其进一步包括从所述培养物的所述细胞克隆出经遗传修饰的动物,其中所述细胞在被所述talen特异性结合的dna位点处包含遗传修饰。
实施方案21.实施方案20的方法,其中所述细胞用于通过体细胞核转移或染色质转移来克隆所述动物。
实施方案22.实施方案1的方法,其中所述遗传修饰选自下组:插入、缺失、外源核酸片段的插入、倒位、基因转化成天然等位基因、基因转化成合成等位基因、和基因转化成新等位基因。
实施方案23.实施方案1的方法,其进一步包括将重组酶投递至所述细胞或胚胎。
实施方案24.实施方案23的方法,其中所述重组酶作为蛋白质、作为mrna、或通过编码所述重组酶的载体来投递。
实施方案25.实施方案23的方法,其中所述重组酶与核酸组合以形成纤丝,其中所述核酸提供同源性依赖性修复的模板。
实施方案26.实施方案23的方法,其中所述重组酶选自下组:reca、reca803、uvsx、reca突变体、reca样重组酶、cre重组酶、hin重组酶、rad51、tre、flp、ruvc、dst2、kem1、xrn1、stpa/dstl、和hpp-1。
实施方案27.实施方案1的方法,其中所述细胞选自下组:家畜细胞、偶蹄动物细胞、培养细胞、原代细胞、原代体细胞、受精卵、原始生殖细胞、或干细胞和受精卵,其中所述胚胎是囊胚。
实施方案28.一种细胞或胚胎,其包含编码talen的核酸片段和位于被talen特异性结合的dna位点处的遗传修饰,其中所述细胞包含自动物组织分离的原代细胞,且其中所述细胞和所述胚胎选自下组:偶蹄动物、猪、牛、鱼、兔、和家畜。
实施方案29.实施方案28的细胞或胚胎,其包含所述细胞,其中所述原代细胞暴露于体外培养的所述核酸。
实施方案30.实施方案28的细胞或胚胎,其中所述编码talen的核酸包括mrna。
实施方案31.实施方案28的细胞或胚胎,其进一步包含编码报告物的载体。
实施方案32.实施方案28的细胞或胚胎,其进一步包含编码选择标志物的载体。
实施方案33.实施方案31的细胞或胚胎,其中所述报告物包含选择标志物。
实施方案34.实施方案28的细胞或胚胎,其进一步包含编码报告物的载体,和编码所述talen的载体。
实施方案35.实施方案34的细胞或胚胎,其进一步包含编码转座酶的载体,其中所述报告物和所述talen是由其相应载体编码的转座子。
实施方案36.实施方案28的细胞或胚胎,其包含所述胚胎,其中所述胚胎在所述遗传修饰方面是纯合双等位的。
实施方案37.实施方案28的细胞或胚胎,其进一步包含外源重组酶。
实施方案38.一种经遗传修饰的家畜动物,其包含选自下组的遗传修饰:插入、缺失、外源核酸片段的插入、倒位、基因转化成天然等位基因、基因转化成合成等位基因、和基因转化成新等位基因,其中所述动物没有外源报告物基因。
实施方案39.实施方案38的家畜动物,其选自下组:牛、猪、绵羊、山羊、兔、家禽、和鱼。
实施方案40.实施方案38的家畜动物,其中所述遗传修饰包含缺失,该缺失包括500个碱基对的缺失。
实施方案41.实施方案38的家畜动物,其中所述遗传修饰包含缺失,所述缺失包含兆碱基对的缺失。
实施方案42.实施方案38的家畜动物,其中所述遗传修饰包含倒位,该倒位包含兆碱基的染色体dna。
实施方案43.实施方案38的家畜动物,其中所述遗传修饰包含倒位,该倒位包含500个碱基对。
实施方案44.实施方案38的家畜动物,其中所述遗传修饰包含倒位,其中所述倒位编码遗传性状。
实施方案45.实施方案38的家畜动物,其包含不是belgianblue牛的牛系动物,其中所述遗传修饰包含belgianblue牛中流行的肌肉生长抑制素缺失。
实施方案46.一种遗传修饰家畜动物的方法,其包括选择性状,并在用作家畜动物祖先的细胞或胚胎中将编码该性状的dna区倒位。
实施方案47.实施方案46的方法,其中所述倒位包含1000个碱基对。
实施方案48.实施方案46的方法,其中所述倒位包含兆碱基的染色体dna。
实施方案49.一种遗传修饰动物的方法,其包括将祖细胞或胚胎暴露于第一talen和第二talen,所述第一talen在第一dna位点处特异性结合,所述第二talen在第二dna位点处特异性结合,其中所述位点之间的区域是缺失的。
实施方案50.实施方案49的方法,其中所述第一talen包含第一talen对,且所述第二talen包含第二talen对。
实施方案51.实施方案49的方法,其包括所述祖细胞,其中使用自所述祖细胞进行的体细胞核转移或染色质转移来创建所述动物。
实施方案52.实施方案49的方法,其包括将所述talen导入所述祖细胞中,而且还导入包含报告物的载体,并仅在表达所述报告物时选择所述祖细胞或胚胎以创建所述动物。
实施方案53.将等位基因从第一家畜品种转移至第二家畜品种的方法,其包括:
在存在编码所述第一家畜品种的所述等位基因的核酸的情况中将talen导入所述第二家畜品种的细胞或胚胎中,其中将所述等位基因复制到所述细胞或所述胚胎中,并从所述细胞或所述胚胎创建所述第二品种的动物。
实施方案54.实施方案53的方法,其中所述等位基因包括存在于belgianblue牛中的肌肉生长抑制素(myostatin)等位基因。
实施方案55.实施方案53的方法,其中所述talen由mrna或由载体编码。
实施方案56.实施方案53的方法,其中所述talen是右talen,且进一步包含左talen。
实施方案57.实施方案53的方法,其进一步包括不依赖于所述talen而导入报告物载体。
实施方案58.实施方案53的方法,其中所述等位基因与所述第一家畜品种的性状有联系。
实施方案59.实施方案53的方法,其进一步包括将重组酶投递至所述细胞或胚胎。
实施方案60.实施方案59的方法,其中所述重组酶作为蛋白质、作为mrna、或通过编码所述重组酶的载体来投递。
实施方案61.实施方案59的方法,其中所述重组酶与所述核酸组合以形成纤丝。
实施方案62.一种经遗传修饰的家畜动物,其包含:
家畜动物的第一品种,其包含家畜动物的第二品种的等位基因,
其中所述家畜动物的第一品种没有与动物的第二品种的减数分裂重组。
实施方案63.实施方案62的动物,其中所述动物选自下组:猪、牛、绵羊、山羊、鸡、兔、和鱼。
实施方案64.实施方案62的动物,其中所述第一品种是wagyu牛,且所述第二品种是belgianblue牛。
实施方案65.实施方案62的动物,其中所述等位基因选自下组:插入、缺失、多态性、和单核苷酸多态性。
实施方案66.实施方案62的动物,其中所述动物没有外源标志物基因。
实施方案67.实施方案62的动物,其包含多个所述等位基因。
实施方案68.实施方案62的动物,其中所述等位基因与所述第二品种的数量性状或质量性状有联系。
附图简述
图1是talen及其引起的遗传修饰的例示。
图2是在多个dna基因座处运行的talen的例示。
图3:牛胚胎中的talen活性。小图(a)中给出实验概况。talen设计为与dna靶链相反,使得foki核酸酶同二聚化单体能够二聚化,并且切割两个单体间的dna。在第1天(d1)给体外生成的牛受精卵注射talenmrna,并将其在体外培养至囊胚形成。单个的囊胚(blasts)在第8天时收集,对其进行全基因组扩增(wga),通过pcr扩增和cel-i(surveyor核酸酶,transgenomics)处理来分析indel(插入缺失)。小图b)surveyor核酸酶处理以分析由acan12talens介导的牛胚胎中的indel。上文显示了扩增子长度和预测的surveyor切割产物,它们指示这些indel。小图c)surveyor核酸酶处理以分析由p65talens介导的猪受精卵中的indel。
图4a:牛胚胎用acan12talen处理后,对缺失和插入进行测序。显示了野生型序列,其中talen结合位点加下划线。鉴定缺失和插入事件两者。
图4b:猪胚胎用acan12talen处理后,对缺失和插入进行测序。显示了野生型序列,其中talen结合位点加下划线。鉴定缺失和插入事件两者。
图5:比较在家畜成纤维细胞中用于基因编辑的talen支架。小图a)在此实验中测试的talen支架的图。每个支架(+231,christian等2010{christian,2010}和+63,miller等2011{miller,2011})含有sv40核定位信号(nls),并且具有foki同二聚体域的c端融合。相对于该dna结合域编号。在第一个重复可变二残基重复单元(rvd)前的氨基酸标记为“-1”,而最后一个rvd重复单元后的氨基酸标记为“+1”。小图b)成纤维细胞用dmde7.1或acan12talen对转染,然后进行surveyor测定法。在凝胶上方标示支架和处理温度,在下方标示百分比nhej。缩写,nt=未处理。小图c)具有+231或+63支架的四种别的talen对的活性。
图6:细胞用acan12talen处理后,对缺失和插入进行测序。野生型aca12序列以斜体字显示,左侧和右侧(互补性)talen-识别序列加下划线。插入的核苷酸以灰色突出显示,错配的核苷酸以小写字体文本表示。
图7:用于indel富集的转座子共选择事件。在小图(a)中显示实验时间线。第0天(d0),用包含每个talen的表达盒、编码选择标志物的转座子、和转座酶表达盒的质粒混合物转染细胞。talen质粒是每次转染的主要组分(按质量计4倍过量)。将经转染的细胞在30或37摄氏度培养3天,之后分开,收集样品以进行surveyor测定法,并再次铺板以便进一步培养+/-选择转座子的整合。第3天后,所有细胞于37摄氏度培养。收集培养14+天的细胞以进行surveyor测定法,并冻存以备后续应用,即单细胞核转移。小图b)使用阳离子脂质转染成纤维细胞。在第3天时没有观察到活性(由于低转染效率),因此仅有第14+天的群体数据。凝胶上方显示了处理温度、选择和talen编号(用字母a-c指示,如小图(c)中标示的)。小图c)成纤维细胞通过核转染进行转染,并测量第3天的百分比nhej/以及第14+天的非选择(ns)和选择(s)群体中的百分比nhej。在每个矩阵上方标示处理温度。缩写:nd=未检测;wt=野生型扩增子,经surveyor处理的。
图8a:直接pcr测序以鉴定indel。将来自各个成纤维细胞集落的pcr扩增子纯化、测序、并与野生型序列比较。一个等位基因的突变,或者两个等位基因的非重叠突变会导致talen识别位点附近的双重序列(顶部)。可以鉴定重叠的双等位突变,其中可以通过该突变位点侧翼的双峰来鉴定每个等位基因间的差异。具有纯合突变的集落没有展示indel位点附近的双峰。
图8b:野生型和图8a所示具有纯合indel的双等位克隆的序列比较。
图9a:dmd双等位修饰等位基因。显示了具有纯合修饰等位基因(即两个等位基因都含有相同突变)的集落、或在每个等位基因上具有不同突变的双等位突变的集落。对于具有两处indel的集落,在右侧显示每个等位基因的测序次数。在一些情况中,对第三处突变或单一野生型等位基因测序,指示不是所有集落是100%克隆的。移码等位基因已标示,错配核苷酸以小写字母文本表示。
图9b:ldlr双等位修饰等位基因,其中注解如在9a中一样。
图10:talen诱导的缺失和倒位。小图(a)中显示了dmd基因座的示意图。dna取向以黑色v形表示。共转染入雄性猪成纤维细胞中的靶向外显子6和7的talen(黑色箭头)可以在外显子6和7之间产生nhej融合事件。这可以使用引物(黑色箭头)鉴定,其生成约500bp扩增子。小图b)用靶向外显子6和7的talen同时转染的细胞的surveyor测定法揭示两个位点处的nhejindel。在下方显示百分比nhej。小图c)在同时导入外显子6和外显子7talen两者而非单个地转染时,用假设的缺失位点侧翼的引物进行pcr,生成约500个碱基对的产物。小图d)显示了talen靶位点间的序列的倒位事件的预测后果。dna取向以黑色v形表示。还显示了该倒位基因座5'和3'端的假设侧翼位点外部的引物(黑色箭头)及预测的产物大小。仅在同时导入外显子-6和外显子-7talen两者时,在5'连接和3'连接两者处观察到pcr产物。
图11:dmd缺失序列。显示了来自重复转染的dmd缺失连接。上部,外显子6和7序列分别以绿色和黄色突出显示,且talen识别位点加下划线。插入的核苷酸以红色突出显示。
图12:dmd倒位序列。图示了dmd倒位等位基因,其具有经测序分析的5'连接和3'连接(框示)。下方,显示了每个融合物的预测序列,其在对应于所述talen对的每个间隔臂的中央融合。talen识别位点加下划线。还显示了来自转染群体的经过测序的倒位等位基因。右侧标示了每个等位基因的测序次数。插入的核苷酸加下划线。错配核苷酸以小写字母文本表示。
图13:牛成纤维细胞中的hdr诱导。小图a)talen(btgdf83.1,箭头)和dsdna模板(bb-hdr)设计为,通过双链断裂诱导的同源重组,将11个碱基对的缺失引入牛gdf8的外显子3中(belgiumblue突变)。左talen的结合位点的一半在bb-hdr模板中缺失,并因此应当对talen切割有抗性。小图b)surveyor测定法证明btgdf83.1talen于37和30摄氏度两者的活性。使用引物b和b'(小图a中显示)生成此测定法的pcr产物。这些重复中不包括bb-hdr模板,因为它可以混淆btgdf83.1活性的评估。小图c)等位基因特异性pcr证明hdr诱导依赖于talen和bb-hdr模板的共转染。开发pcr测定法以使用引物c和c'(小图a显示)特异性检测经hdr修饰的gdf8等基因。引物c'的3'端跨越11个碱基对的缺失,并且不能扩增野生型等位基因(wt)。每个pcr反应中包括500份细胞等同物,包括阳性对照“c”。通过实验性反应和对照反应之间的比较性密度测定法来测定百分比hdr。
图14:通过测序确认belgianblue的渐渗(introgression)。图示了wagyu野生型gdf8和belgianblue模板(bb-hdr)。用位于同源臂外部的引物(c和d)对5个pcr阳性集落进行pcr,接着进行克隆及用引物b'测序。与野生型序列的比较显示,在5个集落之4个中。有预期的belgianblue等位基因(杂合)特征性的11个碱基对的缺失。
图15:talen介导的hdr的示意图和凝胶。将靶向猪的第四个外显子低密度脂蛋白受体(ldlr)基因的talen对(ldlr2.1)与超螺旋质粒ldlr-e4n-终止子(其含有与猪ldlr基因对应的同源臂和能通过hdr表达新霉素磷酸转移酶的基因陷阱)共转染。
图16:图15的talen介导的hdr的示意图和凝胶。
图17:通过图15和16的过程创建的转基因猪。
发明详述
将talen成功用于在高等动物诸如猪或牛中产生遗传修饰呈现相当大的挑战。本文中列出了生成此类动物的组合物和方法。这些中的一些方法牵涉从原代偶蹄动物细胞克隆,这也呈现相当大的挑战。此外,呈现了用于鉴定已经用talen修饰的细胞或胚胎的方法,及用于富集经talen处理的细胞或胚胎的方法。出乎意料地,观察到通过talen对一条染色体的遗传修饰经常引起另一条染色体的互补基因座也被细胞机制修饰。
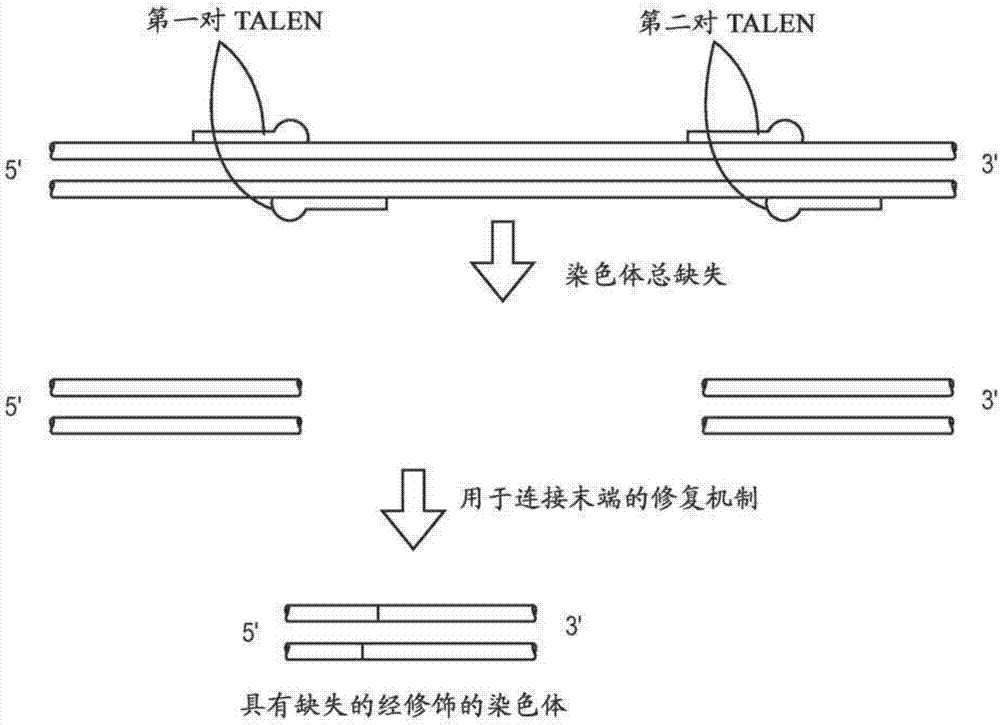
此外,还发现了可以使用talen来在多个位点处产生染色体总缺失(grosschromosomaldeletion,gcd)。图2显示了此方法,其牵涉针对第一基因座的第一talen对和针对第二基因座的第二talen对。令人惊讶地,还发现了可以使用talen对来创建较大染色体序列的倒位。倒位的一种用途是创建具有固定遗传性状的偶蹄动物或其他建立者动物,或者创建缺失品系。
经由talen技术创建经遗传修饰的家畜;确认talen修饰;共转染共选择以选择经修饰的细胞;从经遗传修饰的家畜消除报告物基因。
产生经talen修饰的家畜的障碍之一是对动物细胞做出修饰的效率在常规的最佳实践的情况中仅是几个百分比。在意图位点处实现缺失或插入不必然意味着成功,因为它可以实际上不产生意图的效应,诸如表达外源蛋白质或停止外源蛋白质的表达。即使较低的效率对于创建经遗传修饰的低等动物诸如果蝇或小鼠可以是有用的,因为它们具有较短的且多产的繁殖周期,这提供了创建、测试、并筛选数百个动物以测定是否有少数几个已经成功修饰。然而,常规能实现的这些效率水平不适合于家畜偶蹄动物,其具有长得多的妊娠时间和每次妊娠相当少的后代。
使用talen来修饰家畜的另一个障碍是原代细胞中talen介导的对dna的修饰是困难的,因为细胞是不稳定的。实际上,本文中显示了经talen修饰的细胞的效率在缺乏富集或选择方法的情况中随时间显著降低。不限于具体的理论,推理在非意图位点处的dna切割可以通过诱导凋亡或使非靶基因失能而危害细胞的稳定性。术语原代细胞指自活体动物分离的细胞,其中所述细胞从其自组织分离起已经经历0-2次复制。
因此,常规用于创建和测试经转化细胞以实现成功遗传修饰的技术不能用于原代细胞,这是由于其衰老倾向。通常破坏经talen修饰的细胞以测定其遗传修饰,或者从一个亲本分离以培养具有许多相同细胞的克隆系。然而,原代细胞是本质上不稳定的,并且在尝试遗传修饰并克隆扩充它们时通常经历遗传变化、衰老、和/或细胞死亡。经talen修饰的细胞甚至更不太稳定性,如本文中第一次证明的。因此,在使用牵涉修饰原代细胞以进行体细胞核转移或其它动物克隆技术的常规方法时预期高成功率是不合理的。然而,如本文中报告的,已经使用talen来生成经遗传修饰的偶蹄动物原代细胞。这些修饰适合于通过克隆生成经遗传修饰的动物系的建立者。本文中还描述了用于修饰受精卵的直接胚胎注射,其中经修饰的受精卵适合于植入替代雌性中以实现建立者动物系的妊娠和分娩。
一种用于测试实际talen介导的插入/缺失事件的典型方法是对经修饰的细胞或受精卵测序,这是一个破坏性过程。如此,在对代替物植入前修饰受精卵或胚胎时,在动物出生前,不能以任何便利程度确认其修饰。不能按常规预测,通过克隆法生成经遗传修饰的动物的实际生产国车会受益于测试遗传修饰存在的方法。本文提到发明,它们提供了在单细胞、受精卵或卵母细胞阶段对遗传修饰的指示。如本文中显示的,不与talen修饰偶联的报告物基因的表达,不管是不是报告物基因表达盒的一部分,一般都预示所期望的遗传修饰。更具体地,报告物基因的表达指示核酸被更有效投递,并且在细胞或胚胎中表达;报告物表达细胞或胚胎更有可能已经经历基于talen的修饰。
另一种用于生成经修饰的生物体的技术是使用共转染、共选择技术。选择表达报告物的细胞,且可以使用它来生成经遗传修饰的动物。报告物可以选那些需要转座酶活性者。不限于具体的理论,认为已经历转座的细胞是:1)已被转染的,并且2)有双链dna修复能力,如此在选定克隆中提高基于talen的修饰的可能性。这也促进了对经转座的细胞的富集/选择(且通过延伸经talen修饰的细胞)。转座子与talen修饰可操作但非物理连锁的实情容许其通过育种彼此分开。共转染策略的一个益处是,可以在与经talen修饰的染色体不同的染色体上放置一种或多种报告物。此过程提供了没有报告物基因的建立者动物的创建。例如,通过使用携带报告物基因的质粒生成一些动物,所述报告物基因不依赖于遗传修饰,其在细胞中分开协调。此方案基于如下的运行理论,即掺入新报告物基因的细胞也会掺入遗传修饰。例如,本文中提供的数据显示了可以用四种不同质粒转染细胞,并且一种质粒的基因产物的成功掺入预测其它质粒基因产物的成功掺入以及还有talen介导的变化的成功。
家畜胚胎中的talen功能用体外制备的(ivp)牛和猪胚胎来调查。实施例1描述了将talens(左talen和右talen)直接注射到牛胚胎中以生成在talen特异性结合的位点处具有修饰的经遗传修饰的动物。修饰包括纯合双等位和杂合双等位修饰。talenmrna被直接注射到胚胎中,观察到成功的遗传修饰。实施例2描述了其它实验,其中将报告物mrna与talenmrna共注射。报告物的表达预测成功的遗传修饰,其中约35%胚胎表达报告物,且这些动物约30%具有基于talen的indel。在具有indel的动物中,约35%是纯合或杂合双等位突变体(图4)。如此,显示了使用talens进行的直接胚胎修饰是对于家畜基因组修饰可行的办法。如此,可以产生胚胎,并使用公知的方法植入替代雌性中以实现动物建立者系的妊娠和分娩。此外,有可能使用报告物来选择细胞(例如原代细胞、受精卵、卵母细胞、囊胚)以进一步用于克隆或其它过程。
还开发了通过克隆的talen介导对家畜的遗传修饰的方法。实施例3描述了合适的talens的开发及对猪和牛的原代体细胞的talen修饰。成功修饰的效率是略低的,且未使用衡量修饰成功与否的报告物。核转染是一种用于将外来核酸以高效力导入细胞中的手段,但是它是昂贵的,产生高水平的细胞毒性,而且对于许多研究人员是不可用的。因此,使用常见的阳离子脂质转染试剂作为遗传修饰的媒介物。如实施例4中显示的,尽管用阳离子脂质的转染效率小于5%,修饰水平通过转座子共选择显著富集。虽然基因修饰在第3天群体(数据未显示)和第14天群体中在没有转座子介导的选择的情况中低于检测,但是选定群体中的修饰水平对于dmd7.1,dmd6和ldlr2.1分别达到31、13和20%(图7)。然后,将转座子共选择应用于通过核转染来转染的细胞,其中>90%转染效率是常规的。转座子共选择对于维持通过核转染来转染的经修饰的细胞是有效的,然而,除了acan12外,经过3天,核转染未能显著富集经修饰的细胞(图7)。如此,转座子共选择在转染效率较低时是一种有效的富集方法,而在转染效率较高是是一种有效的维持方法。共选择方法在使用饲养细胞时也是有效的,如实施例5中证明的。观察到高得出乎意料的双等位修饰比例(约17%至约35%,取决于talen对)。
本发明的一个实施方案是使用talen来遗传修饰家畜诸如偶蹄动物的组合物和方法。上文已经讨论了使用常规方法生成这些动物的许多问题。例如,遗传修饰可以选自下组:插入、缺失、外源核酸片段的插入、倒位、和基因转化成天然的或新的等位基因。例如,可以用正常序列替换染色体或染色体对中不想要的突变。一般地,鉴定靶dna位点,并创建talen对,其会特异性结合所述位点。将talen例如以蛋白质、mrna或者通过编码talen的载体投递至细胞或胚胎。talen切割dna以生成双链断裂,然后,其得到修复,经常导致indel的创建,或掺入伴随外源核酸中含有的序列或多态性,所述伴随外源核酸被插入或充当用经修饰的序列修复断裂的模板。术语外源核酸指对细胞或胚胎添加的核酸,而不管核酸是相同的还是与天然在细胞中的核酸序列不同。术语核酸片段是较宽的,且包括染色体、表达盒、基因、dna、rna、mrna、或其部分。例如,细胞或胚胎可以选自下组:家畜、偶蹄动物、牛、猪、绵羊、山羊、鸡、兔、和鱼。术语家畜意指作为食物或生物学材料商品饲养的家养动物。术语“偶蹄动物”意指在每只脚上具有偶数数目的趾(通常两个或者有时四个)的偶蹄目(artiodactyla)的有蹄哺乳动物,其包括牛、鹿、骆驼、河马、绵羊、和山羊。
一个实施方案涉及生成经遗传修饰的家畜和/或偶蹄动物的组合物或方法,其包括将talen对导入家畜和/或偶蹄动物细胞或胚胎中,并自所述细胞生成家畜动物/偶蹄动物,所述细胞或胚胎在talen对特异性结合的位点处对细胞或胚胎的dna做出遗传修饰。或者,可以使用多种用于导入蛋白质、rna、mrna、dna或载体的已知技术之任一将talen和/或其它因子导入细胞中。可以依照已知方法,例如将胚胎植入妊娠宿主中,或者各种克隆方法自胚胎或细胞生成经遗传修饰的动物。短语“在被talen特异性结合的位点处对细胞dna的遗传修饰”,等等,意指当talen特异性结合其靶位点时,talen上被核酸酶切割处的遗传修饰。核酸酶并不是精确切割talen对所结合的地方,而是切割两个结合位点之间的限定位点。
另一个此类实施方案牵涉可用于克隆动物的细胞的组合物或处理。细胞可以是家畜和/或偶蹄动物细胞、培养的细胞、原代细胞、原代体细胞、受精卵、生殖细胞、原始生殖细胞、或干细胞。例如,一个实施方案是创建遗传修饰的组合物或方法,其包括将培养物中的多个细胞暴露于talen蛋白或编码一种或多种talen的核酸。可以将talen以蛋白质或以核酸片段导入,例如由mrna或载体中的dna序列编码。
动物的遗传修饰也可以包括用报告物转染。如上文讨论的,观察到原代细胞由于由talen和/或talen导入介导的细胞修饰而是不稳定的。因此,原代细胞(等等)的修饰,和/或自此类细胞创建新家畜系的成功与否不是使用常规手段理性预期的。不限于具体理论,推理自第一载体表达基因盒的细胞也有可能被通过mrna或另一种载体独立投递的talen成功修饰。报告物的表达容许消除不表达报告物的细胞。或者,它容许通过克隆或其它转基因动物技术移动来自培养物的表达报告物的细胞以在动物中使用,或者移入第二培养物中,用于进一步培养和/或数目扩充和/或添加别的载体和/或核酸和/或talen和/或其它遗传修饰。如本文中使用的,术语报告物包括报告物和选择标志物。
如本文中使用的,术语“选择标志物”指赋予容许通过正或负存活选择标准分离的性状的遗传表达生物分子。报告物可以是例如荧光标志物,例如绿色荧光蛋白和黄色荧光蛋白。报告物可以是选择标志物,例如嘌呤霉素、更昔洛韦(ganciclovir)、腺苷脱氨酶(ada)、氨基糖苷磷酸转移酶(neo,g418,aph)、二氢叶酸还原酶(dhfr)、潮霉素-b-磷酸转移酶、胸苷激酶(tk)、或叶黄素-鸟嘌呤磷酸核糖转移酶(xgprt)。
本发明的实施方案包括将报告物(例如通过使用载体)和talen(例如通过独立载体或mrna)导入细胞或胚胎中。细胞可以是家畜和/或偶蹄动物细胞。可以例如通过注射或其它手段(例如牵涉细胞培养物)将talen和/或报告物直接导入。可以生成细胞培养物,其包含培养的细胞(原代细胞、受精卵、卵母细胞s、永生化细胞)、编码talen的第一核酸,例如mrna或具有编码talen的dna的载体、和具有编码报告物的dna序列的独立载体。mrna或第一载体不编码任何报告物,而第二载体不编码任何tal,并且不编码任何talen。
用于报告物、选择标志物和/或一种或多种talen的载体可以是质粒、转座子、转座酶、病毒或其它载体,例如如本文中详述的。可以使用转座酶。一个牵涉转座酶的实施方案提供编码转座酶的载体。其它载体编码转座酶识别的转座子,并且具有感兴趣的核酸片段,例如,报告物、选择标志物、用于插入或作为修饰模板的外源核酸、或一种或多种talen。因而,可以用例如1-约6个的多个载体转染细胞或胚胎;技术人员会立即领会,涵盖明确叙述范围内的所有范围和数值,例如2、3、4、5、和6。可以使用更多载体。可以使用报告物来鉴定有可能已经经历talen修饰的细胞。或者,可以使用选择标志物通过破坏不表达选择标志物的细胞或胚胎来富集经talen修饰的细胞的比例。
本发明的一个实施方案是暴露于多种载体,或用多种载体注射的细胞或胚胎培养物。第一载体包含talen或talen对;或者,存在有两个独立提供左侧和右talen的talen载体。第二载体包含报告物。报告物可以提供非破坏性鉴定或者可以是选择标志物。编码选择标志物的载体可以作为报告物载体的备选或者在报告物载体外使用。别的载体可以编码外源核酸。
用于生成经talen修饰的细胞、胚胎、或动物的方法包括对暴露于talen的细胞或胚胎测定报告物的表达,并且在用于生成经遗传修饰的家畜和/或偶蹄动物的方法或组合物中使用所述细胞或胚胎。例如,可以将原代细胞从细胞培养物取出,并用于克隆。或者,可以将原代细胞从培养物取出,并在第二培养物中放置以生成克隆系或用于别的方法。或者,可以使用表达报告物的胚胎或受精卵以植入替代母兽(surrogatedam)中或者可以用于克隆,而不表达报告物的其它胚胎或受精卵不用于克隆。在一些实施方案中,报告物是选择标志物,其用于选择表达标志物的细胞或胚胎。
总的染色体缺失和倒位;经遗传修饰的动物
用针对多个dna位点的talen实施实验。各位点以几千个碱基对分开。观察到,可以用位点间的整个区域的缺失重新连接dna。实施方案包括,例如,相距1-5兆碱基、或50%-80%染色体、或约100-约1,000,000个碱基对的位点;技术人员会立即领会涵盖明确叙述的范围内的所有范围和数值,例如约1,000至约10,000碱基对或约500至约500,000碱基对。或者,可以将外源dna添加至细胞或胚胎,用于插入外源dna,或对所述位点间的dna进行模板驱动的修复。可以使用多个位点处的修饰来生成经遗传修饰的细胞、胚胎、偶蹄动物、和家畜。实施例6描述了几千个碱基对的dna的缺失,这些末端被重新连接后经生化方法予以确认。
出乎意料地,在分开位点处的talen切割还导致talen靶物间整个区域的频繁倒位。另外令人惊讶的是,如实施例6中详述的,这些倒位以较大的保真性实现。43个测试倒位之41个在5'和3'连接处呈阳性。并且,pcr产物的测序确认了在连接处添加或缺失非常少的核苷酸的的缺失和倒位事件两者(图11,12)。具有这些缺失或倒位的细胞或胚胎作为遗传学的测定工具具有许多用途。
这些细胞也可用于生成动物、家畜、和动物模型。较大的缺失提供了基因失活。还有,例如,可以对细胞、家畜、或动物模型生成缺失品系。将缺失品系与携带突变的生物体杂交,以便与野生型比较,从而快速且方便地定位并鉴定突变基因座。缺失品系是这些领域中公知的,并且牵涉用一系列基因缺失生成的一组生物。缺失作图涉及将具有基因点突变的品系与缺失品系杂交。无论在两种品系间何处发生重组以生成野生型(+)基因,突变不能位于缺失区内。若重组不能生成任何野生型基因,则合理得出结论,点突变和缺失存在于dna的相同区段内。这可以例如用于鉴定成因型突变,或者用于鉴定基于多个性状基因座的多态性。
具有倒位的细胞、胚胎、家畜、偶蹄动物和动物模型也可用于在生物体或动物系或动物品种的后代中固定遗传性状。通常在减数分裂期间在匹配染色体的同源区之间发生重组。染色体区的倒位破坏同源性,并且抑制减数分裂重组。可以使用本文中描述的方法和组合物来生成此类生物体或动物。例如,talen可以在多个基因座处切割牛或猪体细胞中的dna,并且可以将具有倒位的细胞分离,或者可以使用表达报告物的细胞作为可能进行成功倒位的候选物。可以使用细胞来克隆携带不能减数分裂重组的染色体区的动物。或者,预期也会在多个位点处在用多个talen对处理的胚胎中以合理的频率发生倒位。
此方法的一个实施方案是鉴定编码遗传性状的dna区,并用多个talen在所编码性状的位点处每侧切割细胞或胚胎中的dna。可以使用经修饰的细胞或胚胎来创建经遗传修饰的动物。所述方法可以包括分离具有倒位的经遗传修饰的动物。
等位基因移动
一些家畜性状涉及等位基因诸如多态性(或大或小)、单核苷酸多态性、缺失、插入、或其它变异。例如,已知来自belgianblue牛的肌肉生长抑制素(myostatin)等位基因(11-bp缺失)引起双重肌肉化(double-muscling)表型。实施例7显示了使用belgianblue等位基因,如何通过同源性依赖性修复(hdr)将特定等位基因从一个家畜品种精确转移至另一种。牛成纤维细胞接受等位基因,并且可以容易地用于生成转基因牛。此牛不干扰正常的发育,并且本文中教导的方法精确地且在不破坏其它基因或掺入外源基因的情况下放置等位基因。如已经讨论的,本文中呈现的结果显示了家畜成纤维细胞中的等位基因倒位的频率在姐妹染色单体用于hdr模板时是较高的,因此,通常可以预期对一个姐妹染色单体的等位基因渐渗导致纯合性。
本发明的一个实施方案是一种将等位基因从第一家畜系或品种转移至第二家畜系或品种的方法,其包括在存在编码第一家畜系/品种的等位基因的核酸的情况中在第二家畜系/品种的细胞或胚胎中用talen对切割dna。可以使用胚胎或细胞来创建具有第一系/品种的等位基因的第二系/品种的动物。编码等位基因的dna为同源性依赖性修复提供模板。作为模板,它在切割每侧的dna的各部分具有同源性,并且还含有期望的等位基因。本发明的实施方案包括将多个等位基因从一个品种移动至另一品种。如本文中别处列出的,可以将talen以蛋白质或投递由核酸编码,例如mrna或载体编码。也可以将报告物转染入细胞或胚胎中,并且用作选择经talen修饰的细胞的基础。报告物可以非破坏性测定和/或可以包含选择标志物。术语品种意指具有将其与相同物种的其它动物或植物区分的具有同质外观、行为和其它特征的家养动物或植物组。属于特定品种的动物是这些领域中实践的技术人员已知的。
生物体的群体或物种通常在各个个体间每个基因座处包含多个等位基因。基因座处的等位变异以存在的等位基因(多态性)数目,或群体中杂受精卵比例可测量。例如,在人abo血型碳水化合物抗原的基因基因座处,经典的遗传学认可决定输血相容性的三种等位基因,即ia、ib和io。等位基因是意指两种或更多种基因形式之一的术语。
在家畜中,已知许多等位基因与多个性状诸如生产性状、类型性状、可加工性(workability)性状、和其它功能性状有联系。技术人员习惯于监测和量化这些性状,例如visscheretal.,livestockproductionscience,40(1994)123-137,us7,709,206,us2001/0016315,us2011/0023140,和us2005/0153317。因而,转移的等位基因可以与性状有联系或选自下组中的性状:生产性状、类型性状、可加工性性状、生育力性状、育儿(mothering)性状和疾病抗性性状。
在遗传修饰的上下文中术语天然等位基因意指经过修饰的生物体的相同物种中天然存在的等位基因。术语新的等位基因意指非天然等位基因。放入山羊中的人等位基因是新的等位基因。术语合成等位基因意指在自然界中找不到的等位基因。如此,天然等位基因是可以品种间杂交的物种内已经存在的变异。并且,新的等位基因是可以品种间杂交的物种内不存在的等位基因。
通过常规育种方法将等位基因从一种品种移动至另一种涉及在品种间交换许多等位基因。在减数分裂期间的重组不可避免交换品种间的遗传基因座。比较而言,经talen修饰的家畜和其它动物没有减数分裂重组事件。因此,可以容易地将经talen修饰的动物与通过有性繁殖创建的动物区分。
组合物和试剂盒
本发明还提供了组合物和试剂盒,其含有例如编码talen的核酸分子、talen多肽、含有此类核酸分子或多肽的组合物、或talen工程化细胞系、可以使用此类物品,例如作为研究工具,或者治疗性使用。
重组酶
本发明的实施方案包括与重组酶一起施用talen或talen。重组酶与核酸片段形成纤丝,并且实质上搜索细胞dna以寻找与序列基本上同源的dna序列。talen-重组酶实施方案的一个实施方案包括组合重组酶与核酸序列,该核酸序列充当hdr的模板。hdr模板序列与通过talen/talen对靶向切割的位点具有实质性同源性。如本文中描述的,hdr模板通过放置等位基因、创建indel、插入外源dna提供向天然dna的变化,或者提供其它变化。通过本文中描述的方法以蛋白质、mrna、或者通过载体的使用在细胞或胚胎中放置talen。将重组酶与hdr组合以形成纤丝,并放入细胞中。可以将重组酶和/或与重组酶组合的hdr模板以蛋白质、mrna或者用编码重组酶的载体置于细胞或胚胎中。美国公开文本2011/0059160(美国流水号12/869,232)的公开内容在此通过提及并入本文用于所有目的;在冲突的情况中,以说明书为准。术语重组酶指在细胞中酶促催化两个相对较长的dna链间相对较短的dna片的连接的遗传重组酶。重组酶包括cre重组酶、hin重组酶、reca、rad51、cre、和flp。cre重组酶是来自p1噬菌体的i型拓扑异构酶,其催化loxp位点间dna的位点特异性重组。hin重组酶是一种在细菌沙门氏菌(salmonella)中找到的由198个氨基酸组成的21kd蛋白质。hin属于dna转化酶的丝氨酸重组酶家族,其中它依赖于活性位点丝氨酸来启动dna切割和重组。rad51是一种人基因。由此基因编码的蛋白质是rad51蛋白家族的成员,其帮助修复dna双链断裂。rad51家族成员与细菌reca和酵母rad51是同源的。cre重组酶是一种实验用酶,其在实验室测试中已经成功除去由来自受感染细胞的hiv插入的dna。所述酶是为了鉴定hiv标志物经由选择性突变自cre重组酶衍生的,所述hiv标志物不以loxp位点为界,并且因此不容许尝试cre-lox重组。flp指源自面包师用的酵母酿酒酵母(saccharomycescerevisiae)的2μ质粒的翻转酶重组酶(flippaserecombinationenzyme,flp或flp)。
reca以其重组酶活性以在双链断裂修复期间通过同源重组催化链交换而为人所知(mcgrewandknight,2003)radding,etal.,1981;seitzetal.,1998)。还已经显示了reca催化蛋白水解,例如对lexa和λ阻抑物蛋白质的蛋白水解,以及加工dna依赖性atp酶活性。在通过电离辐射或一些其它攻击发生双链断裂后,外切核酸酶以5'至3'咀嚼回到dna末端,由此暴露dna的一条链(cox,1999;mcgrewandknight,2003)。单链dna通过单链结合蛋白(ssb)被稳定化。在结合ssb后,reca结合单链(ss)dna,并且形成螺旋核蛋白纤丝(称为纤丝或联会前纤丝)。在dna修复期间,reca的同源性搜索功能将纤丝引导至同源dna,某并催化同源碱基配对和链交换。这导致形成dna异源双链体。在链侵入后,dna聚合酶基于同源dna模板延伸ssdna以修复dna断裂,并且形成交叉结构或霍利迪连接(hollidayjunction)。reca还显示发动机功能,其参与交叉结构的迁移(campbellanddavis,1999)。
重组酶活性包含许多不同功能。例如,具有重组酶活性的多肽序列能够以非序列特异性方式结合单链dna以形成核蛋白纤丝。此类重组酶结合的核蛋白纤丝能够以非序列特异性方式与双链dna分子相互作用,搜索双链分子中与纤丝中的细菌同源的序列,并且在找到此类序列时,置换双链分子的一条链以容许纤丝中的序列与双链分子的一条链中的互补序列之间的碱基匹配。此类步骤统称为“联会(synapsis)”。
已经对reca和reca样蛋白(在许多真核物种中称作rad51)检查在多种真核系统中刺激基因靶向和同源重组。在烟草细胞中,含有核定位信号(nls)的细菌reca的表达将通过同源重组和体细胞染色体内重组(同源染色体间的重组)的丝裂霉素c诱导的dna损伤修复提高3至10倍(reissetal,1996)。烟草中nlsreca的表达还可以相对于野生型水平将姐妹染色单体替换刺激2.4倍(reissetal.,2000)。在哺乳动物体细胞中,nlsreca的过表达将通过同源重组的基因靶向刺激10倍(shcherbakovaetal.,2000)。然而,在人细胞中,reca的人同源物hrad51的过表达在抗生素选择下相对于野生型水平仅将重组刺激2至3倍(yanezandporter,1999)。在斑马鱼中,通过直接注射ssdna-reca纤丝以低频率校正增强型绿色荧光蛋白(egfp)的突变体形式(cuietal.,2003)。rad52,即rad51上位性组的成员也在使用突变寡核苷酸的斑马鱼中促进单链退火和低水平基因破坏(takahashianddawid,2005)。总之,这些研究指示reca或rad51的异位表达导致同源重组的适度刺激,但是提高的水平不足以可用于基因靶向。
如此,重组酶活性包括但不限于单链dna结合、联会、同源性搜索、单链dna的双链体侵入、异源双链体形成、atp水解和蛋白水解。原型重组酶是来自大肠杆菌(e.coli)的reca蛋白。参见例如美国专利no.4,888,274。原核reca样蛋白也已经记载于沙门氏菌、芽孢杆菌和变形菌(proteus)物种。来自嗜热水生菌(thermitsaquaticus)的热稳定性reca蛋白已经记载于美国专利no.5,510,473。已经描述了reca的噬菌体t4同源物uvsx蛋白。具有改变的重组酶活性的reca突变体已经记载于例如美国专利no.6,774,213;7,176,007和7,294,494。植物reca同源物记载于例如美国专利no.5,674,992;6,388,169和6,809,183。含有重组酶活性的reca片段已经记载于美国专利no.5,731,411。已经描述了具有增强的重组酶活性的突变体reca蛋白诸如例如reca803。参见例如madirajuetal.(1988)proc.natl.acad.sci.usa85:6592-6596。
reca的真核同源物(其液拥有重组酶活性)是第一次在酵母酿酒酵母中鉴定的rad51蛋白。参见bishopetal.,(1992)cell69:439-56及shinoharaetal,(1992)cell:457-70aboussekhraetal,(1992)mol.cell.biol.72,3224-3234.basileetal,(1992)mol.cell.biol.12,3235-3246。植物rad51序列记载于美国专利no.6,541,684;6,720,478;6,905,857和7,034,117。与reca同源的另一种酵母蛋白是dmc1蛋白。已经描述了在与大肠杆菌和酿酒酵母不同的生物体中的reca/rad51同源物。moritaetal.(1993)proc.natl.acad.sci.usa90:6577-6580;shinoharaetal.(1993)naturegenet.4:239-243;heyer(1994)experientia50:223-233;maeshimaetal.(1995)gene160:195-200;美国专利no.6,541,684和6,905,857。
在本文中,“reca”或“reca蛋白”指具有基本上所有或大多数相同功能的reca样重组蛋白家族,所述功能特别是:(i)能将寡核苷酸或多核苷酸正确定位在它们的同源靶物上以便用dna聚合酶进行随后的延伸;(ii)能地制备拓扑的双链核酸以进行dna合成;及(iii)reca/寡核苷酸或reca/多核苷酸复合物能有效寻找并结合互补序列。最佳表征的reca蛋白来自大肠杆菌;在蛋白质的初始等位形式外,已经鉴定出许多突变体reca样蛋白,例如reca803。此外,许多生物体具有reca样链转移蛋白,包括例如酵母、果蝇、哺乳动物,包括人,和植物。这些蛋白质包括例如reel,rec2,rad51,rad51b,rad51c,rad51d,rad51e,xrcc2和dmcl。重组蛋白的一个实施方案是大肠杆菌的reca蛋白。或者,reca蛋白可以是大肠杆菌的突变体reca-803蛋白、来自另一种细菌来源的reca蛋白或来自另一种生物体的同源重组蛋白。
具有重组酶活性的蛋白质的别的描述参见例如fugisawaetal.(1985)niicl.acidsres.13:7473;hsiehetal.(1986)cell44:885;hsiehetal.(1989)j.biol.chem.264:5089;fisheletal.(1988)proc.natl.acad.sci.usa85:3683;cassutoetal.(1987)mol.gen.genet.208:10;ganeaetal.(1987)mo/.cellbiol.7:3124;mooreetal.(1990)jbiol.chem.:11108;keeneetal.(1984)nucl.acidsres.12:3057;kimiec(1984)coldspringharborsymp.48:675;kimeic(1986)cell44:545;kolodnerelal.(1987)proc.natl.acad.sci.usa84:5560;suginoetal.(1985)proc.natl.acad,sci.usa85:3683;flalbrooketal.(1989)jbiol.chem,264:21403;eisenetal.(1988)proc.natl.acad.sci.usa85:7481;mccarthyetal.(1988)proc.natl.acad.sci.usa85:5854;及lowenhauptetal.(1989)jbiol.chem,264:20568,其通过提及并入本文。还可见brendeletal.(1997)j.mol.evol.44:528。
具有重组酶活性的蛋白质的例子包括reca,reca803,uvsx,和其它reca突变体和reca样重组酶(roca(1990)crit.rev.biochem.molec.biol.25:415),(kolodneretal.(1987)proc.natl.acad.sci.u.s.a.84:5560;tishkoffetal.(1991)molec.cell.biol.11:2593),ruvc(dunderdaleetal.(1991)nature354:506),dst2,kem1和xrn1(dykstraetal.(1991)molec.cell.biol.11:2583),stpa/dst1(clarketal.(1991)molec.cell.biol.11:2576),hpp-1(mooreetal.(1991)proc.natl.acad.sci.u.s.a.88:9067),其它真核重组酶(bishopetal.(1992)cell69:439;及shinoharaetal.(1992)cell69:457);其通过提及并入本文。
具有重组酶活性的蛋白质体外演化的已经记载于美国专利no.6,686,515。涉及重组酶的别的出版物包括例如美国专利no.7,732,585;7,361,641和7,144,734。关于重组酶的综述,参见cox(2001)proc.natl.acad,sci,usa98:8173-8180。
可以形成核蛋白纤丝,或纤丝。术语“纤丝(filament)”在与核酸酶形成结构的上下文中是这些领域技术人员已知的术语。然后,可以使如此形成的核蛋白纤丝例如与另一种核酸接触或者导入细胞中。用于形成核蛋白纤丝(其中该纤丝包含具有重组酶活性的多肽序列和核酸)的方法是本领域中公知的。参见例如cuietal.(2003)marinebiotechnol.5:174-184及美国专利nos.4,888,274;5,763,240;5,948,653和7,199,281,其公开内容通过提及并入本文用于披露使重组酶结合核酸以形成核蛋白纤丝的例示性技术。
一般地,使具有重组酶活性的分子与线性单链核酸接触。线性单链核酸可以是探针。制备此类单链核酸的方法是已知的。反应混合物通常含有镁离子。任选地,反应混合物是缓冲的,并且任选地还含有atp、datp或不可水解的atp类似物,诸如例如γ-硫代-atp(atp-γ-s)或γ-硫代-gtp(gtp-γ-s)。任选地,反应混合物还可以含有atp生成系统。可以在纤丝形成之前或期间将双链dna分子变性(例如通过加热或碱进行)。重组酶与核酸的摩尔比率的优化在本领域技术内。例如,可以将重组的不同浓度系列添加至恒定量的核酸,并且通过琼脂糖或丙烯酰胺凝胶中的迁移率测定纤丝形成。由于结合的蛋白质延迟多横扫的电泳迁移率,通过核酸的延迟迁移率证明纤丝形成。可以使用最大程度的延迟,或者最大量的以延迟迁移率迁移的核酸来指示最佳的重组酶:核酸比率。也可以通过测量多核苷酸结合硝酸纤维素的能力来定量蛋白质-dna联合。
talens
如本文中使用的,术语talen是涵盖较宽的,包括能在无另一talen帮助的情况下切割双链dna的单体talen。术语talen也可以指经改造能一起工作以便在同一位点处切割dna的一对talen中的一个或全部两个成员。一起工作的talen可以称为左talen和右talen,其参照dna的手性。
milleretal.(milleretal.(2011)naturebiotechnol29:143)报告了通过将tal截短变体与foki核酸酶的催化域连接,生成talen,用于位点特异性核酸酶构造。显示了所得的talen依靠两种主要的真核dna修复途径,即非同源末端连接(nhej)和同源性指导的修复在永生化人细胞中诱导基因修饰。可以为了特异性结合而工程化改造talen。特异性结合(如该术语在生物学领域中通常使用)指与非靶组织相比以相对较高的亲和力结合靶物的分子,并且一般牵涉多种非共价相互作用,诸如静电相互作用、范德华(vanderwaals)相互作用、氢键键合等等。特异性结合相互作用表征抗体-抗原结合、酶-底物结合、和特异性结合蛋白质-受体相互作用。
已经报告了tal的密码(pct申请wo2011/072246),其中每个dna结合重复单元负责识别靶dna序列中的一个碱基对。这些残基经过装配可以靶向dna序列,其中:(a)hd用于识别c/g;(b)ni用于识别a/t;(c)ng用于识别t/a;(d)ns用于识别c/g或a/t或t/a或g/c;(e)nn用于30识别g/c或a/t;(f)ig用于识别t/a;(g)n用于识别c/g;(h)hg用于识别c/g或t/a;(i)h用于识别t/a;及(j)nk用于识别g/c。简言之,talen结合的靶位点是确定的,并且创建了融合分子,所述融合分子包含核酸酶和一系列识别靶位点的rvd。在结合后,核酸酶切割dna,从而细胞修复机制可以运行以在切割末端产生遗传修饰。术语talen意指包含转录激活物样(tal)效应器结合域和核酸酶域的蛋白质,并且包括本身功能性的单体talen及需要与另一单体talen二聚化的其它talen。二聚化可以在两个单体talen相同时生成同二聚体talen或者可以在单体talen不同时生成异二聚体talen。
在一些实施方案中,可以使用单体talen。talen通常作为具有间隔臂的跨两部分识别位点的二聚体来发挥功能,使得两个tal效应域各自与foki限制酶的催化域融合,所得每个talen的dna识别位点被间隔臂序列分开,每个talen单体对该识别位点的结合容许foki二聚化,并且在该间隔臂内创建双链断裂。然而,也可以构建单体talen,使得单一tal效应器与不需要二聚化来发挥功能的核酸酶融合。例如,一种此类核酸酶是foki的单链变体,其中以单一多肽表达两个单体。其它天然存在的或工程化改造的单体核酸酶也可以满足此作用。用于单体talen的dna识别域可以源自天然存在的tal效应器。或者,可以将dna识别域工程化改造成识别特定dna靶物。工程化的单链talen可以更容易构建并展开,因为它们仅需要一个工程化的dna识别域。可以使用两种不同dna结合域(例如,一个tal效应器结合域和一个来自另一类分子的结合域)生成二聚体dna序列特异性核酸酶。talen可以作为具有间隔臂的跨两部分识别位点的二聚体发挥功能。此核酸酶构造也可以用于靶物特异性核酸酶,其例如自一个talen单体和一个锌指核酸酶单体生成。在此类情况中,talen和锌指核酸酶单体的dna识别位点可以被合适长度的间隔臂分开。两个单体的结合可以容许foki二聚化,在该间隔臂序列内创建双链断裂。与锌指不同的dna结合域,诸如同源结构域(homeodomain)、myb重复单元或亮氨酸拉链也可以与foki融合,并且充当talen单体内的配偶体以创建功能性核酸酶。
在一些实施方案中,可以使用tal效应器将其它蛋白质域(例如非核酸酶蛋白质域)靶向至特定的核苷酸序列。例如,tal效应器可以与蛋白质域连接,所述蛋白质域来自但不限于dna20相互作用酶(例如,甲基化酶、拓扑异构酶、整合酶、转座酶、或连接酶)、转录激活物或阻抑物、或与其它蛋白质诸如组氨酸相互作用或者修饰其它蛋白质诸如组氨酸的蛋白质。此类tal效应器融合物的应用包括例如创建或修饰外遗传(epigenetic)调节元件,生成位点特异性插入、缺失、或dna中的修复,控制基因表达,并修饰染色质结构。
可以选择或改变靶序列的间隔臂以调控talen特异性和活性。间隔臂长度的柔性指示间隔臂长度可以选择为以高特异性靶向特定序列。此外,已经对不同间隔臂长度观察到活性的变化,指示间隔臂长度可以选择为实现期望水平的talen活性。
术语核酸酶包括外切核酸酶和内切核酸酶。术语内切核酸酶指能够水解(切割)在dna或rna分子,优选dna分子内的核酸间的键的任何野生型或变体酶。内切核酸酶的非限制性例子包括ii型限制性内切核酸酶诸如foki,hhai,hindlii,noti,bbvcl,ecori,bglii和alwi。内切核酸酶还包括罕见的切割内切核酸酶,其通常具有长度约12-45个碱基对(bp),更优选14-45bp的多核苷酸识别位点。罕见的切割内切核酸酶在限定的基因座处诱导dna双链断裂(dsb)。罕见的切割内切核酸酶可以例如是归巢内切核酸酶、源自工程化锌指域与限制酶诸如foki催化域融合的嵌合锌指核酸酶(zfn)或化学内切核酸酶。在化学内切核酸酶中,将化学或肽切割剂与核酸聚合物或与识别特定靶序列的另一dna缀合,由此将切割活性靶向至特定序列。化学内切核酸酶还涵盖合成的核酸酶如邻二氮菲的缀合物、dna切割分子、和三联体形成寡核苷酸(tfo),已知其结合特定的dna序列。依照本发明,术语内切核酸酶中包括此类化学内切核酸酶。此类内切核酸酶的例子包括i-seei,i-chuli-crei,i-csmi,pi-seelpi-ttilpi-mtui,i-ceui,i-seeil1-seeiii,ho,pi-civi,pi-ctrlpi-aaei,pi-bsui,pi-dhai,pi-dralpi-mavlpi-mehi,pi-mfulpi-mfli,pi-mgalpi-mgoi,pi-minlpi-mkalpi-mlepi-mmai,pi-30mshlpi-msmi,pi-mthi,pi-mtui,pi-mxei,pi-npui,pi-pfulpi-rmai,pi-spbi,pi-ssplpi-faelpi-mjai,pi-pholpi-taglpi-thyi,pi-tkopi-tspi-msoi。
经遗传修饰的动物
可以使用本领域中已知的多种技术来将核酸构建体导入非人动物中以生成建立者系,其中核酸构建体整合入基因组中。此类技术包括但不限于原核(pronuclear)显微注射(美国专利no.4,873,191)、逆转录病毒介导的到种系的基因转移(vanderputtenetal.(1985)proc.natl.acad.sci.usa82,6148-1652)、对胚胎干细胞的基因靶向(thompsonetal.(1989)cell56,313-321)、胚胎的电穿孔(lo(1983)mol.cell.biol.3,1803-1814)、精子介导的基因转移(lavitranoetal.(2002)proc.natl.acad.sci.usa99,14230-14235;lavitranoetal.(2006)reprod.fert.develop.18,19-23)、及体细胞诸如卵丘细胞或乳房细胞、或成年、胎儿、或胚胎干细胞的体外转化,接着进行核移植(wilmutetal.(1997)nature385,810-813;及wakayamaetal.(1998)nature394,369-374)。原核显微注射、精子介导的基因转移、和体细胞核转移是特别有用的技术。
通常,在原核显微注射中,将核酸构建体导入受精卵中;使用1或2个细胞受精卵作为含有来自精子头的遗传物质的原核,并且卵在原生质内是可见的。可以在体外或在体内(即,从供体动物输卵管用手术回收)获得原核阶段的受精卵。可以如下生成体外受精卵。例如,可以在屠宰场收集猪卵巢,并在运输期间于22-28℃维持。可以将卵巢清洗,并分离,用于卵泡吸出,并且可以使用18号针且在真空下将范围为4-8mm的卵泡吸入50ml圆锥离心管中。将卵泡液和吸出的卵母细胞用商业tl-hepes(minitube,verona,wi)漂洗通过预滤器。可以将以紧密的卵丘块(cumulusmass)围绕的卵母细胞选择,并放入补充有0.1mg/ml半胱氨酸,10ng/ml表皮生长因子,10%猪卵泡液、50μm2-巯基乙醇、0.5mg/mlcamp、10iu/ml每种孕马血清促性腺激素(pregnantmareserumgonadotropin,pmsg)和人绒毛膜促性腺激素(hcg)的tcm-199卵母细胞成熟培养基(minitube,verona,wi)中,在湿润的空气中于38.7℃和5%co2持续约22小时。随后,可以将卵母细胞移至新鲜的tcm-199成熟培养基,其不会含有camp、pmsg或hcg,并且再温育22小时。可以通过在0.1%透明质酸酶中涡旋振荡1分钟对成熟的卵母细胞剥离其卵丘细胞。
对于猪,可以在minitube5孔受精盘中在500μlminitubeporcproivf培养基系统(minitube,verona,wi)中使成熟的卵母细胞受精。在体外受精(ivf)的制备中,可以将新鲜收集的或冷冻的公猪精子清洗,并在porcproivf培养基中重悬至4x105个精子。可以通过计算机辅助精子分析(spermvision,minitube,verona,wi)分析精子浓度。可以在10μl体积中以终浓度约40个运动型精子/卵母细胞(这取决于公猪)实施最终的体外授精。于38.7℃在5.0%co2气氛中将所有受精的卵母细胞温育6小时。授精后6小时,将假定的受精卵在ncsu-23中清洗两次,并移至0.5ml相同培养基。此系统可以在大多数公猪间以10-30%多精入卵授精率常规生成20-30%囊胚。
可以将线性化核酸构建体注射入原核之一中。然后,可以将注射的卵转移至接受体雌性(例如,转移至接受体雌性的输卵管中),并且容许在接受体雌性中发育以生成转基因动物。具体地,将体外受精的胚胎以15,000xg离心5分钟以沉淀脂质,这容许显现原核。可以使用eppendorffemtojet注射器注射胚胎,并且可以将其培养,直到囊胚形成。可以记录胚胎分裂率和囊胚形成和质量。
可以将胚胎用手术转移至异步(asynchronous)接受体的子宫中。通常,可以使用5.5英寸
在体细胞核转移中,可以将包含上文描述的核酸构建体的转基因偶蹄动物细胞(例如转基因猪细胞或牛细胞)诸如胚胎分裂球、胎儿成纤维细胞、成年耳成纤维细胞、或颗粒细胞导入去核的卵母细胞中以建立组合细胞。可以如下将卵母细胞去核,即在极体附近的部分带切开(partialzonadissection),然后在切开区处压出细胞质。通常,使用具有锋利的有斜面的尖端的注射移液管来将转基因细胞注射到阻滞于减数分裂2的去核卵母细胞中。在一些实施方案中,阻滞于减数分裂2的卵母细胞称作“卵”。在生成猪或牛胚胎(例如通过融合并活化卵母细胞进行)后,在活化后约20至24小时,将胚胎转移至接受体雌性的输卵管。参见例如cibellietal.(1998)science280,1256-1258及美国专利no.6,548,741。对于猪,可以在转移胚胎后20-21天约对接受体雌性检查妊娠。
可以使用标准的育种技术来创建在来自初始杂合建立者动物的靶核酸方面纯合的动物。然而,可以不需要纯合性。可以将本文中描述的转基因猪与感兴趣的其它猪育种。
在一些实施方案中,可以以不相等的量在分开的转座子上提供并且对胚胎或细胞提供感兴趣的核酸和选择标志物,其中含有选择标志物的转座子的量远远超过(5-10倍过量)含有感兴趣核酸的转座子。可以基于选择标志物的存在和表达分离表达感兴趣核酸的转基因细胞或动物。由于转座子会以精确的且不相联的方式(独立转座事件)整合入基因组中,感兴趣的核酸和选择标志物不是遗传连锁的,并且可以容易地经由标准育种通过遗传分离分开。如此,可以生成转基因动物,其不受在后续世代中保留选择标志物(即一个从公共安全性观点看有些顾虑的问题)约束。
一旦生成了转基因动物,可以使用标准技术评估靶核酸的表达。可以通过southern印迹分析实现初始筛选以测定构建体整合发生与否。关于southern分析的描述,参见sambrooketal,1989,molecularcloning,alaboratorymanualsecondedition,coldspringharborpress,plainview;ny的部分9.37-9.52。也可以在初始筛选中使用聚合酶链式反应(pcr)技术。pcr指扩增靶核酸的规程或技术。一般地,采用来自感兴趣区域末端或超出的序列信息来设计与要扩增的模板的相反链在序列上相同或相似的寡核苷酸引物。可以使用pcr从dna及rna扩增特定序列,包括来自总基因组dna或总细胞rna的序列。引物的长度通常是14至40个核苷酸,但是长度范围可以是10个核苷酸至几百个核苷酸。pcr记载于例如pcrprimer:alaboratorymanual,ed.dieffenbachanddveksler,coldspringharborlaboratorypress,1995。也可以通过连接酶链式反应、链置换扩增、自身维持的序列复制、或基于核酸序列的扩增来扩增核酸。见例如lewis(1992)geneticengineeringnews12,1;guatellietal.(1990)proc.natl.acad.sci.usa87:1874;andweiss(1991)science254:1292。在囊胚阶段,可以将胚胎单个地加工以通过pcr、southern杂交和splinkerettepcr分析(参见例如dupuyetal.procnatlacadsciusa(2002)99:4495)。
可以使用技术评估转基因猪的组织中编码多肽的核酸序列表达,所述技术包括例如对自动物获得的组织样品的northern印迹分析、原位杂交分析、western分析、免疫测定法诸如酶联免疫吸附测定法、和逆转录酶pcr(rt-pcr)。
载体和核酸
可以将多种核酸导入偶蹄动物或其它细胞中(出于敲除目的)或者以为了其它目的获得基因表达。可以用于生成转基因动物的核酸构建体包括靶核酸序列。如本文中使用的,术语核酸包括dna、rna、和核酸类似物,以及双链或单链(即有义或反义单链)的核酸。可以在碱基模块、糖模块、或磷酸模块处修饰核酸类似物以改善例如核酸的稳定性、水解或溶解度。碱基模块处的修饰包括脱氧尿苷(对于脱氧胸苷)、和5-甲基-2'-脱氧胞苷和5-溴代-2'-脱氧胞苷(对于脱氧胞苷)。糖模块的修饰包括核糖糖的2'羟基的修饰以形成2'-o-甲基或2'-o-烯丙基糖。可以将脱氧核糖磷酸主链修饰以生成吗啉代核酸(其中每个碱基模块与六个成员的吗啉代环连接)或肽核酸(其中脱氧磷酸酯主链用假肽(pseudopeptide)主链替换,并且保留四个碱基)。参见summertonandweller(1997)antisensenucleicaciddrugdev.7(3):187;及hyrupetal.(1996)bioorgan,med.chem.4:5。另外,可以用例如硫代磷酸酯或二硫代磷酸酯主链、亚磷酰胺或烷基磷酸三酯主链替换脱氧磷酸酯主链。
靶核酸序列可以与调节区诸如启动子可操作连接。调节区可以是猪调节区或者可以来自其它物种。如本文中使用的,可操作连接指以如下的方式相对于核酸序列定位调节区,使得容许或促进靶核酸的转录。
任何类型的启动子可以与靶核酸序列可操作连接。启动子的例子包括但不限于组织特异性启动子、组成性启动子、和响应或不响应特定刺激物的启动子。合适的组织启动子可以导致在beta细胞中核酸转录物的优先表达,并且包括例如人胰岛素启动子。其它组织特异性启动子可以导致在例如干细胞或心脏组织中优先表达,并且分别可以包括清蛋白或alpha-肌球蛋白重链启动子。在其它实施方案中,可以使用不以显著组织或时间特异性促进核酸分子表达的启动子(即,组成性启动子)。例如,可以使用beta-肌动蛋白启动子诸如鸡beta-肌动蛋白基因启动子、泛素启动子、minicag启动子、甘油醛-3-磷酸脱氢酶(gapdh)启动子、或3-磷酸甘油酸激酶(pgk)启动子,以及病毒启动子诸如单纯疱疹病毒胸苷激酶(hsv-tk)启动子、sv40启动子、或巨细胞病毒(cmv)启动子。在一些实施方案中,使用鸡beta肌动蛋白基因启动子和cmv增强子的融合作为启动子。参见例如xuetal.(2001)hum.genether.12:563;及kiwakietal.(1996)hum.genether.7:821。
诱导型启动子的例子是四环素(tet)开启启动子系统,其可以用于调节核酸的转录。在此系统中,突变的tet阻抑物(tetr)与单纯疱疹病毒vp16反式激活物蛋白的激活域融合以创建四环素控制的转录激活物(tta),其受到tet或多西环素(dox)调节。在缺乏抗生素的情况中,转录是最小程度的,而在存在tet或dox的情况中,转录得到诱导。备选的诱导型系统包括蜕皮激素或雷怕霉素(rapamycin)系统。蜕皮激素是一种昆虫蜕皮激素,其生成受到蜕皮激素受体和超气门蛋白(ultraspiracle)基因(usp)产物的异二聚体控制。通过用蜕皮激素或蜕皮激素类似物诸如muristeronea处理诱导表达。对动物施用以触发诱导型系统的药剂称为诱导剂。
可以用于核酸构建体的别的调节区包括但不限于多腺苷酸化序列、翻译控制序列(例如,内部核糖体进入区段,ires)、增强子、可诱导元件、或内含子。此类调节区可以不是必要的,尽管它们可以通过影响转录、mrna的稳定性、转录效率等等提高表达。可以在想要时在核酸构建体中纳入此类调节区以在细胞中获得核酸的最佳表达。然而,足够的表达有时可以在没有此类别的元件的情况下获得。
可以使用编码信号肽或选择标志物的核酸构建体。可以使用信号肽,使得将编码的多肽引导至特定的细胞位置(例如,细胞表面)。选择标志物的非限制性例子包括嘌呤霉素、更昔洛韦、腺苷脱氨酶(ada)、氨基葡糖苷磷酸转移酶(neo,g418,aph)、二氢叶酸还原酶(dhfr)、潮霉素-b-磷酸转移酶、胸苷激酶(tk)、和叶黄素-鸟嘌呤磷酸核糖转移酶(xgprt)。此类标志物可用于选择培养物中的稳定转化体。其它选择标志物包括荧光多肽,诸如绿色荧光蛋白或黄色荧光蛋白。
在一些实施方案中,编码选择标志物的序列侧翼可以有重组酶诸如例如cre或flp的识别序列。例如,选择标志物侧翼可以有loxp识别位点(由cre重组酶识别的34-bp识别位点)或frt识别位点,使得可以从构建体切除选择标志物。见orban,etal,proc.natl.acad.sci.(1992)89:6861(关于cre/lox技术的综述),及brandanddymecki,dev.cell(2004)6:7。也可以使用含有以选择标志物基因中断的cre或flp可激活转基因的转座子来获得具有转基因的条件表达的转基因动物。例如,驱动标志物/转基因表达的启动子可以遍在的或组织特异性的,其会导致标志物在f0动物(例如猪)中的遍在或组织特异性表达。例如,可以如下实现转基因的组织特异性活化,即将遍在表达标志物中断转基因的猪与以组织特异性方式表达cre或flp的猪杂交,或者将以组织特异性方式表达标志物中断转基因的猪与遍在表达cre或flp重组酶的猪杂交。转基因的受控表达或标志物的受控表达容许转基因的表达。
在一些实施方案中,靶核酸编码多肽。编码多肽的核酸序列可以包含编码设计用于促进编码多肽的随后操作(例如以促进定位或检测)的标签的标签序列。可以在编码多肽的核酸序列中插入标签序列,使得编码的标签位于多肽的羧基或氨基端。编码标签的非限制性例子包括谷胱甘肽s-转移酶(gst)和flagtm标签(kodak,newhaven,ct)。
在其它实施方案中,靶核酸序列诱导针对靶核酸的rna干扰,从而诱导靶核酸的表达。例如,靶核酸序列可以诱导针对编码囊性纤维化跨膜传导调节物(cftr)多肽的核酸的rna干扰。例如,与cftrdna同源的双链小干扰rna(sirna)或小发夹rna(shrna)可以用于诱导所述dna的表达。可以生成用于sirna的构建体,如记载于例如fireetal.(1998)nature391:806;romanoandmasino(1992)mol.microbiol.6:3343;cogonietal.(1996)emboj.15:3153;cogoniandmasino(1999)nature399:166;misquittaandpaterson(1999)proc.natl.acad.sci.usa96:1451;及kennerdellandcarthew(1998)cell95:1017。可以生成用于shrna的构建体,如由mclntyreandfanning(2006)bmcbiotechnology6:1描述的。一般地,shrna转录为含有互补区的单链rna分子,其可以退火并形成短的发夹。
可以使用sssicpg甲基化酶(newenglandbiolabs,ipswich,ma)使核酸构建体甲基化。一般地,可以将核酸构建体与缓冲液中的s-腺苷甲硫氨酸和sssicpg甲基化酶于37℃一起温育。可以通过将构建体与一个单位的hinp1i内切核酸酶于37℃一起温育1小时,并通过琼脂糖凝胶电泳测定来确认超甲基化。
可以使用多种技术将核酸构建体导入任何类型的胚胎、胎儿、或成年偶蹄动物细胞,包括例如生殖细胞诸如卵母细胞或卵、祖细胞、成年或胚胎干细胞、原始生殖细胞、肾细胞诸如pk-15细胞、胰岛细胞、beta细胞、肝细胞、或成纤维细胞诸如皮肤成纤维细胞中。技术的非限制性例子包括使用转座子系统、可以感染细胞的重组病毒、或者脂质体或其它非病毒方法诸如电穿孔、显微注射、或磷酸钙沉淀,其能够将核酸投递至细胞。
在转座子系统中,核酸构建体的转录单元,即可操作连接的调节区和靶核酸序列侧翼有转座子的反向重复。已经开发出几种转座子系统,包括例如sleepingbeauty(见美国专利no.6,613,752和美国公开文本no.2005/0003542);frogprince(miskeyetal.(2003)nucleicacidsres.31:6873);toll(kawakami(2007)genomebiology8(suppl.l):s7;minos(pavlopoulosetal.(2007)genomebiology8(suppl.l):s2);hsmar1(miskeyetal.(2007))molcellbiol.27:4589);和passport以将核酸导入细胞,包括小鼠、人和猪细胞中。sleepingbeauty转座子是特别有用的。转座子可以以蛋白质投递,在与靶核酸相同的核酸构建体上编码,可以在分开的核酸构建体上引入,或者以mrna(例如,在体外转录的且加帽的mrna)提供。
也可以在核酸构建体中纳入绝缘子元件以维持靶核酸的表达以及抑制宿主基因的不想要转录。参见例如美国公开文本no.2004/0203158。通常,绝缘子元件在转录单元的每侧侧翼,并且在转座子的反向重复内部。绝缘子元件的非限制性例子包括基质附着元件(mar)型绝缘子元件和边界型绝缘子元件。参见例如美国专利no.6,395,549;5,731,178;6,100,448;和5,610,053,及美国公开文本no.2004/0203158。
可以将核酸引入载体中。载体是一个涵盖较宽的术语,其包含设计用于从载体移入靶dna中的任何特定dna段。载体可以称为表达载体,或载体系统,其是引起dna插入基因组或其它靶向dna序列诸如中需要的组件集,诸如附加体、质粒、或甚至病毒/噬菌体dna段。用于在动物中投递基因的载体系统诸如病毒载体(例如,逆转录病毒、腺伴随病毒和整合噬菌体病毒)、和非病毒载体(例如转座子)具有两种基本元件:1)由dna(或逆转录成cdna的rna)组成的载体和2)转座酶、重组酶、或其它整合酶酶,其识别载体和dna靶序列两者,并且将载体插入靶dna序列中。载体最常含有一种或多种表达盒,其包含一种或多种表达控制序列,其中表达控制序列是分别控制和调节另一dna序列或mrna的转录和/或翻译的dna序列。
许多不同类型的载体是已知的。例如质粒和病毒载体,例如逆转录病毒载体是已知的。哺乳动物表达载体通常具有复制起点、合适的启动子和任选的增强子,而且还有任何必要的核糖体结合位点、多聚腺苷酸化位点、剪接供体和接受位点、转录终止序列、和5'侧翼非转录序列。载体的例子包括:质粒(其也可以是另一类载体的载体)、腺病毒、腺伴随病毒(aav)、慢病毒(例如,经修饰的hiv-1,siv或fiv)、逆转录病毒(例如asv,alv或momlv)、和转座子(例如sleepingbeauty,p元件,tol-2,frogprince,piggybac)。
如本文中使用的,术语核酸指rna和dna两者,包括例如cdna、基因组dna、合成的(例如化学合成的)dna、及天然存在的和经化学修饰的核酸,例如合成的碱基或备选主链。核酸分子可以是双链或单链(即,有义或反义单链)。术语转基因在本文中广泛使用,指经遗传修饰的生物体或经遗传工程化改造的生物体,其遗传物质已经使用遗传工程化技术改造。如此,无论外源基因或核酸是否在动物或其后代中表达,敲除偶蹄动物是转基因的。
实施例1:通过直接注射talen生成经遗传修饰的偶蹄动物家畜(牛):
设计并装配三种talen对,如记载于cermaket.al.(2011)nuc.acidsres.39:e82(图3)的,并将mrna注射入受精后约19小时的牛胚胎的细胞质中。将注射过的胚胎在体外培养,并囊胚阶段时收集(图3)。将各个囊胚基因组dna通过全基因组扩增(wga)予以扩增(carlsonetal.(2011)trangenicres.20:29),并使用surveyor核酸酶测定法筛选indel(图3,4a,和4b)。在10%注射过的胚胎中观察到指示talen活性的裂解产物(例如,图3,4a,和4b)。在2个用acan11或acan12talen对注射的囊胚中确认预测区中的各个突变(图3)。没有观察到经talen注射的胚胎的发育能力的显著降低。然后,以范围为10-100ng/μl的mrna剂量使用acan12talen对实施第二轮注射。轮次1(33%)和2(5%)(10ng/μl条件)之间囊胚形成率的比较揭示较差的胚胎质量(表s2)。尽管胚胎质量较差,使用surveyor测定法鉴定出12个推定的突变体(占注射的27%)。用来自克隆pcr产物的14次测序读数分析每个surveyor阳性胚胎的基因型。测序揭示基因修饰中的镶嵌性(mosaicism)。在4个surveyor阳性胚胎中鉴定出多个indel,并且这些之中,indel阳性序列读数占每个胚胎总读数的7-29%。这些结果证明talen在偶蹄动物胚胎中发挥功能。已经对模式生物体报告了用锌指核酸酶(zfn)和talen进行的胚胎基因修饰,其通过直接注射编码核酸酶对的zfn或talenmrna进行geurtsetal.(2009)science325:433;carberyetal,(2010)genetics186:451;mashimoetal.(2010)plosone5:e8870;tessonetal.(2011)naturebiotechnol.29:695。
实施例2:通过直接注射talen生成的经遗传修饰的偶蹄动物家畜(猪)。
还使用靶向猪rela基因(p65)的talens在猪ivp胚胎中进行单组注射,对此已经提出针对非洲猪霍乱的耐受性等位基因(palgraveetal.(2011)jvirol.85:6008)。两个小组报告了用天然tale支架的截短变体实现的talen活性增强(mussolinoetal.(2011)nucleicacidsres.39:9283;milleretal.(2011)naturebiotechnol.29:143{mussolino,2011#151;miller,2011#42}。因此,与使用+231支架(bamhi片段,christianet.al.(2010)genetics186:757)的上述实验形成对比,选择p65talen支架进行截短以模拟milleret.al.2011(同上)(还可见图5)中描述的+63型式。给受精卵注射包含左侧和右talen各10ng/μl以及5ng/μlegfpmrna(作为成功注射的指标)的mrna混合物。鉴定出75个egfp阳性受精卵(214个接受注射者中,35%),并对其进行wgaindel分析。从56个egfp阳性胚胎成功进行pcr扩增,这些中的16个(29%)通过surveyor(图3)或序列分析揭示indel。与牛相比,镶嵌性在猪受精卵中看来是降低的。实际上,三分之一的突变体(6/16)是纯合或杂合双等位突变体(图4)。这些结果证明talen在偶蹄动物胚胎中发挥功能。基于经修饰的胚胎创建动物建立者系的方法是公知的。
实施例3:通过牛和猪体细胞的遗传修饰生成的经遗传修饰的偶蹄动物家畜。
针对基于生物医学或农业相关性选择的猪和牛中的靶物装配几种别的talen对。在两种先前描述的talen支架(+231,christianet.al.2010(同上)和+63,milleret.al.2011(同上))的背景中放置六种talen对的结合域(图5)。将每个talen对转染到原代家畜成纤维细胞中,并通过surveyor测定法(guschin,etal.(2010)methodsmol.biol.649:247)在第3天测量基因组修饰。最具活性的talen对dmde7.1和acan12分别展现出对38%和25%染色体的切割,并且sanger测序揭示nhej介导的dna修复特征性的indel的分类(图5和图6)。talen支架对成纤维细胞中的活性具有显著影响。总共,用+63同工型靶向的6个基因座中的4个以3.5%或更高水平切割,而仅dmde7.1talen对在+231支架中以高于1%的水平切割(图5)。如先前研究中记录的doyonetal.(2010)naturemethods8:74;miller,(2011)同上),转染后于30℃温育72小时对活性也具有正向影响,并且是3种talen对的活性需要的。生成活性talen对的成功率已经是较高的。直到提交时收集的数据显示,36个中的23个(64%)talen对,在猪和牛基因组中分散的15个基因处,对于常染色体及x和y染色体两者是可检出有活性的(>1.0%nhej)(表s3)。四分之三的活性对以高效率(19-40%)切割,平均修饰水平是25%。基于经修饰的成纤维细胞创建动物建立者系的克隆方法是公知的。
实施例4:进一步培养和通过转座子共转染进行indel富集。
将talen对转染入成纤维细胞中,并且在有或没有转座子共选择的情况下将细胞培养14+天,之后测量修饰水平。图7中汇总了结果。在第0天(d0),用质粒混合物转染细胞,所述质粒混合物包括每种talen的表达盒(两种质粒)、编码选择标志物的转座子(第三质粒,编码嘌呤霉素)、和转座酶表达盒(第四质粒)。这些talen质粒都是每次转染的主要元件(4倍质量过量)。将经转染的细胞于30或37摄氏度培养3天,之后分开,收集样品以进行surveyor测定法,并且再铺板以进一步培养+/-选择转座子整合。在第3天后将所有细胞于37摄氏度培养。收集培养14+天的细胞以进行surveyor测定法,并且冷冻保存以进行后续应用,即scnt。为了比较,通过核转染来转染其它成纤维细胞,并在第3天,以及在第14+天非选择的(ns)和选择的(s)群体中测量百分比nhej。为了比较,还使用阳离子脂质转染成纤维细胞。
实施例5:单和双等位ko克隆的分离。
可以将转基因原代成纤维细胞有效扩增,并与非转基因成纤维细胞(饲养细胞)一起以标准密度(大于150个细胞/cm2)铺板,用上文应用的转座子共选择技术进行药物选择(carlsonetal.(2011)transgenicres.20:1125),从而分离出集落。为了评估此方法,将用六种talen对处理的细胞的嘌呤霉素抗性集落分离,并通过surveyor测定法或跨越靶位点的pcr产物的直接测序评估其基因型(图8a和8b)。总体上,indel阳性克隆的比例与基于第3天修饰水平做出的预测相似。对6种talen对之5种鉴定出双等位敲除克隆,在多至35%的indel阳性细胞中发生(表1)。在大多数例子中(23之15),indel是纯合的(每个等位基因上的indel相同),而不是每个等位基因上有独特的indel,这提示了姐妹染色单体为模板的修复是常见的(图9)。值得注意的是,在经修饰的克隆中,若染色体切割以独立事件处理,则大多数talen对的双等位修饰的频率(17-35%)超出基于第3天修饰的预测(10-15.6%)。用zfn进行的先前研究(kimetal.(2009)genomeres.19:1279;leeetal.(2010)genomeres.20:81;perezetal.(2008)naturebiotechnol.26:808)支持这些结果。这些先前的研究提示在几乎三分之一的经修饰的细胞中发生双等位修饰,其不依赖于核酸酶活性水平。
实施例6:用talen进行的染色体缺失和倒位。
据推测,靶向相同染色体的两种talen对的同时投递可以诱导较大的染色体缺失或倒位。测试了talen对dmde6和dmde7.1,因为它们有较高的活性,还因为高百分比duchene氏肌营养不良由总体缺失引起的事实(blake,2002),使得猪模型会匹配人的状况。图10中汇总了结果。第3天基因修饰水平对于每个talen对是较高的(对于dmde6为24%,而对于dmde7.1为23%),尽管在个别转染任一talen对时略低(图10)。为了测定是否已经删除两个talen对间的序列,用跨越talen靶位点的引物尝试pcr。若已经除去6.5kb序列,则预期约500bp的条带,而用选择的pcr条件不会扩增7kb的野生型条带。一式两份观察到接近500bp的条带,其中两个talen对均被引入,但是在单独引入任一talen对时是缺乏的(图10)。
接着,通过跨假定的新5'和3'连接的pcr对细胞群体测定倒位事件。仅在引入两种talen对时对假定倒位的5'和3'连接两者观察到预期大小的产物(图10)。为了进一步确认倒位,使用转座子共选择策略生成集落,并对其筛选缺失和倒位事件。以高频率获得(recovered)缺失和倒位事件两者(分别是10.3%和4.1%;n>1000)(表s4)。以显著的保真性发生缺失和倒位事件。43倒位阳性集落中的41个在5'和3'连接处呈阳性。最后,对pcr产物的测序确认了缺失和倒位事件两者,在它们的连接处添加或缺失了非常少的核苷酸(图11,12)。
实施例7:talen诱导的同源重组消除了对关联的选择标志物的需要。
将来自belgianblue牛的肌肉生长抑制素等位基因突变(缺失11bp)放入野生型wagyu牛的基因组(grobetetal.(1997)naturegenet.17:71)中(图13)。单独转染时,btgdf8.1talen对在靶基因座处切割多至16%的染色体(图13)。talen(btgdf83.1)和dsdna模板(bb-hdr)设计为通过dsb诱导的同源重组在牛gdf8(belgiumblue突变)的外显子3中引入11bp缺失。左talen的结合位点的一半在bb-hdr模板中缺失,使其对talen切割有抗性。surveyor测定法于37和30摄氏度证明了btgdf83.1talens的活性。使用引物b和b'(图13的小图a中显示)生成用于此测定法的pcr产物。这些重复中不包括bb-hdr模板,因为它会混淆对btgdf83.1活性的评估。等位基因特异性pcr证明,hdr诱导依赖于talen和bb-hdr模板的共转染。开发了pcr测定法以使用引物c和c'(图13的小图a中显示)来特异性检测经hdr修饰的gdf8等位基因。引物c'的3'端跨越所述11-bp缺失,并且不能扩增野生型等位基因“wt”。包含阳性对照“c”的每个pcr反应中包含500个细胞等同物。通过实验反应和对照反应之间的比较密度计量学测定百分比hdr。与含有belgianblue牛1623bpdna片段的超螺旋dna模板共转染,在没有选择期望的事件的情况下,产生多至5%的基因转化频率(hdr),如在第3天通过半定量pcr提示的(图13)。这些结果证明,可以使用talens在家畜中有效放置外源核酸序列,包括等位基因-且不含标志物。为了评估各个集落中放置的频率,实施转座子共选择策略以分离并扩充各个集落,用于dna测序。在经pcr检查的366个中的5个集落中,检出了用来自belgianblue牛的模板进行的基因倒位。用belgianbluehdr模板以外的引物进行的扩增,以及测序,都确认了4个集落中预期的11bp缺失的存在(图14)。
实施例8:talen介导的dna切割作为家畜细胞中hdr的靶物。
将靶向猪低密度脂蛋白受体(ldlr)基因的第四个外显子的talen对(ldlr2.1)与超螺旋质粒ldlr-e4n-终止子(其含有与猪ldlr基因对应的同源臂和能通过hdr表达新霉素磷酸转移酶的基因陷阱)共转染(图15)。在培养3天后,pcr分析揭示,即使没有抗生素选择,也可以于30℃在靶定基因座处检出与hdr事件对应的条带(第4道)。用遗传霉素(g418)选择培养14天的细胞群体导致hdr细胞的显著富集(第11道和第13道)。通过体细胞核转移克隆此类经修饰的细胞,代孕母猪目前正孕育胚胎。
参考文献
在此通过提及将本文中所列的专利申请、专利、出版物、和期刊文章并入本文用于所有目的;在冲突的情况中,以说明书为准。
m.christian,etal,targetingdnadouble-strandbreakswithtaleffectornucleases,186genetics(2010).
j.c.miller,etal,atalenucleasearchitectureforefficientgenomeediting,29naturebiotech.(2011).
d.a.mcgrew&k.l.knight,moleculardesignandfunctionalorganizationoftherecaprotein,38gritrevbiochemmolbiol(2003).
m.m.cox,recombinationaldnarepairinbacteriaandtherecaprotein,63prognucleicacidresmolbiol(1999).
b.reiss,etal.,recaproteinstimulateshomologousrecombinationinplants,93procnatlacadsciusa(l996).
b.reiss,etal.,recastimulatessisterchromatidexchangeandthejidelityofdouble-strandbreakrepair,butnotgenetargeting,inplantstransformedbyagrobacterium,97procnatlacadsciusa(2000).
o.g.shcherbakova,etal.,overexpressionofbacterialrecaproteinstimulateshomologousrecombinationinsomaticmammaliancells,459mutatres(2000).
r.j.yanez&a.c.porter,genetargetingisenhancedinhumancellsoverexpressinghradsl,6genether(1999).
z.cui,etal.,reca-mediated,targetedmutagenesisinzebrafish,5marbiotechnol(ny)(2003).
n.takahashi&i.b.dawid,characterizationofzebrafishrad52andreplicationproteinaoroligonucleotide-mediatedmutagenesis,33nucleicacidsres(2005).t.cermak,etal.,efficientdesignandassemblyofcustomtalenandothertaleffector-basedconstructsfordnatargeting,(inpress)nucl.acidsres.(2011).d.f.carlson,etal.,strategiesforselectionmarker-freeswinetransgenesisusingthesleepingbeautytransposonsystem,20transgenicres(2011).
a.m.geurts,etal.,knockoutratsviaembryomicroinjectionofzinc-fingernucleases,325science(2009).
i.d.carbery,etal.,targetedgenomemodificationinmiceusingzinc-fingernucleases,186genetics(2010).
t.mashimo,etal.,generationofknockoutratswithx-linkedseverecombinedimmunodeficiency(x-scid)usingzinc-fingernucleases,5plosone(2010).
l.tesson,etal.,knockoutratsgeneratedbyembryomicroinjectionoftalens,29natbiotechnol(2011).
c.j.palgrave,etal.,species-specificvariationinrelaunderliesdifferencesinnf-kappabactivity:apotentialroleinafricanswinefeverpathogenesis,85jvirol(2011).
c.mussolino,etal.,anoveltalenucleasescaffoldenableshighgenomeeditingactivityincombinationwithlowtoxicity,nucleicacidsres(2011).
d.y.guschin,etal,arapidandgeneralassayformonitoringendogenousgenemodification,649methodsmolbiol(2010).
y.doyon,etal,transientcoldshockenhanceszinc-fingernuclease-mediatedgenedisruption,7natmethods(2010).
h.j.klm,etal.,targetedgenomeeditinginhumancellswithzincfingernucleasesconstructedviamodularassembly,19genomeres.(2009).
h.j.lee,etal.,targetedchromosomaldeletionsinhumancellsusingzincfingernucleases,20genomeres(2010).
e.e.perez,etal.,establishmentofhiv-1resistanceincd4+tcellsbygenomeeditingusingzinc-fingernucleases,26natbiotechnol(2008).
d.j.blake,etal.,functionandgeneticsofdystrophinanddystrophin-relatedproteinsinmuscle,82physiolrev(2002).
l.grobet,etal.,adeletiointhebovinemyostatingenecausesthedouble-muscledphenotypeincattle,17natgenet(1997).
r.kambadur,etal.,mutationsinmyostatin(gdf8)indouble-muscledbelgianblueandpiedmontesecattle,7genomeres(1997).
序列表
<110>重组股份有限公司(recombineticsinc.)
<120>经遗传修饰的动物及其生成方法
<130>5054.10us02
<140>13/404,662
<141>2012-02-24
<150>61/446,651
<151>2011-02-25
<160>128
<170>patentinversion3.5
<210>1
<211>71
<212>dna
<213>家牛(bostaurus)
<400>1
ccctcttcctttcctccagggatccctcctacatggcctcccactggagaagcaacagag60
gagcacacaga71
<210>2
<211>71
<212>dna
<213>人工序列
<220>
<223>indel序列1
<400>2
ccctcttcctttcctccagggatccctcctacagggactcccactggagaagcaacagag60
gagcacacaga71
<210>3
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列1
<400>3
ccctcttcctttcctccagggatccctcctacctcccactggagaagcaacagaggagca60
cacaga66
<210>4
<211>71
<212>dna
<213>家牛(bostaurus)
<400>4
ccctcttcctttcctccagggatccctcctacatggcctcccactggagaagcaacagag60
gagcacacaga71
<210>5
<211>59
<212>dna
<213>人工序列
<220>
<223>indel序列4
<400>5
ccttcttcctttcctccagggatccctcccactggagaagcaacagaggagcacacaga59
<210>6
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列4
<400>6
ccctcttcctttcctccagggatccctcctacttcccactggagaagcaacagaggagca60
cacaga66
<210>7
<211>70
<212>dna
<213>猪(sus)
<400>7
gcccccccacacagctgagcccatgctgatggagtaccctgaggctataactcgcttggt60
gacagggtcc70
<210>8
<211>91
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>8
gcccccccacacagctgagccacaagtcgagggtctcagagtcactgagtactgagtgct60
gatggagtaccctgaggctataactcgcttg91
<210>9
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>9
gcccccccacacagctgagccctgatggagtaccctgaggctataactcgcttggtgaca60
gggtcc66
<210>10
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>10
gcccccccacacagctgagccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>11
<211>65
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>11
gcccccccacacagctgagcccaatggagtaccctgaggctataactcgcttggtgacag60
ggtcc65
<210>12
<211>65
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>12
gcccccccacacagctgagcacacaggagtaccctgaggctataactcgcttggtgacag60
ggtcc65
<210>13
<211>70
<212>dna
<213>猪(sus)
<400>13
gcccccccacacagctgagcccatgctgatggagtaccctgaggctataactcgcttggt60
gacagggtcc70
<210>14
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>14
gcccccccacacagctgacccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>15
<211>71
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>15
gcccccccacacagctgagcccattgctgatggagtaccctgaggctataactcgcttgg60
tgacagggtcc71
<210>16
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>16
gcccccccacacagctgagcccagctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>17
<211>67
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>17
gcccccccacacagctgagcccatgatggagtaccctgaggctataactcgcttggtgac60
agggtcc67
<210>18
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>18
gcccccccacacagctgagcccagctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>19
<211>65
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>19
gcccccccacacagctgagcccaatggagtaccctgaggctataactcgcttggtgacag60
ggtcc65
<210>20
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>20
gcccccccacacagctgagccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>21
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>21
gcccccccacacagctgagccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>22
<211>67
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>22
gcccccccacacagctgagcccatgatggagtaccctgaggctataactcgcttggtgac60
agggtcc67
<210>23
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>23
gcccccccacacagctgagcccatgtgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>24
<211>70
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>24
gcccccccacacagctgagccatgcctgatggagtaccctgaggctataactcgcttggt60
gacagggtcc70
<210>25
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>25
gcccccccacacagctgagccctgatggagtaccctgaggctataactcgcttggtgaca60
gggtcc66
<210>26
<211>97
<212>dna
<213>家牛(bostaurus)
<400>26
tcgccttccctcttcctttcctccagggatccctcctacatggcctcccactggagaagc60
aacagaggagcacacagaaggcccttctgcaacggaa97
<210>27
<211>97
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>27
tcgccttccctcttcctttcctccagggatccctcctacatggcttggcctcccactgga60
gaagcaacagaggagcacacagaaggcccttctgcaa97
<210>28
<211>106
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>28
tcgccttccctcttcctttcctccagggatccctcctacatggctcccactggctcccac60
tggagaagcaacagaggagcacacagaaggcccttctgcaacggaa106
<210>29
<211>96
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>29
tcgccttccctcttcctttcctccagggatccctcctacatggctggcctcccactggag60
aagcaacagaggagcacacagaaggcccttctgcaa96
<210>30
<211>92
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>30
tcgccttccctcttcctttcctccagggatccctcctacgcctcccactggagagcaaca60
gaggagcacacagaggcccttctgcaacggaa92
<210>31
<211>91
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>31
tcgccttccctcttcctttcctccagggatccctcctacttcccactggagaagcaacag60
aggagcacacagaggcccttctgcaacggaa91
<210>32
<211>89
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>32
tcgccttccctcttcctttcctccagggatccctcctactcccactggagaagcaacaga60
ggagcacacagaggcccttctgcacggaa89
<210>33
<211>91
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>33
tcgccttccctcttcctttcctccagggatccctcctgctcccactggagaagcaacaga60
ggagcacacagaaggcccttctgcaacggaa91
<210>34
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>34
tcaccttccttcttcctttcctccagggatcccactggagaagcaacagaggagcacaca60
gaaggcccttctgcacgga79
<210>35
<211>81
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>35
tcgccttccctcttcctttcctccaggcctcccactggagaagcaacagaggagcacaca60
gaaggcccttctgcaacggaa81
<210>36
<211>71
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>36
tcgccttccctcttcctttcctccagggatccctcacagaggagcacacagaaggccctt60
ctgcaacggaa71
<210>37
<211>97
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>37
tcgccttccctcttcctttcctccagggatccctcctacattgcctcccactggagaagc60
aacagaggagcacacagaaggcccttctgcaacggaa97
<210>38
<211>97
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>38
tcgccttccctcttcctttcctccagggatccctcctacagggatcttcccactggagaa60
gcacagaggagcacacagaaggcccttctgcaacgga97
<210>39
<211>17
<212>dna
<213>猪(sus)
<400>39
ctcctacaagtggattt17
<210>40
<211>53
<212>dna
<213>猪(sus)
<400>40
gacgggaaatgcatctcctacaagtgcacgaacagtccctataaacccctgga53
<210>41
<211>53
<212>dna
<213>猪(sus)
<400>41
gacgggaaatgcatctcctacaagtgcacgaacagtccctataaacccctgga53
<210>42
<211>26
<212>dna
<213>猪(sus)
<400>42
gacgggaaatgcatctcctacaagtg26
<210>43
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>43
aaatgcatctcctacaagtgcatttttgtttaaattaagatgtgggaggagaa53
<210>44
<211>54
<212>dna
<213>猪(sus)
<400>44
aaatgcatctcctacaagtgcatttttgtttaaattaagatgtgggaaggagaa54
<210>45
<211>25
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>45
aaatgcatctcctacaagtggattt25
<210>46
<211>29
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>46
catctcctacaagtgcaaggacgggtccg29
<210>47
<211>29
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>47
catctcctacaagtgcaaggacgggtccg29
<210>48
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>48
catctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtccg53
<210>49
<211>23
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>49
ctcctacaaggacgggtccgatg23
<210>50
<211>23
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>50
ctcctacaaggacgggtccgatg23
<210>51
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>51
ctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtccgatg53
<210>52
<211>40
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>52
cctacaagtggtttgtgagtgcaaggacgggtccgatgag40
<210>53
<211>40
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>53
cctacaagtggtttgtgagtgcaaggacgggtccgatgag40
<210>54
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>54
cctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtccgatgag53
<210>55
<211>81
<212>dna
<213>猪(sus)
<400>55
wtgccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagaga60
aactactggatcctgaaggtt81
<210>56
<211>75
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>56
gccacacaacgactggaacatgcattcaacatcgccatcagttaggcatagagaaactac60
tggatcctgaaggtt75
<210>57
<211>72
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>57
gccacacaacgactggaacatgcattcaacatcgccagttaggcatagagaaactactgg60
atcctgaaggtt72
<210>58
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>58
gccacacaacgactggaacatgcattcaacatcagttaggcatagagaaactactggatc60
ctgaaggtt69
<210>59
<211>72
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>59
gccacacaacgactggaacatgcattcaacatcgccagttaggcatagagaaactactgg60
atcctgaaggtt72
<210>60
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>60
gccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagagaaa60
ctactggatcctgaaggtt79
<210>61
<211>79
<212>dna
<213>猪(sus)
<400>61
gccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagagaaa60
ctactggatcctgaaggtt79
<210>62
<211>81
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>62
cccacacaacgactggaacatgcattcaacatcgcataatatatcagttaggcatagaga60
aactactggatcctgaaggtt81
<210>63
<211>77
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>63
gccacacaacgactggaacatgcattcaacatcgccaaatcagttaggcatagagaaact60
actggatcctgaaggtt77
<210>64
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>64
gccacacaacgactggaacatgcattcaacatcagttaggcatagagaaactactggatc60
ctgaaggtt69
<210>65
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>65
gccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagagaaa60
ctactggatcctgaaggtt79
<210>66
<211>76
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>66
gccacacaacgactggaacatgcattcaacatcgccaatcagttaggcatagagaaacta60
ctggatcctgaaggtt76
<210>67
<211>75
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>67
gccacacaacgactggaacatgcattcaacatcgccaagtattaggcatagagaaactac60
tggatcctgaaggtt75
<210>68
<211>78
<212>dna
<213>猪(sus)
<400>68
gggaaatgcatctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtcc60
gatgagtccctggagacg78
<210>69
<211>82
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>69
gggaaatgcatctcctacaagtggatttgtgatgggggaacaccgagtgcagggacgggt60
cataagagtccctggagacgtg82
<210>70
<211>74
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>70
gggaaatgcatctcctacaagtggatttgtggaacaccgagtgcaaggacgggtccgatg60
agtccctggagacg74
<210>71
<211>74
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>71
gggaaatgcatctcctacaagtggatttgtggaacaccgagtgcaaggacgggtccgatg60
agtccctggagacg74
<210>72
<211>74
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>72
gggaaatgcatctcctacaagtggatttgtggaacaccgagtgcaaggacgggtccgatg60
agtccctggagacg74
<210>73
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>73
gggaaatgcatctcctacaagtggatttgcaccgagtgcaaggacgggtccgatgagtcc60
ctggagacg69
<210>74
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>74
gggaaatgcatctcctacaagtggacttgcaccgagtgcaaggacgggtccgatgagtcc60
ctggagacg69
<210>75
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>75
gggaaatgcatctcctacaagtggatttgtgagtgcaaggacgggtccgatgagtccctg60
gagacg66
<210>76
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>76
gggaaatgcatctcctacaagtggatttgtgagtgcaaggacgggtccgatgagtccctg60
gagacg66
<210>77
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>77
gggaaatgcatctcctacaagtgcaaggacgggtccgatgagtccctggagacg54
<210>78
<211>48
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>78
gggaaatgcatctcctacaaggacgggtccgatgagtccctggagaag48
<210>79
<211>50
<212>dna
<213>猪(sus)
<400>79
gggaaatgcatctcctacaagtggacgggtccgatgagtccctggagacg50
<210>80
<211>50
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>80
tgctgatcctggcactgatcaaggacgggtccgatgagtccctggagacg50
<210>81
<211>50
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>81
gggaaatgcatctcctacaagtggacgggtccgatgagtccctggagacg50
<210>82
<211>50
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>82
tgctgatcctggcactgatcaaggacgggtccgatgagtccctggagacg50
<210>83
<211>41
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>83
tagacacagggagtatggtcacttgctgattcccaccgagt41
<210>84
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>84
gggaaatgcatctcctacaagtggatttgcaccgagtgcaaggacgggtccgatgagtcc60
ctggagacg69
<210>85
<211>70
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>85
gggaaatgcatctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtcc60
gatgagtccc70
<210>86
<211>77
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>86
gggaaatgcatctcctacaagtgcaaggagtgcaaggacggaatgcaccgagtgcaagga60
cgggtccgatgagtccc77
<210>87
<211>56
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>87
gggaaatgcatctcctacaagttgcattccgctgtgaattaggatgtgggcggaga56
<210>88
<211>75
<212>dna
<213>猪(sus)
<400>88
gggaaatgcatctcctacaagtggatttgtgatgacaccgagtgcaaggacgggtccgat60
gagtccctggagacg75
<210>89
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列89
<400>89
ggctcatttctcagcttgcagtgcaaggacgggtccggtgagtccctggagacg54
<210>90
<211>76
<212>dna
<213>人工序列
<220>
<223>indel序列89
<400>90
gggaaatgcatctcctacaagtggatttgtgatacacaccgagtgcaaggacgggtccga60
tgagtccctggagacg76
<210>91
<211>62
<212>dna
<213>猪(sus)
<400>91
tctatacctaggtcaaaaatgtaatgaagaaagtatcagttaggcatagagaaactactg60
ga62
<210>92
<211>52
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>92
tctatacctaggtcaaaaatgtaattcagttaggcatagagaaactactgga52
<210>93
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>93
tctatacctaggtcaaaaatgtaatgatcagttaggcatagagaaactactgga54
<210>94
<211>62
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>94
tctatacctaggtcaaaaatgtaatgagacaggtatcagttaggcatagagaaactactg60
ga62
<210>95
<211>44
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>95
tctatacctaggtcaaaaatgttaggcatagagaaactactgga44
<210>96
<211>43
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>96
tctatacctaggtcaaaaatgtaggcatagagaaactactgga43
<210>97
<211>51
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>97
tctatacctaggtcaaaaatgtaatcagttaggcatagagaaactactgga51
<210>98
<211>62
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>98
tctatacctaggtcaaaaatgtaatgaagaaagtatcagttaggcatagagaaactactg60
ga62
<210>99
<211>61
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>99
tctatacctaggtcaaaaatgtaatgaagaaatatcagttaggcatagagaaactactgg60
a61
<210>100
<211>62
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>100
tctatacctaggtcaaaaatgtaatgaattaaatatcagttaggcatagagaaactactg60
ga62
<210>101
<211>60
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>101
tctatacctaggtcaaaaatgtaatgaagagtatcagttaggcatagagaaactactgga60
<210>102
<211>56
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>102
tctatacctaggtcaaaaatgtaaaggtatcagttaggcatagagaaactactgga56
<210>103
<211>63
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>103
tctatacctaggtcaaaaatgtaaagaaaaggggtatcagttaggcatagagaaactact60
gga63
<210>104
<211>49
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>104
tctatacctaggtcaaaaattatcagttaggcatagagaaactactgga49
<210>105
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>105
tctatacctaggtcaaaaatgtaagaatcagttaggcatagagaaactactgga54
<210>106
<211>81
<212>dna
<213>猪(sus)
<400>106
gaaattttctttctatacctatgtcaaaaatgtaatgaaacttggcgatgttgaatgcat60
gttccagtcgttgtgtggctg81
<210>107
<211>77
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>107
gaaattttctttctatacctatgtcaaaaatgtaatgatggcgatgttgaatgcatgttc60
cagtcgttgtgtggctg77
<210>108
<211>60
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>108
gaaattttctttctatacctagggcgatgttgaatgcatgttccagtcgttgtgtggctg60
<210>109
<211>73
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>109
gaaattttctttctatacctaggtcaaaaatgtatggcgatgttgaatgcatgttccagt60
cgttgtgtggctg73
<210>110
<211>82
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>110
gaaattttctttctatacctaggtcaaaaatgtaatgaagacttggcgatgttgaatgca60
tgttccagtcgctgtgtggctg82
<210>111
<211>86
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>111
gaaattttctttcgatacctaggtcaaaaatgtaatgaagatctacttggcgatgttgaa60
tgcatgttccagtcgttgtgtggctg86
<210>112
<211>87
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>112
gaaattttctttctatacctaggtcaaaaatgtaatgaagatgatacttggcgatgttga60
atgcatgttccagtcgttgtgtggctg87
<210>113
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>113
ttttcactgttagtttgttgcaatccagccatgatattcatcagttaggcatagagaaac60
tactggatcctgaaggtta79
<210>114
<211>84
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>114
ttttcactgttagtttgttgcaatccagccatgatattcttagtatcagttaggcataga60
gaaactactggatcctgaaggtta84
<210>115
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>115
ttttcactgttggtttgttgcaatccagccaagatatatatcagttaggcatagagcaac60
tactggatcctgaaggtta79
<210>116
<211>59
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>116
tgtgatgaacactccacagaatctcgatgctgtcgttaccctctaactgtggattttga59
<210>117
<211>48
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>117
tgtgacagaatctcgatgctgtcgttaccctctaactgtggattttga48
<210>118
<211>59
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>118
ttgggcttgattgtgacagaatctcgatgctgtcgttaccctctaactgtggattttga59
<210>119
<211>11
<212>dna
<213>猪(sus)
<400>119
atgaacactcc11
<210>120
<211>33
<212>dna
<213>猪(sus)
<400>120
cttgattgtgatgaacactccacagaatctcga33
<210>121
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>121
cttgattgtgacagaatctcga22
<210>122
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>122
cttgattgtgacagaatctcga22
<210>123
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>123
cttgattgtgacagaatctcga22
<210>124
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>124
cttgattgtgacagaatctcga22
<210>125
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>125
cttgattgtgacagaatctcga22
<210>126
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>126
cttgattgtgacagaatctcga22
<210>127
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>127
cttgattgtgacagaatctcga22
<210>128
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>128
cttgattgtgacagaatctcga22
- 还没有人留言评论。精彩留言会获得点赞!