单细胞RNA和突变分析PCR(SCRM‑PCR):用于在单细胞水平上同时分析DNA和RNA的方法与流程
相关申请的交叉引用
本申请要求2015年1月21日提交的新加坡专利申请号10201500472r的优先权的权益,该新加坡专利申请的内容在此以引用方式整体并入,用于所有目的。
本发明大体上涉及聚合酶链式反应(pcr)。具体地说,本发明涉及同时分析rna和dna的方法。
背景技术:
1983年由karymullis开发的聚合酶链式反应(pcr)技术提供了快速扩增少量特定靶dna的方法。扩增的dna可用于促进对于存在dna序列变异、突变、限制性酶切割或寡核苷酸对的连接的分析。pcr已成为在医学和生物研究实验室中广泛应用的一种常见的且经常不可缺少的工具。
这些应用中的一些越来越指向需要将特定的dna谱与其基因表达相关联。例如,已变得越来越有用的是,确定在血流中循环的癌细胞的突变,并且将该特异性dna谱与其基因表达相关联以确定所述细胞的类型和来源,以便促进各种人癌症的诊断、预后、预防和治疗。对临床样品或其它样品如法医样品的此类分析的一个问题在于所述样品经常含有低数目的细胞(如1-100个细胞),或者靶核酸以非常有限的量存在。
在下一代测序领域中,如在rna测序(rna-seq)中,来源于大规模转录组学的数据和发现经常需要通过靶向测定来验证。然而,来源于rna-seq数据的dna序列由于逆转录酶缺乏校正活性而经常容易出错。在对低数目的细胞进行rna-seq实验的情况下,这是特别成问题的。
因此,需要提供克服或至少改善上述一个或多个缺点的从单细胞或有限量的核酸同时分析rna和dna的方法。
需要以有成本效益的方式与最少的程序步骤提供这种方法。
技术实现要素:
在第一方面,提供了同时分析样品中的rna和dna的方法,所述方法包括以下步骤:
(a)使所述样品与来自第一引物对的反向引物和逆转录酶接触,以实现将所述rna逆转录为cdna,来自所述第一引物对的所述反向引物指向靶rna区域;
(b)随后使所述样品与以下接触:
(i)来自所述第一引物对的正向引物,来自所述第一引物对的所述正向引物指向靶cdna区域,
(ii)来自第二引物对的反向引物和正向引物,来自所述第二引物对的所述反向引物和正向引物指向靶dna区域,和
(ⅲ)dna聚合酶
以同时扩增所述靶cdna区域和所述靶dna区域;和
(c)分析所述扩增的靶cdna区域和/或所述扩增的靶dna区域。
所述第一方面的所述方法的一个实施方案进一步包括使用步骤(a)中的所述反向引物或步骤(b)(i)中的所述正向引物和在所述扩增的靶cdna区域内结合的巢式引物,使来自步骤(b)的所述样品经受半巢式pcr的步骤。
在第二方面,提供了同时分析样品中的rna和dna的方法,所述方法包括以下步骤:
(a)溶解所述样品中的细胞;
(b)使溶解的细胞的样品与来自第一引物对的反向引物和逆转录酶接触,以实现将所述rna逆转录为cdna,来自所述第一引物对的所述反向引物指向靶rna区域;
(c)随后使所述溶解的细胞的样品与以下接触:
(i)来自所述第一引物对的正向引物,来自所述第一引物对的所述正向引物指向靶cdna区域,
(ii)来自第二引物对的反向引物和正向引物,来自所述第二引物对的所述反向引物和正向引物指向靶dna区域,和
(ⅲ)dna聚合酶
以在预扩增步骤中同时扩增所述靶cdna区域和所述靶dna区域;
(d)使用步骤(b)中的所述反向引物或步骤(c)(i)中的所述正向引物、在所述预扩增的靶cdna区域内结合的巢式引物和dna聚合酶,使来自步骤(c)的所述样品经受半巢式pcr,以进一步扩增所述靶cdna区域,和/或使用在所述预扩增的靶dna区域内结合的巢式引物对和dna聚合酶,使来自步骤(c)的所述样品经受巢式pcr,以进一步扩增所述靶dna区域;和
(e)分析所述进一步扩增的靶cdna区域和/或所述进一步扩增的靶dna区域。
在第三方面,提供了根据第一或第二方面的方法用于确定单细胞或多个细胞的dna谱和/或基因表达谱、或来源于单细胞或多个细胞的无细胞rna和/或dna的dna谱和/或基因表达谱的用途。
在第四方面,提供了根据第一或第二方面的方法用于验证大规模转录组学数据集的用途。
在第五方面,提供了用于进行根据第一或第二方面的方法或用于根据第三或第四方面的用途的试剂盒,其中所述试剂盒包含:
(a)选自由以下组成的组的引物:
i.所述第一方面的所述方法的步骤(a)的所述反向引物或所述第二方面的所述方法的步骤(b)的所述反向引物,
ii.所述第一方面的所述方法的步骤(b)(i)的所述正向引物或所述第二方面的所述方法的步骤(c)(i)的所述正向引物,
iii.所述第一方面的所述方法的步骤(b)(ii)的所述引物对或所述第二方面的所述方法的步骤(c)(ii)的所述引物对,和
iv.所述第一方面的所述方法的所述巢式引物和巢式引物对或所述第二方面的所述方法的步骤(d)的所述巢式引物和巢式引物对;
(b)选自由以下组成的组的一种或多种试剂:
i.逆转录酶和一种或多种适合的反应缓冲液,用于所述第一方面的所述方法的步骤(a)中的所述逆转录或所述第二方面的所述方法的步骤(b)中的所述逆转录,
ii.dna聚合酶和一种或多种适合的反应缓冲液,用于所述第一方面的所述方法的步骤(b)中的所述扩增或所述第一方面的所述方法的所述半巢式或巢式pcr中的所述扩增或所述第二方面的所述方法的步骤(c)或(d)中的所述扩增,和
iii.一种或多种经标记或未经标记的选自由datp、dctp、dgtp和dttp或dutp组成的组的脱氧核糖核苷酸;和
(c)使用说明,用于进行根据第一或第二方面的方法或用于根据第三或第四方面的用途。
附图说明
当结合非限制性实施例和附图考虑时,参考具体实施方式将更好地理解本发明,在所述附图中:
图1
[图1]提供了本公开的scrmpcr方法的概要。该方法可用于检测和定量rna转录物并对来自相同的单细胞的dna进行测序。该方法的示意图包含于图1中,并且由以下步骤构成:
1)引物设计:引物组被设计用于半巢式pcr(rna转录物)或巢式pcr(基因组dna)。在一些情况下,引物来源于文献(表1)。2)逆转录:将多重反向引物连同逆转录酶一起添加到溶解的单细胞中(图1中的步骤ii)。3)预扩增:将多重正向引物连同对dna序列特异的引物添加到所述混合物中,并且使用优化的退火温度进行预扩增步骤(图1中的步骤iii)。4)清除:通过纯化柱(spincolumn)或酶消化去除未使用的引物。5)样品的稀释。6)对于rna分析,使用半巢式方法进行qpcr,其中一种引物被设计成结合预扩增产物内在的序列,并且先前已在预扩增步骤期间使用另一引物(图1中的步骤iv)7)对于dna分析:使用巢式方法进行pcr,其中两种引物均被设计成结合预扩增产物内在的序列(图1中的步骤iv)。
图2
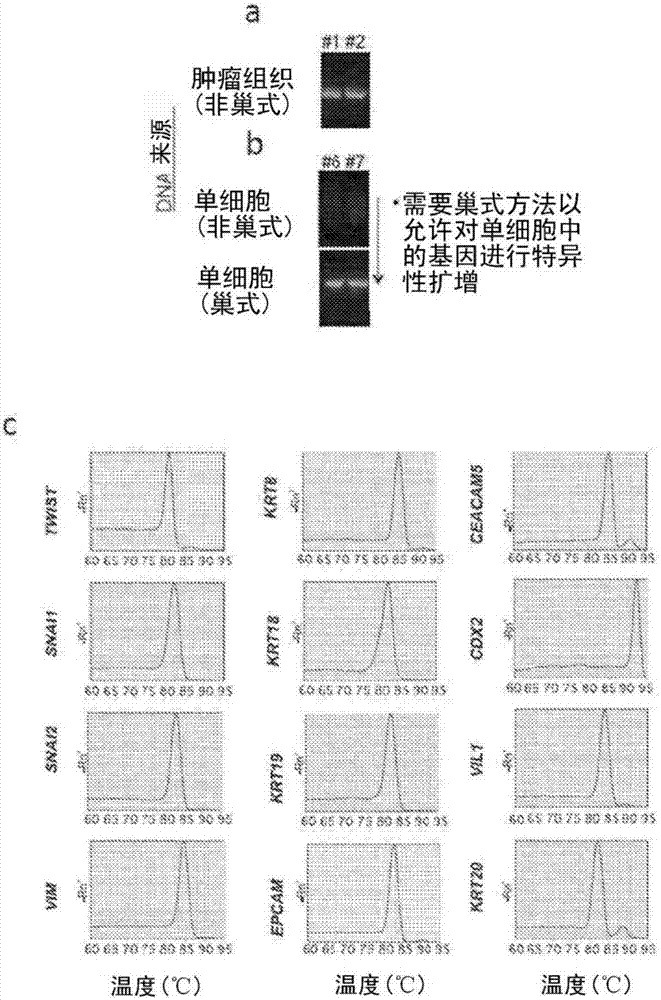
[图2]显示来自单细胞的pcr产物的特异性扩增需要巢式pcr。在(a)中,用于tp53外显子6的引物被优化以扩增组织dna,并获得可被测序的特异性产物。在(b)中,使用相同的引物对,tp53外显子6被预扩增,并将反应以2等量分开。第一部分用于使用相同引物对的第二轮pcr。这导致非特异性且低效的扩增(上图)。然后通过使用巢式引物对进行第二次扩增获得特异性pcr产物(下图)。(c)显示由qpcr进行rna检测的若干半巢式测定的解链曲线,表明靶产物的特异性扩增。
图3
[图3]提供了本公开的scrmpcr方法的原理证据。在5μl2x反应缓冲液(celldirect试剂盒)中显微操作单个dld1和rko细胞(结直肠癌细胞系)。然后如本文所述进行scrmpcr,结果示于(a)中。扩增属于tp53、kras和braf基因的基因组区域。对pcr产物进行桑格(sanger)测序,且如(b)中所示检测先前已在两个细胞系中表征的已知热点突变。同时,来自相同细胞的若干转录物被扩增并显示在两个细胞系中均具有可变的基因表达。如(c)中所示,通过解链曲线峰值温度并且通过单个峰值的存在验证基因表达特异性。
图4
[图4]使用从临床样品分离的单细胞和单细胞簇提供本公开的scrmpcr方法的原理证据。对从结直肠癌患者血液分离的未知来源的单细胞或单个簇进行显微操作并进行scrmpcr,如本文所述。在(a)中,若干上皮和间充质标志物的基因表达显示单个簇的主要间充质性质。还显示了ptprc(cd45)的表达。颜色表示从不存在(深灰色)到最大(浅灰色)的基因表达。ntc,无模板对照。在(b)中,匹配的结直肠癌组织中突变的靶向基因区域的桑格测序结果显示(a)中所示的相同簇不含突变,因此与肿瘤上皮无关。(c)显示使用最新的scrmpcr方案,其含有另外的测定来定量来自一组簇的转录物,以显示簇表达属于内皮谱系的转录物。热图表示对照单细胞和14个ctm(n=4位患者)中的scrmpcr基因表达。色标指示从低或不存在(深灰色)到高(浅灰色)的基因表达。
图5
[图5]显示使用微滤装置对循环的肿瘤细胞的回收。(a)显示装置设置。封闭硅微筛(插图,比例尺=10μm)的微滤装置连接到用于流速控制的蠕动泵。(b)显示用于各种下游应用的微滤程序。数字指示每个步骤的程序时间。(c)显示用于下游单细胞显微操作的回收效率和纯度的优化。各散点图表示使用各种流速和微筛孔径的实验。黑色虚线矩形指示用于被回收细胞的最佳下游处置的>90%回收效率和>5×103个wbc耗尽的靶区域。数据点是在每种条件下进行三个独立实验的平均值±s.e.m.。
图6
[图6]显示ctm表达emt标志物,但不反映原发性肿瘤突变或染色体异常。(a)显示来自具有已知的原发性肿瘤突变的4位crc患者的9个ctm的图像,所述ctm在用于下游scrmpcr的单个管中被显微操作。(b)ctmacgh显示来自具有已知的染色体异常的代表性crc患者的三个ctm的图像。(c)显示(b)中所示的的ctm的acgh分析,匹配正常组织和肿瘤组织。
图7
[图7]显示ctm是肿瘤来源的成熟内皮细胞。(a)显示从单ctm的rna-seq数据的谱系推断。归一化的比值比比较针对每个样品(行)和细胞类型(列)富集相对于随机富集的基因数。mep,巨核细胞-红系祖细胞。在(b)中,免疫荧光研究确认了ctm的内皮谱系。针对所示抗体染色的代表性的ctm,每个染色均有内部对照。插图中图,cd45+wbc。插图右图,cd41/cd42b+血小板凝集物。该表指示对所示免疫荧光为阳性或阴性的ctm的定量。(c)显示针对ctm的epc克隆形成实验的结果(n=5位患者)。散点图表示ctm(灰色符号)或huvec细胞(黑色圆圈)的初始(x轴)和最终(y轴)数目。相同的灰色符号是技术重复。huvec细胞数据表示为技术重复的平均值±s.e.m。显示了两个代表性实验中的一个。(d)显示基于主要组分分析的ctm和组织rnaseq数据的秩相关。(e)显示术前0-24h和手术后24-72h的cd31+cd45-ctm计数的梯形图。线条连接来自同一患者的数据。双尾威氏(wilcoxon)符号秩检验,***p=0.0006,效应值r=0.54。(f)显示单个患者的所示组织、pbmc和ctm的folh1表达。条形代表平均值。在组织中,误差条是来自独立数据点的s.e.m.(n=3位患者)。(g)显示正常组织、肿瘤组织中和单ctm中来自rna-seq数据的cd276。(h)显示具有低(≤10ctm/2ml)和高(>10ctm/2ml)ctm计数的患者中的微血管密度计数(n=17位患者)。箭头和白色虚线指示代表性图像中的微血管腔。具有邦弗朗尼(bonferroni)校正的双尾威-曼-怀三氏(wilcoxon-mann-whitney)u检验,*p=0.03,效应值r=0.57,
图8
[图8]显示ctm在crc患者中普遍存在,反映了治疗干预并指示早期未治疗(treatment-naive)患者中存在结直肠癌。(a)是血液中的ctm的病例对照研究。显示每个健康对照(n=45)和crc病例(n=80)的ctm计数。双尾威-曼-怀三氏u检验,***p=7.31×10-15,效应值r=0.65,
图9
[图9]图解说明硅微筛、微滤装置及其回收效率和纯度。(a)显示硅微筛的明场合成图像(左图)和扫描电子显微照片(右图)。(b)显示具有部件的微滤装置。(c)显示硅微筛允许对被捕获细胞的有效回收。来自全血的hct116细胞的捕获效率,指示可被回收用于下游测定(浅灰色条)或不可被回收(暗灰色条)的被捕获细胞的%。显示了六个独立实验。(d)显示sw620(浅灰色线)的大小分布,(n=50)。从coumans,f等,2013年报道的分别从结直肠癌、前列腺癌和乳腺癌患者分离的wbc和ctc的中值大小。(e)显示使用图5b和5c中所示的优化方案的临床样品中污染的有核细胞(n=13)。(f)显示使用图5b和5c中所示的优化方案的从另外的细胞系的回收效率。条形表示平均值±s.e.m。
图10
[图10]显示ctm的表征和定义。(a)显示10μl洗涤缓冲液中cd45-ctm(p7-ctm3,还参见图1、图4a和图6a)的代表性未裁剪图像,表明适于下游分析的样品的纯度,并且显示在最终显微操作(参见方法)之前,靶cd45-ctm和稀疏rbc和wbc的存在。比例尺=50μm。(b)显示从5位crc患者回收的ctm的cd45和ep-cam的代表性免疫荧光。该表指示每个染色和患者的cd45-ctm计数。比例尺=10μm。(c)ctm的代表性细胞形态。比例尺=10μm。dic:微分干涉对比。
图11
[图11]显示使用上皮样(dld-1)和间充质样(rko)crc细胞系的scrmpcr原理证据。(a)显示所示的基因和单细胞或大批细胞的dna扩增(凝胶电泳)和rna定量(热图)。(b)显示来自rko和dld-1单细胞中已知突变等位基因的dna热点的序列色谱图。(c)显示来自qpcr反应的所示基因的解链曲线图。sc,单细胞。ntc,无模板对照。rn,归一化的报道信号
图12
[图12]显示ctm表达emt标志物,但具有正常的染色体结构。(a)显示cd45、波形蛋白(vim)、泛角蛋白(pan-keratin)(ck)和dapi的两种ctm的代表性4色免疫荧光,表明异源间充质标志物和上皮标志物的表达。(b)显示对照实验,以评估全基因组扩增(wga)对使用单细胞的acgh实验的影响。(c-e)各自显示与(b)中所示的正常组织dna类似的所示患者的单ctm的acgh。
图13
[图13]显示用于rna-seq的ctm的细胞形态学。(a-g)各自显示用于每个相应患者的rna-seq的ctm的微分干涉对比(dic)和hoechst33342图像。患者p1无可用图像。
图14
[图14]显示谱系推断工作流程。(a)是谱系推断工作流程的流程图。图(b)显示使用biogps(wu等)验证了对代表性谱系具有最高特异性指数的所选基因(c)显示通常用于ctc研究中以表示上皮细胞的标志物的基因表达水平。注意内皮谱系中的krt18表达和造血细胞中的epcam表达。
图15
[图15]显示谱系推断算法验证。(a)显示比较针对每个样品(行)和谱系(列)富集的相对于随机富集的基因数的热图。样品是来自所选谱系的已公布的rna-seq数据。每个有色盒表示相应的费希尔(fisher)精确检验的0(黑色)到1(浅灰色)的归一化比值比。(b)与(a)中相同,不同之处在于使用全组织或复杂细胞混合物如pbmc、皮肤和脑的数据集。
图16
[图16]显示aceto等报道的ctm和ctc-簇之间的比较。来自aceto等的ctc簇数据的谱系推断显示上皮细胞类型的富集。从该分析中去除血小板和红细胞信号。显示了本研究中分析的ctm的rna-seq数据用于比较。mcf7和huvecrna-seq显示为阳性对照。
图17
[图17]显示ctm中表达的肿瘤内皮标志物。使用rna-seq数据,另外的肿瘤内皮标志物在正常组织、肿瘤组织和ctm中表达。plxdc1,含有丛状蛋白结构域者1(plexindomaincontaining1)(肿瘤内皮标志物3/7)(stcroix等);mmp2,基质金属肽酶2(stcroix等);nid1,巢蛋白1(stcroix等);mmp11(stcroix等),基质金属肽酶11;clec14a,c型凝集素结构域家族14,成员a(mura等);postn,骨膜素(borgia等);vwf,冯·威利布兰德(vonwillebrand)因子(zanetta等);ecscr,内皮细胞表面表达的趋化性和细胞凋亡调控物(herbert等)。
图18
[图18]显示ctm计数与炎症标志物或其它变量不相关。(a-c)显示ctm数目分别与所示的肿瘤特征、患者特征和血液测试值的关联性。相关性被显示为点图,并使用肯德尔(kendall)τ系数和其导出的p值进行测量。二分变量的比较被显示为盒形图,并且使用来自双尾精确威-曼-怀三氏u检验的p值来定量差异。
具体实施方式
本公开的方法可用于同时定量rna转录物和测序dna区域,所述rna转录物和dna区域来自单细胞。所述方法可被应用于其它样品,例如核酸经常以非常有限的量存在的临床或法医样品,或者可被应用于含有低数目的细胞例如2-100个细胞的实验样品。
在第一方面,提供了同时分析样品中的rna和dna的方法,所述方法包括以下步骤:
(a)使所述样品与来自第一引物对的反向引物和逆转录酶接触,以实现将所述rna逆转录为cdna,来自所述第一引物对的所述反向引物指向靶rna区域;
(b)随后使所述样品与以下接触:
(i)来自所述第一引物对的正向引物,来自所述第一引物对的所述正向引物指向靶cdna区域,
(ii)来自第二引物对的反向引物和正向引物,来自所述第二引物对的所述反向引物和正向引物指向靶dna区域,和
(ⅲ)dna聚合酶
以同时扩增所述靶cdna区域和所述靶dna区域;和
(c)分析所述扩增的靶cdna区域和/或所述扩增的靶dna区域。
有利地,步骤(b)中靶cdna区域和靶dna区域的同时扩增可形成预扩增步骤,其增加cdna和/或dna的量,所述cdna和/或dna作为在分析前进一步扩增靶cdna和/或靶dna区域的模板。所述靶dna区域可以是靶基因组dna区域。
“引物”意指当与dna或rna链配对时能够在适合的聚合剂存在下起始引物延伸产物的合成的寡核苷酸。引物出于最大的扩增效率优选单链,但或者可以是双链。引物必须足够长以在聚合剂存在下备好延伸产物的合成。引物的长度取决于许多因素,包括应用、待采用的温度、模板反应条件、其它试剂和引物来源。例如,取决于靶序列的复杂性,寡核苷酸引物通常含有15到35个或更多个核苷酸,尽管它可能含有更少的核苷酸。引物可以是大的多核苷酸,如约200个核苷酸到数千碱基或更多。引物可被选择为与模板上的序列“基本上互补”,其被设计成与所述模板杂交并且用作合成起始用位点。例如,并非引物中的所有碱基都需要反映将与引物杂交的模板分子的序列-引物只需要含有足够的互补碱基以使引物能够与模板杂交。引物可包含额外的碱基,例如在5'端呈限制酶识别序列的形式,以促进扩增的dna的克隆。引物还可包含一个或多个位置处的错配碱基,所述错配碱基不与模板中的碱基互补,而是被设计成在碱基延伸或扩增时将变化引入到dna中。
术语“扩增(amplification或amplify)”涉及核酸的额外拷贝的产生。扩增可使用聚合酶链式反应(pcr)技术或本领域内已知的其它核酸扩增技术进行。
“引物对”可用于核酸的扩增(和鉴别),例如通过聚合酶链式反应(pcr)。“引物对”可包含“正向引物”和“反向引物”。在pcr反应中,双链dna的两条链均被扩增。“正向引物”可结合dna的一条链,并允许从5'到3'方向合成引物延伸产物。“反向引物”可结合到dna的互补链,并且也允许在互补dna链的5'到3'方向上合成引物延伸产物。在逆转录反应中,“反向引物”可结合到rna链,并且允许在逆转录酶存在下在cdna链的5'到3'方向上合成互补dna(cdna)链。随后可将“反向引物”连同“正向引物”一起用于扩增合成的cdna链。pcr引物对可从已知序列得到,例如,通过使用意在用于该目的的计算机程序,如primer(0.5版,1991,whiteheadinstituteforbiomedicalresearch(怀特黑德生物医学研究所),剑桥,马萨诸塞州)和本文所公开的实施例中所使用的那些(例如,primerblast、ncbi引物设计工具、ensembl基因组浏览器、netprimer)。使用本领域内已知的用于该目的的软件选择用作引物的寡核苷酸。例如,oligo4.06软件可用于选择各自最多达30-100个核苷酸的pcr引物对,以及用于从最多达32千碱基的输入多核苷酸序列分析最多达5,000个核苷酸的寡核苷酸和更大的多核苷酸。
用于pcr扩增反应、限制酶消化和随后的片段拆分以及核酸测序中的方法和试剂是本领域技术人员众所周知的。在每种情况下,适合的方案和试剂将在很大程度上取决于各个情况。可从各种来源获得指导,如例如sambrook等,分子克隆:实验室手册(molecularcloning:alaboratorymanual),纽约冷泉港实验室,1989和ausubel等,分子生物学中的现行方案(currentprotocolsinmolecularbiology),greenepubl.assoc.andwiley-intersciences,1992。本领域技术人员将容易地了解,可在不影响实现期望产品的能力的情况下改变这些程序的各种参数。例如,在pcr扩增的情况下,盐浓度可以变化。类似地,用作模板的dna的量也可取决于可用的dna的量或有效扩增所需的最佳模板量而变化。
本领域技术人员将能够理解,“逆转录酶”是可用于基于rna模板合成cdna的酶。本领域技术人员还将理解,“dna聚合酶”是可基于dna模板合成dna分子的酶。
通过“接触”,可使引物与样品物理缔合。这允许例如引物对与存在于样品中的dna退火,并随后通过pcr扩增所述dna。这还允许引物与存在于样品中的rna链退火,以允许使用如本领域技术人员已知的逆转录酶合成cdna。
如本文所定义的方法允许“同时”扩增样品或单细胞中的rna和dna。术语“同时”意味着能够扩增存在于同一样品或单细胞中的rna和dna两者。它还可能意味着能够分析来自同一样品或单细胞的rna和dna两者。
据本发明人所知,这是允许在同一单细胞中靶向分析dna突变和rna转录两者的第一种方法。该方法已经历若干优化步骤。
本发明人已发现,对dna和rna两者的特异性扩增只可通过至少使用用于rna的半巢式方法和用于dna分子的完全巢式方法(例如参见图2)来实现。如本文所用的术语“半巢式pcr”是指其中使用一个“巢式引物”来减少由于意外结合位点的扩增所导致的非特异性结合的经改进的pcr技术。“完全巢式方法”是指其中在模板dna的两侧使用两个巢式引物的经改进的pcr技术。“巢式引物”的使用允许使用第一组引物扩增的pcr产物的特异性识别,从而消除来自不想要的产物如引物二聚体、发夹和替代引物靶序列的污染。本发明人还发现dna和rna分子的扩增受到预扩增步骤中的退火温度差异性地影响。因此,需要设置取舍条件(trade-off)以扩增两种分子。
因此,在一个实施方案,根据第一方面的方法进一步包括以下步骤:使用步骤(a)中的反向引物或步骤(b)(i)中的正向引物和在扩增的靶cdna区域内结合的巢式引物,使来自步骤(b)的样品经受半巢式pcr。所述巢式引物可以是匹配或对应于步骤(a)中的反向引物或步骤(b)(i)中的正向引物的引物。在另一实施方案中,根据第一方面的方法进一步包括以下步骤:使用在扩增的靶dna区域内结合的巢式引物对,使来自步骤(b)的样品经受巢式pcr。在一个实施方案中,步骤(a)和(b)在相同的反应混合物中进行。
在一个实施方案中,同时对于一个或多个靶rna区域和/或一个或多个靶cdna区域和/或一个或多个靶dna区域进行根据第一方面的方法。因此,各自对靶rna区域具有相同或不同特异性的一种或多种反向引物可用于步骤(a)中,各自对靶cdna区域具有相同或不同特异性的一种或多种正向引物可用于步骤(b)(i)中,各自对靶dna区域具有相同或不同特异性的一种或多种引物对可用于步骤(b)(ii)中,可使用结合到靶cdna区域的一种或多种巢式引物和结合到靶dna区域的一种或多种巢式引物对。
有利地,根据第一方面的方法可用于在有限量的样品可用的情况下分析rna和dna。
在一个实施方案中,样品包含单细胞或多个细胞(例如,低数目的细胞,包括约2到约100个细胞、约2到约90个细胞、约2到约80个细胞、约2到约70个细胞、约2到约60个细胞、约2到约50个细胞、约2到约40个细胞、约2到约30个细胞、约2到约20个细胞、约2到约10个细胞、约2到约5个细胞、约5到约100个细胞、约10到约100个细胞、约20到约100个细胞、约30到约100个细胞、约40到约100个细胞、约50到约100个细胞、约60到约100个细胞、约70到约100个细胞、约80到约100个细胞或约90到约100个细胞)。在步骤(a)之前,单细胞或多个细胞可被溶解以释放所述细胞(或多个细胞)内所含的rna和dna。
在另一实施方案中,样品包含无细胞的rna或无细胞的dna。
rna或dna可以少量存在,例如约1pg到约10ng、约5pg到约10ng、约5pg到约5ng、约5pg到约1ng、约5pg到约500pg、约5pg到约250pg、约5pg到约125pg、约5pg到约100pg或约5pg到约50pg。
样品可以是从受试者分离的组织、细胞、小囊泡如外来体、体液和其分离物等样品。样品的示例包括:全血、血液流体(例如血清和血浆)、淋巴和囊液、痰、粪便、眼泪、粘液、毛发、皮肤、腹水液、囊液、尿液、乳头渗出物、乳头抽吸物、精液、阴道液、汗液、外来体或其它小囊泡、组织切片(如活组织检查和尸体剖检样品、为组织学目的而取出的冷冻切片)、档案样品(archivalsample)、来源于患者组织的外植体和原代和/或转化的细胞培养物、临床样品、来自犯罪现场的法医样品、土壤样品等。
单细胞或多个细胞可以是真核细胞、细菌细胞或古细菌细胞、来自植入前胚胎的细胞、干细胞、疑似癌细胞、疑似肿瘤来源的细胞、疑似胚胎细胞、来自病原生物体的细胞或从犯罪现场获得的细胞。
在一个实施方案中,第一引物对包含跨越外显子-外显子边界的引物或由相应dna区域上的至少一个内含子隔开的引物。
在一个实施方案中,第二引物对包含与靶dna区域的内含子区域结合的引物。
术语“外显子”是指基因组dna的一部分,该部分成为转化为成熟信使mrna的基因组dna的一部分。术语“内含子”或“内含子区域”是指基因组dna的通过rna剪接被去除且因此不会存在于最终的成熟mrna中的一部分。
术语“分析(analyze或analyzing)”是指通过本领域内已知的各种技术研究或检查扩增的靶cdna区域和/或扩增的靶dna区域。可研究扩增的cdna区域和/或扩增的靶dna区域,针对其基因表达或可能存在的突变。
在一个实施方案中,步骤(c)中的分析包括针对基因表达分析扩增的靶cdna(例如在基因表达分析中)。基因表达分析可以使用本领域内已知的任何技术进行,如定量pcr、数字pcr、微阵列等。
在一个实施方案中,步骤(c)中的分析包括针对突变分析扩增的靶dna(例如在突变分析中)。突变分析可使用本领域内已知的任何技术进行,如桑格测序、maxam-gilbert测序、焦磷酸测序、鸟枪法(shot-gun)测序、高通量dna测序、等位基因特异性pcr(aspcr)或高分辨率解链温度pcr(hrm)。
根据第一方面的方法的步骤(b)中的预扩增可包括一个或多个循环步骤。每个循环步骤可包含在预定温度下持续预定的持续时间的一个或多个扩增循环(即,变性、退火和伸长)。应当了解,循环步骤数、变性、退火和伸长的循环数、进行循环的温度以及施加每个温度的持续时间将取决于用于扩增反应中的试剂、靶cdna或dna区域、所用的引物、待扩增的样品。在一个实施方案中,扩增不包括最终延伸步骤。
在一个实施方案中,步骤(b)包括约1到约50个循环步骤、约1到约40个循环步骤、约1到约30个循环步骤、约1到约25个循环步骤、约1到约20个循环步骤、约1到约10个循环步骤、约1到约5个循环步骤、约1到约4个循环步骤、约1到约3个循环步骤、约1个循环步骤、约2个循环步骤或约3个循环步骤。在一个实施方案中,步骤(b)包括3个循环步骤。
在一个实施方案中,每个循环步骤包括约1到约50个循环、约1到约40个循环、约1到约30个循环、约1到约25个循环、约1到约20个循环、约1到约18个循环、约1到约15个循环、约1到约10个循环、约1到约6个循环、约2个循环、约4个循环、约6个循环、约8个循环、约10个循环、约20个循环、约30个循环、约40个循环或约50个循环的变性、退火和伸长。在一个实施方案中,每个循环步骤包括6个循环的变性、退火和伸长。
在一个实施方案中,循环中的退火和/或伸长温度为约40℃到约75℃、约40℃到约70℃、约40℃到约65℃、约40℃到约60℃、约40℃到约55℃、约40℃到约50℃、约40℃、约45℃、约50℃、约55℃、约60℃、约65℃、约70℃或约75℃。
用于连续循环步骤的退火和/或伸长温度可降低约1℃到约10℃、约1℃到约9℃、约1℃到约8℃、约1℃到到约7℃、约1℃到约6℃、约1℃到约5℃、约1℃到约4℃、约1℃到约3℃或约1℃到约2℃。
在一个实施方案中,退火和/或伸长进行约10秒到约10分钟、约10秒到约8分钟、约10秒到约6分钟、约10秒到约4分钟、约10秒到约2分钟、约10秒到约1分钟、约1分钟、约2分钟、约4分钟、约6分钟、约8分钟或约10分钟。
在一个实施方案中,在约75℃到约120℃、约75℃到约105℃、约75℃到约95℃、约75℃到约90℃、约75℃到约85℃、约75℃到约80℃、约75℃、约80℃、约85℃、约90℃、约95℃、约100℃、约105℃、约110℃、约115℃或约120℃的温度下进行变性。变性可以进行约1秒到约10分钟、约1秒到约5分钟、约1秒到约4分钟、约1秒到约3分钟、约1秒到约2分钟、约1秒约1分钟、约1秒、约10秒、约20秒、约30秒、约40秒、约50秒、约1分钟、约2分钟、约3分钟、约4分钟、约5分钟或约10分钟。在一个实施方案中,根据第一方面的方法的步骤(b)包括:
60℃持续4分钟、之后95℃持续1分钟的6个循环,
55℃持续4分钟、之后95℃持续1分钟的6个循环,和
50℃持续4分钟、之后95℃持续1分钟的6个循环。
在第二方面,提供了同时分析样品中的rna和dna的方法,所述方法包括以下步骤:
(a)溶解所述样品中的细胞;
(b)使所述溶解的细胞样品与来自第一引物对的反向引物和逆转录酶接触,以实现将所述rna逆转录为cdna,来自所述第一引物对的所述反向引物指向靶rna区域;
(c)随后使所述溶解的细胞样品与以下接触:
(i)来自所述第一引物对的正向引物,来自所述第一引物对的所述正向引物指向靶cdna区域,
(ii)来自第二引物对的反向引物和正向引物,来自所述第二引物对的所述反向引物和正向引物指向靶dna区域,和
(ⅲ)dna聚合酶
以在预扩增步骤中同时扩增所述靶cdna区域和所述靶dna区域;
(d)使用步骤(b)中的所述反向引物或步骤(c)(i)中的所述正向引物、在所述预扩增的靶cdna区域内结合的巢式引物和dna聚合酶,使来自步骤(c)的所述样品经受半巢式pcr,以进一步扩增所述靶cdna区域,和/或使用在所述预扩增的靶dna区域内结合的巢式引物对和dna聚合酶,使来自步骤(c)的所述样品经受巢式pcr,以进一步扩增所述靶dna区域;和
(e)分析所述进一步扩增的靶cdna区域和/或所述进一步扩增的靶dna区域。
在第二方面的方法的一个实施方案中,步骤(d)中的巢式引物可以是匹配或对应于步骤(b)中的反向引物或步骤(c)(i)中的正向引物的引物。在第二方面的方法的一个实施方案中,步骤(b)和(c)在相同的反应混合物中进行。
与第一方面的方法一样,也可同时对于一个或多个靶rna区域和/或一个或多个靶cdna区域和/或一个或多个靶dna区域(如一个或多个靶基因组dna区域)进行根据第二方面的方法。因此,各自对靶rna区域具有相同或不同特异性的一种或多种反向引物可用于根据第二方面的方法的步骤(b)中,各自对靶cdna区域具有相同或不同特异性的一种或多种正向引物可用于根据第二方面的方法的步骤(c)(i)中,各自对靶dna区域具有相同或不同特异性的一种或多种引物对可用于根据第二方面的方法的步骤(c)(ii)中,与靶cdna区域结合的一种或多种巢式引物和与靶dna区域结合的一种或多种巢式引物对可用于第二方面的方法的步骤(d)中。
术语“溶解”是指分解细胞的膜。这允许细胞中的dna或rna可被例如一种或多种引物和酶接触。
第二方面的方法的步骤(a)中的溶解可使用本领域内已知的任何技术进行,例如使用溶解缓冲液、超声处理、冷冻-解冻、机械破碎等。
在一个实施方案中,第二方面的方法的步骤(c)中的预扩增不包括最终延伸步骤。
如上文所述用于第一方面的方法的样品、引物、引物对、分析技术和预扩增条件(如循环步骤、其中所用的温度和持续时间)也可用于第二方面的方法中。
在第三方面,提供了根据第一或第二方面的方法用于确定单细胞或多个细胞(例如,低数目的细胞,包括约2到约100个细胞)或来源于单细胞或多个细胞的无细胞的rna和/或dna(例如,少量的dna和/或rna,包括约1pg到约10ngdna和/或rna)的dna谱和/或基因表达谱的用途。
术语“dna谱”是指一个细胞或多个细胞内一系列基因的dna序列的表征。术语“基因表达谱”是指一个细胞或细胞内一系列基因的表达水平的表征。
该用途可被应用于包含来自受试者的异源细胞类型或少量无细胞的rna和/或dna的生物样品。生物样品可选自由从受试者分离的植入前胚胎、组织、细胞(例如干细胞、疑似癌细胞)、体液和其分离物等组成的组。生物样品的示例包括:全血、血液流体(例如血清和血浆)、淋巴和囊液、痰、粪便、眼泪、粘液、毛发、皮肤、腹水液、囊液、尿液、乳头渗出物、乳头抽吸物、精液、阴道液、汗液、外来体或其它小囊泡、组织切片(如活组织检查和尸体剖检样品、为组织学目的而取出的冷冻切片)、档案样品、来源于患者组织的外植体和原代和/或转化的细胞培养物、临床样品、来自犯罪现场的法医样品、土壤样品等。
受试者可以是哺乳动物,如人。单细胞或多个细胞的dna谱与基因表达谱的关联性、或少量无细胞的dna和/或无细胞的rna的dna谱与基因表达谱的关联性可用于优化用于受试者的抗癌疗法或用于诊断/预后目的,如本文所述的实施例中所说明的。或者,该用途可被应用于来自病原生物体的细胞、或从犯罪现场获得的细胞、或来源于病原生物体或犯罪现场的少量无细胞的rna和/或无细胞的dna。
在第四方面,提供了根据第一或第二方面的方法用于验证大规模转录组学数据集的用途。例如,该用途可被用于确认已使用小规模样品(如包含单细胞或低数目细胞(例如约2到约100个细胞)或少量rna的生物样品)产生的通过高通量rna测序实验获得的特定结果。排除假阳性结果并使用独立方法验证所述结果是重要的。该方法可用于验证通过高通量rna测序产生的rna表达和/或突变谱结果。
在第五方面,提供了用于进行根据第一或第二方面的方法或用于根据第三或第四方面的用途的试剂盒,其中所述试剂盒包含:
(a)选自由以下组成的组的引物:
i.所述第一方面的所述方法的步骤(a)的所述反向引物或所述第二方面的所述方法的步骤(b)的所述反向引物,
ii.所述第一方面的所述方法的步骤(b)(i)的所述正向引物或所述第二方面的所述方法的步骤(c)(i)的所述正向引物,
iii.所述第一方面的所述方法的步骤(b)(ii)的所述引物对或所述第二方面的所述方法的步骤(c)(ii)的所述引物对,和
iv.所述第一方面的所述方法的所述巢式引物和巢式引物对或所述第二方面的所述方法的步骤(d)的所述巢式引物和巢式引物对;
(b)选自由以下组成的组的一种或多种试剂:
i.逆转录酶和一种或多种适合的反应缓冲液,其用于所述第一方面的所述方法的步骤(a)中的所述逆转录或所述第二方面的所述方法的步骤(b)中的所述逆转录,
ii.dna聚合酶和一种或多种适合的反应缓冲液,其用于所述第一方面的所述方法的步骤(b)或所述半巢式或巢式pcr中的所述扩增或所述第二方面的所述方法的步骤(c)或(d)中的所述扩增,和
iii.一种或多种经标记或未经标记的选自由datp、dctp、dgtp和dttp或dutp组成的组的脱氧核糖核苷酸;和
(c)使用说明,其用于进行根据第一或第二方面的方法或用于根据第三或第四方面的用途。
根据第五方面的试剂盒可包括一种或多种溶解缓冲液,用于在第一方面的方法之前或在第二方面的方法的步骤(a)中溶解样品中的细胞。可使用本领域通常使用的溶解缓冲液,例如碱性溶解缓冲液或含有蛋白酶k的细胞溶解缓冲液、或只是这样的缓冲液,其含有将破坏细胞并允许其核酸在溶液中释放的洗涤剂或化合物和/或酶。
根据第五方面的试剂盒还可包括用于定量实时pcr的探针或染料。示例性的探针和染料包括但不限于sybr绿色染料、evagreen、dsgreen、taqman探针、杂交探针等。
试剂盒还可包括用于设计第五方面的试剂盒的组分(a)的一种或多种引物和/或优化第一方面的方法的步骤(b)或第二方面的方法的步骤(c)的预扩增循环条件的使用说明。
在一个实施方案中,将引物和/或试剂以适于上述溶解、预扩增和扩增步骤的组合预混合。在另一实施方案中,将引物以适于分析基因表达谱或突变标记(signature)的组合预混合。引物可以是已被设计用于扩增一个或多个目标靶基因的引物。
在又一实施方案中,试剂盒包括一个或多个容器,所述容器包含用于进行上述方法和/或用途的一种或多种反应缓冲液。在一些实施方案中,试剂盒包括可提供于cd上的用于商业pcr仪器(如lifetechnologies7500fastdx或cepheid
除非上下文另有明确规定,否则如本文所用的单数形式“一(a、an)”和“所述”包括复数指示物。例如,术语“引物”包括多种引物,包括其混合物。
词语“基本上”并不排除“完全”,例如“基本上不含”y的组合物可完全不含y。在需要的情况下,可从本发明的定义中省略词语“基本上”。
除非另有说明,否则术语“包含(comprising和comprise)”和其语法变体意在表示“开放”或“包含性”用语,使得它们包括列举的要素,而且还允许包括额外的、未提及的要素。
如本文所用的术语“约”在制剂的组分的浓度的背景下,通常意指所述值的+/-5%,更通常所述值的+/-4%,更通常所述值的+/-3%,更通常所述值的+/-2%,甚至更通常所述值的+/-1%,且甚至更通常所述值的+/-0.5%。
在本公开通篇内,某些实施方案可以范围形式被公开。应当理解,呈范围形式的描述仅仅是为了方便和简洁且不应当被解释为对所公开范围的范畴的不可改变的限制。因此,对一个范围的描述应当被认为已经具体地公开了所述范围内的所有可能的子范围以及单个数值。例如,对一个范围(例如1到6)的描述应当被认为已经公开了子范围(例如1到3、1到4、1到5、2到4、2到6、3到6等)以及所述范围内的单个数字(例如,1、2、3、4、5和6)。不管所述范围的大小如何,这都是适用的。
某些实施方案也可在本文中被宽泛地和一般地描述。落入该一般公开的每个较窄的类和次一般的组合也构成本公开的一部分。这包括带有将任何主题从一般性中去除的附带条件或负面限制来一般性地描述实施方案,而不管被去除的内容是否在本文中明确述及。
实施例
实施例1-引物设计和scrmpcr方案
在来自其它地方已经描述的来源的方法中已经实施了以下方案步骤或材料:
1)peixoto等中所述的条件,具体地说:
a.使用反向特异性引物进行逆转录酶反应。
b.使用半巢式pcr进行rna转录物定量。
2)来自cellsdirecttm一步法qrt-pcr试剂盒(invitrogen)的材料,具体地说:
a.2x反应混合物目录编号11753-100
b.目录编号11753-100中所含的
3)来自biomarkhd系统(fluidigm)(sanchez-freire等)和http://www.fluidigm.com/home/fluidigm/docs/appnote_2v1step_pf9.pdf)中的方案pn100-4109所述的条件,
具体地说:
a.在2x反应混合物中沉积单细胞。
b.预扩增步骤的退火时间(4分钟)。
引物设计方案
用于在多重测试(一直到单细胞检测)中检测少量rna的引物设计方法
为了设计在单细胞规模的基因组dna存在下用于mrna扩增的引物,在基因组dna上引物之间必须存在内含子序列,或者至少一个引物必须位于外显子-外显子接合部。此外,引物需要是特异性的,不应当扩增不想要的产物。
该方案可用于依照peixoto等的一般方法(http://www.ncbi.nlm.nih.gov/pubmed/15466292)来产生用于半巢式/巢式pcr的引物。rna来源的基因转录物的稳健扩增需要半巢式pcr。基因组dna的稳健扩增需要巢式pcr。
程序:
使用以下工具(通过在firefox中打开):
1)位于http://www.ncbi.nlm.nih.gov/tools/primer-blast/的ncbi引物设计工具
该软件基于用于最佳引物对查找的primer3和blast。它还可以在外显子-外显子接合部处设计引物。
半巢式pcr:对于外部引物对,取决于样品类型和研究目的,所用的参数可相应变化。
2)位于http://www.ensembl.org/homo_sapiens/info/index的ensembl基因组浏览器
该网页工具允许进行直接的基因选择和保管式(curated)id检索。
3)位于http://www.premierbiosoft.com/jsp/marketing/freetoollogin.jsp?pid=3的netprimer
该网页工具允许通过对引物相应地评级来检查其质量。
使用上述网页工具应用以下步骤:
a)-在ensembl中搜索目标基因
b)-检查正确的鉴别,并点击“外部参考”
c)-向下滚动并鉴别refseq标识符
d)-将标识符复制并粘贴在ncbi底物设计工具窗口上
e)-使用上文提供的链接或定制的严格性来寻找引物
f)-在netprimer应用程序上检查引物,并通过评级来选择最佳引物对。
g)-如果没有返回引物,则使用较不严格的参数。
实施例:cd45引物的设计.
正向引物aagacaacagtggagaaaggacgca(seqidno:137)
反向引物cagtgggggaaggtgttgggct(seqidno:138)
产物长度159
模板nm_002838.3上的外显子接合部131/132(正向引物)
为了寻找内部探针,可使用具有更高评级的引物进行与上述相同的搜索,并限制pcr产物长度来取得位于pcr产物中的引物。
对于基因组区域上的引物,应用以下步骤:
a)在ensembl中寻找目标基因
b)点击序列
c)选择用于测序的基因组区域,包括内含子区域
d)复制并粘贴在primer-blast上
e)寻找靶向目标区域的引物
设计了两对引物,一对退火温度(t)为60℃,一对退火t为65℃。
对于内部探针的设计,通过引物组一选择扩增区域,将其复制并粘贴在primer-blast中。选择退火温度为60℃的引物。
引物设计:
使用primer-blast(ye等和bbenek等)并使用上述引物设计方案设计引物。对于每个rna转录物,设计了跨越外显子-外显子边界的引物或由相应基因组dna区域上的至少一个内含子隔开的引物。这允许引物特异性扩增rna转录物,而dna区域不会被扩增。此外,设计用于突变分析的引物以结合靶基因的内含子区域。这将避免可能已由非特异性或特异性逆转录引起的cdna转录物的扩增。(参见引物设计方案(上文)和表1a到1c)。
表1a:用于scrm-pcr测定(预扩增步骤)中的引物
参考文献和图例说明
(1)来源于hesse等(2001)j.cellsci.114,2569
(2)来源于sakaizawa等br.j.cancer(2012)106,939
图例说明:
c:结肠特异的
emt:上皮-间充质转化标志物
l:谱系标志物
g:基因
qc:质量控制
tec:肿瘤内皮细胞标志物
s:干细胞标志物
ep:上皮标志物
he:造血细胞标志物
en:内皮细胞标志物
me:巨核细胞/血小板标志物
mo:单核细胞-巨噬细胞标志物
er:红细胞标志物
表1b:用于巢式和半巢式扩增(扩增步骤)的scrm-pcr测定中的引物
参考文献和图例说明
(1)来源于hesse等(2001)j.cellsci.114,2569
(2)来源于sakaizawa等br.j.cancer(2012)106,939
表1c:用于scrm-pcr测定中的引物(扩增产物和qpcr熔解曲线峰值)
参考文献和图例说明
(1)来源于hesse等(2001)j.cellsci.114,2569
(2)来源于sakaizawa等br.j.cancer(2012)106,939
scrmpcr方案
1)将单细胞在5μl2x反应缓冲液(cellsdirecttm一步法qrtpcr试剂盒(invitrogen))中显微操作并储存在-80℃直到使用。
2)使用
3)接下来通过添加正向引物混合物(匹配步骤2中添加的反向引物)和对dna区域特异性的引物对进行预扩增轮次。
4)类似于如来自biomarkhd系统(fluidigm)的protocolpn100-4109所述的进行预扩增,其中使用4分钟退火时间,但将退火温度改变为以下循环步骤:
6x循环:4',60℃-1',95℃
6x循环:4',55℃-1',95℃
6x循环:4',50℃-1',95℃
5)使用标准方法(例如纯化柱如axygenaxypreppcr清除试剂盒-目录编号14-223-018)从样品中清除引物。
6)将样品以1/20-1/50稀释并储存在-20℃直到进一步使用。这产生至少200μl的预扩增样品,这足以进行100次反应。
7)对于rna转录物定量,依照制造商方案,使用来自5)的2ul预扩增反应物、根据靶转录物的半巢式引物对(表1b)和sensifastsybrlo-rox试剂盒(bioline)在viia7仪器(appliedbiosystems)上进行定量pcr。为了分析所选的dna突变热点,依照制造商说明书,通过使用来自5)的2ul预扩增反应物、巢式pcr引物对(表1b)和含有校正聚合酶的主混合物(kodmastermix,emdmillipore)进行pcr。在琼脂糖凝胶上分离pcr产物,切下特异性条带并使用桑格法测序。可使用任何qpcr方法和列出的引物进行步骤7。可使用具有校正活性的任何taq聚合酶进行用于dna序列的pcr,并且还可使用诸如等位基因特异性pcr(aspcr)或高分辨率解链温度pcr(hrm)的qpcr技术进行突变检测。
实施例2-早期结直肠癌患者中普遍存在的肿瘤来源的内皮微栓子
患者样品和临床数据。所有受试者均已给出参与的知情书面同意。根据由新加坡国立大学、富斯外科医院(fortissurgicalhospital)和新加坡保健集团(singaporehealthservices)(singhealth)的机构审查委员会(institutionalreviewboard)(irb)批准的方案,于2012年7月到2014年4月间获得了临床样品。来自82位crc患者的连续血液样品由富斯外科医院(fsh)和新加坡国家癌症中心(nationalcancercenter,singapore)(ncc)提供。来自45位健康受试者的血液样品由新加坡队列研究协会(singaporeconsortiumofcohortstudies)(sccs)提供。将所有样品收集在edta真空采血管(vacutainertube)(becton-dickinson)中,并在生物工程和纳米技术研究所(instituteofbioengineeringandnanotechnology)于6小时内处理。两例由于微滤装置的技术故障而被排除分析。在可能的任何情况下,将匹配的肿瘤和转移性样品在切除后立即冷冻,并储存在-80℃直到使用。参与受试者的临床病理数据描述于补充表6中,并在ctm计数完成后被回溯性地收集。在不事先知晓ctm计数的情况下进行临床数据收集。同样,除了fsh样品的诊断和术前状态之外,在ctm计数时对crc患者的临床数据尚不了解。通过宽度×长度计算肿瘤面积。
细胞系和培养。hct116、colo201、sw480、sw620、dld-1和rko结直肠癌细胞系、bj-5ta永生化人包皮成纤维细胞和huvec来自atcc。在第1次传代和第2次传代使用huvec,并在egm-2培养基(lonza)中培养huvec。将所有其它细胞系在补充有10%fbs的dmem(lifetechnologies)中培养。将细胞维持在存在5%co2的37℃的加湿培育箱中。
装置制作和组装。如所述(lim等)制作硅微筛。简单地说,微筛由具有7.3mm的总直径
微滤。为了优化血液微滤,将5μmcelltracker(lifetechnologies)标记的细胞以每毫升全血10-50个细胞添加到供体血液中。借助蠕动泵(ismatec)以各种流速对血液进行过滤。在使用pbs、0.5%bsa和2mmedta洗涤6次后,将细胞再悬浮于培养基中。随后,使用hoechst33342(lifetechnologies)对细胞核进行染色,并回收细胞以确定污染性wbc的回收效率和倍数耗尽。在一些实验中,还对残留在微筛上的celltracker阳性细胞进行计数。
回收效率百分比计算如下:
回收效率%=(回收的细胞)×100/(掺入的细胞)
来自1ml全血的倍数耗尽计算如下:
倍数耗尽=(全血中的wbc)/(微滤液中的wbc)
微滤液中的wbc计数被定义为在实验富集情况下的任何hoechst33342阳性、celltracker阴性事件的数目或在临床样品分析情况下基于任何cd45阳性事件。使用图5b和c中描述的优化参数,立即针对所示的下游应用处理所有临床样品。为了估计理想的目标wbc耗尽,进行了含有50个celltracker阳性hct116细胞的pbmc的连续稀释的显微操作。五千倍的耗尽允许对没有污染白细胞的纯hct116细胞进行显微操作。基于对现有无标签ctc分离装置的文献搜索(cima等)来选择理想的目标回收效率。使用用于每个装置的2ml全血和优化的微滤条件进行临床样品的微滤。
筛上免疫荧光(on-sieveimmunofluorescence)。在含有0.5%bsa、2mmedta和人fcr封闭试剂(miltenyibiotec)的pbs中洗涤5次后,使用以下荧光标记的抗体将悬浮细胞直接“筛上”染色30min):抗-cd451:200(克隆2d1;ebioscience)、抗-ep-cam1:20(9c4,biolegend)、抗-cd311:20(wm59,biolegend)、抗-cd1441:10(55-7h1,bd)、抗-cd411:20(hip8,biolegend)、抗-cd42b1:20(hip1,biolegend)。对于细胞内抗原,使用内部染色试剂盒(insidestainkit)(miltenyibiotec)和人fcr封闭试剂与以下抗体:抗-vwf1:200(兔多克隆a0082,dako,其使用lifetechnologiesapex抗体标记试剂盒在内部缀合到alexa488或alexa555)、抗波形蛋白(v9,santacruzbiotechnology)、抗泛细胞角蛋白(anti-pancytokeratin)(c11,cellsignalingtechnology)。使用hoechst33342(lifetechnologies)对核进行染色。在一些实验中,使用钙黄绿素am(lifetechnologies)来鉴别活细胞。在洗涤步骤后,在倒置荧光显微镜(ix81,olympus)下在悬浮液中回收细胞并使其显现,以供用于成像、计数和/或显微操作。使用metamorph软件(moleculardevices)与coolsnaphq2ccd照相机(photometrics)记录图像。
ctm定义和计数。本研究中通过“主轴>10μm且具有至少2个明显区分的核的任何cd45-细胞或cd45-细胞簇”来定义ctm。在本文所述的研究中,所有这样定义的细胞均对内皮标志物cd31、cd144或vwf染色呈阳性(图7b)。重要的是,排除具有大而分叶状的单核或大而圆的单核、属于巨核细胞谱系的细胞群体。这些细胞具有容易与ctm辨别的特征性细胞形态,对cd41和cd42b染色呈阳性,并且主要在经受治疗的crc患者中观察到,而且也在一些健康志愿者和未治疗crc患者中观察到。单个内皮细胞由于直径较小而允许它们通过微筛而也被排除在分析之外。通过将从2ml全血获得的微滤液添加到96孔板的孔中,通过应用这些纳入和排除准则来对ctm进行计数。在短暂的离心步骤后,通过使用20×物镜手动扫描靶孔三次来鉴别ctm并对于其进行计数。通过检测到至少一个ctm来定义阳性样品。
靶细胞鉴别、显微操作和储存。使用连接到25-ml注射器的口吸移液器手动微量吸移靶细胞。简单地说,借助明场图像、核染色和特异性荧光信号从总回收细胞中鉴别细胞。然后将靶单细胞或ctm微量吸移在10μl的洗涤缓冲液微滴中,随后沉积在0.2-mlpcr管中,所述管含有适当缓冲液:用于scrmpcr的5μl2×反应缓冲液(cellsdirect一步式qrt-pcr试剂盒,lifetechnologies)、用于全基因组扩增的2μlpbs或用于低输入rna-seq的2μlsuperblock缓冲液(thermoscientific)。将细胞立即储存在-80℃直到使用。在一些情况下,将全部微滤液离心沉淀下来,并储存在-80℃直到进一步使用。
单细胞rna和突变分析pcr(scrmpcr)。使用primer-blast(ye等)设计引物。对于每个rna转录物,设计了跨越外显子-外显子边界的引物或由相应基因组dna区域上的至少一个内含子隔开的引物。设计用于突变分析的引物以结合靶基因的内含子区域(表1a-c)。如以上实施例1中所述,图1中突出显示了scrmpcr的方法。该方法可用于在同一细胞中同时检测和定量rna转录物和对dna热点进行测序。简单地说,使用superscriptiii逆转录酶(invitrogen)和500nm靶反向引物混合物,将单细胞rna转录物在50℃逆转录30min。然后使用platinumtaqdna聚合酶(invitrogen)、通过添加针对转录物特异性反向引物的正向引物的匹配混合物和用于靶向基因组区域的引物对来进行预扩增轮次(表1a)。如下通过无延伸地交替退火和变性步骤进行预扩增循环:60℃,4min,95℃,1min,6×循环;55℃,4min,95℃,1min,6×循环;50℃,4min,95℃,1min,6×循环。使用axypreppcr清除试剂盒(axygen)进行引物清除。将样品以1/20稀释并储存在-20℃直到进一步使用。对于rna转录物定量,依照制造商方案,使用2μl预扩增反应物、根据靶转录物的半巢式引物对(表1b)和sensifastsybrlo-rox试剂盒(bioline)在viia7仪器(appliedbiosystems)上进行定量pcr。使用actb作为参考基因将相对基因表达归一化。为了分析所选的dna突变热点,依照制造商说明书,通过使用2μl预扩增反应物、巢式pcr引物对(表1b)和含有校正聚合酶的主混合物(kodhotstartmastermix,emdmillipore)进行pcr。对于肿瘤和正常组织中的kras外显子2测序(图4b),使用以下正向引物tttgtattaaaaggtactggtggag(seqidno:135)和反向引物cctttatctgtatcaaagaatggtc(seqidno:136)进行pcr扩增。在琼脂糖凝胶上分离pcr产物;切下特异性条带并使用桑格法测序。
核酸提取。依照制造商说明书,使用rnaqueous-micro总rna分离试剂盒(ambion)对全部微滤液或分离的细胞进行rna提取。使用rneasy小型试剂盒(qiagen)分离来自组织的总rna。使用dneasy小型试剂盒(qiagen)分离来自组织的dna。
ctm靶向重测序和阵列比较基因组杂交(acgh)。使用genomeplex单细胞全基因组扩增试剂盒(sigma)并依照制造商说明书对单ctm进行全基因组扩增。使用相同的程序扩增组织dna(50pg)样品。为了靶向重测序,设计靶向用于nras、ctnnb1、pik3ca、egfr、braf、pten、kras、akt1和tp53基因的外显子的一组定制基因(约6.1kb)。使用ionampliseqlibrarykit2.0(lifetechnologies)与10ng输入dna构建文库。在iontorrentpersonalgenomemachine(pgm)测序仪(lifetechnologies)上进行靶向重测序运行。在高严格性设置中使用iontorrentvariantcallerplugin识别(call)变体。利用制造商说明书和试剂,通过将250ngdna杂交到cytoscan750k阵列(affymetrix)来进行acgh。使用chas软件2.1版(affymetrix)对数据进行分析和可视化。
ctm和组织cdna合成和rna-seq。使用长距离pcr(ldpcr)分别利用25个循环和18个循环利用smarter超低rna试剂盒(clontechlaboratories)从单ctm和10pg组织rna合成cdna。对于每个样品,使用adaptivefocusedacoustics系统(covaris)剪切cdna。使用nebnextdnalibraryprepmastermix试剂盒(newenglandbiolabs)构建文库。使用唯一索引对所有文库进行条形编码,并将其汇集以供在illuminahiseq2000平台上运行rna测序。使用tophat(第2版)(trapnell等,2009)将数据映射到人类基因组第19版(hg19)。cufflinks(第2.2版)(trapnell,c.等,2010)用于将基因表达定量为fpkm(每百万映射读段中每千碱基转录物的片段数)。
rna-seq数据主要组分分析。进行对完整rna-seq数据集(图16)的主要组分分析。通过选择由它们在第1到第3主要组分中的最大载荷所分选的前300个基因来计算秩相关系数。从该列表中,计算每个ctm和组织的斯皮尔曼(spearman)秩相关系数(ρ),并将所得数据绘制为热图。通过平均连锁聚类生成树状图。
rna-seq数据谱系推断。用于谱系推理的工作流程呈现于图14a中,并以r脚本实现。简单地说,获得原代细胞atlas图谱数据集(gse49910)(mabbott等),并选择来自298个不同实验的表达数据,其对应于n=42种不同的细胞类型或“谱系”。对于每种谱系l中的每个基因g,基于香农(shannon)信息熵和由schug等引入的q统计量来计算“特异性指数”s,
其中p(l|g)是基因g在谱系l中的相对表达。通过使用biogps显现具有高特异性指数的基因的表达数据来确认基因特异性(图14b)。对于每种谱系,选择具有最高特异性指数的前80个基因(“特异性基因”)。选择80个基因,因为这提供了本文所报告的分析中的最佳分辨率。接下来,对于每个rna-seq样品,计算每种谱系所特有的基因的数目。同时,从affymetrixhg-u133_plus_2基因列表生成了80个随机选择的基因的1,000×列表(“随机基因”),并确定了每个实验rna-seq谱中偶然存在的基因的平均数目。最后,通过对每个实验样品中的每种测试谱系进行费希尔精确检验,检查富集的特异性基因的数目是否等于随机富集的基因的数量。将每个测试的比值比在包含所有测试谱系的热图中进行平均值中心化、缩放和可视化。最终结果用于基于归一化的比值比的分布产生关于细胞谱系的假设。该算法使用从各种细胞类型和组织产生的已公开的rna-seq数据集(图15)进行验证。
内皮祖细胞(epc)测定。如先前所述(kalka等,colombo等)进行集落形成epc测定。简单地说,通过cd144和钙黄绿素am荧光染色在2ml微滤液中对活的内皮ctm进行计数。然后在egm-2细胞培养基(lonza)的存在下,将来自第二装置的2ml血液的未染色的微滤液置于用纤连蛋白(1μg/cm2)(sigma-aldrich)涂覆的96孔板上用于培养。孵育前通过明场显微镜检查确认ctm的存在。如下使用huvec作为阳性对照:将10,000个huvec掺入2ml供体血液中,并通过微滤使用两个装置加以分离。在一个装置中,通过cd144和钙黄绿素am染色来定量所回收的huvec。将从另一装置回收的huvec以限定数目(5个、10个、20个、40个、80个和160个细胞)接种于一式八份孔中。2天后,通过每隔一天更换一半的培养基来更换培养基并使细胞生长总共30天。每周在明场显微镜检查下监测集落的存在和活力。30天后,通过胰蛋白酶消化(trypsinisation)将细胞分离,使用cd144抗体、钙黄绿素am和hoechst33342染色,并在ix81(olympus)倒置荧光显微镜下定量。
微血管密度和管腔(lumen)计数。使用如先前所述(wild等,gupta等)使用cd31染色的组织切片的免疫荧光图像并使用imagej(schneider等)进行微血管密度(mvd)计数。简单地说,将新鲜组织包埋在tissue-teko.c.t化合物(sakura)中并储存在-80℃直到进一步使用。从所有可用的组织,在聚-l-赖氨酸载玻片上切割5微米恒冷箱切片,将其在含有4%多聚甲醛的pbs中固定8min,在pbs中洗涤,并使用pe-抗cd抗体(1:20,克隆wm59,biolegend)染色。借助ix71显微镜系统(olympus)和metamorph软件(moleculardevices),利用10×物镜对每个组织切片的整个肿瘤区域进行成像。在成像之前以及整个mvd和管腔计数中,患者id被设盲以避免数据采集和分析期间的主观偏差。
从新鲜组织分离内皮细胞。如先前所述(vanbeijnum等),在对所述方案进行微小修改的情况下,从正常结肠和肿瘤组织分离内皮细胞。简单地说,如所述将新鲜的组织切碎并在37℃使用胶原酶、分散酶和dna酶消化60分钟。在菲科-帕克(ficoll-paque)密度离心步骤后,依照制造商说明书,使用macs试剂和材料(miltenyibiotec)进行两步磁选。首先,在用抗cd45磁珠和人fcr封闭试剂标记细胞后,通过ld柱上的阴性选择来耗尽表达cd45的细胞。接下来收集耗尽cd45的级分,并通过添加抗cd31磁珠和人fcr封闭试剂进行第二次标记。在使用ms柱进行阳性选择后,将具有富集的cd31+cd45-细胞的级分储存在-80℃直到进一步使用。
统计分析。在r环境(第3.1.0版)(rcoreteam等)中进行统计分析。在多重比较的情况下,使用双尾威-曼-怀三氏u检验和邦弗朗尼校正来测试未配对的样品。对于每个测试,使用“coin”包(zeileis等)计算具有位置参数(霍奇-斯莱曼(hodges-lehmann)估计值
分析结果
循环肿瘤微栓子(ctm)是从癌症患者取样的血液中通常观察到的细胞簇。50多年来,ctm已被普遍描述为恶性实体,并且它们的调查可能获得对于肿瘤进展和临床结果的深刻见解。然而,这些结构的综合表征尚未被呈现。这里显示,与目前的共识相反,从结直肠癌患者分离的ctm不是癌性的,而是代表了肿瘤来源的内皮细胞的不同群体。ctm不反映匹配肿瘤的遗传变异,但ctm表达上皮和间充质转录物,这与先前关于循环肿瘤细胞分型的报道一致。单ctm的转录组分析揭示它们作为内皮细胞的特征,进一步的结果指示它们的肿瘤来源和成熟表型。在从术前的早期癌症患者取样的血液中发现内皮ctm的广泛存在,但在健康供体中未发现,表明内皮ctm计数作为用于结直肠癌的潜在指标。内皮ctm不应与真正的循环肿瘤细胞混淆,尽管它们的分析可能在诊断上有用,并提供关于在治疗和疾病进程期间潜在肿瘤血管系统的直接信息。
为了分析循环肿瘤细胞(ctc)的转录谱和遗传谱,开发了能够实现ctc的回收和下游显微操作两者的、无标签的、基于大小的微滤装置(图5a、图5b和图9a-c)。通过向1ml供体血掺加30个sw620细胞(中值大小与ctc类似的结直肠癌(crc)细胞系)来优化ctc的富集和回收效率(图9d)。使用0.25ml/min-1的流速和9-10μm的孔径,获得了回收效率和细胞纯度之间的最佳权衡(tradeoff)。这产生了>90%的sw620回收效率与白细胞的>5×103倍耗尽(图5c),从而允许除细胞计数之外的各种下游应用(图5b、图9f和图10a)。使用另外的crc细胞系获得了类似的结果(图9f)。
接下来测试来源于5位患者的先导系列的微滤液,并针对上皮细胞粘附分子(ep-cam)(典型的ctc标志物)和cd45(白细胞的标志物)染色。未检测到ep-cam阳性细胞。然而,注意到存在ep-cam-/cd45-细胞的大的异常簇(图10b)。首次描述于1959年,许多研究已报道了恶性循环细胞簇或循环肿瘤微栓子(ctm)(补充表1)。已分离的ctm展示与恶性肿瘤一致的细胞形态,如不典型的核、明显的核仁和高核质比(图10a-c)。另外,缺乏ep-cam染色表明来自上皮表型的漂变(drift)(先前针对ctc所报告的事件)。因此假设ctm带有反映原发性肿瘤的dna改变。为了确认在经历上皮-间充质转化(emt)的单细胞中存在dna突变,建立了如本文所述的聚合酶链式反应(pcr)方案,用于在单细胞规模上同时定量rna转录物和检测dna突变(单细胞rna和突变分析pcr或“scrmpcr”)(图1、图11、表1a-1c)。来源于4位患者的9个ctm中的scrmpcr揭示上皮和间充质标志物(包括serpine1、foxc1和krt8)的存在,这与先前针对乳腺癌ctc所报道的上皮-间充质谱一致(图4a和图6a)。通过panck和波形蛋白免疫染色(图11a和cima等)来确认这些结果。接下来针对相应原发性肿瘤中存在的突变对这些ctm进行测序。令人惊讶的是,所有测试的dna序列热点均匹配野生型等位基因(图4b)。将靶向高通量dna测序进一步应用于从16个单ctm(6位患者)和匹配的肿瘤组织扩增的dna中的8个共同突变基因。再次,不能检测到肿瘤组织和相关ctm之间的匹配突变(补充表2和3)。使用来自12个ctm(4位患者)的扩增dna,接下来进行阵列比较基因组杂交(acgh)。实际上,来自肺癌患者的ctc已被证明可再现地反映癌组织拷贝数变异(ni.x等)。这里,与匹配的原发性肿瘤相对比,ctm反而具有正常的细胞遗传学特征(图6b-c和图11b-c)。总之,来自10位患者的26个ctm的单细胞规模分析尽管展示了上皮-间充质标志物表达,但并未反映匹配肿瘤组织中发现的dna异常。这表明与肿瘤上皮无关的ctm来源。
接下来,通过高通量测序(rna-seq)对来自8位患者的18个单ctm和匹配的正常结肠和肿瘤组织进行rna表达谱分析(图12、图16、补充表4)。进一步开发了用于从转录谱推断细胞谱系的工作流程(图14、图15)。在包括42种不同细胞类型的比较中,所有ctm转录组均与内皮谱系的细胞类型相关(图7a)。在另外的14个ctm中通过scrmpcr确认了一系列内皮谱系标记物与一般emt标志物的存在(图4c)。内皮细胞被认为是特化的上皮,并且已知其表达波形蛋白(通常用作间充质标志物)和各种角蛋白(上皮的经典标记物)。所有ctm,包括具有恶性细胞形态的那些,均无例外地对内皮标志物如cd31、vwf或cd144染色(图7b),但对cd45或巨核细胞谱系标志物cd41和cd42b为阴性。这表明在crc患者中,检测到的所有ctm均为内皮来源。另外,如先前其它地方所报道的,未检测到ctm内的单个肿瘤细胞。当前的发现与el-heliebi等(他们报道了来自肾癌患者的循环非血液细胞(cnhc)上的cd31表达)一致,但不同于描述恶性来源的ctc簇的最近报道(aceto等)。aceto等描述的从ctc-簇的rna-seq数据的谱系推断实际上指示上皮来源的细胞的存在(图15)。因此本研究中所表征的ctm代表crc患者中循环内皮细胞簇的不同群体。
内皮祖细胞克隆形成实验(kalka等和colombo等)揭示了ctm的成熟表型,活的ctm在30天时期内未能在纤连蛋白底物上增殖(图7c)。ctm来源的rna-seq数据的主要组分分析显示相对于正常结肠组织转录组,与肿瘤组织转录组的更强相关性(图7d)。为了测试ctm是否为肿瘤来源,在手术肿瘤切除前0-24h和手术肿瘤切除后24-72h收集来自17位crc患者的配对样品(n=34)。肿瘤去除引起内皮ctm急剧下降,支持肿瘤与ctm之间的直接联系(图7e和补充表5)。叶酸水解酶(folh1)(编码前列腺特异性膜抗原(psma)的基因)在各种癌症类型的肿瘤血管系统中特异性表达,但在正常血管系统和外周血中不存在。folh1确实在从新鲜crc组织分离的cd31+cd45-细胞和从7/10crc患者的血液分离的ctm中表达,但不在从正常组织分离的内皮细胞或健康供体外周血单核细胞(pbmc)中表达。该结果进一步支持了内皮ctm的肿瘤来源(图7f)。另外,ctm的rna-seq数据揭示了几种肿瘤内皮标志物(包括cd276)的表达(图7g和图17)。进一步通过对来源于具有低或高ctm计数的患者的肿瘤组织中的血管进行计数来探询ctm数目是否可能与潜在的肿瘤血管系统的特征相关联。尽管血管单位的中位数没有差异,但管腔的中位数在具有高ctm计数的患者中显著更高(图7h)。总之,显示结直肠癌患者中的ctm不是恶性实体,而是肿瘤来源的成熟内皮细胞簇。
由于ctm与原发性肿瘤之间的上述相关性,因此接下来探询内皮ctm是否为crc的信息指标。对来自125位受试者(45位对照健康志愿者和一系列连续80位crc患者,包括来自上述患者的ctm计数)的总共141份临床标本的内皮ctm进行计数。在76.2%(61/80)的crc患者中,但仅在2.2%(1/45)的健康个体中观察到至少一个内皮ctm(图8a)。发现未治疗患者与经历用于crc的治疗干预的患者相比,呈现出显著更高的内皮ctm计数(n=80)(图8b)。然而,内皮ctm计数与临床参数如肿瘤分期、分级或远端转移的存在(图8b、补充表6和7)或其它变量(包括炎症标志物)(图18)无关。具体地说,时间序列分析中的内皮ctm数目指示手术切除事件对ctm分布具有最强影响,从而确认了图7e中的结果,并且进一步支持内皮ctm与原发性肿瘤的存在的相关性(图8c)。86.5%的未治疗患者(45/52)中存在内皮ctm,但只有2.2%的健康对照(1/45)中存在内皮ctm指示ctm计数可能在辅助crc诊断是有用的。比较未治疗患者和健康对照组的接受操作者特征(receivingoperatorcharacteristic)(roc)曲线的曲线下面积(auc)为0.930(图8d)。引人注目的是,具有低病理性肿瘤分期(分期≤iia)的crc患者在86.4%(19/22)的病例中也对内皮ctm为阳性,auc=0.922(图8e)。总之,这些结果进一步确认了内皮ctm计数与原发性肿瘤的存在之间的相关性。此外,内皮细胞ctm在未治疗患者而不在健康个体中的普遍存在指示内皮ctm计数作为用于crc的诊断辅助手段的潜在用途。
总之,本文报告了来自结直肠癌患者的单循环肿瘤微塞子(ctm)的分离、回收和分析。首次呈现了单ctm的转录组谱分析(profiling),并且与目前的共识相对比,提供了ctm的肿瘤内皮来源的几条证据。内皮ctm被检测为多细胞结构。因此,ctm可能是从经历病理血管发生(crc肿瘤进展中公认的早期事件)的混乱肿瘤血管系统脱落的。临床前模型可能揭示循环中的肿瘤内皮细胞脱落的潜在机制,并且目前处于研究之中。与经常在晚期疾病患者中检测到的ctc相反,ctm是在早期和术前crc患者中普遍存在的肿瘤来源的实体。因此,内皮ctm计数是用于早期crc检测的有吸引力的方式。在本研究中,未如aceto等所报道的检测到ctc簇的存在。这可能是患者谱(profile)差异的结果。实际上,aceto等分析了来自终末乳腺癌患者的血液样品,而本研究中的血液样品主要来源于术前crc患者。进一步的研究将需要解决各种疾病中循环内皮细胞簇的特异性。有趣的是,组织特异性分子标记已在来自各种器官的内皮细胞中被证实,表明ctm可基于特定基因集的表达追溯到它们的来源器官。由于它们暗示恶性肿瘤的细胞形态、角蛋白表达和混合的上皮和间充质标志物,内皮ctm不应与经历emt的真正恶性ctc混淆。同时,内皮ctm分析可能有助于早期结直肠癌检测,并提供关于治疗和疾病进程期间潜在肿瘤血管系统的直接信息。
补充表1:包含描述为癌性实体的循环肿瘤细胞簇或ctm的所选出版物(1959-2014)
补充表4:rna-seq数据,到hg19外显子的独特映射读段
图例说明:
p,患者
t,肿瘤组织
c,中央
d,深部
s,浅表
n,正常组织
met,转移
ctm,循环肿瘤微栓子
补充表5:术前和术后ctm计数。来自图8e的数据
术前:术前0-24hr血液中的ctm计数
术后:术后24-72hr血液中的ctm计数
补充表6:基线患者和健康供体特征
*不包括来自图3e的术后数据
补充表7:本研究中分析的各基线样品类型的ctm计数和单ctm的数目
补充表7:本研究中分析的各基线样品类型的ctm计数和单ctm的数目
补充表7:本研究中分析的各基线样品类型的ctm计数和单ctm的数目
图例说明:
nuh,新加坡国立大学医院
ibh,新加坡生物工程和纳米技术研究所
fsh,新加坡富斯外科医院
ncc,新加坡国家癌症中心
crc,结直肠癌
早期,分期≤iia
*不包括来自图3f的术后样品
参考文献
aboulafia,d.m.carcinocythemia.转移性乳腺癌的终末表现(aterminalmanifestationofmetastaticbreastcancer).west.j.med.157,672–674(1992).
allard,w.j.等,肿瘤细胞在所有主要癌症的外周血中循环,但不在健康受试者或非恶性疾病患者中循环(tumorcellscirculateintheperipheralbloodofallmajorcarcinomasbutnotinhealthysubjectsorpatientswithnonmalignantdiseases).clin.cancerres.10,6897–6904(2004).
aceto,n.等,循环肿瘤细胞簇是乳腺癌转移的寡克隆前体(circulatingtumorcellclustersareoligoclonalprecursorsofbreastcancermetastasis).cell158,1110–1122(2014).
bbenek,k.,kunkel,a.t.,(1993).“逆转录病毒逆转录酶的保真性(thefidelityofretroviralreversetranscriptases)”.inskalka,m.a.,goff,p.s.reversetranscriptase.newyork:coldspringharborlaboratorypress.第85页.isbn0-87969-382-7.
borgia,b.等,用于鉴别肝转移的血管标志物的蛋白质组学方法(aproteomicapproachfortheidentificationofvascularmarkersoflivermetastasis).cancerres.70,309–318(2010).
champely,s.pwr:用于检定力分析的基本函数(pwr:basicfunctionsforpoweranalysis).(rfoundationforstatisticalcomputing,vienna,2009).
cima,i.等,微流体装置中循环肿瘤细胞的无标记分离:目前的研究和观点(label-freeisolationofcirculatingtumorcellsinmicrofluidicdevices:currentresearchandperspectives).biomicrofluidics7,011810(2013).
cohen,j.用于行为科学的统计检定力分析(statisticalpoweranalysisforthebehavioralsciences).(l.erlbaumassociates,1988).
cole,w.h.,roberts,s.s.,webb,r.s.,strehl,f.w.和oates,g.d.特别强调血管扩散和植入的癌症播散(disseminationofcancerwithspecialemphasisonvascularspreadandimplantation).ann.surg.161,753–770(1965).
colombo,e.,calcaterra,f.,cappelletti,m.,mavilio,d.和dellabella,s.纤连蛋白和胶原在支持内皮祖细胞从人成年外周血的分离和扩增方面的比较(comparisonoffibronectinandcollageninsupportingtheisolationandexpansionofendothelialprogenitorcellsfromhumanadultperipheralblood).plosone8,e66734(2013).
coumans,f.a.w.,vandalum,g.,beck,m.和terstappen,l.w.m.m.影响来自全血的循环肿瘤细胞富集的过滤特征(filtercharacteristicsinfluencingcirculatingtumorcellenrichmentfromwholeblood).plosone8,e61770(2013).
cho,e.h.等,在上皮肿瘤患者中鉴别的循环肿瘤细胞聚集体的表征(characterizationofcirculatingtumorcellaggregatesidentifiedinpatientswithepithelialtumors).phys.biol.9,016001(2012).
desitter,i.等,一种用于通过稀有循环肿瘤细胞的大小和表征快速分离的新装置(anewdeviceforrapidisolationbysizeandcharacterizationofrarecirculatingtumorcells).anticancerres.31,427–441(2011).
ejeckam,g.c.,sogbein,s.k.和mcleish,w.a.由于肺转移性燕麦细胞癌引起的癌细胞血症(carcinocythemiaduetometastaticoat-cellcarcinomaofthelung).can.med.assoc.j.120,336–338(1979).
engell,h.c.血液中的癌细胞;5到9年的随访研究(cancercellsintheblood;afivetonineyearfollowupstudy).ann.surg.149,457–461(1959).
el-heliebi,a.等,形态学准则足以鉴别肾癌中的循环肿瘤细胞吗(aremorphologicalcriteriasufficientfortheidentificationofcirculatingtumorcellsinrenalcancer)?j.transl.med.11,214(2013).
finkel,g.c.和tishkoff,g.h.外周血涂片中的恶性细胞:病例报告(malignantcellsinaperipheralbloodsmear:reportofacase).n.engl.j.med.262,187–188(1960).
glaves,d.,huben,r.p.和weiss,l.来自人肾腺癌的细胞的血源性播散(haematogenousdisseminationofcellsfromhumanrenaladenocarcinomas).br.j.cancer57,32–35(1988).
griffiths,j.d.,mckinna,j.a.,rowbotham,h.d.,tsolakidis,p.和salsbury,a.j.结肠和直肠癌:循环恶性细胞和5年存活(carcinomaofthecolonandrectum:circulatingmalignantcellsandfive-yearsurvival).cancer31,226–236(1973).
gupta,g.p.等,被选择用于肺转移中的依序步骤的血管重塑介质(mediatorsofvascularremodellingco-optedforsequentialstepsinlungmetastasis).nature446,765–770(2007).
herbert,j.m.j.,stekel,d.,sanderson,s.,heath,v.l.和bicknell,r.使用应用于肿瘤内皮基因鉴别的多个cdna文库的新型差异基因表达分析方法(anovelmethodofdifferentialgeneexpressionanalysisusingmultiplecdnalibrariesappliedtotheidentificationoftumourendothelialgenes).bmcgenomics9,153(2008).
hofman,v.j.等,使用通过上皮肿瘤细胞大小法加以分离对循环肿瘤细胞的细胞病理学检测:前景和误区(cytopathologicdetectionofcirculatingtumorcellsusingtheisolationbysizeofepithelialtumorcellmethod:promisesandpitfalls).am.j.clin.pathol.135,146–156(2011).
hofman,v.等,使用通过上皮肿瘤细胞大小法加以分离对肺癌患者进行术前循环肿瘤细胞检测是一种新的预后生物标志(preoperativecirculatingtumorcelldetectionusingtheisolationbysizeofepithelialtumorcellmethodforpatientswithlungcancerisanewprognosticbiomarker).clin.cancerres.17,827–835(2011).
hou,j.-m.等,循环肿瘤细胞、计数和其它(circulatingtumorcells,enumerationandbeyond).cancers(basel).2,1236–1250(2010).
hou,j.-m.等,循环肿瘤细胞作为肺癌中的转移生物学的窗口(circulatingtumorcellsasawindowonmetastasisbiologyinlungcancer).am.j.pathol.178,989–996(2011).
hou,j.m.等,循环肿瘤细胞和循环肿瘤微栓子在小细胞肺癌患者中的临床意义和分子特征(clinicalsignificanceandmolecularcharacteristicsofcirculatingtumorcellsandcirculatingtumormicroemboliinpatientswithsmall-celllungcancer).j.clin.oncol.30,525–532(2012).
kalka,c.等,用于治疗性新生血管形成的离体扩增的内皮祖细胞的移植(transplantationofexvivoexpandedendothelialprogenitorcellsfortherapeuticneovascularization).proc.natl.acad.sci.usa97,3422–3427(2000).
khoja,l.等,探讨胰腺癌中作为新型生物标志物的循环肿瘤细胞的先导研究(apilotstudytoexplorecirculatingtumourcellsinpancreaticcancerasanovelbiomarker).br.j.cancer106,508–16(2012).
kling,j.不止是对肿瘤细胞计数(beyondcountingtumorcells).nat.biotechnol.30,578–580(2012).
krebs,m.g.等,使用上皮标志物依赖和非依赖性方法对非小细胞肺癌患者中的循环肿瘤细胞的分析(analysisofcirculatingtumorcellsinpatientswithnon-smallcelllungcancerusingepithelialmarker-dependentand-independentapproaches).j.thorac.oncol.7,306–315(2012).
lim,l.s.等,用于循环肿瘤细胞的快速计数和荧光原位杂交的微筛实验室芯片装置(microsievelab-chipdeviceforrapidenumerationandfluorescenceinsituhybridizationofcirculatingtumorcells).labonachip12,4388–4396(2012).
mabbott,n.a.,baillie,j.k.,brown,h.,freeman,t.c.和hume,d.a.人原代细胞的表达图谱:从共表达网络推断基因功能(anexpressionatlasofhumanprimarycells:inferenceofgenefunctionfromcoexpressionnetworks).bmcgenomics14,632(2013).
marrinucci,d.等,转移性前列腺癌、胰腺癌和乳腺癌患者中的流体活组织检查(fluidbiopsyinpatientswithmetastaticprostate,pancreaticandbreastcancers).phys.biol.9,016003(2012).
molnar,b.,ladanyi,a.,tanko,l.,sréter,l.和tulassay,z.结直肠癌患者的外周血中的循环肿瘤细胞簇(circulatingtumorcellclustersintheperipheralbloodofcolorectalcancerpatients).clin.cancerres.7,4080–4085(2001).
mura,m.等,新型肿瘤内皮标志物clec14a的鉴别和血管生成作用(identificationandangiogenicroleofthenoveltumorendothelialmarkerclec14a).oncogene31,293–305(2012).
ni,x.等,肺癌患者的单循环肿瘤细胞中可再现的拷贝数变异模式(reproduciblecopynumbervariationpatternsamongsinglecirculatingtumorcellsoflungcancerpatients).proc.natl.acad.sci.usa110,21083–21088(2013).
paterlini-brechot,p.和benali,n.l.循环肿瘤细胞(ctc)检测:临床影响和未来方向(circulatingtumorcells(ctc)detection:clinicalimpactandfuturedirections).cancerlett.253,180–204(2007).
peixoto,a.,monteiro,m.,rocha,b.和veiga-fernandes,h.个体细胞中多基因表达的定量(quantificationofmultiplegeneexpressioninindividualcells).genomeres.14,1938–1947(2004).
rcoreteam.r:用于统计计算的语言和环境(r:alanguageandenvironmentforstatisticalcomputing).(rfoundationforstatisticalcomputing,vienna,2005).
rice,m.e.和harris,g.t.比较随访研究中的效应值:roc面积、cohen'sd和r(comparingeffectsizesinfollow-upstudies:rocarea,cohen’sd,andr).lawhum.behav.29,615–620(2005).
robin,x.等,proc:分析和比较roc曲线的r和s+的开源包(proc:anopen-sourcepackageforrands+toanalyzeandcompareroccurves).bmcbioinformatics12,77(2011).
rosenthal,r.用于社会研究的元分析程序(meta-analyticproceduresforsocialresearch).(sagepublications,1991).
salsbury,a.j.循环癌细胞的意义(thesignificanceofthecirculatingcancercell).cancertreat.rev.2,55–72(1975).
sanchez-freire,v.,ebert,a.d.,kalisky,t.,quake,s.r.和wu,j.c.用于基因表达模式的比较分析的微流体单细胞实时pcr(microfluidicsingle-cellreal-timepcrforcomparativeanalysisofgeneexpressionpatterns).natureprotocols7,829-38(2012).
schneider,c.a.,rasband,w.s.和eliceiri,k.w.nihimage到imagej:25年的图像分析(nihimagetoimagej:25yearsofimageanalysis).naturemethods9,671–675(2012).
schug,j.等,如通过香农熵测量的与组织特异性有关的启动子特征(promoterfeaturesrelatedtotissuespecificityasmeasuredbyshannonentropy).genomebiol.6,r33(2005).
seal,s.h.a用于从血液分离癌细胞和其它大细胞的筛(sievefortheisolationofcancercellsandotherlargecellsfromtheblood).cancer17,637–642(1964).
sellwood,r.a.,kuper,s.w.,burn,j.i.和wallace,e.n.循环癌细胞(circulatingcancercells).br.med.j.1,1683–1686(1964).
song,j.,from,p.,morrissey,w.j.和sams,j.循环癌细胞:化疗前和化疗后的观察(circulatingcancercells:pre-andpost-chemotherapyobservations).cancer28,553–561(1971).
stcroix,b.等,在人肿瘤内皮中表达的基因(genesexpressedinhumantumorendothelium).science289,1197–1202(2000).
stott,s.l.等,使用产生微涡的人字形芯片对循环肿瘤细胞的分离(isolationofcirculatingtumorcellsusingamicrovortex-generatingherringbone-chip).proc.natl.acad.sci.usa107,18392–18397(2010).
trapnell,c.,pachter,l.和salzberg,s.l.tophat:利用rna-seq发现剪接点(tophat:discoveringsplicejunctionswithrna-seq).bioinformatics25,1105–1111(2009).
trapnell,c.等,通过rna-seq进行的转录物组装和定量揭示在细胞分化期间未注释的转录物和亚型转换(transcriptassemblyandquantificationbyrna-seqrevealsunannotatedtranscriptsandisoformswitchingduringcelldifferentiation).nat.biotechnol.28,511–515(2010).
wild,r.,ramakrishnan,s.,sedgewick,j.和griffioen,a.w.通过计算机辅助数字图像分析对血管发生和肿瘤血管构筑的定量评估:vegf-毒素缀合物对肿瘤微血管密度的影响(quantitativeassessmentofangiogenesisandtumorvesselarchitecturebycomputer-assisteddigitalimageanalysis:effectsofvegf-toxinconjugateontumormicrovesseldensity).microvasc.res.59,368–376(2000).
wu,c.等,biogps:用于查询和组织基因注释资源的可扩展和可定制的门户(biogps:anextensibleandcustomizableportalforqueryingandorganizinggeneannotationresources).genomebiol.10,r130(2009).
ye,j.等,primer-blast:设计用于聚合酶链式反应的靶特异性引物的工具(primer-blast:atooltodesigntarget-specificprimersforpolymerasechainreaction).bmcbioinformatics13,134(2012).
yu,m.等,循环乳腺肿瘤细胞展现出上皮和间充质组成的动态变化(circulatingbreasttumorcellsexhibitdynamicchangesinepithelialandmesenchymalcomposition).science339,580–584(2013).
vanbeijnum,j.r.,rousch,m.,castermans,k.,vanderlinden,e.和griffioen,a.w.从新鲜组织分离内皮细胞(isolationofendothelialcellsfromfreshtissues).nat.protoc.3,1085–1091(2008).
vona,g.等,通过上皮细胞的大小加以分离:用于循环肿瘤细胞的免疫形态学和分子表征的新方法(isolationbysizeofepithelialtumourcells:anewmethodfortheimmunomorphologicalandmolecularcharacterizationofcirculatingtumorcells).am.j.pathol.156,57–63(2000).
vona,g.590等,肝癌患者中循环肿瘤细胞的细胞形态学检测的影响(impactofcytomorphologicaldetectionofcirculatingtumorcellsinpatientswithlivercancer).hepatology39,792–797(2004).
zanetta,l.等,冯·威利布兰德因子(内皮细胞标志物)的表达由血管发生因子上调:用于肿瘤血管发生的客观评估的潜在方法(expressionofvonwillebrandfactor,anendothelialcellmarker,isup-regulatedbyangiogenesisfactors:apotentialmethodforobjectiveassessmentoftumorangiogenesis).int.j.cancer85,281–288(2000)
zeileis,a.,wiel,m.,hornik,k.和hothorn,t.实施一类置换检验:coin包(implementingaclassofpermutationtests:thecoinpackage).j.stat.softw.28,1–23(2008).
序列表
<110>新加坡科技研究局
<120>单细胞rna和突变分析pcr(scrm-pcr):用于在单细胞水平上同时分析dna和rna的方法
<130>9869sg3572
<160>138
<170>patentinversion3.5
<210>1
<211>24
<212>dna
<213>人工序列
<220>
<223>ceacam5正向引物
<400>1
cccaagccctccatctccagcaac24
<210>2
<211>23
<212>dna
<213>人工序列
<220>
<223>ceacam5反向引物
<400>2
gcatccgggccatagaggacatt23
<210>3
<211>22
<212>dna
<213>人工序列
<220>
<223>cdx2正向引物
<400>3
tgggctgctgcaaacgctcaac22
<210>4
<211>25
<212>dna
<213>人工序列
<220>
<223>cdx2反向引物
<400>4
tttcgtcctggttttcacttggctg25
<210>5
<211>20
<212>dna
<213>人工序列
<220>
<223>krt20正向引物
<400>5
aaaatggccatgcagaacct20
<210>6
<211>20
<212>dna
<213>人工序列
<220>
<223>krt20反向引物
<400>6
gaagtcctcagcagccagtt20
<210>7
<211>16
<212>dna
<213>人工序列
<220>
<223>fn1正向引物
<400>7
ccacgggagcctcgaa16
<210>8
<211>20
<212>dna
<213>人工序列
<220>
<223>fn1反向引物
<400>8
taaaacctcggcttcctcca20
<210>9
<211>20
<212>dna
<213>人工序列
<220>
<223>cdh2正向引物
<400>9
ggatgaagatggcatggtgt20
<210>10
<211>20
<212>dna
<213>人工序列
<220>
<223>cdh2反向引物
<400>10
cccagtctctcttctgcctt20
<210>11
<211>17
<212>dna
<213>人工序列
<220>
<223>serpine1正向引物
<400>11
gccaagagcgctgtcaa17
<210>12
<211>19
<212>dna
<213>人工序列
<220>
<223>serpine1反向引物
<400>12
cagcagacccttcaccaaa19
<210>13
<211>25
<212>dna
<213>人工序列
<220>
<223>twist1正向引物
<400>13
acctagatgtcattgtttccagaga25
<210>14
<211>20
<212>dna
<213>人工序列
<220>
<223>twist1反向引物
<400>14
gcagaggtgtgaggatggtg20
<210>15
<211>20
<212>dna
<213>人工序列
<220>
<223>snai1正向引物
<400>15
gaccccaatcggaagcctaa20
<210>16
<211>17
<212>dna
<213>人工序列
<220>
<223>snai1反向引物
<400>16
gcggtggggttgaggat17
<210>17
<211>20
<212>dna
<213>人工序列
<220>
<223>vim正向引物
<400>17
gatgtttccaagcctgacct20
<210>18
<211>19
<212>dna
<213>人工序列
<220>
<223>vim反向引物
<400>18
cagtggactcctgctttgc19
<210>19
<211>21
<212>dna
<213>人工序列
<220>
<223>snai2正向引物
<400>19
agcgaactggacacacataca21
<210>20
<211>20
<212>dna
<213>人工序列
<220>
<223>snai2反向引物
<400>20
aggaggtgtcagatggagga20
<210>21
<211>19
<212>dna
<213>人工序列
<220>
<223>foxc2正向引物
<400>21
cgcctaaggacctggtgaa19
<210>22
<211>20
<212>dna
<213>人工序列
<220>
<223>foxc2反向引物
<400>22
gaagcggtccatgatgaact20
<210>23
<211>20
<212>dna
<213>人工序列
<220>
<223>foxc1正向引物
<400>23
cacaccctcaaagccgaact20
<210>24
<211>20
<212>dna
<213>人工序列
<220>
<223>foxc1反向引物
<400>24
aaagtggaggtggctctgaa20
<210>25
<211>17
<212>dna
<213>人工序列
<220>
<223>cdh1正向引物
<400>25
gagagaggccgcgtcct17
<210>26
<211>22
<212>dna
<213>人工序列
<220>
<223>cdh1反向引物
<400>26
ggccttttgactgtaatcacac22
<210>27
<211>20
<212>dna
<213>人工序列
<220>
<223>krt18(1)正向引物
<400>27
tgctcaccacacagtctgat20
<210>28
<211>20
<212>dna
<213>人工序列
<220>
<223>krt18(1)反向引物
<400>28
cactttgccatccactagcc20
<210>29
<211>20
<212>dna
<213>人工序列
<220>
<223>krt8(2)正向引物
<400>29
aaggatgccaacgccaagtt20
<210>30
<211>20
<212>dna
<213>人工序列
<220>
<223>krt8(2)反向引物
<400>30
ccgctggtggtcttcgtatg20
<210>31
<211>20
<212>dna
<213>人工序列
<220>
<223>krt19正向引物
<400>31
cagccactactacacgacca20
<210>32
<211>18
<212>dna
<213>人工序列
<220>
<223>krt19反向引物
<400>32
cgttgatgtcggcctcca18
<210>33
<211>17
<212>dna
<213>人工序列
<220>
<223>epcam正向引物
<400>33
gcaggtcctcgcgttcg17
<210>34
<211>22
<212>dna
<213>人工序列
<220>
<223>epcam反向引物
<400>34
tctcccaagttttgagccattc22
<210>35
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子5(3)正向
<400>35
tgttcacttgtgccctgact20
<210>36
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子5(3)反向引物
<400>36
cagccctgtcgtctctccag20
<210>37
<211>19
<212>dna
<213>人工序列
<220>
<223>tp53外显子6(4)正向引物
<400>37
tggttgcccagggtcccca19
<210>38
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子6(4)反向引物
<400>38
tggagggccactgacaacca20
<210>39
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子7(5)正向引物
<400>39
cttgccacaggtctccccaa20
<210>40
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子7(5)反向引物
<400>40
aggggtcagaggcaagcaga20
<210>41
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子8(6)正向引物
<400>41
ttccttactgcctcttgctt20
<210>42
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子8(6)反向引物
<400>42
aggcataactgcacccttgg20
<210>43
<211>21
<212>dna
<213>人工序列
<220>
<223>braf外显子15(7)正向引物
<400>43
cataatgcttgctctgatagg21
<210>44
<211>22
<212>dna
<213>人工序列
<220>
<223>braf外显子15(7)反向引物
<400>44
ggccaaaaatttaatcagtgga22
<210>45
<211>25
<212>dna
<213>人工序列
<220>
<223>kras外显子2(8)正向引物
<400>45
ttataaggcctgctgaaaatgactg25
<210>46
<211>25
<212>dna
<213>人工序列
<220>
<223>kras外显子2(8)反向引物
<400>46
tcatgaaaatggtcagagaaacctt25
<210>47
<211>19
<212>dna
<213>人工序列
<220>
<223>egfr正向引物
<400>47
gtcgggctctggaggaaaa19
<210>48
<211>22
<212>dna
<213>人工序列
<220>
<223>egfr反向引物
<400>48
ctctggaggctgagaaaatgat22
<210>49
<211>24
<212>dna
<213>人工序列
<220>
<223>ptprc正向引物
<400>49
gacatcatcacctagcagttcatg24
<210>50
<211>19
<212>dna
<213>人工序列
<220>
<223>ptprc反向引物
<400>50
cagtgggggaaggtgttgg19
<210>51
<211>19
<212>dna
<213>人工序列
<220>
<223>vwf正向引物
<400>51
acacagggggaccaaagag19
<210>52
<211>19
<212>dna
<213>人工序列
<220>
<223>vwf反向引物
<400>52
gagatgcccgttcacacca19
<210>53
<211>19
<212>dna
<213>人工序列
<220>
<223>pecam1正向引物
<400>53
tctcaacggtgacttgtgg19
<210>54
<211>21
<212>dna
<213>人工序列
<220>
<223>pecam1反向引物
<400>54
gttcttcccattttgcaccgt21
<210>55
<211>20
<212>dna
<213>人工序列
<220>
<223>col1a1正向引物
<400>55
tgttcagctttgtggacctc20
<210>56
<211>19
<212>dna
<213>人工序列
<220>
<223>col1a1反向引物
<400>56
ggtttccacacgtctcggt19
<210>57
<211>21
<212>dna
<213>人工序列
<220>
<223>cd68正向引物
<400>57
cattctttcaccagctgtcca21
<210>58
<211>15
<212>dna
<213>人工序列
<220>
<223>cd68反向引物
<400>58
gcaccagggcgagga15
<210>59
<211>18
<212>dna
<213>人工序列
<220>
<223>mcam正向引物
<400>59
ctcggtcccaggagtacc18
<210>60
<211>21
<212>dna
<213>人工序列
<220>
<223>mcam反向引物
<400>60
tgtacaaaccactcgactcca21
<210>61
<211>20
<212>dna
<213>人工序列
<220>
<223>fcgr3a正向引物
<400>61
cccttgccagacttcagact20
<210>62
<211>20
<212>dna
<213>人工序列
<220>
<223>fcgr3a反向引物
<400>62
gggagatcttcagtccgcat20
<210>63
<211>21
<212>dna
<213>人工序列
<220>
<223>gypa正向引物
<400>63
ctagcaggctaaggtcagaca21
<210>64
<211>20
<212>dna
<213>人工序列
<220>
<223>gypa反向引物
<400>64
gtgtcccgtttgtgcgtatc20
<210>65
<211>19
<212>dna
<213>人工序列
<220>
<223>itga2b正向引物
<400>65
cttctatgcaggccccaat19
<210>66
<211>22
<212>dna
<213>人工序列
<220>
<223>itga2b反向引物
<400>66
agcctacatttcgggtctcatc22
<210>67
<211>21
<212>dna
<213>人工序列
<220>
<223>dcstamp正向引物
<400>67
ctcccgctgaataaggaggaa21
<210>68
<211>25
<212>dna
<213>人工序列
<220>
<223>dcstamp反向引物
<400>68
tcttgagttccttgtttctctccgt25
<210>69
<211>25
<212>dna
<213>人工序列
<220>
<223>cd34正向引物
<400>69
ccttctgggttcatgagtcttgaca25
<210>70
<211>25
<212>dna
<213>人工序列
<220>
<223>cd34反向引物
<400>70
tgtcgtttctgtgatgtttgttgtg25
<210>71
<211>20
<212>dna
<213>人工序列
<220>
<223>actb正向引物
<400>71
ctggcaccacaccttctaca20
<210>72
<211>20
<212>dna
<213>人工序列
<220>
<223>actb反向引物
<400>72
tagcacagcctggatagcaa20
<210>73
<211>23
<212>dna
<213>人工序列
<220>
<223>folh1正向引物
<400>73
cggatattgtaccacctttcagt23
<210>74
<211>20
<212>dna
<213>人工序列
<220>
<223>folh1反向引物
<400>74
agcagggtcggagtagagaa20
<210>75
<211>22
<212>dna
<213>人工序列
<220>
<223>eng正向引物
<400>75
gtgacggtgaaggtggaactga22
<210>76
<211>20
<212>dna
<213>人工序列
<220>
<223>eng反向引物
<400>76
ttgaggtgtgtctgggagct20
<210>77
<211>23
<212>dna
<213>人工序列
<220>
<223>kdr正向引物
<400>77
gaaatgacactggagcctacaag23
<210>78
<211>21
<212>dna
<213>人工序列
<220>
<223>kdr反向引物
<400>78
aatggacccgagacatggaat21
<210>79
<211>22
<212>dna
<213>人工序列
<220>
<223>cdh5正向引物
<400>79
gttcacgcatcggttgttcaat22
<210>80
<211>20
<212>dna
<213>人工序列
<220>
<223>cdh5反向引物
<400>80
gcctgcttctctcggtccaa20
<210>81
<211>23
<212>dna
<213>人工序列
<220>
<223>tek正向引物
<400>81
cttatttctgtgaagggcgagtt23
<210>82
<211>22
<212>dna
<213>人工序列
<220>
<223>tek反向引物
<400>82
ctcccttgtccacagtcatagt22
<210>83
<211>25
<212>dna
<213>人工序列
<220>
<223>angpt2正向引物
<400>83
aacactccctctcgacaaacaaatt25
<210>84
<211>24
<212>dna
<213>人工序列
<220>
<223>angpt2反向引物
<400>84
ctgtagttggatgatgtgcttgtc24
<210>85
<211>20
<212>dna
<213>人工序列
<220>
<223>hprt1正向引物
<400>85
tgaccagtcaacaggggaca20
<210>86
<211>21
<212>dna
<213>人工序列
<220>
<223>hprt1反向引物
<400>86
ggtccttttcaccagcaagct21
<210>87
<211>20
<212>dna
<213>人工序列
<220>
<223>ubc正向引物
<400>87
tcggccttagaaccccagta20
<210>88
<211>23
<212>dna
<213>人工序列
<220>
<223>ubc反向引物
<400>88
acgaagatctgcattgtcaagtg23
<210>89
<211>24
<212>dna
<213>人工序列
<220>
<223>ceacam5(外显子接合部上的反向引物)正向引物
<400>89
tgtggtgggtaaacaatcagagcc24
<210>90
<211>20
<212>dna
<213>人工序列
<220>
<223>cdx2(外显子接合部上的反向引物)正向引物
<400>90
cggcggaacctgtgcgagtg20
<210>91
<211>23
<212>dna
<213>人工序列
<220>
<223>krt20(dna序列中的2141bp内含物)正向引物
<400>91
gcgactacagtgcatattacaga23
<210>92
<211>20
<212>dna
<213>人工序列
<220>
<223>fn1(dna序列中的830bp内含子)正向引物
<400>92
caagcccggttgttatgaca20
<210>93
<211>23
<212>dna
<213>人工序列
<220>
<223>cdh2(dna序列中的1690bp内含子)正向引物
<400>93
aagttcctgatatatgcccaaga23
<210>94
<211>23
<212>dna
<213>人工序列
<220>
<223>serpine1(dna序列中的1148bp内含子)正向引物
<400>94
agaacttcaggatgcagatgtct23
<210>95
<211>24
<212>dna
<213>人工序列
<220>
<223>twist1(外显子接合部上的正向引物)正向引物
<400>95
ccagagaaggagaaaatggacagt24
<210>96
<211>20
<212>dna
<213>人工序列
<220>
<223>snai1(dna序列中的682bp内含子)反向引物
<400>96
gtagggctgctggaaggtaa20
<210>97
<211>21
<212>dna
<213>人工序列
<220>
<223>vim(dna序列中的761bp内含子)反向引物
<400>97
tgtaccattcttctgcctcct21
<210>98
<211>20
<212>dna
<213>人工序列
<220>
<223>snai2(dna序列中的745bp内含子)反向引物
<400>98
gtggaatggagcagcggtag20
<210>99
<211>19
<212>dna
<213>人工序列
<220>
<223>foxc2(na(单个外显子编码基因))正向引物
<400>99
gctcatcaccatggccatc19
<210>100
<211>22
<212>dna
<213>人工序列
<220>
<223>foxc1(na(单个外显子编码基因))反向引物
<400>100
gagggatattctgttcgctggt22
<210>101
<211>24
<212>dna
<213>人工序列
<220>
<223>cdh1(dna序列中的63258bp内含子)正向引物
<400>101
ggcagagtgaattttgaagattgc24
<210>102
<211>20
<212>dna
<213>人工序列
<220>
<223>krt18(dna序列中的641bp内含子)正向引物
<400>102
tggaggaccgctacgcccta20
<210>103
<211>20
<212>dna
<213>人工序列
<220>
<223>krt18(dna序列中的641bp内含子)反向引物
<400>103
ccaaggcatcaccaagacta20
<210>104
<211>17
<212>dna
<213>人工序列
<220>
<223>krt8(dna序列中的159bp内含子)正向引物
<400>104
gctggagggcgaggaga17
<210>105
<211>20
<212>dna
<213>人工序列
<220>
<223>krt19(dna序列中的2745bp内含子)正向引物
<400>105
tgcgggacaagattcttggt20
<210>106
<211>20
<212>dna
<213>人工序列
<220>
<223>epcam(dna序列中的4118bp内含子)正向引物
<400>106
ccgcagctcaggaagaatgt20
<210>107
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子5(内含子序列上的引物)正向引物
<400>107
ttcaactctgtctccttcct20
<210>108
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子6(内含子序列上的引物)正向引物
<400>108
gcctctgattcctcactgat20
<210>109
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子6(内含子序列上的引物)反向引物
<400>109
ttaacccctcctcccagaga20
<210>110
<211>21
<212>dna
<213>人工序列
<220>
<223>tp53外显子7(内含子序列上的引物)正向引物
<400>110
aaggcgcactggcctcatctt21
<210>111
<211>21
<212>dna
<213>人工序列
<220>
<223>tp53外显子8(内含子序列上的引物)正向引物
<400>111
agtggtaatctactgggacgg21
<210>112
<211>20
<212>dna
<213>人工序列
<220>
<223>tp53外显子8(内含子序列上的引物)反向引物
<400>112
acctcgcttagtgctccctg20
<210>113
<211>20
<212>dna
<213>人工序列
<220>
<223>braf外显子15(内含子序列上的引物)反向引物
<400>113
tagcctcaattcttaccatc20
<210>114
<211>25
<212>dna
<213>人工序列
<220>
<223>kras外显子2(内含子序列上的引物)反向引物
<400>114
caaagaatggtcctgcaccagtaat25
<210>115
<211>22
<212>dna
<213>人工序列
<220>
<223>egfr(dna序列中的122920bp内含子)正向引物
<400>115
tctggaggaaaagaaagtttgc22
<210>116
<211>21
<212>dna
<213>人工序列
<220>
<223>ptprc(dna序列中的53092bp内含子)正向引物
<400>116
caacagtggagaaaggacgca21
<210>117
<211>20
<212>dna
<213>人工序列
<220>
<223>vwf(外显子接合部上的正向引物)正向引物
<400>117
tgcctccaaagggctgtatc20
<210>118
<211>21
<212>dna
<213>人工序列
<220>
<223>pecam1(dna序列中的12457bp内含子)正向引物
<400>118
cagtcttcactctcaggatgc21
<210>119
<211>20
<212>dna
<213>人工序列
<220>
<223>col1a1(dna序列中的1463bp内含子)反向引物
<400>119
gtgggatgtcttcgtcttgg20
<210>120
<211>20
<212>dna
<213>人工序列
<220>
<223>cd68(dna序列中的311bp内含子)正向引物
<400>120
aaagtttctcctgccccagt20
<210>121
<211>22
<212>dna
<213>人工序列
<220>
<223>mcam(dna序列中的1724bp内含子)反向引物
<400>121
cggccattcttgtaccagatga22
<210>122
<211>17
<212>dna
<213>人工序列
<220>
<223>fcgr3a(外显子接合部上的反向引物和dna序列中的内含子)
反向引物
<400>122
tttccccagcccctcca17
<210>123
<211>21
<212>dna
<213>人工序列
<220>
<223>gypa(dna序列中的20010bp内含子)正向引物
<400>123
caggaaccagctcatgatctc21
<210>124
<211>16
<212>dna
<213>人工序列
<220>
<223>itga2b(dna序列中的3242bp内含子)正向引物
<400>124
ggcggcgtgttcctgt16
<210>125
<211>20
<212>dna
<213>人工序列
<220>
<223>dcstamp(外显子接合部上的反向引物)正向引物
<400>125
acctggggctgtttttcctc20
<210>126
<211>22
<212>dna
<213>人工序列
<220>
<223>cd34(外显子接合部上的正向引物和反向引物)正向引物
<400>126
ctaccccagagttacctaccca22
<210>127
<211>20
<212>dna
<213>人工序列
<220>
<223>folh1(dna序列中的6811bp内含子)正向引物
<400>127
ccagagggcgatctagtgta20
<210>128
<211>24
<212>dna
<213>人工序列
<220>
<223>eng(dna序列中的256bp内含子)反向引物
<400>128
agtattctccagtggtccagatct24
<210>129
<211>21
<212>dna
<213>人工序列
<220>
<223>kdr(dna序列中的3192bp内含子)反向引物
<400>129
tgttggtcactaacagaagca21
<210>130
<211>20
<212>dna
<213>人工序列
<220>
<223>cdh5(dna序列中的2143bp内含子)正向引物
<400>130
cacgcctctgtcatgtacca20
<210>131
<211>21
<212>dna
<213>人工序列
<220>
<223>tek(dna序列中的10352bp内含子)反向引物
<400>131
gtagctggtaggaaggaagct21
<210>132
<211>24
<212>dna
<213>人工序列
<220>
<223>angpt2(dna序列中的6144bp内含子)正向引物
<400>132
ggaccagaccagtgaaataaacaa24
<210>133
<211>21
<212>dna
<213>人工序列
<220>
<223>hprt1(外显子接合部上的反向引物和dna序列中的内含子)
正向引物
<400>133
tgacactggcaaaacaatgca21
<210>134
<211>20
<212>dna
<213>人工序列
<220>
<223>ubc(外显子接合部上的反向引物)正向引物
<400>134
aaagtagtcccttctcggcg20
<210>135
<211>25
<212>dna
<213>人工序列
<220>
<223>用于kras外显子2测序的引物-正向引物
<400>135
tttgtattaaaaggtactggtggag25
<210>136
<211>25
<212>dna
<213>人工序列
<220>
<223>用于kras外显子2测序的引物-反向引物
<400>136
cctttatctgtatcaaagaatggtc25
<210>137
<211>25
<212>dna
<213>人工序列
<220>
<223>cd45引物(正向引物)
<400>137
aagacaacagtggagaaaggacgca25
<210>138
<211>22
<212>dna
<213>人工序列
<220>
<223>cd45引物(反向引物)
<400>138
cagtgggggaaggtgttgggct22
- 还没有人留言评论。精彩留言会获得点赞!