具有整合可控功能的嵌合抗原受体的制作方法
本发明涉及细胞免疫治疗领域,更具体地,本发明涉及新一代嵌合抗原受体(car),其允许通过与小分子的相互作用来控制被赋予这种car的免疫细胞。更具体地,本发明涉及嵌合抗原受体,其在至少一个胞外域中包含控制受体的抗原结合功能的分子开关。因此,本发明提供了被赋予更受控的和潜在更安全的工程化的car的免疫细胞,例如t淋巴细胞。
背景技术:
过继性免疫治疗涉及离体产生的自体抗原特异性t细胞的转移,是治疗病毒感染和癌症的有前景的策略。可以通过扩增抗原特异性t细胞或通过基因工程重定向t细胞而产生用于过继性免疫治疗的t细胞(park,rosenbergetal.2011)。病毒抗原特异性t细胞的转移是用于治疗移植相关病毒感染和罕见病毒相关恶性肿瘤的完善的程序。类似地,已证实肿瘤特异性t细胞的分离和转移在治疗黑素瘤方面是成功的。
已通过转基因t细胞受体或嵌合抗原受体(car)的遗传转移成功地产生了t细胞中的新特异性(jena,dottietal.2010)。car是由与单个融合分子中的一个或多个信号结构域关联的靶向部分组成的合成受体。通常,car的结合部分由单链抗体(scfv)的抗原结合结构域组成,其包含通过柔性接头连接的单克隆抗体的轻链和重链可变片段。也已成功应用基于受体或配体结构域的结合部分。第一代car的信号结构域衍生自cd3ζ或fc受体γ链的细胞质区域。已证实第一代car成功地重定向t细胞的细胞毒性,然而它们未能在体内提供延长的扩增和抗肿瘤活性。已单独(第二代)或组合地(第三代)添加来自共刺激分子(包括cd28、ox-40(cd134)、icos和4-1bb(cd137))的信号结构域来增强car修饰的t细胞的存活并增加其增殖。car已成功允许t细胞针对来自各种恶性肿瘤包括淋巴瘤和实体瘤在肿瘤细胞表面的表达的抗原进行重定向(jena,dottietal.2010)。
然而,尽管大量的临床研究已经证明cart细胞的过继转移用于癌症治疗的潜力,但它们也提高了与细胞因子释放综合征(crs)和“脱靶(on-targetoff-tumor)”效应相关的风险。迄今为止,开发了几种药物控制car工程t细胞的策略,并且主要依靠自杀机制。导致彻底根除工程t细胞的这种自杀策略将导致治疗的提前结束。
因此,非常需要实施对工程化的cart细胞的非致死性控制以改善cart细胞技术及其安全性。
技术实现要素:
在此,本发明人开发了一种系统,其中在加入小分子后,可以获得含有抗原结合结构域的car的细胞外部分的构象的受控变化。该整合系统在开启/关闭状态之间转换抗原和抗原结合结构域之间的相互作用。特别地,通过能够控制car的细胞外部分的确证,可以直接调节被赋予这种嵌合抗原受体的免疫细胞的下游功能,例如t细胞的细胞毒性。这个系统提供了新的更受控和潜在更安全的工程化的car赋予的免疫细胞。
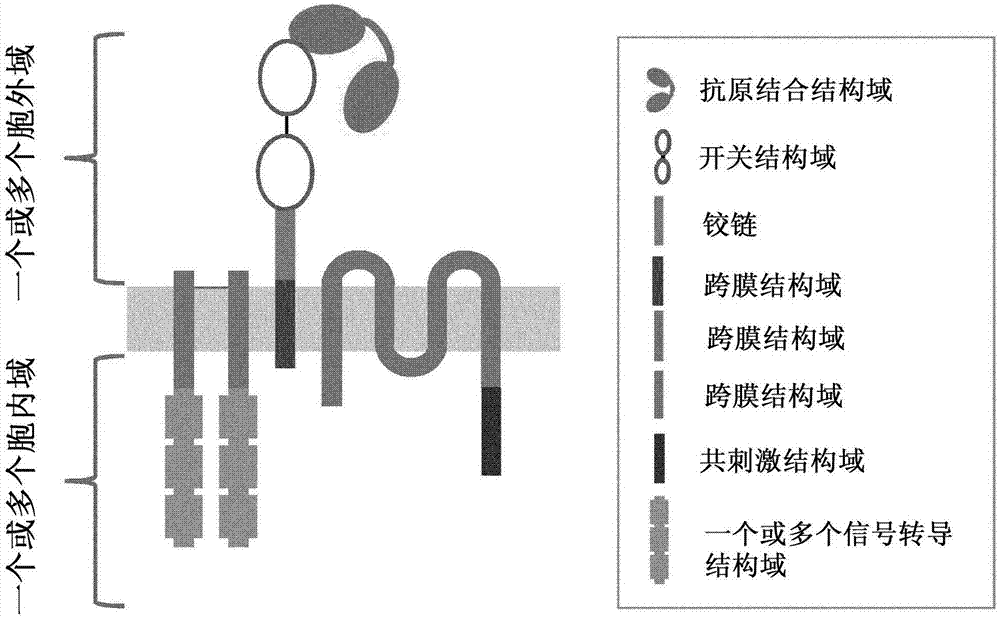
因此本发明在第一方面提供了嵌合抗原受体(car),其特征在于它包含:
a)至少一个胞外域,其包含:
i)细细胞外抗原结合结构域;和
ii)包含至少第一多聚化配体结合结构域和第二多聚化配体结合结构域的开关结构域,该第一多聚化配体结合结构域和第二多聚化配体结合结构域能够结合预定的多价配体以形成包含所述两个结合结构域和它们能够与其结合的多价配体的多聚体;
b)至少一个跨膜结构域;和
c)至少一个包含信号转导结构域和可选的共刺激结构域的胞内域(endodomain);
其中开关结构域位于细胞外抗原结合结构域和跨膜结构域之间。
本发明在另外的方面提供了包含一个或多个编码本发明的嵌合抗原受体的核酸序列的多核苷酸和载体。
本发明在另外的方面提供了一种免疫细胞,更具体地是一种分离的免疫细胞,其包含(例如,在其表面上表达)至少一种本发明的嵌合抗原受体。
本发明在另外的方面提供了用于工程化本发明的免疫细胞的方法。
本发明在另外的方面提供了本发明的免疫细胞在治疗中的用途,例如用于治疗癌症。
由于构成分子开关的第一多聚化配体结合结构域和第二多聚化配体结合结构域存在于同一多肽分子上,因此本发明的car的结构提供了可靠的功能性。如果两者都存在于不同的多肽分子上,则这种结构使得本发明的car系统独立于否则可能影响配体结合结构域在细胞表面上的正确表达和定位的因素,其中的一些难以控制。
附图说明
图1.(a)根据本发明的单链嵌合抗原受体的示意图。(b)实施例中使用的car胞外域的示意图。根据本发明,两个下部结构均结合frb和fkbp结构域。
图2.根据本发明的多链嵌合抗原受体的示意图。
图3.(a)针对媒介物(dmso)或雷帕霉素(rapamycin)的存在,对mccar功能的表面检测呈阳性的活细胞百分比。示出了scfv的fab’2区域的检测。(b)如在整个活细胞群体中所描绘的,加入小分子雷帕霉素后,中值荧光强度(mfi)的倍数增加。
图4.如在整个活细胞群体中所描绘的,加入与t2098l突变体fkbp/frb*构建体结合使用的小分子ap21967后,中值荧光强度(mfi)的倍数增加。
图5.(a)在基于流动的细胞毒性测定中评估ap21967雷帕霉素类似物(rapalog)对cart细胞向模型抗原递呈细胞的细胞溶解能力的影响。在存在或不存在ap21967的情况下,在与工程化的cart细胞共培养后,测量cd19pos(cd19阳性)和cd19neg(cd19阴性)靶细胞活力。效应物/靶标比被设定为20:1。nt代表未转染的t细胞。(b)显示当使用增加浓度的根据本发明的雷帕霉素类似物ap21967来诱导car活性时,daudicd19阳性细胞裂解的百分比的图。
图6.如实施例3所描述的,ap21967(10nm)和他克莫司(0-500nm)之间的竞争实验。
图7.如在整个活细胞群体中所描绘的,加入与t2098l突变体fkbp/frb*构建体结合使用的小分子ap21967后,中值荧光强度(mfi)的倍数增加。
图8.(a)用靶向cd19的工程化的car测定ap21967ec50。用增加量的ap21967雷帕霉素合成类似物处理已经用三种剂量(d、d1/2和d1/4)的编码工程化的car的mrna转染的t细胞。检测scfv的fab’2区域。(b)用靶向cd123的工程化的mccar测定ap21967ec50。用增加量的ap21967雷帕霉素类似物处理已经用三种剂量(d、d1/2和d1/4)的编码工程化的mccar的mrna转染的t细胞。使用与fc片段融合的重组cd123检测scfv。
图9.针对增加剂量的ap21967的存在,对fkbp/frb*-mccar功能的表面检测呈阳性的活细胞的百分比。
图10.如实施例8中所描述的,用t细胞carcd123+与重复剂量的雷帕霉素类似物ap21967联合处理的molm-13-luc的治疗方案。
图11.如图10和实施例8所示,处理后的molm-13-luc小鼠的存活曲线。
图12.如图10和实施例8所示,处理后的molm-13-luc小鼠的体重图。
具体实施方式
除非本文具体地定义,否则使用的所有技术和科学术语具有与基因治疗、生物化学、遗传学和分子生物学领域的技术人员通常理解的相同含义。
除非另有说明,否则实施本发明会采用细胞生物学、细胞培养、分子生物学、转基因生物学、微生物学、重组dna和免疫学的常规技术,它们均在本领域技术范围内。这些技术在文献中得到充分解释。参见例如,currentprotocolsinmolecularbiology(frederickm.ausubel,2000,wileyandsoninc,libraryofcongress,usa);molecularcloning:alaboratorymanual,thirdedition,(sambrook等人,2001,coldspringharbor,newyork:coldspringharborlaboratorypress);oligonucleotidesynthesis(m.j.gait编辑,1984);mullis等人,美国专利号4,683,195;nucleicacidhybridization(b.d.harries&s.j.higgins编辑.1984);transcriptionandtranslation(b.d.hames&s.j.higgins编辑.1984);cultureofanimalcells(r.i.freshney,alanr.liss,inc.,1987);immobilizedcellsandenzymes(irlpress,1986);b.perbal,apracticalguidetomolecularcloning(1984);theseries,methodsinenzymology(j.abelsonandm.simon主编,academicpress,inc.,newyork),具体地,第154和155卷(wu等人编辑)和第185卷,“geneexpressiontechnology”(d.goeddel编辑);genetransfervectorsformammaliancells(j.h.miller和m.p.calos编辑,1987,coldspringharborlaboratory);immunochemicalmethodsincellandmolecularbiology(mayer和walker编辑,academicpress,london,1987);handbookofexperimentalimmunology,第i-iv卷(d.m.weir和c.c.blackwell编辑,1986)和manipulatingthemouseembryo,(coldspringharborlaboratorypress,coldspringharbor,n.y.,1986)。
本发明的嵌合抗原受体
因此本发明在第一方面提供了嵌合抗原受体(car),其特征在于它包含:
a)至少一个胞外域,其包含:
i)细细胞外抗原结合结构域;和
ii)包含至少第一多聚化配体结合结构域和第二多聚化配体结合结构域的开关结构域,该第一多聚化配体结合结构域和第二多聚化配体结合结构域能够结合预定的多价配体以形成包含所述两个结合结构域和它们能够与其结合的多价配体的多聚体;
b)至少一个跨膜结构域;和
c)至少一个包含信号转导结构域和可选的共刺激结构域的胞内域(endodomain);
其中开关结构域位于细胞外抗原结合结构域和跨膜结构域之间。
在多价配体同时结合至第一多聚化配体结合结构域和第二多聚化配体结合结构域时,形成多聚体复合物如二聚体复合物,其使得嵌合抗原受体的胞外域内的构象变化,其反过来改善抗原结合结构域的表面暴露。
“多聚化配体”或“多聚物”是多价配体,其能够同时结合至少两个结合伴侣,例如第一多聚化配体结合结构域和第二多聚化配体结合结构域,引起结合时结合伴侣的多聚化,例如二聚化。更特别地,“多价配体”是分子,优选为小分子,其具有至少两个允许至少两个结合伴侣如第一多聚化配体结合结构域和第二多聚化配体结合结构域的同时结合的结合位点。术语“多聚化配体”、“多聚体”和“多价配体”在本文可互换使用,并分别包括术语“二聚化配体”、“二聚体”和“二价配体”。“二聚化配体”、“二聚体”或“二价配体”是分子,优选为小分子,其具有两个结合位点,允许两个配体结合伴侣如第一多聚化配体结合结构域和第二多聚化配体结合结构域的结合,由此引起其二聚化。
如在本文中所使用的,“小分子”是低分子量(<2000道尔顿)的有机化合物。可用于本发明的小分子的非限制性实例包括大环内酯类雷帕霉素和其类似物,统称为“雷帕霉素类似物(rapalog)”,如ap21967,deforolimus(ap23573)、依维莫司(rad001)和西罗莫司(cci-779)。可用于本发明的小分子的其他非限制性实例包括他克莫司(fk506)、fk506衍生物,如fk1012、fk506类似物,如ap1903。可用于本发明的小分子的其他非限制性实例包括香豆霉素、赤霉素、haxs、ap1510、ap20187和ap21967。
“多聚化配体”还可以是肽或核酸(天然或合成的)。
根据某些实施方式,第一多聚化配体结合结构域和第二多聚化配体结合结构域来源于化学诱导二聚化(cid)系统。
cid系统基于这样的机制:两种多肽仅在某些小分子或其他二聚化试剂的存在下通过该机制结合。各种cid系统是已知的并且可以根据本发明使用。下表1中提供了合适的cid系统的非限制性实例。
ap21967是具有异源二聚化活性的雷帕霉素类似物。二聚体ap21967将fkbp12与含有单个氨基酸替换(t2098l)的frb变体异源二聚化。
ap1903是具有同源二聚化活性的他克莫司类似物。二聚体ap1903将含有单个氨基酸替换(f36v)的fkbp12变体同源二聚化。
根据某些实施方式,可以使用他克莫司类似物如ap1903或其他大环内酯类免疫抑制剂来进一步调节根据本发明的免疫细胞的car介导的激活。它们可以充当小分子竞争者,提供工程化的cart细胞的另外的控制。作为调整以开启状态锁定在细胞表面处的car的量的可能性的说明,本发明人使用他克莫司(fk506)作为已知与fkbp12结合而不能与frb形成复合物的小分子(参见实施例4和图6)。ap21967(或雷帕霉素)和他克莫司具有相同的fkbp12结合核并竞争fkbp部分中的相同结合位点(wilson,k.p.等人,1995,comparativex-raystructuresofthemajorbindingproteinfortheimmunosuppressantfk506(tacrolimus)inunligandedformandincomplexwithfk506andrapamycin.actacrystallogrdbiolcrystallogr51,511-521)。如图9所示,用固定量的ap21967和增加量的他克莫司(0至10mm,优选为0至500nm)孵育的工程化的car转染的t细胞显示减少的car表面检测。因此,旨在接通car诱导的免疫细胞激活的根据本发明的方法可以包括另外的步骤,其中通过使所述免疫细胞与他克莫司类似物或大环内酯类免疫抑制剂在体内或体外接触来调节所述诱导。这可以通过本发明的car客体来调节、减少或抑制免疫细胞的激活来进行。
二聚体haxs描述于例如erhart,d.等人(2013),“chemicaldevelopmentofintracellularproteinheterodimerizers”,chemistry&biology20:549–557中。
二聚体sc描述于例如gaultier,a.等人(2009),“selectivecross-linkingofinteractingproteinsusingself-labelingtags”,jamchemsoc.131(49):17954-62中。
二聚体bismtx描述于例如,kopytek,s.j.等人(2000),“chemicallyinduceddimerizationofdihydrofolatereductasebyahomobifunctionaldimerofmethotrexate”,chemistry&biology7:313–321中。
二聚体dex-mtx描述于例如,lin,h.n.等人(2000),“dexamethasone-methotrexate:anefficientchemicalinducerofproteindimerizationinvivo”,journaloftheamericanchemicalsociety,122(17):4247-4248中。
二聚体dex-tmp描述于例如,gallagher,s.s.等人(2007)"anorthogonaldexamethasone-trimethoprimyeastthree-hybridsystem",analbiochem.363(1):160-2中。
第一多聚化配体结合结构域和第二多聚化配体结合结构域可以是相同的或不同的。因此,根据某些实施方式,第一多聚化配体结合结构域和第二多聚化配体结合结构域是相同的。根据其他某些实施方式,第一多聚化配体结合结构域和第二多聚化配体结合结构域是不同的。
根据某些实施方式,第一多聚化配体结合结构域和第二多聚化配体结合结构域是选自由以下组成的多聚化配体结合结构域的对:fk506结合蛋白(fkbp12):mtor的fkbp-雷帕霉素结合结构域(frb)、fkbp12:fkbp12、fkbp(f36v):fkbp(f36v)、fkbp12:frb(t2098l)、fkbp12:钙调磷酸酶a(cna)、fkbp12:亲环素(cyp)、gyrb:gyrb、gai:gid1a、gai:gid1b、gai:gid1c、snap标签:halo标签、snap标签:clip标签、dhfr:dhfr、糖皮质激素受体配体结合结构域:dhfr和pyl1:abi1。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1)或其与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1)。
根据其他特定的实施方式,第一多聚化配体结合结构域是与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fkbp12的变体。
根据某些实施方式,第二多聚化配体结合结构域也是fkbp12(seqidno:1)或其与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第二多聚化配体结合结构域是fkbp12(seqidno:1)。
根据其他特定的实施方式,第二多聚化配体结合结构域是与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fkbp12的变体。
根据某些实施方式,第二多聚化配体结合结构域是frb(seqidno:2)或其与frb(seqidno:2)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第二多聚化配体结合结构域是frb(seqidno:2)。
根据其他特定的实施方式,第二多聚化配体结合结构域是与frb(seqidno:2)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的frb的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1)或其与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是frb(seqidno:2)或其与frb(seqidno:2)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是frb(seqidno:2)或其与frb(seqidno:2)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他更特定的实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是frb(seqidno:2)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是与frb(seqidno:2)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的frb的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fkbp12的变体1,并且第二多聚化配体结合结构域是frb(seqidno:2)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fkbp12的变体,并且第二多聚化配体结合结构域是与frb(seqidno:2)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的frb的变体。
fkbp12的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于fkbp12(seqidno:1)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于fkbp12(seqidno:1)。
合适地,fkbp12的变体能够结合雷帕霉素或雷帕霉素类似物,如ap21967、ap23573、rad001或cci-779。更具体地,这种变体包含雷帕霉素或雷帕霉素类似物结合序列。
fkbp12变体的非限制性实例是含有如seqidno:3所示的单个氨基酸替换(f36v)的变体。例如,这种fkbp12变体可以用作第一多聚化配体结合结构域和第二多聚化配体结合结构域,在结合二聚体ap1903时形成同源二聚体。
因此,根据某些实施方式,第一多聚化配体结合结构域是具有如seqidno:3所示的氨基酸序列的fkbp12(f36v),并且第二多聚化配体结合结构域是具有如seqidno:3所示的氨基酸序列的fkbp12(f36v)。
frb的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于frb(seqidno:2)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于frb(seqidno:2)。
合适地,frb的变体能够结合雷帕霉素或雷帕霉素类似物,如ap21967、ap23573、rad001或cci-779。更具体地,这种变体包含雷帕霉素或雷帕霉素类似物结合序列。
frb变体的非限制性实例包括包含在选自l2031、e2032、s2035、r2036、f2039、g2040、t2098、w2101、d2102、y2105和f2108的氨基酸位置处的一个或多个氨基酸替换的变体。根据某些实施方式,frb的变体包含在t2098处的氨基酸替换,其中t2098被亮氨酸(t2098l)替换,例如,seqidno:4,或被苯丙氨酸(t2098f)替换。根据某些其他实施方式,frb的变体包括在e2032处的氨基酸替换,其中e2032被以下替换:苯丙氨酸(e2032f)、甲硫氨酸(e2032m)、精氨酸(e2032r)、缬氨酸(e2032v)、酪氨酸(e2032y)、异亮氨酸(e2032i)或亮氨酸(e2032l)。根据某些其他实施方式,frb的变体包含在e2032和在t2098处的氨基酸替换,其中e2032被任何氨基酸替换,并且其中t2098被任何氨基酸替换。根据某些其他实施方式,frb的变体包含e2032i和t2098l替换。根据某些其他实施方式,frb的变体包含e2032l和t2098l替换。
具有t2098l替换的frb的变体可以例如用作第二多聚化配体结合结构域,在结合二聚体ap21967时与fkbp12或其变体形成异源二聚体。因此,根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是包含在t2098处的氨基酸替换的frb的变体,其中t2098被亮氨酸(seqidno:4)替换。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1)或其与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是钙调磷酸酶a(cna)(seqidno:5)或其与cna(seqidno:5)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是钙调磷酸酶a(cna)(seqidno:5)或其与cna(seqidno:5)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fkbp12的变体,并且第二多聚化配体结合结构域是钙调磷酸酶a(cna)(seqidno:5)或其与cna(seqidno:5)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是钙调磷酸酶a(cna)(seqidno:5)。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是与cna(seqidno:5)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的钙调磷酸酶a(cna)的变体。
钙调磷酸酶a(cna)的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cna(seqidno:5)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cna(seqidno:5)。适当地,cna的变体能够结合fk506。更具体地,这种变体包括fk506结合序列。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1)或其与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是亲环素(cyp)(seqidno:6)或其与cyp(seqidno:6)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体6)。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1)并且第二多聚化配体结合结构域是亲环素(cyp)(seqidno:6)或其与cyp(seqidno:6)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是与fkbp12(seqidno:1)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fkbp12的变体,并且第二多聚化配体结合结构域是亲环素(cyp)(seqidno:6)或其与cyp(seqidno:6)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是亲环素(cyp)(seqidno:6)。
根据某些实施方式,第一多聚化配体结合结构域是fkbp12(seqidno:1),并且第二多聚化配体结合结构域是与cyp(seqidno:6)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的亲环素(cyp)的变体。
亲环素(cyp)的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cyp(seqidno:6)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cyp(seqidno:6)。适当地,cyp的变体能够结合fkcsa。更具体地,这种变体包含fkcsa结合序列。
根据某些实施方式,第一多聚化配体结合结构域和第二多聚化配体结合结构域选自gyrb(seqidno:7)、其香豆霉素结合片段,或其与gyrb(seqidno:7)或其香豆霉素结合片段具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是gyrb(seqidno:7),并且第二多聚化配体结合结构域选自gyrb(seqidno:7)、其香豆霉素结合片段,或其与gyrb(seqidno:7)或其香豆霉素结合片段具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是gyrb的香豆霉素结合片段,并且第二多聚化配体结合结构域选自gyrb(seqidno:7)、其香豆霉素结合片段,或其与gyrb(seqidno:7)或其香豆霉素结合片段具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些实施方式,第一多聚化配体结合结构域是gyrb(seqidno:7)或其与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gyrb(seqidno:7)或其与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是gyrb(seqidno:7),并且第二多聚化配体结合结构域是gyrb(seqidno:7)或其与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gyrb的变体,并且第二多聚化配体结合结构域是gyrb(seqidno:7)或其与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是gyrb,并且第二多聚化配体结合结构域是gyrb(seqidno:7)。
根据其他特定的实施方式,第一多聚化配体结合结构域是gyrb(seqidno:7),并且第二多聚化配体结合结构域是与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gyrb的变体。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gyrb的变体,并且第二多聚化配体结合结构域是与gyrb(seqidno:7)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gyrb的变体。
根据某些实施方式,第一多聚化配体结合结构域是gyrb的香豆霉素结合片段或其与gyrb的香豆霉素结合片段具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gyrb的香豆霉素结合片段或其与gyrb(seqidno:7)或其香豆霉素结合片段具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是gyrb的香豆霉素结合片段,并且第二多聚化配体结合结构域是gyrb的香豆霉素结合片段或其与gyrb的香豆霉素结合片段具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与gyrb的香豆霉素结合片段具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gyrb的香豆霉素结合片段的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是gyrb的香豆霉素结合片段,并且第二多聚化配体结合结构域是gyrb的香豆霉素结合片段。
gyrb的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gyrb(seqidno:7)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gyrb(seqidno:7)。适当地,gyrb的变体能够结合香豆霉素。更具体地,这种变体包含香豆霉素结合序列。
gyrb的香豆霉素结合片段的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gyrb的香豆霉素结合片段。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gyrb的香豆霉素结合片段。适当地,这种变体能够结合香豆霉素。更具体地,这种变体包含香豆霉素结合序列。
可以根据本发明使用的香豆霉素结合片段可以是gyrb的24kda氨基末端亚结构域。
根据某些实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gid1a(seqidno:9)或其与gid1a(seqidno:9)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8),并且第二多聚化配体结合结构域是gid1a(seqidno:9)或其与gid1a(seqidno:9)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gai的变体,并且第二多聚化配体结合结构域是gid1a(seqidno:9)或其与gid1a(seqidno:9)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gid1a(seqidno:9)。
根据其他特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与gid1a(seqidno:9)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gid1a的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8),并且第二多聚化配体结合结构域是gid1a(seqidno:9)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gai的变体,并且第二多聚化配体结合结构域是与gid1a(seqidno:9)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gid1a的变体。
gai的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gai(seqidno:8)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gai(seqidno:8)。适当地,gai的变体能够结合赤霉素。更具体地,这种变体包含赤霉素结合序列。
gid1a的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gid1a(seqidno:9)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gid1a(seqidno:9)。适当地,gid1a的变体能够结合赤霉素。更具体地,这种变体包含赤霉素结合序列。
根据某些实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gid1b(seqidno:10)或其与gid1b(seqidno:10)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:并且第二多聚化配体结合结构域是gid1b(seqidno:10)或其与gid1b(seqidno:10)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gai的变体,并且第二多聚化配体结合结构域是gid1b(seqidno:10)或其与gid1b(seqidno:10)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gid1b(seqidno:10)。
根据其他特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与gid1b(seqidno:10)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gid1b的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8),并且第二多聚化配体结合结构域是gid1b(seqidno:10)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gai的变体,并且第二多聚化配体结合结构域是与gid1b(seqidno:10)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gid1b的变体。
gid1b的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gid1b(seqidno:10)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gid1b(seqidno:10)。适当地,gid1b的变体能够结合赤霉素。更具体地,这种变体包含赤霉素结合序列。
根据某些实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gid1c(seqidno:11)或其与gid1c(seqidno:11)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8),并且第二多聚化配体结合结构域是gid1c(seqidno:11)或其与gid1c(seqidno:11)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gai的变体,并且第二多聚化配体结合结构域是gid1c(seqidno:11)或其与gid1c(seqidno:11)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是gid1c(seqidno:11)。
根据其他特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8)或其与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与gid1c(seqidno:11)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gid1c的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是gai(seqidno:8),并且第二多聚化配体结合结构域是gid1c(seqidno:11)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与gai(seqidno:8)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gai的变体,并且第二多聚化配体结合结构域是与gid1c(seqidno:11)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的gid1c的变体。
gid1c的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gid1c(seqidno:11)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于gid1c(seqidno:11)。适当地,gid1c的变体能够结合赤霉素。更具体地,这种变体包含赤霉素结合序列。
根据某些实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12)或其与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是halo标签(seqidno:13)或其与halo标签(seqidno:13)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12),并且第二多聚化配体结合结构域是halo标签(seqidno:13)或其与halo标签(seqidno:13)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的snap标签的变体,并且第二多聚化配体结合结构域是halo标签(seqidno:13)或其与halo标签(seqidno:13)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12)或其与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是halo标签(seqidno:13)。
根据其他特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12)或其与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与halo标签(seqidno:13)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的halo标签的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12),并且第二多聚化配体结合结构域是halo标签(seqidno:13)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的snap标签的变体,并且第二多聚化配体结合结构域是与halo标签(seqidno:13)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的halo标签的变体。
snap标签的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于snap标签(seqidno:12)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于snap标签(seqidno:12)。适当地,snap标签的变体能够结合haxs。更具体地,这种变体包含haxs结合序列。
halo标签的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于halo标签(seqidno:13)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于halo标签(seqidno:13)。适当地,halo标签的变体能够结合haxs。更具体地,这种变体包含haxs结合序列。
根据某些实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12)或其与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是clip标签(seqidno:14)或其与clip标签(seqidno:14)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:并且第二多聚化配体结合结构域是clip标签(seqidno:14)或其与clip标签(seqidno:14)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的snap标签的变体,并且第二多聚化配体结合结构域是clip标签(seqidno:14)或其与clip标签(seqidno:14)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12)或其与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是clip标签(seqidno:14)。
根据其他特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12)或其与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与clip标签(seqidno:14)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的clip标签的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是snap标签(seqidno:12),并且第二多聚化配体结合结构域是clip标签(seqidno:14)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与snap标签(seqidno:12)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的snap标签的变体,并且第二多聚化配体结合结构域是与clip标签(seqidno:14)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的clip标签的变体。
clip标签的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于clip标签(seqidno:14)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于clip标签(seqidno:14)。适当地,clip标签的变体能够结合sc。更具体地,这种变体包含sc结合序列。
根据某些实施方式,第一多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是dhfr(seqidno:15),并且第二多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的dhfr的变体,并且第二多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是dhfr(seqidno:15)。
根据其他特定的实施方式,第一多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的dhfr的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是dhfr(seqidno:15),并且第二多聚化配体结合结构域是dhfr(seqidno:15)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的dhfr的变体,并且第二多聚化配体结合结构域是与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的dhfr的变体。
dhfr的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于dhfr(seqidno:15)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于dhfr(seqidno:15)。适当地,dhfr的变体能够结合bismtx。更具体地,这种变体包含bismtx结合序列。
根据某些实施方式,第一多聚化配体结合结构域是糖皮质激素受体配体结合结构域(seqidno:16)或其与所述糖皮质激素受体配体结合结构域(seqidno:16)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是糖皮质激素受体配体结合结构域(seqidno:16),并且第二多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与糖皮质激素受体配体结合结构域(seqidno:16)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的糖皮质激素受体配体结合结构域的变体,并且第二多聚化配体结合结构域是dhfr(seqidno:15)或其与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是糖皮质激素受体配体结合结构域(seqidno:16)或其与糖皮质激素受体配体结合结构域(seqidno:16)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是dhfr(seqidno:15)。
根据其他特定的实施方式,第一多聚化配体结合结构域是糖皮质激素受体配体结合结构域(seqidno:16)或其与糖皮质激素受体配体结合结构域(seqidno:16)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的dhfr的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是糖皮质激素受体配体结合结构域(seqidno:16),并且第二多聚化配体结合结构域是dhfr(seqidno:15)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与糖皮质激素受体配体结合结构域(seqidno:16)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的糖皮质激素受体配体结合结构域的变体,并且第二多聚化配体结合结构域是与dhfr(seqidno:15)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的dhfr的变体。
糖皮质激素受体配体结合结构域(seqidno:16)的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于糖皮质激素受体配体结合结构域(seqidno:16)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于dhfr(seqidno:15)。适当地,糖皮质激素受体配体结合结构域(seqidno:16)的变体能够结合dex-mtx或dex-tmp。更具体地,这种变体包含dex-mtx或dex-tmp结合序列。
根据某些实施方式,第一多聚化配体结合结构域是pyl1(seqidno:17)或其与pyl1(seqidno:17)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是abi1(seqidno:18)或其与abi1(seqidno:18)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据特定的实施方式,第一多聚化配体结合结构域是pyl1(seqidno:17),并且第二多聚化配体结合结构域是abi1(seqidno:18)或其与abi1(seqidno:18)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是与pyl1(seqidno:17)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的pyl1的变体,并且第二多聚化配体结合结构域是abi1(seqidno:18)或其与abi1(seqidno:18)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据其他特定的实施方式,第一多聚化配体结合结构域是pyl1(seqidno:17)或其与pyl1(seqidno:17)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是abi1(seqidno:18)。
根据其他特定的实施方式,第一多聚化配体结合结构域是pyl1(seqidno:17)或其与pyl1(seqidno:17)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体,并且第二多聚化配体结合结构域是与abi1(seqidno:18)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的abi1的变体。
根据更特定的实施方式,第一多聚化配体结合结构域是pyl1(seqidno:17),并且第二多聚化配体结合结构域是abi1(seqidno:18)。
根据其他更特定的实施方式,第一多聚化配体结合结构域是与pyl1(seqidno:17)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的pyl1的变体,并且第二多聚化配体结合结构域是与abi1(seqidno:18)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的abi1的变体。
pyl1的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于pyl1(seqidno:17)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于pyl1(seqidno:17)。适当地,pyl1的变体能够结合s-(+)-脱落酸(aba)。更具体地,这种变体包含s-(+)-脱落酸(aba)结合序列。
abi1的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于abi1(seqidno:18)。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于abi1(seqidno:18)。适当地,abi1的变体能够结合s-(+)-脱落酸(aba)。更具体地,这种变体包含s-(+)-脱落酸(aba)结合序列。
第一多聚化配体结合结构域和第二多聚化配体结合结构域可以彼此直接融合或可以由肽接头分开。
因此,根据某些实施方式,第一多聚化配体结合结构域和第二多聚化配体结合结构域由肽接头分开。肽接头可以由最高达50个氨基酸组成,例如最高达25个氨基酸。根据某些实施方式,肽接头由5至25个氨基酸组成。可以根据本发明使用的肽接头的非限制性实例包括四-eaaar-接头(seqidno:19)、–gs-4x-eaaar-接头(seqidno:20)及其变体。因此,根据特定的实施方式,肽接头是四-eaaar-接头(seqidno:19)或其与四-eaaar-接头(seqidno:19)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。根据其他特定的实施方式,肽接头是–gs-4x-eaaar-接头(seqidno:20)或其与–gs-4x-eaaar-接头(seqidno:20)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
根据某些其他实施方式,第一多聚化配体结合结构域和第二多聚化配体结合结构域直接融合(例如,第一多聚化配体结合结构域的c末端与第二多聚化配体结合结构域的n末端直接融合)。”直接融合”意指在第一多聚化配体结合结构域和第二多聚化配体结合结构域之间不存在肽接头,即两个结构域通过碳-氮键连接。
第一多聚化配体结合结构域和第二多聚化配体结合结构域可以以任何可能的顺序排列,即第一多聚化配体结合结构域可以位于第二多聚化配体结合结构域的n末端,或者第一多聚化配体结合结构域可以位于第二多聚化配体结合结构域的c-末端。因此,根据某些实施方式,第一多聚化配体结合结构域位于第二多聚化配体结合结构域的n-末端。根据其他某些实施方式,第一多聚化配体结合结构域位于第二多聚化配体结合结构域的c末端。
根据本发明的嵌合抗原受体还可以在至少一个胞外域中包含铰链。铰链适当地位于开关结构域和跨膜结构域之间。
本文使用的术语“铰链”或“铰链区”通常意指用于将跨膜结构域连接至开关结构域的任何寡肽或多肽。特别地,铰链用于为细胞外配体结合结构域提供更多的柔性和可接近性。铰链区可包含最高达300个氨基酸,优选为10至100个氨基酸,并且最优选为25至50个氨基酸。铰链区可衍生自天然存在的分子的全部或部分,例如衍生自cd8、cd4或cd28的胞外区的全部或部分,或衍生自抗体恒定区的全部或部分。可替代地,铰链区可以是对应于天然存在的铰链序列的合成序列,或者可以是完全合成的铰链序列。可以根据本发明使用的铰链的非限制性实例包括以下的部分:人cd8α链、fcγriiiα受体或igg1。
根据某些实施方式,铰链选自由cd8α铰链、igg1铰链和fcγriiiα铰链组成的组。
根据特定的实施方式,铰链是cd8α铰链(seqidno:21)或其与cd8α铰链(seqidno:21)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。根据更特定的实施方式,铰链是cd8α铰链(seqidno:21)。根据其他更特定的实施方式,铰链是与cd8α铰链(seqidno:21)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的cd8α铰链的变体。cd8α铰链的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cd8α铰链。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cd8α铰链。
根据其他特定的实施方式,铰链是igg1铰链(seqidno:22)或其与igg1铰链(seqidno:22)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。根据更特别定的实施方式,铰链是igg1铰链(seqidno:22)。根据其他更特定的实施方式,铰链是与igg1铰链(seqidno:22)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的igg1铰链的变体。igg1铰链的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于igg1铰链。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于igg1铰链。
根据其他特定的实施方式,铰链是fcγriiiα铰链(seqidno:23)或其与fcγriiiα铰链(seqidno:23)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。根据更特定的实施方式,铰链是fcγriiiα铰链(seqidno:23)。根据其他更特定的实施方式,铰链是与fcγriiiα铰链(seqidno:23)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fcγriiiα铰链的变体。fcγriiiα铰链的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于fcγriiiα铰链。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于fcγriiiα铰链。
由嵌合抗原受体的胞外域组成的“细胞外抗原结合结构域”可以是能够结合配体,更具体为抗原的任何寡肽或多肽。优选地,该结构域能够与细胞表面分子相互作用。例如,可以选择细胞外配体结合结构域以识别用作与特定疾病状态相关的靶细胞上的细胞表面标志物的配体。因此,可用作配体的细胞表面标志物的实例包括与病毒、细菌和寄生虫感染、自体免疫疾病和癌细胞相关的那些。特别地,胞外配体结合结构域可以包含衍生自针对靶抗原的抗体的抗原结合片段。
因此,根据某些实施方式,细胞外抗原结合结构域包含抗体或其抗原结合片段。抗原结合片段可以是例如scfv或fab。
根据特定的实施方式,细胞外抗原结合结构域是scfv,优选地衍生自针对靶抗原的单克隆抗体的scfv。更具体地,胞外配体结合结构域可以包含单链抗体片段(scfv),该单链抗体片段(scfv)包含靶抗原特异性单克隆抗体的轻链(vl)和重链(vh)可变片段,可选地由肽接头连接,该肽接头由例如5至25个氨基酸组成(例如seqidno:24所示的ggggsggggsggggs-接头)。
根据其他特定的实施方式,细胞外抗原结合结构域是fab,优选地衍生自针对靶抗原的单克隆抗体的fab。
作为非限制性实例,靶抗原可以是任何分化分子的簇(分化分子例如,cd16、cd64、cd78、cd96、cll1、cd116、cd117、cd71、cd45、cd71、cd123和cd138)、肿瘤相关表面抗原如erbb2(her2/neu)、癌胚抗原(cea)、上皮细胞粘附分子(epcam)、表皮生长因子受体(egfr)、egfr变体iii(egfrviii)、cd19、cd20、cd30、cd40、双唾液酸神经节苷脂gd2、导管-上皮粘蛋白(mucine)、gp36、tag-72、鞘糖脂、胶质瘤相关抗原、β-人体绒毛膜促性腺激素、甲胎蛋白(afp)、凝集素-反应性afp、甲状腺球蛋白、rage-1、mn-caix、人端粒酶逆转录酶、ru1、ru2(as)、肠羧基酯酶、m-csf、前列腺酶、前列腺特异性抗原(psa)、pap、ny-eso-1、laga-1a、p53、前列腺特异性蛋白(prostein)、psma、存活和端粒酶、前列腺癌肿瘤抗原-1(pcta-1)、mage、elf2m、中性白细胞弹性蛋白酶、肝配蛋白b2、cd22、胰岛素生长因子(igf1)-i、igf-ii、igfi受体、间皮素、呈递肿瘤特异性肽表位的主要组织相容性复合体(mhc)分子、5t4、ror1、nkp30、nkg2d、肿瘤基质抗原、纤连蛋白的额外结构域a(eda)和额外结构域b(edb)和腱生蛋白-c的a1结构域(tnca1)和成纤维细胞相关蛋白(fap);谱系特异性或组织特异性抗原如cd3、cd4、cd8、cd24、cd25、cd33、cd34、cd133、cd138、ctla-4、b7-1(cd80)、b7-2(cd86)、gm-csf、细胞因子受体、内皮糖蛋白、主要组织相容性复合体(mhc)分子、bcma(cd269、tnfrsf17),或病毒特异性表面抗原如hiv特异性抗原(例如hivgp120);hbv特异性抗原、ebv特异性抗原、cmv特异性抗原、hpv特异性抗原、拉塞(lasse)病毒特异性抗原、流感病毒特异性抗原、真菌特异性抗原或细菌特异性抗原以及这些表面标志物的任何衍生物或变体。抗原不一定是表面标记抗原,但也可以是由细胞表面处的hlai类呈递的内源小抗原。
根据某些实施方式,细胞外抗原结合结构域可以针对cd19。这样的细胞外抗原结合结构域可以是来源于cd19单克隆抗体的scfv,例如4g7(peipp,saul等人,2004)。根据特定的实施方式,所述scfv包含cd19单克隆抗体4g7免疫球蛋白γ1重链(seqidno:25)的可变片段和cd19单克隆抗体4g7免疫球蛋白κ轻链(seqidno:26或seqidno:27)的可变片段,可选地由肽接头连接。
根据某些实施方式,细胞外抗原结合结构域针对cd123。这样的细胞外抗原结合结构域可以是来源于cd123单克隆抗体的scfv。
根据其他某些实施方式,细胞外抗原结合结构域针对ror1。这样的细细胞外抗原结合结构域可以是来源于ror1单克隆抗体的scfv。
根据其他某些实施方式,细胞外抗原结合结构域针对bcma。这样的细细胞外抗原结合结构域可以是来源于bcma单克隆抗体的scfv。
根据其他某些实施方式,细胞外抗原结合结构域可以针对cd20。这样的细细胞外抗原结合结构域可以是来源于cd20单克隆抗体的scfv。
根据其他某些实施方式,细胞外抗原结合结构域可以针对cd33。这样的细细胞外抗原结合结构域可以是来源于cd33单克隆抗体的scfv。
根据本发明的嵌合抗原受体包含至少一个包含信号转导结构域的胞外域和可选的共刺激结构域。
本发明的car的信号转导结构域或细胞内信号转导结构域负责胞外配体结合结构域与靶标结合后的细胞内信号转导,获得免疫细胞和免疫应答的激活。换言之,信号转导结构域负责其中表达car的免疫细胞的至少一种正常效应子功能的激活。例如,t细胞的效应子功能可以是细胞溶解活性或包括细胞因子分泌的辅助活性。
在本申请中,术语“信号转导结构域”是指转导效应物信号功能信号并指导细胞执行特化功能的蛋白质部分。
用于单链或多链car的信号转导结构域的优选实例可以是fc受体或t细胞受体的细胞质序列和共同起作用以在抗原受体衔接后启动信号转导的共受体,以及这些序列的任何衍生物或变体以及作为相同功能性能的任何合成序列。信号转导结构域包含两种不同类型的细胞质信号转导序列,即启动抗原依赖性初级激活的那些,以及以抗原非依赖性方式起作用以提供二级信号或共刺激信号的那些。初级细胞质信号转导序列可以包含已知为itam的基于免疫受体酪氨酸的激活基序的信号转导基序。itam是用作syk/zap70类酪氨酸激酶的结合位点的各种受体的胞质内尾区中发现的定义明确的信号基序。可以根据本发明使用的itam的非限制性实例可以包括衍生自tcrζ、fcrγ、fcrβ、fcrε、cd3γ、cd3δ、cd3ε、cd3ζ、cd5、cd22、cd79a、cd79b和cd66d的那些。
根据某些实施方式,信号转导结构域包含cd3ζ信号转导结构域或fcεriβ或γ链的胞质内结构域。
根据特定的实施方式,信号转导结构域包含cd3ζ信号转导结构域。根据更特定的实施方式,信号转导结构域包含如seqidno:28列出的cd3ζ信号转导结构域。根据其他更特定的实施方式,信号转导结构域包含如seqidno:28列出的cd3ζ信号转导结构域的变体,其与如seqidno:28列出的cd3ζ信号转导结构域具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性。cd3ζ信号转导结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cd3ζ信号转导结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于cd3ζ信号转导结构域。合适的,变体是具有与针对cd3ζ信号转导结构域(seqidno:28)所见的相同或相似的功能和活性的变体。
根据其他特定的实施方式,信号转导结构域包含fcεriβ或γ链的胞质内结构域或其变体。
根据更特定的实施方式,信号转导结构域包含fcεriβ链(seqidno:29)的胞质内结构域。根据其他更特定的实施方式,信号转导结构域包含fcεriβ链(seqidno:29)的胞质内结构域的变体,其与fcεriβ链(seqidno:29)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性。fcεriβ链的胞质内结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述胞质内结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于胞质内结构域。合适的,变体是具有与针对fcεriβ链(seqidno:29)所见的相同或相似的功能和活性的变体。
根据其他更特定的实施方式,信号转导结构域包含fcεriγ链(seqidno:30)的胞质内结构域。根据其他更特定的实施方式,信号转导结构域包含与fcεriγ链(seqidno:30)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fcεriγ链(seqidno:30)的胞质内结构域的变体。fcεriγ链的胞质内结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于胞质内结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于胞质内结构域。合适的,变体是具有与针对fcεriγ链(seqidno:30)所见的相同或相似的功能和活性的变体。
根据某些实施方式,本发明的car在至少一个胞内域中包含共刺激结构域。
共刺激结构域可以是共刺激分子的任何胞质结构域。共刺激分子是除抗原受体以外的细胞表面分子或其有效免疫应答所需的配体。
“共刺激配体”是指特异性结合t细胞上的同源共刺激分子的抗原呈递细胞上的分子,从而提供一种信号,除了由例如tcr/cd3复合物与载有肽的mhc分子的结合提供的原始信号之外,该信号介导t细胞应答,包括但不限于增殖激活、分化等。共刺激配体可以包括但不限于cd7、b7-1(cd80)、b7-2(cd86)、pd-l1、pd-l2、4-1bbl、ox40l、诱导型共刺激配体(icos-l)、细胞间粘附分子(icam、cd30l、cd40、cd70、cd83、hla-g、mica、m1cb、hvem、淋巴毒素β受体、3/tr6、ilt3、ilt4、结合toll配体受体的激动剂或抗体以及与b7-h3特异性结合的配体。共刺激配体还特别地包括与t细胞上存在的共刺激分子特异性结合的抗体,如但不限于cd27、cd28、4-ibb、ox40、cd30、cd40、pd-1、icos、淋巴细胞功能相关抗原-1(lfa-1)、cd2、cd7、ltght、nkg2c、b7-h3、与cd83特异性结合的配体。
“共刺激分子”是指与共刺激配体特异性结合的t细胞上的同源结合伴侣,从而介导通过细胞的共刺激反应,例如但不限于增殖。共刺激分子包括但不限于mhci类分子、btla和toll配体受体。共刺激分子的实例包括cd27、cd28、cd8、4-1bb(cd137)、ox40、cd30、cd40、pd-1、icos、淋巴细胞功能相关抗原-1(lfa-1)、cd2、cd7、light、nkg2c、b7-h3和与cd83特异性结合的配体等。
如在本文中所使用的,“共刺激信号”是指与原始信号相结合的信号,如tcr/cd3连接,其使得关键分子的t细胞增殖和/或上调或下调。
例如,共刺激结构域可以是来自选自以下各项的共刺激分子的胞质结构域:cd27、cd28、4-1bb、ox40、cd30、cd40、pd-1、icos、淋巴细胞功能相关抗原-1(lfa-1)、cd2、cd7、cd8、light、nkg2c、b7-h3、与cd83特异性结合的配体和它们的任何组合。
因此,根据某些实施方式,共刺激结构域是来自4-1bb的共刺激结构域。根据特定的实施方式,共刺激结构域是来自如在seqidno:31中列出的4-1bb的共刺激结构域。根据其他特定的实施方式,共刺激结构域是来自如在seqidno:31中列出的4-1bb的共刺激结构域的变体,其与来自如在seqidno:31中列出的4-1bb的共刺激结构域具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性。来自4-1bb的共刺激结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述共刺激结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述共刺激结构域。合适的,变体是具有与针对来自4-1bb(seqidno:31)的共刺激结构域所见的相同或相似的功能和活性的变体。
根据本发明的嵌合抗原受体包含至少一个跨膜结构域。合适的跨膜结构域的区别特征包括在细胞、优选地在本发明中的免疫细胞、特别是淋巴细胞或自然杀伤(nk)细胞的表面表达的能力,以及一起相互作用以指导免疫细胞针对预定义的靶细胞的细胞应答的能力。至少一个跨膜结构域可以来源于天然来源或合成来源。至少一个跨膜结构域可以来源于任何膜结合蛋白或跨膜蛋白。作为非限制性实例,至少一个跨膜结构域可以是t细胞受体的亚基如α、β、γ或δ,构成cd3复合体的多肽,il2受体p55(α链)、p75(β链)或γ链,fc受体的亚基链、特别是fcγ受体iii或cd蛋白。可替代地,至少一个跨膜结构域可以是合成的并且可以主要包含疏水性残基如亮氨酸和缬氨酸。
至少一个跨膜结构域可以例如来源于cd8α链。因此,根据某些实施方式,至少一个跨膜结构域是cd8α跨膜结构域。根据特定的实施方式,至少一个跨膜结构域是cd8α跨膜结构域(seqidno:32)。根据其他特定的实施方式,至少一个跨膜结构域是cd8α跨膜结构域(seqidno:32)的变体,其与人cd8α跨膜结构域(seqidno:32)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性。cd8α跨膜结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。合适的,变体是具有与针对cd8α跨膜结构域(seqidno:32)所见的相同或相似的功能的变体。
可替代地,至少一个跨膜结构域可以来源于4-1bb。因此,根据某些实施方式,至少一个跨膜结构域是4-1bb跨膜结构域(seqidno:33)。根据特定的实施方式,至少一个跨膜结构域是4-1bb跨膜结构域(seqidno:33)。根据其他特定的实施方式,至少一个跨膜结构域是4-1bb跨膜结构域(seqidno:33)的变体,其与4-1bb跨膜结构域(seqidno:33)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性。4-1bb跨膜结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。合适的,变体是具有与针对4-1bb跨膜结构域(seqidno:33)所见的相同或相似的功能的变体。
可替代地,至少一个跨膜结构域可以来源于fcε受体链或其变体。特别地,跨膜结构域可以选自fcεriα、β和γ链、片段或其变体的跨膜结构域。因此,根据某些实施方式,至少一个跨膜结构域是来自高亲和力ige受体(fcεri)的α链(seqidno:34)的跨膜结构域。根据某些其他实施方式,至少一个跨膜结构域是与fcεriα(seqidno:34)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的fcεriα链的跨膜结构域的变体。fcεriα链的跨膜结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。合适的,变体是具有与针对fcεriα链(seqidno:34)的跨膜结构域所见的相同或相似的功能的变体。
在嵌合抗原受体是由至少两条各自含有至少一个跨膜结构域的不同多肽链组成的多链car的情况下,跨膜结构域可以例如选自fcεriα、β和γ链、片段的跨膜结构域或其变体。
因此,由含有至少一个根据本发明的胞外域的第一多肽链所包含的至少一个跨膜结构域可以是来自高亲和力ige受体(fcεri)的α链(seqidno:34)的跨膜结构域,或其与fcεriα(seqidno:34)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。由含有至少一个根据本发明的胞内域的第二多肽链组成的至少一个跨膜结构域可以是来自fcεri的γ链或β链(分别为seqidno:35和36)的跨膜结构域,或其与fcεriγ或β(分别为seqidno:35和36)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。由包含至少一个根据本发明的胞内域的第三多肽链组成的至少一个跨膜结构域可以是来自fcεri的γ链或β链(分别为seqidno:35和36)的跨膜结构域,或其与fcεriγ或β(分别为seqidno:35和36)具有至少80%如至少85%、至少90%、至少95%、至少98%或至少99%序列同一性的变体。
fcεriγ或β链的跨膜结构域的变体可能由于替换一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。可替代地或另外地,这种变体可能由于添加或删除一个或多个(例如1至10、1至5或1至3个)氨基酸残基而不同于所述跨膜结构域。合适的,变体是具有与针对fcεriγ或β链(分别为seqidno:35和36)所见的相同或相似的功能的变体。
根据本发明的嵌合抗原受体可以是单链car。单链car是嵌合抗原受体,其中构成所述car的所有结构域位于一条多肽链上。图1a中示出根据本发明的单链car的非限制性说明。
可替代地,根据本发明的嵌合抗原受体可以是多链car。图2中示出根据本发明的多链car的非限制性说明。根据这种结构,在不同的多肽链上产生至少一个胞外域和至少一个胞内域。不同的多肽链以极为贴近的方式锚定在膜中,从而允许彼此相互作用。在这样的结构中,信号转导结构域和共刺激结构域可以位于近膜位置中(即,与其内侧上的细胞膜相邻),这被认为可以改善共刺激结构域的功能。多亚基结构还提供了设计具有对t细胞激活的更好控制的car的更多灵活性和可能性。例如,可以包括几个具有不同特异性的细胞外抗原识别结构域以获得多特异性car结构。还可以控制进入多链car中的不同亚基之间的相对比例。最近申请人在pct/us2013/058005中描述了这种类型的结构。
因此,根据本发明的多链car可以是包括以下各项的多链car:
a)第一多肽链,包含
a)至少一个胞外域,其包含:
i)细胞外抗原结合结构域;和
ii)包含至少第一多聚化配体结合结构域和第二多聚化配体结合结构域的开关结构域,该第一多聚化配体结合结构域和第二多聚化配体结合结构域能够结合预定的多价配体以形成包含所述两个结合结构域和它们能够与其结合的多价配体的多聚体;和
aa)至少一个跨膜结构域;和
b)第二多肽链,其包含
b)至少一个包含信号转导结构域和可选的共刺激结构域的胞内域;
bb)至少一个跨膜结构域。
根据某些实施方式,本发明的多链car还可包含:
c)第三多肽链,其包含
c)至少一种包含共刺激结构域的胞内域;和
cc)至少一个跨膜结构域。
例如,通过使用用于ige(fcεri)的高亲和力受体的不同α、β和γ链,将不同链组装为单个多链car的部分是可能的(metzger,alcarazetal.1986)。通过用本文详述的胞外域替换fcεriα链的高亲和力ige结合结构域,可以从fcεri得到这样的多链car,而fcεriβ和/或γ链的n末端尾区和/或c末端尾区被分别融合到本文详述的包含信号转导结构域和共刺激结构域的胞外域。胞外配体结合结构域具有将t细胞特异性重定向到细胞靶标的作用,而信号转导结构域激活免疫细胞应答。衍生自fcεri的α、β和γ多肽的不同的多肽链是位于近膜位置的跨膜多肽的事实为car提供更柔性的结构,提高针对抗原靶标的特异性并降低免疫细胞的背景激活。
因此,根据特定的实施方式,包含胞外域的第一多肽链(a)包含来自高亲和力ige受体(fcεri)的α链的跨膜结构域,而包含含有信号转导结构域的胞内域的第二多肽链(b)包含来自fcεri的γ链或β链的跨膜结构域,例如来自fcεri的γ链的跨膜结构域。如果存在,则包含含有共刺激结构域的胞内域的第三多肽链(c)包含来自fcεri的γ链或β链的跨膜结构域,例如来自fcεri的β链的跨膜结构域。
嵌合抗原受体的至少一个胞外域的构象优选为使得在不存在相应多聚化配体的情况下,胞外结合结构域不能结合被靶向的抗原。然后,多聚化配体与开关结构域的结合导致构象变化,其以允许其结合被靶向的抗原的方式暴露胞外结合结构域(该机制可被称为接通)。可以基于表达所述car的免疫细胞的细胞溶解活性(细胞毒性)来确定适当的构象。“细胞溶解活性”意指由表达所述car的免疫细胞赋予的靶细胞的细胞裂解百分比。
下面描述了测定细胞毒性的方法:
使用贴壁靶细胞:将2x104特异性靶抗原(sta)阳性或sta阴性细胞以每孔0.1ml接种在96孔板中。制板后的次日,用celltracecfse标记sta阳性和sta阴性细胞并与4x105个t细胞共培养4小时。然后,收获细胞,用可固定活性染料(ebioscience)染色并使用macsquant流式细胞仪(miltenyi)分析。
使用下式计算特异性裂解的百分比:
使用悬浮液靶细胞:分别用celltracecfse和celltraceviolet标记sta阳性和sta阴性细胞。在96孔板中以0.1ml/孔将约2x104个ror1阳性细胞与2x104个sta阴性细胞以及4x105个t细胞共培养。在孵育4小时后,收获细胞,用可固定活性染料(ebioscience)染色并使用macsquant流式细胞仪(miltenyi)分析。
使用上式计算特异性裂解的百分比。
“特异性靶抗原(sta)阳性细胞”意指表达嵌合抗原受体对其显示特异性的靶抗原的细胞,而“sta阴性细胞”意指不表达特异性靶抗原的细胞。作为非限制性实例,如果car针对cd19,那么特异性靶抗原是cd19。因此,cd19阳性和cd19阴性细胞将被用于测定细胞溶解活性。
因此,取决于由免疫细胞表达的嵌合抗原受体的抗原特异性,必须调整上述细胞毒性测定以适应相应的靶细胞。
用于测定细胞溶解活性的类似方法也描述于例如valton等人,(2015)或poirot等人(2015)中。
根据某些实施方式,与不存在多聚化配体的情况下的免疫细胞的细胞溶解活性相比,根据本发明的嵌合抗原受体在存在相应多聚化配体的情况下为表达其的所述免疫细胞赋予调节的细胞溶解活性。
根据特定的实施方式,与不存在多聚化配体的情况下的免疫细胞的细胞溶解活性相比,根据本发明的嵌合抗原受体在存在相应多聚化配体的情况下为表达其的免疫细胞赋予增加的细胞溶解活性。“增加的细胞溶解活性”是指:与在不存在多聚化配体的情况下由免疫细胞赋予的靶细胞的%细胞裂解相比,在存在多聚化配体的情况下由表达所述car的免疫细胞赋予的靶细胞的%细胞裂解增加至少10%,例如至少20%、至少30%、至少40%、至少50%、至少50%、至少60%、至少70%、至少80%、至少90%或至少100%。
根据其他特定的实施方式,与不存在多聚化配体的情况下的免疫细胞的细胞溶解活性相比,根据本发明的嵌合抗原受体在存在相应多聚化配体的情况下为表达其的免疫细胞赋予降低的细胞溶解活性。“降低的细胞溶解活性”是指与在不存在多聚化配体的情况下由免疫细胞赋予的靶细胞的%细胞裂解相比,在存在多聚化配体的情况下由表达所述car的免疫细胞赋予的靶细胞的%细胞裂解减少至少10%,例如至少20%、至少30%、至少40%、至少50%、至少50%、至少60%、至少70%、至少80%、至少90%或至少100%。
“相应的多聚化配体”意指通过第一多聚化配体结合结构域和第二多聚化配体结合结构域二者结合的多聚化配体,并因此促进第一多聚化配体结合结构域和第二多聚化配体结合结构域之间的多聚化(例如二聚化)。作为非限制性实例,如果第一多聚化配体结合结构域是kbp12并且第二多聚化配体结合结构域是frb,那么”相应的配体结合结构域”可以是雷帕霉素。
多核苷酸、载体:
本发明还涉及包含编码根据本发明的嵌合抗原受体的一个或多个核苷酸序列的多核苷酸和载体。本发明提供了包含编码嵌合抗原受体的一个或多个核苷酸序列的多核苷酸,包括dna和rna分子。在嵌合抗原受体是多链car的情况下,提供至少一种多核苷酸,其包含两个或多个编码包含根据本发明的多链car的多肽链的核苷酸序列。根据某些实施方式,提供了包含含有编码第一多肽链的核苷酸序列的第一多核苷酸和含有编码第二多肽链的核苷酸序列的第二多核苷酸的组合物。可选地,组合物包含含有编码第三多肽链的核苷酸序列的第三多核苷酸。
可以由表达盒或表达载体(例如,用于引入细菌宿主细胞的质粒,或用于转染昆虫宿主细胞的病毒载体如杆状病毒载体,或质粒或病毒载体如用于转染哺乳动物宿主细胞的慢病毒)包含一种或多种多核苷酸。
根据某些实施方式,不同的核苷酸序列可以被包括在一个多核苷酸或载体中,其包含编码核糖体跳读序列的核苷酸序列,例如编码2a肽的序列。在小核糖核酸病毒的口蹄疫病毒(aphthovirus)亚群中识别的2a肽导致从一个密码子核糖体“跳读”到下一个密码子,而不在由密码子编码的两个氨基酸之间形成肽键(参见donnelly等人,j.ofgeneralvirology82:1013-1025(2001);donnelly等人,j.ofgen.virology78:13-21(1997);doronina等人,mol.and.cell.biology28(13):4227-4239(2008);atkins等人,rna13:803-810(2007))。“密码子”意指mrna(或dna分子的有义链)上的三个核苷酸,其由核糖体翻译成一个氨基酸残基。因此,当多肽被非移码(inframe)的2a寡肽序列分开时,两个多肽可由mrna内的单个邻接开放读码框架合成。这种核糖体跳读机制是本领域公知的,并且已知被几种载体使用用于表达几种由单个信使rna编码的蛋白质。作为非限制性实例,在本发明中,2a肽已用于在细胞中表达多链car的不同多肽。
为了将跨膜多肽如fcεr引入宿主细胞的分泌途径,在多核苷酸序列或载体序列中提供分泌信号序列(还称为前导序列、前原(prepro)序列或前(pre)序列)。分泌信号序列可以是fcεr的分泌信号序列,或者可以来源于另一种分泌蛋白(例如,t-pa)或是从头合成的。分泌信号序列与跨膜核酸序列可操作地连接,即两个序列以正确的读码框形式连接并定位以将新合成的多肽引导到宿主细胞的分泌途径中。分泌信号序列通常位于编码感兴趣的多肽的核酸序列的5’,尽管某些分泌信号序列可以位于感兴趣的核酸序列中的其他位置(参见例如,welch等人,美国专利号5,037,743;holland等人,美国专利号5,143,830)。在一个优选的实施方式中,信号肽包含fcεriα链的残基1至25。
本领域技术人员会认识到,考虑到遗传密码的简并性,在这些多核苷酸分子中可能存在相当多的序列变异。优选地,本发明的核苷酸序列是针对在哺乳动物细胞中表达、优选用于在人类细胞中表达被密码子优化的。密码子优化是指由给定物种的高度表达的基因中通常常见的密码子交换在这种物种的高度表达的基因中通常罕见的感兴趣的密码子的序列,这种密码子作为正在交换的密码子编码氨基酸。
工程化免疫细胞的方法
本发明还涉及制备用于免疫疗法的免疫细胞的方法,包括将根据本发明的car引入所述免疫细胞中并使所述细胞扩增。特别地,提供了一种用于工程化免疫细胞的方法,所述方法包括:
(i)提供免疫细胞,例如t细胞;和
(ii)在所述免疫细胞表面表达至少一种根据本发明的嵌合抗原受体。
根据某些实施方式,方法包括:
(a)提供免疫细胞;
(b)将至少一种根据本发明的多核苷酸或载体引入所述细胞中;和
(c)在所述细胞中表达本发明的嵌合抗原受体。
在优选的实施方式中,考虑到在细胞中稳定表达,所述多核苷酸被包括在慢病毒载体中。
例如,为了增强抗肿瘤效果,考虑在免疫细胞的表面上进一步表达至少一种共刺激受体。因此,用于工程化免疫细胞的方法可以包括(iii)在免疫细胞的表面上表达至少一种共刺激受体。
根据某些实施方式,方法还包括:
(d)将至少一种包含编码共刺激受体的核苷酸序列的多核苷酸引入所述细胞中;和
(e)表达所述至少一种共刺激受体。
如在本文中所使用的,“共刺激受体”意指调节通过t细胞受体(tcr)对t淋巴细胞的激活的受体家族的成员。受体响应于在抗原呈递细胞上发现的一种或多种b7抗原有,并且取决于特异性配体-受体组合调节各种t细胞功能,例如克隆扩增的速率、细胞存活和细胞因子产生。根据本发明待由免疫细胞表达的合适的共刺激受体的非限制性实例包括nkg2d(uniprotkb:p26718)和dap10(uniprotkb:q9ubk5)。
根据某些实施方式,免疫细胞在其表面上至少表达nkg2d。
根据某些其他实施方式,免疫细胞在其表面上至少表达dap10。
至少一种共刺激受体的表达可以是瞬时的或组成型的。因此,根据某些实施方式,至少一种共刺激受体由免疫细胞瞬时表达。根据其他某些实施方式,至少一种共刺激受体由免疫细胞组成型表达。
递送方法
上述不同的方法涉及将car引入细胞中。作为非限制性实例,可以作为由一个质粒载体编码的转基因引入所述car。所述质粒载体还可含有选择标志物,其提供对于接收所述载体的细胞的识别和/或选择。
作为将编码所述多肽的多核苷酸引入到细胞中的结果,可以在细胞中原位合成多肽。或者,所述多肽可以在细胞外产生,然后被引入其中。将多核苷酸构建体引入到细胞中的方法是本领域已知的,并且包括作为非限制性实例的其中将多核苷酸构建体整合到细胞的基因组中的稳定转化方法,其中未将多核苷酸构建体整合到细胞的基因组中的瞬时转化方法和病毒介导的方法。可以通过例如重组病毒载体(例如,逆转录病毒、腺病毒)、脂质体等将所述多核苷酸引入到细胞中。例如,瞬时转化方法包括例如显微注射、电穿孔或粒子轰击。考虑到在细胞中表达,所述多核苷酸可以包括在载体中,更特别地包括在质粒或病毒中。
工程化免疫细胞
本发明还涉及免疫细胞,例如易于通过所述工程化细胞的方法获得的分离的免疫细胞或细胞系。
具体地,根据本发明的免疫细胞,例如分离的免疫细胞包含至少一种本发明的car。根据某些实施方式,免疫细胞例如分离的免疫细胞包含car群体,每一种car包含不同的胞外配体结合结构域。特别地,所述免疫细胞,例如分离的免疫细胞包含编码构成至少一个car的一种或多种多肽的一个或多个外源多核苷酸序列。本发明的基因修饰的免疫细胞独立于抗原结合机制而被激活和增殖。
如本文所提及的,“免疫细胞”意指功能上参与先天和/或适应性免疫应答的启动和/或执行的造血起源的细胞。根据本发明的免疫细胞可衍生自干细胞。干细胞可是成体干细胞、非人胚胎干细胞,更特别为非人类干细胞、脐带血干细胞、祖细胞、骨髓干细胞、诱导性多能干细胞、全能干细胞或造血干细胞。代表性的人类细胞是cd34+细胞。所述免疫细胞还可以是选自炎性t淋巴细胞、细胞毒性t淋巴细胞、调节性t淋巴细胞或辅助性t淋巴细胞组成的组的树突状细胞、杀手树突状细胞、肥大细胞、nk细胞、b细胞或t细胞。根据特定的实施方式,所述免疫细胞可以来源于由cd4+t淋巴细胞和cd8+t淋巴细胞组成的组。根据更特定的实施方式,所述免疫细胞可以来源于cd4+t淋巴细胞。
根据某些实施方式,免疫细胞是人免疫细胞,例如人t-淋巴细胞。
由于雷帕霉素通过与胞质蛋白fk结合蛋白12(fkbp12)相互作用,然后通过fkbp12/雷帕霉素复合物抑制mtor(哺乳动物雷帕霉素靶标)而直接抑制免疫细胞如t细胞,因此可能期望抑制内源性fkbp12/雷帕霉素/mtor复合物的形成。
例如,可以通过在mtor蛋白质序列(ncbi参考序列:np_004949.1;seqidno:37)内引入一个或多个氨基酸替换(包括2035位的氨基酸替换)来实现内源性fkbp12/雷帕霉素/mtor复合物形成的抑制。用于在蛋白质的氨基酸序列内引入氨基酸替换的技术是本领域技术人员熟知的,并且包括作为非限制性实例的定向诱变。
因此,可以进一步修饰本发明的免疫细胞以在内源性mtor蛋白质内包含至少2035位的氨基酸替换,其中丝氨酸被另一种氨基酸替换。因此,根据某些实施方式,免疫细胞在内源性mtor蛋白质的氨基酸序列内包含一个或多个氨基酸替换,包括在位置2035位的氨基酸替换,其中丝氨酸被另一种氨基酸如ile替换。
例如,还可以通过使内源性fkbp12基因失活来实现内源性fkbp12/雷帕霉素/mtor复合物形成的抑制。“使基因失活(inactivatingagene)”或“失活基因(inactivationofagene)”意图是感兴趣的基因(例如,kfbp12基因)不以功能蛋白形式表达。用于使基因失活的技术是本领域技术人员熟知的,并且作为非限制性实例包括使用靶向该基因的特异性稀切核酸内切酶,如tal核酸酶、大范围核酸酶、锌指核酸酶(zfn)或rna指导的核酸内切酶。另外的非限制性实例包括例如分别在wo2014/191128和swarts等人(2014)中公开的基于cas9/crispr或argonaute(ago)的系统。
因此,可以进一步修饰本发明的免疫细胞以使内源性fkbp12基因失活。因此,根据某些实施方式,免疫细胞包含失活的fkbp12基因。因此,这种免疫细胞不表达fkpb12蛋白质。
为了使免疫细胞保持增殖状态,避免早期重新施用新的工程化免疫细胞,使用病毒特异性t细胞(vst)可能是合适的。它们的天然形式不具有细胞毒性,vst通过衔接它们的天然受体被内源性病毒抗原刺激,然后被允许增殖。不管是否存在car靶抗原,都会发生扩增和持久生存。当根据本发明进行工程化时,即带有胞外域开关系统时,vst可以从它们的增殖性能中受益而不存在car靶抗原,而非vstt细胞不会增殖并最终死亡。工程化以表达cd19特异性嵌合抗原受体及其生成的供体来源的病毒特异性t细胞描述于cruz等人,(2013)中。
因此,根据某些实施方式,免疫细胞是病毒特异性t细胞(vst),优选从供体分离的。
在本发明的免疫细胞的扩增和基因修饰之前,可以通过各种非限制性方法从受试者获得细胞来源。可以从许多非限制性来源获得细胞,包括外周血单核细胞、骨髓、淋巴结组织、脐带血、胸腺组织、来自感染部位的组织、腹水、胸腔积液、脾组织和肿瘤。在本发明的某些实施方式中,可以使用本领域技术人员可获得和已知的任何数量的t细胞系。在另一个实施方式中,所述细胞可衍生自健康供体、诊断患有癌症的患者或诊断患有感染的患者。在另一个实施方式中,所述细胞是呈现不同表型特征的混合细胞群的一部分。在本发明的范围内还包括根据前述方法从转化的t细胞获得的细胞系。对免疫抑制治疗有抗性且易于通过前述方法获得的修饰细胞包括在本发明的范围内。
根据本发明的免疫细胞可被进一步修饰成同种异体的。因此,根据某些实施方式,免疫细胞还包含至少一种失活基因,其选自由cd52、gr、tcrα、tcrβ、hla基因、免疫检查点基因如pd1和ctla-4组成的组,或者可以表达ptα转基因。更特别地,免疫细胞可以包含至少一种选择的tcrα或tcrβ基因的失活基因。这种失活使得tcr在细胞中没有功能。该策略对于避免移植物抗宿主病(gvhd)是特别有用的。使基因失活的方法是本领域已知的,并且包括使用能够通过dna切割(优选地通过双链断裂)使感兴趣的一个或多个基因选择性地失活的稀切核酸内切酶。因此,基因可以通过用包含编码能够通过dna切割选择性失活的稀切核酸内切酶的核苷酸序列的多核苷酸,优选地通过双链断裂选自cd52、gr、tcrα、tcrβ、hla基因、免疫检查点基因如pd1和ctla-4的基因转化免疫细胞而被失活。所述稀切核酸内切酶可以是大范围核酸酶、锌指核酸酶或tale核酸酶。根据特定的实施方式,所述稀切核酸内切酶是tale核酸酶。wo2013176915中描述了优选的方法和相关的tale核酸酶。根据其他特定的实施方式,所述稀切核酸内切酶是rna指导的核酸内切酶,例如cas9或dna引导的核酸内切酶,例如wo2014189628中描述的基于argonaute的技术。
根据本发明的免疫细胞可被进一步修饰成对化疗药物有耐药性的。因此,根据某些实施方式,免疫细胞还包含至少一种失活的负责细胞对药物敏感的基因(一种或多种药物致敏基因),如dck和/或hprt基因。使基因失活的方法是本领域已知的,并且包括使用能够通过dna切割、优选地通过双链断裂选择性地使一种或多种感兴趣的基因失活的稀切核酸内切酶。因此,所述一种或多种基因可以通过用包含编码能够通过dna切割选择性失活的稀切核酸内切酶的核苷酸序列的多核苷酸、优选通过双链断裂至少一种负责细胞对药物(一种或多种药物致敏基因)敏感的基因转化免疫细胞来失活。所述稀切核酸内切酶可以是大范围核酸酶、锌指核酸酶或tale核酸酶。根据特定的实施方式,所述稀切核酸内切酶是tale核酸酶。wo2013176915中描述了优选的方法和相关的tale核酸酶。根据其他特定的实施方式,所述稀切核酸内切酶是rna指导的核酸内切酶,例如cas9或dna引导的核酸内切酶,例如wo2014189628中的基于argonaute的技术。
可替代地,通过表达抗药性基因,可将对药物的抗性赋予免疫细胞如t细胞。根据本发明已经识别了几种基因如二氢叶酸还原酶(dhfr)、肌苷单磷酸脱氢酶2(impdh2)、钙调磷酸酶或甲基鸟嘌呤转移酶(mgmt)的变体等位基因赋予免疫细胞抗药性。
t细胞的激活和扩增
无论在免疫细胞的基因修饰之前或之后,即使本发明的基因修饰的免疫细胞独立于抗原结合机制而被激活和增殖,也可通常使用例如在美国专利6,352,694;6,534,055;6,905,680;6,692,964;5,858,358;6,887,466;6,905,681;7,144,575;7,067,318;7,172,869;7,232,566;7,175,843;5,883,223;6,905,874;6,797,514;6,867,041和美国专利申请公开号20060121005中描述的方法进一步激活和扩增本发明的免疫细胞,特别是t细胞。可以在体外或体内扩增t细胞。
通常,通过与刺激t细胞表面上的cd3tcr复合物和共刺激分子的试剂接触而扩增本发明的免疫细胞以产生t细胞的激活信号。
例如,可以使用化学物质如钙离子载体a23187、佛波醇12-肉豆蔻酸酯13-乙酸酯(pma)或促有丝分裂凝集素如植物血球凝集素(pha)来产生t细胞的激活信号。
作为非限制性实例,可以在体外刺激t细胞群体,例如通过与抗cd3抗体或其抗原结合片段或固定在表面上的抗cd2抗体接触、或者通过与结合钙离子载体的蛋白激酶c激活剂(例如,苔藓抑素)接触。为了共刺激t细胞表面上的辅助分子,使用结合辅助分子的配体。例如,在适于刺激t细胞增殖的条件下,可将t细胞群体与抗cd3抗体和抗cd28抗体接触。适用于t细胞培养的条件包括可能含有增殖和活力所必需的因子的合适培养基(例如,最低必需培养基或rpmi培养基1640或x-vivo5,(lonza)),所述因子包括血清(例如,胎牛或人类血清)、白细胞介素-2(il-2)、胰岛素、ifn-g、1l-4、1l-7、gm-csf、gm-10、gm-2、1l-15、tgfβ和tnf-或本领域技术人员已知的用于细胞生长的任何其他添加剂。用于细胞生长的其他添加剂包括但不限于表面活性剂、人血浆蛋白粉和还原剂如n-乙酰基-半胱氨酸和2-巯基乙醇。培养基可以包括rpmi1640、a1m-v、dmem、mem、a-mem、f-12、x-vivo1和x-vivo20,优化剂,添加氨基酸、丙酮酸钠和维生素,无血清或补充有适量的血清(或血浆)或一组定义的激素,和/或一定量的足以使t细胞生长和扩增的细胞因子。抗生素例如青霉素和链霉素仅包括在实验培养物中,而不是在要输注到受试者的细胞的培养物中。靶细胞保持在支持生长所必需的条件下,例如适当的温度(例如,37℃)和气氛(例如,空气加5%的co2)。已暴露于不同刺激时间的t细胞可能表现出不同的特征。
根据某些实施方式,可以通过与组织或细胞共培养来扩增细胞。还可以在体内扩增所述细胞,例如在将所述细胞施用于受试者之后在受试者的血液中。
治疗应用
根据本发明可获得的免疫细胞旨在用作药物,并且特别地用于治疗有需要的患者(例如,人类患者)中的癌症。因此,本发明提供了用作药物的免疫细胞。特别地,本发明提供了用于治疗癌症的免疫细胞。还提供了组合物,特别是药物组合物,其包含至少一种本发明的免疫细胞。在某些实施方式中,组合物可以包含本发明的免疫细胞群体。
治疗可以是改善、治愈或预防。它可能是自体免疫治疗的一部分或同种异体免疫治疗的一部分。自体意思是用于治疗患者的细胞、细胞系或细胞群源自所述患者或人类白细胞抗原(hla)相容的供体。同种异体意思是用于治疗患者的细胞或细胞群不是源自所述患者而是源自供体。
本发明特别适用于同种异体免疫治疗,只要它能够将通常从供体获得的免疫细胞如t细胞转化为非同种异体反应性细胞。这可以在标准方案下完成,并根据需要可多次再现。可将所得的修饰的免疫细胞汇集并施用于一个或几个患者,可作为”现成的”治疗产品获得。
治疗主要是治疗被诊断患有癌症的患者。根据本发明要治疗的具体癌症是那些具有实体瘤但也可能涉及液体瘤的癌症。还包括成人肿瘤/癌症和儿科肿瘤/癌症。
根据某些实施方式,一种或多种免疫细胞或者组合物用于治疗癌症,更具体地用于治疗实体瘤或液体瘤。根据特定的实施方式,一种或多种免疫细胞或者组合物用于治疗实体瘤。根据其他特定的实施方式,一种或多种免疫细胞或者组合物用于治疗液体瘤。
根据特定的实施方式,一种或多种免疫细胞或者组合物用于治疗选自肺癌、小细胞肺癌(小肺癌,smalllungcancer)、乳癌、子宫癌、前列腺癌、肾癌、结肠癌、肝癌、胰腺癌和皮肤癌的癌症。根据更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗肺癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗小细胞肺癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗乳癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗子宫癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗前列腺癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗肾癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗结肠癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗肝癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗胰腺癌。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗皮肤癌。
根据其他特定的实施方式,一种或多种免疫细胞或者组合物用于治疗肉瘤。
根据其他特定的实施方式,一种或多种免疫细胞或者组合物用于治疗肿瘤(carcinoma)。根据更特定的实施方式,免疫细胞或组合物用于治疗肾肿瘤、肺肿瘤或结肠肿瘤。
根据其他特定的实施方式,一种或多种免疫细胞或者组合物用于治疗白血病,例如急性淋巴细胞白血病(all)、急性髓性白血病(aml)、慢性淋巴细胞白血病(cll)、慢性髓细胞性白血病(cml)和慢性粒单核细胞白血病(cmml)。根据更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗急性淋巴细胞白血病(all)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗急性髓性白血病(aml)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗慢性淋巴细胞白血病(cll)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗慢性髓细胞性白血病(cml)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗慢性粒单核细胞白血病(cmml)。
根据其他特定的实施方式,一种或多种免疫细胞或者组合物用于治疗淋巴瘤,例如b-细胞淋巴瘤。根据更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗原发性cns淋巴瘤。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗霍奇金淋巴瘤。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗非霍奇金淋巴瘤。根据更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗弥漫性大b细胞淋巴瘤(dlbcl)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗滤泡性淋巴瘤。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗边缘带淋巴瘤(mzl)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗黏膜相关性淋巴组织淋巴瘤(malt)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗小细胞淋巴细胞性淋巴瘤。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗套细胞淋巴瘤(mcl)。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗伯基特淋巴瘤。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗原发性纵隔(胸腺)大b细胞淋巴瘤。根据其他更特定的实施方式,一种或多种免疫细胞或者组合物用于治疗瓦尔登斯特伦
根据某些实施方式,一种或多种免疫细胞或者组合物用于治疗病毒性感染,例如hiv感染或hbv感染。
根据某些实施方式,来源于患者(例如,人患者)的免疫细胞将被治疗。根据某些其他实施方式,免疫细胞来源于至少一个供体。
治疗可联合一种或多种选自抗体治疗、化疗、细胞因子治疗、树突细胞治疗、基因治疗、激素治疗、激光治疗和放射治疗的组中的疗法进行。
根据某些实施方式,本发明的免疫细胞在施用于患者后可以进行强健的体内免疫细胞扩增,并且可以在体液中持续延长的时间量,优选持续一周,更优选持续2周,甚至更优选持续至少一个月。尽管预期根据本发明的免疫细胞在这些时间段内持续存在,但意图它们在患者体内的寿命不超过1年,优选为6个月,更优选为2个月,甚至更优选为1个月。
根据本发明的免疫细胞或组合物的施用可以任何方便的方式进行,包括通过雾化吸入、注射、摄取、输血、植入或移植。本文的免疫细胞或组合物可以通过静脉内或淋巴管内注射或腹膜内皮下、皮内、瘤内、结内,髓内、肌内施用给患者。
根据某些实施方式,通过静脉内注射施用免疫细胞或组合物。
根据其他某些实施方式,肠胃外施用一种或多种免疫细胞或者组合物。
根据某些其他实施方式,瘤内施用一种或多种免疫细胞或者组合物。所述施用可以通过直接注射到肿瘤中或与其相邻来完成。
细胞或细胞群的施用可以包括施用104-109个细胞/千克体重,优选为105至106个细胞/千克体重,包括在那些范围内的所有整数值的细胞数。可以一种或多种剂量施用细胞或细胞群。在另一个实施方式中,以单一剂量形式给予所述有效量的细胞。在另一个实施方式中,在一段时间内以多于一种剂量施用所述有效量的细胞。施用时机在管理医师的判断之内,并且取决于患者的临床状况。可以从任何来源获得细胞或细胞群,例如血库或供者。虽然个体需要变化,但确定用于特定疾病或病况的给定细胞类型的有效量的最佳范围在本领域的技术范围内。有效量是指提供治疗或预防益处的量。施用的剂量取决于受体的年龄、健康和体重,同期治疗(如有的话)的种类、治疗频率和所需效果的性质。
根据某些实施方式,可结合任何数量的相关治疗方式(例如,之前、同时或之后)将免疫细胞施用给患者,其包括但不限于用诸如抗病毒治疗、西多福韦和白介素-2、阿糖胞苷(还称为ara-c)的药剂治疗或针对ms患者的那他珠单抗(nataliziimab)治疗或针对银屑病患者的依法利珠单抗(efaliztimab)治疗或针对pml患者的其他治疗。在另外的实施方式中,可联合化学疗法、辐射、免疫抑制剂如环孢菌素、硫唑嘌呤、甲氨蝶呤、霉酚酸酯和fk506抗体或其他免疫烧蚀剂如campath、抗cd3抗体或其他抗体疗法,细胞毒素、氟达拉滨、环孢菌素、fk506、雷帕霉素、霉酚酸、类固醇、fr901228、细胞因子和辐照使用本发明的t细胞。这些药物抑制钙依赖性磷酸酶钙调磷酸酶(环孢菌素和fk506)或抑制对生长因子诱导的信号传导(雷帕霉素)重要的p70s6激酶(liu等人,cell66:807-815,11;henderson等人,immun.73:316-321,1991;bierer等人,citrr.opin.mmn.5:763-773,93)。在另一个实施方式中,联合骨髓移植(例如,之前、同时或之后)、使用化疗剂如氟达拉滨、体外放射线治疗(xrt)、环磷酰胺或抗体如okt3或阿仑单抗(campath)的t细胞消除治疗将本发明的细胞组合物施用于患者。在另一个实施方式中,在b细胞消除治疗后施用本发明的细胞组合物,例如与cd20反应的药剂如利妥昔单抗。例如,在一个实施方式中,受试者可以通过高剂量化疗,随后进行外周血干细胞移植进行标准治疗。在某些实施方式中,在移植后,受试者接受本发明的扩增的基因工程化免疫细胞的输注。在另外的实施方式中,在手术之前或之后施用扩增的细胞。
还包括在本发明的这个方面中的是治疗有需要的患者的方法,其包括a)提供至少一种本发明的免疫细胞,优选为免疫细胞的群体;和b)将免疫细胞或群体施用给患者。
还包括在本发明的这个方面中的是使用至少一种本发明的免疫细胞,优选为免疫细胞的群体来制备药物的方法。因此,本发明提供了至少一种本发明的免疫细胞、优选为免疫细胞的群体在制备药物中的用途。优选地,这样的药物用于治疗上述指出的疾病。
特别设想的是本发明的免疫细胞与能够结合第一多聚化配体结合结构域和第二多聚化配体结合结构域的多价配体组合使用(或用于使用)。在这方面,本发明考虑将有效量的第一多聚化配体结合结构域和第二多聚化配体结合结构域的多价配体施用给患者。
例如,多价配体如雷帕霉素可以约0.01至10mg/kg体重的剂量施用给患者。根据某些实施方式,以约0.01至5mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.01至4mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.01至3mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.01至2mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.01至1mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.05至5mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.05至4mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.05至3mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.05至2mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.05至1mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.1至5mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.1至4mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.1至3mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.1至2mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.1至1mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.5至5mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.5至4mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.5至3mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.5至2mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约0.5至1mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约1至5mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以约2至5mg/kg体重的剂量施用多价配体。根据某些其他实施方式,以一定剂量或约2.5至5mg/kg体重的剂量施用多价配体。
可以任何方便的方式进行本发明的多价配体的施用,方式包括通过雾化吸入、注射、摄取、输血、植入或移植。多价配体可以通过皮下、皮内、瘤内、结内,髓内、肌内、通过静脉内或淋巴管内注射或腹膜内施用给患者。
根据某些实施方式,通过静脉内注射施用多价配体。
根据某些其他实施方式,瘤内施用多价配体,可选地与根据本发明的免疫细胞或免疫细胞群体一起。这样的方法防止或限制待治疗的肿瘤(例如,实体瘤)外的含car的免疫细胞的激活(接通)。
其他定义
-“胞外域”是指延伸到细胞外空间(细胞外部的空间)的本发明的嵌合抗原受体的一部分。
-“胞内域”是指延伸到细胞的细胞质中的本发明的嵌合抗原受体的一部分。
-多肽序列中的氨基酸残基在本文根据单字母密码指定,其中例如,q是指gln或谷氨酰胺残基,r是指arg或精氨酸残基,并且d是指asp或天冬氨酸残基。
-“替换”或“替换的”是指通过用另一个代替一个氨基酸残基的多肽修饰,例如用多肽序列中的谷氨酰胺残基代替精氨酸残基是氨基酸替换。
-“保守替换”是指用具有相似侧链的不同残基替换氨基酸残基,因此通常涉及用相同或相似类别的氨基酸内的氨基酸替换多肽中的氨基酸。作为举例而非限制,具有脂肪族侧链的氨基酸可被另一脂肪族氨基酸例如丙氨酸、缬氨酸、亮氨酸和异亮氨酸替换;具有羟基侧链的氨基酸被具有羟基侧链的另一氨基酸例如丝氨酸和苏氨酸替换;具有芳香族侧链的氨基酸被具有芳香族侧链的另一氨基酸例如苯丙氨酸、酪氨酸、色氨酸和组氨酸替换;具有碱性侧链的氨基酸被具有碱性侧链的另一氨基酸例如赖氨酸和精氨酸替换;具有酸性侧链的氨基酸被具有酸性侧链的另一氨基酸例如天冬氨酸或谷氨酸替换;并且疏水或亲水氨基酸分别被另一疏水或亲水氨基酸替换。
-“非保守替换”是指用具有显著不同侧链性质的氨基酸替换多肽中的氨基酸。非保守替换可以在定义的基团之间而不是在定义的基团之内使用氨基酸,并且影响(a)替换区域中的肽骨架的结构(例如,针对甘氨酸的脯氨酸)(b)电荷或疏水性,或(c)侧链的大小。作为实例而非限制,示例性的非保守替换可以是用碱性或脂肪族氨基酸替换酸性氨基酸;用小氨基酸替换芳香族氨基酸;和用疏水性氨基酸替换亲水性氨基酸。
-“缺失(deletion)”或“缺失的(deleted)”是指通过去除参照多肽中的一个或多个氨基酸来修饰多肽。缺失可以包括去除构成多肽的1个或多个氨基酸、2个或多个氨基酸、5个或多个氨基酸、10个或多个氨基酸、15个或多个氨基酸或者20个或多个氨基酸、最高达总数的10%的氨基酸,或最高达总数的20%的氨基酸,同时保持多肽功能。缺失可以针对多肽的内部部分和/或末端部分。在各种实施方式中,缺失可以包含连续片段或可以是不连续的。
-“插入(insertion)”或“插入的(inserted)”是指通过向参照多肽添加一个或多个氨基酸来修饰多肽。插入可以包括添加1个或多个氨基酸、2个或多个氨基酸、5个或多个氨基酸、10个或多个氨基酸、15个或多个氨基酸或者20个或多个氨基酸。插入可以位于多肽的内部,或插入到羧基或氨基末端。插入可以是连续的氨基酸片段,或者被参考多肽中的一个或多个氨基酸分开。
-核苷酸指定如下:单字母密码用于指定核苷的碱基:a是腺嘌呤、t是胸腺嘧啶、c是胞嘧啶,并且g是鸟嘌呤。对于简并的核苷酸,r表示g或a(嘌呤核苷酸),k表示g或t,s表示g或c,w表示a或t,m表示a或c,y表示t或c(嘧啶核苷酸),d表示g、a或t,v表示g、a或c,b表示g、t或c,h表示a、t或c,并且n表示g、a、t或c。
-如在本文中所使用的,“核酸”或“多核苷酸”是指核苷酸和/或多核苷酸,例如脱氧核糖核酸(dna)或核糖核酸(rna)、寡核苷酸、通过聚合酶链反应(pcr)产生的片段,以及由连接、裂解、核酸内切酶作用和外切核酸酶作用中的任一种产生的片段。核酸分子可由天然存在的核苷酸的单体(例如,dna和rna)或天然存在的核苷酸的类似物(例如,天然存在的核苷酸的对映体形式)或两者的组合组成。修饰的核苷酸可以在糖部分和/或嘧啶或嘌呤碱基部分中具有改变。糖修饰包括例如,用卤素、烷基、胺和叠氮基取代一个或多个羟基,或糖可被官能化为醚或酯。另外地,整个糖部分可用空间和电子相似的结构代替,例如氮杂-糖和碳环糖类似物。碱基部分中的修饰的实例包括烷基化嘌呤和嘧啶、酰化嘌呤或嘧啶或其他众所周知的杂环取代基。核酸单体可以通过磷酸二酯键或这种键的类似物连接。核酸可以是单链或双链的。
-“递送载体(deliveryvector)”或“多种递送载体(deliveryvectors)”是指可用于本发明中以使细胞接触(即“接触”)或将本发明所需的药剂/化学物质和分子(蛋白质或核酸)递送到细胞内或亚细胞区室(即“引入”)的任何递送载体。它包括但不限于脂质体递送载体、病毒递送载体、药物递送载体、化学载体、聚合物载体、脂质复合物、多聚复合物、树状聚合物、微泡(超声造影剂)、纳米颗粒、乳剂或其他合适的转移载体。这些递送载体允许递送分子、化学物质、大分子(基因、蛋白质)或其他载体如质粒或穿膜肽。在这些后面的情况下,递送载体是分子载体。
-术语“载体(vector)”或“多种载体(vectors)”是指能够转运与其连接的另一个核酸的核酸分子。本发明中的“载体”包括但不限于可由染色体、非染色体、半合成或合成的核酸组成的病毒载体、质粒、rna载体或线性或环状dna或rna分子。优选的载体是能够自主复制(附加型载体)和/或表达与它们连接的核酸(表达载体)的那些。大量合适的载体是本领域技术人员已知的并且可商购。
病毒载体包括逆转录病毒、腺病毒、细小病毒(例如,腺相关病毒)、冠状病毒、负链rna病毒如正粘病毒(例如,流感病毒)、弹状病毒(例如,狂犬病和水泡性口炎病毒)、副粘病毒(例如,麻疹和仙台病毒)、正链rna病毒如小核糖核酸病毒和甲病毒,以及双链dna病毒,包括腺病毒、疱疹病毒(例如,1型和2型单纯疱疹病毒、艾普斯登-巴尔病毒、巨细胞病毒)和痘病毒(例如,牛痘、禽痘和金丝雀痘)。其他病毒包括例如,诺沃克病毒、披膜病毒、黄病毒、呼肠孤病毒、乳多空病毒、嗜肝dna病毒和肝炎病毒。逆转录病毒的实例包括:禽白血病肉瘤、哺乳动物c型、b型病毒、d型病毒、htlv-blv组、慢病毒、泡沫病毒(coffin,j.m.,逆转录病毒:病毒及其复制(retroviridae:thevirusesandtheirreplication),infundamentalvirology,第三版,b.n.fields等人编辑,lippincott-ravenpublishers,philadelphia,1996)。
-“慢病毒载体”是指基于hiv的慢病毒载体,因为它们具有相对较大的包装能力,降低的免疫原性及其以高效率稳定地转导大范围不同细胞类型的能力,因此其对于基因递送是非常有前景的。通常在将三种(包装、包膜和转移)或更多质粒瞬时转染入生产细胞后产生慢病毒载体。与hiv一样,慢病毒载体通过病毒表面糖蛋白与细胞表面上的受体的相互作用进入靶细胞。进入时,病毒rna经历逆转录,其由病毒逆转录酶复合物介导。逆转录产物是双链线性病毒dna,其是用于感染细胞的dna中的病毒整合的底物。“整合慢病毒载体(或lv)”意指能够整合靶细胞基因组的作为非限制性实例的这种载体。相反,“非整合慢病毒载体(或nilv)”意指不能通过病毒整合酶的作用整合靶细胞的基因组的有效的基因递送载体。
-递送载体和载体可与任何细胞透化技术(例如,声致穿孔或电穿孔或这些技术的衍生物)相关联或组合。
-“细胞(cell)”或“多个细胞(cells)”是指衍生自这些生物体的任何真核活细胞、原代细胞和细胞系用于体外培养。
-“原代细胞(primarycell)”或“多个原代细胞(primarycells)”是指从活组织(即,活检材料)直接取出并建立用于体外生长的细胞,其经历非常少的群体倍增值,因此与连续致瘤或人工永生化细胞系相比,从其衍生它们的组织的主要功能成分和特征更具代表性。
作为非限制性实例,细胞系可选自cho-k1细胞;hek293细胞;caco2细胞;u2-os细胞;nih3t3细胞;nso细胞;sp2细胞;cho-s细胞;dg44细胞;k-562细胞、u-937细胞;mrc5细胞;imr90细胞;jurkat细胞;hepg2细胞;hela细胞;ht-1080细胞;hct-116细胞;hu-h7细胞;huvec细胞;molt4细胞。
所有这些细胞系可以通过本发明的方法进行修饰以提供细胞系模型从而产生、表达、定量、检测、研究感兴趣的基因或蛋白质;作为非限制性实例,这些模型也可用于筛选研究和生产以及诸如化学、生物燃料、治疗和农学的各个领域中的感兴趣的生物活性分子。
-“干细胞”意指具有自我更新能力和产生分化细胞能力的细胞。更明确地,干细胞是能够产生与它们的母细胞相同的子细胞(自我更新)并且能够产生更受限的潜能的后代(分化细胞)的细胞。
-“nk细胞”意指自然杀伤细胞。nk细胞定义为大的颗粒状淋巴细胞,并且构成从产生b淋巴细胞和t淋巴细胞的普通淋巴样祖细胞中分化出来的第三种细胞。
-“突变”是指替换、删除、插入最高达一个、二个、三个、四个、五个、七个、八个、九个、十个、十一个、十二个、十三个、十四个、十五个、二十个、二十五个、三十个、四十个、五十个或更多的多核苷酸(cdna、基因)或多肽序列中的核苷酸/氨基酸。突变可影响基因或其调控序列的编码序列。它也可能影响基因组序列的结构或编码的mrna的结构/稳定性。
-“一个或多个变体”是指通过在母体分子的氨基酸序列中突变或替换至少一个残基获得的多肽变体。
-“基因”是指遗传的基本单位,由编码特定蛋白质或蛋白质片段的沿染色体以线性方式排列的dna片段组成。基因通常包括启动子、5’非翻译区、一个或多个编码序列(外显子)、可选的内含子、3’非翻译区。基因还可包含终止子、增强子和/或沉默子。
-如在本文中所使用的,术语“基因座”是染色体上的dna序列(例如,基因)的具体物理位置。术语“基因座”可以是指染色体上的稀切核酸内切酶靶序列的具体物理位置。这种基因座可包含被根据本发明的稀切核酸内切酶识别和/或切割的靶序列。应当理解,本发明的感兴趣的基因座不仅可限定存在于细胞的遗传物质(即,染色体)的主体中的核酸序列,而且可限定可独立于遗传物质的主体存在的遗传物质的一部分如质粒、游离基因、病毒、转座子或细胞器如线粒体的主体作为非限制性实例。
-“融合蛋白”是指本领域公知的方法的结果,该方法包括两个或多个最初编码单独的蛋白质或它们的一部分的基因的连接,“融合基因”的翻译导致衍生自每种原始蛋白质的功能性质的单一多肽。
-“同一性”,“序列同一性百分比”,“百分比序列同一性”和“百分比同一性”在本文中用于指氨基酸序列与参考氨基酸序列之间的比较。如在本文中所使用的,“百分比序列同一性”由两个氨基酸序列计算如下:使用geneticcomputinggroup的gap(全局比对程序)的版本9、使用默认的blosum62矩阵对序列进行比对,其中,空位开放罚分为-12(对于空位的第一个空值)且空位延伸罚分为-4(对于空位的每个另外的空值)。比对后,百分比同一性通过将匹配数目表示为参考氨基酸序列中的氨基酸数目的百分比来计算。例如,考虑了与本文的特异性多肽具有至少80%、85%、90%、95%、98%或99%的同一性并且优选显示基本上相同功能的多肽,以及编码这种多肽的多核苷酸。
-“参考序列”或“参考氨基酸序列”是指与另一序列进行比较的定义的序列。
如在本文中所使用的,术语“受试者”或“患者”包括动物界的所有成员,包括非人类灵长类和人类。
本发明的上述书面描述提供了制造和使用它的方式和方法,使得本领域的技术人员能够制造和使用它们,特别是为了所附权利要求书的主题而提供该可实施性,其构成原始描述的一部分。
在本文列出数字限制或范围的情况下,包括端点。另外地,数值限制或范围内的所有值和子范围都被具体地包括,如同明确地写出一样。
一般地已描述了本发明,可以通过参考某些具体实施例获得进一步的理解,在本文提供这些具体实施例仅为了说明的目的,除非另有说明,否则不旨在限制。
实施例
实施例1:开发小分子(雷帕霉素)接通mccar19和mccar123-mrna递送-表面检测
构建体和mrna制备
所有构建体来源于pcls24707(seqidno:38),其编码多链car(mccar)的α链(seqidno:39)、β链(seqidno:40)和γ链(seqidno:41)。从头合成(genecust)编码frb结构域(seqidno:2)和fkbp结构域(seqidno:1)的序列。通过pcr进一步扩增scfv、铰链-跨膜-胞质内α链结构域、frb和fkbp以产生金门(goldengate)组装相容片段(seqidno:42至45)。另外地,使用四-eaaar-接头(seqidno:19)或–gs-4x-eaaar-接头(seqidno:20)和标准的分子生物学程序生成frb-fkbp和fkbp-frb片段(seqidno:46至47)。然后,使用一轮限制酶切(restriction)和连接将片段组装成pcls26563、pcls26564、pcls26881和pcls27123(seqidno:48至51)。由这些构建体编码的相应的氨基酸序列显示在seqidno:52至55中。
在mrna合成之前,通过使用寡核苷酸对α链-f/α链-r、β链-f/β链-r和γ链-f/γ链-r(seqidno:56至61)的pcr扩增所有单个链。根据制造商的说明,从pcr产物体外转录编码不同α链、β链、γ链的mrna并使用mmessagemmachinet7ultra试剂盒(lifetechnologies)进行聚腺苷酸化。用rneasy柱(qiagen)纯化rna,在cytoporation培养基t中洗脱,并通过使用nanodropnd-1000分光光度计在260nm下测量吸光度来定量。在变性甲醛/mops琼脂糖凝胶上验证rna的质量。
转染
激活后3至6天,使用agilepulsemax系统(harvardapparatus)通过电转移信使rna来转染t淋巴细胞。去除激活珠后,将细胞制粒,以>28x106个细胞/ml重新悬浮于cytoporation培养基t中。将5x106个细胞与6.9μg的总rna(2.5μg的α链,1.9μg的β链和2.5μg的γ链)或与8.4μg的总rna(4μg的修饰α链、1.9μg的β链和2.5μg的γ链)混合到0.4cm的比色皿中。电穿孔由1200v下的两个0.1ms脉冲和130v下的四个0.2ms脉冲组成。电穿孔后,将细胞稀释到2ml培养基中并在37℃/5%co2下孵育。mrna电转移后2小时,加入媒介物(dmso)或雷帕霉素(100nm)时间为19小时。
流式细胞术
在4℃下,在pbsfbs2%、edta2mm、叠氮化物0.1%中使用抗fab’2-生物素(山羊抗小鼠igg、fab’2片段特异性,115-066-072,jacksonimmunoresearch)进行用于α链检测的第一次标记,时间为20分钟,然后是用pbsfbs2%、edta2mm、叠氮化物0.1%的两个洗涤步骤。在4℃下,在pbsfbs2%、edta2mm、叠氮化物0.1%中使用链霉亲和素蛋白-apc进行第二次标记,时间为20分钟,然后在pbsfbs2%edta2mm叠氮化物0.1%中的洗涤步骤,并在pbs中的洗涤步骤。在4℃下,在pbs中使用efluor450(ebioscience65-0863-14)监测细胞活力,时间为20分钟,然后是用pbsfbs2%、edta2mm、叠氮化物0.1%的洗涤步骤并固定在2%的pfa中。使用macsquant(miltenyibiotec)进行流式细胞术,并使用flowjo软件进行数据分析。
所获得的数据清楚地表明,当frb-fkbp或fkbp-frb结构域被引入α链时,在雷帕霉素的存在下表面暴露改善(图3a和b)。
实施例2:开发小分子(ap21967)接通mccar19-mrna递送-表面检测
构建体、mrna制备和流式细胞术
为了设计整合系统以在打开/关闭状态之间转换scfv/抗原相互作用,将frb、fkbp12或frb和fkbp12的融合物插入cd8a铰链和scfv结构域之间(图1b)。作为起始实验,转染具有编码多链car(mccar)的每条链的mrna的原代t细胞。在添加雷帕霉素后,通过追踪靶向cd19的scfv的fab’2结构域(100nm,20h)监测在胞外铰链结构域的检测方面的变化。在没有小分子(雷帕霉素)的情况下,发现只有野生型mccar和fkbp-mccar具有高于90%的具有总体高的mfi的阳性细胞才能实现高水平的表面检测,如图3a所示。茎部(stalk)区域中的fkbp和frb二者的存在实际上废除了cd19scfv的表面检测,与它们的相互位置无关(低于40%的阳性细胞,与mccar相比mfi降低高达40倍)。有趣地,尽管雷帕霉素的添加几乎不影响mccar、frb-mccar和fkb-mccar构建体,但当考虑到阳性细胞或mfi的百分比(图3b)时,其强烈提高fkbp/frb-mccar和frb/fkbp-mccar构建体的表面检测(当考虑mfi时为15倍,并且当考虑阳性细胞的百分比时为3倍),使系统从关闭状态变为开启状态。加入雷帕霉素后检测的这种变化可能由不同的因素引起,包括含有接通组分的car链的稳定化。然而,必须注意的是,总是需要小分子以有效地开启在t细胞表面处的car的fkbp/frb-car的检测。
还测试了合成的非免疫抑制性ap21967雷帕霉素合成类似物,其结合fkbp12但不促进与mtor的frb结构域的结合。因此,在frb结构域中引入t2098l突变(称为frb*)以允许形成fkbp/ap21967/frb*复合物。
使用可商购的试剂盒(agilent)和标准分子生物学程序将frb结构域中的t2098l突变引入fkbp-frb结构域中,产生fkbp-frb*结构域(seqidno:62)。含有scfv、fkbp-frb*结构域、铰链-跨膜-胞质内结构域的α链的组装产生pcls27039(seqidno:63)。由该构建体编码的氨基酸序列显示在seqidno:64中。frb*是指具有t2098l突变的frb的变体(seqidno:4)。
如实施例1中所描述的使用ap21967而不是雷帕霉素进行mrna制备、转染和表面呈递的流式细胞术测量。
获得的数据清楚地表明,当fkbp-frb*结构域结合到α链中时,在ap21697存在下表面暴露改善(图4)。
为了评估接通系统的ap21967可用剂量范围,进行剂量反应测定(图8a)。获得的结果表明在100nm下的最大信号诱导和约10nm(8.2-10.1nm)的ec50值,其与转染的工程化的car的量无关。为了验证接通方法的可移植性,还工程化靶向cd123的car。如通过10nm的相似ec50值(7.3-8.7nm,图8b)所证实的,发现scfv的性质不影响接通性质。值得注意的是,ec50与在患者的外周血或肿瘤组织中报道的雷帕霉素浓度范围内,表明接通系统可能对临床相关浓度敏感。
实施例3:开发小分子(ap21967)接通mccar-mrna递送-诱导的细胞毒性
使用基于流式细胞术的细胞毒性测定来评估被赋予来自实施例2的fkbp-frb*mccarcd19的工程化t细胞的细胞溶解活性。在此测定中,用celltracetmcfse标记呈递car靶抗原(daudicd19阳性)的靶细胞并且用celltracetm紫罗兰标记对照细胞。在媒介物(乙醇)或ap21967(100nm)的存在下,在37℃下将混合的靶细胞群体(1:1比例)与各种比例的工程化的效应物cart细胞(效应物/靶标比例为20:1)在最终体积100ul的x-vivo-15培养基中共同孵育固定的时间段(5h)。
在用2%pfa固定之前,回收全部细胞群体,在pbs中洗涤并用efluor780活力标志物标记。通过流式细胞术分析固定的细胞以确定它们的活力。如实施例1中所描述的进行流式细胞术和数据分析。
获得的数据清楚地表明在ap21697存在下改善的接通细胞溶解活性(图5a)。
还对ap21967进行了剂量反应(0、1、5、10、33、100nm),并测量了所得到的工程化的cart细胞的细胞溶解能力。正如预期的,它导致了靶细胞杀伤水平与ap21967的变化相关(图5b)。计算的ec50为约10nm(12.7nm),在使用表面检测确定的范围内。靶向细胞杀伤水平也与car检测水平相关(图9)。
总而言之,此处呈现的结果提供了工程化car分子的铰链结构域的原理的证明,以创建用于逻辑门控策略的整合接通系统。
实施例4:开发小分子(ap21967)接通mccar-mrna递送-其他小分子竞争调整
如实施例2中所描述的进行mrna制备、转染和表面呈递的流式细胞术测量,同时使用10nm的ap21967和增加量的他克莫司孵育转染的t细胞。
条件:ap21967:10nm,他克莫司:0nm、10nm、30nm、100nm或500nm。在37℃/5%的co2下将细胞孵育20小时。
获得的数据清楚地表明,当将fkbp-frb*结构域引入α链中后,由于小分子ap21967与第二小分子(他克莫司)调节工程化的car的表面呈递的可能性(图6)。
实施例5:开发小分子(ap21967)接通sccar-mrna递送-表面检测
使用实施例2中出现的car胞外结构域(α链)作为模板来制备编码单链car(sccar)的质粒dna。cd8α跨膜结构域(seqidno:32)、cd3-t细胞受体的ζ链的胞质内信号转导区域(seqidno:28)和来自共刺激4-1bb(cd137)(seqidno:31)的信号转导结构域用于完成car。根据标准分子生物学程序,使用限制和连接的循环通过金门(goldengate)克隆装配sccar,产生pcls27572(fkbp-frb*)(seqidno:65)、pcls27603(fkbp)(seqidno:66)、pcls27604(frb*)(seqidno:67)。由这些构建体编码的相应的氨基酸序列显示在seqidno:68至70中。
如实施例1中所描述的使用ap21967而不是雷帕霉素进行mrna制备(使用分别位于car中和质粒上的寡核苷酸对sccar-f(seqidno:71)和sccar-r(seqidno:72))和转染以及表面呈递的流式细胞术测量。在4℃下,在pbsfbs2%、edta2mm、叠氮化物0.1%中用fc标记的重组cd123(lakepharma)进行用于sccars的检测的初级标记,时间为20分钟,然后是用pbsfbs2%、edta2mm、叠氮化物0.1%的两次洗涤步骤。在4℃下,在pbsfbs2%、edta2mm、叠氮化物0.1%中使用pe标记的山羊抗小鼠igg(子类1+2a+2b+3)fcγ片段特异性(jacksonimmunoresearch)进行第二次标记,时间为20分钟,然后是在pbsfbs2%、edta2mm、叠氮化物0.1%中的洗涤步骤和在pbs中的洗涤步骤。在细胞外标记之后,在4℃下,在pbs中使用efluor450或efluor780(ebioscience)监测细胞活力,时间为20分钟,然后是用pbsfbs2%edta2mm叠氮化物0.1%的洗涤步骤并固定在pfa2%中。
获得的数据清楚地表明,当fkbp-frb*结构域被引到α链中时,在ap21697存在下表面暴露改善(图7)。
实施例6:开发与mtor基因组编辑结合的小分子(雷帕霉素)接通mccar
雷帕霉素通过与胞质蛋白fk结合蛋白12(fkbp12)相互作用直接抑制t细胞,然后通过fkbp12/雷帕霉素复合物抑制mtor。
设计和构建/产生设计核酸酶(designernucleases),其产生靶向编码丝氨酸/苏氨酸蛋白激酶mtor的氨基酸(丝氨酸)2035的三联体周围的序列(或三联体本身)的单链或双链断裂。为了进行2035位的基因修正/突变,设计了含有由内源序列的两个同源臂包围的一个或多个期望突变碱基的供体dna。添加另外的沉默突变以防止供体dna的切割或通过设计核酸酶的修正/突变的基因组dna。
用编码设计核酸酶和供体dna的遗传物质转染或转导t细胞。然后,选择或分离在内源基因座处含有期望的突变的t细胞。记录在雷帕霉素存在下改进的工程t细胞的扩增性能,并与非工程化的t细胞比较。
然后,在新工程化的t细胞中实施在实施例1中呈现的接通car,并且记录在雷帕霉素存在下的car的改善的表面呈现和细胞溶解性质。
实施例7:开发与fkbp12基因组编辑(敲除)结合的小分子(雷帕霉素)接通mccar
雷帕霉素通过与胞质蛋白fk结合蛋白12(fkbp12)相互作用直接抑制t细胞,然后通过fkbp12/雷帕霉素复合物抑制mtor。
设计和构建/产生设计核酸酶,其产生靶向fkbp12基因的序列的双链断裂。用编码设计核酸酶的遗传物质转染或转导t细胞。然后,选择或分离在内源基因座处含有期望的敲除的t细胞。记录在雷帕霉素存在下改进的工程t细胞的扩增性能,并与非工程化的t细胞比较。
然后,在新工程化的t细胞中实施在实施例1中呈现的接通car,并且记录在雷帕霉素存在下的car的改善的表面呈现和细胞溶解性质。
实施例8:人类修饰的诱导型cd123car+t细胞在静脉注射molm13-luc肿瘤细胞的nog小鼠中的抗肿瘤活性研究
该研究的目的是证明由cellectis基因修饰的人类t细胞在体内的抗肿瘤活性,以表达针对人cd123抗原的诱导型嵌合抗原受体(car)。该诱导系统与非免疫抑制性雷帕霉素合成类似物(ap21967,由ariad开发,#635055)一起使用。
在静脉内注射molm13-luc肿瘤细胞的nog小鼠中评估在实施例1中描述的表达接通mccar-cd123的人类t细胞的抗肿瘤活性。如图10(3mg/kg/inj)所示,将雷帕霉素合成类似物ap21967重复注射到小鼠的腹腔(腹膜内,ip)中。
为了建立molm13-luc细胞系,用编码gfp和萤火虫萤光素酶(amsbiolvp438-pbs)的慢病毒转导molm13细胞(dsmzacc554)。用新霉素(参考10131-027,gibco,lifetechnologies,saint-aubin,france)选择gfp阳性细胞。在潮湿的气氛(5%的co2,95%的空气)中在37℃下将molm13-luc细胞悬浮生长到补充有15%胎牛血清(参考:3302,lonza,verviers,belgium)、100u/ml的青霉素和100μg/ml的链霉素(参考:de17-602e,lonza)的含有2.05mml-谷氨酰胺(参考:be12-702f,lonza)的培养基rpmi1640中。在血细胞计数器中将细胞计数,并通过0.25%的台盼蓝拒染实验评估它们的存活力,并在新鲜培养基中每周传代(pass)两次(0.8百万/ml)。
在注射到小鼠中的当天,将用接通mccar-cd123car转化的冷冻人类t细胞评估为在每毫升1-2.106个活细胞的范围内。
从taconic(ry,danemark)获得18只(18)的7-8周龄的健康雌性nog(nod.cg-prkdcscidii2rgtm1sug/jictac)小鼠,并根据实验室动物的护理和使用的nrc指南繁殖。
在d-7进行molm13-luc细胞(0.25x106个细胞/小鼠)的静脉注射。
细胞注射和治疗计划如下:
在d0,第1组(未转导的t细胞/媒介物)接受单次iv注射xno转导的t细胞(200μl,在rpmi1640中),然后连续10天每天2次ip注射媒介物(2q1dx10)。
在d0,第2组(未转导的t细胞/ap21967)接受单次iv注射xno转导的t细胞(200μl,在rpmi1640中),然后连续10天每天2次ip注射3mg/kg/inj的ap21967(2q1dx10)。
在d0,第3组(cart细胞/媒介物)接受单次iv注射包含xcar阳性t细胞的x修饰的cart细胞(200μl,在rpmi1640中),然后连续10天每天2次ip注射媒介物(2q1dx10)。
在d0,第4组(cart细胞/ap21967)接受单次iv注射包含xcar阳性t细胞的x修饰的cart细胞(200μl,在rpmi1640中),然后连续10天每天2次ip注射ap21967(2q1dx10)。
t细胞注射发生在d0、d2、d4、d7、d10、d15和d21,而小鼠每天注射两次雷帕霉素类似物。
每天监测小鼠的体重测量、临床和死亡率记录,并在vivo
参考文献
cruz,c.r.etal.(2013),″infusionofdonor-derivedcd19-redirectedvirus-specifictcellsforb-cellmalignanciesrelapsedafterallogeneicstemcelltransplant:aphase1study″,blood,122(17):2965-73
erhart,d.etal.(2013),″chemicaldevelopmentofintracellularproteinheterodimerizers″,chemistry&biology,20:549-557.
gaultier,a.etal.(2009),″selectivecross-linkingofinteractingproteinsusingself-labelingtags″,jamchemsoc,.131(49):17954-62.
gallagher,s.s.etal.(2007)″anorthogonaldexamethasone-trimethoprimyeastthree-hybridsystem″,analbiochem.363(1):160-2.
jena,b.,g.dotti,etal.(2010).″redirectingt-cellspecificitybyintroducingatumor-specificchimericantigenreceptor.″blood,116(7):1035-44.
kopytek,s.j.(2000),″chemicallyinduceddimerizationofdihydrofolatereductasebyahomobifunctionaldimerofmethotrexate″,chemistry&biology,7:313-321.
lin,h.n.,etal.(2000),″dexamethasone-methotrexate:anefficientchemicalinducerofproteindimerizationinvivo″,journaloftheamericanchemicalsociety,122(17):4247-4248.park,t.s.,s.a.rosenberg,etal.(2011).″treatingcancerwithgeneticallyengineeredtcells.″trendsbiotechnol,29(11):550-7.
peipp,m.,d.saul,etal.(2004).″efficienteukaryoticexpressionoffluorescentscfvfusionproteinsdirectedagainstcdantigensforfacsapplications.″jimmunolmethods,285(2):265-80.
poirot,l.etal.(2015),″multiplexgenomeeditedt-cellmanufacturingplatformfor″off-the-shelf″adoptivet-cellimmunotherapies″,j.cancerres.2015jul16.[epubaheadofprint]
swarts,d.c.etal.(2014),”theevoiutionaryjourneyofargonauteproteins”.natstructmolbiol.,21(9):743-53
valton,j.etal.(2015),″amultidrug-resistantengineeredcartcellforallogeneiccombinationimmunotherapy”,molther.2015jun10.[epubaheadofprint]
wilson,k.p.etal.(1995),”comparativex-raystructuresofthemajorbindingproteinfortheimmunosuppressantfk506(tacrolimus)inunligandedformandincomplexwithfk506andrapamycin″,acta.crystallogr.dbiol.crystallogr.51:511-521
序列表
<110>塞勒克提斯公司(cellectis)
<120>具有整合可控功能的嵌合抗原受体
<130>p81502686pct00
<150>pa201570545
<151>2015-08-24
<150>ep15202592
<151>2015-12-23
<160>72
<170>patentinversion3.5
<210>1
<211>108
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>fkbp12
<400>1
metglyvalglnvalgluthrileserproglyaspglyargthrphe
151015
prolysargglyglnthrcysvalvalhistyrthrglymetleuglu
202530
aspglylyslyspheaspserserargaspargasnlysprophelys
354045
phemetleuglylysglngluvalileargglytrpglugluglyval
505560
alaglnmetservalglyglnargalalysleuthrileserproasp
65707580
tyralatyrglyalathrglyhisproglyileileproprohisala
859095
thrleuvalpheaspvalgluleuleulysleuglu
100105
<210>2
<211>94
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>frb
<400>2
argvalalaileleutrphisglumettrphisgluglyleugluglu
151015
alaserargleutyrpheglygluargasnvallysglymetpheglu
202530
valleugluproleuhisalametmetgluargglyproglnthrleu
354045
lysgluthrserpheasnglnalatyrglyargaspleumetgluala
505560
glnglutrpcysarglystyrmetlysserglyasnvallysaspleu
65707580
thrglnalatrpaspleutyrtyrhisvalpheargargile
8590
<210>3
<211>107
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>fkbp12(f36v)
<400>3
glyvalglnvalgluthrileserproglyaspglyargthrphepro
151015
lysargglyglnthrcysvalvalhistyrthrglymetleugluasp
202530
glylyslysvalaspserserargaspargasnlysprophelysphe
354045
metleuglylysglngluvalileargglytrpglugluglyvalala
505560
glnmetservalglyglnargalalysleuthrileserproasptyr
65707580
alatyrglyalathrglyhisproglyileileproprohisalathr
859095
leuvalpheaspvalgluleuleulysleuglu
100105
<210>4
<211>94
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>frbt2098l
<400>4
argvalalaileleutrphisglumettrphisgluglyleugluglu
151015
alaserargleutyrpheglygluargasnvallysglymetpheglu
202530
valleugluproleuhisalametmetgluargglyproglnthrleu
354045
lysgluthrserpheasnglnalatyrglyargaspleumetgluala
505560
glnglutrpcysarglystyrmetlysserglyasnvallysaspleu
65707580
leuglnalatrpaspleutyrtyrhisvalpheargargile
8590
<210>5
<211>344
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>钙调磷酸酶a(cna)
<400>5
gluvalpheaspasnaspglylysproargvalaspileleulysala
151015
hisleumetlysgluglyargleuglugluservalalaleuargile
202530
ilethrgluglyalaserileleuargglnglulysasnleuleuasp
354045
ileaspalaprovalthrvalcysglyaspilehisglyglnphephe
505560
aspleumetlysleuphegluvalglyglyserproalaasnthrarg
65707580
tyrleupheleuglyasptyrvalaspargglytyrpheserileglu
859095
cysvalleutyrleutrpalaleulysileleutyrprolysthrleu
100105110
pheleuleuargglyasnhisglucysarghisleuthrglutyrphe
115120125
thrphelysglnglucyslysilelystyrsergluargvaltyrasp
130135140
alacysmetaspalapheaspcysleuproleualaalaleumetasn
145150155160
glnglnpheleucysvalhisglyglyleuserprogluileasnthr
165170175
leuaspaspilearglysleuaspargphelysgluproproalatyr
180185190
glyprometcysaspileleutrpseraspproleugluaspphegly
195200205
asnglulysthrglngluhisphethrhisasnthrvalargglycys
210215220
sertyrphetyrsertyrproalavalcysglupheleuglnhisasn
225230235240
asnleuleuserileleuargalahisglualaglnaspalaglytyr
245250255
argmettyrarglysserglnthrthrglypheproserleuilethr
260265270
ilepheseralaproasntyrleuaspvaltyrasnasnlysalaala
275280285
valleulystyrgluasnasnvalmetasnileargglnpheasncys
290295300
serprohisprotyrtrpleuproasnphemetaspvalphethrtrp
305310315320
serleuprophevalglyglulysvalthrglumetleuvalasnval
325330335
leuasnilecysseraspaspglu
340
<210>6
<211>165
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>亲环素(cyp)
<400>6
metvalasnprothrvalphepheaspilealavalaspglyglupro
151015
leuglyargvalserphegluleuphealaasplysvalprolysthr
202530
alagluasnpheargalaleuserthrglyglulysglypheglytyr
354045
lysglysercysphehisargileileproglyphemetcysglngly
505560
glyaspphethrarghisasnglythrglyglylysseriletyrgly
65707580
glulysphegluaspgluasnpheileleulyshisthrglyprogly
859095
ileleusermetalaasnalaglyproasnthrasnglyserglnphe
100105110
pheilecysthralalysthrglutrpleuaspglylyshisvalval
115120125
pheglylysvallysgluglymetasnilevalglualametgluarg
130135140
pheglyserargasnglylysthrserlyslysilethrilealaasp
145150155160
cysglyglnleuglu
165
<210>7
<211>804
<212>prt
<213>大肠杆菌(escherichiacoli)
<220>
<221>chain
<223>gyrb
<400>7
metserasnsertyraspserserserilelysvalleulysglyleu
151015
aspalavalarglysargproglymettyrileglyaspthraspasp
202530
glythrglyleuhishismetvalphegluvalvalaspasnalaile
354045
aspglualaleualaglyhiscyslysgluileilevalthrilehis
505560
alaaspasnservalservalglnaspaspglyargglyileprothr
65707580
glyilehisproglugluglyvalseralaalagluvalilemetthr
859095
valleuhisalaglyglylyspheaspaspasnsertyrlysvalser
100105110
glyglyleuhisglyvalglyvalservalvalasnalaleusergln
115120125
lysleugluleuvalileglnarggluglylysilehisargglnile
130135140
tyrgluhisglyvalproglnalaproleualavalthrglygluthr
145150155160
glulysthrglythrmetvalargphetrpproserleugluthrphe
165170175
thrasnvalthrglupheglutyrgluileleualalysargleuarg
180185190
gluleuserpheleuasnserglyvalserileargleuargasplys
195200205
argaspglylysgluasphisphehistyrgluglyglyilelysala
210215220
phevalglutyrleuasnlysasnlysthrproilehisproasnile
225230235240
phetyrpheserthrglulysaspglyileglyvalgluvalalaleu
245250255
glntrpasnaspglypheglngluasniletyrcysphethrasnasn
260265270
ileproglnargaspglyglythrhisleualaglypheargalaala
275280285
metthrargthrleuasnalatyrmetasplysgluglytyrserlys
290295300
lysalalysvalseralathrglyaspaspalaarggluglyleuile
305310315320
alavalvalservallysvalproaspprolyspheserserglnthr
325330335
lysasplysleuvalsersergluvallysseralavalgluglngln
340345350
metasngluleuleualaglutyrleuleugluasnprothraspala
355360365
lysilevalvalglylysileileaspalaalaargalaarggluala
370375380
alaargargalaargglumetthrargarglysglyalaleuaspleu
385390395400
alaglyleuproglylysleualaaspcysglngluargaspproala
405410415
leusergluleutyrleuvalgluglyaspseralaglyglyserala
420425430
lysglnglyargasnarglysasnglnalaileleuproleulysgly
435440445
lysileleuasnvalglulysalaargpheasplysmetleuserser
450455460
glngluvalalathrleuilethralaleuglycysglyileglyarg
465470475480
aspglutyrasnproasplysleuargtyrhisserileileilemet
485490495
thraspalaaspvalaspglyserhisileargthrleuleuleuthr
500505510
phephetyrargglnmetprogluilevalgluargglyhisvaltyr
515520525
ilealaglnproproleutyrlysvallyslysglylysglnglugln
530535540
tyrilelysaspaspglualametaspglntyrglnileserileala
545550555560
leuaspglyalathrleuhisthrasnalaseralaproalaleuala
565570575
glyglualaleuglulysleuvalserglutyrasnalathrglnlys
580585590
metileasnargmetgluargargtyrprolysalametleulysglu
595600605
leuiletyrglnprothrleuthrglualaaspleuseraspglugln
610615620
thrvalthrargtrpvalasnalaleuvalsergluleuasnasplys
625630635640
gluglnhisglyserglntrplyspheaspvalhisthrasnalaglu
645650655
glnasnleuphegluproilevalargvalargthrhisglyvalasp
660665670
thrasptyrproleuasphisglupheilethrglyglyglutyrarg
675680685
argilecysthrleuglyglulysleuargglyleuleuglugluasp
690695700
alapheilegluargglygluargargglnprovalalaserpheglu
705710715720
glnalaleuasptrpleuvallysgluserargargglyleuserile
725730735
glnargtyrlysglyleuglyglumetasnprogluglnleutrpglu
740745750
thrthrmetaspprogluserargargmetleuargvalthrvallys
755760765
aspalailealaalaaspglnleuphethrthrleumetglyaspala
770775780
valgluproargargalapheileglugluasnalaleulysalaala
785790795800
asnileaspile
<210>8
<211>92
<212>prt
<213>拟南芥(arabidopsisthaliana)
<220>
<221>chain
<223>gai
<400>8
metlysargasphishishishishishisglnasplyslysthrmet
151015
metmetasnglugluaspaspglyasnglymetaspgluleuleuala
202530
valleuglytyrlysvalargserserglumetalaaspvalalagln
354045
lysleugluglnleugluvalmetmetserasnvalglngluaspasp
505560
leuserglnleualathrgluthrvalhistyrasnproalagluleu
65707580
tyrthrtrpleuaspsermetleuthraspleuasn
8590
<210>9
<211>345
<212>prt
<213>拟南芥(arabidopsisthaliana)
<220>
<221>chain
<223>gid1a
<400>9
metalaalaseraspgluvalasnleuilegluserargthrvalval
151015
proleuasnthrtrpvalleuileserasnphelysvalalatyrasn
202530
ileleuargargproaspglythrpheasnarghisleualaglutyr
354045
leuasparglysvalthralaasnalaasnprovalaspglyvalphe
505560
serpheaspvalleuileaspargargileasnleuleuserargval
65707580
tyrargproalatyralaaspglngluglnproproserileleuasp
859095
leuglulysprovalaspglyaspilevalprovalileleuphephe
100105110
hisglyglyserphealahisserseralaasnseralailetyrasp
115120125
thrleucysargargleuvalglyleucyslyscysvalvalvalser
130135140
valasntyrargargalaprogluasnprotyrprocysalatyrasp
145150155160
aspglytrpilealaleuasntrpvalasnserargsertrpleulys
165170175
serlyslysaspserlysvalhisilepheleualaglyaspserser
180185190
glyglyasnilealahisasnvalalaleuargalaglyglusergly
195200205
ileaspvalleuglyasnileleuleuasnprometpheglyglyasn
210215220
gluargthrgluserglulysserleuaspglylystyrphevalthr
225230235240
valargaspargasptrptyrtrplysalapheleuprogluglyglu
245250255
asparggluhisproalacysasnpropheserproargglylysser
260265270
leugluglyvalserpheprolysserleuvalvalvalalaglyleu
275280285
aspleuileargasptrpglnleualatyralagluglyleulyslys
290295300
alaglyglngluvallysleumethisleuglulysalathrvalgly
305310315320
phetyrleuleuproasnasnasnhisphehisasnvalmetaspglu
325330335
ileseralaphevalasnalaglucys
340345
<210>10
<211>358
<212>prt
<213>拟南芥(arabidopsisthaliana)
<220>
<221>chain
<223>gid1b
<400>10
metalaglyglyasngluvalasnleuasnglucyslysargileval
151015
proleuasnthrtrpvalleuileserasnphelysleualatyrlys
202530
valleuargargproaspglyserpheasnargaspleualagluphe
354045
leuasparglysvalproalaasnserpheproleuaspglyvalphe
505560
serpheasphisvalaspserthrthrasnleuleuthrargiletyr
65707580
glnproalaserleuleuhisglnthrarghisglythrleugluleu
859095
thrlysproleuserthrthrgluilevalprovalleuilephephe
100105110
hisglyglyserphethrhisserseralaasnseralailetyrasp
115120125
thrphecysargargleuvalthrilecysglyvalvalvalvalser
130135140
valasptyrargargserprogluhisargtyrprocysalatyrasp
145150155160
aspglytrpasnalaleuasntrpvallysserargvaltrpleugln
165170175
serglylysaspserasnvaltyrvaltyrleualaglyaspserser
180185190
glyglyasnilealahisasnvalalavalargalathrasnglugly
195200205
vallysvalleuglyasnileleuleuhisprometpheglyglygln
210215220
gluargthrglnserglulysthrleuaspglylystyrphevalthr
225230235240
ileglnaspargasptrptyrtrpargalatyrleuprogluglyglu
245250255
aspargasphisproalacysasnpropheglyproargglyglnser
260265270
leulysglyvalasnpheprolysserleuvalvalvalalaglyleu
275280285
aspleuvalglnasptrpglnleualatyrvalaspglyleulyslys
290295300
thrglyleugluvalasnleuleutyrleulysglnalathrilegly
305310315320
phetyrpheleuproasnasnasphisphehiscysleumetgluglu
325330335
leuasnlysphevalhisserilegluaspserglnserlysserser
340345350
provalleuleuthrpro
355
<210>11
<211>344
<212>prt
<213>拟南芥(arabidopsisthaliana)
<220>
<221>chain
<223>gid1c
<400>11
metalaglyserglugluvalasnleuilegluserlysthrvalval
151015
proleuasnthrtrpvalleuileserasnphelysleualatyrasn
202530
leuleuargargproaspglythrpheasnarghisleualagluphe
354045
leuasparglysvalproalaasnalaasnprovalasnglyvalphe
505560
serpheaspvalileileaspargglnthrasnleuleuserargval
65707580
tyrargproalaaspalaglythrserproserilethraspleugln
859095
asnprovalaspglygluilevalprovalilevalphephehisgly
100105110
glyserphealahisserseralaasnseralailetyraspthrleu
115120125
cysargargleuvalglyleucysglyalavalvalvalservalasn
130135140
tyrargargalaprogluasnargtyrprocysalatyraspaspgly
145150155160
trpalavalleulystrpvalasnsersersertrpleuargserlys
165170175
lysaspserlysvalargilepheleualaglyaspserserglygly
180185190
asnilevalhisasnvalalavalargalavalgluserargileasp
195200205
valleuglyasnileleuleuasnprometpheglyglythrgluarg
210215220
thrgluserglulysargleuaspglylystyrphevalthrvalarg
225230235240
aspargasptrptyrtrpargalapheleuprogluglygluasparg
245250255
gluhisproalacysserpropheglyproargserlysserleuglu
260265270
glyleuserpheprolysserleuvalvalvalalaglyleuaspleu
275280285
ileglnasptrpglnleulystyralagluglyleulyslysalagly
290295300
glngluvallysleuleutyrleugluglnalathrileglyphetyr
305310315320
leuleuproasnasnasnhisphehisthrvalmetaspgluileala
325330335
alaphevalasnalaglucysgln
340
<210>12
<211>182
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>snap标签
<400>12
metasplysaspcysglumetlysargthrthrleuaspserproleu
151015
glylysleugluleuserglycysgluglnglyleuhisargileile
202530
pheleuglylysglythrseralaalaaspalavalgluvalproala
354045
proalaalavalleuglyglyprogluproleumetglnalathrala
505560
trpleuasnalatyrphehisglnproglualaileglugluphepro
65707580
valproalaleuhishisprovalpheglnglngluserphethrarg
859095
glnvalleutrplysleuleulysvalvallyspheglygluvalile
100105110
sertyrserhisleualaalaleualaglyasnproalaalathrala
115120125
alavallysthralaleuserglyasnprovalproileleuilepro
130135140
cyshisargvalvalglnglyaspleuaspvalglyglytyrglugly
145150155160
glyleualavallysglutrpleuleualahisgluglyhisargleu
165170175
glylysproglyleugly
180
<210>13
<211>297
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>halo标签
<400>13
metalagluileglythrglyphepropheaspprohistyrvalglu
151015
valleuglygluargmethistyrvalaspvalglyproargaspgly
202530
thrprovalleupheleuhisglyasnprothrsersertyrvaltrp
354045
argasnileileprohisvalalaprothrhisargcysilealapro
505560
aspleuileglymetglylysserasplysproaspleuglytyrphe
65707580
pheaspasphisvalargphemetaspalapheileglualaleugly
859095
leuglugluvalvalleuvalilehisasptrpglyseralaleugly
100105110
phehistrpalalysargasnprogluargvallysglyilealaphe
115120125
metglupheileargproileprothrtrpaspglutrpprogluphe
130135140
alaarggluthrpheglnalapheargthrthraspvalglyarglys
145150155160
leuileileaspglnasnvalpheilegluglythrleuprometgly
165170175
valvalargproleuthrgluvalglumetasphistyrargglupro
180185190
pheleuasnprovalasparggluproleutrpargpheproasnglu
195200205
leuproilealaglygluproalaasnilevalalaleuvalgluglu
210215220
tyrmetasptrpleuhisglnserprovalprolysleuleuphetrp
225230235240
glythrproglyvalleuileproproalaglualaalaargleuala
245250255
lysserleuproasncyslysalavalaspileglyproglyleuasn
260265270
leuleuglngluaspasnproaspleuileglysergluilealaarg
275280285
trpleuserthrleugluilesergly
290295
<210>14
<211>182
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>clip标签
<400>14
metasplysaspcysglumetlysargthrthrleuaspserproleu
151015
glylysleugluleuserglycysgluglnglyleuhisargileile
202530
pheleuglylysglythrseralaalaaspalavalgluvalproala
354045
proalaalavalleuglyglyprogluproleuileglnalathrala
505560
trpleuasnalatyrphehisglnproglualaileglugluphepro
65707580
valproalaleuhishisprovalpheglnglngluserphethrarg
859095
glnvalleutrplysleuleulysvalvallyspheglygluvalile
100105110
sergluserhisleualaalaleuvalglyasnproalaalathrala
115120125
alavalasnthralaleuaspglyasnprovalproileleuilepro
130135140
cyshisargvalvalglnglyaspseraspvalglyprotyrleugly
145150155160
glyleualavallysglutrpleuleualahisgluglyhisargleu
165170175
glylysproglyleugly
180
<210>15
<211>159
<212>prt
<213>大肠杆菌(escherichiacoli)
<220>
<221>chain
<223>dhfr
<400>15
metileserleuilealaalaleualavalaspargvalileglymet
151015
gluasnalametprotrpasnleuproalaaspleualatrpphelys
202530
argasnthrleuasnlysprovalilemetglyarghisthrtrpglu
354045
serileglyargproleuproglyarglysasnileileleuserser
505560
glnproglythraspaspargvalthrtrpvallysservalaspglu
65707580
alailealaalacysglyaspvalprogluilemetvalileglygly
859095
glyargvaltyrgluglnpheleuprolysalaglnlysleutyrleu
100105110
thrhisileaspalagluvalgluglyaspthrhispheproasptyr
115120125
gluproaspasptrpgluservalphesergluphehisaspalaasp
130135140
alaglnasnserhissertyrcysphegluileleugluargarg
145150155
<210>16
<211>278
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>糖皮质激素受体配体结合域
<400>16
ileglnglnalathrthrglyvalserglngluthrsergluasnpro
151015
glyasnlysthrilevalproalathrleuproglnleuthrprothr
202530
leuvalserleuleugluvalilegluprogluvalleutyralagly
354045
tyraspserservalproaspserthrtrpargilemetthrthrleu
505560
asnmetleuglyglyargglnvalilealaalavallystrpalalys
65707580
alaileproglypheargasnleuhisleuaspaspglnmetthrleu
859095
leuglntyrsertrpmetpheleumetalaphealaleuglytrparg
100105110
sertyrargglnserseralaasnleuleucysphealaproaspleu
115120125
ileileasngluglnargmetthrleuprocysmettyraspglncys
130135140
lyshismetleutyrvalsersergluleuhisargleuglnvalser
145150155160
tyrgluglutyrleucysmetlysthrleuleuleuleuserserval
165170175
prolysaspglyleulysserglngluleupheaspgluileargmet
180185190
thrtyrilelysgluleuglylysalailevallysarggluglyasn
195200205
serserglnasntrpglnargphetyrglnleuthrlysleuleuasp
210215220
sermethisgluvalvalgluasnleuleuasntyrcyspheglnthr
225230235240
pheleuasplysthrmetserileglupheproglumetleualaglu
245250255
ileilethrasnglnileprolystyrserasnglyasnilelyslys
260265270
leuleuphehisglnlys
275
<210>17
<211>176
<212>prt
<213>拟南芥(arabidopsisthaliana)
<220>
<221>chain
<223>pyl1
<400>17
glnaspgluphethrglnleuserglnserilealagluphehisthr
151015
tyrglnleuglyasnglyargcysserserleuleualaglnargile
202530
hisalaproprogluthrvaltrpservalvalargargpheasparg
354045
proglniletyrlyshispheilelyssercysasnvalsergluasp
505560
pheglumetargvalglycysthrargaspvalasnvalilesergly
65707580
leuproalaasnthrserarggluargleuaspleuleuaspaspasp
859095
argargvalthrglypheserilethrglyglygluhisargleuarg
100105110
asntyrlysservalthrthrvalhisargpheglulysglugluglu
115120125
glugluargiletrpthrvalvalleuglusertyrvalvalaspval
130135140
progluglyasnserglugluaspthrargleuphealaaspthrval
145150155160
ileargleuasnleuglnlysleualaserilethrglualametasn
165170175
<210>18
<211>298
<212>prt
<213>拟南芥(arabidopsisthaliana)
<220>
<221>chain
<223>abi1
<400>18
valproleutyrglyphethrserilecysglyargargproglumet
151015
gluaspalavalserthrileproargpheleuglnsersersergly
202530
sermetleuaspglyargpheaspproglnseralaalahisphephe
354045
glyvaltyraspglyhisglyglyserglnvalalaasntyrcysarg
505560
gluargmethisleualaleualaglugluilealalysglulyspro
65707580
metleucysaspglyaspthrtrpleuglulystrplyslysalaleu
859095
pheasnserpheleuargvalaspsergluilegluservalalapro
100105110
gluthrvalglyserthrservalvalalavalvalpheproserhis
115120125
ilephevalalaasncysglyaspserargalavalleucysarggly
130135140
lysthralaleuproleuservalasphislysproasparggluasp
145150155160
glualaalaargileglualaalaglyglylysvalileglntrpasn
165170175
glyalaargvalpheglyvalleualametserargserileglyasp
180185190
argtyrleulysproserileileproaspprogluvalthralaval
195200205
lysargvallysgluaspaspcysleuileleualaseraspglyval
210215220
trpaspvalmetthraspgluglualacysglumetalaarglysarg
225230235240
ileleuleutrphislyslysasnalavalalaglyaspalaserleu
245250255
leualaaspgluargarglysgluglylysaspproalaalametser
260265270
alaalaglutyrleuserlysleualaileglnargglyserlysasp
275280285
asnileservalvalvalvalaspleulys
290295
<210>19
<211>20
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>4-eaaar-接头
<400>19
glualaalaalaargglualaalaalaargglualaalaalaargglu
151015
alaalaalaarg
20
<210>20
<211>22
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>gs-4x-eaaar-接头
<400>20
glyserglualaalaalaargglualaalaalaargglualaalaala
151015
argglualaalaalaarg
20
<210>21
<211>45
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>cd8α铰链
<400>21
thrthrthrproalaproargproprothrproalaprothrileala
151015
serglnproleuserleuargproglualacysargproalaalagly
202530
glyalavalhisthrargglyleuaspphealacysasp
354045
<210>22
<211>231
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>igg1铰链
<400>22
gluprolysserproasplysthrhisthrcysproprocysproala
151015
proprovalalaglyproservalpheleupheproprolysprolys
202530
aspthrleumetilealaargthrprogluvalthrcysvalvalval
354045
aspvalserhisgluaspprogluvallyspheasntrptyrvalasp
505560
glyvalgluvalhisasnalalysthrlysproargglugluglntyr
65707580
asnserthrtyrargvalvalservalleuthrvalleuhisglnasp
859095
trpleuasnglylysglutyrlyscyslysvalserasnlysalaleu
100105110
proalaproileglulysthrileserlysalalysglyglnproarg
115120125
gluproglnvaltyrthrleuproproserargaspgluleuthrlys
130135140
asnglnvalserleuthrcysleuvallysglyphetyrproserasp
145150155160
ilealavalglutrpgluserasnglyglnprogluasnasntyrlys
165170175
thrthrproprovalleuaspseraspglyserphepheleutyrser
180185190
lysleuthrvalasplysserargtrpglnglnglyasnvalpheser
195200205
cysservalmethisglualaleuhisasnhistyrthrglnlysser
210215220
leuserleuserproglylys
225230
<210>23
<211>16
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>fcγriiiα铰链
<400>23
glyleualavalserthrileserserphepheproproglytyrgln
151015
<210>24
<211>15
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>肽接头
<400>24
glyglyglyglyserglyglyglyglyserglyglyglyglyser
151015
<210>25
<211>121
<212>prt
<213>小鼠(musmusculus)
<220>
<221>chain
<223>抗人cd19单克隆抗体4g7免疫球蛋白γ1重链的片段-(残基20-140)
<400>25
gluvalglnleuglnglnserglyprogluleuilelysproglyala
151015
servallysmetsercyslysalaserglytyrthrphethrsertyr
202530
valmethistrpvallysglnlysproglyglnglyleuglutrpile
354045
glytyrileasnprotyrasnaspglythrlystyrasnglulysphe
505560
lysglylysalathrleuthrserasplysserserserthralatyr
65707580
metgluleuserserleuthrsergluaspseralavaltyrtyrcys
859095
alaargglythrtyrtyrtyrglyserargvalpheasptyrtrpgly
100105110
glnglythrthrleuthrvalserser
115120
<210>26
<211>115
<212>prt
<213>小鼠(musmusculus)
<220>
<221>chain
<223>抗人cd19单克隆抗体4g7免疫球蛋白κ轻链的片段-(残基21-130)
<400>26
aspilevalmetthrglnalaalaproserileprovalthrprogly
151015
gluservalserilesercysargserserlysserleuleuasnser
202530
asnglyasnthrtyrleutyrtrppheleuglnargproglyglnser
354045
proglnleuleuiletyrargmetserasnleualaserglyvalpro
505560
aspargpheserglyserglyserglythralaphethrleuargile
65707580
serargvalglualagluaspvalglyvaltyrtyrcysmetglnhis
859095
leuglutyrprophethrpheglyalaglythrlysleugluleulys
100105110
argalaasp
115
<210>27
<211>116
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>抗人cd19单克隆抗体4g7免疫球蛋白κ轻链的片段
<400>27
aspilevalmetthrglnalaalaproserileprovalthrprogly
151015
gluservalserilesercysargserserlysserleuleuasnser
202530
asnglyasnthrtyrleutyrtrppheleuglnargproglyglnser
354045
proglnleuleuiletyrargmetserasnleualaserglyvalpro
505560
aspargpheserglyserglyserglythralaphethrleuargile
65707580
serargvalglualagluaspvalglyvaltyrtyrcysmetglnhis
859095
leuglutyrprophethrpheglyalaglythrlysleugluleulys
100105110
argserasppro
115
<210>28
<211>112
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>cd3ζ信号转导结构域
<400>28
argvallyspheserargseralaaspalaproalatyrglnglngly
151015
glnasnglnleutyrasngluleuasnleuglyargarggluglutyr
202530
aspvalleuasplysargargglyargaspproglumetglyglylys
354045
proargarglysasnproglngluglyleutyrasngluleuglnlys
505560
asplysmetalaglualatyrsergluileglymetlysglygluarg
65707580
argargglylysglyhisaspglyleutyrglnglyleuserthrala
859095
thrlysaspthrtyraspalaleuhismetglnalaleuproproarg
100105110
<210>29
<211>215
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>fcεriβ链
<400>29
metaspthrgluserasnargargalaasnleualaleuproglnglu
151015
proserservalproalaphegluvalleugluileserproglnglu
202530
valserserglyargleuleulysseralaserserproproleuhis
354045
thrtrpleuthrvalleulyslysgluglnglupheleuglyvalthr
505560
glnileleuthralametilecysleucyspheglythrvalvalcys
65707580
servalleuaspileserhisilegluglyaspilepheserserphe
859095
lysalaglytyrprophetrpglyalailephepheserilesergly
100105110
metleuserileilesergluargargasnalathrtyrleuvalarg
115120125
glyserleuglyalaasnthralaserserilealaglyglythrgly
130135140
ilethrileleuileileasnleulyslysserleualatyrilehis
145150155160
ilehissercysglnlysphephegluthrlyscysphemetalaser
165170175
pheserthrgluilevalvalmetmetleupheleuthrileleugly
180185190
leuglyseralavalserleuthrilecysglyalaglyglugluleu
195200205
lysglyasnlysvalproglu
210215
<210>30
<211>43
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>fcεriγ链
<400>30
leuglygluproglnleucystyrileleuaspalaileleupheleu
151015
tyrglyilevalleuthrleuleutyrcysargleulysileglnval
202530
arglysalaalailethrsertyrglulysser
3540
<210>31
<211>42
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>4-1bb的共刺激结构域
<400>31
lysargglyarglyslysleuleutyrilephelysglnprophemet
151015
argprovalglnthrthrglnglugluaspglycyssercysargphe
202530
proglugluglugluglyglycysgluleu
3540
<210>32
<211>24
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>人cd8α跨膜结构域
<400>32
iletyriletrpalaproleualaglythrcysglyvalleuleuleu
151015
serleuvalilethrleutyrcys
20
<210>33
<211>27
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>4-1bb跨膜结构域
<400>33
ileileserphepheleualaleuthrserthralaleuleupheleu
151015
leuphepheleuthrleuargpheservalval
2025
<210>34
<211>52
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>fcεriα链
<400>34
phepheileproleuleuvalvalileleuphealavalaspthrgly
151015
leupheileserthrglnglnglnvalthrpheleuleulysilelys
202530
argthrarglysglypheargleuleuasnprohisprolysproasn
354045
prolysasnasn
50
<210>35
<211>43
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>fcεri的γ链
<400>35
leuglygluproglnleucystyrileleuaspalaileleupheleu
151015
tyrglyilevalleuthrleuleutyrcysargleulysileglnval
202530
arglysalaalailethrsertyrglulysser
3540
<210>36
<211>215
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>fcεri的β链
<400>36
metaspthrgluserasnargargalaasnleualaleuproglnglu
151015
proserservalproalaphegluvalleugluileserproglnglu
202530
valserserglyargleuleulysseralaserserproproleuhis
354045
thrtrpleuthrvalleulyslysgluglnglupheleuglyvalthr
505560
glnileleuthralametilecysleucyspheglythrvalvalcys
65707580
servalleuaspileserhisilegluglyaspilepheserserphe
859095
lysalaglytyrprophetrpglyalailephepheserilesergly
100105110
metleuserileilesergluargargasnalathrtyrleuvalarg
115120125
glyserleuglyalaasnthralaserserilealaglyglythrgly
130135140
ilethrileleuileileasnleulyslysserleualatyrilehis
145150155160
ilehissercysglnlysphephegluthrlyscysphemetalaser
165170175
pheserthrgluilevalvalmetmetleupheleuthrileleugly
180185190
leuglyseralavalserleuthrilecysglyalaglyglugluleu
195200205
lysglyasnlysvalproglu
210215
<210>37
<211>2549
<212>prt
<213>智人(homosapiens)
<220>
<221>chain
<223>mtor蛋白质序列,np_004949.1
<400>37
metleuglythrglyproalaalaalathrthralaalathrthrser
151015
serasnvalservalleuglnglnphealaserglyleulysserarg
202530
asnglugluthrargalalysalaalalysgluleuglnhistyrval
354045
thrmetgluleuargglumetserglnglugluserthrargphetyr
505560
aspglnleuasnhishisilephegluleuvalserserseraspala
65707580
asngluarglysglyglyileleualailealaserleuileglyval
859095
gluglyglyasnalathrargileglyargphealaasntyrleuarg
100105110
asnleuleuproserasnaspprovalvalmetglumetalaserlys
115120125
alaileglyargleualametalaglyaspthrphethralaglutyr
130135140
valgluphegluvallysargalaleuglutrpleuglyalaasparg
145150155160
asngluglyargarghisalaalavalleuvalleuarggluleuala
165170175
ileservalprothrphephepheglnglnvalglnprophepheasp
180185190
asnilephevalalavaltrpaspprolysglnalaileargglugly
195200205
alavalalaalaleuargalacysleuileleuthrthrglnargglu
210215220
prolysglumetglnlysproglntrptyrarghisthrphegluglu
225230235240
alaglulysglypheaspgluthrleualalysglulysglymetasn
245250255
argaspaspargilehisglyalaleuleuileleuasngluleuval
260265270
argilesersermetgluglygluargleuarggluglumetgluglu
275280285
ilethrglnglnglnleuvalhisasplystyrcyslysaspleumet
290295300
glypheglythrlysproarghisilethrprophethrserphegln
305310315320
alavalglnproglnglnserasnalaleuvalglyleuleuglytyr
325330335
serserhisglnglyleumetglypheglythrserproserproala
340345350
lysserthrleuvalgluserargcyscysargaspleumetgluglu
355360365
lyspheaspglnvalcysglntrpvalleulyscysargasnserlys
370375380
asnserleuileglnmetthrileleuasnleuleuproargleuala
385390395400
alapheargproseralaphethraspthrglntyrleuglnaspthr
405410415
metasnhisvalleusercysvallyslysglulysgluargthrala
420425430
alapheglnalaleuglyleuleuservalalavalargsergluphe
435440445
lysvaltyrleuproargvalleuaspileileargalaalaleupro
450455460
prolysaspphealahislysargglnlysalametglnvalaspala
465470475480
thrvalphethrcysilesermetleualaargalametglyprogly
485490495
ileglnglnaspilelysgluleuleugluprometleualavalgly
500505510
leuserproalaleuthralavalleutyraspleuserargglnile
515520525
proglnleulyslysaspileglnaspglyleuleulysmetleuser
530535540
leuvalleumethislysproleuarghisproglymetprolysgly
545550555560
leualahisglnleualaserproglyleuthrthrleuprogluala
565570575
seraspvalglyserilethrleualaleuargthrleuglyserphe
580585590
gluphegluglyhisserleuthrglnphevalarghiscysalaasp
595600605
hispheleuasnsergluhislysgluileargmetglualaalaarg
610615620
thrcysserargleuleuthrproserilehisleuileserglyhis
625630635640
alahisvalvalserglnthralavalglnvalvalalaaspvalleu
645650655
serlysleuleuvalvalglyilethraspproaspproaspilearg
660665670
tyrcysvalleualaserleuaspgluargpheaspalahisleuala
675680685
glnalagluasnleuglnalaleuphevalalaleuasnaspglnval
690695700
phegluilearggluleualailecysthrvalglyargleuserser
705710715720
metasnproalaphevalmetpropheleuarglysmetleuilegln
725730735
ileleuthrgluleugluhisserglyileglyargilelysglugln
740745750
seralaargmetleuglyhisleuvalserasnalaproargleuile
755760765
argprotyrmetgluproileleulysalaleuileleulysleulys
770775780
aspproaspproaspproasnproglyvalileasnasnvalleuala
785790795800
thrileglygluleualaglnvalserglyleuglumetarglystrp
805810815
valaspgluleupheileileilemetaspmetleuglnaspserser
820825830
leuleualalysargglnvalalaleutrpthrleuglyglnleuval
835840845
alaserthrglytyrvalvalgluprotyrarglystyrprothrleu
850855860
leugluvalleuleuasnpheleulysthrgluglnasnglnglythr
865870875880
argargglualaileargvalleuglyleuleuglyalaleuasppro
885890895
tyrlyshislysvalasnileglymetileaspglnserargaspala
900905910
seralavalserleusergluserlysserserglnaspserserasp
915920925
tyrserthrserglumetleuvalasnmetglyasnleuproleuasp
930935940
gluphetyrproalavalsermetvalalaleumetargilephearg
945950955960
aspglnserleuserhishishisthrmetvalvalglnalailethr
965970975
pheilephelysserleuglyleulyscysvalglnpheleuprogln
980985990
valmetprothrpheleuasnvalileargvalcysaspglyalaile
99510001005
argglupheleupheglnglnleuglymetleuvalserpheval
101010151020
lysserhisileargprotyrmetaspgluilevalthrleumet
102510301035
arggluphetrpvalmetasnthrserileglnserthrileile
104010451050
leuleuilegluglnilevalvalalaleuglyglygluphelys
105510601065
leutyrleuproglnleuileprohismetleuargvalphemet
107010751080
hisaspasnserproglyargilevalserilelysleuleuala
108510901095
alaileglnleupheglyalaasnleuaspasptyrleuhisleu
110011051110
leuleuproproilevallysleupheaspalaproglualapro
111511201125
leuproserarglysalaalaleugluthrvalaspargleuthr
113011351140
gluserleuaspphethrasptyralaserargileilehispro
114511501155
ilevalargthrleuaspglnserprogluleuargserthrala
116011651170
metaspthrleuserserleuvalpheglnleuglylyslystyr
117511801185
glnilepheileprometvalasnlysvalleuvalarghisarg
119011951200
ileasnhisglnargtyraspvalleuilecysargilevallys
120512101215
glytyrthrleualaaspgluglugluaspproleuiletyrgln
122012251230
hisargmetleuargserglyglnglyaspalaleualasergly
123512401245
provalgluthrglyprometlyslysleuhisvalserthrile
125012551260
asnleuglnlysalatrpglyalaalaargargvalserlysasp
126512701275
asptrpleuglutrpleuargargleuserleugluleuleulys
128012851290
aspserserserproserleuargsercystrpalaleualagln
129513001305
alatyrasnprometalaargaspleupheasnalaalapheval
131013151320
sercystrpsergluleuasngluaspglnglnaspgluleuile
132513301335
argserilegluleualaleuthrserglnaspilealagluval
134013451350
thrglnthrleuleuasnleualagluphemetgluhisserasp
135513601365
lysglyproleuproleuargaspaspasnglyilevalleuleu
137013751380
glygluargalaalalyscysargalatyralalysalaleuhis
138513901395
tyrlysgluleuglupheglnlysglyprothrproalaileleu
140014051410
gluserleuileserileasnasnlysleuglnglnprogluala
141514201425
alaalaglyvalleuglutyralametlyshispheglygluleu
143014351440
gluileglnalathrtrptyrglulysleuhisglutrpgluasp
144514501455
alaleuvalalatyrasplyslysmetaspthrasnlysaspasp
146014651470
progluleumetleuglyargmetargcysleuglualaleugly
147514801485
glutrpglyglnleuhisglnglncyscysglulystrpthrleu
149014951500
valasnaspgluthrglnalalysmetalaargmetalaalaala
150515101515
alaalatrpglyleuglyglntrpaspsermetgluglutyrthr
152015251530
cysmetileproargaspthrhisaspglyalaphetyrargala
153515401545
valleualaleuhisglnaspleupheserleualaglnglncys
155015551560
ileasplysalaargaspleuleuaspalagluleuthralamet
156515701575
alaglyglusertyrserargalatyrglyalametvalsercys
158015851590
hismetleusergluleuglugluvalileglntyrlysleuval
159516001605
progluargarggluileileargglniletrptrpgluargleu
161016151620
glnglycysglnargilevalgluasptrpglnlysileleumet
162516301635
valargserleuvalvalserprohisgluaspmetargthrtrp
164016451650
leulystyralaserleucysglylysserglyargleualaleu
165516601665
alahislysthrleuvalleuleuleuglyvalaspproserarg
167016751680
glnleuasphisproleuprothrvalhisproglnvalthrtyr
168516901695
alatyrmetlysasnmettrplysseralaarglysileaspala
170017051710
pheglnhismetglnhisphevalglnthrmetglnglnglnala
171517201725
glnhisalailealathrgluaspglnglnhislysglngluleu
173017351740
hislysleumetalaargcyspheleulysleuglyglutrpgln
174517501755
leuasnleuglnglyileasngluserthrileprolysvalleu
176017651770
glntyrtyrseralaalathrgluhisaspargsertrptyrlys
177517801785
alatrphisalatrpalavalmetasnpheglualavalleuhis
179017951800
tyrlyshisglnasnglnalaargaspglulyslyslysleuarg
180518101815
hisalaserglyalaasnilethrasnalathrthralaalathr
182018251830
thralaalathralathrthrthralaserthrgluglyserasn
183518401845
sergluserglualagluserthrgluasnserprothrproser
185018551860
proleuglnlyslysvalthrgluaspleuserlysthrleuleu
186518701875
mettyrthrvalproalavalglnglyphepheargserileser
188018851890
leuserargglyasnasnleuglnaspthrleuargvalleuthr
189519001905
leutrppheasptyrglyhistrpproaspvalasnglualaleu
191019151920
valgluglyvallysalaileglnileaspthrtrpleuglnval
192519301935
ileproglnleuilealaargileaspthrproargproleuval
194019451950
glyargleuilehisglnleuleuthraspileglyargtyrhis
195519601965
proglnalaleuiletyrproleuthrvalalaserlysserthr
197019751980
thrthralaarghisasnalaalaasnlysileleulysasnmet
198519901995
cysgluhisserasnthrleuvalglnglnalametmetvalser
200020052010
glugluleuileargvalalaileleutrphisglumettrphis
201520202025
gluglyleugluglualaserargleutyrpheglygluargasn
203020352040
vallysglymetphegluvalleugluproleuhisalametmet
204520502055
gluargglyproglnthrleulysgluthrserpheasnglnala
206020652070
tyrglyargaspleumetglualaglnglutrpcysarglystyr
207520802085
metlysserglyasnvallysaspleuthrglnalatrpaspleu
209020952100
tyrtyrhisvalpheargargileserlysglnleuproglnleu
210521102115
thrserleugluleuglntyrvalserprolysleuleumetcys
212021252130
argaspleugluleualavalproglythrtyraspproasngln
213521402145
proileileargileglnserilealaproserleuglnvalile
215021552160
thrserlysglnargproarglysleuthrleumetglyserasn
216521702175
glyhisgluphevalpheleuleulysglyhisgluaspleuarg
218021852190
glnaspgluargvalmetglnleupheglyleuvalasnthrleu
219522002205
leualaasnaspprothrserleuarglysasnleuserilegln
221022152220
argtyralavalileproleuserthrasnserglyleuilegly
222522302235
trpvalprohiscysaspthrleuhisalaleuileargasptyr
224022452250
argglulyslyslysileleuleuasnilegluhisargilemet
225522602265
leuargmetalaproasptyrasphisleuthrleumetglnlys
227022752280
valgluvalphegluhisalavalasnasnthralaglyaspasp
228522902295
leualalysleuleutrpleulysserprosersergluvaltrp
230023052310
pheaspargargthrasntyrthrargserleualavalmetser
231523202325
metvalglytyrileleuglyleuglyasparghisproserasn
233023352340
leumetleuaspargleuserglylysileleuhisileaspphe
234523502355
glyaspcysphegluvalalametthrargglulyspheproglu
236023652370
lysilepropheargleuthrargmetleuthrasnalametglu
237523802385
valthrglyleuaspglyasntyrargilethrcyshisthrval
239023952400
metgluvalleuarggluhislysaspservalmetalavalleu
240524102415
glualaphevaltyraspproleuleuasntrpargleumetasp
242024252430
thrasnthrlysglyasnlysargserargthrargthraspser
243524402445
tyrseralaglyglnservalgluileleuaspglyvalgluleu
245024552460
glygluproalahislyslysthrglythrthrvalprogluser
246524702475
ilehisserpheileglyaspglyleuvallysproglualaleu
248024852490
asnlyslysalaileglnileileasnargvalargasplysleu
249525002505
thrglyargasppheserhisaspaspthrleuaspvalprothr
251025152520
glnvalgluleuleuilelysglnalathrserhisgluasnleu
252525302535
cysglncystyrileglytrpcysprophetrp
25402545
<210>38
<211>2541
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls24707
<400>38
atgatcccagccgtggtcctgctgctgctgctgctggtggagcaggcagctgcactggga60
gaaccccagctgtgctacatcctggacgccattctgttcctgtacggcattgtgctgaca120
ctgctgtattgtaggctgaagatccaggtccgcaaagccgctattacttcatatgagaag180
agccgcgtgaagttcagccgatccgctgacgcaccagcataccagcagggacagaaccag240
ctgtataacgagctgaatctgggacggagagaggaatacgacgtcctggataagaggcgc300
ggcagggatcctgaaatgggcgggaagcctcgacggaaaaacccacaggaggggctgtac360
aatgaactgcagaaggacaaaatggctgaggcatatagtgaaatcggaatgaagggcgag420
agaaggcgcgggaaaggacacgatggcctgtaccaggggctgtccaccgccacaaaagac480
acttatgatgcactgcatatgcaggccctgccccctcgcggcagcggggccaccaacttc540
tccctgctgaagcaggctggagacgtggaggaaaatcccggccctatggctcccgcaatg600
gagtcccctacactgctgtgcgtggcactgctgttctttgcaccagatggcgtgctggca660
gaggtccagctgcagcagtcaggaccagaactgatcaaacccggagcatctgtgaaaatg720
agttgtaaggcctcaggctatactttcacctcttacgtgatgcactgggtcaagcagaaa780
cctggacagggcctggagtggatcggctatattaatccatacaacgacgggaccaagtac840
aacgaaaagtttaaaggcaaggcaacactgactagtgataagagctcctctactgcttac900
atggagctgagttcactgaccagcgaagactccgctgtgtactattgcgcaagaggaacc960
tactattacggctctagggtgttcgattactgggggcagggaaccacactgacagtcagc1020
tccggaggaggaggatccggaggaggagggtctggaggcgggggaagtgacatcgtgatg1080
acacaggccgctcctagcattccagtgactcccggcgagtcagtcagcatctcctgtcgg1140
tctagtaagagcctgctgaactccaatggaaacacatatctgtactggtttctgcagagg1200
cctggccagtccccacagctgctgatctatcgcatgtctaacctggccagtggcgtgccc1260
gatcggttctctggcagtgggtcaggaaccgcctttacactgaggattagccgcgtcgag1320
gctgaagacgtgggggtctattactgcatgcagcatctggagtaccctttcacatttggc1380
gccgggactaaactggaactgaagcgcgccgatactaccacaccagctccacgaccacct1440
actcctgcaccaaccattgcttcacagcctctgagcctgcgaccagaagcttgccggcca1500
gcagcaggaggagcagtgcacaccagaggcctggacttcgcctgtgatttctttatcccc1560
ctgctggtggtcattctgttcgccgtggacactgggctgtttatctccacccagcagcag1620
gtcacattcctgctgaaaattaagcggaccagaaagggcttccggctgctgaatccccat1680
cctaaaccaaaccccaagaacaatggaagcggagagggacgaggatccctgctgacctgc1740
ggggacgtggaggaaaacccaggacctatggacactgagtctaaccggagagccaatctg1800
gctctgccacaggaacccagctccgtgcccgcattcgaggtcctggaaatctctcctcag1860
gaggtgtctagtgggcgcctgctgaagagtgcctcaagcccccctctgcacacttggctg1920
accgtgctgaagaaagagcaggaattcctgggagtcacccagatcctgacagctatgatt1980
tgcctgtgttttggcacagtggtctgcagtgtgctggacatctcacatattgagggggat2040
atcttctcctcttttaaggctgggtaccctttttggggagcaatcttctttagcatttcc2100
ggaatgctgtcaatcattagcgaaaggcgcaacgcaacatatctggtgcgaggaagcctg2160
ggcgcaaatactgccagttcaatcgccggcgggacaggcatcactattctgatcattaac2220
ctgaagaaaagcctggcttacatccacattcattcctgccagaagttctttgagactaaa2280
tgtttcatggcctcttttagtaccgaaatcgtggtcatgatgctgttcctgaccattctg2340
gggctgggatccgccgtgtctctgacaatctgcggcgctggggaggaactgaagggcaac2400
aaggtcccagagaagcgagggcggaagaaactgctgtatattttcaaacagccttttatg2460
agaccagtgcagaccacacaggaggaagatggctgctcctgtaggtttcccgaggaagag2520
gaaggaggctgtgagctgtga2541
<210>39
<211>373
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>多链嵌合抗原受体的α链
<400>39
metalaproalametgluserprothrleuleucysvalalaleuleu
151015
phephealaproaspglyvalleualagluvalglnleuglnglnser
202530
glyprogluleuilelysproglyalaservallysmetsercyslys
354045
alaserglytyrthrphethrsertyrvalmethistrpvallysgln
505560
lysproglyglnglyleuglutrpileglytyrileasnprotyrasn
65707580
aspglythrlystyrasnglulysphelysglylysalathrleuthr
859095
serasplysserserserthralatyrmetgluleuserserleuthr
100105110
sergluaspseralavaltyrtyrcysalaargglythrtyrtyrtyr
115120125
glyserargvalpheasptyrtrpglyglnglythrthrleuthrval
130135140
serserglyglyglyglyserglyglyglyglyserglyglyglygly
145150155160
seraspilevalmetthrglnalaalaproserileprovalthrpro
165170175
glygluservalserilesercysargserserlysserleuleuasn
180185190
serasnglyasnthrtyrleutyrtrppheleuglnargproglygln
195200205
serproglnleuleuiletyrargmetserasnleualaserglyval
210215220
proaspargpheserglyserglyserglythralaphethrleuarg
225230235240
ileserargvalglualagluaspvalglyvaltyrtyrcysmetgln
245250255
hisleuglutyrprophethrpheglyalaglythrlysleugluleu
260265270
lysargalaaspthrthrthrproalaproargproprothrproala
275280285
prothrilealaserglnproleuserleuargproglualacysarg
290295300
proalaalaglyglyalavalhisthrargglyleuaspphealacys
305310315320
aspphepheileproleuleuvalvalileleuphealavalaspthr
325330335
glyleupheileserthrglnglnglnvalthrpheleuleulysile
340345350
lysargthrarglysglypheargleuleuasnprohisprolyspro
355360365
asnprolysasnasn
370
<210>40
<211>257
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>多链嵌合抗原受体的β链
<400>40
metaspthrgluserasnargargalaasnleualaleuproglnglu
151015
proserservalproalaphegluvalleugluileserproglnglu
202530
valserserglyargleuleulysseralaserserproproleuhis
354045
thrtrpleuthrvalleulyslysgluglnglupheleuglyvalthr
505560
glnileleuthralametilecysleucyspheglythrvalvalcys
65707580
servalleuaspileserhisilegluglyaspilepheserserphe
859095
lysalaglytyrprophetrpglyalailephepheserilesergly
100105110
metleuserileilesergluargargasnalathrtyrleuvalarg
115120125
glyserleuglyalaasnthralaserserilealaglyglythrgly
130135140
ilethrileleuileileasnleulyslysserleualatyrilehis
145150155160
ilehissercysglnlysphephegluthrlyscysphemetalaser
165170175
pheserthrgluilevalvalmetmetleupheleuthrileleugly
180185190
leuglyseralavalserleuthrilecysglyalaglyglugluleu
195200205
lysglyasnlysvalproglulysargglyarglyslysleuleutyr
210215220
ilephelysglnprophemetargprovalglnthrthrglngluglu
225230235240
aspglycyssercysargpheproglugluglugluglyglycysglu
245250255
leu
<210>41
<211>173
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>多链嵌合抗原受体的γ链
<400>41
metileproalavalvalleuleuleuleuleuleuvalgluglnala
151015
alaalaleuglygluproglnleucystyrileleuaspalaileleu
202530
pheleutyrglyilevalleuthrleuleutyrcysargleulysile
354045
glnvalarglysalaalailethrsertyrglulysserargvallys
505560
pheserargseralaaspalaproalatyrglnglnglyglnasngln
65707580
leutyrasngluleuasnleuglyargarggluglutyraspvalleu
859095
asplysargargglyargaspproglumetglyglylysproargarg
100105110
lysasnproglngluglyleutyrasngluleuglnlysasplysmet
115120125
alaglualatyrsergluileglymetlysglygluargargarggly
130135140
lysglyhisaspglyleutyrglnglyleuserthralathrlysasp
145150155160
thrtyraspalaleuhismetglnalaleuproproarg
165170
<210>42
<211>852
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>scfv-gg
<400>42
gaagacacatggctcccgcaatggagtcccctacactgctgtgcgtggcactgctgttct60
ttgcaccagatggcgtgctggcagaggtccagctgcagcagtcaggaccagaactgatca120
aacccggagcatctgtgaaaatgagttgtaaggcctcaggctatactttcacctcttacg180
tgatgcactgggtcaagcagaaacctggacagggcctggagtggatcggctatattaatc240
catacaacgacgggaccaagtacaacgaaaagtttaaaggcaaggcaacactgactagtg300
ataagagctcctctactgcttacatggagctgagttcactgaccagcgaggactccgctg360
tgtactattgcgcaagaggaacctactattacggctctagggtgttcgattactgggggc420
agggaaccacactgacagtcagctccggaggaggagggtccggaggaggagggtctggag480
gcgggggaagtgacatcgtgatgacacaggccgctcctagcattccagtgactcccggcg540
agtcagtcagcatctcctgtcggtctagtaagagcctgctgaactccaatggaaacacat600
atctgtactggtttctgcagaggcctggccagtccccacagctgctgatctatcgcatgt660
ctaacctggccagtggcgtgcccgatcggttctctggcagtgggtcaggaaccgccttta720
cactgaggattagccgcgtcgaggctgaggacgtgggggtctattactgcatgcagcatc780
tggagtaccctttcacatttggcgccgggactaaactggaactgaagcgctcggatcccg840
gaagacgtcttc852
<210>43
<211>315
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>铰链-跨膜-胞质内-gg
<400>43
gaagacactcccactaccacaccagctccacgaccacctactcctgcaccaaccattgct60
tcacagcctctgagcctgcgaccagaagcctgccggccagcagcaggaggagcagtgcac120
accagaggcctggacttcgcctgtgatttctttatccccctgctggtggtcattctgttc180
gccgtggacactgggctgtttatctccacccagcagcaggtcacattcctgctgaaaatt240
aagcggaccagaaagggcttccggctgctgaatccccatcctaaaccaaaccccaagaac300
aatgaatacgtcttc315
<210>44
<211>324
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>frb-gg
<400>44
gaagacacgaagcggaggcagggttgctatcctttggcatgaaatgtggcatgaggggct60
cgaggaggccagtcggctgtacttcggagaaagaaatgtgaagggtatgttcgaggtctt120
ggaacccctgcacgccatgatggagcgagggcctcaaaccctcaaggagacatcttttaa180
ccaggcatatggtagggatctcatggaagcccaggaatggtgcagaaaatacatgaaatc240
cggcaacgtgaaggacctgactcaggcttgggacctgtattaccacgtatttcggcgcat300
tggcagcggggctcccacgtcttc324
<210>45
<211>360
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>fkbp-gg
<400>45
gaagacacgaagcggaggcgtgcaagtcgagactatttcaccaggcgacggtagaacttt60
tccaaaacggggtcagacctgcgtcgttcattacacaggcatgctcgaggatggcaagaa120
gtttgactctagtagggatcgtaacaaacccttcaagttcatgctgggtaagcaggaggt180
gatccgcggctgggaggaaggggtggcacagatgtctgtgggacagcgagccaagctgac240
catcagccctgattatgcttacggagccaccgggcaccccggtatcatacctccccacgc300
tacactggttttcgacgtagaactccttaaattggaaggcagcggggctcccacgtcttc360
<210>46
<211>223
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>frb-fkbp片段,包含gs-4x-eaaar-接头
<400>46
argvalalaileleutrphisglumettrphisgluglyleugluglu
151015
alaserargleutyrpheglygluargasnvallysglymetpheglu
202530
valleugluproleuhisalametmetgluargglyproglnthrleu
354045
lysgluthrserpheasnglnalatyrglyargaspleumetgluala
505560
glnglutrpcysarglystyrmetlysserglyasnvallysaspleu
65707580
thrglnalatrpaspleutyrtyrhisvalpheargargileglyser
859095
glualaalaalaargglualaalaalaargglualaalaalaargglu
100105110
alaalaalaargglyvalglnvalgluthrileserproglyaspgly
115120125
argthrpheprolysargglyglnthrcysvalvalhistyrthrgly
130135140
metleugluaspglylyslyspheaspserserargaspargasnlys
145150155160
prophelysphemetleuglylysglngluvalileargglytrpglu
165170175
gluglyvalalaglnmetservalglyglnargalalysleuthrile
180185190
serproasptyralatyrglyalathrglyhisproglyileilepro
195200205
prohisalathrleuvalpheaspvalgluleuleulysleuglu
210215220
<210>47
<211>222
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>fkbp-frb片段,包含四-eaaar-接头
<400>47
glyvalglnvalgluthrileserproglyaspglyargthrphepro
151015
lysargglyglnthrcysvalvalhistyrthrglymetleugluasp
202530
glylyslyspheaspserserargaspargasnlysprophelysphe
354045
metleuglylysglngluvalileargglytrpglugluglyvalala
505560
glnmetservalglyglnargalalysleuthrileserproasptyr
65707580
alatyrglyalathrglyhisproglyileileproprohisalathr
859095
leuvalpheaspvalgluleuleulysleugluglualaalaalaarg
100105110
glualaalaalaargglualaalaalaargglualaalaalaarggly
115120125
argvalalaileleutrphisglumettrphisgluglyleugluglu
130135140
alaserargleutyrpheglygluargasnvallysglymetpheglu
145150155160
valleugluproleuhisalametmetgluargglyproglnthrleu
165170175
lysgluthrserpheasnglnalatyrglyargaspleumetgluala
180185190
glnglutrpcysarglystyrmetlysserglyasnvallysaspleu
195200205
thrglnalatrpaspleutyrtyrhisvalpheargargile
210215220
<210>48
<211>1470
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls26563
<400>48
atggctcccgcaatggagtcccctacactgctgtgcgtggcactgctgttctttgcacca60
gatggcgtgctggcagaggtccagctgcagcagtcaggaccagaactgatcaaacccgga120
gcatctgtgaaaatgagttgtaaggcctcaggctatactttcacctcttacgtgatgcac180
tgggtcaagcagaaacctggacagggcctggagtggatcggctatattaatccatacaac240
gacgggaccaagtacaacgaaaagtttaaaggcaaggcaacactgactagtgataagagc300
tcctctactgcttacatggagctgagttcactgaccagcgaggactccgctgtgtactat360
tgcgcaagaggaacctactattacggctctagggtgttcgattactgggggcagggaacc420
acactgacagtcagctccggaggaggagggtccggaggaggagggtctggaggcggggga480
agtgacatcgtgatgacacaggccgctcctagcattccagtgactcccggcgagtcagtc540
agcatctcctgtcggtctagtaagagcctgctgaactccaatggaaacacatatctgtac600
tggtttctgcagaggcctggccagtccccacagctgctgatctatcgcatgtctaacctg660
gccagtggcgtgcccgatcggttctctggcagtgggtcaggaaccgcctttacactgagg720
attagccgcgtcgaggctgaggacgtgggggtctattactgcatgcagcatctggagtac780
cctttcacatttggcgccgggactaaactggaactgaagcgctcggatcccggaagcgga840
ggcgtgcaagtcgagactatttcaccaggcgacggtagaacttttccaaaacggggtcag900
acctgcgtcgttcattacacaggcatgctcgaggatggcaagaagtttgactctagtagg960
gatcgtaacaaacccttcaagttcatgctgggtaagcaggaggtgatccgcggctgggag1020
gaaggggtggcacagatgtctgtgggacagcgagccaagctgaccatcagccctgattat1080
gcttacggagccaccgggcaccccggtatcatacctccccacgctacactggttttcgac1140
gtagaactccttaaattggaaggcagcggggctcccactaccacaccagctccacgacca1200
cctactcctgcaccaaccattgcttcacagcctctgagcctgcgaccagaagcctgccgg1260
ccagcagcaggaggagcagtgcacaccagaggcctggacttcgcctgtgatttctttatc1320
cccctgctggtggtcattctgttcgccgtggacactgggctgtttatctccacccagcag1380
caggtcacattcctgctgaaaattaagcggaccagaaagggcttccggctgctgaatccc1440
catcctaaaccaaaccccaagaacaatgaa1470
<210>49
<211>1434
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls26564
<400>49
atggctcccgcaatggagtcccctacactgctgtgcgtggcactgctgttctttgcacca60
gatggcgtgctggcagaggtccagctgcagcagtcaggaccagaactgatcaaacccgga120
gcatctgtgaaaatgagttgtaaggcctcaggctatactttcacctcttacgtgatgcac180
tgggtcaagcagaaacctggacagggcctggagtggatcggctatattaatccatacaac240
gacgggaccaagtacaacgaaaagtttaaaggcaaggcaacactgactagtgataagagc300
tcctctactgcttacatggagctgagttcactgaccagcgaggactccgctgtgtactat360
tgcgcaagaggaacctactattacggctctagggtgttcgattactgggggcagggaacc420
acactgacagtcagctccggaggaggagggtccggaggaggagggtctggaggcggggga480
agtgacatcgtgatgacacaggccgctcctagcattccagtgactcccggcgagtcagtc540
agcatctcctgtcggtctagtaagagcctgctgaactccaatggaaacacatatctgtac600
tggtttctgcagaggcctggccagtccccacagctgctgatctatcgcatgtctaacctg660
gccagtggcgtgcccgatcggttctctggcagtgggtcaggaaccgcctttacactgagg720
attagccgcgtcgaggctgaggacgtgggggtctattactgcatgcagcatctggagtac780
cctttcacatttggcgccgggactaaactggaactgaagcgctcggatcccggaagcgga840
ggcagggttgctatcctttggcatgaaatgtggcatgaggggctcgaggaggccagtcgg900
ctgtacttcggagaaagaaatgtgaagggtatgttcgaggtcttggaacccctgcacgcc960
atgatggagcgagggcctcaaaccctcaaggagacatcttttaaccaggcatatggtagg1020
gatctcatggaagcccaggaatggtgcagaaaatacatgaaatccggcaacgtgaaggac1080
ctgactcaggcttgggacctgtattaccacgtatttcggcgcattggcagcggggctccc1140
actaccacaccagctccacgaccacctactcctgcaccaaccattgcttcacagcctctg1200
agcctgcgaccagaagcctgccggccagcagcaggaggagcagtgcacaccagaggcctg1260
gacttcgcctgtgatttctttatccccctgctggtggtcattctgttcgccgtggacact1320
gggctgtttatctccacccagcagcaggtcacattcctgctgaaaattaagcggaccaga1380
aagggcttccggctgctgaatccccatcctaaaccaaaccccaagaacaatgaa1434
<210>50
<211>1815
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls26881
<400>50
atggctcccgcaatggagtcccctacactgctgtgcgtggcactgctgttctttgcacca60
gatggcgtgctggcagaggtccagctgcagcagtcaggaccagaactgatcaaacccgga120
gcatctgtgaaaatgagttgtaaggcctcaggctatactttcacctcttacgtgatgcac180
tgggtcaagcagaaacctggacagggcctggagtggatcggctatattaatccatacaac240
gacgggaccaagtacaacgaaaagtttaaaggcaaggcaacactgactagtgataagagc300
tcctctactgcttacatggagctgagttcactgaccagcgaggactccgctgtgtactat360
tgcgcaagaggaacctactattacggctctagggtgttcgattactgggggcagggaacc420
acactgacagtcagctccggaggaggagggtccggaggaggagggtctggaggcggggga480
agtgacatcgtgatgacacaggccgctcctagcattccagtgactcccggcgagtcagtc540
agcatctcctgtcggtctagtaagagcctgctgaactccaatggaaacacatatctgtac600
tggtttctgcagaggcctggccagtccccacagctgctgatctatcgcatgtctaacctg660
gccagtggcgtgcccgatcggttctctggcagtgggtcaggaaccgcctttacactgagg720
attagccgcgtcgaggctgaggacgtgggggtctattactgcatgcagcatctggagtac780
cctttcacatttggcgccgggactaaactggaactgaagcgctcggatcccggaagcgga840
ggcgtgcaagtcgagactatttcaccaggcgacggtagaacttttccaaaacggggtcag900
acctgcgtcgttcattacacaggcatgctcgaggatggcaagaagtttgactctagtagg960
gatcgtaacaaacccttcaagttcatgctgggtaagcaggaggtgatccgcggctgggag1020
gaaggggtggcacagatgtctgtgggacagcgagccaagctgaccatcagccctgattat1080
gcttacggagccaccgggcaccccggtatcatacctccccacgctacactggttttcgac1140
gtagaactccttaaattggaagaagcagcggctcgcgaagctgcagcgcgagaggctgct1200
gcccgggaggcggctgcacggggcagggttgctatcctttggcatgaaatgtggcatgag1260
gggctcgaggaggccagtcggctgtacttcggagaaagaaatgtgaagggtatgttcgag1320
gtcttggaacccctgcacgccatgatggagcgagggcctcaaaccctcaaggagacatct1380
tttaaccaggcatatggtagggatctcatggaagcccaggaatggtgcagaaaatacatg1440
aaatccggcaacgtgaaggacctgactcaggcttgggacctgtattaccacgtatttcgg1500
cgcattggcagcggggctcccactaccacaccagctccacgaccacctactcctgcacca1560
accattgcttcacagcctctgagcctgcgaccagaagcctgccggccagcagcaggagga1620
gcagtgcacaccagaggcctggacttcgcctgtgatttctttatccccctgctggtggtc1680
attctgttcgccgtggacactgggctgtttatctccacccagcagcaggtcacattcctg1740
ctgaaaattaagcggaccagaaagggcttccggctgctgaatccccatcctaaaccaaac1800
cccaagaacaatgaa1815
<210>51
<211>1821
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls27123
<400>51
atggctcccgcaatggagtcccctacactgctgtgcgtggcactgctgttctttgcacca60
gatggcgtgctggcagaggtccagctgcagcagtcaggaccagaactgatcaaacccgga120
gcatctgtgaaaatgagttgtaaggcctcaggctatactttcacctcttacgtgatgcac180
tgggtcaagcagaaacctggacagggcctggagtggatcggctatattaatccatacaac240
gacgggaccaagtacaacgaaaagtttaaaggcaaggcaacactgactagtgataagagc300
tcctctactgcttacatggagctgagttcactgaccagcgaggactccgctgtgtactat360
tgcgcaagaggaacctactattacggctctagggtgttcgattactgggggcagggaacc420
acactgacagtcagctccggaggaggagggtccggaggaggagggtctggaggcggggga480
agtgacatcgtgatgacacaggccgctcctagcattccagtgactcccggcgagtcagtc540
agcatctcctgtcggtctagtaagagcctgctgaactccaatggaaacacatatctgtac600
tggtttctgcagaggcctggccagtccccacagctgctgatctatcgcatgtctaacctg660
gccagtggcgtgcccgatcggttctctggcagtgggtcaggaaccgcctttacactgagg720
attagccgcgtcgaggctgaggacgtgggggtctattactgcatgcagcatctggagtac780
cctttcacatttggcgccgggactaaactggaactgaagcgctcggatcccggaagcgga840
ggcagggttgctatcctttggcatgaaatgtggcatgaggggctcgaggaggccagtcgg900
ctgtacttcggagaaagaaatgtgaagggtatgttcgaggtcttggaacccctgcacgcc960
atgatggagcgagggcctcaaaccctcaaggagacatcttttaaccaggcatatggtagg1020
gatctcatggaagcccaggaatggtgcagaaaatacatgaaatccggcaacgtgaaggac1080
ctgactcaggcttgggacctgtattaccacgtatttcggcgcattggcagcgaagcagcg1140
gctcgcgaagctgcagcgcgagaggctgctgcccgggaggcggctgcacggggcgtgcaa1200
gtcgagactatttcaccaggcgacggtagaacttttccaaaacggggtcagacctgcgtc1260
gttcattacacaggcatgctcgaggatggcaagaagtttgactctagtagggatcgtaac1320
aaacccttcaagttcatgctgggtaagcaggaggtgatccgcggctgggaggaaggggtg1380
gcacagatgtctgtgggacagcgagccaagctgaccatcagccctgattatgcttacgga1440
gccaccgggcaccccggtatcatacctccccacgctacactggttttcgacgtagaactc1500
cttaaattggaaggcagcggggctcccactaccacaccagctccacgaccacctactcct1560
gcaccaaccattgcttcacagcctctgagcctgcgaccagaagcctgccggccagcagca1620
ggaggagcagtgcacaccagaggcctggacttcgcctgtgatttctttatccccctgctg1680
gtggtcattctgttcgccgtggacactgggctgtttatctccacccagcagcaggtcaca1740
ttcctgctgaaaattaagcggaccagaaagggcttccggctgctgaatccccatcctaaa1800
ccaaaccccaagaacaatgaa1821
<210>52
<211>490
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls26563(编码的氨基酸序列)
<400>52
metalaproalametgluserprothrleuleucysvalalaleuleu
151015
phephealaproaspglyvalleualagluvalglnleuglnglnser
202530
glyprogluleuilelysproglyalaservallysmetsercyslys
354045
alaserglytyrthrphethrsertyrvalmethistrpvallysgln
505560
lysproglyglnglyleuglutrpileglytyrileasnprotyrasn
65707580
aspglythrlystyrasnglulysphelysglylysalathrleuthr
859095
serasplysserserserthralatyrmetgluleuserserleuthr
100105110
sergluaspseralavaltyrtyrcysalaargglythrtyrtyrtyr
115120125
glyserargvalpheasptyrtrpglyglnglythrthrleuthrval
130135140
serserglyglyglyglyserglyglyglyglyserglyglyglygly
145150155160
seraspilevalmetthrglnalaalaproserileprovalthrpro
165170175
glygluservalserilesercysargserserlysserleuleuasn
180185190
serasnglyasnthrtyrleutyrtrppheleuglnargproglygln
195200205
serproglnleuleuiletyrargmetserasnleualaserglyval
210215220
proaspargpheserglyserglyserglythralaphethrleuarg
225230235240
ileserargvalglualagluaspvalglyvaltyrtyrcysmetgln
245250255
hisleuglutyrprophethrpheglyalaglythrlysleugluleu
260265270
lysargseraspproglyserglyglyvalglnvalgluthrileser
275280285
proglyaspglyargthrpheprolysargglyglnthrcysvalval
290295300
histyrthrglymetleugluaspglylyslyspheaspserserarg
305310315320
aspargasnlysprophelysphemetleuglylysglngluvalile
325330335
argglytrpglugluglyvalalaglnmetservalglyglnargala
340345350
lysleuthrileserproasptyralatyrglyalathrglyhispro
355360365
glyileileproprohisalathrleuvalpheaspvalgluleuleu
370375380
lysleugluglyserglyalaprothrthrthrproalaproargpro
385390395400
prothrproalaprothrilealaserglnproleuserleuargpro
405410415
glualacysargproalaalaglyglyalavalhisthrargglyleu
420425430
aspphealacysaspphepheileproleuleuvalvalileleuphe
435440445
alavalaspthrglyleupheileserthrglnglnglnvalthrphe
450455460
leuleulysilelysargthrarglysglypheargleuleuasnpro
465470475480
hisprolysproasnprolysasnasnglu
485490
<210>53
<211>478
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls26564(编码的氨基酸序列)
<400>53
metalaproalametgluserprothrleuleucysvalalaleuleu
151015
phephealaproaspglyvalleualagluvalglnleuglnglnser
202530
glyprogluleuilelysproglyalaservallysmetsercyslys
354045
alaserglytyrthrphethrsertyrvalmethistrpvallysgln
505560
lysproglyglnglyleuglutrpileglytyrileasnprotyrasn
65707580
aspglythrlystyrasnglulysphelysglylysalathrleuthr
859095
serasplysserserserthralatyrmetgluleuserserleuthr
100105110
sergluaspseralavaltyrtyrcysalaargglythrtyrtyrtyr
115120125
glyserargvalpheasptyrtrpglyglnglythrthrleuthrval
130135140
serserglyglyglyglyserglyglyglyglyserglyglyglygly
145150155160
seraspilevalmetthrglnalaalaproserileprovalthrpro
165170175
glygluservalserilesercysargserserlysserleuleuasn
180185190
serasnglyasnthrtyrleutyrtrppheleuglnargproglygln
195200205
serproglnleuleuiletyrargmetserasnleualaserglyval
210215220
proaspargpheserglyserglyserglythralaphethrleuarg
225230235240
ileserargvalglualagluaspvalglyvaltyrtyrcysmetgln
245250255
hisleuglutyrprophethrpheglyalaglythrlysleugluleu
260265270
lysargseraspproglyserglyglyargvalalaileleutrphis
275280285
glumettrphisgluglyleugluglualaserargleutyrphegly
290295300
gluargasnvallysglymetphegluvalleugluproleuhisala
305310315320
metmetgluargglyproglnthrleulysgluthrserpheasngln
325330335
alatyrglyargaspleumetglualaglnglutrpcysarglystyr
340345350
metlysserglyasnvallysaspleuthrglnalatrpaspleutyr
355360365
tyrhisvalpheargargileglyserglyalaprothrthrthrpro
370375380
alaproargproprothrproalaprothrilealaserglnproleu
385390395400
serleuargproglualacysargproalaalaglyglyalavalhis
405410415
thrargglyleuaspphealacysaspphepheileproleuleuval
420425430
valileleuphealavalaspthrglyleupheileserthrglngln
435440445
glnvalthrpheleuleulysilelysargthrarglysglyphearg
450455460
leuleuasnprohisprolysproasnprolysasnasnglu
465470475
<210>54
<211>605
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls26881(编码的氨基酸序列)
<400>54
metalaproalametgluserprothrleuleucysvalalaleuleu
151015
phephealaproaspglyvalleualagluvalglnleuglnglnser
202530
glyprogluleuilelysproglyalaservallysmetsercyslys
354045
alaserglytyrthrphethrsertyrvalmethistrpvallysgln
505560
lysproglyglnglyleuglutrpileglytyrileasnprotyrasn
65707580
aspglythrlystyrasnglulysphelysglylysalathrleuthr
859095
serasplysserserserthralatyrmetgluleuserserleuthr
100105110
sergluaspseralavaltyrtyrcysalaargglythrtyrtyrtyr
115120125
glyserargvalpheasptyrtrpglyglnglythrthrleuthrval
130135140
serserglyglyglyglyserglyglyglyglyserglyglyglygly
145150155160
seraspilevalmetthrglnalaalaproserileprovalthrpro
165170175
glygluservalserilesercysargserserlysserleuleuasn
180185190
serasnglyasnthrtyrleutyrtrppheleuglnargproglygln
195200205
serproglnleuleuiletyrargmetserasnleualaserglyval
210215220
proaspargpheserglyserglyserglythralaphethrleuarg
225230235240
ileserargvalglualagluaspvalglyvaltyrtyrcysmetgln
245250255
hisleuglutyrprophethrpheglyalaglythrlysleugluleu
260265270
lysargseraspproglyserglyglyvalglnvalgluthrileser
275280285
proglyaspglyargthrpheprolysargglyglnthrcysvalval
290295300
histyrthrglymetleugluaspglylyslyspheaspserserarg
305310315320
aspargasnlysprophelysphemetleuglylysglngluvalile
325330335
argglytrpglugluglyvalalaglnmetservalglyglnargala
340345350
lysleuthrileserproasptyralatyrglyalathrglyhispro
355360365
glyileileproprohisalathrleuvalpheaspvalgluleuleu
370375380
lysleugluglualaalaalaargglualaalaalaargglualaala
385390395400
alaargglualaalaalaargglyargvalalaileleutrphisglu
405410415
mettrphisgluglyleugluglualaserargleutyrpheglyglu
420425430
argasnvallysglymetphegluvalleugluproleuhisalamet
435440445
metgluargglyproglnthrleulysgluthrserpheasnglnala
450455460
tyrglyargaspleumetglualaglnglutrpcysarglystyrmet
465470475480
lysserglyasnvallysaspleuthrglnalatrpaspleutyrtyr
485490495
hisvalpheargargileglyserglyalaprothrthrthrproala
500505510
proargproprothrproalaprothrilealaserglnproleuser
515520525
leuargproglualacysargproalaalaglyglyalavalhisthr
530535540
argglyleuaspphealacysaspphepheileproleuleuvalval
545550555560
ileleuphealavalaspthrglyleupheileserthrglnglngln
565570575
valthrpheleuleulysilelysargthrarglysglypheargleu
580585590
leuasnprohisprolysproasnprolysasnasnglu
595600605
<210>55
<211>607
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls27123(编码的氨基酸序列)
<400>55
metalaproalametgluserprothrleuleucysvalalaleuleu
151015
phephealaproaspglyvalleualagluvalglnleuglnglnser
202530
glyprogluleuilelysproglyalaservallysmetsercyslys
354045
alaserglytyrthrphethrsertyrvalmethistrpvallysgln
505560
lysproglyglnglyleuglutrpileglytyrileasnprotyrasn
65707580
aspglythrlystyrasnglulysphelysglylysalathrleuthr
859095
serasplysserserserthralatyrmetgluleuserserleuthr
100105110
sergluaspseralavaltyrtyrcysalaargglythrtyrtyrtyr
115120125
glyserargvalpheasptyrtrpglyglnglythrthrleuthrval
130135140
serserglyglyglyglyserglyglyglyglyserglyglyglygly
145150155160
seraspilevalmetthrglnalaalaproserileprovalthrpro
165170175
glygluservalserilesercysargserserlysserleuleuasn
180185190
serasnglyasnthrtyrleutyrtrppheleuglnargproglygln
195200205
serproglnleuleuiletyrargmetserasnleualaserglyval
210215220
proaspargpheserglyserglyserglythralaphethrleuarg
225230235240
ileserargvalglualagluaspvalglyvaltyrtyrcysmetgln
245250255
hisleuglutyrprophethrpheglyalaglythrlysleugluleu
260265270
lysargseraspproglyserglyglyargvalalaileleutrphis
275280285
glumettrphisgluglyleugluglualaserargleutyrphegly
290295300
gluargasnvallysglymetphegluvalleugluproleuhisala
305310315320
metmetgluargglyproglnthrleulysgluthrserpheasngln
325330335
alatyrglyargaspleumetglualaglnglutrpcysarglystyr
340345350
metlysserglyasnvallysaspleuthrglnalatrpaspleutyr
355360365
tyrhisvalpheargargileglyserglualaalaalaarggluala
370375380
alaalaargglualaalaalaargglualaalaalaargglyvalgln
385390395400
valgluthrileserproglyaspglyargthrpheprolysarggly
405410415
glnthrcysvalvalhistyrthrglymetleugluaspglylyslys
420425430
pheaspserserargaspargasnlysprophelysphemetleugly
435440445
lysglngluvalileargglytrpglugluglyvalalaglnmetser
450455460
valglyglnargalalysleuthrileserproasptyralatyrgly
465470475480
alathrglyhisproglyileileproprohisalathrleuvalphe
485490495
aspvalgluleuleulysleugluglyserglyalaprothrthrthr
500505510
proalaproargproprothrproalaprothrilealaserglnpro
515520525
leuserleuargproglualacysargproalaalaglyglyalaval
530535540
histhrargglyleuaspphealacysaspphepheileproleuleu
545550555560
valvalileleuphealavalaspthrglyleupheileserthrgln
565570575
glnglnvalthrpheleuleulysilelysargthrarglysglyphe
580585590
argleuleuasnprohisprolysproasnprolysasnasnglu
595600605
<210>56
<211>55
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸引物(α链正向)
<400>56
gcatcgtaatacgactcactatagggcaggccaccatggctcccgcaatggagtc55
<210>57
<211>22
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸引物(α链反向)
<400>57
tcaattgttcttggggtttggt22
<210>58
<211>54
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸引物(β链正向)
<400>58
gcatcgtaatacgactcactatagggcaggccaccatggacactgagtctaacc54
<210>59
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸引物(β链反向)
<400>59
tcacagctcacagcctcctt20
<210>60
<211>55
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸引物(γ链正向)
<400>60
gcatcgtaatacgactcactatagggcaggccaccatgatcccagccgtggtcct55
<210>61
<211>20
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸引物(γ链反向)
<400>61
tcagcgagggggcagggcct20
<210>62
<211>222
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>fkbp-frb*结构域,包含四-eaaar-接头;frb*指的是
是指具有t2098l突变的frb的变体(seqidno:4)
<400>62
glyvalglnvalgluthrileserproglyaspglyargthrphepro
151015
lysargglyglnthrcysvalvalhistyrthrglymetleugluasp
202530
glylyslyspheaspserserargaspargasnlysprophelysphe
354045
metleuglylysglngluvalileargglytrpglugluglyvalala
505560
glnmetservalglyglnargalalysleuthrileserproasptyr
65707580
alatyrglyalathrglyhisproglyileileproprohisalathr
859095
leuvalpheaspvalgluleuleulysleugluglualaalaalaarg
100105110
glualaalaalaargglualaalaalaargglualaalaalaarggly
115120125
argvalalaileleutrphisglumettrphisgluglyleugluglu
130135140
alaserargleutyrpheglygluargasnvallysglymetpheglu
145150155160
valleugluproleuhisalametmetgluargglyproglnthrleu
165170175
lysgluthrserpheasnglnalatyrglyargaspleumetgluala
180185190
glnglutrpcysarglystyrmetlysserglyasnvallysaspleu
195200205
leuglnalatrpaspleutyrtyrhisvalpheargargile
210215220
<210>63
<211>1815
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls27039
<400>63
atggctcccgcaatggagtcccctacactgctgtgcgtggcactgctgttctttgcacca60
gatggcgtgctggcagaggtccagctgcagcagtcaggaccagaactgatcaaacccgga120
gcatctgtgaaaatgagttgtaaggcctcaggctatactttcacctcttacgtgatgcac180
tgggtcaagcagaaacctggacagggcctggagtggatcggctatattaatccatacaac240
gacgggaccaagtacaacgaaaagtttaaaggcaaggcaacactgactagtgataagagc300
tcctctactgcttacatggagctgagttcactgaccagcgaggactccgctgtgtactat360
tgcgcaagaggaacctactattacggctctagggtgttcgattactgggggcagggaacc420
acactgacagtcagctccggaggaggagggtccggaggaggagggtctggaggcggggga480
agtgacatcgtgatgacacaggccgctcctagcattccagtgactcccggcgagtcagtc540
agcatctcctgtcggtctagtaagagcctgctgaactccaatggaaacacatatctgtac600
tggtttctgcagaggcctggccagtccccacagctgctgatctatcgcatgtctaacctg660
gccagtggcgtgcccgatcggttctctggcagtgggtcaggaaccgcctttacactgagg720
attagccgcgtcgaggctgaggacgtgggggtctattactgcatgcagcatctggagtac780
cctttcacatttggcgccgggactaaactggaactgaagcgctcggatcccggaagcgga840
ggcgtgcaagtcgagactatttcaccaggcgacggtagaacttttccaaaacggggtcag900
acctgcgtcgttcattacacaggcatgctcgaggatggcaagaagtttgactctagtagg960
gatcgtaacaaacccttcaagttcatgctgggtaagcaggaggtgatccgcggctgggag1020
gaaggggtggcacagatgtctgtgggacagcgagccaagctgaccatcagccctgattat1080
gcttacggagccaccgggcaccccggtatcatacctccccacgctacactggttttcgac1140
gtagaactccttaaattggaagaagcagcggctcgcgaagctgcagcgcgagaggctgct1200
gcccgggaggcggctgcacggggcagggttgctatcctttggcatgaaatgtggcatgag1260
gggctcgaggaggccagtcggctgtacttcggagaaagaaatgtgaagggtatgttcgag1320
gtcttggaacccctgcacgccatgatggagcgagggcctcaaaccctcaaggagacatct1380
tttaaccaggcatatggtagggatctcatggaagcccaggaatggtgcagaaaatacatg1440
aaatccggcaacgtgaaggacctgctccaggcttgggacctgtattaccacgtatttcgg1500
cgcattggcagcggggctcccactaccacaccagctccacgaccacctactcctgcacca1560
accattgcttcacagcctctgagcctgcgaccagaagcctgccggccagcagcaggagga1620
gcagtgcacaccagaggcctggacttcgcctgtgatttctttatccccctgctggtggtc1680
attctgttcgccgtggacactgggctgtttatctccacccagcagcaggtcacattcctg1740
ctgaaaattaagcggaccagaaagggcttccggctgctgaatccccatcctaaaccaaac1800
cccaagaacaatgaa1815
<210>64
<211>605
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls27039(编码的氨基酸序列)
<400>64
metalaproalametgluserprothrleuleucysvalalaleuleu
151015
phephealaproaspglyvalleualagluvalglnleuglnglnser
202530
glyprogluleuilelysproglyalaservallysmetsercyslys
354045
alaserglytyrthrphethrsertyrvalmethistrpvallysgln
505560
lysproglyglnglyleuglutrpileglytyrileasnprotyrasn
65707580
aspglythrlystyrasnglulysphelysglylysalathrleuthr
859095
serasplysserserserthralatyrmetgluleuserserleuthr
100105110
sergluaspseralavaltyrtyrcysalaargglythrtyrtyrtyr
115120125
glyserargvalpheasptyrtrpglyglnglythrthrleuthrval
130135140
serserglyglyglyglyserglyglyglyglyserglyglyglygly
145150155160
seraspilevalmetthrglnalaalaproserileprovalthrpro
165170175
glygluservalserilesercysargserserlysserleuleuasn
180185190
serasnglyasnthrtyrleutyrtrppheleuglnargproglygln
195200205
serproglnleuleuiletyrargmetserasnleualaserglyval
210215220
proaspargpheserglyserglyserglythralaphethrleuarg
225230235240
ileserargvalglualagluaspvalglyvaltyrtyrcysmetgln
245250255
hisleuglutyrprophethrpheglyalaglythrlysleugluleu
260265270
lysargseraspproglyserglyglyvalglnvalgluthrileser
275280285
proglyaspglyargthrpheprolysargglyglnthrcysvalval
290295300
histyrthrglymetleugluaspglylyslyspheaspserserarg
305310315320
aspargasnlysprophelysphemetleuglylysglngluvalile
325330335
argglytrpglugluglyvalalaglnmetservalglyglnargala
340345350
lysleuthrileserproasptyralatyrglyalathrglyhispro
355360365
glyileileproprohisalathrleuvalpheaspvalgluleuleu
370375380
lysleugluglualaalaalaargglualaalaalaargglualaala
385390395400
alaargglualaalaalaargglyargvalalaileleutrphisglu
405410415
mettrphisgluglyleugluglualaserargleutyrpheglyglu
420425430
argasnvallysglymetphegluvalleugluproleuhisalamet
435440445
metgluargglyproglnthrleulysgluthrserpheasnglnala
450455460
tyrglyargaspleumetglualaglnglutrpcysarglystyrmet
465470475480
lysserglyasnvallysaspleuleuglnalatrpaspleutyrtyr
485490495
hisvalpheargargileglyserglyalaprothrthrthrproala
500505510
proargproprothrproalaprothrilealaserglnproleuser
515520525
leuargproglualacysargproalaalaglyglyalavalhisthr
530535540
argglyleuaspphealacysaspphepheileproleuleuvalval
545550555560
ileleuphealavalaspthrglyleupheileserthrglnglngln
565570575
valthrpheleuleulysilelysargthrarglysglypheargleu
580585590
leuasnprohisprolysproasnprolysasnasnglu
595600605
<210>65
<211>2169
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls27572
<400>65
atggctctgcctgtcaccgccctgctgctgcctctggccctgctgctgcacgccgcacgc60
cctcagattcagctggtccagtccgggcctgagctgaagaaaccaggcgaaaccgtgaag120
atctcttgcaaagctagtggatacattttcacaaactatggcatgaattgggtcaagcag180
gccccagggaagagctttaaatggatgggatggatcaacacttacaccggcgagagtacc240
tattcagctgacttcaagggccgcttcgcattttccctggaaacctcagcaagcacagcc300
tacctgcacattaacgacctgaagaacgaggatacagctacttacttctgcgcacgatcc360
ggaggatacgacccaatggattattggggccaggggacatctgtgactgtcagctccgga420
ggaggaggatccggcggaggaggctctgggggaggcgggagtgacatcgtgctgactcag480
agcccagcttccctggcagtctcactgggacagcgcgcaaccattagctgtcgagcctcc540
gagtctgtggataactacggcaatacattcatgcattggtatcagcagaagcctgggcag600
ccccctaaactgctgatctaccgcgcctcaaatctggaaagcggcattccagctcgcttc660
agtggatcaggcagccggactgactttaccctgacaatcaaccccgtggaggcagacgat720
gtcgccacctactattgccagcagagtaatgaggatccacccacttttggggcaggaacc780
aagctggaactgaaacgatcggatcccggaagcggaggcgtgcaagtcgagactatttca840
ccaggcgacggtagaacttttccaaaacggggtcagacctgcgtcgttcattacacaggc900
atgctcgaggatggcaagaagtttgactctagtagggatcgtaacaaacccttcaagttc960
atgctgggtaagcaggaggtgatccgcggctgggaggaaggggtggcacagatgtctgtg1020
ggacagcgagccaagctgaccatcagccctgattatgcttacggagccaccgggcacccc1080
ggtatcatacctccccacgctacactggttttcgacgtagaactccttaaattggaagaa1140
gcagcggctcgcgaagctgcagcgcgagaggctgctgcccgggaggcggctgcacggggc1200
agggttgctatcctttggcatgaaatgtggcatgaggggctcgaggaggccagtcggctg1260
tacttcggagaaagaaatgtgaagggtatgttcgaggtcttggaacccctgcacgccatg1320
atggagcgagggcctcaaaccctcaaggagacatcttttaaccaggcatatggtagggat1380
ctcatggaagcccaggaatggtgcagaaaatacatgaaatccggcaacgtgaaggacctg1440
ctccaggcttgggacctgtattaccacgtatttcggcgcattggcagcggggctcccacc1500
acaacccccgctccaaggccccctacccccgcaccaactattgcctcccagccactctca1560
ctgcggcctgaggcctgtcggcccgctgctggaggcgcagtgcatacaaggggcctcgat1620
ttcgcctgcgatatttacatctgggcacccctcgccggcacctgcggggtgcttctcctc1680
tccctggtgattaccctgtattgcagacggggccggaagaagctcctctacatttttaag1740
cagcctttcatgcggccagtgcagacaacccaagaggaggatgggtgttcctgcagattc1800
cctgaggaagaggaaggcgggtgcgagctgagagtgaagttctccaggagcgcagatgcc1860
cccgcctatcaacagggccagaaccagctctacaacgagcttaacctcgggaggcgcgaa1920
gaatacgacgtgttggataagagaagggggcgggaccccgagatgggaggaaagccccgg1980
aggaagaaccctcaggagggcctgtacaacgagctgcagaaggataagatggccgaggcc2040
tactcagagatcgggatgaagggggagcggcgccgcgggaaggggcacgatgggctctac2100
caggggctgagcacagccacaaaggacacatacgacgccttgcacatgcaggcccttcca2160
ccccgggaa2169
<210>66
<211>1824
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls27603
<400>66
atggctctgcctgtcaccgccctgctgctgcctctggccctgctgctgcacgccgcacgc60
cctcagattcagctggtccagtccgggcctgagctgaagaaaccaggcgaaaccgtgaag120
atctcttgcaaagctagtggatacattttcacaaactatggcatgaattgggtcaagcag180
gccccagggaagagctttaaatggatgggatggatcaacacttacaccggcgagagtacc240
tattcagctgacttcaagggccgcttcgcattttccctggaaacctcagcaagcacagcc300
tacctgcacattaacgacctgaagaacgaggatacagctacttacttctgcgcacgatcc360
ggaggatacgacccaatggattattggggccaggggacatctgtgactgtcagctccgga420
ggaggaggatccggcggaggaggctctgggggaggcgggagtgacatcgtgctgactcag480
agcccagcttccctggcagtctcactgggacagcgcgcaaccattagctgtcgagcctcc540
gagtctgtggataactacggcaatacattcatgcattggtatcagcagaagcctgggcag600
ccccctaaactgctgatctaccgcgcctcaaatctggaaagcggcattccagctcgcttc660
agtggatcaggcagccggactgactttaccctgacaatcaaccccgtggaggcagacgat720
gtcgccacctactattgccagcagagtaatgaggatccacccacttttggggcaggaacc780
aagctggaactgaaacgatcggatcccggaagcggaggcgtgcaagtcgagactatttca840
ccaggcgacggtagaacttttccaaaacggggtcagacctgcgtcgttcattacacaggc900
atgctcgaggatggcaagaagtttgactctagtagggatcgtaacaaacccttcaagttc960
atgctgggtaagcaggaggtgatccgcggctgggaggaaggggtggcacagatgtctgtg1020
ggacagcgagccaagctgaccatcagccctgattatgcttacggagccaccgggcacccc1080
ggtatcatacctccccacgctacactggttttcgacgtagaactccttaaattggaaggc1140
agcggggctcccaccacaacccccgctccaaggccccctacccccgcaccaactattgcc1200
tcccagccactctcactgcggcctgaggcctgtcggcccgctgctggaggcgcagtgcat1260
acaaggggcctcgatttcgcctgcgatatttacatctgggcacccctcgccggcacctgc1320
ggggtgcttctcctctccctggtgattaccctgtattgcagacggggccggaagaagctc1380
ctctacatttttaagcagcctttcatgcggccagtgcagacaacccaagaggaggatggg1440
tgttcctgcagattccctgaggaagaggaaggcgggtgcgagctgagagtgaagttctcc1500
aggagcgcagatgcccccgcctatcaacagggccagaaccagctctacaacgagcttaac1560
ctcgggaggcgcgaagaatacgacgtgttggataagagaagggggcgggaccccgagatg1620
ggaggaaagccccggaggaagaaccctcaggagggcctgtacaacgagctgcagaaggat1680
aagatggccgaggcctactcagagatcgggatgaagggggagcggcgccgcgggaagggg1740
cacgatgggctctaccaggggctgagcacagccacaaaggacacatacgacgccttgcac1800
atgcaggcccttccaccccgggaa1824
<210>67
<211>1788
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>构建体pcls27604
<400>67
atggctctgcctgtcaccgccctgctgctgcctctggccctgctgctgcacgccgcacgc60
cctcagattcagctggtccagtccgggcctgagctgaagaaaccaggcgaaaccgtgaag120
atctcttgcaaagctagtggatacattttcacaaactatggcatgaattgggtcaagcag180
gccccagggaagagctttaaatggatgggatggatcaacacttacaccggcgagagtacc240
tattcagctgacttcaagggccgcttcgcattttccctggaaacctcagcaagcacagcc300
tacctgcacattaacgacctgaagaacgaggatacagctacttacttctgcgcacgatcc360
ggaggatacgacccaatggattattggggccaggggacatctgtgactgtcagctccgga420
ggaggaggatccggcggaggaggctctgggggaggcgggagtgacatcgtgctgactcag480
agcccagcttccctggcagtctcactgggacagcgcgcaaccattagctgtcgagcctcc540
gagtctgtggataactacggcaatacattcatgcattggtatcagcagaagcctgggcag600
ccccctaaactgctgatctaccgcgcctcaaatctggaaagcggcattccagctcgcttc660
agtggatcaggcagccggactgactttaccctgacaatcaaccccgtggaggcagacgat720
gtcgccacctactattgccagcagagtaatgaggatccacccacttttggggcaggaacc780
aagctggaactgaaacgatcggatcccggaagcggaggcagggttgctatcctttggcat840
gaaatgtggcatgaggggctcgaggaggccagtcggctgtacttcggagaaagaaatgtg900
aagggtatgttcgaggtcttggaacccctgcacgccatgatggagcgagggcctcaaacc960
ctcaaggagacatcttttaaccaggcatatggtagggatctcatggaagcccaggaatgg1020
tgcagaaaatacatgaaatccggcaacgtgaaggacctgctccaggcttgggacctgtat1080
taccacgtatttcggcgcattggcagcggggctcccaccacaacccccgctccaaggccc1140
cctacccccgcaccaactattgcctcccagccactctcactgcggcctgaggcctgtcgg1200
cccgctgctggaggcgcagtgcatacaaggggcctcgatttcgcctgcgatatttacatc1260
tgggcacccctcgccggcacctgcggggtgcttctcctctccctggtgattaccctgtat1320
tgcagacggggccggaagaagctcctctacatttttaagcagcctttcatgcggccagtg1380
cagacaacccaagaggaggatgggtgttcctgcagattccctgaggaagaggaaggcggg1440
tgcgagctgagagtgaagttctccaggagcgcagatgcccccgcctatcaacagggccag1500
aaccagctctacaacgagcttaacctcgggaggcgcgaagaatacgacgtgttggataag1560
agaagggggcgggaccccgagatgggaggaaagccccggaggaagaaccctcaggagggc1620
ctgtacaacgagctgcagaaggataagatggccgaggcctactcagagatcgggatgaag1680
ggggagcggcgccgcgggaaggggcacgatgggctctaccaggggctgagcacagccaca1740
aaggacacatacgacgccttgcacatgcaggcccttccaccccgggaa1788
<210>68
<211>723
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls27572(编码的氨基酸序列)
<400>68
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglnileglnleuvalglnserglyprogluleu
202530
lyslysproglygluthrvallysilesercyslysalaserglytyr
354045
ilephethrasntyrglymetasntrpvallysglnalaproglylys
505560
serphelystrpmetglytrpileasnthrtyrthrglygluserthr
65707580
tyrseralaaspphelysglyargphealapheserleugluthrser
859095
alaserthralatyrleuhisileasnaspleulysasngluaspthr
100105110
alathrtyrphecysalaargserglyglytyraspprometasptyr
115120125
trpglyglnglythrservalthrvalserserglyglyglyglyser
130135140
glyglyglyglyserglyglyglyglyseraspilevalleuthrgln
145150155160
serproalaserleualavalserleuglyglnargalathrileser
165170175
cysargalasergluservalaspasntyrglyasnthrphemethis
180185190
trptyrglnglnlysproglyglnproprolysleuleuiletyrarg
195200205
alaserasnleugluserglyileproalaargpheserglysergly
210215220
serargthraspphethrleuthrileasnprovalglualaaspasp
225230235240
valalathrtyrtyrcysglnglnserasngluaspproprothrphe
245250255
glyalaglythrlysleugluleulysargseraspproglysergly
260265270
glyvalglnvalgluthrileserproglyaspglyargthrphepro
275280285
lysargglyglnthrcysvalvalhistyrthrglymetleugluasp
290295300
glylyslyspheaspserserargaspargasnlysprophelysphe
305310315320
metleuglylysglngluvalileargglytrpglugluglyvalala
325330335
glnmetservalglyglnargalalysleuthrileserproasptyr
340345350
alatyrglyalathrglyhisproglyileileproprohisalathr
355360365
leuvalpheaspvalgluleuleulysleugluglualaalaalaarg
370375380
glualaalaalaargglualaalaalaargglualaalaalaarggly
385390395400
argvalalaileleutrphisglumettrphisgluglyleugluglu
405410415
alaserargleutyrpheglygluargasnvallysglymetpheglu
420425430
valleugluproleuhisalametmetgluargglyproglnthrleu
435440445
lysgluthrserpheasnglnalatyrglyargaspleumetgluala
450455460
glnglutrpcysarglystyrmetlysserglyasnvallysaspleu
465470475480
leuglnalatrpaspleutyrtyrhisvalpheargargileglyser
485490495
glyalaprothrthrthrproalaproargproprothrproalapro
500505510
thrilealaserglnproleuserleuargproglualacysargpro
515520525
alaalaglyglyalavalhisthrargglyleuaspphealacysasp
530535540
iletyriletrpalaproleualaglythrcysglyvalleuleuleu
545550555560
serleuvalilethrleutyrcysargargglyarglyslysleuleu
565570575
tyrilephelysglnprophemetargprovalglnthrthrglnglu
580585590
gluaspglycyssercysargpheproglugluglugluglyglycys
595600605
gluleuargvallyspheserargseralaaspalaproalatyrgln
610615620
glnglyglnasnglnleutyrasngluleuasnleuglyargargglu
625630635640
glutyraspvalleuasplysargargglyargaspproglumetgly
645650655
glylysproargarglysasnproglngluglyleutyrasngluleu
660665670
glnlysasplysmetalaglualatyrsergluileglymetlysgly
675680685
gluargargargglylysglyhisaspglyleutyrglnglyleuser
690695700
thralathrlysaspthrtyraspalaleuhismetglnalaleupro
705710715720
proargglu
<210>69
<211>608
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls27603(编码的氨基酸序列)
<400>69
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglnileglnleuvalglnserglyprogluleu
202530
lyslysproglygluthrvallysilesercyslysalaserglytyr
354045
ilephethrasntyrglymetasntrpvallysglnalaproglylys
505560
serphelystrpmetglytrpileasnthrtyrthrglygluserthr
65707580
tyrseralaaspphelysglyargphealapheserleugluthrser
859095
alaserthralatyrleuhisileasnaspleulysasngluaspthr
100105110
alathrtyrphecysalaargserglyglytyraspprometasptyr
115120125
trpglyglnglythrservalthrvalserserglyglyglyglyser
130135140
glyglyglyglyserglyglyglyglyseraspilevalleuthrgln
145150155160
serproalaserleualavalserleuglyglnargalathrileser
165170175
cysargalasergluservalaspasntyrglyasnthrphemethis
180185190
trptyrglnglnlysproglyglnproprolysleuleuiletyrarg
195200205
alaserasnleugluserglyileproalaargpheserglysergly
210215220
serargthraspphethrleuthrileasnprovalglualaaspasp
225230235240
valalathrtyrtyrcysglnglnserasngluaspproprothrphe
245250255
glyalaglythrlysleugluleulysargseraspproglysergly
260265270
glyvalglnvalgluthrileserproglyaspglyargthrphepro
275280285
lysargglyglnthrcysvalvalhistyrthrglymetleugluasp
290295300
glylyslyspheaspserserargaspargasnlysprophelysphe
305310315320
metleuglylysglngluvalileargglytrpglugluglyvalala
325330335
glnmetservalglyglnargalalysleuthrileserproasptyr
340345350
alatyrglyalathrglyhisproglyileileproprohisalathr
355360365
leuvalpheaspvalgluleuleulysleugluglyserglyalapro
370375380
thrthrthrproalaproargproprothrproalaprothrileala
385390395400
serglnproleuserleuargproglualacysargproalaalagly
405410415
glyalavalhisthrargglyleuaspphealacysaspiletyrile
420425430
trpalaproleualaglythrcysglyvalleuleuleuserleuval
435440445
ilethrleutyrcysargargglyarglyslysleuleutyrilephe
450455460
lysglnprophemetargprovalglnthrthrglnglugluaspgly
465470475480
cyssercysargpheproglugluglugluglyglycysgluleuarg
485490495
vallyspheserargseralaaspalaproalatyrglnglnglygln
500505510
asnglnleutyrasngluleuasnleuglyargarggluglutyrasp
515520525
valleuasplysargargglyargaspproglumetglyglylyspro
530535540
argarglysasnproglngluglyleutyrasngluleuglnlysasp
545550555560
lysmetalaglualatyrsergluileglymetlysglygluargarg
565570575
argglylysglyhisaspglyleutyrglnglyleuserthralathr
580585590
lysaspthrtyraspalaleuhismetglnalaleuproproargglu
595600605
<210>70
<211>596
<212>prt
<213>人工序列(artificialsequence)
<220>
<221>chain
<223>构建体pcls27604(编码的氨基酸序列)
<400>70
metalaleuprovalthralaleuleuleuproleualaleuleuleu
151015
hisalaalaargproglnileglnleuvalglnserglyprogluleu
202530
lyslysproglygluthrvallysilesercyslysalaserglytyr
354045
ilephethrasntyrglymetasntrpvallysglnalaproglylys
505560
serphelystrpmetglytrpileasnthrtyrthrglygluserthr
65707580
tyrseralaaspphelysglyargphealapheserleugluthrser
859095
alaserthralatyrleuhisileasnaspleulysasngluaspthr
100105110
alathrtyrphecysalaargserglyglytyraspprometasptyr
115120125
trpglyglnglythrservalthrvalserserglyglyglyglyser
130135140
glyglyglyglyserglyglyglyglyseraspilevalleuthrgln
145150155160
serproalaserleualavalserleuglyglnargalathrileser
165170175
cysargalasergluservalaspasntyrglyasnthrphemethis
180185190
trptyrglnglnlysproglyglnproprolysleuleuiletyrarg
195200205
alaserasnleugluserglyileproalaargpheserglysergly
210215220
serargthraspphethrleuthrileasnprovalglualaaspasp
225230235240
valalathrtyrtyrcysglnglnserasngluaspproprothrphe
245250255
glyalaglythrlysleugluleulysargseraspproglysergly
260265270
glyargvalalaileleutrphisglumettrphisgluglyleuglu
275280285
glualaserargleutyrpheglygluargasnvallysglymetphe
290295300
gluvalleugluproleuhisalametmetgluargglyproglnthr
305310315320
leulysgluthrserpheasnglnalatyrglyargaspleumetglu
325330335
alaglnglutrpcysarglystyrmetlysserglyasnvallysasp
340345350
leuleuglnalatrpaspleutyrtyrhisvalpheargargilegly
355360365
serglyalaprothrthrthrproalaproargproprothrproala
370375380
prothrilealaserglnproleuserleuargproglualacysarg
385390395400
proalaalaglyglyalavalhisthrargglyleuaspphealacys
405410415
aspiletyriletrpalaproleualaglythrcysglyvalleuleu
420425430
leuserleuvalilethrleutyrcysargargglyarglyslysleu
435440445
leutyrilephelysglnprophemetargprovalglnthrthrgln
450455460
glugluaspglycyssercysargpheproglugluglugluglygly
465470475480
cysgluleuargvallyspheserargseralaaspalaproalatyr
485490495
glnglnglyglnasnglnleutyrasngluleuasnleuglyargarg
500505510
gluglutyraspvalleuasplysargargglyargaspproglumet
515520525
glyglylysproargarglysasnproglngluglyleutyrasnglu
530535540
leuglnlysasplysmetalaglualatyrsergluileglymetlys
545550555560
glygluargargargglylysglyhisaspglyleutyrglnglyleu
565570575
serthralathrlysaspthrtyraspalaleuhismetglnalaleu
580585590
proproargglu
595
<210>71
<211>56
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸sccar-f
<400>71
gcatcgtaatacgactcactatagggcaggccaccatggctttgcctgtcactgcc56
<210>72
<211>26
<212>dna
<213>人工序列(artificialsequence)
<220>
<221>misc_feature
<223>寡核苷酸sccar-r
<400>72
ctagaggatcgctcgagttaggtctc26
- 还没有人留言评论。精彩留言会获得点赞!
- 金黄色葡萄球菌肠毒素C2的核酸适配体C203及其筛选方法和应用与制造工艺
- 一种全人源化EGFRvIII嵌合抗原受体T细胞及其制备方法与制造工艺
- 嵌合抗原受体及制备方法与制造工艺
- 检测外周血CAR‑T细胞的TaqMan实时荧光定量PCR试剂盒的制造方法与工艺
- CD20特异性嵌合抗原受体及其应用的制造方法与工艺
- 一种用于辣根过氧化物酶染色的染色剂及其制备方法、染色组合、应用及标记酶染色试剂盒与制造工艺
- 对SSEA4抗原具有特异性的嵌合抗原受体的制造方法与工艺
- 具有针对CD79和CD22的特异性的分子的制造方法与工艺
- 具有针对CD45和CD79的特异性的分子的制造方法与工艺
- 一种检测嵌合抗原受体T细胞对肝癌细胞抑制作用的模型和方法与制造工艺
- 抗cd33嵌合抗原受体、编码基因、重组表达载体及其构建方法和应用
- 抗cd138嵌合抗原受体、编码基因、重组表达载体及其构建方法和应用
- 用修饰的嵌合抗原受体靶向肿瘤新生血管的制作方法
- Cd19靶向性的嵌合抗原受体和nkt细胞及其制法和应用
- 表达嵌合抗原受体的脐血有核细胞及其应用
- 嵌合抗原受体及其基因和重组表达载体、工程化her2靶向性的nkt细胞及其应用
- 嵌合抗原受体及其基因和重组表达载体、工程化cd33靶向性的nkt细胞及其应用
- 嵌合抗原受体及其基因和重组表达载体、工程化cd138靶向性的nkt细胞及其应用
- 嵌合抗原受体及其基因和重组表达载体、工程化cd138靶向性的nkt细胞及其应用
- 嵌合抗原受体及其基因和重组表达载体、工程化cd19靶向性的nkt细胞及其应用