酸脱羧酶的蛋白与蛋白相互作用的控制的制作方法
大多数酶在较窄的ph范围内发挥最佳作用,因为它们是两性分子。周围环境的ph直接影响组成酶的氨基酸的酸性和碱性基团上的电荷。电荷的这些变化影响酶的净电荷、活性位点的pka和酶表面上的电荷分布。结果,ph的变化可以影响酶的活性、溶解性和稳定性。
已知为酸脱羧酶的一类蛋白是催化氨基酸例如碱性氨基酸如赖氨酸、精氨酸、鸟氨酸的脱羧反应以便产生产物例如多胺(作为许多微生物中酸应激反应的部分)的一组酶。大肠杆菌(escherichiacoli)具有6种吡哆醛5′-磷酸(plp)-诱导型酸脱羧酶:cada、ldcc、adia、spea、spec、spef、gada和gadb。所有这些酶均在较窄的ph范围内发挥作用,并且酶的活性在该ph范围外显著降低(kanjeeetal.,biochemistry50,9388-9398,2011)。以前已观察到这些plp依赖性脱羧酶二聚化以形成完整的活性位点。在某些情况下,如cada,二聚体形成十聚体,聚集为最佳功能需要的较高分子量蛋白复合物。较高分子量蛋白复合物形成的抑制(例如,在最佳ph外的条件下)导致功能的显著下降(kanjeeetal.,theembojournal30,931-944,2011)。
以前对多胺生成的研究集中在各种酸脱羧酶的过表达。但是,没有任何在各种应激如碱性ph下增加酶活性的稳定性的研究。酸脱羧酶对碱性ph的耐受很重要,因为它们作为产物产生的多胺增加反应环境的ph。因此,酸脱羧酶的活性通常随着更多多胺的产生而降低,当反应环境的ph超过酸脱羧酶耐受的ph范围时这可以引起脱羧反应过早终止。
产生多胺(例如,尸胺)的典型过程使用与用来产生氨基酸(例如,赖氨酸)的相似的过程和发酵培养基(qianetal.,biotechnol.bioeng.108,93-103,2010)。例如,由于其提供氮的能力和略微酸性(0.1m溶液具有ph5.5),硫酸铵是主要氮源。酸性ph保持酸脱羧酶的ph范围,如赖氨酸脱羧酶,例如,cada,以便发挥功能。但是,使用硫酸铵在培养基中留下硫酸根离子,其成为副产物,是发酵过程中的盐废物。耐受碱性ph的能力允许使用替代氮源,并且在发酵过程中产生较少盐废物。
已鉴定朊病毒是引起传染性海绵状脑病的传染物(与“疯牛病”,绵羊瘙痒症,人库鲁病和克雅病相似)。对朊病毒的综述,参见derkatch&liebman,prion1:3,161-169,2007。朊病毒是传染性的蛋白构象。当它们不在朊病毒构象中时,蛋白在细胞中可以具有其他作用。形成朊病毒构象的蛋白不是同源的,但是一些富含谷氨酰胺或天冬酰胺残基。朊病毒聚集体是高度有序的,并且通常通过β-链之间的分子间相互作用形成。因此,朊病毒构象富含β-折叠,并且组装成类似淀粉样蛋白纤维的结构。参见,例如,derkatch&liebman,prion1:3,161-169,2007。
还已在酵母中鉴定朊病毒,并且首先在两个酵母决定簇中观察到:[psi+]和[ure3](kushnirov&ter-avanesyan,cell94,13-16,1998)。观察到朊病毒的形成可以通过蛋白sup35或ure2的过表达诱导。发现具有[psi+]或[ure3]表型的酵母细胞中的sup35和ure2蛋白表现出增加的蛋白酶抗性,并且发现处于高分子量聚集状态。除了sup35和ure2蛋白,在酵母中鉴定了其他两种蛋白具有形成朊病毒构象的能力–new1和rnq1(osherovichetal.,plosbiology2,442-451,2004)。像sup35和ure2,new1和rnq1也具有富含谷氨酰胺和天冬酰胺的长系列序列。以前,sup35已融合至pgex-4t-3中的gst(25kd蛋白)(onoetal.,biosci.biotechnol.biochem.70,2813-2823,2006),并且sup35、new1和rnq1已融合至gfp(27kd蛋白)(garrityetal.,pnas107,10596-10601,2010)。但是,这些蛋白相对较小。酸脱羧酶单体为约81kd(比gst大3倍以上),并且形成较高分子量结构,例如,大于1620kd(比gst大60倍以上),以便发挥功能。没有研究评价朊病毒是否可以融合至大得多的蛋白而不影响功能。
发明概述
这个发明部分是基于令人惊讶的发现,将朊病毒蛋白融合至酸脱羧酶增加各种应激下的酶活性的稳定性,所述应激通常引起蛋白复合物从高寡聚状态转变为低寡聚状态(例如,碱性ph和高温)。
在一方面,本公开因此提供遗传修饰的宿主细胞,其包含编码酸脱羧酶融合蛋白的核酸,所述酸脱羧酶融合蛋白包含连接至朊病毒亚基的酸脱羧酶多肽,所述朊病毒亚基融合至所述酸脱羧酶多肽的羧基端,其中相对于未连接至所述朊病毒亚基的酸脱羧酶多肽,酸脱羧酶融合多肽具有增加的活性。在一些实施方案中,朊病毒亚基长度为至少50个氨基酸,长度为至少75个氨基酸或长度为至少100个氨基酸,但是长度为500个氨基酸或更少。朊病毒亚基通常具有10%或更多谷氨酰胺和/或天冬酰胺残基的氨基酸组成。在一些实施方案中,朊病毒亚基包含具有至少20%谷氨酰胺和/或天冬酰胺残基的氨基酸组成。在一些实施方案中,朊病毒亚基与sup35、new1、ure2或rnq1氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含sup35、new1、ure2或rnq1氨基酸序列。在一些实施方案中,朊病毒亚基与seqidno:3或seqidno:4中示出的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:3或seqidno:4的氨基酸序列。在进一步的实施方案中,朊病毒亚基在羧基末端连接至bst片段、λci片段或reca片段,例如具有氨基酸序列rrfgeassaf、asqwpeetfg或egvaetnedf的片段。在一些实施方案中,朊病毒亚基在c-末端连接至bst片段并且与seqidno:15除接头区外的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:15除接头区外的氨基酸序列。在一些实施方案中,朊病毒亚基在c-末端连接至λci片段并且与seqidno:16除接头区外的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:16除接头区外的氨基酸序列。在一些实施方案中,朊病毒亚基在c-末端连接至reca片段并且与seqidno:19除接头区外的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:19除接头区外的氨基酸序列。在一些实施方案中,酸脱羧酶是赖氨酸脱羧酶、鸟氨酸脱羧酶、谷氨酸脱羧酶或精氨酸脱羧酶。在一些实施方案中,酸脱羧酶是cada、ldcc、adia、spea、spec、spef、gada或gadb多肽。例如,在一些实施方案中,酸脱羧酶是赖氨酸脱羧酶,如cada赖氨酸脱羧酶多肽或ldcc多肽。在一些实施方案中,其中酸脱羧酶是赖氨酸脱羧酶,将宿主细胞遗传修饰以过表达一种或多种赖氨酸生物合成多肽。在一些实施方案中,编码酸脱羧酶融合蛋白的核酸由引入细胞的表达载体编码,其中所述表达载体包含可操作地连接至启动子的编码酸脱羧酶融合蛋白的核酸。在可选实施方案中,将编码酸脱羧酶融合蛋白的核酸整合至宿主染色体中。宿主细胞可以是细菌,如来自埃希氏菌属(escherichia)或哈夫尼菌属(hafnia)的细菌。在一些实施方案中,宿主细胞是大肠杆菌或蜂房哈夫尼菌(hafniaalvei)。
在另一方面,本发明提供一种制备酸脱羧酶融合蛋白的方法,所述方法包括在表达酸脱羧酶融合蛋白的条件下培养如前段所述的宿主细胞。在另一方面,本发明提供一种制备氨基酸或氨基酸衍生物的方法,所述方法包括在表达酸脱羧酶融合多肽的条件下培养如前段所述的宿主细胞。
本发明额外地提供一种在酸性ph和/或高温下提高体外酸脱羧酶活性的方法。在一些实施方案中,所述方法包括将朊病毒亚基融合至酸脱羧酶的羧基末端并使融合蛋白经受碱性ph。在一些实施方案中,所述方法包括将朊病毒亚基融合至酸脱羧酶的羧基末端并使融合蛋白经受高温。
在额外方面,本发明提供一种酸脱羧酶融合蛋白,其包含融合至朊病毒亚基的酸脱羧酶多肽,其中如通过多胺生成测量的,在升高的温度和/或碱性ph下,所述融合蛋白在体外相对于缺少朊病毒亚基的对应融合蛋白具有提高的酸脱羧酶活性。在一些实施方案中,朊病毒亚基长度为30个氨基酸,长度为至少50个氨基酸,长度为至少75个氨基酸或长度为至少100个氨基酸,但是长度为1200个氨基酸或更少。在一些实施方案中,朊病毒亚基包含具有至少20%谷氨酰胺和/或天冬酰胺残基的氨基酸组成。在进一步的实施方案中,朊病毒亚基与sup35、new1、ure2或rnq1氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含sup35、new1、ure2或rnq1氨基酸序列。在其他实施方案中,朊病毒亚基与seqidno:3或seqidno:4中示出的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:3或seqidno:4的氨基酸序列。在其他实施方案中,朊病毒亚基与seqidno:7、8、11、12、13、14、15或19的朊病毒亚基区的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:7、8、11、12、13、14、15或19的朊病毒亚基区。在一些实施方案中,朊病毒亚基连接至酸脱羧酶的c-末端。在一些实施方案中,朊病毒亚基在羧基末端连接至稳定性片段,例如bst片段、λci片段或reca片段,如具有氨基酸序列rrfgeassaf、asqwpeetfg或egvaetnedf的片段。在一些实施方案中,朊病毒亚基在c-末端连接至bst片段并且与seqidno:15除接头区外的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:15除接头区外的氨基酸序列。在一些实施方案中,朊病毒亚基在c-末端连接至λci片段并且与seqidno:16除接头区外的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:16除接头区外的氨基酸序列。在一些实施方案中,朊病毒亚基在c-末端连接至reca片段并且与seqidno:19除接头区外的氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含seqidno:19除接头区外的氨基酸序列。在一些实施方案中,酸脱羧酶是赖氨酸脱羧酶、鸟氨酸脱羧酶、精氨酸脱羧酶或谷氨酸脱羧酶。在其他实施方案中,酸脱羧酶是cada、ldcc、adia、spea、spec、spef、gada或gadb多肽。在一些实施方案中,酸脱羧酶是赖氨酸脱羧酶,如cada赖氨酸脱羧酶,并且如通过尸胺生成测量的,在升高的温度和/或碱性ph下,融合蛋白相对于缺少朊病毒亚基的对应融合蛋白具有提高的体外赖氨酸脱羧酶活性。在一些实施方案中,将融合蛋白固定在固体支持物上。
在其他方面,本发明提供编码如本文所述的融合蛋白的多核苷酸以及包含这类多核苷酸的表达载体。
本发明的其他方面在下文中进一步描述。
附图说明
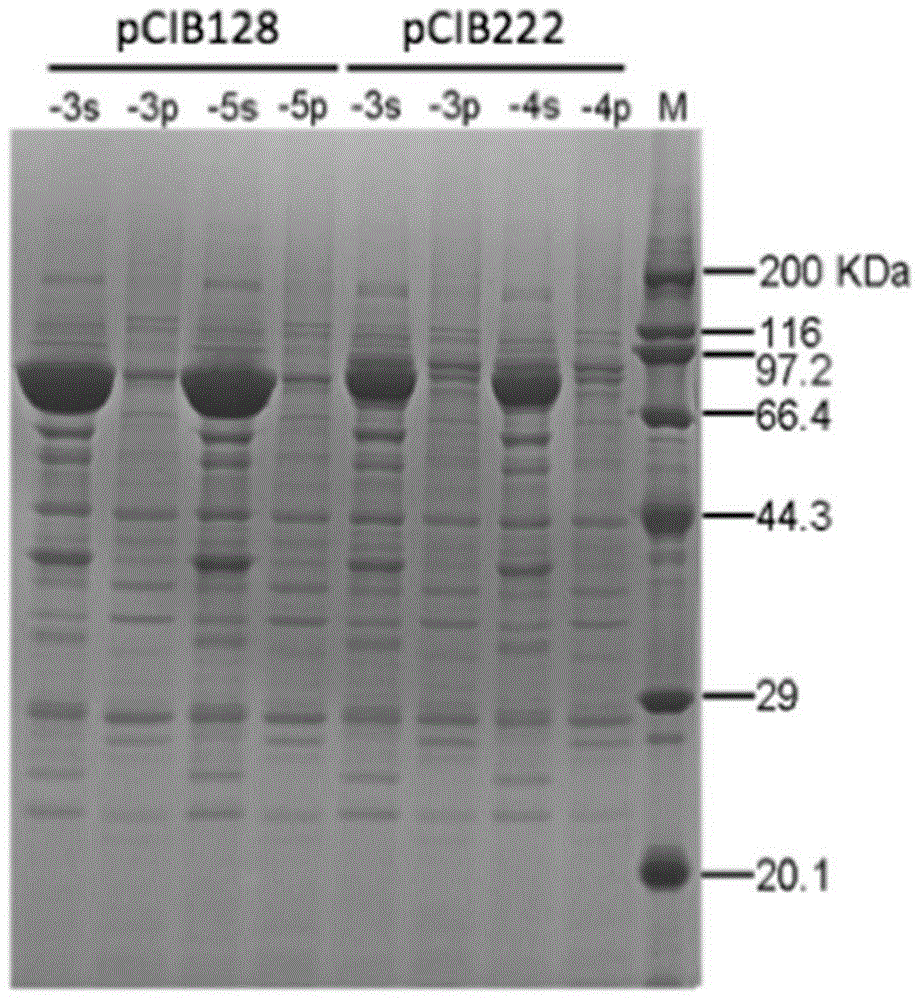
图1.sds-page结果,示出来自用pcib128或pcib222转化的蜂房哈夫尼菌(h.avlei)的裂解的细胞培养物的可溶性(s)和沉淀(p)级分。对每个转化子示出来自两个不同菌落的结果。
图2.sds-page结果,示出来自用pcib222或其截短变体之一转化的大肠杆菌(e.coli)bl21的裂解的细胞培养物的总蛋白。对每个转化子示出来自两个不同菌落的结果。
发明详述
在描述本发明之前,应当理解本发明并不限于描述的特定实施方案,因此当然可以变化。还应当理解本文中使用的术语仅仅是为了描述特定实施方案的目的而不是为了进行限制。
除非另有定义,本文所用的所有技术和科学术语均具有与本发明所属领域的技术人员通常理解的相同的含义。尽管与本文所述的方法和材料相似或等价的任何方法和材料可以用于本发明的实施或测试,但是现在描述优选的方法和材料。本文提到的所有出版物和登录号均通过公开并描述引用出版物相关的方法和/或材料加入本文参考。
术语
“朊病毒”是指包含可以触发正常蛋白折叠为多种、结构不同构象的蛋白物质的传染物(对朊病毒的综述,参见derkatch&liebman,prion1:3,161-169,2007)。分离自酿酒酵母(s.cerevisiae)的酵母朊病毒蛋白的说明性实例包括sup35、ure2、new1、rnq1、swi1、cyc8、mot3、spf1和mod5。分离自podosporaanserine的丝状真菌朊病毒蛋白的说明性实例是het-s。如本领域常用的术语“朊病毒变体”是指具有不同特性的朊病毒分离株,尽管是基于具有相同序列的朊病毒蛋白。如本发明所用的“朊病毒序列变体”是指不同于天然朊病毒氨基酸序列的氨基酸序列但在融合至酸脱羧酶时保留天然朊病毒氨基酸序列的活性的朊病毒氨基酸序列。
如本文所用,术语“朊病毒亚基”是指朊病毒蛋白的最小氨基酸序列,其按照本发明融合至酸脱羧酶,并且与未融合至朊病毒亚基时的酸脱羧酶相比增加酸脱羧酶的活性。包含朊病毒亚基的最小氨基酸序列(和相应的多核苷酸序列)一般可以在朊病毒蛋白如ure2、sup35、new1或rnq1的n-末端区找到。在一个实施方案中,朊病毒亚基长度为至少30个氨基酸,长度为至少40个氨基酸,长度为至少50个氨基酸,长度为至少60个氨基酸,长度为至少70个氨基酸,长度为至少75个氨基酸,长度为至少100个氨基酸,长度为至少150个氨基酸,长度为至少200个氨基酸,或者长度为至少300个氨基酸,或者更多,但是长度为1200个氨基酸或更少。在一些实施方案中,朊病毒亚基长度为1000个氨基酸或更少。在一些实施方案中,朊病毒亚基长度为500个氨基酸或更少。在另一实施方案中,朊病毒亚基长度为30-200个氨基酸,长度为40-150个氨基酸,或者长度为50-120个氨基酸。与未融合至朊病毒亚基的酸脱羧酶相比,融合至酸脱羧酶的朊病毒亚基在应答应激条件如碱性ph或升高的温度时,提高酸脱羧酶的活性。
如本公开上下文所用,“酸脱羧酶”是指催化碱性氨基酸(例如赖氨酸、精氨酸、鸟氨酸、谷氨酸)的脱羧反应产生多胺的多肽。酸脱羧酶包括赖氨酸脱羧酶,例如cada、ldcd;精氨酸脱羧酶,例如adia;鸟氨酸脱羧酶,例如spec、spef;以及谷氨酸脱羧酶,例如gada、gadb;其是折叠型i吡哆醛5′-磷酸(plp)依赖性脱羧酶的原核鸟氨酸脱羧酶亚类的部分。这类蛋白通常包含n-末端翼结构域(n-terminalwingdomain)、核心结构域和c-末端结构域。核心结构域包含plp-结合亚结构域。酸脱羧酶spea也是plp依赖性脱羧酶,但是属于plp依赖性脱羧酶的不同折叠家族,其包含tim桶结构域、β-夹层、插入和c-末端结构域(forouhar,etal.,acta.cryst.f66,1562-1566,2010)。酸脱羧酶单体可以形成各种大小的多聚体,取决于酸脱羧酶。例如,酸脱羧酶cada、ldcc和adia形成双对称二聚体,完成每个单体的活性位点。5个二聚体关联形成十聚体,具有五重对称的双环状结构。十聚体可以与其他十聚体在有利的ph条件下关联形成高阶寡聚体。不是所有酸脱羧酶均形成十聚体以发挥功能。例如,酸脱羧酶gada和gadb形成六聚体,而spea形成四聚体。根据kanjeeetal.,2011,酸脱羧酶cada、ldcc、adia、spec和spef具有相同的结构折叠,并且至少作为同源二聚体存在。晶体结构分析显示gada和gadb还具有相同的plp依赖性酶的i型折叠,如cada、ldcc、adia、spec和spef(capitani,etal.,theembojournal22,4027-4037,2003)。ldcc十聚体和cada十聚体之间的相似性在kandia,etal.,sci.rep.6,24601,2016中描述。adia十聚体形成在boekerea&snellee,j.biol.chem.243,1678-1684,1968和andrell,etal.,biochemistry48,3915-3927,2009中描述。gada和gadb的结构描述在capitani,etal.,theembojournal22,4027-4037,2003中描述。说明性酸脱羧酶的结构的蛋白数据库id是:3n75(cada)、5fkz(ldccd)和2vyc(adia)。其他大肠杆菌酸脱羧酶如spec和spef形成同源二聚体。spea形成同源四聚体(pdbid:3nzq),而gada(pdbid:1xey)和gadb(pdbid:1pmm)形成同源六聚体。
术语“酸脱羧酶”涵盖本文所述特定多肽的生物活性变体、等位基因、突变体和种间同系物。编码酸脱羧酶的核酸是指基因、pre-mrna、mrna等,包括编码本文所述特定氨基酸序列的变体、等位基因、突变体和种间同系物的核酸。
如本文所用的“酸脱羧酶融合多肽”是指包含融合至朊病毒亚基的酸脱羧酶的多肽。“酸脱羧酶融合多核苷酸”或“酸脱羧酶融合基因”是指编码酸脱羧酶融合多肽的核酸。
赖氨酸脱羧酶是指将l-赖氨酸转化为尸胺的酶。该酶分类为e.c.4.1.1.18。赖氨酸脱羧酶多肽是良好表征的酶,其结构是本领域公知的(参见例如kanjee,etal.,emboj.30:931-944,2011;以及lemmonier&lane,microbiology144;751-760,1998的综述;以及其中描述的参考文献)。说明性赖氨酸脱羧酶序列是cada同系物,来自克雷伯氏菌属(klebsiellasp.),wp012968785.1;产气肠杆菌(enterobacteraerogenes),yp004592843.1;肠道沙门氏菌(salmonellaenterica),wp020936842.1;沙雷氏菌属(serratiasp.),wp033635725.1;和解鸟氨酸拉乌尔菌(raoultellaornithinolytica),yp007874766.1;以及ldcc同系物,来自志贺氏菌属(shigellasp.),wp001020968.1;柠檬酸杆菌属(citrobactersp.),wp016151770.1;和肠道沙门氏菌,wp001021062.1。如本文所用,赖氨酸脱羧酶包括具有赖氨酸脱羧酶酶促活性的天然赖氨酸脱羧酶的变体。pct/cn2014/080873和pct/cn2015/072978中描述了额外的赖氨酸脱羧酶。
“cada”多肽是指具有seqidno:2的氨基酸序列的大肠杆菌cada多肽,或者其具有酸脱羧酶活性的生物活性变体。生物活性变体包括大肠杆菌cada多肽的等位基因、突变体和种间同系物。cada包含n-末端翼结构域、核心结构域和c-末端结构域。来自其他物种的说明性cada多肽包括肠道沙门氏菌,蛋白序列登录号wp001021062.1。在一些实施方案中,“cada”多肽优选与seqidno:2的cada多肽的至少约50、100或更多个氨基酸的区域或者全长具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。如本文所用的“cada多核苷酸”是指编码cada多肽的多核苷酸。
如本文所用,术语“碱性ph”是指具有大于7.5的ph的溶液或周围环境。在一个实施方案中,碱性ph是指具有至少8.0或至少8.5、或者更高的ph的溶液或周围环境。
如本文所用,术语“升高的温度”或“高温”是指约35℃或更高的温度。在一些实施方案中,较高的温度是至少37℃、至少40℃、至少42℃、至少45℃、至少48℃、至少50℃、至少52℃、至少55℃或更高。在一个实施方案中,升高的温度是指至少42℃但低于60℃的温度。
如本文所用,在氨基酸例如赖氨酸或者赖氨酸衍生物例如尸胺生成的上下文中,术语“增强”或“提高”是指与对照对应细胞相比,如野生型菌株的细胞或者表达酸脱羧酶蛋白但不融合至朊病毒亚基的相同菌株的细胞,宿主细胞产生的氨基酸或氨基酸衍生物的生成增加,所述宿主细胞表达酸脱羧酶融合多肽,其包含(例如在酸脱羧酶多肽的羧基端)融合至朊病毒亚基的酸脱羧酶多肽。在一个实施方案中,酸脱羧酶融合蛋白(例如其中朊病毒亚基融合至酸脱羧酶的羧基端)的酸脱羧酶活性与表达缺少朊病毒亚基的酸脱羧酶的对应细胞的酸脱羧酶活性相比,提高至少5%,通常至少10%、15%、20%、30%、40%、50%、60%、70%、80%、90%、100%或更大,其中活性通过测量在相同条件下宿主细胞和对照细胞产生的多胺如尸胺以及赖氨酸的生产来评价。在一些实施方案中,在升高的温度下测量来自宿主细胞培养物的裂解提取物的活性。在一些实施方案中,在碱性条件下测量来自宿主细胞培养物的裂解提取物的活性。例如,可以通过与来自表达未融合至朊病毒亚基的酸脱羧酶的相同菌株的对应宿主细胞的培养物的相应等分试样相比,评价用酸脱羧酶融合多肽转化的宿主细胞的培养物的等分试样,从而评价本发明的酸脱羧酶融合多肽的活性。举例而言,与未融合至朊病毒亚基的对应赖氨酸脱羧酶相比,本发明的赖氨酸脱羧酶融合多肽的活性可以通过评价裂解样品的反应速率确定,例如在8.0的ph在100ml样品中。可以利用nmr测量反应速率,通过在总计20分钟中约每1.5分钟采样在plp的存在下转化为尸胺的赖氨酸的量并取收率曲线的线性部分的斜率进行。将样品稀释,从而在ph6.0和35℃测量的裂解样品的反应速率/体积(u)相同。在8的初始ph利用相同的u测量每个裂解样品的赖氨酸的动力学常数vmax和km。通过对u归一化,每个样品中的活性酶的浓度相同。
当在给定氨基酸或多核苷酸序列编号的上下文中使用时,术语“参考编号”或“对应于”或“参考确定”是指当给定氨基酸或多核苷酸序列与参考序列相比时,指定参考序列残基的编号。例如,当片段在最大比对中与seqidno:4比对时,朊病毒亚基多肽序列的片段“对应于”seqidno:4中的片段。
术语“多核苷酸”和“核酸”可交换使用,是指从5'至3'端阅读的脱氧核糖核苷酸或核糖核苷酸碱基的单链或双链聚合物。如本发明所用的核酸一般包含磷酸二酯键,虽然在某些情况下,可以使用核酸类似物,其可以具有替代骨架,包含例如氨基磷酸酯、硫代磷酸酯、二硫代磷酸酯或o-甲基亚磷酰胺酯键(参见eckstein,oligonucleotidesandanalogues:apracticalapproach,oxforduniversitypress);正骨架;非离子骨架,以及非核糖骨架。核酸或多核苷酸还可以包括允许聚合酶正确通读的修饰的核苷酸。“多核苷酸序列”或“核酸序列”包括作为单独单链或双链体的核酸的正义和反义链。本领域技术人员会理解,单链的描述还定义互补链的序列;因此本文描述的序列还提供序列的补体。除非另有说明,特定核酸序列还暗中涵盖其变体(例如简并密码子取代)和互补序列以及明确指明的序列。核酸可以是dna,基因组和cdna,rna或杂合体,其中核酸可以包含脱氧核糖核苷酸和核糖核苷酸的组合,以及碱基的组合,包括尿嘧啶、腺嘌呤、胸腺嘧啶、胞嘧啶、鸟嘌呤、肌苷、黄嘌呤、次黄嘌呤、异胞嘧啶、异鸟嘌呤等。
在两个核酸或多肽的上下文中使用的术语“基本上相同”是指与参考序列具有至少40%、45%或50%序列相同性的序列。百分比相同性可以是50%-100%的任何整数。一些实施方案包括利用本文描述的程序与参考序列相比至少:50%、55%、60%、65%、70%、75%、80%、85%、90%、91%、92%、93%、94%、95%、96%、97%、98%或99%,优选使用标准参数的blast,如下文所述。
如果在如下文所述在最大对应比对时两个序列中的核苷酸或氨基酸残基的序列分别是相同的,则说两个核酸序列或多肽序列是“相同的”。在两个或更多个核酸或多肽序列的上下文中,术语“相同”或百分比“相同性”是指如利用以下序列比较算法之一或者通过手动比对和目视检查测量的,在相对比较窗进行最大对应性比较和比对时,相同或者具有特定百分比的相同氨基酸残基或核苷酸的两个或更多个序列或子序列。当序列相同性的百分比用于蛋白或肽时,应当认识到不相同的残基位置通常由于保守氨基酸取代而不同,其中氨基酸残基被取代为具有相似化学特性(例如电荷或疏水性)的其他氨基酸残基,因此不改变分子的功能特性。当序列的不同之处在于保守取代时,可以将百分比序列相同性上调以对取代的保守性质进行修正。进行这种调整的方法是本领域技术人员熟知的。通常这包括将保守取代评分为部分而不是完全错配,从而增加百分比序列相同性。因此,例如,当相同氨基酸给出1的评分并且非保守取代给出0的评分时,保守取代给出在0和1之间的评分。
对于序列比较,通常一个序列用作参考序列,测试序列与其进行比较。当利用序列比较算法时,将测试和参考序列输入计算机,如果必要,指定子序列坐标,并且指定序列算法程序参数。可以使用默认程序参数,或者可以指定可选参数。然后序列比较算法基于程序参数计算测试序列相对于参考序列的百分比序列相同性。
可以用来确定酸脱羧酶融合多肽是否与序列如seqidno:2;或者seqidno:21-28任一或另一多肽参考序列具有序列相同性的一种算法是blast算法,其在altschuletal.,1990,j.mol.biol.215:403-410中描述,其并入本文参考。进行blast分析的软件可通过美国国家生物技术信息中心公开获得(在ncbi.nlm.nih.gov/的万维网上)。对于氨基酸序列,blastp程序使用3的字长(w)、10的预期值(e)和blosum62评分矩阵作为默认值(参见henikoff&henikoff,1989,proc.natl.acad.sci.usa89:10915)。可以使用的其他程序包括needleman-wunsch方法,j.moi.biol.48:443-453(1970),利用blosum62、7的缺口起始罚分和1的缺口延伸罚分;以及缺口blast2.0(参见altschul,etal.1997,nucleicacidsres.,25:3389-3402)。
如本文所用,“比较窗”包括引用选自20-600,通常约50-约200,更通常约100-约150的连续位置数量的任一个的片段,其中可以在将两个序列最佳比对之后,将序列与相同连续位置数量的参考序列进行比较。用于比较的序列的比对方法是本领域熟知的。用于比较的序列的最佳比对可以这样进行,例如通过smith&waterman,adv.appl.math.2:482(1981)的局部同源性算法,通过needleman&wunsch,j.mol.biol.48:443(1970)的同源性比对算法,通过pearson&lipman,proc.nat'l.acad.sci.usa85:2444(1988)的搜索相似性方法,通过这些算法的计算机化实施(wisconsingeneticssoftwarepackage,geneticscomputergroup,575sciencedr.,madison,wi中的gap、bestfit、fasta和tfasta),或者通过手动比对和目视检查。
与参考序列基本上相同的核酸或蛋白序列包括“保守修饰的变体”。关于特定核酸序列,保守修饰的变体是指编码相同或基本上相同的氨基酸序列的那些核酸,或者当核酸不编码氨基酸序列时,是指基本上相同的序列。因为遗传密码的简并性,大量功能相同的核酸编码任何给定蛋白。例如,密码子gca、gcc、gcg和gcu全部编码氨基酸丙氨酸。因此,在通过密码子指定丙氨酸的每个位置,密码子可以改变为所述的任何相应密码子而不改变编码的多肽。这类核酸变化为“沉默变化”,其为保守修饰的变化的一个种类。本文中编码多肽的每个核酸序列还描述核酸的每个可能的沉默变化。技术人员会认识到可以修饰核酸中的每个密码子(除了aug,其通常是甲硫氨酸的唯一密码子)以获得功能上相同的分子。因此,编码多肽的核酸的每个沉默变化隐含在每个所述序列中。
如本文所用的术语“多肽”包括含有天然存在的氨基酸和氨基酸骨架以及非天然存在的氨基酸和氨基酸类似物的多肽。
对于氨基酸序列,技术人员会认识到核酸、肽、多肽或蛋白序列中改变编码序列中单一氨基酸或一小部分氨基酸的各个取代(其中改变导致一个氨基酸用化学上相似的氨基酸取代)是“保守修饰的变体”。提供功能上相似的氨基酸的保守取代表是本领域熟知的。以这种方式定义的氨基酸组的实例可以包括:“带电荷/极性组”,包括glu(谷氨酸或e)、asp(天冬氨酸或d)、asn(天冬酰胺或n)、gln(谷氨酰胺或q)、lys(赖氨酸或k)、arg(精氨酸或r)和his(组氨酸或h);“芳香或环状组”,包括pro(脯氨酸或p)、phe(苯丙氨酸或f)、tyr(酪氨酸或y)和trp(色氨酸或w);以及“脂肪族组”,包括gly(甘氨酸或g)、ala(丙氨酸或a)、val(缬氨酸或v)、leu(亮氨酸或l)、ile(异亮氨酸或i)、met(甲硫氨酸或m)、ser(丝氨酸或s)、thr(苏氨酸或t)和cys(半胱氨酸或c)。在每组内,还可以鉴定亚组。例如,带电荷/极性氨基酸的组可以细分为亚组,包括:“带正电荷的亚组”,包含lys、arg和his;“带负电荷的亚组”,包含glu和asp;以及“极性亚组”,包含asn和gln。在另一实例中,芳香或环状组可以细分为亚组,包括:“氮环亚组”,包含pro、his和trp;以及“苯基亚组”,包含phe和tyr。在另一实例中,脂肪族组可以细分为亚组,包括:“大脂肪族非极性亚组”,包含val、leu和ile;“脂肪族微极性亚组”,包含met、ser、thr和cys;以及“小残基亚组”,包含gly和ala。保守突变的实例包括上述亚组内氨基酸的氨基酸取代,例如但不限于:lys取代arg或反之亦然,从而可以保持正电荷;glu取代asp或反之亦然,从而可以保持负电荷;ser取代thr或反之亦然,从而可以保持游离的--oh;以及gln取代asn或反义亦然,从而可以保持游离的--nh2。以下6组每个包含互相进一步提供说明性保守取代的氨基酸。1)ala、ser、thr;2)asp、glu;3)asn、gln;4)arg、lys;5)ile、leu、met、val;以及6)phe、try和trp(参见例如creighton,proteins(1984))。
如本文所用,术语“启动子”是指能够驱动细胞中dna序列转录的多核苷酸序列。因此,本发明的多核苷酸构建体中使用的启动子包括顺式和反式作用转录控制元件和调制序列,参与调节或调控基因转录的时间和/或速率。例如,启动子可以是顺式作用转录控制元件,包括增强子、阻遏物结合序列等。这些顺式作用序列通常与蛋白或其他生物分子相互作用以进行(开启/关闭、调节、调控等)基因转录。核心启动子序列通常在翻译起始位点的1-2kb内,更常在1kbp内,并且常在翻译起始位点的500bp或200bp或更少内。按照惯例,启动子序列通常作为其控制的基因的编码链上的序列提供。在本申请的上下文中,启动子通常通过其天然调节表达的基因的名称来表示。本发明的表达构建体中使用的启动子通过基因名称命名。通过名称引用启动子包括野生型、天然启动子以及保留诱导表达能力的启动子的变体。通过名称引用启动子并不限于特定物种,而是还涵盖来自其他物种中的相应基因的启动子。
在本发明的上下文中,“组成型启动子”是指能够在细胞中在大多数条件下,例如在诱导分子不存在下起始转录的启动子。“诱导型启动子”在诱导分子的存在下起始转录。
多核苷酸对于一个生物体或第二多核苷酸序列而言,如果其源自外来物种或者源自相同物种但是从其原来形式加以修饰,则其与所述生物体或第二多核苷酸序列是“异源的”。例如,当编码多肽序列的多核苷酸据说可操作地连接至异源启动子时,这表示编码多肽的多核苷酸编码序列源自一个物种,而启动子序列源自另一不同物种;或者,如果两者源自相同物种,则编码序列与启动子不是天然相关的(例如,是遗传工程化的编码序列,例如来自相同物种的不同基因,或者来自不同生态型或品种的等位基因)。相似地,如果天然野生型宿主细胞不产生多肽,则所述多肽对于宿主细胞是“异源的”。
术语“外源”一般是指不天然存在于野生型细胞或生物体中的多核苷酸序列或多肽,但是通常通过分子生物学技术引入细胞,即工程化以产生重组微生物。“外源”多核苷酸的实例包括载体、质粒和/或编码期望的蛋白或酶的人造核酸构建体。
术语“内源”是指可以在给定野生型细胞或生物体中找到的天然存在的多核苷酸序列或多肽。在这方面,还注意到尽管生物体可以包含给定多核苷酸序列或基因的内源拷贝,但是引入编码该序列的质粒或载体如用于过表达或另外调节所编码的蛋白的表达,表示该基因或多核苷酸序列的“外源”拷贝。本文描述的任何途径、基因或酶可以利用或依赖“内源”序列,其可以一个或多个“外源”多核苷酸序列提供,或者两者。
如本文所用的“重组核酸”或“重组多核苷酸”是指核酸的聚合物,其中以下至少一个是正确的:(a)核酸的序列对于给定宿主细胞是外源的(即未天然发现);(b)序列可以在给定宿主细胞中天然发现,但是不是天然(例如大于预期)的量;或者(c)核酸的序列包含两个或更多个子序列,其在天然中未发现彼此处于相同关系中。例如,关于情况(c),重组核酸序列具有来自无关基因的两个或更多个序列排列以产生新的功能核酸。
术语“可操作地连接”是指两个或更多个多核苷酸(例如dna)片段之间的功能关系。通常,其指转录调节序列与转录的序列的功能关系。例如,如果其刺激或调节适当宿主细胞或其他表达系统中的dna或rna序列的转录,则启动子或增强子序列是可操作地连接至dna或rna序列。一般来说,可操作地连接至转录序列的启动子转录调节序列与转录序列在物理上是连续的,即它们是顺式作用的。但是,一些转录调节序列如增强子不必与其增强转录的编码序列在物理上连续或靠近。
术语“表达盒”或“dna构建体”或“表达构建体”是指核酸构建体,当引入宿主细胞时,其分别导致rna或多肽的转录和/或翻译。在转基因表达的情况下,技术人员会认识到插入的多核苷酸序列不必相同,但是可以仅与其起源的基因序列基本上相同。如本文解释的,参考特定核酸序列特别涵盖这些基本上相同的变体。表达盒的一个实例是多核苷酸构建体,其包含编码本发明的多肽的多核苷酸序列,其可操作地连接至启动子,例如其天然启动子,其中将所述表达盒引入异源微生物。在一些实施方案中,表达盒包含编码本发明的多肽的多核苷酸序列,其中所述多核苷酸靶向微生物基因组中的位置,从而多核苷酸序列的表达通过微生物中存在的启动子驱动。
如本发明的上下文中使用的术语“宿主细胞”是指微生物并且包括可以是或已经是本发明的任何重组载体或分离的多核苷酸的受体的各个细胞或细胞培养物。宿主细胞包括单个宿主细胞的子代,并且由于天然、偶然或人为的突变和/或改变,所述子代可以不必与原始的亲代细胞(在形态学上或在总dna互补上)完全相同。宿主细胞包括已引入本发明的重组载体或多核苷酸的细胞,包括通过转化、转染等。
术语“分离的”是指基本上或根本上不含在其天然状态下通常伴随它的组分的物质。例如,如本文所用,“分离的多核苷酸”可以指分离自在其天然存在或基因组状态下在其侧翼的序列的多核苷酸,例如已从通常与dna片段相邻的序列移出的所述片段,如通过克隆至载体中。例如,如果克隆至不是天然环境的一部分的载体中,或者如果以不同于其天然存在的状态的方式人工引入细胞的基因组,则认为多核苷酸是分离的。或者,如本文所用,“分离的肽”或“分离的多肽”等是指不含细胞的其他组分的多肽分子,即其与体内物质不相关。
本发明采用各种常规重组核酸技术。一般来说,下文描述的重组dna技术中的命名法和实验室程序常用于本领域。为进行重组dna操作提供指导的许多手册是可用的,例如sambrook&russell,molecularcloning,alaboratorymanual(3rded,2001);和currentprotocolsinmolecularbiology(ausubel,etal.,johnwileyandsons,newyork,2009-2016)。
发明概述
本公开部分基于这样的发现,如在各种应激条件如碱性ph或升高的温度下通过多胺生成测量的,与表达缺少朊病毒亚基的酸脱羧酶相比,将朊病毒亚基例如在羧基末端融合至酸脱羧酶以产生酸脱羧酶融合蛋白并在宿主细胞中表达该融合蛋白提高了酸脱羧酶融合蛋白的活性。
此外,本发明部分基于这样的发现,如在升高的温度或碱性ph下通过融合蛋白的寡聚化测量的,当与缺少朊病毒亚基的对应酸脱羧酶相比时,将朊病毒亚基例如在羧基末端融合至酸脱羧酶以产生融合蛋白增加了酸脱羧酶的稳定性。
此外,本公开部分基于这样的发现,将朊病毒亚基例如在羧基末端融合至酸脱羧酶以产生融合蛋白增加了酸脱羧酶在升高的温度或碱性ph的溶解度。因此,如在碱性ph下通过酸脱羧酶活性测量的,本公开的融合蛋白相对于缺少相应朊病毒亚基的酸脱羧酶蛋白通常具有提高的溶解度。
本发明的酸脱羧酶融合蛋白耐受碱性ph的能力还允许在发酵反应中使用具有较高ph值的替代氮源,如尿素和氨(1m溶液具有ph11.6),以产生期望产物,例如多胺。这些替代氮源产生较少的盐废物副产物。
朊病毒亚基
朊病毒是自我增殖和可传染的蛋白同种型。具有改变的构象的正常细胞蛋白可以是传染性的,导致疾病。虽然在酵母中没有蛋白与prpc具有同源性,但是已鉴定几种酵母朊病毒蛋白。
首先在酵母中鉴定的朊病毒[psi+]和[ure3]分别确定为ure2和sup35蛋白的朊病毒形式。从那时开始,在酿酒酵母(saccharomycescerevisiae)中已鉴定其他酵母朊病毒,包括[pin+]/[rnq+]、[swi+]、[oct+]、[mot+]、[isp+]、[beta]、[mod+]以及podosporaanserina中鉴定的真菌朊病毒[het-s](参见wickneretal.,microbiologyandmolecularbiologyreviews,2015)。
在一些实施方案中,朊病毒亚基可以定义为朊病毒多肽或其片段,其中天冬酰胺(n)和谷氨酰胺(q)的百分比组成为10%或更大。例如,sup35朊病毒亚基中q/n的百分比是154个氨基酸的25%,并且new1的是253个氨基酸的27%。参考认为具有朊病毒活性的融合多肽的部分,确定10%,即仅在朊病毒亚基序列的背景下考虑确定的。
适合作为朊病毒亚基用于本发明的朊病毒多肽序列包括如seqidno:3或4中说明的朊病毒多肽的氨基酸序列,或者其基本上相同的序列变体。这样的序列变体通常与seqidno:3或4或者例如seqidno:3或4的同系物之一具有至少50%,或者至少60%、70%、75%、80%、85%或90%相同性。在一些实施方案中,朊病毒亚基包含seqidno:7、8、11、12、13或14的朊病毒区的氨基酸序列;或者与seqidno:7、8、11、12、13或14的朊病毒区具有至少50%,或者具有至少60%、70%、75%、80%、85%或90%相同性。如本文所用,术语“序列变体”涵盖相对于朊病毒多肽参考序列如seqidno:3或4具有一个或多个取代、缺失或插入的生物活性多肽。因此,术语“序列变体”包括生物活性片段以及取代变体。
在一个实施方案中,适合用于本发明的朊病毒亚基多肽序列包括编码ure2、sup35、new1、rnq1、swi1、cyc8、mot3或sfp1或者其基本上相同的变体的氨基酸序列。ure2多肽的说明性实例包括来自酿酒酵母蛋白序列登录号aam93184;白色假丝酵母(candidaalbicans)蛋白序列登录号aam91946;巴扬氏糖酵母(s.bayanus)蛋白序列登录号aam91939;以及eremotheciumgossypii蛋白序列登录号aam91943的那些。sup35多肽的说明性实例包括来自酿酒酵母蛋白序列登录号ajv18122;鲍氏酵母菌(s.boulardii)蛋白序列登录号koh51638;以及巴扬氏糖酵母蛋白序列登录号aal15027的那些。new1多肽的说明性实例包括来自酿酒酵母蛋白序列登录号ahy77957;鲍氏酵母菌蛋白序列登录号koh47591;以及sugiyamellalignohabitans蛋白序列登录号anb11767的那些。rnq1多肽的说明性实例包括来自酿酒酵母蛋白序列登录号afu61310;以及鲍氏酵母菌蛋白序列登录号koh52602的那些。swi1多肽的说明性实例包括来自酿酒酵母蛋白序列登录号ajp42124;白色假丝酵母蛋白序列登录号aow28823;以及sugiyamellalignohabitans蛋白序列登录号anb13699的那些。cyc8多肽的说明性实例包括来自酿酒酵母蛋白序列登录号caa85069;以及aspergillusnomius蛋白序列登录号kng81485的那些。mot3多肽的说明性实例包括来自酿酒酵母蛋白序列登录号aac49982;以及鲍氏酵母菌蛋白序列登录号kqc41827的那些。sfp1多肽的说明性实例包括来自酿酒酵母蛋白序列登录号aab82343;鲍氏酵母菌蛋白序列登录号koh49283;以及s.arboricola蛋白序列登录号ejs42621的那些。在一个实施方案中,适合用于本发明的多肽序列包括编码能够在体内或体外诱导蛋白寡聚化的朊病毒多肽的氨基酸序列。
在一些实施方案中,适合用于本发明的多核苷酸序列包括编码以下朊病毒蛋白中的一种或多种的核酸序列:ure2、sup35、new1、rnq1、swi1、cyc8、mot3或sfp1或者它们的同系物。此外,用于本发明的合适的多核苷酸包括编码本文提供的任一种说明性朊病毒多肽的核酸序列。在另一实施方案中,合适的多核苷酸包括编码本文公开的能够在体内或体外诱导蛋白寡聚化的说明性朊病毒多肽中的任一种或多种的核酸序列。在一个实施方案中,适合用于本发明的多核苷酸序列包括编码如seqno:5或6中说明的朊病毒多肽或者其基本上相同的变体的核酸序列。这样的变体通常与seqidno:5或6之一具有至少60%,或者至少70%、75%、80%、85%或90%相同性。
在一个实施方案中,本发明涉及一种具有编码酸脱羧酶融合蛋白的核酸序列的遗传修饰的宿主细胞,其中所述酸脱羧酶融合蛋白包含或由例如在酸脱羧酶多肽的羧基末端连接至朊病毒亚基的酸脱羧酶多肽组成,并且其中如通过多胺生成测量的,所述融合蛋白相对于表达未连接至所述朊病毒亚基的酸脱羧酶多肽的对应宿主细胞具有提高的酸脱羧酶活性。在一个实施方案中,朊病毒亚基长度为至少30个氨基酸,长度为至少50个氨基酸,长度为至少75个氨基酸或长度为至少100个氨基酸,但是长度为1200个氨基酸或更少。在一些实施方案中,朊病毒亚基包含具有至少20%q或n残基的氨基酸组成。在另一实施方案中,朊病毒亚基与sup35、new1、ure2或rnq1氨基酸序列具有至少70%、75%、80%、85%、90%或95%相同性;或者包含sup35、new1、ure2或rnq1氨基酸序列。
朊病毒的结构组织
一般来说,酵母朊病毒在溶液中本质上是无序的,并且富含qn。据发现保持氨基酸组成而不是精确序列的sup35和ure2的乱序prd能够在体外产生淀粉样蛋白和在体内产生朊病毒,并且能够传播朊病毒状态,表明氨基酸组成在朊病毒特性中起重要作用(rossetal.,2005)。因此,作为朊病毒亚基用于本发明的酵母朊病毒序列的变体包括sup35或ure2的片段,其与sup35和ure2具有少于50%序列相同性,但是具有10%或更大的qn组成。
sup35
酵母蛋白sup35(685aa)是翻译终止因子的亚基并在终止密码子终止翻译,已观察到残基254-685(sup35c)足以实现基本的翻译终止功能。据观察残基1-253(sup35nm)通过与poly(a)–结合蛋白和poly(a)-降解酶相互作用调节一般mrna周转。残基1-114(sup35n)足以传播原始[psi+]变体,而残基1-61足以传播这种朊病毒的几种变体(changetal.,pnas,2008)。sup35的n-近端prd区包括位于前40个氨基酸内的n-末端qn富集区,以及位于位置41和97的5.5不完全寡肽重复(or)区域。聚集需要的prd片段比传播朊病毒状态需要的片段短,并且主要局限于qn富集区(osherovichetal,2004)。
据观察长达残基137的sup35m结构域(残基115-253)的部分是传播一些强和弱[psi+]变体必需的,并且据观察m结构域内的缺失和取代显著改变[psi+]变体的特征(liuetal.,pnas,2002)。sup35nm细丝的固态核磁共振(ss-nmr)实验显示全部在n末端内的酪氨酸(tyr)残基为对齐的平行β-折叠结构(shewmaker,pnas,2006)。此外,据观察有8个亮氨酸(leu)残基,即残基110、126、144、146、154、212、218和238。ss-nmr数据表明这些leu残基中的4个为对齐的平行结构(shewmakeretal.,biochemistry,2009)。
ure2
酵母蛋白ure2(354aa)的作用是调节氮分解代谢。据发现其生产过剩诱导[ure3]形成的ure2部分是n-末端65个残基,并且证实这个区域足以在分子的剩余部分不存在下传播[ure3](masisonetal.,science,1997)。n-末端朊病毒结构域通常功能是稳定ure2抗降解(shewmakeretal.,genetics,2007)。
rnq1
在酵母蛋白rnq1(405aa)的情况下,在prd内发现4个qn富集区(kadnaretal.,2010)。虽然没有对于朊病毒传播是必要的,但是发现4段中的2段如果单独保留各自支持朊病毒维持。
new1
酵母蛋白new1(1196aa)由n-末端朊病毒区(new1n)和与具有两个atp-结合基序的翻译延长因子同源的c-末端区组成。
一般来说,能够诱导蛋白聚集的朊病毒需要谷氨酰胺(q)和/或天冬酰胺(n)或者(nq)富集区。例如,朊病毒蛋白new1包含序列“qqqrnwkqggnyqqyqsyn”和“snynnynnynnynnynnynnynkyngqgyq”。在朊病毒蛋白sup35中,n-末端包含nq富集区,然后是n结构域重复(nr)区,其包含5个完整拷贝(r1-r5)和一个部分拷贝(r6)的不完全寡肽重复序列“pqggyqqn”。
在一个实施方案中,朊病毒亚基可以包含或由编码朊病毒蛋白的核酸组成,所述朊病毒蛋白选自ure2、sup35、new1、rnq1、swi1、cyc8、mot3和sfp1。在另一实施方案中,朊病毒亚基可以包含或由编码源自ure2、sup35、new1、rnq1、swi1、cyc8、mot3或sfp1的多肽的核酸组成,所述多肽能够在体内或体外诱导蛋白寡聚化。在另一实施方案中,朊病毒亚基可以由以下组成或包含:至少30个氨基酸、至少40个氨基酸、至少50个氨基酸、至少60个氨基酸、至少70个氨基酸、至少80个氨基酸、至少90个氨基酸、至少100个氨基酸、至少200个氨基酸、至少300个氨基酸但少于1200个氨基酸的氨基酸序列,当融合至酸脱羧酶的羧基端时,在相同条件下与缺少朊病毒亚基的对应酸脱羧酶相比,其提高碱性ph和/或升高的温度下酸脱羧酶的脱羧作用。在一个实施方案中,通过酸脱羧酶产生的多胺测量脱羧作用。
在一个实施方案中,朊病毒亚基可以由以下组成或包含:至少30个氨基酸、至少40个氨基酸、至少50个氨基酸、至少60个氨基酸、至少70个氨基酸、至少80个氨基酸、至少90个氨基酸、至少100个氨基酸、至少200个氨基酸、至少300个氨基酸但少于1200个氨基酸的氨基酸序列,当融合至赖氨酸脱羧酶的羧基端时,在相同条件下与缺少朊病毒亚基的对应赖氨酸脱羧酶相比,其提高碱性ph和/或升高的温度下赖氨酸脱羧酶的脱羧作用。在一个实施方案中,通过赖氨酸脱羧酶产生的尸胺测量脱羧作用。
在一个实施方案中,朊病毒亚基可以由以下组成或包含:至少30个氨基酸、至少40个氨基酸、至少50个氨基酸、至少60个氨基酸、至少70个氨基酸、至少80个氨基酸、至少90个氨基酸、至少100个氨基酸、至少200个氨基酸、至少300个氨基酸但少于1200个氨基酸的氨基酸序列,当融合至精氨酸脱羧酶的羧基端时,在相同条件下与缺少朊病毒亚基的对应精氨酸脱羧酶相比,其提高碱性ph和/或升高的温度下精氨酸脱羧酶的脱羧作用。在一个实施方案中,通过精氨酸脱羧酶产生的腐胺测量脱羧作用。
在一个实施方案中,朊病毒亚基可以由以下组成或包含:至少30个氨基酸、至少40个氨基酸、至少50个氨基酸、至少60个氨基酸、至少70个氨基酸、至少80个氨基酸、至少90个氨基酸、至少100个氨基酸、至少200个氨基酸、至少300个氨基酸但少于1200个氨基酸的氨基酸序列,当融合至鸟氨酸脱羧酶的羧基端时,在相同条件下与缺少朊病毒亚基的对应鸟氨酸脱羧酶相比,其提高碱性ph和/或升高的温度下鸟氨酸脱羧酶的脱羧作用。在一个实施方案中,通过鸟氨酸脱羧酶产生的精胺测量脱羧作用。
在一个实施方案中,朊病毒亚基可以由以下组成或包含:至少30个氨基酸、至少40个氨基酸、至少50个氨基酸、至少60个氨基酸、至少70个氨基酸、至少80个氨基酸、至少90个氨基酸、至少100个氨基酸、至少200个氨基酸、至少300个氨基酸但少于1200个氨基酸的氨基酸序列,当融合至谷氨酸脱羧酶的羧基端时,在相同条件下与缺少朊病毒亚基的对应谷氨酸脱羧酶相比,其提高碱性ph和/或升高的温度下谷氨酸脱羧酶的脱羧作用。在一个实施方案中,通过谷氨酸脱羧酶产生的γ-氨基丁酸(gaba)测量脱羧作用。
朊病毒亚基具有至少10%q和n残基,并且通常至少20%、30%、40%、50%或更多q和n残基。在另一实施方案中,朊病毒亚基具有至少10%q残基,并且通常至少20%、30%、40%、50%或更多q残基。在另一实施方案中,朊病毒亚基具有至少10%n残基,并且通常至少20%、30%、40%、50%或更多n残基。在一个实施方案中,与q残基相比,朊病毒亚基包含较高百分比的n残基。在另一实施方案中,与n残基相比,朊病毒亚基包含较高百分比的q残基。在另一实施方案中,融合蛋白中存在的q和n残基的百分比是这样的,能够引起蛋白寡聚化的朊病毒亚基包含至少10%q或n残基。
在一个实施方案中,融合蛋白的朊病毒亚基可以包含或由编码朊病毒蛋白的核酸序列组成,所述朊病毒蛋白选自ure2、sup35、new1、rnq1、swi1、cyc8、mot3和sfp1。在另一实施方案中,朊病毒亚基可以包含或由编码源自ure2、sup35、new1、rnq1、swi1、cyc8、mot3或sfp1的多肽的核酸序列组成,所述多肽能够在体内或体外诱导蛋白寡聚化。在另一实施方案中,朊病毒亚基可以由以下组成或包含:至少30个氨基酸、至少40个氨基酸、至少50个氨基酸、至少60个氨基酸、至少70个氨基酸、至少80个氨基酸、至少90个氨基酸、至少100个氨基酸、至少200个氨基酸、至少300个氨基酸但少于1200个氨基酸的氨基酸序列,当融合至酸脱羧酶的羧基端时,在相同条件下与缺少朊病毒亚基的对应酸脱羧酶相比,其增加碱性ph和/或升高的温度下酸脱羧酶的脱羧作用。在一个实施方案中,通过酸脱羧酶产生的多胺测量脱羧作用。
在另一实施方案中,朊病毒亚基代表提高酸脱羧酶活性必需的最小需要的氨基酸序列(和相应的多核苷酸序列),如通过融合蛋白生成的多胺测量的。在一个实施方案中,朊病毒亚基长度可以为至少30个氨基酸,或者至少40、50、60、70、80、90、100、200、300或更多个氨基酸,但是长度少于1200个氨基酸。
在一个实施方案中,朊病毒亚基可以连接至赋予稳定性的另一短氨基酸序列。在一个实施方案中,短肽选自bst片段、reca片段和λci片段。在另一实施方案中,接头多肽可以包含氨基酸序列rrfgeassaf、asqwpeetfg或egvaetnedf。
在一个实施方案中,朊病毒亚基代表相对于缺少朊病毒亚基的酸脱羧酶提高酸脱羧酶活性必需的最小需要的氨基酸序列(和相应的多核苷酸序列),如通过融合蛋白生成的多胺测量的。在一个实施方案中,朊病毒亚基长度可以为至少30个氨基酸,或者至少40、50、60、70、80、90、100、200、300或更多个氨基酸,但是长度少于1200个氨基酸。
在一个实施方案中,朊病毒亚基能够诱导蛋白聚集(寡聚化)并且朊病毒亚基形成β-折叠结构,如对齐的平行β-折叠结构。在一个实施方案中,朊病毒亚基可能能够诱导融合至朊病毒亚基的一个或多个蛋白单体的蛋白聚集,并且朊病毒亚基形成β-螺旋结构。
朊病毒亚基可以在酸脱羧酶的n-末端,c-末端融合至酸脱羧酶,或者可以在酸脱羧酶蛋白的表面区引入。在某些实施方案中,朊病毒亚基融合在酸脱羧酶的c-末端。朊病毒亚基通常通过接头连接至酸脱羧酶,如包含氨基酸如gly、ser、ala等的柔性接头。
酸脱羧酶
多种酸脱羧酶活性已在结构和功能上良好表征。这些包括cada、ldcc、adia、spea、spec、spef、gada、gadb和它们的同系物。cada的最佳ph是5-6,ldcc是7-8,adia是4.5-5.5,spec是7.5-8.5,并且spef是7-8(kanjeeetal.,biochemistry50,9388-9398,2011)。当环境的ph为2-2.5时,gada和gadb被激活(castanie-cornetetal.,j.bacteriol.181,3525-3535,1999)。但是,碱性氨基酸赖氨酸、精氨酸、鸟氨酸和谷氨酸的脱羧作用导致尸胺、腐胺、精胺和gaba的产生;并且它们的形成涉及质子的消耗。这些是碱性分子,倾向于增加培养基的ph。例如,尸胺的pka是9.1和10.2,并且腐胺的是9.7和11.2。因此,这些碱性分子的产生使反应培养基的ph快速增加至酸脱羧酶最佳ph外的ph。
如本文所述融合至朊病毒亚基的酸脱羧酶包括赖氨酸脱羧酶、精氨酸脱羧酶、鸟氨酸脱羧酶和谷氨酸脱羧酶,如上文详述,它们是折叠型i吡哆醛5′-磷酸(plp)依赖性脱羧酶的原核鸟氨酸脱羧酶亚类的部分。这类蛋白通常包含n-末端翼结构域、核心结构域和c-末端结构域。核心结构域包含plp-结合子结构域和子结构域4。酸脱羧酶spea属于plp依赖性脱羧酶的不同折叠家族。这些享有很少序列相同性,但是结构是公知的。说明性酸脱羧酶结构的蛋白数据库识别号是cada:3n75,ldcc:5fkz,adia:2vyc,spea:3nzq,gada:1xey和gadb:1pmm。
对于cada,n-末端翼结构域(如参考seqidno:2确定的残基1-129)具有黄素氧还蛋白样折叠,由夹在两组两亲性α-螺旋之间的五链平行β-折叠组成。核心结构域(如参考seqidno:2的563确定的残基130-563)包括形成短螺旋束的接头区(seqidno:2的氨基酸残基130-183)、形成3组α-螺旋包围的七链β-折叠核心的plp结合子结构域(seqidno:2的氨基酸184-417)以及由氨基酸残基418-563组成的子结构域4,其形成四链反向平行β-折叠核心,3个α-螺旋朝外。c-末端结构域(对应于参考seqidno:2确定的氨基酸残基564-715)形成两组β折叠,具有α-螺旋外表面(kanjeeetal.,theembojournal30,931-9442011)。
cada蛋白形成双对称二聚体,完成每个单体的活性位点。5个二聚体关联形成十聚体,其由五重对称的双环状结构组成。十聚体与其他十聚体关联形成高阶寡聚体。已显示在酸性条件(ph5)下,cada主要以寡聚体状态存在,并且随着环境变得更加碱性发现较少的寡聚体和十聚体。估计在ph6.5,25%的酶以二聚体存在而75%以十聚体存在,但是在ph8.0,95%的酶以二聚体存在(kanjeeetal.,theembojournal30,931-9442011)。寡聚体形成的这种减少与酶的环境ph增加至5.0以上时观察到的脱羧酶活性减少一致,表明寡聚体形成的减少是脱羧酶活性减少的原因之一。
任何酸脱羧酶,例如赖氨酸脱羧酶、精氨酸脱羧酶、谷氨酸脱羧酶或鸟氨酸脱羧酶,可以按照本发明融合至朊病毒蛋白。合适的酸脱羧酶包括cada、ldcc、adia、spea、spec、spef、gada、gadb和它们的同系物。如本文所用:赖氨酸脱羧酶是指将l-赖氨酸转化为尸胺的酶;该酶分类为e.c.4.1.1.18;精氨酸脱羧酶是指将l-精氨酸转化为鲱精胺的酶,该酶分类为e.c.4.1.1.19,鲱精胺可以通过鲱精胺酶活性进一步转化为purtrescine;鸟氨酸脱羧酶是指将鸟氨酸转化为腐胺的酶,该酶分类为e.c.4.1.1.17,腐胺可以进一步转化为亚精胺和精胺;并且谷氨酸脱羧酶是指将谷氨酸转化为γ-氨基丁酸(gaba)的酶,该酶分类为4.1.1.15。
在一些实施方案中,赖氨酸脱羧酶是来自大肠杆菌的cada或来自另一物种的cada同系物,例如克雷伯氏菌属;产气肠杆菌;肠道沙门氏菌;沙雷氏菌属;和解鸟氨酸拉乌尔菌。在一些实施方案中,赖氨酸脱羧酶是来自大肠杆菌的ldcc或者来自志贺氏菌属、柠檬酸杆菌属和肠道沙门氏菌的ldcc同系物。如本文所用,赖氨酸脱羧酶包括具有赖氨酸脱羧酶酶促活性的天然赖氨酸脱羧酶的变体。其它赖氨酸脱羧酶在pct/cn2014/080873和pct/cn2015/072978中描述。
在一些实施方案中,适合用于本发明的赖氨酸脱羧酶多肽,优选相对seqidno:2的cada多肽的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的赖氨酸脱羧酶多肽,优选相对cada多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的赖氨酸脱羧酶多肽,优选相对seqidno:21的ldcc多肽的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的赖氨酸脱羧酶多肽,优选相对ldcc多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。说明性同系物和多肽序列的登录号是:志贺氏菌属(shigella)多物种(wp001020996.1)、费格森埃希菌(escherichiafergusonii)(wp001021009.1)、无色杆菌属(achromobactersp)atcc35328(cuj86682.1)、肠杆菌科(enterobacteriaceae)多物种(wp058668594.1)、柠檬酸杆菌属(citrobacter)多物种(wp016151770.1)、gammoprobacteria多物种(wp046401634.1)和邦戈尔沙门氏菌(salmonellabongori)(wp038390535.1)。
在一些实施方案中,适合用于本发明的精氨酸脱羧酶多肽是adi或其来自另一物种的同系物。在一些实施方案中,精氨酸脱羧酶多肽,优选相对seqidno:22的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的精氨酸脱羧酶多肽,优选相对adia多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。说明性同系物和多肽序列的登录号是:志贺氏菌属多物种(wp000978677.1)、埃希氏菌属多物种(wp000978709.1)、肠杆菌科多物种(wp032934133.1)、柠檬酸杆菌属多物种(wp008786969.1)、产酸克雷伯氏菌(klebsiellaoxytoca)(sap84601.1)和肠道沙门氏菌(wp048668294.1)。
在一些实施方案中,适合用于本发明的精氨酸脱羧酶多肽是spea或其来自另一物种的同系物。在一些实施方案中,spea多肽,优选相对seqidno:23的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的精氨酸脱羧酶多肽,优选相对spea多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。说明性同系物和多肽序列的登录号是:志贺氏菌属多物种(wp005096955.1)、埃希氏菌属多物种(wp010350365.1)、gammaproteobacteria多物种(wp042998051.1)、沙门氏菌属(salmonella)多物种(wp001278580.1)、无色杆菌属atcc35328(cuj95389.1)、法氏柠檬酸杆菌(citrobacterfarmeri)(wp042324083.1)、合适柠檬酸杆菌(citrobacterkoseri)(wp024130934.1)和无丙二酸柠檬酸杆菌(citrobacteramalonaticus)(wp052746994.1)。
在一些实施方案中,适合用于本发明的酸脱羧酶多肽是鸟氨酸脱羧酶spec或其来自另一物种的同系物。在一些实施方案中,鸟氨酸脱羧酶,优选相对seqidno:24的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的鸟氨酸脱羧酶多肽,优选相对spec多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。说明性同系物和序列同系物的登录号是:志贺氏菌属多物种(wp005085661.1)、埃希氏菌属多物种(wp010352539.1)、柠檬酸杆菌属多物种(wp044255681.1)、肠杆菌科(wp047357853.1)、gammaproteobacteria多物种(wp044327655.1)、无色杆菌属atcc35328(cuj95194.1)、产酸克雷伯氏菌(sbl12331.1)和肺炎克雷伯氏菌(klebsiellapneumonia)(cdk72259.1)。
在一些实施方案中,适合用于本发明的鸟氨酸脱羧酶多肽是spef或其来自另一物种的同系物。在一些实施方案中,鸟氨酸脱羧酶,优选相对seqidno:25的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的鸟氨酸脱羧酶多肽,优选相对spef多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或至少500或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。说明性同系物和多肽序列的登录号是:志贺氏菌属多物种(wp000040203.1)、肠杆菌科多物种(wp049009856.1)、埃希氏菌属多物种(wp001292417.1)、gammaproteobacteria多物种(wp046401512.1)、合适柠檬酸杆菌(wp024130539.1)、无丙二酸柠檬酸杆菌(wp046274704.1)、布氏柠檬酸杆菌(citrobacterbraakii)(wp047501716.1)和肠道沙门氏菌(wp023220629.1)。
在一些实施方案中,适合用于本发明的谷氨酸脱羧酶多肽是gada或其来自另一物种的同系物。在一些实施方案中,谷氨酸脱羧酶,优选相对seqidno:26的长度为至少约50、至少100、至少200、至少300、至少400或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的谷氨酸脱羧酶多肽,优选相对gada多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。说明性同系物和多肽序列的登录号是:埃希氏菌属多物种(wp001517297.1)、志贺氏菌属多物种(wp000358931.1)、耶尔森氏菌属(yersinia)多物种(wp050085789.1)、无色杆菌属atcc35328(wp054518524.1)和rhodococcsgingshengii(kdq00107.1)。
在一些实施方案中,适合用于本发明的谷氨酸脱羧酶多肽是gadb或其来自另一物种的同系物。在一些实施方案中,谷氨酸脱羧酶,优选相对seqidno:27的长度为至少约50、至少100、至少200、至少300、至少400或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。
在一些实施方案中,适合用于本发明的谷氨酸脱羧酶多肽,优选相对gadb多肽的同系物的长度为至少约50、至少100、至少200、至少300、至少400或更多个氨基酸的区域,或者全长,具有至少60%氨基酸序列相同性,优选至少65%、70%、75%、80%、85%、90%,优选91%、92%、93%、94%、95%、96%、97%、98%或99%或更大的氨基酸序列相同性。说明性同系物和多肽序列的登录号是:志贺氏菌属多物种(wp000358931.1)、埃希氏菌属多物种(wp016248697.1)、耶尔森氏菌属多物种(wp050085789.1)、无色杆菌属atcc35328(wp054518524.1)、rhodococcsgingshengii(kdq00107.1)。
编码朊病毒亚基和酸脱羧酶的核酸
酸脱羧酶多核苷酸序列的分离或产生可以通过许多技术完成。在一些实施方案中,基于本文公开序列的寡核苷酸探针可以用来鉴定来自期望细菌物种的cdna或基因组dna文库中的期望多核苷酸。探针可以用来与基因组dna或cdna序列杂交以从不同生物体例如真菌物种或植物物种分离同源基因。
或者,可以利用常规扩增技术从核酸样品扩增感兴趣的核酸。例如,pcr可以用来直接从mrna、cdna、基因组文库或cdna文库扩增基因的序列。pcr和其他体外扩增方法还可以用来例如克隆编码待表达的蛋白的核酸序列,制备核酸用作探针用于检测样品中期望mrna的存在、用于核酸测序或用于其他目的。
用于鉴定细菌中的酸脱羧酶多核苷酸的适当引物和探针可以产生自本文提供的序列的比较。pcr的综述参见pcrprotocols:aguidetomethodsandapplications.(innis,m,gelfand,d.,sninsky,j.andwhite,t.,eds.),academicpress,sandiego(1990)。说明性引物序列在实施例部分中的引物表中示出。
编码用于本公开的酸脱羧酶多肽的核酸序列包括利用说明性核酸序列,例如seqidno:1的cada多核苷酸序列,通过技术如杂交和/或序列分析鉴定和表征的基因和基因产物。在一些实施方案中,通过引入与酸脱羧酶多核苷酸例如seqidno:1的cada多核苷酸具有至少60%相同性,或者至少70%、75%、80%、85%或90%相同性或者100%相同性的核酸序列来遗传修饰宿主细胞。
可以将赋予宿主细胞增加的氨基酸如赖氨酸或氨基酸衍生产物如尸胺产生的按照本发明的编码酸脱羧酶融合蛋白的核酸序列进行额外的密码子优化以用于在期望的宿主细胞中表达。可以采用的方法和数据库是本领域已知的。例如,关于单个基因、一组共同功能或起源的基因、高表达基因中的密码子使用、整个生物体的聚集蛋白编码区中的密码子频率、相关生物体的聚集蛋白编码区中的密码子频率或者它们的组合,可以确定优选密码子。参见例如,henautanddanchinin"escherichiacoliandsalmonella,"neidhardt,etal.eds.,asmpres,washingtond.c.(1996),pp.2047-2066;nucleicacidsres.20:2111-2118;nakamuraetal.,2000,nucl.acidsres.28:292)。
重组载体的制备
可以利用本领域公知的方法制备用于表达酸脱羧酶融合蛋白的重组载体。例如,可以将编码酸脱羧酶融合多肽的dna序列(在下文中进一步详细描述)与指导序列在预期细胞,例如细菌细胞如大肠杆菌中从基因转录的转录和其他调节序列组合。在一些实施方案中,包含表达盒(包含编码酸脱羧酶融合多肽的基因)的表达载体进一步包含可操作地连接至编码酸脱羧酶融合多肽的核酸序列的启动子。在其他实施方案中,指导酸脱羧酶融合多肽序列基因转录的启动子和/或其他调节元件对于宿主细胞是内源的,并且例如通过同源重组引入包含酸脱羧酶融合基因的表达盒,从而将外源基因可操作地连接至内源启动子并通过内源启动子驱动表达。
如上所述,编码酸脱羧酶融合蛋白的多核苷酸的表达可以通过许多调节序列控制,包括启动子,其可以是组成型或诱导型的;以及如果需要的话,包括任选存在的阻遏物序列。特别是在细菌宿主细胞中,合适的启动子的实例是获得自大肠杆菌lac操纵子的启动子以及源自参与其他糖如半乳糖和麦芽糖代谢的基因的其他启动子。额外的实例包括启动子如trp启动子、bla启动子、噬菌体λpl和t5。此外,可以使用合成的启动子,如tac启动子(u.s.pat.no.4,551,433)。启动子的进一步实例包括天蓝色链霉菌(streptomycescoelicolor)琼脂糖酶基因(daga)、枯草芽孢杆菌(bacillussubtilis)果聚糖蔗糖酶基因(sacb)、地衣芽孢杆菌(bacilluslicheniformis)α-淀粉酶基因(amyl)、嗜热脂肪芽孢杆菌(bacillusstearothermophilus)生麦芽糖淀粉酶基因(amym)、解淀粉芽孢杆菌(bacillusamyloliquefaciens)α-淀粉酶基因(amyq)、地衣芽孢杆菌青霉素酶基因(penp)、枯草芽孢杆菌xyla和xylb基因。合适的启动子还在ausubel和sambrook&russell中描述,均如上文所示。额外的启动子包括jensen&hammer,appl.environ.microbiol.64:82,1998;shimada,etal.,j.bacteriol.186:7112,2004;和mikschetal.,appl.microbiol.biotechnol.69:312,2005描述的启动子。
在一些实施方案中,可以修饰影响酸脱羧酶多肽表达的启动子以增加表达。例如,可以用与天然启动子相比提供增加的表达的启动子代替内源酸脱羧酶启动子。
表达载体还可以包含影响编码酸脱羧酶融合多肽的多核苷酸表达的额外序列。这类序列包括增强子序列、核糖体结合位点或其他序列如转录终止序列等。
表达编码本发明的酸脱羧酶融合多肽的多核苷酸的载体可以是自主复制载体,即作为染色体外实体存在的载体,其复制独立于染色体复制,例如质粒、染色体外因子、微型染色体或人工染色体。载体可以包含保证自我复制的任何手段。或者,载体可以是这样的,当引入宿主时,其整合至基因组中并与其已整合的染色体一起复制。因此,表达载体可以额外地包含允许载体整合至宿主基因组中的元件。
本发明的表达载体优选包含一或多个选择标记,其允许容易选择转化的宿主。例如,表达载体可以包含赋予重组宿主生物体例如细菌细胞如大肠杆菌抗生素抗性(例如氨苄西林、卡那霉素、氯霉素或四环素抗性)的基因。
虽然任何合适的表达载体均可以用来整合期望序列,但是可容易获得的细菌表达载体包括但不限于:质粒如psclol、pbr322、pbbrlmcs-3、pur、pet、pex、pmrloo、pcr4、pbad24、p15a、pacyc、puc例如puc18或puc19,或者源自这些质粒的质粒;以及噬菌体,如ml3噬菌体和λ噬菌体。但是,本领域技术人员可以通过常规实验容易地确定任何特定表达载体是否适合任何给定宿主细胞。例如,可以将表达载体引入宿主细胞,然后监测其生存力和载体中包含的序列的表达。
可以利用任何数量的公知方法将本发明的表达载体引入宿主细胞,所示方法包括基于氯化钙的方法、电穿孔或本领域已知的任何其他方法。
宿主细胞
本发明提供工程化以表达酸脱羧酶融合多肽的遗传修饰的宿主细胞。本发明的遗传修饰的宿主菌株通常包含至少一个额外的遗传修饰,以便相对于没有所述一个额外的遗传修饰的对照菌株,例如野生型菌株或者没有所述一个额外的遗传修饰的相同菌株的细胞,增加氨基酸或氨基酸衍生物的生成。“增加氨基酸或氨基酸衍生物的生成的额外的遗传修饰”可以是任何遗传修饰。在一些实施方案中,遗传修饰是引入表达参与氨基酸或氨基酸衍生物合成的酶的多核苷酸。在一些实施方案中,宿主细胞包含多个修饰以便相对于野生型宿主细胞增加氨基酸或氨基酸衍生物的生成。
在一些方面,对宿主细胞进行遗传修饰以表达酸脱羧酶融合多肽联合修饰宿主细胞以过表达一种或多种赖氨酸生物合成多肽。
在一些实施方案中,可以将宿主细胞遗传修饰以表达影响赖氨酸生物合成的一种或多种多肽。赖氨酸生物合成多肽的实例包括大肠杆菌基因suca、ppc、aspc、lysc、asd、dapa、dapb、dapd、argd、dape、dapf、lysa、ddh、pntab、cyoabe、gadab、ybje、gdha、glta、succ、gadc、acnb、pflb、thra、acea、aceb、gltb、acee、sdha、mure、spee、speg、puua、puup和ygjg,或者来自其他生物体的相应基因。这类基因是本领域已知的(参见例如shahetal.,j.med.sci.2:152-157,2002;anastassiadia,s.recentpatentsonbiotechnol.1:11-24,2007)。参与尸胺生成的基因的综述还参见kind,etal.,appl.microbiol.biotechnol.91:1287-1296,2011。下文提供编码赖氨酸生物合成多肽的说明性基因。
在一些实施方案中,可以将宿主细胞遗传修饰以减弱或减少影响赖氨酸生物合成的一种或多种多肽的表达。这类多肽的实例包括大肠杆菌基因pck、pgi、dead、cite、mene、poxb、acea、aceb、acee、rpoc和thra,或者来自其他生物体的相应基因。这类基因是本领域已知的(参见例如shahetal.,j.med.sci.2:152-157,2002;anastassiadia,s.recentpatentsonbiotechnol.1:11-24,2007)。减弱基因以增加尸胺生成的综述还参见kind,etal.,appl.microbiol.biotechnol.91:1287-1296,2011。下文提供编码其减弱增加赖氨酸生物合成的多肽的说明性基因。
可以将编码赖氨酸生物合成多肽的核酸与酸脱羧酶融合多核苷酸一起引入宿主细胞,例如在单个表达载体上编码,或者在多个表达载体中同时引入。或者,可以将宿主细胞遗传修饰以在遗传修饰的宿主细胞表达酸脱羧酶融合多肽之前或之后过表达一种或多种赖氨酸生物合成多肽。
工程化以表达酸脱羧酶融合多肽的宿主细胞通常是细菌宿主细胞。在典型的实施方案中,细菌宿主细胞是革兰氏阴性细菌宿主细胞。在本发明的一些实施方案中,细菌是肠细菌。在本发明的一些实施方案中,细菌是棒杆菌属(corynebacterium)、埃希氏菌属、假单胞菌属(pseudomonas)、单胞发酵菌属(zymomonas)、希瓦氏菌属(shewanella)、沙门氏菌属、志贺氏菌属、肠杆菌属(enterobacter)、柠檬酸杆菌属、cronobacter、欧文氏菌属(erwinia)、沙雷氏菌属(serratia)、变形杆菌属(proteus)、哈夫尼菌属(hafnia)、耶尔森氏菌属、摩根菌属(morganella)、爱德华氏菌属(edwardsiella)或克雷伯氏菌属(klebsiella)分类学类别的物种。在一些实施方案中,宿主细胞是埃希氏菌属、哈夫尼菌属或棒杆菌属的成员。在一些实施方案中,宿主细胞是大肠杆菌、蜂房哈夫尼菌或谷氨酸棒杆菌(corynebacteriumglutamicum)宿主细胞。
在一些实施方案中,宿主细胞是革兰氏阳性细菌宿主细胞,如芽孢杆菌(bacillussp.),例如枯草芽孢杆菌或地衣芽孢杆菌;或者另一芽孢杆菌如嗜碱芽孢杆菌(b.alcalophilus)、嗜胺芽孢杆菌(b.aminovorans)、解淀粉芽孢杆菌(b.amyloliquefaciens)、热溶芽孢杆菌(b.caldolyticus)、环状芽孢杆菌(b.circulans)、嗜热脂肪芽孢杆菌(b.stearothermophilus)、热葡糖苷酶芽胞杆菌(b.thermoglucosidasius)、苏云金芽孢杆菌(b.thuringiensis)或b.vulgatis。
如本文所述,可以针对增加的赖氨酸或赖氨酸衍生物如尸胺的生成筛选按照本发明修饰的宿主细胞。
在一些实施方案中,可以从表达融合蛋白的宿主细胞回收本发明的酸脱羧酶融合蛋白。在一些实施方案中,可以将回收的融合蛋白固定在固体基底或惰性材料上以形成固定化的酶。在一个实施方案中,固定化的酶可以具有比融合蛋白的可溶形式提高的热稳定性和/或操作稳定性。在一个实施方案中,融合蛋白包含在c-末端融合至朊病毒亚基的赖氨酸、精氨酸、鸟氨酸或谷氨酸脱羧酶。
制备赖氨酸或赖氨酸衍生物的方法
遗传修饰以表达酸脱羧酶融合多肽的宿主细胞可以用来制备赖氨酸或赖氨酸的衍生物。在一些实施方案中,宿主细胞产生尸胺。为制备赖氨酸或赖氨酸衍生物,可以将如本文所述遗传修饰以表达酸脱羧酶融合多肽的宿主细胞在合适的条件下培养,所述条件允许表达所述多肽以及表达编码用来产生赖氨酸或赖氨酸衍生物的酶的基因。相对于表达未融合至朊病毒亚基的酸脱羧酶的未修饰的对应宿主细胞,按照本发明的被修饰表达酸脱羧酶融合多肽的宿主细胞提供较高收率的赖氨酸或赖氨酸衍生物。
可以利用公知技术培养宿主细胞(参见例如实施例部分中提供的说明性条件)。
在一些实施方案中,利用不是盐(例如硫酸铵或氯化铵)的氮源如氨或尿素培养宿主细胞。在细胞生长或酶生成期间可以在碱性ph培养宿主细胞。
然后利用已知技术分离并纯化赖氨酸或赖氨酸衍生物。然后按照本发明制备的赖氨酸或赖氨酸衍生物如尸胺可以用于任何已知方法,例如制备聚酰胺。
在一些实施方案中,可以利用化学催化剂或者利用酶和化学催化剂将赖氨酸转化为己内酰胺。
通过具体实施例更详细地描述本发明。为了说明目的提供以下实施例,而不意图以任何方式限制本发明。本领域技术人员会容易地认识到各种非关键参数,其可以改变或修改以获得基本上相同的结果。
实施例
实施例1:编码cada的质粒载体的构建
利用pcr引物cada-f和cada-r从大肠杆菌mg1655k12基因组dna扩增包含编码赖氨酸脱羧酶cada(seqidno:2)的野生型大肠杆菌cada(seqidno:1)的质粒载体(图1),利用限制性酶saci和xbai消化,并且连接至puc18中产生质粒pcib60。利用pcr引物cada-f2和cada-r2优化cada基因上游的5’序列产生pcib71。利用引物cat-hindiii-f和cat-ndei-r扩增氯霉素抗性基因cat,并且克隆至pcib71中cada之后产生pcib128。
实施例2:密码子优化的new1和sup35朊病毒片段的合成
基于osherovichetal.,plosbiology2:4,2004确定new1和sup35朊病毒活性必需的最小多肽片段。因此,将new1(seqidno:3)的氨基酸2-100和sup35(seqidno:4)的氨基酸2-103经密码子优化以在大肠杆菌中异源表达(seqidno:5和6)。此外,将由氨基酸gsgsg组成的短多肽接头序列添加至seqidno:3和4的开始(seqidno:7和8),并且它们相应的dna序列在seqidno:9和10中示出。根据hooverdm&lubkowskij,nucleicacidsresearch30:10,2002进行密码子优化和dna组装。
实施例3:编码融合cada的3’的new1片段的多核苷酸的构建
利用引物cadat-xbai-r和cat-xbai-f去除pcib128中cada的终止密码子,产生质粒pcib138。利用引物new1-xbai-f和new1-hindiii-r扩增由具有多肽接头的new1的朊病毒结构域组成的dna片段,利用限制性酶xbai和hindiii消化,并且连接至pcib138产生pcib222。
实施例4:编码融合cada的3’的sup35的片段的多核苷酸的构建
利用引物35-xbai-f和35-hindiii-r扩增由具有多肽接头的sup35的朊病毒结构域组成的dna片段,利用限制性酶xbai和hindiii消化,并且连接至pcib138产生pcib223。
实施例5:由融合cada的3’的new1或sup35朊病毒结构域片段组成的新多肽的赖氨酸脱羧酶活性
用pcib128、pcib222和pcib223转化蜂房哈夫尼菌(h.alvei)。将来自每个转化的3个单克隆在4ml具有氨苄西林(100μg/ml)的lb培养基中于37℃生长过夜。第二天,将0.7ml的每个过夜培养物添加至0.3ml的赖氨酸-hcl和plp,其终浓度分别为120g/l和0.1mm。将每个混合物在37℃温育2小时。利用nmr定量来自每个样品的尸胺生成,并且通过将产生的尸胺的摩尔量除以添加的赖氨酸的摩尔量计算收率/od。2小时之后来自每个样品的收率在表1中示出。
表1.过量产生由朊病毒结构域片段和赖氨酸脱羧酶组成的融合多肽的蜂房哈夫尼菌菌株的尸胺生成。
如表1所示,由融合cada的3’的new1或sup35朊病毒结构域片段组成的两种多肽均表现出显著的赖氨酸脱羧酶活性。当赖氨酸脱羧酶自身表达而不融合至朊病毒结构域片段时,赖氨酸脱羧酶活性低于对照。活性的减少可以由许多因素引起,其中一些是细胞密度和总蛋白减少,赖氨酸脱羧酶活性减少,功能性可溶性酶的量减少,或者因为融合多肽具有折叠困难而不溶性蛋白部分增加。
实施例6:编码融合cada的5’的new1片段的多核苷酸的构建
利用引物cada-xbai-f和cada-hindiii-r扩增cada基因,利用限制性酶xbai和hindiii消化,并且连接至pcib128产生具有两个拷贝的cada基因的质粒pcib146。利用引物rbs2-saci-f和rbs2-saci-r将saci限制性位点添加至启动子后的第一个cada基因的5’,构建puib149。利用引物new1-saci-f和new1-xbai-r扩增new1朊病毒片段,利用限制性酶saci和xbai消化,并且连接至pcib149产生pcib241。
实施例7:编码融合cada的5’的sup35片段的多核苷酸的构建
利用引物sup35-saci-f和sup35-xbai-r扩增sup35朊病毒片段,利用限制性酶saci和xbai消化,并且连接至pcib149产生pcib242。
实施例8:由融合cada的5’的new1或sup35朊病毒结构域片段组成的新多肽的赖氨酸脱羧酶活性
用pcib128、pcib241和pcib242转化蜂房哈夫尼菌。将来自每个转化的3个单克隆在4ml具有氨苄西林(100μg/ml)的lb培养基中于37℃生长过夜。第二天,将0.7ml的每个过夜培养物添加至0.3ml的赖氨酸-hcl和plp,其终浓度分别为120g/l和0.1mm。将每个混合物在37℃温育2小时。利用nmr定量来自每个样品的尸胺生成,并且通过将产生的尸胺的摩尔量除以添加的赖氨酸的摩尔量计算收率/od。2小时之后来自每个样品的收率在表2中示出。
表2.过量产生由朊病毒结构域片段和赖氨酸脱羧酶组成的融合多肽的蜂房哈夫尼菌菌株的尸胺生成。
如表2所示,由融合cada的5’的new1或sup35朊病毒结构域片段组成的两种多肽均表现出非常少的赖氨酸脱羧酶活性。当赖氨酸脱羧酶自身表达而不融合至朊病毒结构域片段时,赖氨酸脱羧酶活性低于对照。与朊病毒结构域片段融合cada的3’的那些相比,这些酶的赖氨酸脱羧酶活性也较低。这表明合成的融合多肽在朊病毒结构域融合c-末端结构域之后的酸脱羧酶的3’时更具功能性,并且在朊病毒结构域融合n-末端翼结构域之前的酸脱羧酶的5’时功能性较少。
实施例9:过表达融合至cada的new1朊病毒结构域的蜂房哈夫尼菌的细胞密度和酶生成
为确定在new1朊病毒结构域片段融合赖氨酸脱羧酶的3’时观察到的总活性减少的原因,在abs600测量过夜培养物的细胞密度,并且将培养物裂解以利用bradford法确定总蛋白含量。利用sds-page分析裂解的细胞培养物以确定总蛋白的多少由合成的融合多肽组成。利用冻融和溶菌酶的组合进行细胞裂解。用dnase处理裂解的样品以去除大部分dna。od和总蛋白数据在表3中示出。sds-page的结果在图1中示出。
表3.由朊病毒结构域片段和赖氨酸脱羧酶组成的融合多肽的od和总蛋白数据。
如表3所示,与产生单独的赖氨酸脱羧酶(pcib128)相比,当蜂房哈夫尼菌产生融合多肽(pcib222)时观察到的od和总蛋白均较低。od减少17%,而总蛋白减少38%。sds-page结果显示背景蛋白未显著改变;相反,靶蛋白显著减少(在80kda处的黑色条带)。此外,发现大部分靶蛋白在可溶性部分中,这表明蛋白溶解度不是问题。因此,总活性的减少不是由于不溶性的失活蛋白的积累。
实施例10:融合至cada的new1朊病毒结构域的不同大小片段的构建
据观察当与没有朊病毒结构域片段的对照相比时,朊病毒结构域片段与赖氨酸脱羧酶的融合物影响宿主细胞的生长,并且减少达到的最终细胞密度(在abs600测量的od)。如上文证实的,细胞密度的减少会减少产生的可溶性酶的量和总活性。如sds-page结果所示,od的减少不是由于不良折叠的酶的积累。但是,细胞密度的减少可以是由新融合多肽折叠中的挑战(例如较慢的动力学)引起的。
产生new1朊病毒结构域的不同大小的片段以确定截短一部分朊病毒结构域是否会减少细胞的负担并提高生长和最终细胞密度。将new1朊病毒结构域片段的n-末端截短各种长度的氨基酸以确定q/n残基较少的片段部分对于融合多肽的赖氨酸脱羧酶功能是否重要,以及它们的去除是否可以提高宿主的生长。利用pcr引物222-de-r以及各自的pcr引物222-de-9-f、222-de18-f、222-de36-f和222-de45-f截短pcib222中new1朊病毒结构域片段的前9、18、36和45个氨基酸(seqidno:11、12、13和14)。
实施例11:过表达融合至cada的new1朊病毒结构域变体的蜂房哈夫尼菌的细胞密度的提高
用pcib222或者new1朊病毒结构域片段的前9、18、27、36或45个氨基酸截短的pcib222的变体转化大肠杆菌bl21。将来自每个转化的3个单克隆在4ml具有氨苄西林(100μg/ml)的lb培养基中于37℃生长过夜。第二天,在abs600测量过夜培养物的吸光度。每个样品的od在表4中示出,并且每个样品的sds-page分析在图2中示出。
表4.由朊病毒结构域片段和赖氨酸脱羧酶组成的融合多肽的截短片段的od。
表4显示与具有整个朊病毒结构域片段的对照相比,new1朊病毒结构域片段的截短版本提高大肠杆菌bl21宿主的生长。产生不同版本的截短酶的菌株之间最终od没有显著差异。尽管基于od数据在截短版本之间不能看到差异,但是sds-page分析显示总蛋白显著增加,并且对具有36个氨基酸截短的版本观察到靶酶。与对照相比,具有9、18和45个氨基酸截短的酶的版本表现出较少的总蛋白和靶酶。
实施例12:编码融合至cada的new1片段的多核苷酸的c-末端氨基酸的修饰
折叠缓慢或不良的合成蛋白在过表达时被降解是常见的。因此,减少蛋白降解以增加多肽折叠的时间会增加细胞中功能蛋白的量。以前的工作(trepodcm&mottje,jbiotechnol84,273-284,2000)报道短多肽的特定序列当融合至蛋白的c-末端时可以增加蛋白稳定性并减少胞内蛋白降解。这些短多肽源自蛋白如牛生长激素(bst)、rpoc、λci、rho、reca、bla和tufa的c-末端。
利用各自的pcr引物222-c2-f、222-c2-r、222-c4-f、222-c4-r、222-c6-f和222-c6-r将源自基因bst(c2)、大肠杆菌λci(c4)和大肠杆菌reca(c6)的3个不同短多肽融合至pcib222中new1朊病毒结构域片段的c-末端。bst片段由氨基酸序列rrfgeassaf组成(seqidno:15和16),λci片段由氨基酸序列asqwpeetfg组成(seqidno:17和18),并且reca片段由氨基酸序列egvaetnedf组成(seqidno:19和20)。
实施例13:过表达融合至cada的new1朊病毒结构域的c-末端变体的大肠杆菌的总酶活性的提高
用pcib222或pcib222的变体(其中将bst、λci或reca片段融合至new1朊病毒结构域片段的c-末端)转化大肠杆菌bl21。将来自每个转化的3个单克隆在4ml具有氨苄西林(100μg/ml)的lb培养基中于37℃生长过夜。第二天,将0.6ml的每个过夜培养物添加至0.4ml的赖氨酸-hcl和plp,其终浓度分别为160g/l和0.1mm。将每个混合物在37℃温育2小时。利用nmr定量来自每个样品的尸胺生成,并且通过将产生的尸胺的摩尔量除以添加的赖氨酸的摩尔量计算收率。2小时之后来自每个样品的收率在表5中示出。
表5.产生由朊病毒结构域片段和赖氨酸脱羧酶组成的融合多肽的c-末端变体的大肠杆菌的尸胺生成。
表5显示用源自λci的额外的短多肽修饰new1朊病毒结构域片段的c-末端提高来自赖氨酸的尸胺收率。用源自bst或reca的额外的短多肽修饰朊病毒结构域片段在尸胺收率中表现出较不显著的改变。在包含λcic-末端片段的菌株中观察到的总活性增加表明与缺少λci多肽的双融合多肽相比,由cada、new1朊病毒结构域蛋白和λci多肽组成的三融合多肽的稳定性增加。
实施例14:添加山梨醇对由融合至cada的new1组成的多肽的赖氨酸脱羧酶活性的影响
可以在宿主中过表达蛋白分子伴侣以提高细胞中产生的功能蛋白的量,特别是在折叠不好或对于宿主不是天然的合成蛋白的情况下。例如,大肠杆菌中使用的常见分子伴侣蛋白系统是groel/groes、dnak/dnaj/grpe、clpb和热休克蛋白/ibpa/ibpb(demarcoetal.,bmcbiotechnol7:32,2007)。还已证实可以增加产生的可溶性和功能蛋白的量的化学分子伴侣(prasad,etal.,applenvironmicrobiol77,4603-4609,2011)。
为改善由融合至赖氨酸脱羧酶cada的new1朊病毒结构域组成的合成多肽的折叠,在蛋白生成期间添加山梨醇。用pcib222转化大肠杆菌bl21和蜂房哈夫尼菌。使来自每个转化的3个单克隆在4ml具有氨苄西林(100μg/ml)和足够的山梨醇(达到终浓度为0、0.2或0.8m)的lb培养基中于37℃生长过夜。第二天,将0.6ml的每个过夜培养物添加至0.4ml的赖氨酸-hcl和plp,其终浓度分别为160g/l和0.1mm。将每个混合物在37℃温育2小时。利用nmr定量来自每个样品的尸胺生成,并且通过将产生的尸胺的摩尔量除以添加的赖氨酸的摩尔量再除以在abs600过夜培养物的吸光度计算收率/od。来自每个样品的收率/od在表6中示出。
表6.用山梨醇生长并过表达融合多肽的大肠杆菌和蜂房哈夫尼菌菌株的尸胺生成。
如表6所示,山梨醇的添加根据使用的菌株不同地影响酶活性。在大肠杆菌和蜂房哈夫尼菌中,与0m相比,山梨醇在0.8m的浓度下负面影响生长,并且在0.2m下对生长的影响较不显著。当蜂房哈夫尼菌是宿主时,山梨醇的添加增加收率/od,而当大肠杆菌是宿主时,山梨醇的添加减少收率/od。虽然在蜂房哈夫尼菌中山梨醇的添加减少od,但是山梨醇增加赖氨酸脱羧酶活性/细胞并增加总活性/培养体积。
实施例15:由融合至cada的new1组成的多肽在不同ph的体外赖氨酸脱羧酶活性和动力学
根据文献,由于从高寡聚体状态至低寡聚体状态的结构改变,赖氨酸脱羧酶在ph8的活性显著少于其在ph6的活性。在ph6和ph8比较有和无融合至其c-末端的new1朊病毒片段的赖氨酸脱羧酶的活性,以确定朊病毒片段是否可以增加多肽对碱性条件的耐受性。
用frenchpress裂解用pcib128或pcib222转化的蜂房哈夫尼菌的100ml样品。将裂解的样品离心,并且分离上清液和沉淀以进行体外实验。利用nmr测量每个裂解样品的反应速率,通过在总计20分钟中每1.6分钟采样在plp的存在下转化为尸胺的赖氨酸的量,并且取收率曲线的线性部分的斜率进行。将样品稀释,从而裂解样品的反应速率/体积(u)相同。在6或ph8的初始ph下利用相同的u测量每个裂解样品的赖氨酸的动力学常数vmax和km。通过对u归一化,每个样品中活性酶的浓度相同。两个样品的动力学分析的结果在表7中示出。
表7.ph对产生有和无new1朊病毒结构域片段的赖氨酸脱羧酶的蜂房哈夫尼菌的裂解样品的影响的动力学分析。
按照文献,与ph6相比,野生型赖氨酸脱羧酶(cib128)在ph8丢失大量活性。与ph6相比,在ph8的活性减少33%。令人惊讶的是,当初始ph为8时,与6相比,融合至new1朊病毒结构域的赖氨酸脱羧酶(cib222)仅丢失其活性的10%。即使初始ph改变,野生型或修饰的赖氨酸脱羧酶对赖氨酸的km不改变。因为添加的活性酶的量通过u归一化,修饰的酶更好地耐受碱性ph的能力可能是由于其与野生型酶相比保持较高寡聚体状态的能力。
实施例16:由融合至cada的new1组成的多肽在不同温度的体外赖氨酸脱羧酶活性和动力学
对高温的耐受是生物酶在大规模生产系统中的另一有益性状,特别是在夏季的几个月中,以便减少冷却反应器的成本。大多数酶在狭窄的温度范围内工作,并且比该范围高的温度倾向于引起酶变性或不以功能必需的结构状态存在。与new1朊病毒结构域片段融合的赖氨酸脱羧酶表现出对高ph增加的耐受性。可能朊病毒结构域片段提供的增加的结构稳定性不仅对耐受高ph有用,而且还对耐受高温有用。在37℃、45℃和55℃温育不同时间之后确定有和无new1朊病毒结构域片段的赖氨酸脱羧酶的活性,以确定酶是否能够在高温下保持对于功能是必需的结构完整性。
用frenchpress裂解用pcib128或pcib222转化的蜂房哈夫尼菌的100ml样品。将裂解的样品离心,并且分离上清液和沉淀以进行体外实验。利用nmr在plp的存在下测量每个裂解样品的反应速率,通过在总计20分钟中每1.6分钟采样转化为尸胺的赖氨酸的量,并且取收率曲线的线性部分的斜率进行。将样品稀释,从而裂解样品的反应速率/体积(u)相同。将基于u等量的酶在37℃、45℃和55℃温育0、1、2、4和20小时。在特定温度和时间温育之后,在37℃确定每个酶样品的反应速率。温度和时间对两种不同酶的影响在表8中示出。
表8.在不同温度温育不同时间之后有或无new1朊病毒结构域片段的赖氨酸脱羧酶的相对活性。
表8示出令人惊讶的发现,new1朊病毒结构域片段不仅增加赖氨酸脱羧酶对碱性ph的耐受,而且其还增加酶对高温的耐受。在55℃温育1小时之后,野生型赖氨酸脱羧酶(cib128)几乎失去其所有活性。但是,与new1朊病毒结构域片段融合的赖氨酸脱羧酶(cib222)能够保持其活性的93%。此外,当在55℃温育4小时之后未观察到野生型酶的可检测的活性时,融合多肽仍保持其活性的71%。因此,通过将其与朊病毒结构域片段融合赋予酸脱羧酶的增加的稳定性不是特异性耐受单一环境应激的,并且可以使得新酶能够在可用于工业生产的更宽范围的操作条件下发挥功能。
实施例中使用的质粒表
实施例中使用的引物序列表
说明性序列:
seqidno:1大肠杆菌cada核酸序列
atgaacgttattgcaatattgaatcacatgggggtttattttaaagaagaacccatccgtgaacttcatcgcgcgcttgaacgtctgaacttccagattgtttacccgaacgaccgtgacgacttattaaaactgatcgaaaacaatgcgcgtctgtgcggcgttatttttgactgggataaatataatctcgagctgtgcgaagaaattagcaaaatgaacgagaacctgccgttgtacgcgttcgctaatacgtattccactctcgatgtaagcctgaatgacctgcgtttacagattagcttctttgaatatgcgctgggtgctgctgaagatattgctaataagatcaagcagaccactgacgaatatatcaacactattctgcctccgctgactaaagcactgtttaaatatgttcgtgaaggtaaatatactttctgtactcctggtcacatgggcggtactgcattccagaaaagcccggtaggtagcctgttctatgatttctttggtccgaataccatgaaatctgatatttccatttcagtatctgaactgggttctctgctggatcacagtggtccacacaaagaagcagaacagtatatcgctcgcgtctttaacgcagaccgcagctacatggtgaccaacggtacttccactgcgaacaaaattgttggtatgtactctgctccagcaggcagcaccattctgattgaccgtaactgccacaaatcgctgacccacctgatgatgatgagcgatgttacgccaatctatttccgcccgacccgtaacgcttacggtattcttggtggtatcccacagagtgaattccagcacgctaccattgctaagcgcgtgaaagaaacaccaaacgcaacctggccggtacatgctgtaattaccaactctacctatgatggtctgctgtacaacaccgacttcatcaagaaaacactggatgtgaaatccatccactttgactccgcgtgggtgccttacaccaacttctcaccgatttacgaaggtaaatgcggtatgagcggtggccgtgtagaagggaaagtgatttacgaaacccagtccactcacaaactgctggcggcgttctctcaggcttccatgatccacgttaaaggtgacgtaaacgaagaaacctttaacgaagcctacatgatgcacaccaccacttctccgcactacggtatcgtggcgtccactgaaaccgctgcggcgatgatgaaaggcaatgcaggtaagcgtctgatcaacggttctattgaacgtgcgatcaaattccgtaaagagatcaaacgtctgagaacggaatctgatggctggttctttgatgtatggcagccggatcatatcgatacgactgaatgctggccgctgcgttctgacagcacctggcacggcttcaaaaacatcgataacgagcacatgtatcttgacccgatcaaagtcaccctgctgactccggggatggaaaaagacggcaccatgagcgactttggtattccggccagcatcgtggcgaaatacctcgacgaacatggcatcgttgttgagaaaaccggtccgtataacctgctgttcctgttcagcatcggtatcgataagaccaaagcactgagcctgctgcgtgctctgactgactttaaacgtgcgttcgacctgaacctgcgtgtgaaaaacatgctgccgtctctgtatcgtgaagatcctgaattctatgaaaacatgcgtattcaggaactggctcagaatatccacaaactgattgttcaccacaatctgccggatctgatgtatcgcgcatttgaagtgctgccgacgatggtaatgactccgtatgctgcattccagaaagagctgcacggtatgaccgaagaagtttacctcgacgaaatggtaggtcgtattaacgccaatatgatccttccgtacccgccgggagttcctctggtaatgccgggtgaaatgatcaccgaagaaagccgtccggttctggagttcctgcagatgctgtgtgaaatcggcgctcactatccgggctttgaaaccgatattcacggtgcataccgtcaggctgatggccgctataccgttaaggtattgaaagaagaaagcaaaaaataa
seqidno:2cada多肽序列
mnviailnhmgvyfkeepirelhralerlnfqivypndrddllkliennarlcgvifdwdkynlelceeiskmnenlplyafantystldvslndlrlqisffeyalgaaediankikqttdeyintilppltkalfkyvregkytfctpghmggtafqkspvgslfydffgpntmksdisisvselgslldhsgphkeaeqyiarvfnadrsymvtngtstankivgmysapagstilidrnchkslthlmmmsdvtpiyfrptrnaygilggipqsefqhatiakrvketpnatwpvhavitnstydgllyntdfikktldvksihfdsawvpytnfspiyegkcgmsggrvegkviyetqsthkllaafsqasmihvkgdvneetfneaymmhtttsphygivastetaaammkgnagkrlingsieraikfrkeikrlrtesdgwffdvwqpdhidttecwplrsdstwhgfknidnehmyldpikvtlltpgmekdgtmsdfgipasivakyldehgivvektgpynllflfsigidktkalsllraltdfkrafdlnlrvknmlpslyredpefyenmriqelaqnihklivhhnlpdlmyrafevlptmvmtpyaafqkelhgmteevyldemvgrinanmilpyppgvplvmpgemiteesrpvleflqmlceigahypgfetdihgayrqadgrytvkvlkeeskk
seqidno:3new1朊病毒亚基多肽序列
fppkkfkdlnsflddqpkdpnlvaspfggyfknpaadagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyq
seqidno:4sup35朊病毒亚基多肽序列
sdsnqgnnqqnyqqysqngnqqqgnnryqgyqaynaqaqpaggyyqnyqgysgyqqggyqqynpqggyqqdagyqqqynpqggyqqynpqggyqqqfnpqgg
seqidno:5new1朊病毒亚基核酸序列
tttccgccgaaaaagttcaaagacctgaactctttcctggacgaccagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcggcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactggaaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaactacaacaactacaataactataataactacaacaactacaacaattataacaaatacaacggtcagggctaccag
seqidno:6sup35朊病毒亚基核酸序列
tctgactctaaccaaggtaataaccagcagaactaccaacaatactctcagaacggcaaccagcagcagggcaacaaccgctatcaaggctaccaagcgtacaacgcgcaggcacagccagcaggtggctactaccagaattaccagggttactctggttaccagcaaggtggttatcaacagtataatccgcagggcggctatcagcaggacgcaggttaccagcaacaatataaccctcagggcggctatcagcaatacaacccgcaaggcggttatcaacaacagtttaacccacagggtggc
seqidno:7new1朊病毒亚基和接头多肽序列。接头序列下划线
gsgsgfppkkfkdlnsflddqpkdpnlvaspfggyfknpaadagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyq
seqidno:8sup35朊病毒亚基和接头多肽序列
gsgsgsdsnqgnnqqnyqqysqngnqqqgnnryqgyqaynaqaqpaggyyqnyqgysgyqqggyqqynpqggyqqdagyqqqynpqggyqqynpqggyqqqfnpqgg
seqidno:9new1朊病毒亚基和接头核酸序列。编码接头的区域下划线。
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgaccagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcggcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactggaaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaactacaacaactacaataactataataactacaacaactacaacaattataacaaatacaacggtcagggctaccag
seqidno:10sup35朊病毒亚基和接头核酸序列。编码接头的区域下划线。
ggtagcggctctggctctgactctaaccaaggtaataaccagcagaactaccaacaatactctcagaacggcaaccagcagcagggcaacaaccgctatcaaggctaccaagcgtacaacgcgcaggcacagccagcaggtggctactaccagaattaccagggttactctggttaccagcaaggtggttatcaacagtataatccgcagggcggctatcagcaggacgcaggttaccagcaacaatataaccctcagggcggctatcagcaatacaacccgcaaggcggttatcaacaacagtttaacccacagggtggc
seqidno:11具有9个氨基酸截短的new1朊病毒亚基和接头多肽序列。接头下划线。
gsgsgnsflddqpkdpnlvaspfggyfknpaadagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyq
seqidno:12具有18个氨基酸截短的new1朊病毒亚基和接头多肽序列。接头下划线。
gsgsgdpnlvaspfggyfknpaadagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyq
seqidno:13具有36个氨基酸截短的new1朊病毒亚基和接头多肽序列。接头下划线。
gsgsgdagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyq
seqidno:14具有45个氨基酸截短的new1朊病毒亚基和接头多肽序列。接头下划线。
gsgsgkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyq
seqidno:15具有bstc-末端片段的new1朊病毒结构域片段和接头多肽序列。接头下划线。
gsgsgfppkkfkdlnsflddqpkdpnlvaspfggyfknpaadagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyqrrfgeassaf
seqidno:16具有bstc-末端片段的new1朊病毒亚基和接头核酸序列。编码接头的区域下划线。
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgaccagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcggcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactggaaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaactacaacaactacaataactataataactacaacaactacaacaattataacaaatacaacggtcagggctaccagcgtcgtttcggcgaagcgagcagcgcgttc
seqidno:17具有λcic-末端片段的new1朊病毒亚基和接头多肽序列。接头下划线。
gsgsgfppkkfkdlnsflddqpkdpnlvaspfggyfknpaadagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyqasqwpeetfg
seqidno:18具有λcic-末端片段的new1朊病毒结构域片段和接头核酸序列。编码接头的区域下划线。
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgaccagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcggcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactggaaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaactacaacaactacaataactataataactacaacaactacaacaattataacaaatacaacggtcagggctaccaggcgagccagtggccggaagaaaccttcggc
seqidno:19具有recac-末端片段的new1朊病毒结构域片段和接头多肽序列。接头下划线。
gsgsgfppkkfkdlnsflddqpkdpnlvaspfggyfknpaadagsnnaskkssyqqqrnwkqggnyqqggyqsydsnynnynnynnynnynnynnynkyngqgyqegvaetnedf
seqidno:20具有recac-末端片段的new1亚基和接头核酸序列。编码接头的区域下划线。
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgaccagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcggcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactggaaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaactacaacaactacaataactataataactacaacaactacaacaattataacaaatacaacggtcagggctaccaggaaggcgtggcggaaaccaacgaagatttc
seqidno:21ldcc多肽序列
mniiaimgphgvfykdepikelesalvaqgfqiiwpqnsvdllkfiehnpricgvifdwdeysldlcsdinqlneylplyafinthstmdvsvqdmrmalwffeyalgqaediairmrqytdeyldnitppftkalftyvkerkytfctpghmggtayqkspvgclfydffggntlkadvsisvtelgslldhtgphleaeeyiartfgaeqsyivtngtstsnkivgmyaapsgstllidrnchkslahllmmndvvpvwlkptrnalgilggiprreftrdsieekvaattqaqwpvhavitnstydgllyntdwikqtldvpsihfdsawvpythfhpiyqgksgmsgervagkvifetqsthkmlaalsqaslihikgeydeeafneafmmhtttspsypivasvetaaamlrgnpgkrlinrsveralhfrkevqrlreesdgwffdiwqppqvdeaecwpvapgeqwhgfndadadhmfldpvkvtiltpgmdeqgnmseegipaalvakfldergivvektgpynllflfsigidktkamgllrgltefkrsydlnlriknmlpdlyaedpdfyrnmriqdlaqgihklirkhdlpglmlrafdtlpemimtphqawqrqikgevetialeqlvgrvsanmilpyppgvpllmpgemltkesrtvldfllmlcsvgqhypgfetdihgakqdedgvyrvrvlkmag
seqidno:22adia多肽序列
mkvliveseflhqdtwvgnaverladalsqqnvtvikstsfddgfailssneaidclmfsyqmehpdehqnvrqligklherqqnvpvfllgdrekalaamdrdllelvdefawiledtadfiagravaamtryrqqllpplfsalmkysdiheyswaapghqggvgftktpagrfyhdyygenlfrtdmgiertslgslldhtgafgesekyaarvfgadrswsvvvgtsgsnrtimqacmtdndvvvvdrnchksieqglmltgakpvymvpsrnrygiigpiypqemqpetlqkkisespltkdkagqkpsycvvtnctydgvcynakeaqdllektsdrlhfdeawygyarfnpiyadhyamrgepgdhngptvfathsthkllnalsqasyihvregrgainfsrfnqaymmhattsplyaicasndvavsmmdgnsglsltqevideavdfrqamarlykeftadgswffkpwnkevvtdpqtgktydfadaptkllttvqdcwvmhpgeswhgfkdipdnwsmldpikvsilapgmgedgeleetgvpaalvtawlgrhgivptrttdfqimflfsmgvtrgkwgtlvntlcsfkrhydantplaqvmpelveqypdtyanmgihdlgdtmfawlkennpgarlneaysglpvaevtpreaynaivdnnvelvsienlpgriaansvipyppgipmllsgenfgdknspqvsylrslqswdhhfpgfehetegteiidgiyhvmcvka
seqidno:23spea多肽序列
msddmsmglpssagehgvlrsmqevamssqeaskmlrtyniawwgnnyydvnelghisvcpdpdvpearvdlaqlvktreaqgqrlpalfcfpqilqhrlrsinaafkraresygyngdyflvypikvnqhrrvieslihsgeplgleagskaelmavlahagmtrsvivcngykdreyirlaligekmghkvylviekmseiaivldeaerlnvvprlgvrarlasqgsgkwqssggekskfglaatqvlqlvetlreagrldslqllhfhlgsqmanirdiatgvresarfyvelhklgvniqcfdvggglgvdyegtrsqsdcsvnyglneyanniiwaigdaceenglphptvitesgravtahhtvlvsniigverneytvptapaedapralqsmwetwqemhepgtrrslrewlhdsqmdlhdihigyssgifslqerawaeqlylsmchevqkqldpqnrahrpiidelqermadkmyvnfslfqsmpdawgidqlfpvlplegldqvperravllditcdsdgaidhyidgdgiattmpmpeydpenppmlgffmvgayqeilgnmhnlfgdteavdvfvfpdgsvevelsdegdtvadmlqyvqldpktlltqfrdqvkktdldaelqqqfleefeaglygytylede
seqidno:24spec多肽序列
mksmniaasselvsrlsshrrvvalgdtdftdvaavvitaadsrsgilallkrtgfhlpvflysehavelpagvtavingneqqwlelesaacqyeenllppfydtltqyvemgnstfacpghqhgaffkkhpagrhfydffgenvfradmcnadvklgdllihegsakdaqkfaakvfhadktyfvlngtsaankvvtnalltrgdlvlfdrnnhksnhhgaliqagatpvyleasrnpfgfiggidahcfneeylrqqirdvapekadlprpyrlaiiqlgtydgtvynarqvidtvghlcdyilfdsawvgyeqfipmmadssplllelnendpgifvtqsvhkqqagfsqtsqihkkdnhirgqarfcphkrlnnafmlhastspfyplfaaldvnakihegesgrrlwaecveigiearkailarcklfrpfippvvdgklwqdyptsvlasdrrffsfepgakwhgfegyaadqyfvdpcklllttpgidaetgeysdfgvpatilahylrengivpekcdlnsilflltpaesheklaqlvamlaqfeqhieddsplvevlpsvynkypvryrdytlrqlcqemhdlyvsfdvkdlqkamfrqqsfpsvvmnpqdahsayirgdvelvrirdaegriaaegalpyppgvlcvvpgevwggavqryflaleegvnllpgfspelqgvysetdadgvkrlygyvlk
seqidno:25spef多肽序列
msklkiavsdscpdcfttqreciyinesrnidvaaivlslndvtcgkldeidatgygipvfiatenqervpaeylprisgvfencesrrefygrqletaashyetqlrppffralvdyvnqgnsafdcpghqggeffrrhpagnqfveyfgealfradlcnadvamgdllihegapciaqqhaakvfnadktyfvlngtsssnkvvlnalltpgdlvlfdrnnhksnhhgallqagatpvyletarnpygfiggidahcfeesylreliaevapqrakearpfrlaviqlgtydgtiynarqvvdkighlcdyilfdsawvgyeqfipmmadcspllldlnendpgilvtqsvhkqqagfsqtsqihkkdshikgqqryvphkrmnnafmmhastspfyplfaalninakmhegvsgrnmwmdcvvnginarklildncqhirpfvpelvdgkpwqsyetaqiavdlrffqfvpgehwhsfegyaenqyfvdpcklllttpgidarngeyeafgvpatilanflrengvvpekcdlnsilflltpaedmaklqqlvallvrfekllesdaplaevlpsiykqheeryagytlrqlcqemhdlyarhnvkqlqkemfrkehfprvsmnpqeanyaylrgevelvrlpdaegriaaegalpyppgvlcvvpgeiwggavlryfsaleeginllpgfapelqgvyieehdgrkqvwcyvikprdaqstllkgekl
seqidno:26gada多肽序列
mdqklltdfrselldsrfgakaistiaeskrfplhemrddvafqiindelyldgnarqnlatfcqtwddenvhklmdlsinknwidkeeypqsaaidlrcvnmvadlwhapapkngqavgtntigsseacmlggmamkwrwrkrmeaagkptdkpnlvcgpvqicwhkfarywdvelreipmrpgqlfmdpkrmieacdentigvvptfgvtytgnyefpqplhdaldkfqadtgididmhidaasggflapfvapdivwdfrlprvksisasghkfglaplgcgwviwrdeealpqelvfnvdylggqigtfainfsrpagqviaqyyeflrlgregytkvqnasyqvaayladeiaklgpyefictgrpdegipavcfklkdgedpgytlydlserlrlrgwqvpaftlggeatdivvmrimcrrgfemdfaellledykaslkylsdhpklqgiaqqnsfkht
seqidno:27gadb多肽序列
mdkkqvtdlrselldsrfgaksistiaeskrfplhemrddvafqiindelyldgnarqnlatfcqtwddenvhklmdlsinknwidkeeypqsaaidlrcvnmvadlwhapapkngqavgtntigsseacmlggmamkwrwrkrmeaagkptdkpnlvcgpvqicwhkfarywdvelreipmrpgqlfmdpkrmieacdentigvvptfgvtytgnyefpqplhdaldkfqadtgididmhidaasggflapfvapdivwdfrlprvksisasghkfglaplgcgwviwrdeealpqelvfnvdylggqigtfainfsrpagqviaqyyeflrlgregytkvqnasyqvaayladeiaklgpyefictgrpdegipavcfklkdgedpgytlydlserlrlrgwqvpaftlggeatdivvmrimcrrgfemdfaellledykaslkylsdhpklqgiaqqnsfkht
序列表
<110>上海凯赛生物技术研发中心有限公司
cibt美国公司
<120>酸脱羧酶的蛋白与蛋白相互作用的控制
<130>097080-1025710-000600pc
<160>68
<170>patentinversion3.5
<210>1
<211>2148
<212>dna
<213>escherichiacoli
<400>1
atgaacgttattgcaatattgaatcacatgggggtttattttaaagaagaacccatccgt60
gaacttcatcgcgcgcttgaacgtctgaacttccagattgtttacccgaacgaccgtgac120
gacttattaaaactgatcgaaaacaatgcgcgtctgtgcggcgttatttttgactgggat180
aaatataatctcgagctgtgcgaagaaattagcaaaatgaacgagaacctgccgttgtac240
gcgttcgctaatacgtattccactctcgatgtaagcctgaatgacctgcgtttacagatt300
agcttctttgaatatgcgctgggtgctgctgaagatattgctaataagatcaagcagacc360
actgacgaatatatcaacactattctgcctccgctgactaaagcactgtttaaatatgtt420
cgtgaaggtaaatatactttctgtactcctggtcacatgggcggtactgcattccagaaa480
agcccggtaggtagcctgttctatgatttctttggtccgaataccatgaaatctgatatt540
tccatttcagtatctgaactgggttctctgctggatcacagtggtccacacaaagaagca600
gaacagtatatcgctcgcgtctttaacgcagaccgcagctacatggtgaccaacggtact660
tccactgcgaacaaaattgttggtatgtactctgctccagcaggcagcaccattctgatt720
gaccgtaactgccacaaatcgctgacccacctgatgatgatgagcgatgttacgccaatc780
tatttccgcccgacccgtaacgcttacggtattcttggtggtatcccacagagtgaattc840
cagcacgctaccattgctaagcgcgtgaaagaaacaccaaacgcaacctggccggtacat900
gctgtaattaccaactctacctatgatggtctgctgtacaacaccgacttcatcaagaaa960
acactggatgtgaaatccatccactttgactccgcgtgggtgccttacaccaacttctca1020
ccgatttacgaaggtaaatgcggtatgagcggtggccgtgtagaagggaaagtgatttac1080
gaaacccagtccactcacaaactgctggcggcgttctctcaggcttccatgatccacgtt1140
aaaggtgacgtaaacgaagaaacctttaacgaagcctacatgatgcacaccaccacttct1200
ccgcactacggtatcgtggcgtccactgaaaccgctgcggcgatgatgaaaggcaatgca1260
ggtaagcgtctgatcaacggttctattgaacgtgcgatcaaattccgtaaagagatcaaa1320
cgtctgagaacggaatctgatggctggttctttgatgtatggcagccggatcatatcgat1380
acgactgaatgctggccgctgcgttctgacagcacctggcacggcttcaaaaacatcgat1440
aacgagcacatgtatcttgacccgatcaaagtcaccctgctgactccggggatggaaaaa1500
gacggcaccatgagcgactttggtattccggccagcatcgtggcgaaatacctcgacgaa1560
catggcatcgttgttgagaaaaccggtccgtataacctgctgttcctgttcagcatcggt1620
atcgataagaccaaagcactgagcctgctgcgtgctctgactgactttaaacgtgcgttc1680
gacctgaacctgcgtgtgaaaaacatgctgccgtctctgtatcgtgaagatcctgaattc1740
tatgaaaacatgcgtattcaggaactggctcagaatatccacaaactgattgttcaccac1800
aatctgccggatctgatgtatcgcgcatttgaagtgctgccgacgatggtaatgactccg1860
tatgctgcattccagaaagagctgcacggtatgaccgaagaagtttacctcgacgaaatg1920
gtaggtcgtattaacgccaatatgatccttccgtacccgccgggagttcctctggtaatg1980
ccgggtgaaatgatcaccgaagaaagccgtccggttctggagttcctgcagatgctgtgt2040
gaaatcggcgctcactatccgggctttgaaaccgatattcacggtgcataccgtcaggct2100
gatggccgctataccgttaaggtattgaaagaagaaagcaaaaaataa2148
<210>2
<211>715
<212>prt
<213>escherichiacoli
<400>2
metasnvalilealaileleuasnhismetglyvaltyrphelysglu
151015
gluproilearggluleuhisargalaleugluargleuasnphegln
202530
ilevaltyrproasnaspargaspaspleuleulysleuilegluasn
354045
asnalaargleucysglyvalilepheasptrpasplystyrasnleu
505560
gluleucysglugluileserlysmetasngluasnleuproleutyr
65707580
alaphealaasnthrtyrserthrleuaspvalserleuasnaspleu
859095
argleuglnileserphepheglutyralaleuglyalaalagluasp
100105110
ilealaasnlysilelysglnthrthraspglutyrileasnthrile
115120125
leuproproleuthrlysalaleuphelystyrvalarggluglylys
130135140
tyrthrphecysthrproglyhismetglyglythralapheglnlys
145150155160
serprovalglyserleuphetyraspphepheglyproasnthrmet
165170175
lysseraspileserileservalsergluleuglyserleuleuasp
180185190
hisserglyprohislysglualagluglntyrilealaargvalphe
195200205
asnalaaspargsertyrmetvalthrasnglythrserthralaasn
210215220
lysilevalglymettyrseralaproalaglyserthrileleuile
225230235240
aspargasncyshislysserleuthrhisleumetmetmetserasp
245250255
valthrproiletyrpheargprothrargasnalatyrglyileleu
260265270
glyglyileproglnserglupheglnhisalathrilealalysarg
275280285
vallysgluthrproasnalathrtrpprovalhisalavalilethr
290295300
asnserthrtyraspglyleuleutyrasnthrasppheilelyslys
305310315320
thrleuaspvallysserilehispheaspseralatrpvalprotyr
325330335
thrasnpheserproiletyrgluglylyscysglymetserglygly
340345350
argvalgluglylysvaliletyrgluthrglnserthrhislysleu
355360365
leualaalapheserglnalasermetilehisvallysglyaspval
370375380
asnglugluthrpheasnglualatyrmetmethisthrthrthrser
385390395400
prohistyrglyilevalalaserthrgluthralaalaalametmet
405410415
lysglyasnalaglylysargleuileasnglyserilegluargala
420425430
ilelysphearglysgluilelysargleuargthrgluseraspgly
435440445
trpphepheaspvaltrpglnproasphisileaspthrthrglucys
450455460
trpproleuargseraspserthrtrphisglyphelysasnileasp
465470475480
asngluhismettyrleuaspproilelysvalthrleuleuthrpro
485490495
glymetglulysaspglythrmetserasppheglyileproalaser
500505510
ilevalalalystyrleuaspgluhisglyilevalvalglulysthr
515520525
glyprotyrasnleuleupheleupheserileglyileasplysthr
530535540
lysalaleuserleuleuargalaleuthraspphelysargalaphe
545550555560
aspleuasnleuargvallysasnmetleuproserleutyrargglu
565570575
aspprogluphetyrgluasnmetargileglngluleualaglnasn
580585590
ilehislysleuilevalhishisasnleuproaspleumettyrarg
595600605
alaphegluvalleuprothrmetvalmetthrprotyralaalaphe
610615620
glnlysgluleuhisglymetthrglugluvaltyrleuaspglumet
625630635640
valglyargileasnalaasnmetileleuprotyrproproglyval
645650655
proleuvalmetproglyglumetilethrglugluserargproval
660665670
leuglupheleuglnmetleucysgluileglyalahistyrprogly
675680685
phegluthraspilehisglyalatyrargglnalaaspglyargtyr
690695700
thrvallysvalleulysglugluserlyslys
705710715
<210>3
<211>100
<212>prt
<213>saccharomycescerevisiae
<400>3
pheproprolyslysphelysaspleuasnserpheleuaspaspgln
151015
prolysaspproasnleuvalalaserpropheglyglytyrphelys
202530
asnproalaalaaspalaglyserasnasnalaserlyslysserser
354045
tyrglnglnglnargasntrplysglnglyglyasntyrglnglngly
505560
glytyrglnsertyraspserasntyrasnasntyrasnasntyrasn
65707580
asntyrasnasntyrasnasntyrasnasntyrasnlystyrasngly
859095
glnglytyrgln
100
<210>4
<211>102
<212>prt
<213>saccharomycescerevisiae
<400>4
seraspserasnglnglyasnasnglnglnasntyrglnglntyrser
151015
glnasnglyasnglnglnglnglyasnasnargtyrglnglytyrgln
202530
alatyrasnalaglnalaglnproalaglyglytyrtyrglnasntyr
354045
glnglytyrserglytyrglnglnglyglytyrglnglntyrasnpro
505560
glnglyglytyrglnglnaspalaglytyrglnglnglntyrasnpro
65707580
glnglyglytyrglnglntyrasnproglnglyglytyrglnglngln
859095
pheasnproglnglygly
100
<210>5
<211>300
<212>dna
<213>artificialsequence
<220>
<223>new1prionsubunitnucleicacidsequence.codonoptimized.
<400>5
tttccgccgaaaaagttcaaagacctgaactctttcctggacgaccagccgaaagacccg60
aacctggttgcgtctccgttcggtggctacttcaaaaacccagcggcggacgcgggttct120
aacaacgcgtctaagaaatcttcttaccagcagcagcgtaactggaaacagggtggcaac180
tatcagcaaggtggttaccagtcttacgactctaattacaacaactacaacaactacaat240
aactataataactacaacaactacaacaattataacaaatacaacggtcagggctaccag300
<210>6
<211>306
<212>dna
<213>artificialsequence
<220>
<223>sup35prionsubunitnucleicacidsequence.codonoptimized
<400>6
tctgactctaaccaaggtaataaccagcagaactaccaacaatactctcagaacggcaac60
cagcagcagggcaacaaccgctatcaaggctaccaagcgtacaacgcgcaggcacagcca120
gcaggtggctactaccagaattaccagggttactctggttaccagcaaggtggttatcaa180
cagtataatccgcagggcggctatcagcaggacgcaggttaccagcaacaatataaccct240
cagggcggctatcagcaatacaacccgcaaggcggttatcaacaacagtttaacccacag300
ggtggc306
<210>7
<211>105
<212>prt
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkerpolypeptidesequence.thelinker
sequenceisgsgsg(inthefrontofthesequence).
<400>7
glyserglyserglypheproprolyslysphelysaspleuasnser
151015
pheleuaspaspglnprolysaspproasnleuvalalaserprophe
202530
glyglytyrphelysasnproalaalaaspalaglyserasnasnala
354045
serlyslyssersertyrglnglnglnargasntrplysglnglygly
505560
asntyrglnglnglyglytyrglnsertyraspserasntyrasnasn
65707580
tyrasnasntyrasnasntyrasnasntyrasnasntyrasnasntyr
859095
asnlystyrasnglyglnglytyrgln
100105
<210>8
<211>107
<212>prt
<213>artificialsequence
<220>
<223>sup35prionsubunitandlinkerpolypeptidesequence.thelinker
polypeptidesequenceisgsgsg(inthefrontofthesequence)
<400>8
glyserglyserglyseraspserasnglnglyasnasnglnglnasn
151015
tyrglnglntyrserglnasnglyasnglnglnglnglyasnasnarg
202530
tyrglnglytyrglnalatyrasnalaglnalaglnproalaglygly
354045
tyrtyrglnasntyrglnglytyrserglytyrglnglnglyglytyr
505560
glnglntyrasnproglnglyglytyrglnglnaspalaglytyrgln
65707580
glnglntyrasnproglnglyglytyrglnglntyrasnproglngly
859095
glytyrglnglnglnpheasnproglnglygly
100105
<210>9
<211>315
<212>dna
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkernucleicacidsequence.theregion
encodingthelinkerisggttctggctctggt(inthefrontofsequence)
<400>9
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgac60
cagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcg120
gcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactgg180
aaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaac240
tacaacaactacaataactataataactacaacaactacaacaattataacaaatacaac300
ggtcagggctaccag315
<210>10
<211>321
<212>dna
<213>artificialsequence
<220>
<223>sup35prionsubunitandlinkernucleicacidsequence.theregion
encodingthelinkerisggtagcggctctggc(inthefrontofthe
sequence).
<400>10
ggtagcggctctggctctgactctaaccaaggtaataaccagcagaactaccaacaatac60
tctcagaacggcaaccagcagcagggcaacaaccgctatcaaggctaccaagcgtacaac120
gcgcaggcacagccagcaggtggctactaccagaattaccagggttactctggttaccag180
caaggtggttatcaacagtataatccgcagggcggctatcagcaggacgcaggttaccag240
caacaatataaccctcagggcggctatcagcaatacaacccgcaaggcggttatcaacaa300
cagtttaacccacagggtggc321
<210>11
<211>96
<212>prt
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkerwith9aminoacidtruncation
polypeptidesequence.thelinkerisgsgsg(inthefrontofthe
sequence).
<400>11
glyserglyserglyasnserpheleuaspaspglnprolysasppro
151015
asnleuvalalaserpropheglyglytyrphelysasnproalaala
202530
aspalaglyserasnasnalaserlyslyssersertyrglnglngln
354045
argasntrplysglnglyglyasntyrglnglnglyglytyrglnser
505560
tyraspserasntyrasnasntyrasnasntyrasnasntyrasnasn
65707580
tyrasnasntyrasnasntyrasnlystyrasnglyglnglytyrgln
859095
<210>12
<211>87
<212>prt
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkerwith18aminoacidtruncation
polypeptidesequence.thelinkerisgsgsg(inthefrontofthe
sequence).
<400>12
glyserglyserglyaspproasnleuvalalaserpropheglygly
151015
tyrphelysasnproalaalaaspalaglyserasnasnalaserlys
202530
lyssersertyrglnglnglnargasntrplysglnglyglyasntyr
354045
glnglnglyglytyrglnsertyraspserasntyrasnasntyrasn
505560
asntyrasnasntyrasnasntyrasnasntyrasnasntyrasnlys
65707580
tyrasnglyglnglytyrgln
85
<210>13
<211>69
<212>prt
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkerwith36aminoacidtruncation
polypeptidesequence.thelinkerisgsgsg(inthefrontofthe
sequence).
<400>13
glyserglyserglyaspalaglyserasnasnalaserlyslysser
151015
sertyrglnglnglnargasntrplysglnglyglyasntyrglngln
202530
glyglytyrglnsertyraspserasntyrasnasntyrasnasntyr
354045
asnasntyrasnasntyrasnasntyrasnasntyrasnlystyrasn
505560
glyglnglytyrgln
65
<210>14
<211>60
<212>prt
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkerwith45aminoacidtruncation
polypeptidesequence.thelinkerisgsgsg(inthefrontofthe
sequence).
<400>14
glyserglyserglylyssersertyrglnglnglnargasntrplys
151015
glnglyglyasntyrglnglnglyglytyrglnsertyraspserasn
202530
tyrasnasntyrasnasntyrasnasntyrasnasntyrasnasntyr
354045
asnasntyrasnlystyrasnglyglnglytyrgln
505560
<210>15
<211>115
<212>prt
<213>artificialsequence
<220>
<223>new1priondomainfragmentandlinkerwithbstc-terminal
fragmentpolypeptidesequence.thelinkerisgsgsg(inthefrontof
thesequence).
<400>15
glyserglyserglypheproprolyslysphelysaspleuasnser
151015
pheleuaspaspglnprolysaspproasnleuvalalaserprophe
202530
glyglytyrphelysasnproalaalaaspalaglyserasnasnala
354045
serlyslyssersertyrglnglnglnargasntrplysglnglygly
505560
asntyrglnglnglyglytyrglnsertyraspserasntyrasnasn
65707580
tyrasnasntyrasnasntyrasnasntyrasnasntyrasnasntyr
859095
asnlystyrasnglyglnglytyrglnargargpheglyglualaser
100105110
seralaphe
115
<210>16
<211>345
<212>dna
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkerwithbstc-terminalfragment
nucleicacidsequence.theregionencodingthelinkeris
ggttctggctctggt(inthefrontofthesequence).
<400>16
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgac60
cagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcg120
gcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactgg180
aaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaac240
tacaacaactacaataactataataactacaacaactacaacaattataacaaatacaac300
ggtcagggctaccagcgtcgtttcggcgaagcgagcagcgcgttc345
<210>17
<211>115
<212>prt
<213>artificialsequence
<220>
<223>new1prionsubunitandlinkerwithλcic-terminalfragment
polypeptidesequence.thelinkerisgsgsg(inthefrontofthe
sequence).
<400>17
glyserglyserglypheproprolyslysphelysaspleuasnser
151015
pheleuaspaspglnprolysaspproasnleuvalalaserprophe
202530
glyglytyrphelysasnproalaalaaspalaglyserasnasnala
354045
serlyslyssersertyrglnglnglnargasntrplysglnglygly
505560
asntyrglnglnglyglytyrglnsertyraspserasntyrasnasn
65707580
tyrasnasntyrasnasntyrasnasntyrasnasntyrasnasntyr
859095
asnlystyrasnglyglnglytyrglnalaserglntrpprogluglu
100105110
thrphegly
115
<210>18
<211>345
<212>dna
<213>artificialsequence
<220>
<223>new1priondomainfragmentandlinkerwithλcic-terminal
fragmentnucleicacidsequence.theregionencodingthelinkeris
ggttctggctctggt(inthefrontofthesequence).
<400>18
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgac60
cagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcg120
gcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactgg180
aaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaac240
tacaacaactacaataactataataactacaacaactacaacaattataacaaatacaac300
ggtcagggctaccaggcgagccagtggccggaagaaaccttcggc345
<210>19
<211>115
<212>prt
<213>artificialsequence
<220>
<223>new1priondomainfragmentandlinkerwithrecac-terminal
fragmentpolypeptidesequence.thelinkerisgsgsg(inthefrontof
thesequence).
<400>19
glyserglyserglypheproprolyslysphelysaspleuasnser
151015
pheleuaspaspglnprolysaspproasnleuvalalaserprophe
202530
glyglytyrphelysasnproalaalaaspalaglyserasnasnala
354045
serlyslyssersertyrglnglnglnargasntrplysglnglygly
505560
asntyrglnglnglyglytyrglnsertyraspserasntyrasnasn
65707580
tyrasnasntyrasnasntyrasnasntyrasnasntyrasnasntyr
859095
asnlystyrasnglyglnglytyrglngluglyvalalagluthrasn
100105110
gluaspphe
115
<210>20
<211>345
<212>dna
<213>artificialsequence
<220>
<223>new1subunitandlinkerwithrecac-terminalfragmentnucleic
acidsequence.theregionencodingthelinkeris
ggttctggctctggt(inthefrontofthesequence).
<400>20
ggttctggctctggttttccgccgaaaaagttcaaagacctgaactctttcctggacgac60
cagccgaaagacccgaacctggttgcgtctccgttcggtggctacttcaaaaacccagcg120
gcggacgcgggttctaacaacgcgtctaagaaatcttcttaccagcagcagcgtaactgg180
aaacagggtggcaactatcagcaaggtggttaccagtcttacgactctaattacaacaac240
tacaacaactacaataactataataactacaacaactacaacaattataacaaatacaac300
ggtcagggctaccaggaaggcgtggcggaaaccaacgaagatttc345
<210>21
<211>713
<212>prt
<213>escherichiacoli
<400>21
metasnileilealailemetglyprohisglyvalphetyrlysasp
151015
gluproilelysgluleugluseralaleuvalalaglnglyphegln
202530
ileiletrpproglnasnservalaspleuleulyspheilegluhis
354045
asnproargilecysglyvalilepheasptrpaspglutyrserleu
505560
aspleucysseraspileasnglnleuasnglutyrleuproleutyr
65707580
alapheileasnthrhisserthrmetaspvalservalglnaspmet
859095
argmetalaleutrpphepheglutyralaleuglyglnalagluasp
100105110
ilealaileargmetargglntyrthraspglutyrleuaspasnile
115120125
thrproprophethrlysalaleuphethrtyrvallysgluarglys
130135140
tyrthrphecysthrproglyhismetglyglythralatyrglnlys
145150155160
serprovalglycysleuphetyraspphepheglyglyasnthrleu
165170175
lysalaaspvalserileservalthrgluleuglyserleuleuasp
180185190
histhrglyprohisleuglualagluglutyrilealaargthrphe
195200205
glyalagluglnsertyrilevalthrasnglythrserthrserasn
210215220
lysilevalglymettyralaalaproserglyserthrleuleuile
225230235240
aspargasncyshislysserleualahisleuleumetmetasnasp
245250255
valvalprovaltrpleulysprothrargasnalaleuglyileleu
260265270
glyglyileproargarggluphethrargaspserilegluglulys
275280285
valalaalathrthrglnalaglntrpprovalhisalavalilethr
290295300
asnserthrtyraspglyleuleutyrasnthrasptrpilelysgln
305310315320
thrleuaspvalproserilehispheaspseralatrpvalprotyr
325330335
thrhisphehisproiletyrglnglylysserglymetserglyglu
340345350
argvalalaglylysvalilephegluthrglnserthrhislysmet
355360365
leualaalaleuserglnalaserleuilehisilelysglyglutyr
370375380
aspgluglualapheasnglualaphemetmethisthrthrthrser
385390395400
prosertyrproilevalalaservalgluthralaalaalametleu
405410415
argglyasnproglylysargleuileasnargservalgluargala
420425430
leuhisphearglysgluvalglnargleuargglugluseraspgly
435440445
trpphepheaspiletrpglnproproglnvalaspglualaglucys
450455460
trpprovalalaproglygluglntrphisglypheasnaspalaasp
465470475480
alaasphismetpheleuaspprovallysvalthrileleuthrpro
485490495
glymetaspgluglnglyasnmetserglugluglyileproalaala
500505510
leuvalalalyspheleuaspgluargglyilevalvalglulysthr
515520525
glyprotyrasnleuleupheleupheserileglyileasplysthr
530535540
lysalametglyleuleuargglyleuthrgluphelysargsertyr
545550555560
aspleuasnleuargilelysasnmetleuproaspleutyralaglu
565570575
aspproaspphetyrargasnmetargileglnaspleualaglngly
580585590
ilehislysleuilearglyshisaspleuproglyleumetleuarg
595600605
alapheaspthrleuproglumetilemetthrprohisglnalatrp
610615620
glnargglnilelysglygluvalgluthrilealaleugluglnleu
625630635640
valglyargvalseralaasnmetileleuprotyrproproglyval
645650655
proleuleumetproglyglumetleuthrlysgluserargthrval
660665670
leuasppheleuleumetleucysservalglyglnhistyrprogly
675680685
phegluthraspilehisglyalalysglnaspgluaspglyvaltyr
690695700
argvalargvalleulysmetalagly
705710
<210>22
<211>755
<212>prt
<213>escherichiacoli
<400>22
metlysvalleuilevalgluserglupheleuhisglnaspthrtrp
151015
valglyasnalavalgluargleualaaspalaleuserglnglnasn
202530
valthrvalilelysserthrserpheaspaspglyphealaileleu
354045
serserasnglualaileaspcysleumetphesertyrglnmetglu
505560
hisproaspgluhisglnasnvalargglnleuileglylysleuhis
65707580
gluargglnglnasnvalprovalpheleuleuglyaspargglulys
859095
alaleualaalametaspargaspleuleugluleuvalaspgluphe
100105110
alatrpileleugluaspthralaasppheilealaglyargalaval
115120125
alaalametthrargtyrargglnglnleuleuproproleupheser
130135140
alaleumetlystyrseraspilehisglutyrsertrpalaalapro
145150155160
glyhisglnglyglyvalglyphethrlysthrproalaglyargphe
165170175
tyrhisasptyrtyrglygluasnleupheargthraspmetglyile
180185190
gluargthrserleuglyserleuleuasphisthrglyalaphegly
195200205
gluserglulystyralaalaargvalpheglyalaaspargsertrp
210215220
servalvalvalglythrserglyserasnargthrilemetglnala
225230235240
cysmetthraspasnaspvalvalvalvalaspargasncyshislys
245250255
serilegluglnglyleumetleuthrglyalalysprovaltyrmet
260265270
valproserargasnargtyrglyileileglyproiletyrprogln
275280285
glumetglnprogluthrleuglnlyslysilesergluserproleu
290295300
thrlysasplysalaglyglnlysprosertyrcysvalvalthrasn
305310315320
cysthrtyraspglyvalcystyrasnalalysglualaglnaspleu
325330335
leuglulysthrseraspargleuhispheaspglualatrptyrgly
340345350
tyralaargpheasnproiletyralaasphistyralametarggly
355360365
gluproglyasphisasnglyprothrvalphealathrhisserthr
370375380
hislysleuleuasnalaleuserglnalasertyrilehisvalarg
385390395400
gluglyargglyalaileasnpheserargpheasnglnalatyrmet
405410415
methisalathrthrserproleutyralailecysalaserasnasp
420425430
valalavalsermetmetaspglyasnserglyleuserleuthrgln
435440445
gluvalileaspglualavalasppheargglnalametalaargleu
450455460
tyrlysgluphethralaaspglysertrpphephelysprotrpasn
465470475480
lysgluvalvalthraspproglnthrglylysthrtyrasppheala
485490495
aspalaprothrlysleuleuthrthrvalglnaspcystrpvalmet
500505510
hisproglyglusertrphisglyphelysaspileproaspasntrp
515520525
sermetleuaspproilelysvalserileleualaproglymetgly
530535540
gluaspglygluleuglugluthrglyvalproalaalaleuvalthr
545550555560
alatrpleuglyarghisglyilevalprothrargthrthraspphe
565570575
glnilemetpheleuphesermetglyvalthrargglylystrpgly
580585590
thrleuvalasnthrleucysserphelysarghistyraspalaasn
595600605
thrproleualaglnvalmetprogluleuvalgluglntyrproasp
610615620
thrtyralaasnmetglyilehisaspleuglyaspthrmetpheala
625630635640
trpleulysgluasnasnproglyalaargleuasnglualatyrser
645650655
glyleuprovalalagluvalthrproargglualatyrasnalaile
660665670
valaspasnasnvalgluleuvalserilegluasnleuproglyarg
675680685
ilealaalaasnservalileprotyrproproglyileprometleu
690695700
leuserglygluasnpheglyasplysasnserproglnvalsertyr
705710715720
leuargserleuglnsertrpasphishispheproglyphegluhis
725730735
gluthrgluglythrgluileileaspglyiletyrhisvalmetcys
740745750
vallysala
755
<210>23
<211>658
<212>prt
<213>escherichiacoli
<400>23
metseraspaspmetsermetglyleuproserseralaglygluhis
151015
glyvalleuargsermetglngluvalalametserserglngluala
202530
serlysmetleuargthrtyrasnilealatrptrpglyasnasntyr
354045
tyraspvalasngluleuglyhisileservalcysproaspproasp
505560
valproglualaargvalaspleualaglnleuvallysthrargglu
65707580
alaglnglyglnargleuproalaleuphecyspheproglnileleu
859095
glnhisargleuargserileasnalaalaphelysargalaargglu
100105110
sertyrglytyrasnglyasptyrpheleuvaltyrproilelysval
115120125
asnglnhisargargvalilegluserleuilehisserglyglupro
130135140
leuglyleuglualaglyserlysalagluleumetalavalleuala
145150155160
hisalaglymetthrargservalilevalcysasnglytyrlysasp
165170175
argglutyrileargleualaleuileglyglulysmetglyhislys
180185190
valtyrleuvalileglulysmetsergluilealailevalleuasp
195200205
glualagluargleuasnvalvalproargleuglyvalargalaarg
210215220
leualaserglnglyserglylystrpglnserserglyglyglulys
225230235240
serlyspheglyleualaalathrglnvalleuglnleuvalgluthr
245250255
leuargglualaglyargleuaspserleuglnleuleuhisphehis
260265270
leuglyserglnmetalaasnileargaspilealathrglyvalarg
275280285
gluseralaargphetyrvalgluleuhislysleuglyvalasnile
290295300
glncyspheaspvalglyglyglyleuglyvalasptyrgluglythr
305310315320
argserglnseraspcysservalasntyrglyleuasnglutyrala
325330335
asnasnileiletrpalaileglyaspalacysglugluasnglyleu
340345350
prohisprothrvalilethrgluserglyargalavalthralahis
355360365
histhrvalleuvalserasnileileglyvalgluargasnglutyr
370375380
thrvalprothralaproalagluaspalaproargalaleuglnser
385390395400
mettrpgluthrtrpglnglumethisgluproglythrargargser
405410415
leuargglutrpleuhisaspserglnmetaspleuhisaspilehis
420425430
ileglytyrserserglyilepheserleuglngluargalatrpala
435440445
gluglnleutyrleusermetcyshisgluvalglnlysglnleuasp
450455460
proglnasnargalahisargproileileaspgluleuglngluarg
465470475480
metalaasplysmettyrvalasnpheserleupheglnsermetpro
485490495
aspalatrpglyileaspglnleupheprovalleuproleuglugly
500505510
leuaspglnvalprogluargargalavalleuleuaspilethrcys
515520525
aspseraspglyalaileasphistyrileaspglyaspglyileala
530535540
thrthrmetprometproglutyraspprogluasnproprometleu
545550555560
glyphephemetvalglyalatyrglngluileleuglyasnmethis
565570575
asnleupheglyaspthrglualavalaspvalphevalpheproasp
580585590
glyservalgluvalgluleuseraspgluglyaspthrvalalaasp
595600605
metleuglntyrvalglnleuaspprolysthrleuleuthrglnphe
610615620
argaspglnvallyslysthraspleuaspalagluleuglnglngln
625630635640
pheleugluglupheglualaglyleutyrglytyrthrtyrleuglu
645650655
aspglu
<210>24
<211>711
<212>prt
<213>escherichiacoli
<400>24
metlyssermetasnilealaalasersergluleuvalserargleu
151015
serserhisargargvalvalalaleuglyaspthraspphethrasp
202530
valalaalavalvalilethralaalaaspserargserglyileleu
354045
alaleuleulysargthrglyphehisleuprovalpheleutyrser
505560
gluhisalavalgluleuproalaglyvalthralavalileasngly
65707580
asngluglnglntrpleugluleugluseralaalacysglntyrglu
859095
gluasnleuleuproprophetyraspthrleuthrglntyrvalglu
100105110
metglyasnserthrphealacysproglyhisglnhisglyalaphe
115120125
phelyslyshisproalaglyarghisphetyraspphepheglyglu
130135140
asnvalpheargalaaspmetcysasnalaaspvallysleuglyasp
145150155160
leuleuilehisgluglyseralalysaspalaglnlysphealaala
165170175
lysvalphehisalaasplysthrtyrphevalleuasnglythrser
180185190
alaalaasnlysvalvalthrasnalaleuleuthrargglyaspleu
195200205
valleupheaspargasnasnhislysserasnhishisglyalaleu
210215220
ileglnalaglyalathrprovaltyrleuglualaserargasnpro
225230235240
pheglypheileglyglyileaspalahiscyspheasngluglutyr
245250255
leuargglnglnileargaspvalalaproglulysalaaspleupro
260265270
argprotyrargleualaileileglnleuglythrtyraspglythr
275280285
valtyrasnalaargglnvalileaspthrvalglyhisleucysasp
290295300
tyrileleupheaspseralatrpvalglytyrgluglnpheilepro
305310315320
metmetalaaspserserproleuleuleugluleuasngluasnasp
325330335
proglyilephevalthrglnservalhislysglnglnalaglyphe
340345350
serglnthrserglnilehislyslysaspasnhisileargglygln
355360365
alaargphecysprohislysargleuasnasnalaphemetleuhis
370375380
alaserthrserprophetyrproleuphealaalaleuaspvalasn
385390395400
alalysilehisgluglygluserglyargargleutrpalaglucys
405410415
valgluileglyileglualaarglysalaileleualaargcyslys
420425430
leupheargpropheileproprovalvalaspglylysleutrpgln
435440445
asptyrprothrservalleualaseraspargargphepheserphe
450455460
gluproglyalalystrphisglyphegluglytyralaalaaspgln
465470475480
tyrphevalaspprocyslysleuleuleuthrthrproglyileasp
485490495
alagluthrglyglutyrserasppheglyvalproalathrileleu
500505510
alahistyrleuarggluasnglyilevalproglulyscysaspleu
515520525
asnserileleupheleuleuthrproalagluserhisglulysleu
530535540
alaglnleuvalalametleualaglnphegluglnhisilegluasp
545550555560
aspserproleuvalgluvalleuproservaltyrasnlystyrpro
565570575
valargtyrargasptyrthrleuargglnleucysglnglumethis
580585590
aspleutyrvalserpheaspvallysaspleuglnlysalametphe
595600605
argglnglnserpheproservalvalmetasnproglnaspalahis
610615620
seralatyrileargglyaspvalgluleuvalargileargaspala
625630635640
gluglyargilealaalagluglyalaleuprotyrproproglyval
645650655
leucysvalvalproglygluvaltrpglyglyalavalglnargtyr
660665670
pheleualaleuglugluglyvalasnleuleuproglypheserpro
675680685
gluleuglnglyvaltyrsergluthraspalaaspglyvallysarg
690695700
leutyrglytyrvalleulys
705710
<210>25
<211>732
<212>prt
<213>escherichiacoli
<400>25
metserlysleulysilealavalseraspsercysproaspcysphe
151015
thrthrglnargglucysiletyrileasngluserargasnileasp
202530
valalaalailevalleuserleuasnaspvalthrcysglylysleu
354045
aspgluileaspalathrglytyrglyileprovalpheilealathr
505560
gluasnglngluargvalproalaglutyrleuproargilesergly
65707580
valphegluasncysgluserargarggluphetyrglyargglnleu
859095
gluthralaalaserhistyrgluthrglnleuargproprophephe
100105110
argalaleuvalasptyrvalasnglnglyasnseralapheaspcys
115120125
proglyhisglnglyglygluphepheargarghisproalaglyasn
130135140
glnphevalglutyrpheglyglualaleupheargalaaspleucys
145150155160
asnalaaspvalalametglyaspleuleuilehisgluglyalapro
165170175
cysilealaglnglnhisalaalalysvalpheasnalaasplysthr
180185190
tyrphevalleuasnglythrserserserasnlysvalvalleuasn
195200205
alaleuleuthrproglyaspleuvalleupheaspargasnasnhis
210215220
lysserasnhishisglyalaleuleuglnalaglyalathrproval
225230235240
tyrleugluthralaargasnprotyrglypheileglyglyileasp
245250255
alahiscysphegluglusertyrleuarggluleuilealagluval
260265270
alaproglnargalalysglualaargpropheargleualavalile
275280285
glnleuglythrtyraspglythriletyrasnalaargglnvalval
290295300
asplysileglyhisleucysasptyrileleupheaspseralatrp
305310315320
valglytyrgluglnpheileprometmetalaaspcysserproleu
325330335
leuleuaspleuasngluasnaspproglyileleuvalthrglnser
340345350
valhislysglnglnalaglypheserglnthrserglnilehislys
355360365
lysaspserhisilelysglyglnglnargtyrvalprohislysarg
370375380
metasnasnalaphemetmethisalaserthrserprophetyrpro
385390395400
leuphealaalaleuasnileasnalalysmethisgluglyvalser
405410415
glyargasnmettrpmetaspcysvalvalasnglyileasnalaarg
420425430
lysleuileleuaspasncysglnhisileargprophevalproglu
435440445
leuvalaspglylysprotrpglnsertyrgluthralaglnileala
450455460
valaspleuargphepheglnphevalproglygluhistrphisser
465470475480
phegluglytyralagluasnglntyrphevalaspprocyslysleu
485490495
leuleuthrthrproglyileaspalaargasnglyglutyrgluala
500505510
pheglyvalproalathrileleualaasnpheleuarggluasngly
515520525
valvalproglulyscysaspleuasnserileleupheleuleuthr
530535540
proalagluaspmetalalysleuglnglnleuvalalaleuleuval
545550555560
argpheglulysleuleugluseraspalaproleualagluvalleu
565570575
proseriletyrlysglnhisglugluargtyralaglytyrthrleu
580585590
argglnleucysglnglumethisaspleutyralaarghisasnval
595600605
lysglnleuglnlysglumetphearglysgluhispheproargval
610615620
sermetasnproglnglualaasntyralatyrleuargglygluval
625630635640
gluleuvalargleuproaspalagluglyargilealaalaglugly
645650655
alaleuprotyrproproglyvalleucysvalvalproglygluile
660665670
trpglyglyalavalleuargtyrpheseralaleuglugluglyile
675680685
asnleuleuproglyphealaprogluleuglnglyvaltyrileglu
690695700
gluhisaspglyarglysglnvaltrpcystyrvalilelysproarg
705710715720
aspalaglnserthrleuleulysglyglulysleu
725730
<210>26
<211>466
<212>prt
<213>escherichiacoli
<400>26
metaspglnlysleuleuthrasppheargsergluleuleuaspser
151015
argpheglyalalysalaileserthrilealagluserlysargphe
202530
proleuhisglumetargaspaspvalalapheglnileileasnasp
354045
gluleutyrleuaspglyasnalaargglnasnleualathrphecys
505560
glnthrtrpaspaspgluasnvalhislysleumetaspleuserile
65707580
asnlysasntrpileasplysgluglutyrproglnseralaalaile
859095
aspleuargcysvalasnmetvalalaaspleutrphisalaproala
100105110
prolysasnglyglnalavalglythrasnthrileglyserserglu
115120125
alacysmetleuglyglymetalametlystrpargtrparglysarg
130135140
metglualaalaglylysprothrasplysproasnleuvalcysgly
145150155160
provalglnilecystrphislysphealaargtyrtrpaspvalglu
165170175
leuarggluileprometargproglyglnleuphemetaspprolys
180185190
argmetileglualacysaspgluasnthrileglyvalvalprothr
195200205
pheglyvalthrtyrthrglyasntyrglupheproglnproleuhis
210215220
aspalaleuasplyspheglnalaaspthrglyileaspileaspmet
225230235240
hisileaspalaalaserglyglypheleualaprophevalalapro
245250255
aspilevaltrpasppheargleuproargvallysserileserala
260265270
serglyhislyspheglyleualaproleuglycysglytrpvalile
275280285
trpargaspgluglualaleuproglngluleuvalpheasnvalasp
290295300
tyrleuglyglyglnileglythrphealaileasnpheserargpro
305310315320
alaglyglnvalilealaglntyrtyrglupheleuargleuglyarg
325330335
gluglytyrthrlysvalglnasnalasertyrglnvalalaalatyr
340345350
leualaaspgluilealalysleuglyprotyrglupheilecysthr
355360365
glyargproaspgluglyileproalavalcysphelysleulysasp
370375380
glygluaspproglytyrthrleutyraspleusergluargleuarg
385390395400
leuargglytrpglnvalproalaphethrleuglyglyglualathr
405410415
aspilevalvalmetargilemetcysargargglypheglumetasp
420425430
phealagluleuleuleugluasptyrlysalaserleulystyrleu
435440445
serasphisprolysleuglnglyilealaglnglnasnserphelys
450455460
histhr
465
<210>27
<211>466
<212>prt
<213>escherichiacoli
<400>27
metasplyslysglnvalthraspleuargsergluleuleuaspser
151015
argpheglyalalysserileserthrilealagluserlysargphe
202530
proleuhisglumetargaspaspvalalapheglnileileasnasp
354045
gluleutyrleuaspglyasnalaargglnasnleualathrphecys
505560
glnthrtrpaspaspgluasnvalhislysleumetaspleuserile
65707580
asnlysasntrpileasplysgluglutyrproglnseralaalaile
859095
aspleuargcysvalasnmetvalalaaspleutrphisalaproala
100105110
prolysasnglyglnalavalglythrasnthrileglyserserglu
115120125
alacysmetleuglyglymetalametlystrpargtrparglysarg
130135140
metglualaalaglylysprothrasplysproasnleuvalcysgly
145150155160
provalglnilecystrphislysphealaargtyrtrpaspvalglu
165170175
leuarggluileprometargproglyglnleuphemetaspprolys
180185190
argmetileglualacysaspgluasnthrileglyvalvalprothr
195200205
pheglyvalthrtyrthrglyasntyrglupheproglnproleuhis
210215220
aspalaleuasplyspheglnalaaspthrglyileaspileaspmet
225230235240
hisileaspalaalaserglyglypheleualaprophevalalapro
245250255
aspilevaltrpasppheargleuproargvallysserileserala
260265270
serglyhislyspheglyleualaproleuglycysglytrpvalile
275280285
trpargaspgluglualaleuproglngluleuvalpheasnvalasp
290295300
tyrleuglyglyglnileglythrphealaileasnpheserargpro
305310315320
alaglyglnvalilealaglntyrtyrglupheleuargleuglyarg
325330335
gluglytyrthrlysvalglnasnalasertyrglnvalalaalatyr
340345350
leualaaspgluilealalysleuglyprotyrglupheilecysthr
355360365
glyargproaspgluglyileproalavalcysphelysleulysasp
370375380
glygluaspproglytyrthrleutyraspleusergluargleuarg
385390395400
leuargglytrpglnvalproalaphethrleuglyglyglualathr
405410415
aspilevalvalmetargilemetcysargargglypheglumetasp
420425430
phealagluleuleuleugluasptyrlysalaserleulystyrleu
435440445
serasphisprolysleuglnglyilealaglnglnasnserphelys
450455460
histhr
465
<210>28
<211>19
<212>prt
<213>saccharomycescerevisiae
<400>28
glnglnglnargasntrplysglnglyglyasntyrglnglntyrgln
151015
sertyrasn
<210>29
<211>30
<212>prt
<213>saccharomycescerevisiae
<400>29
serasntyrasnasntyrasnasntyrasnasntyrasnasntyrasn
151015
asntyrasnasntyrasnlystyrasnglyglnglytyrgln
202530
<210>30
<211>8
<212>prt
<213>saccharomycescerevisiae
<400>30
proglnglyglytyrglnglnasn
15
<210>31
<211>9
<212>prt
<213>saccharomycescerevisiae
<400>31
pheproprolyslysphelysaspleu
15
<210>32
<211>18
<212>prt
<213>saccharomycescerevisiae
<400>32
pheproprolyslysphelysaspleuasnserpheleuaspaspgln
151015
prolys
<210>33
<211>36
<212>prt
<213>saccharomycescerevisiae
<400>33
pheproprolyslysphelysaspleuasnserpheleuaspaspgln
151015
prolysaspproasnleuvalalaserpropheglyglytyrphelys
202530
asnproalaala
35
<210>34
<211>45
<212>prt
<213>saccharomycescerevisiae
<400>34
pheproprolyslysphelysaspleuasnserpheleuaspaspgln
151015
prolysaspproasnleuvalalaserpropheglyglytyrphelys
202530
asnproalaalaaspalaglyserasnasnalaserlys
354045
<210>35
<211>10
<212>prt
<213>bovineadenovirustype1
<400>35
argargpheglyglualaserseralaphe
1510
<210>36
<211>10
<212>prt
<213>escherichiacoli
<400>36
alaserglntrpproglugluthrphegly
1510
<210>37
<211>10
<212>prt
<213>escherichiacoli
<400>37
gluglyvalalagluthrasngluaspphe
1510
<210>38
<211>52
<212>dna
<213>artificialsequence
<220>
<223>primercada-f
<400>38
ggcgagctcacacaggaaacagaccatgaacgttattgcaatattgaatcac52
<210>39
<211>28
<212>dna
<213>artificialsequence
<220>
<223>primercada-r
<400>39
ggctctagaccacttcccttgtacgagc28
<210>40
<211>44
<212>dna
<213>artificialsequence
<220>
<223>primercada-f2
<400>40
atttcacacaggaaacagctatgaacgttattgcaatattgaat44
<210>41
<211>20
<212>dna
<213>artificialsequence
<220>
<223>primercada-r2
<400>41
agctgtttcctgtgtgaaat20
<210>42
<211>33
<212>dna
<213>artificialsequence
<220>
<223>primercat-hindiii-f
<400>42
ggcaagcttgagaaaaaaatcactggatatacc33
<210>43
<211>29
<212>dna
<213>artificialsequence
<220>
<223>primercat-ndei-r
<400>43
ggccatatgtaagggcaccaataactgcc29
<210>44
<211>31
<212>dna
<213>artificialsequence
<220>
<223>primercadat-xbai-r
<400>44
ggctctagatttgctttcttctttcaatacc31
<210>45
<211>33
<212>dna
<213>artificialsequence
<220>
<223>primercat-xbai-f
<400>45
ggctctagagagaaaaaaatcactggatatacc33
<210>46
<211>30
<212>dna
<213>artificialsequence
<220>
<223>primernew1-xbai-f
<400>46
ggctctagaggttctggctctggttctccg30
<210>47
<211>31
<212>dna
<213>artificialsequence
<220>
<223>primernew1-hindiii-r
<400>47
ggcaagcttttactggtagccctgaccgttg31
<210>48
<211>29
<212>dna
<213>artificialsequence
<220>
<223>primersup35-xbai-f
<400>48
ggctctagaggtagcggctctggctctga29
<210>49
<211>32
<212>dna
<213>artificialsequence
<220>
<223>primersup35-hindiii-r
<400>49
ggcaagcttttagccaccctgtgggttaaact32
<210>50
<211>29
<212>dna
<213>artificialsequence
<220>
<223>primercada-xbai-f
<400>50
ggctctagaatttcacacaggaaacagct29
<210>51
<211>29
<212>dna
<213>artificialsequence
<220>
<223>primercada-hindiii-r
<400>51
ggcaagcttcacttcccttgtacgagcta29
<210>52
<211>34
<212>dna
<213>artificialsequence
<220>
<223>primerrbs2-saci-f
<400>52
ggcgagctcatgaacgttattgcaatattgaatc34
<210>53
<211>25
<212>dna
<213>artificialsequence
<220>
<223>primerrbs2-saci-r
<400>53
ggcgagctcctcctgtgtgaaattg25
<210>54
<211>29
<212>dna
<213>artificialsequence
<220>
<223>primernew1-saci-f
<400>54
ggcgagctcatgggttctggctctggttc29
<210>55
<211>28
<212>dna
<213>artificialsequence
<220>
<223>primernew1-xbai-r
<400>55
ggctctagactggtagccctgaccgttg28
<210>56
<211>27
<212>dna
<213>artificialsequence
<220>
<223>primersup35-saci-f
<400>56
ggcgagctcatgggtagcggctctggc27
<210>57
<211>29
<212>dna
<213>artificialsequence
<220>
<223>primersup35-xbai-r
<400>57
ggctctagagccaccctgtgggttaaact29
<210>58
<211>28
<212>dna
<213>artificialsequence
<220>
<223>primer222-de-r
<400>58
tctagatttgctttcttctttcaatacc28
<210>59
<211>46
<212>dna
<213>artificialsequence
<220>
<223>primer222-de-9-f
<400>59
gaagaaagcaaatctagaaagttcaaagacctgaactctttcggtg46
<210>60
<211>42
<212>dna
<213>artificialsequence
<220>
<223>primer222-de-18-f
<400>60
gaagaaagcaaatctagagacgaccagccgaaagacccgaac42
<210>61
<211>41
<212>dna
<213>artificialsequence
<220>
<223>primer222-de-36-f
<400>61
gaagaaagcaaatctagaaaaaacccagcggcggacgcggg41
<210>62
<211>41
<212>dna
<213>artificialsequence
<220>
<223>primer222-de-45-f
<400>62
gaagaaagcaaatctagaaacaacgcgtctaagaaatcttc41
<210>63
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer222-c2-f
<400>63
tttcggcgaagcgagcagcgcgttctaaaagcttaagagacaggatg47
<210>64
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer222-c2-r
<400>64
cgctgctcgcttcgccgaaacgacgctggtagccctgaccgttgtat47
<210>65
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer222-c4-f
<400>65
ccagtggccggaagaaaccttcggctaaaagcttaagagacaggatg47
<210>66
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer222-c4-r
<400>66
aggtttcttccggccactggctcgcctggtagccctgaccgttgtat47
<210>67
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer222-c6-f
<400>67
cgtggcggaaaccaacgaagatttctaaaagcttaagagacaggatg47
<210>68
<211>47
<212>dna
<213>artificialsequence
<220>
<223>primer222-c6-r
<400>68
cttcgttggtttccgccacgccttcctggtagccctgaccgttgtat47
- 还没有人留言评论。精彩留言会获得点赞!