紫花苜蓿ILP分子标记引物及在苜蓿品种鉴定中的应用的制作方法
本发明属于生物技术领域,具体涉及紫花苜蓿ILP分子标记引物及在苜蓿品种鉴定中的应用。
背景技术:
紫花苜蓿(Medicago sativa L.)是全球种植面积最大的豆科牧草,具有广泛的生态适应性、稳定的生产力及较高的营养价值,具有“牧草之王”的称号,在我国农牧业生产和生态经济建设中发挥着巨大作用。近年来,随着畜牧业的快速发展,我国在苜蓿品种(包括杂花苜蓿)培育和审定工作方面也取得了重要进展。目前,苜蓿新品种的审定和品种真实性的鉴定方法主要是通过田间种植,根据其形态学特征进行鉴别。但这种方法费时费力、容易受环境和主观因素的影响。另外,由于苜蓿品种选育过程中所用的亲本较为集中,从而导致育成品种间性状差异日趋狭窄,使得通过形态学特征进行品种鉴定变得更加困难。因此,建立一套快速、精确、廉价的苜蓿品种鉴定技术,显得非常必要。
随着现代分子生物学的发展,分子标记越来越多的被应用于品种鉴定中,如以PCR反应为基础的RAPD、SCAR、SSR、ILP等。与其它分子标记相比,ILP标记具有许多独特的优点,例如,ILP标记引物设计于相对保守的外显子区域,具有较强的通用性、特异性、共显性、中性、可靠性以及功能相关性等特点。此外,ILP标记变异适中,可以用于品种鉴定、系统发育分析、遗传多样性分析以及遗传连锁作图等方面。
作为一种新型的分子标记,迄今为止,ILP标记主要在水稻、玉米、大豆等模式植物中中得到开发并主要应用于遗传多样性分析和遗传连锁图谱构建。如赵建亚等利用ILP标记研究了52份籼粳杂交水稻的遗传多样性(参见《石河子大学学报》,2009年第27卷第3期,《利用ILP分子标记分析籼粳杂交水稻的遗传多样性》)。但在非模式植物苜蓿中,ILP引物的开发以及在品种鉴定中的应用尚未见报道。因此,在全基因组水平开发紫花苜蓿ILP分子标记并研究其在苜蓿品种鉴定中的应用,对苜蓿品种鉴定以及分子标记辅助育种具有重要意义。
技术实现要素:
为解决上述技术问题,本发明采用如下技术方案:
紫花苜蓿ILP分子标记引物,所述分子标记引物包括如实施例所述36对。
紫花苜蓿ILP分子标记引物在苜蓿品种鉴定中的应用。
其中所述的苜蓿品种包括以下表1:
表1苜蓿品种信息
一种鉴定苜蓿品种的试剂盒,包含所述的紫花苜蓿ILP分子标记引物。
本发明的有益效果是:在不计算DNA提取过程所消耗的时间的情况下,本发明的检测方法可利用较少的引物组合在4h之内完成对供试苜蓿品种与已知苜蓿品种的比对鉴定,具有准确、高效、快速、成本低以及操作方便等优势,本申请具有较高的灵敏度和精确度。
附图说明
图1是本发明具体实施步骤流程图;
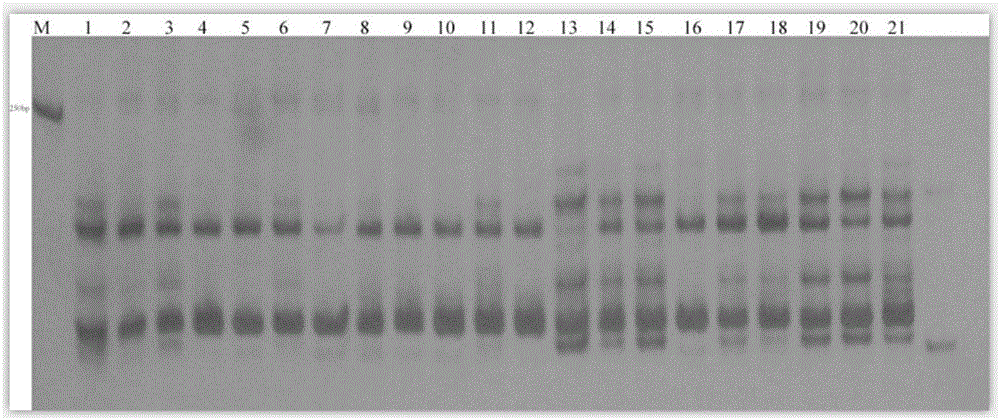
图2是引物MsILP-07对苜蓿品种的扩增结果图;
图3是引物MsILP-20对苜蓿品种的扩增结果图。
具体实施方式
下面更详细地描述本发明的具体实施例。应当理解,可以以各种形式实现本发明而不应被这里阐述的实施例所限制。相反,提供这些实施例是为了能够更透彻地理解本发明,并且能够将本发明的范围完整的传达给本领域的技术人员。
如在通篇说明书及权利要求当中所提及的“包含”或“包括”为一开放式用语,故应解释成“包含但不限定于”。说明书后续描述为实施本发明的较佳实施方式,然所述描述乃以说明书的一般原则为目的,并非用以限定本发明的范围。本发明的保护范围当视所附权利要求所界定者为准。
实施例:
1.鉴定材料DNA提取
参照SDS法,具体操作步骤如下(见图1):
1)选取40粒饱满、健康种子,利用双层滤纸法20℃萌发72h;
2)将以上种子混合于研钵中充分研磨,加入600μL DNA buffer,继续研磨混匀。将研磨液转移至2.0mL离心管;
3)将上述离心管转移至65℃水浴10min,此过程中摇匀1-2次;
4)在通风厨中加入氯仿:异戊醇体积比例为24∶1的混合液600μL,上下颠倒混匀,室温条件下12000转离心10min,吸取360μL上清液转入1.5ml离心管中;
5)加入预冷的3mol/L NaAc(pH=5.2)36μL和异丙醇240μL后混合均匀,室温放置10min后,室温12000转离心10min,弃上清液,离心30s,用10μL移液枪将残夜吸出;
6)加入500μL的75%乙醇,将沉淀弹起,12000转离心5min,倒出乙醇。离心30s,用10μL移液枪将残夜吸出,此步骤重复2次,在通风厨中吹干残留乙醇;
7)加入20μL灭菌去离子水,充分溶解后,用1%琼脂糖电泳检测DNA分子量大小,利用超微量紫外分光光度计测定DNA浓度,并稀释到25ng/μL,4℃保存待用。
2.鉴定材料DNA的ILP-PCR扩增
引物的合成过程是本发明最为困难、过程最为复杂的部分,其中最重要的是前期的序列比对,而Primer3.0进行引物设计是常规方法。
从M.sativa gene index 1.2 http://plantgrn.noble.org/AGED/(AGED)数据库下载紫花苜蓿高通量转录组测序序列共112626条conting序列。从https://www.arabidopsis.org/index.jsp(TAIR 10)下载拟南芥全基因组序列。根据物种间外显子序列保守性较强的原理,分别以拟南芥基因组序列和紫花苜蓿转录组序列为参考序列和查询序列,利用Perl脚本筛选拟南芥内含子长度≤400bp位置的两端各100bp长度外显子序列,并利用本地Blast程序确定与上述获得的200bp拟南芥外显子序列相似度≥80%的紫花苜蓿转录组序列,最后利用Primer 3.0软件进行紫花苜蓿ILP引物设计。紫花苜蓿ILP正反引物序列应分别位于左右两侧各100bp序列内,以保证扩增产物为内含子序列。
从上述描述可知,待比对的紫花苜蓿序列非常多(112626条),实际设计出来的紫花苜蓿ILP引物也非常多。而本申请中提到的36条引物是经过我们前期大量的创造性筛选最后得到多态性高、重复性好的引物,期间需要大量的创造性工作。
所设计出来的36对引物如表2所示。
表2 36对引物信息
利用本方法给出的36对引物对待鉴定材料DNA进行PCR扩增,反应体系及程序如下:
1)反应体系:10μL体系包括鉴定材料DNA模板25ng,正、反向引物各0.4μmol/L,5μL2×power Taq MasterMix(百泰克),剩余体积用超纯水补足;
2)反应程序:使用降落PCR,首先94℃变性30s,58-61℃退火30s,72℃延伸30s,共5个循环;接着94℃变性30s,55-58℃退火30s,72℃延伸30s,共30个循环;72℃延伸7min,4℃保存。
3.变性聚丙烯酰氨凝胶电泳与银染检测
以使用DYCZ-28C双板夹芯式垂直电泳槽为例,操作步骤如下:
1)清洗玻璃板:用自来水将玻璃板清洗干净,用去离子水冲洗后晾干。用95%乙醇擦洗两遍,吸水纸擦干。操作过程中防止两块玻璃板互相污染。
2)组装玻璃板:待玻璃板彻底干燥后,使用胶条将其组装成电泳胶板。
3)胶板封底:在10mL 6.0%PAGE胶中分别加入12.5μL TEMED和新配制的10%过硫酸铵溶液100μL可用于2板胶封底。
4)制胶:在80mL 6.0%PAGE胶中分别加入54μL TEMED和新配制的10%过硫酸铵溶液800μL可用于灌装2板胶。上述迅速混匀后灌胶,灌胶时应匀速以防止出现气泡。待胶液充满玻璃板夹层,在上部轻轻插入长城齿梳子,聚合至少30min以上。胶聚合后,清理胶板表面溢出的胶液,轻轻拔出梳子,用水清洗干净备用。
5)样品制备:在10μL PCR产物中加入2μL6×加样缓冲液,混匀后,在水浴锅或PCR仪上95℃变性5min,4℃冷却10min以上。
6)电泳:用注射器吸取缓冲液冲洗凝胶顶端几次,清除气泡和凝胶碎片,同时将胶条拨正。每一个加样孔点入1μL~2μL样品。除待测样品外,同时加入参照品种扩增产物。在400V恒压下电泳,电泳1.5h~2h(电泳时间取决于扩增片段的大小范围)。电泳结束后,小心分开两块玻璃板,从玻璃板上取下凝胶准备银染。
7)银染:a.将附着凝胶的长玻璃板置于塑料盒中,加入适量蒸馏水,轻轻晃动洗下胶片,去除玻璃板,将胶片漂洗2次。b.向装有胶片的塑料盒中加入0.08%的硝酸银溶液,使溶液没过胶片,在摇床上60转/分钟摇10min。c.将硝酸银倒入废液桶,用蒸馏水漂洗干净胶片,将胶片转移入含有1.5%的氢氧化钠、0.4%的甲醛显影液中,在摇床上60转/分钟摇数分钟出现清晰带纹为止。d.将胶片转移至灯箱,拍照保存照片。其中,例如引物MsILP-07对苜蓿品种的扩增结果见图2,引物MsILP-20对苜蓿品种的扩增结果见图3。
8)将各鉴定品种同一位点扩增片段的带型和迁移位置进行比较,扩增带型以0、1表示,在相同迁移位置上,有带记为“1”,无带记为“0”,构建“1”、“0”矩阵。各引物对21对品种的区分信息见表3。
表3 21个苜蓿品种基于36个ILP分子标记构建的“0”、“1”矩阵
对于表3中的等位基因,以MsILP-28为例,对应的显示是有11个等位基因,但其实都是扩增的MsILP-28对应的一个基因,之所以会出现多个等位基因的情况,是因为苜蓿是异源四倍体,再加上某个基因可能有多个拷贝(即一个基因可能在基因组中出现多次,且之间的内含子有可能有差异),所以有可能出现多条条带(等位基因)的情况。
在表3中可以看出每个引物扩增结果的情况,36对紫花苜蓿ILP分子标记在21个供试苜蓿品种中都能够扩增出多态性条带,且扩增结果稳定,具有重复性。其中,36对ILP分子标记在21个苜蓿品种中共扩增出169个等位基因条带,平均每个位点为4.7个等位基因条带。36对ILP分子标记的预期杂合度(He)范围为0.16(MsILP-40,MsILP-53,MsILP-54)到0.88(MsILP-75),平均值为0.65。多态性指数(PIC)范围为0.15(MsILP-40,MsILP-53,MsILP-54)到0.87(MsILP-75),平均值为0.60。
9)对每对引物的鉴定结果进行统计分析,不同鉴定品种间差异位点数≥1则判定两品种为不同品种。具体的品种鉴定结果见表4。
表4 36个ILP分子标记区分品种信息(品种编号对应品种信息见表1)
表4种的类群以MsILP-01为例,其对21个苜蓿品种进行PCR扩增,根据每个品种扩增出的条带差异,可以将21个品种分为7个类群(情况)。其中,品种1、18、13和14归属于类群1,是因为他们的扩增条带一样;品种2和3的扩增条带一样,归属于类群2,但他们的条带类型和类群1的条带类型又有差别。其后同理。因此,如果仅仅利用MsILP-01引物的话,仅能将21个品种部分区分开。而如果要完全区分开21个品种的话,就需要利用不同引物的组合。例如,品种2和3我们无法用MsILP-01,但是如果再结合MsILP-05的结果的话,就可以把品种2和3区分开,且上述结果也可证明本申请具有较高的灵敏度和精确度。
以上所述仅为本发明的实施例,并非因此限制本发明的专利范围。
SEQUENCE LISTING
<110> 兰州大学
<120> 紫花苜蓿ILP分子标记引物及在苜蓿品种鉴定中的应用
<130> 2016
<160> 72
<170> PatentIn version 3.5
<210> 1
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 1
agacaggttg ttcacattgc 20
<210> 2
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 2
accagcagta tcccaaatct 20
<210> 3
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 3
cacaactctg gaatgggaa 19
<210> 4
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 4
catctctcgt gttactgaat gc 22
<210> 5
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 5
ggactcaact caaagatgcc 20
<210> 6
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 6
gacccataga caacattcca ag 22
<210> 7
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 7
tagacgcttg acccttgag 19
<210> 8
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 8
gtggtattcc tctgtcccat 20
<210> 9
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 9
attgagcgtt acaccaggg 19
<210> 10
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 10
tgctgaagta gtggctttga 20
<210> 11
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 11
gctgaaacac acaaggacaa 20
<210> 12
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 12
aacaccgtga gttgaagga 19
<210> 13
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 13
ctcacggtgt ttatgaagaa gg 22
<210> 14
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 14
ccaacctggg cttgtaagt 19
<210> 15
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 15
gttaggaagg aggctgagaa 20
<210> 16
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 16
ggagtggcaa acctgaaa 18
<210> 17
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 17
cacaactctg gaatgggaa 19
<210> 18
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 18
catctctcgt gttactgaat gc 22
<210> 19
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 19
ggactcaact caaagatgcc 20
<210> 20
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 20
gacccataga caacattcca ag 22
<210> 21
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 21
tagacgcttg acccttgag 19
<210> 22
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 22
gtggtattcc tctgtcccat 20
<210> 23
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 23
attgagcgtt acaccaggg 19
<210> 24
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 24
tgctgaagta gtggctttga 20
<210> 25
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 25
gctgaaacac acaaggacaa 20
<210> 26
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 26
aacaccgtga gttgaagga 19
<210> 27
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 27
ctcacggtgt ttatgaagaa gg 22
<210> 28
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 28
ccaacctggg cttgtaagt 19
<210> 29
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 29
gaccaacaac accttgacta tc 22
<210> 30
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 30
atactcacat cagcaccagc 20
<210> 31
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 31
atcctttcaa accacccaag 20
<210> 32
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 32
ggcaaatgct accgttactg 20
<210> 33
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 33
gttaggaagg aggctgagaa 20
<210> 34
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 34
ggagtggcaa acctgaaa 18
<210> 35
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 35
tgttccctgc tatcaatgtc 20
<210> 36
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 36
gtgacggcat ccatacaa 18
<210> 37
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 37
aggctgtcct ctccctctat 20
<210> 38
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 38
cggaatccaa cacaatacc 19
<210> 39
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 39
gggctaaagg gcattaca 18
<210> 40
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 40
agttcctcca ccaagtgaat 20
<210> 41
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 41
acgcagacga atgtatggt 19
<210> 42
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 42
cagggaacct caaacaaca 19
<210> 43
<211> 21
<212> DNA
<213> Medicago sativa L.
<400> 43
cttgtctaca ccgtcttctc c 21
<210> 44
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 44
ccagtgattg ggatagtgg 19
<210> 45
<211> 21
<212> DNA
<213> Medicago sativa L.
<400> 45
gcaatgacag ggatagagtt g 21
<210> 46
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 46
ggcgaacaca agcaatact 19
<210> 47
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 47
tgctgtttct cacggtctt 19
<210> 48
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 48
ctcacggtca aatgctaagt tc 22
<210> 49
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 49
gtgttacagc aactgacttg gt 22
<210> 50
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 50
atctacaggg aagaaaccca 20
<210> 51
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 51
ggtccactgc tttccgtaa 19
<210> 52
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 52
ccaaccacac tgtcaggat 19
<210> 53
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 53
tgaagcagag gcagcaat 18
<210> 54
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 54
ttgaacccaa caacaccg 18
<210> 55
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 55
tttctgtgct ggtcgtgt 18
<210> 56
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 56
gaatctctcg tttcctactt gc 22
<210> 57
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 57
tgaaacattc gggtcaactg 20
<210> 58
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 58
gcattccagg tcttctcgt 19
<210> 59
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 59
cctattacaa gcagcggct 19
<210> 60
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 60
caatcaggat gaacaaccc 19
<210> 61
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 61
ggatggcttt gagcgttt 18
<210> 62
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 62
gaatcagttt aggaaccacc ac 22
<210> 63
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 63
ttgctgcctt tgtcccaa 18
<210> 64
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 64
cgaccttgaa cctgactcca 20
<210> 65
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 65
ggcaaggttg aaaggaaca 19
<210> 66
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 66
acaagggcag cacaagatg 19
<210> 67
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 67
ggcaaggttg aaaggaaca 19
<210> 68
<211> 19
<212> DNA
<213> Medicago sativa L.
<400> 68
acaagggcag cacaagatg 19
<210> 69
<211> 22
<212> DNA
<213> Medicago sativa L.
<400> 69
gtgttacagc aactgacttg gt 22
<210> 70
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 70
atctacaggg aagaaaccca 20
<210> 71
<211> 18
<212> DNA
<213> Medicago sativa L.
<400> 71
ccgtgctttc gctttatg 18
<210> 72
<211> 20
<212> DNA
<213> Medicago sativa L.
<400> 72
caccatctcc agaatcaaga 20
- 还没有人留言评论。精彩留言会获得点赞!