一种与奶山羊乳体细胞数性状相关的分子标记及其应用的制作方法
本发明属于动物分子标记制备技术领域,具体涉及一种与奶山羊乳体细胞数性状相关的分子标记的制备与应用。
背景技术:
我国是奶山羊饲养量较多、分布范围较广的国家,20世纪60-80年代曾经是我国奶业的主导产业,90年代以后受到饲养规模小、乳品工业迅速膨胀等因素的影响,奶山羊生产发展迟缓。近十多年来,随着人民生活水平的不断提高和膳食结构改善的需求,人们对山羊奶营养价值和安全性的认同度有所改变,尤其是机器挤奶解决了规模化饲养的瓶颈问题,促进了我国奶山羊产业的复兴和发展。我国奶山羊品种是在二十世纪初由国外传教士带入的奶山羊品种与本地山羊杂交改良,又经过生态适应和系统选育而形成的,已经列入国家品种志或名录的有雅安奶山羊、崂山奶山羊、关中奶山羊和西农萨能奶山羊等。但由于饲养分散、规模较小等原因,我国奶山羊的遗传改良主要采用常规的育种手段,其泌乳性能遗传进展较为缓慢。
随着分子生物学技术的快速发展,分子标记辅助育种已成为动物遗传育种研究的热点和必然趋势。应用分子技术阐明奶山羊泌乳性状的调控机制,结合泌乳性状加强奶山羊的选育,必将大大加快奶山羊育种的进程。分子标记辅助育种技术的核心是寻找决定泌乳性状的主效基因及其分子标记。近年来,国内外学者对奶山羊产奶性状相关基因进行了一些研究,找到了一些功能基因和分子标记,但是有关乳体细胞数性状的研究报道较少。
SPARC基因与细胞生长代谢相关。孙杰(2009)通过差异消减法鉴定到SPARC基因在西农萨能奶山羊泌乳初期和盛期乳腺组织之间存在差异表达,并通过实时荧光定量PCR进行了验证(孙杰,奶山羊乳腺消减文库构建及差异表达基因的研究,石河子大学2009年博士学位论文),提示该基因可能在奶山羊泌乳过程中发挥重要的调控作用,可作为开发奶山羊分子育种标记的候选基因。本发明申请中克隆与奶山羊乳体细胞数性状相关的SPARC基因片段,并找到一个与奶山羊乳体细胞数性状相关的突变位点,为奶山羊育种提供新的分子标记。
技术实现要素:
本发明克隆了与奶山羊乳体细胞数性状相关的SPARC基因片段(该基因片段就是本发明制备的与奶山羊乳体细胞数性状相关的分子标记),并提供了与奶山羊乳体细胞数性状相关的SPARC基因片段的制备方法,该方法通过设计特异性的引物,扩增包括一个突变位点的SPARC基因的部分片段,并且根据NarⅠ-RFLP方法得到的不同酶切图谱确定不同的基因型。
本发明还提供了一种与奶山羊乳体细胞数性状相关的SPARC基因片段在奶山羊分子标记辅助选育中的应用。
具体地,本发明采用以下技术方案:
一、发明人通过基因克隆方法得到了一种与奶山羊乳体细胞数性状相关的SPARC基因片段(该基因片段就是本发明制备的与奶山羊乳体细胞数性状相关的分子标记),其核苷酸序列如下所示:
gctttg ggaatgaggg cgaggctctc tgctttgcca acaggtgtgc
agcaacgaca acaagacctt cgactcttcc tgccacttct ttgccaccaa gtgcacactg
gagggcacca agaagggcca caagctccac ctggactaca tcgggccttg caaatgtgag
tctccttggg ccccgccaag cctcccgtct cagggaagag gcgccaaatt ggcactgctg
ctggctctgc cacctctggg ctgtgtgatc ttgggcacct ctcttcccct gtctgagctt
cagggatccc agttatagaa tgagatgatt gtttatgagc tggtatctgt actcaggacc
cctccaggga aggactgagc agtggaggga acacaggatc agggcagatt t
上述序列203-210位区域(带下划线区域,AGAGGCGC)存在插入/缺失突变(即这八个碱基的有或无),该突变导致NarⅠ-RFLP多态性。
二、发明人设计了一种检测与奶山羊乳体细胞数性状相关的基因片段突变的引物对,其DNA序列如下所示:
正向引物SPARCF6:GCTTTGGGAATGAGGGCGAG
反向引物SPARCR6:AAATCTGCCCTGATCCTGTGTT
三、一种与奶山羊乳体细胞数性状相关的SPARC基因片段的制备方法,其步骤是:
1、引物设计
利用引物设计软件以山羊SPARC基因序列为模板设计出包含部分序列的扩增引物对SPARCF6、SPARCR6,其DNA序列如SEQ ID NO.2-3所示:
SPARCF6:GCTTTGGGAATGAGGGCGAG
SPARCR6:AAATCTGCCCTGATCCTGTGTT
2、PCR产物的纯化和测序
构建PCR反应体系,以奶山羊基因组DNA为模板,利用步骤1所得引物进行PCR扩增,所得PCR产物测序,测序序列与山羊SPARC基因比对,获得与奶山羊乳体细胞数性状相关的分子标记,其DNA序列如SEQ ID NO.1所示,具体地:
在崂山奶山羊群体中随机选取2个及以上个体的基因组DNA,等量混合,以此为模板进行PCR扩增,将所得PCR产物送公司测序。测序序列与山羊SPARC基因比对,成功获得山羊SPARC基因部分序列397bp/389bp的片段,序列如下:
gctttg ggaatgaggg cgaggctctc tgctttgcca acaggtgtgc
agcaacgaca acaagacctt cgactcttcc tgccacttct ttgccaccaa gtgcacactg
gagggcacca agaagggcca caagctccac ctggactaca tcgggccttg caaatgtgag
tctccttggg ccccgccaag cctcccgtct cagggaagag gcgccaaatt ggcactgctg
ctggctctgc cacctctggg ctgtgtgatc ttgggcacct ctcttcccct gtctgagctt
cagggatccc agttatagaa tgagatgatt gtttatgagc tggtatctgt actcaggacc
cctccaggga aggactgagc agtggaggga acacaggatc agggcagatt t
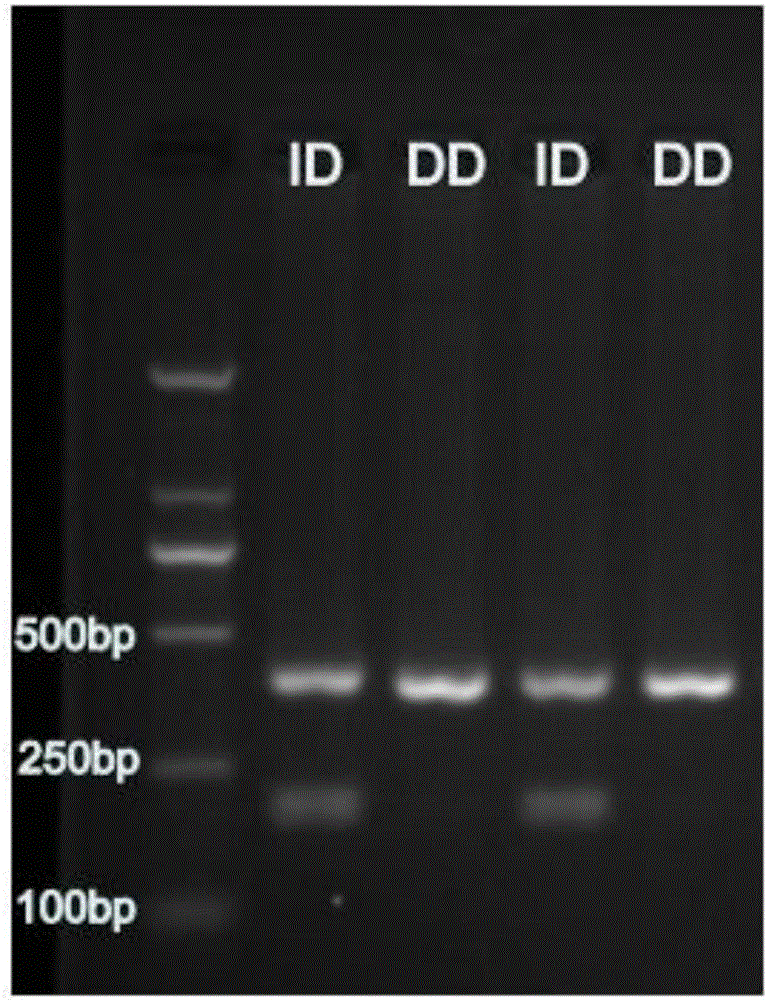
上述序列203-210位(带下划线区域)共8个碱基(AGAGGCGC)出现插入/缺失突变(I/D),该突变导致NarⅠ-RFLP多态性。经NarⅠ酶切后产生389bp、208bp和189bp三条带(其中208bp和189bp两条带大小接近,显示为一条带),说明该个体SPARC两个等位基因203-210位突变区域分别为插入(I)和缺失(D),判定为ID基因型;经NarⅠ酶切后产生389bp一条带(未切开),说明该个体SPARC两个等位基因203-210位突变区域均为缺失(D),判定为DD基因型。
四、一种与奶山羊乳体细胞数性状相关的SPARC基因片段在奶山羊分子标记辅助选育中的应用
选取处于泌乳期的崂山奶山羊种母羊200只,采取颈静脉全血,提取基因组DNA,使用引物对SPARCF6、SPARCR6,对奶山羊群体DNA进行PCR扩增,使用NarⅠ酶切检测每个个体的基因型,发现ID型的乳体细胞数性状显著优于DD型(P<0.05),选择ID型个体留种。
五、本发明制备的引物对可用于奶山羊分子标记辅助选育中。
更详细的技术方案见具体实施方式。
有益效果:山羊产奶性状属于数量性状,传统表型选择的准确性较低,且受山羊生长及繁殖周期的限制,产奶性状需要等到母羊分娩泌乳后才能表现出来,无法进行超早期选择,从而导致育种进展缓慢。本发明采用PCR-RFLP方法检测SPARC基因第六内含子NarⅠ酶切位点多态性,将其多态性与崂山奶山羊产奶性状进行关联分析,发现该多态性与崂山奶山羊乳体细胞数性状显著关联。此多态位点可用于崂山奶山羊产奶性状的辅助选择,提高种羊选留的准确性,加快奶山羊育种进程。本方法在山羊刚出生时就可以检测基因型,选择准确性提高,缩短世代间隔,育种成本显著降低;本方法实验操作简单,需要的仪器设备少,成本低,普通实验室均可进行;基因分型效率高,不同基因型之间区别显著。本发明发现了一个与奶山羊乳体细胞数性状相关联的分子标记,丰富了奶山羊育种的分子标记资源库。
附图说明
序列表SEQ ID NO.1是本发明制备的与奶山羊乳体细胞数性状相关的分子标记,序列长度为397bp/389bp,在该序列203-210位共8个碱基(AGAGGCGC)出现插入/缺失突变(I/D);
图1为崂山奶山羊SPARC基因部分序列扩增的电泳图。片段大小为397bp,最左侧泳道为分子质量标准;
图2为崂山奶山羊SPARC基因部分序列扩增片段经内切酶NarⅠ酶切后的电泳图。最右侧泳道为分子质量标准。
具体实施方式
实施例1崂山奶山羊SPARC基因部分序列的扩增
1、引物设计
利用在线引物设计软件Primer-BLAST(https://www.ncbi.nlm.nih.gov/tools/primer-blast/)以山羊SPARC基因序列(GeneBank登录号:NC_030814.1,序列区间47508621..47530930)为模板设计出包含部分序列的扩增引物,引物由青岛擎科梓熙生物技术有限公司合成,引物对的DNA序列如下:
SPARCF6:GCTTTGGGAATGAGGGCGAG
SPARCR6:AAATCTGCCCTGATCCTGTGTT
2、PCR产物的纯化和测序
PCR扩增反应体系25μL,包括1μL的奶山羊外周血基因组DNA(50ng/μL),12.5μL的2×PCR Mix(上海生工生物工程有限公司),0.75μL的正向引物SPARCF6(10mM),0.75μL的反向引物SPARCR6(10mM),10μL的ddH2O。PCR的反应程序为:95℃5min;94℃30s,60℃30s,72℃30s,30个循环;72℃延伸10min。扩增产物用2%琼脂糖凝胶进行电泳检测(见图1)。
随机选取奶山羊群体10个个体基因组DNA,等量混合,以此为模板进行PCR扩增,将PCR产物送上海生工生物工程有限公司测序。测序序列与山羊SPARC基因(GeneBank登录号:NC_030814.1,序列区间47508621..47530930)比对,成功获得山羊SPARC基因部分序列397bp/389bp的片段,序列如下:
gctttg ggaatgaggg cgaggctctc tgctttgcca acaggtgtgc
agcaacgaca acaagacctt cgactcttcc tgccacttct ttgccaccaa gtgcacactg
gagggcacca agaagggcca caagctccac ctggactaca tcgggccttg caaatgtgag
tctccttggg ccccgccaag cctcccgtct cagggaagag gcgccaaatt ggcactgctg
ctggctctgc cacctctggg ctgtgtgatc ttgggcacct ctcttcccct gtctgagctt
cagggatccc agttatagaa tgagatgatt gtttatgagc tggtatctgt actcaggacc
cctccaggga aggactgagc agtggaggga acacaggatc agggcagatt t
实施例2基因分型
上述序列插入/缺失突变的序列为AGAGGCGC,该突变导致NarⅠ-PFLP多态性。ID基因型经NarⅠ酶切后产生389bp、208bp和189bp三条带(其中208bp和189bp两条带大小接近,显示为一条带),DD基因型经NarⅠ酶切后产生389bp一条带(未切开)。基因分型示意图如附图2所示。
实施例3崂山奶山羊群体基因型与性状关联分析
选取青岛奥特种羊场处于泌乳期的崂山奶山羊种母羊200只,采取颈静脉全血,提取基因组DNA,使用引物对SPARCF6、SPARCR6,对奶山羊群体DNA进行PCR扩增,使用NarⅠ酶切检测每个个体的基因型,发现ID基因型个体为113只(组1),所测得的乳体细胞数平均值为50.33±10.15万个/毫升;DD基因型个体为87只(组2),乳体细胞数平均值为58.49±12.61万个/毫升。使用单因素方差分析对组1和组2两组数据进行统计分析,所得P值为0.028(<0.05),判定ID基因型与DD基因型的乳体细胞数存在显著差异,该NarⅠ-RFLP多态位点可作为奶山羊育种的分子标记使用。
SEQUENCE LISTING
<110> 青岛市畜牧兽医研究所
<120> 一种与奶山羊乳体细胞数性状相关的分子标记及其应用
<160> 3
<170> PatentIn version 3.3
<210> 1
<211> 397
<212> DNA
<213> 山羊(Capra hircus)
<220>
<221>mutation
<222>(203-210)..(203-210)
<223>
<400> 1
gctttgggaa tgagggcgag gctctctgct ttgccaacag gtgtgcagca acgacaacaa 60
gaccttcgac tcttcctgcc acttctttgc caccaagtgc acactggagg gcaccaagaa 120
gggccacaag ctccacctgg actacatcgg gccttgcaaa tgtgagtctc cttgggcccc 180
gccaagcctc ccgtctcagg gaagaggcgc caaattggca ctgctgctgg ctctgccacc 240
tctgggctgt gtgatcttgg gcacctctct tcccctgtct gagcttcagg gatcccagtt 300
atagaatgag atgattgttt atgagctggt atctgtactc aggacccctc cagggaagga 360
ctgagcagtg gagggaacac aggatcaggg cagattt 397
<210> 2
<211> 20
<212> DNA
<213> 人工序列
<400> 2
gctttgggaa tgagggcgag 20
<210> 3
<211> 22
<212> DNA
<213> 人工序列
<400> 3
aaatctgccc tgatcctgtg tt 22
- 还没有人留言评论。精彩留言会获得点赞!