激发植物产生过敏反应的蛋白质及其编码基因的制作方法
本发明属于生物技术领域,尤其是涉及激发植物产生过敏反应的蛋白质及其基因。
背景技术:
植物对病原物侵染的抗性表现在非寄主和寄主抗性两个方面。革兰氏阴性植物病原细菌在非寄主植物上产生过敏反应(Hypersensitive response,HR)是由病原菌中的某些激发子决定的。这种HR是一种抗病免疫反应,相应激发产生HR的蛋白质称为harpin类蛋白。目前已从革兰氏阴性植物病原细菌中鉴定的harpin类蛋白种类有HrpZ,HrpN,HrpW,PopA1,Hpa1,HopAK1和HopP1,并且明确植物病原黄单胞菌中HrpW和Hpa1这两个激发非寄主植物产生HR的harpin类蛋白。这些harpin蛋白具有共同的特点:具亲水性、富含甘氨酸、对热稳定、对蛋白酶敏感、酸性蛋白。
Harpin类蛋白具有重要的生物学功能。harpin类蛋白在植物上产生的HR是一种细胞编程死亡形式,在这个过程中伴随着活性氧迸发、水杨酸积累、病程相关蛋白产生和胼胝质增加等,从而增加植物的抗病免疫性。Harpin类蛋白施用于植物上,可促进植物生长、增加作物产量和诱导植物产生抗病性等。植物病原黄单胞菌harpin类蛋白及其编码基因具有植物抗病育种价值和生防价值,而且具有分离和鉴别植物抗病免疫基因的功能,发掘可使植物产生抗病免疫反应的植物病原黄单胞菌中的harpin类蛋白及其编码基因具有重要的科学价值和实践价值。
经对现有技术的文献检索发现,未发现植物病原黄单胞菌中可激发植物产生抗病免疫反应的XopP蛋白以及相应编码基因xopP的有关报道。
技术实现要素:
本发明的目的就是为了克服现有技术中的不足而提供激发植物产生过敏反应的蛋白质及其编码基因。本发明XopP蛋白能够使病原黄单胞菌的非寄主植物产生过敏反应。
本发明的目的可以通过以下技术方案来实现:
本发明涉及的激发植物产生过敏反应的蛋白质,其对应基因在黄单胞菌基因组上具有两个拷贝,氨基酸的同源性达到70%左右,根据其蛋白质序列号先后顺序分别命名为XopP1和XopP2。
本发明是以稻黄单胞菌水稻条斑病菌Xanthomonas oryzae pv.oryzicola(Xoc)作为研究的模式菌株。
本发明提供的第一种激发植物产生过敏反应的蛋白质,命名为XopP1,其氨基酸序列如SEQ ID NO.2所示,共有713个氨基酸。蛋白质XopP1对应的编码基因命名为xopP1,其核苷酸序列如SEQ ID NO.1,序列长度为2142bp。
本发明提供的第二种激发植物产生过敏反应的蛋白质,命名为XopP2,其氨基酸序列如SEQ ID NO.4所示,共有710个氨基酸。蛋白质XopP2对应的编码基因命名为xopP2,其核苷酸序列如SEQ ID NO.3,序列长度为2133bp。
本发明提供的蛋白质XopP1和蛋白质XopP2,其氨基酸序列中半胱氨酸含量均低于1%,蛋白质等电点为8.8,呈弱碱性,富含丙氨酸和亮氨酸等脂肪族氨基酸以及丝氨酸和精氨酸等碱性氨基酸,分子量77.9kD左右,其中丙氨酸和亮氨酸的总含量达25.7%。本发明提供的蛋白质XopP1和蛋白质XopP2都可以使烟草产生过敏反应。过敏反应是植物抗病性被激活的结果。
本发明还提供两种基因的构建方法。
基因xopP1的构建方法,包括以下步骤:
利用序列如SEQ ID NO.5所示的引物Xopp1-F和序列如SEQ ID NO.6所示的引物Xopp1-R,通过PCR扩增,可从水稻条斑病菌Xoc RS105菌株、水稻白叶枯病菌Xoo PXO99A菌株以及柑橘溃疡病菌Xac 29-1菌株基因组DNA中扩增得到xopP1基因。
基因xopP2的构建方法,包括以下步骤:
利用序列如SEQ ID NO.7所示的引物Xopp2-F和序列如SEQ ID NO.8所示的引物Xopp2-R,通过PCR扩增,可从水稻条斑病菌Xoc RS105菌株、水稻白叶枯病菌Xoo PXO99A菌株以及柑橘溃疡病菌Xac 29-1菌株基因组DNA中扩增得到xopP2基因。
本发明还提供了两种XopP表达工程菌株。
XopP1表达工程菌株,采用以下方法得到:
将基因xopP1,通过Hind III和XbaⅠ限制性酶切位点克隆到pHB载体上,转化到农杆菌EH105株中,并筛选具有利福平和卡那霉素抗性的阳性转化子,该转化子为XopP1表达工程菌株;其中,基因xopP1核苷酸序列如SEQ ID NO.1所示,蛋白质XopP1氨基酸序列如SEQ ID NO.2所示。
XopP2表达工程菌株,采用以下方法得到:
将基因xopP2,通过Hind III和XbaⅠ限制性酶切位点克隆到pHB载体上,转化到农杆菌EH105株中,并筛选具有利福平和卡那霉素抗性的阳性转化子,该转化子为XopP2表达工程菌株;其中,基因xopP2核苷酸序列如SEQ ID NO.3所示,蛋白质XopP2氨基酸序列如SEQ ID NO.4所示。
以上两种农杆菌工程菌株经过诱导表达,注射三生烟,发现能够使烟草产生过敏反应。
本发明中所涉及的水稻条斑病菌Xoc RS105和水稻白叶枯病菌Xoo PXO99A菌株已在文献《Wu Xiaomin,Li Yurong,Zou Lifang,and Chen Gongyou.2007.Gene-for-gene relationships between rice and diverse avrBs3/pthA avirulence genes in Xanthomonas oryzae pv.oryzae.Plant Pathology,56(1):26-34》中公开;涉及的柑橘溃疡病菌Xac 29-1菌株已在文献《Zhou,T.,Rou,W.,Song,X.,et al.2015.Molecular study on the carAB operon reveals that carB gene is required for swimming and biofilm formation in the Xanthomanas citri subsp.citri.BMC Microbiology(2015)15:255》中公开。这些菌株均为常用的工程菌株。
本发明中所涉及的农杆菌EH105菌株为公知公用菌株,特征见文献:TAKARA公司产品说明书。
与现有技术相比,本发明具有以下优点及有益效果:
本发明获得的激发子蛋白XopP1和XopP2具有harpin类蛋白的特性,能激发烟草产生HR反应和系统获得抗病性,这为设计诱导植物产生系统获得抗病性的基因工程药剂以及转基因植物提供了重要的基因资源,具有重要的实践价值。
附图说明
图1、不同来源的xopP1和xopP2基因片段PCR扩增(A)和酶切验证图(B)。
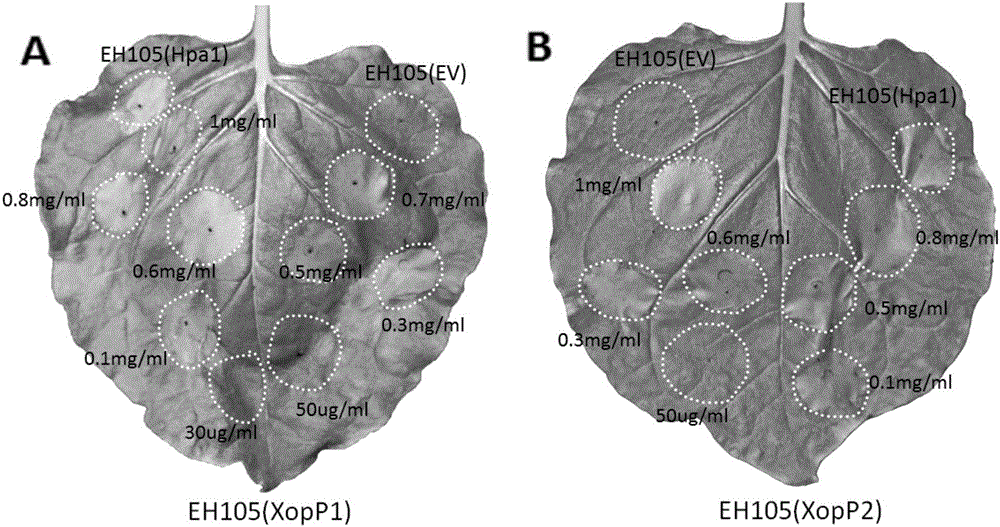
图2、XopP1和XopP2通过农杆菌在烟草上激发过敏反应的最低浓度测试图。
图3、不同植物病原黄单胞菌来源的XopP1和XopP2蛋白在烟草上激发过敏反应图。
具体实施方式
下面结合附图和具体实施例对本发明进行详细说明。
这些实施例仅用于说明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方法,通常按照常规条件,例如Sambrook等分子克隆:实验室手册(New York:Cold Spring Harbor Laboratory Press,1989)中所述的条件,或按照制造厂商所建议的条件。
实施例1、植物病原黄单胞菌xopP基因原核表达载体构建
本实施例从水稻条斑病菌Xoc RS105菌株、水稻白叶枯病菌Xoo PXO99A菌株以及柑橘溃疡病菌Xac 29-1菌株DNA中xopP基因为模版,设计引物xopP1-F/R(SEQ ID NO.3和NO.4)和xopP2-F/R(SEQ ID NO.5和NO.6),进行PCR扩增,PCR扩增条件为:94℃/4min+(94℃/30sec+55℃/30sec+72℃/2min)×32循环+72℃/10min。
利用序列如SEQ ID NO.5所示的引物Xopp1-F和序列如SEQ ID NO.6所示的引物Xopp1-R,通过PCR扩增,可从水稻条斑病菌Xoc RS105菌株、水稻白叶枯病菌Xoo PXO99A菌株以及柑橘溃疡病菌Xac 29-1菌株基因组DNA中扩增得到xopP1基因,其核苷酸序列如SEQ ID NO.1,序列长度为2142bp。并且得到第一种激发植物产生过敏反应的蛋白质,命名为XopP1,其氨基酸序列如SEQ ID NO.2所示,共有713个氨基酸。
利用序列如SEQ ID NO.7所示的引物Xopp2-F和序列如SEQ ID NO.8所示的引物Xopp2-R,通过PCR扩增,可从水稻条斑病菌Xoc RS105菌株、水稻白叶枯病菌Xoo PXO99A菌株以及柑橘溃疡病菌Xac 29-1菌株基因组DNA中扩增得到xopP2基因,其核苷酸序列如SEQ ID NO.3,序列长度为2133bp。并且得到第二种激发植物产生过敏反应的蛋白质,命名为XopP2,其氨基酸序列如SEQ ID NO.4所示,共有710个氨基酸。
蛋白质XopP1和蛋白质XopP2,其氨基酸序列中半胱氨酸含量均低于1%,蛋白质等电点为8.8,呈弱碱性,富含丙氨酸和亮氨酸等脂肪族氨基酸以及丝氨酸和精氨酸等碱性氨基酸,分子量77.9kD左右,其中丙氨酸和亮氨酸的总含量达25.7%。本发明提供的蛋白质XopP1和蛋白质XopP2都可以使烟草产生过敏反应。过敏反应是植物抗病性被激活的结果。
不同来源的xopP1和xopP2基因片段PCR扩增见图1(A),酶切验证图见如1(B)。
最终PCR产物连接在pMD18-T载体,连接产物转化感受态细胞DH5α,筛选具有Amp抗性的重组转化子TxopP1和TxopP2。重组转化子经Hind III和XbaⅠ双酶切回收xopP片段,连接经过相同酶切处理的表达载体pHB载体上,连接产物转化DH5α,获得正确的重组质粒PxopP1和PxopP2,将验证正确的质粒转化到农杆菌EH105菌株中,得到重组菌株EH105(XopP1)和EH105(XopP2)。
所涉及的水稻条斑病菌Xoc RS105和水稻白叶枯病菌Xoo PXO99A菌株已在文献《Wu Xiaomin,Li Yurong,Zou Lifang,and Chen Gongyou.2007.Gene-for-gene relationships between rice and diverse avrBs3/pthA avirulence genes in Xanthomonas oryzae pv.oryzae.Plant Pathology,56(1):26-34》中公开;涉及的柑橘溃疡病菌Xac29-1菌株已在文献《Zhou,T.,Rou,W.,Song,X.,et al.2015.Molecular study on the carAB operon reveals that carB gene is required for swimming and biofilm formation in the Xanthomanas citri subsp.citri.BMC Microbiology(2015)15:255》中公开。这些菌株均为常用的工程菌株。pMD18-T和pHB载体为公知公用载体,农杆菌EH105菌株为公用公知菌株,特征见文献:TAKARA公司产品说明书。所用内切酶为Hind III和XbaⅠ。
实施例2、XopP通过农杆菌介导的烟草上过敏反应的观察
将待测定的农杆菌菌株EH105(XopP1)和EH105(XopP2)在新鲜的平板上活化,EH105(EV)作为阴性对照,EH105(Hpa1)作为阳性对照,分别转接到的LB的液体培养基中,突变体或接合子要添加相对应的抗生素,28℃振荡培养大约12~16h,菌量浓度达到108CFU/mL;低速离心(<6,000rpm)收集菌体,丢弃上清。超纯水洗涤菌体1~2次,定容至所需浓度,一般为OD600=0.6。选取生长适中烟草从温室移出常温下放置24h,在叶片背面用无菌针头轻轻戳出接种口(注意不要挑破叶片),无针头的无菌注射器吸取菌液注射,并在叶片上标注好注射范围,过48h左右观察表型、并拍照。本实施例三生烟为HR测定最常用的烟草品种Nicotiana tabacum cv.xanthi,公知公用。
实施例3、XopP激发烟草过敏反应的最小浓度的测定
本实施例XopP通过调整不同的浓度值,分别接种三生烟,确定最小HR激发浓度,同时用相同浓度的Hpa1注射烟草作为阳性对照,带有空质粒的农杆菌作为阴性对照,48h后观察烟草叶片过敏反应。经测定发现XopP激发HR的最小浓度为0.1mg/ml(见图2)。
实施例4、不同来源的植物病原黄单胞菌的XopP蛋白激发烟草过敏反应的测定
本实施例从水稻条斑病菌Xoc RS105菌株、水稻白叶枯病菌Xoo PXO99A菌株以及柑橘溃疡病菌Xac 29-1菌株等黄单胞菌中克隆出XopP基因,构建在pHB载体上,然后转化进入EH105农杆菌中,调整终浓度OD600=0.6,用相同浓度的XopPXoc菌株为阳性对照,48h后观察烟草叶片过敏反应。发现来自水稻白叶枯病菌Xoo PXO99A菌株以及柑橘溃疡病菌Xac 29-1菌株的XopP蛋白均能够激发烟草产生过敏反应(见图3)。
关于RS105:陈功友、潘小玫、王金生,水稻白叶枯病菌(Xanthomonas oryzae pv.oryzae)化学诱变hrp基因突变体及相关性状的研究,植物病理学报,2001,31(3):199~207;
关于基因重组分子操作:Sambrook J,Russell D.W.,Molelcular cloning,A Laboratory Manual(3rd ed),2001,Spring Harbor Laboratory Press;
关于克隆harpin相关基因的方法:Wen W.G,Wang J.S,Defense response in plants induced by Harpinxoo,an elicitor of Hypersensitive response from Xanthomonas oryzae pv.oryzae,2003,Journal of Agricultural Biotechnology;
关于克隆载体pMD18-T和pHB:TAKARA载体说明书。
上述的对实施例的描述是为便于该技术领域的普通技术人员能理解和使用发明。熟悉本领域技术的人员显然可以容易地对这些实施例做出各种修改,并把在此说明的一般原理应用到其他实施例中而不必经过创造性的劳动。因此,本发明不限于上述实施例,本领域技术人员根据本发明的揭示,不脱离本发明范畴所做出的改进和修改都应该在本发明的保护范围之内。
<110>上海交通大学
<120>激发植物产生过敏反应的蛋白质及其编码基因
<160> 8
<170> PatentIn version 3.5
<210> 1
<211> 2142
<212> DNA
<213> 水稻白叶枯病菌(Xanthomonas oryzae pv. oryzae)
<220>
<221> CDS
<222> (1)..(2142)
<400> 1
gtgcctaaaa ttgaatcgac caagcaggcc ccccctgatc ccgcgcaatt ggttcaaact 60
gcaccgacac catcgggctc ggcatcagtg gcaagcacgt ctgccgacag tgcggtgccc 120
tcagcgcaac tcggcgggct ttccatcagg ccgcggctgt tgggccgagc ctccacagcc 180
aatccctcag cggcatcgtc cagcaacgca aagaacgtca gggcggcgct ctttgccgcg 240
cagtcagttt cgcaagggcc agatcctgcc gaagtattgg tcaaggcctc atccaggctg 300
aaacgcttca acgacttggc acaaaaagta gtgccgacag aaccgtcgga tatcaaagcc 360
ctggaagcaa gactccggtc cggaacgtct gcgctcgaat ccgctcgaca ggcctttcag 420
gcgcttgccg agttgaatat caaaaaacgc attccggtcg acaatatcga agaacacgag 480
aatttcctgg tgccgcagtt cgcaatcgcc gcatccctgc acttcggcgc atgccagaag 540
atgatcgatc agaaaatggc caccgaattg ggccatgtgc ccacccaaag ggtcgagatg 600
gtgacgttgt ccgttgacgc cacccgtctg cagcaaccgg aatcatggct tgcgttacgc 660
gctcatattc tgaattccct tgcagcgatg gaggaggttt gccagcgcat caggtcgccg 720
atgacgggtc aggggccgcg tgactcgctc aaggccaatc tccccgtcgt cagggcgatg 780
atcgcatcgt gccatgaacg catgcagacc gtccgcggaa tgctcgtcga aatatattca 840
aggagactta gcgatcaggc caccgattgc ggcctgctcc aggtgcttga cagactgagg 900
gagctcgaag aaatcgacag ccttagccat gctgaggtga gcgaagcgct caatcatgtg 960
atccaggtcc atatcgaacg tctgtcctca accgagcatt cgctcacacc ggaggcgctg 1020
gccctgtgca aaaggctact cgtcgaatac gccgaggtgc gcggccgagc ggggatgcag 1080
ctgtgtgtag atgccgccgc gctcatcgaa caaggtgagc atgccagaga tcatctatgg 1140
ccaacgatgc tccatcttgc acaggcactt gccgaccaac gtcaggcaat tcttgacatg 1200
tgtgagttcg ccgtccagga cagacagctg attggcacgg cggcggcttc gcccgagcaa 1260
gcaccggcat cgataacacc gagcgtgccg acaacgacag caggcaaatc gagcaggtca 1320
cgcaaggcac gcatgcgcac ctctgatgct tccagctcgg cagcgcccgc gcagcgggtc 1380
attgacgagc gcaacgcagc gcaaaagcag gccgacgaga tactgaaaag cacgcgtctg 1440
gagtcgttgc cggtggcaga actcggcggc gatttcatcg cgctcgccaa gcgccttggc 1500
aaggacacca ccgacataga gcgcctgatc ggcgactcca ggcgtgacgc ggcgacggct 1560
ttcgattttg ccaggacctc catgcaggac tggttcggat cgagtgagcg tctgctgcag 1620
ctcaaaagca agctccgtgc gggagatccg cgtatcgcac aactcgacac gcggctgcgt 1680
cttctgcaga ggatcgagca ggagtttgaa cggcgcgaag ccgatgcgct caagatcgat 1740
ccgcaaccgc gtgcgccgca tctgcagcgc ctgttggcga tgcatgcact ggcacgcgtc 1800
acggcgccga accgccttcc ctcggaggac gaccgtggcg gccgtggcag gctatttgaa 1860
gtgcgtatcg accacacgcc gcagtcaaat ggcgacattc ccgcgccctg gttcgtccac 1920
gtacataccg aaaaaccggt aacgcccacc gggctgcgcg cgctccacta caaggacttg 1980
gccgccgtcc acctgaagac ggccagggag gtcaacctgg gcccacgctg ggaagagatg 2040
atgcgcgccc tcggaaacac cgaggccaag gtgcaccggg caaccatcgg ctcgaagctg 2100
ctgggccagc tttgggcggc tggcgctggt gggcagcgat aa 2142
<210>2
<211>713
<212>PRT
<213> 水稻白叶枯病菌(Xanthomonas oryzae pv. oryzae)
<400>2
Met Pro Lys Ile Glu Ser Thr Lys Gln Ala Pro Pro Asp Pro Ala
5 10 15
Gln Leu Val Gln Thr Ala Pro Thr Pro Ser Gly Ser Ala Ser Val
20 25 30
Ala Ser Thr Ser Ala Asp Ser Ala Val Pro Ser Ala Gln Leu Gly
35 40 45
Gly Leu Ser Ile Arg Pro Arg Leu Leu Gly Arg Ala Ser Thr Ala
50 55 60
Asn Pro Ser Ala Ala Ser Ser Ser Asn Ala Lys Asn Val Arg Ala
65 70 75
Ala Leu Phe Ala Ala Gln Ser Val Ser Gln Gly Pro Asp Pro Ala
80 85 90
Glu Val Leu Val Lys Ala Ser Ser Arg Leu Lys Arg Phe Asn Asp
95 100 105
Leu Ala Gln Lys Val Val Pro Thr Glu Pro Ser Asp Ile Lys Ala
110 115 120
Leu Glu Ala Arg Leu Arg Ser Gly Thr Ser Ala Leu Glu Ser Ala
125 130 135
Arg Gln Ala Phe Gln Ala Leu Ala Glu Leu Asn Ile Lys Lys Arg
140 145 150
Ile Pro Val Asp Asn Ile Glu Glu His Glu Asn Phe Leu Val Pro
155 160 165
Gln Phe Ala Ile Ala Ala Ser Leu His Phe Gly Ala Cys Gln Lys
170 175 180
Met Ile Asp Gln Lys Met Ala Thr Glu Leu Gly His Val Pro Thr
185 190 195
Gln Arg Val Glu Met Val Thr Leu Ser Val Asp Ala Thr Arg Leu
200 205 210
Gln Gln Pro Glu Ser Trp Leu Ala Leu Arg Ala His Ile Leu Asn
215 220 225
Ser Leu Ala Ala Met Glu Glu Val Cys Gln Arg Ile Arg Ser Pro
230 235 240
Met Thr Gly Gln Gly Pro Arg Asp Ser Leu Lys Ala Asn Leu Pro
245 250 255
Val Val Arg Ala Met Ile Ala Ser Cys His Glu Arg Met Gln Thr
260 265 270
Val Arg Gly Met Leu Val Glu Ile Tyr Ser Arg Arg Leu Ser Asp
275 280 285
Gln Ala Thr Asp Cys Gly Leu Leu Gln Val Leu Asp Arg Leu Arg
290 295 300
Glu Leu Glu Glu Ile Asp Ser Leu Ser His Ala Glu Val Ser Glu
305 310 315
Ala Leu Asn His Val Ile Gln Val His Ile Glu Arg Leu Ser Ser
320 325 330
Thr Glu His Ser Leu Thr Pro Glu Ala Leu Ala Leu Cys Lys Arg
335 340 345
Leu Leu Val Glu Tyr Ala Glu Val Arg Gly Arg Ala Gly Met Gln
350 355 360
Leu Cys Val Asp Ala Ala Ala Leu Ile Glu Gln Gly Glu His Ala
365 370 375
Arg Asp His Leu Trp Pro Thr Met Leu His Leu Ala Gln Ala Leu
380 385 390
Ala Asp Gln Arg Gln Ala Ile Leu Asp Met Cys Glu Phe Ala Val
395 400 405
Gln Asp Arg Gln Leu Ile Gly Thr Ala Ala Ala Ser Pro Glu Gln
410 415 420
Ala Pro Ala Ser Ile Thr Pro Ser Val Pro Thr Thr Thr Ala Gly
425 430 435
Lys Ser Ser Arg Ser Arg Lys Ala Arg Met Arg Thr Ser Asp Ala
440 445 450
Ser Ser Ser Ala Ala Pro Ala Gln Arg Val Ile Asp Glu Arg Asn
455 460 465
Ala Ala Gln Lys Gln Ala Asp Glu Ile Leu Lys Ser Thr Arg Leu
470 475 480
Glu Ser Leu Pro Val Ala Glu Leu Gly Gly Asp Phe Ile Ala Leu
485 490 495
Ala Lys Arg Leu Gly Lys Asp Thr Thr Asp Ile Glu Arg Leu Ile
500 505 510
Gly Asp Ser Arg Arg Asp Ala Ala Thr Ala Phe Asp Phe Ala Arg
515 520 525
Thr Ser Met Gln Asp Trp Phe Gly Ser Ser Glu Arg Leu Leu Gln
530 535 540
Leu Lys Ser Lys Leu Arg Ala Gly Asp Pro Arg Ile Ala Gln Leu
545 550 555
Asp Thr Arg Leu Arg Leu Leu Gln Arg Ile Glu Gln Glu Phe Glu
560 565 570
Arg Arg Glu Ala Asp Ala Leu Lys Ile Asp Pro Gln Pro Arg Ala
575 580 585
Pro His Leu Gln Arg Leu Leu Ala Met His Ala Leu Ala Arg Val
590 595 600
Thr Ala Pro Asn Arg Leu Pro Ser Glu Asp Asp Arg Gly Gly Arg
605 610 615
Gly Arg Leu Phe Glu Val Arg Ile Asp His Thr Pro Gln Ser Asn
620 625 630
Gly Asp Ile Pro Ala Pro Trp Phe Val His Val His Thr Glu Lys
635 640 645
Pro Val Thr Pro Thr Gly Leu Arg Ala Leu His Tyr Lys Asp Leu
650 655 660
Ala Ala Val His Leu Lys Thr Ala Arg Glu Val Asn Leu Gly Pro
665 670 675
Arg Trp Glu Glu Met Met Arg Ala Leu Gly Asn Thr Glu Ala Lys
680 685 690
Val His Arg Ala Thr Ile Gly Ser Lys Leu Leu Gly Gln Leu Trp
695 700 705
Ala Ala Gly Ala Gly Gly Gln Arg
710
<210> 3
<211> 2133
<212> DNA
<213> 水稻白叶枯病菌(Xanthomonas oryzae pv. oryzae)
<220>
<221> CDS
<222> (1)..(2133)
<400>3
gtgccgagat ttcgagcgac caatcaaacc ccagtggatc ccacgtcatc cactcagcca 60
gcggcggcac cacaggatgc gggggcaagc gcgagcgtgc ctaccagcat ggggcctccc 120
tccgcgcaac tcagcgggct tgccatcaga gcgtcgagtg aggccagggc ttccagagcc 180
agtgcacccg catcatcatc ggccaacgtc agggcgatgc tctttacccc ggcatctgtc 240
tcgggaggac cagatccgtt tgaggtattg aacagggcct cggccaggct gcagctgttc 300
aatcacctgg tcacaaacgt agcgccgagg gattcagggg acgcaaaatc ccttgaagca 360
agactcaggt ccggcgcttc tgcaatcgaa ttcgctcgac aaggcttgca agcgctgagc 420
gagctcaaga ttcaacgtcg catgtcatcc gccgacatcg aagcccatga gaatcgattg 480
gtttcgtctc tcgcacaagc cgcagcaatg aactatgtgt catgcgaaag aatgatcgat 540
cagaaattgg cccagggatt gggttctgcg cttacggaag gcagcgattt tgcgcagctt 600
gatatccaag ccatccgact gcagcagccg gaatcatggc atgcgttacg tgctcatctt 660
cttgatgcca ttgcggcgtt ggaagaggtt ttccgacgta tcaagtcgcc gatgacgact 720
cggacaccgt gcgactcgct gaaggcccag ctgccgatcg tcaggatgac gatggaatct 780
tgtcacacgc ggatgcaaac cgtgcgtgga gtgctgtacg aaatatattc agtaagactc 840
tccgatcaag taagtagttg ccgcatgcca ctggtgcttg atcgactgcg cgagcttgaa 900
gaagtagaca agtgcatgga cgccaaggtc ggaaaagcgg tcacggagct ggccaagatg 960
catattgaac gctttctctc aaccgagcac tcgctgacgc cggagatgct ggccatgcac 1020
aagaatgtgc ttgtcgaata cgccgagatg cgcggccgag cggcaacgca gctgtgtatg 1080
gatgccgccg cgctcattga ggaaggaggg cattccagag atcacatatg gccagcgatg 1140
ctcgatcttg cacaggcact tgccgaccaa cgtcaggcaa ttcttgacat gtgtgagttc 1200
gccgtccagg acagacagct gattggcgca gcggcggctt cgcccgagca agcaccggca 1260
tcgataacac cgagcgtgcc gacaacgaca gcaggtaaat cgagcaggtc acgcaaggca 1320
cgcatgcgca cctctgatgc ttccagctcg gcagcgcccg cgcagcgggt cattgacgag 1380
cgcaacgcag cgcaaaagca ggccgacgag atactgaaaa gcacgcgtct ggagtcgttg 1440
ccggtggcag aactcggcgg cgatttcatc gcgctcgcca agcgccttgg caaggacacc 1500
accgacatag agcgcctgat cggcgactcc aggcgtgacg cggcgacggc tttcgatttt 1560
gccaggacct ccatgcagga ctggttcgga tcgagtgagc gtctgctgca gctcaaaagc 1620
aagctccgtg cgggagatcc gcgcatcgca caactcgaca cgcggctgcg tcttctgcag 1680
aggatcgagc aggagtttga acggcgcgaa gccgatgcgc tcaagatcga tccgcaaccg 1740
cgtgcgccgc atctgcagcg cctgttggcg atgcatgcac tggcacgcgt cacggcgccg 1800
aaccgccttc cctcggagga cgaccgtggc ggccgtggca ggctatttga agtgcgtatc 1860
gaccacacgc cgcagtcaaa tggcgacatt cccgcgccct ggttcgtcca cgtacatacc 1920
gaaaaaccgg taacgcccac cgggctgcgc gcgctccact acaaggactt ggccgccgtc 1980
cacctgaaga cggccaggga ggtcaacctg ggcccacgct gggaagagat gatgcgcgcc 2040
ctcggaaaca ccgaggccaa ggtgcaccgg gcaaccatcg gctcgaagct gctgggccag 2100
ctttgggcgg ctggcgctgg cgggcaacaa taa 2133
<210>4
<211>710
<212>PRT
<213> 水稻白叶枯病菌(Xanthomonas oryzae pv. oryzae)
<400>4
Met Pro Arg Phe Arg Ala Thr Asn Gln Thr Pro Val Asp Pro Thr
5 10 15
Ser Ser Thr Gln Pro Ala Ala Ala Pro Gln Asp Ala Gly Ala Ser
20 25 30
Ala Ser Val Pro Thr Ser Met Gly Pro Pro Ser Ala Gln Leu Ser
35 40 45
Gly Leu Ala Ile Arg Ala Ser Ser Glu Ala Arg Ala Ser Arg Ala
50 55 60
Ser Ala Pro Ala Ser Ser Ser Ala Asn Val Arg Ala Met Leu Phe
65 70 75
Thr Pro Ala Ser Val Ser Gly Gly Pro Asp Pro Phe Glu Val Leu
80 85 90
Asn Arg Ala Ser Ala Arg Leu Gln Leu Phe Asn His Leu Val Thr
95 100 105
Asn Val Ala Pro Arg Asp Ser Gly Asp Ala Lys Ser Leu Glu Ala
110 115 120
Arg Leu Arg Ser Gly Ala Ser Ala Ile Glu Phe Ala Arg Gln Gly
125 130 135
Leu Gln Ala Leu Ser Glu Leu Lys Ile Gln Arg Arg Met Ser Ser
140 145 150
Ala Asp Ile Glu Ala His Glu Asn Arg Leu Val Ser Ser Leu Ala
155 160 165
Gln Ala Ala Ala Met Asn Tyr Val Ser Cys Glu Arg Met Ile Asp
170 175 180
Gln Lys Leu Ala Gln Gly Leu Gly Ser Ala Leu Thr Glu Gly Ser
185 190 195
Asp Phe Ala Gln Leu Asp Ile Gln Ala Ile Arg Leu Gln Gln Pro
200 205 210
Glu Ser Trp His Ala Leu Arg Ala His Leu Leu Asp Ala Ile Ala
215 220 225
Ala Leu Glu Glu Val Phe Arg Arg Ile Lys Ser Pro Met Thr Thr
230 235 240
Arg Thr Pro Cys Asp Ser Leu Lys Ala Gln Leu Pro Ile Val Arg
245 250 255
Met Thr Met Glu Ser Cys His Thr Arg Met Gln Thr Val Arg Gly
260 265 270
Val Leu Tyr Glu Ile Tyr Ser Val Arg Leu Ser Asp Gln Val Ser
275 280 285
Ser Cys Arg Met Pro Leu Val Leu Asp Arg Leu Arg Glu Leu Glu
290 295 300
Glu Val Asp Lys Cys Met Asp Ala Lys Val Gly Lys Ala Val Thr
305 310 315
Glu Leu Ala Lys Met His Ile Glu Arg Phe Leu Ser Thr Glu His
320 325 330
Ser Leu Thr Pro Glu Met Leu Ala Met His Lys Asn Val Leu Val
335 340 345
Glu Tyr Ala Glu Met Arg Gly Arg Ala Ala Thr Gln Leu Cys Met
350 355 360
Asp Ala Ala Ala Leu Ile Glu Glu Gly Gly His Ser Arg Asp His
365 370 375
Ile Trp Pro Ala Met Leu Asp Leu Ala Gln Ala Leu Ala Asp Gln
380 385 390
Arg Gln Ala Ile Leu Asp Met Cys Glu Phe Ala Val Gln Asp Arg
395 400 405
Gln Leu Ile Gly Ala Ala Ala Ala Ser Pro Glu Gln Ala Pro Ala
410 415 420
Ser Ile Thr Pro Ser Val Pro Thr Thr Thr Ala Gly Lys Ser Ser
425 430 435
Arg Ser Arg Lys Ala Arg Met Arg Thr Ser Asp Ala Ser Ser Ser
440 445 450
Ala Ala Pro Ala Gln Arg Val Ile Asp Glu Arg Asn Ala Ala Gln
455 460 465
Lys Gln Ala Asp Glu Ile Leu Lys Ser Thr Arg Leu Glu Ser Leu
470 475 480
Pro Val Ala Glu Leu Gly Gly Asp Phe Ile Ala Leu Ala Lys Arg
485 490 495
Leu Gly Lys Asp Thr Thr Asp Ile Glu Arg Leu Ile Gly Asp Ser
500 505 510
Arg Arg Asp Ala Ala Thr Ala Phe Asp Phe Ala Arg Thr Ser Met
515 520 525
Gln Asp Trp Phe Gly Ser Ser Glu Arg Leu Leu Gln Leu Lys Ser
530 535 540
Lys Leu Arg Ala Gly Asp Pro Arg Ile Ala Gln Leu Asp Thr Arg
545 550 555
Leu Arg Leu Leu Gln Arg Ile Glu Gln Glu Phe Glu Arg Arg Glu
560 565 570
Ala Asp Ala Leu Lys Ile Asp Pro Gln Pro Arg Ala Pro His Leu
575 580 585
Gln Arg Leu Leu Ala Met His Ala Leu Ala Arg Val Thr Ala Pro
590 595 600
Asn Arg Leu Pro Ser Glu Asp Asp Arg Gly Gly Arg Gly Arg Leu
605 610 615
Phe Glu Val Arg Ile Asp His Thr Pro Gln Ser Asn Gly Asp Ile
620 625 630
Pro Ala Pro Trp Phe Val His Val His Thr Glu Lys Pro Val Thr
635 640 645
Pro Thr Gly Leu Arg Ala Leu His Tyr Lys Asp Leu Ala Ala Val
650 655 660
His Leu Lys Thr Ala Arg Glu Val Asn Leu Gly Pro Arg Trp Glu
665 670 675
Glu Met Met Arg Ala Leu Gly Asn Thr Glu Ala Lys Val His Arg
680 685 690
Ala Thr Ile Gly Ser Lys Leu Leu Gly Gln Leu Trp Ala Ala Gly
695 700 705
Ala Gly Gly Gln Gln
710
<210>5
<211>32
<212>DNA
<213> 人工序列
<400>5
cccaagcttg tgcctaaaat tgaatcgacc aa 32
<210>6
<211>26
<212>DNA
<213>人工序列
<400>6
gctctagatt atcgctgccc accagcgcca gc 32
<210>7
<211>32
<212>DNA
<213> 人工序列
<400>7
cccaagcttg tgccgagatt tcgagcgacc aa 32
<210>8
<211>32
<212>DNA
<213> 人工序列
<400>8
gctctagatt attgttgccc gccagcgcca gc 32
- 还没有人留言评论。精彩留言会获得点赞!
- 腈水解酶突变体及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白AsTMK1及其编码基因和应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种花狭口蛙的基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种泽陆蛙基因及其编码的多肽和该多肽的用途的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 蛋白质及其编码基因在调控植物抗黄萎病性中的应用的制造方法与工艺
- 甘薯耐逆相关蛋白IbFBA2、编码基因、重组载体与应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 植物抗旱相关蛋白EtSnRK2.2及其编码基因和应用的制造方法与工艺
- OsSAPK7蛋白及其编码基因在提高水稻白叶枯病抗性中的应用的制造方法与工艺
- 一种抗除草剂抗虫融合基因、编码蛋白及应用的制造方法与工艺
- 毛果杨PtrZFP103基因及其编码蛋白和应用的制造方法与工艺
- 大豆bHLH转录因子基因GmFER及其编码蛋白和应用的制造方法与工艺
- 一种水稻MIT1基因、其编码蛋白及应用的制造方法与工艺
- 蛋白StTrxF、编码基因及其在提高植物淀粉含量中的应用的制造方法与工艺
- 蛋白质及其编码基因在调控植物抗黄萎病性中的应用的制造方法与工艺
- 植物抗旱相关蛋白ZmNAC111及其编码基因与应用的制造方法与工艺
- 核盘菌凝集素SSL‑6蛋白及其编码基因与应用的制造方法与工艺