一种快速简易的基因多态性检测方法及试剂盒和应用与流程
本发明涉及分子生物学技术领域,具体涉及一种快速简易的基因多态性检测方法及试剂盒和应用。
背景技术:
多态性是指在一个生物群体中,同时和经常存在两种或多种不连续的变异型或基因型(genotype)或等位基因(allele),亦称遗传多态性(genetic polymorphism)或基因多态性。人类遗传基因多态性在阐明人体对疾病、毒物的易感性与耐受性、疾病临床表现的多样性以及对药物治疗的反应性上都起着重要的作用。疾病基因多态性的研究不但可以揭示同一种疾病不同临床表型的实质,也是个体化治疗的基础。
目前用于检测基因多态性的方法很多,主要有选择性扩增方法与选择性检测方法两类,其中选择性检测方法主要以TaqManTM探针为代表,Taqman探针是一种经典的定量PCR技术,现阶段得到广泛应用,在PCR反应体系中加入一对引物和一个特异性荧光探针,探针只与两条引物之间特异性模板结合,利用荧光信号积累实时监测整个PCR过程。探针的5’端连接报告荧光,3’端连接淬灭荧光,随后进行实时定量PCR。如果探针能够与DNA杂交,则在PCR用引物延伸时,TaqDNA聚合酶5’到3’的外切酶活性会将探针序列上5’端的报告荧光切下,淬灭荧光不再能对报告荧光进行抑制,使得报告荧光发光。针对突变位点分别设计两个不同荧光标记突变探针、野生探针,根据突变探针、野生探针荧光信号强度进行SNP位点基因型判读。该方法的缺点在于:首先该方法不存在选择性扩增机制,不同基因型结果差异不明显,每次做qPCR检测必须设置三个基因型对照,很难在单个样品检测的情况下进行准确精确基因分型。此外,由于突变型所占比例十分稀少,易导致信号淹没在与待检测目的序列突变型变异体相似的野生型变异体所产生的信号中,其选择性没有选择性扩增法的选择性高。
因此,等位基因选择性扩增允许我们选择性的扩增并检测目的核酸序列的基因多态性。在一个成功的选择性扩增系统中,只有目的核酸序列中的突变型变异体被扩增,而目的核酸序列的野生型变异体不被扩增、或者目的核酸序列的野生型变异体的扩增效率远远低于目的核酸序列中突变型变异体的扩增效率。为了达到选择性扩增的目的,多种方法被开发出来,如下述的PCR夹子方法、RFLP-PCR方法、数字PCR方法、低变性温度下共扩增PCR方法、等位基因特异性PCR法(ARMS-PCR法)等。
1、PCR夹子方法(PCR clamping method)通过抑制目的核酸序列的野生型变异体的扩增来达到选择性扩增待检测目的片段的目的。使用肽核酸(PNA)或使用锁核酸(LNA)的方法分别在文献[Henrik et al.,Nucleic Acid Research 21:5332-5336(1993)]和[Luo et al.,Nucleic Acid Research Vol.34,No 2 e12(2006)]中公开。但是,与目的核酸序列的野生型变异体与突变型变异体通常只有一个碱基与待检测目的片段不同,这种方法并不能完美的抑制住这些非目的片段的扩增。
2、RFLP分析法:基于基因突变导致的限制性内切酶识别位点的改变,如位点丢失或产生新位点,通过PCR扩增某一特定片段,再利用限制性内切酶进行酶切反应,电泳观察片段的大小,但绝大多数变异位点并不涉及酶切位点的改变,因此此方法不具备通用性,同样由于操作步骤多而难以进行高通量平行分析。
3、数字PCR(digital PCR)通过稀释模板和增加PCR反应的数目来检测少量目的核酸序列突变型变异体[Vogelstein B,Kinzler KW.Digital PCR.ProcNatlAcad SciUSA1999;96:9236-41]。理论上,当核酸模板稀释到每个PCR反应中仅有一个或没有核酸模板时,这个PCR反应中要么没有扩增,要么扩增野生型模板或扩增突变型模板。结合相应的检测方法即可以达到检测少量突变型变异体的目的。理论上,该方法通过增加PCR反应的数量选择性是无限的。但是在实际操作中,选择性不仅受限于Taq DNA聚合酶的保真性限制,也受限于同时能进行的PCR数目。虽然有报道基于数字PCR的方法具有高选择性,如[Bielas JH,Loeb LA.Quantification of random genomic mutations.Nat Methods 2005;2:285-90]所公开的内容。但该法通常需要专门的仪器并借助芯片技术,过程非常繁琐复杂,成本较高。
4、低变性温度下共扩增PCR(Coamplification at lower denaturation temperature PCR,Cold-PCR)是一种通过调节PCR反应的热循环条件来富集目的扩增片段的方法。在低变形温度下(Tc),目的核酸序列的突变型变异体或者突变型变异体与野生型变异体形成的异源二聚体相较于野生型变异体更容易变性而被进一步扩增并富集。该方法能提供一定的选择性,但是这种选择性仍然是不够的,如[Li J,Wang L,Mamon H,Kulke MH,Berbeco R,Makrigiorgos GM.Replacing PCR with COLD-PCR enriches variant DNA sequences and redefines the sensitivity of genetic testing.Nat Med 2008;14:579–84]所公开的内容。
5、ARMS-PCR法:ARMS-PCR(amplification refractory mutation system)又叫等位基因特异PCR,Taqman聚合酶缺少3’→5’外切酶活性,在一定条件下PCR产物3’末端的错配导致产物的急剧减少,针对不同的已知突变,分别设计突变引物、野生引物,2个引物主要是3’末端碱基不同。该方法的主要限制因素是:a)若突变的位点为弱错配碱基序列,ARMS引物不能有效区分目标核酸序列的野生型变异体和突变型变异体,从而使该方法的选择性受到影响,也会因此产生假阴性或假阳性结果。b)ARMS-PCR只含有选择性扩增机制而不具备选择性检测,无法进行基因型特异性检测,因此当选择性扩增出现错误时会导致完全错误的结论,即出现假阴性及假阳性结果。c)ARMS-PCR引物设计必须涵盖多态性位点区域,当这些区域有特殊序列或结构时,扩增(效率、特异性、选择性)会被影响。
以上所述方法都需对样本进行繁琐的DNA提取、整个检测耗时长。而在临床上,一些急性疾病如急性冠脉综合症、冠脉支架术和冠心病的患者在发病时需要快速服药,以免危及生命,由于不同个体对药物的代谢能力不同,需要在给药前进行药物代谢相关基因的检测,这就需要在尽量短的时间内得到测试结果。虽然现有的核酸提取方案很多,但是基因多态性的检测对于核酸样本的要求比较严格,核酸提取方案需要较为严格地适配采用的方法,否则会影响最终检测结果,因此有必要建立一种快速、简易和准确的基因多态性检测方法,为医院及其它医疗机构提供一种操作简单、特异性强、灵敏度高、通量高、可靠性强和成本低的检测方案,节省操作时间,快速获得实验结果并用于指导临床用药。
技术实现要素:
有鉴于此,本发明的目的在于提供一种快速简易的基因多态性检测方法,省去繁琐又费时的DNA提取步骤,将样品与样品处理液混合处理后,直接对所述的样品和样品处理液的混合液进行选择性扩增及选择性检测从而实现快速、可靠的基因多态性检测。
为了实现上述目的,本发明提供如下技术方案:
一种快速简易的基因多态性检测方法,该方法包括:
(1)将待测样品与样品处理液处理获得含有核酸的溶液;
(2)选择性扩增所述含有核酸的溶液中的目标核酸序列;
同时对目标核酸序列进行基因型特异性选择性检测,根据检测结果判断待测样品基因型;
所述样品处理液包括处理缓冲液、金属离子络合剂。
针对现有用于基因多态性检测的方法普遍需要进行繁琐的DNA提取过程的问题,本发明提供了一种快速简易的基因多态性检测方法,其中的样品处理液可快速实现对待测样品的简易处理,无需对样品进行DNA提取,省去了繁琐费时的DNA提取步骤,在不长于7min即可完成样品的快速处理,并直接对处理后的样品进行选择性扩增及基因型特异性选择性检测,并且满足检测对样品核酸的要求,保证检测结果的准确性。同时,本发明在此基础上提供了一种适配该核酸样品处理液的目标核酸序列选择性扩增引物,用于基因多态性的检测,样品处理到获取最终检测数据整个过程在1h内完成。
作为优选,所述处理缓冲液与扩增检测体系兼容,包括但不限于磷酸盐缓冲液、柠檬酸缓冲液、碳酸盐缓冲液、醋酸缓冲液、巴比妥酸缓冲液和Tris-HCl缓冲液;在本发明具体实施过程中可采用Tris-HCl缓冲液,所述Tris-HCl缓冲液浓度为10~100mM,pH7.0~10.0;更具体地,所述Tris-HCl的浓度为30mM,pH8.2。
作为优选,所述金属离子络合剂包括但不限于氨基羧酸盐类络合剂、羟基羧酸盐类络合剂、有机膦酸盐类络合剂和聚丙烯酸类络合剂。其中,所述氨基羧酸盐类络合剂可选择为EDTA。在本发明的具体实施过程中,所述EDTA浓度为1~20mM,pH7.5~8.5;更具体地,所述EDTA浓度为3mM,pH8.2。
作为优选,所述样品处理液还包括DNA释放促进剂,所述DNA释放促进剂包括但不限于去垢剂(阴离子、阳离子、兼性去垢剂)、蛋白酶K、胍盐或Chelex。在本发明的具体实施过程中可采用蛋白酶K,所述蛋白酶K浓度为0.01~20mg/ml;更具体地,所述蛋白酶K浓度为2mg/ml。
作为优选,步骤(1)为:将待测样品用样品处理液浸泡或混合,然后经56℃保温5min,98℃保温2min,获得含有核酸的溶液。本发明所述待测样本适用于人或动物的生理/病理体液(分泌物、渗出液等);人或动物的细胞悬液(血液、淋巴液、滑膜液、精液、唾液等);植物的生理/病理体液或者细胞悬液;细菌、真菌、质粒、病毒、寄生虫等的提取液或者悬液;人或动物机体组织(肝脏、肾脏)提取液或者组织匀浆;其他任何可以提供遗传物质的物质。在本发明具体实施过程中,所述待测样本为血液样本或刮取口腔内粘膜细胞的唾液试子样本。
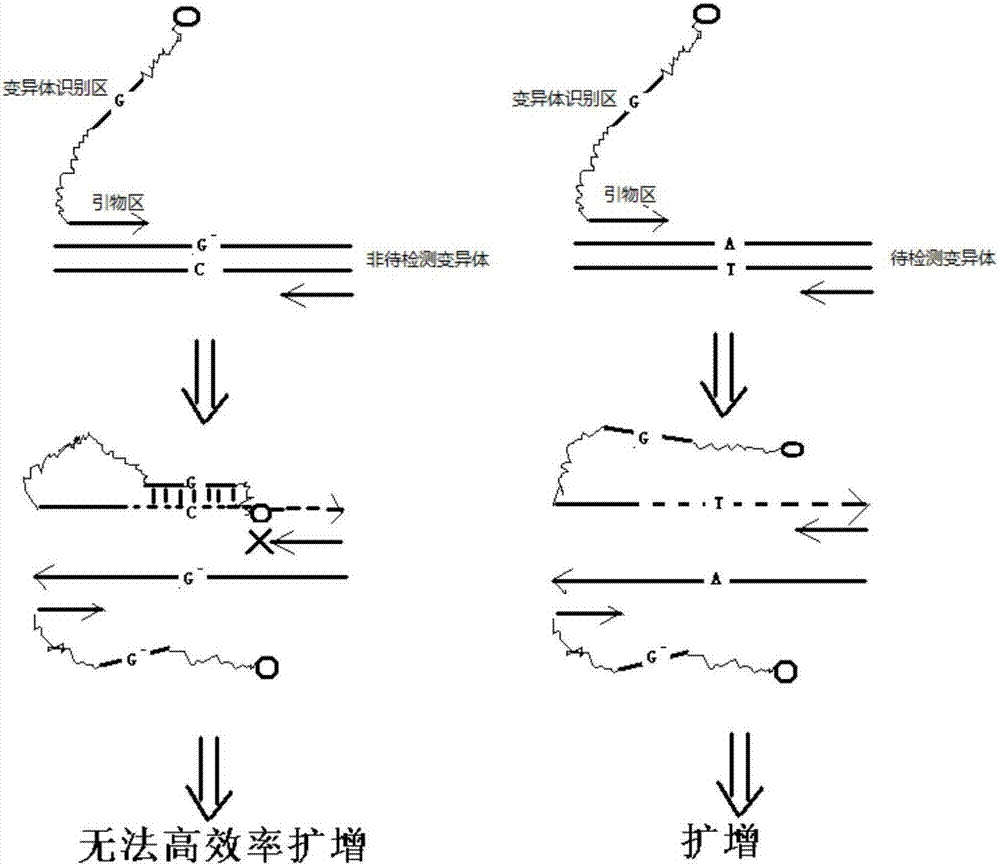
作为优选,所述选择性扩增采用选择性扩增引物,所述选择性扩增引物能有效区分目标核酸序列变异体与非目标核酸序列变异体。更进一步地,所述选择性扩增引物包括突变型扩增引物、野生型扩增引物,所述突变型扩增引物和野生型扩增引物均包含以共价相连或直接相连的引物区序列和变异体识别区序列,所述引物区序列在3’端,变异体识别区序列在5’端,所述变异体识别区序列连接有核酸双链稳定因子或含有不能被核酸酶水解的修饰因子,所述引物区序列能够从目标核酸基因多态性位点前开始扩增,扩增的位置一般为基因多态性位点前5-15bp,所述变异体识别区序列长度为10-20bp,突变型扩增引物变异体识别区序列包含目标核酸基因多态性位点处的正常碱基,野生型扩增引物变异体识别区序列包含目标核酸基因多态性位点处的突变碱基,两种变异体识别区序列均与所述引物区序列扩增延伸产生的序列形成发夹结构。
以突变性扩增引物为例,当野生型核酸(在图2中为非待检测变异体)作为模板时,突变性扩增引物变异体识别区与引物区延伸产生的序列,形成一种发夹结构(见图1);当突变型核酸(在图2中为待检测变异体)作为模板时,变异体识别区与引物区延伸产生的序列无法形成稳定的发夹结构。上述发夹结构的存在有效抑制了进一步的扩增,突变型核酸扩增过程中由于无法形成稳定的发夹结构,因此可以得到有效扩增从而实现选择性扩增(见图2)。在本发明中,即使野生型核酸在PCR一轮反应中得到了扩增,但是扩增产物在下一轮反应中仍然容易形成发夹结构而无法高效扩增,从而高效的抑制了野生型核酸的扩增效率,实现选择性扩增,不会出现现有特异性扩增方法中使用3’末端错配的引物一旦错误的延伸就相当于人为的引入突变型的变异体的现象。
同样地原理,以野生型扩增引物为例,当突变型核酸作为模板时,野生型扩增引物变异体识别区与引物区延伸产生的序列,形成一种发夹结构,无法高效扩增;当野生型核酸作为模板时,变异体识别区与引物区延伸产生的序列无法形成稳定的发夹结构,可以高效扩增。
由于SNP位点的突变位置、突变碱基在本领域已被广泛报道,本发明所述的突变型扩增引物、野生型扩增引物,可根据需要检测已被报道的目标基因的突变型序列以及野生型序列进行常规设计。在本发明具体实施例中,本发明就某些目标核酸给出了本发明的较优引物序列。
作为优选,所述修饰因子包括碱基类似物、核酸骨架、脱氧核糖类似物、肽核酸或硫代磷酸酯;所述核酸双链稳定因子包括锁核酸、肽核酸或DNA小沟结合物/类似物中的一种或一种以上的组合。在本发明具体实施过程中,所述核酸双链稳定因子可选自DNA小沟结合物,如MGB等。
作为优选,所述基因型特异性选择性检测使用Taqman荧光检测探针检测,包含突变型检测探针和野生型检测探针,该探针对于不同基因型有差异性结合,从而实现差异性检测。突变型检测探针和野生型检测探针可根据已被报道的目标基因的突变型序列以及野生型序列进行常规设计。在本发明具体实施例中,本发明就某些目标核酸给出了本发明的较优探针序列。
作为优选,本发明步骤(2)为:
采用荧光定量PCR选择性扩增所述含有核酸的溶液中的目标核酸序列;
同时对目标核酸序列进行基因型特异性选择性检测,根据PCR扩增结果的Ct值判断待测样品基因型,即Ct值<36±0.5为高效扩增,Ct值>38±0.5为非高效扩增,Ct的具体数值会因基因多态性检测位点、反应体系、样品来源、机器种类等差异而有差异。
在本发明步骤(2)中,突变型扩增引物和野生型扩增引物分别与公共扩增引物组成一对扩增引物,同时各自附加对应探针进行PCR扩增。采用突变型扩增引物进行PCR的一组,当Ct值<36±0.5时,表明能够高效扩增,即为突变型;同时,采用野生型扩增引物进行PCR的一组,当Ct值>38±0.5时,表明不能高效扩增,即待测样本基因型为突变型;
采用野生型扩增引物进行PCR的一组,当Ct值<36±0.5时,表明能够高效扩增,即为野生型;同时,采用突变型扩增引物进行PCR的一组,当Ct值>38±0.5时,表明不能高效扩增,即待测样本基因型为野生型;
采用野生型扩增引物进行PCR的一组,当Ct值<36±0.5时,表明能够高效扩增,即为野生型;同时,采用突变型扩增引物进行PCR的一组,当Ct值<36±0.5时,表明能够高效扩增,即待测样本基因型为杂合性(即同时存在野生型序列和突变型序列);
在本发明的试验中,本发明发现,不同的样品处理液处理后的待测样品在采用本发明上述方法进行检测时,出现了不同检测结果,完全采用本发明检测方法不仅能够对样品基因型分型,而且结果准确,更换了其他核酸样品处理液无法对样品基因型分型。同样地,同是本发明样品处理液处理的样本,采用不同的检测方法检测,本发明检测方法更加准确。两方面结果表明,基因多态性的检测对于核酸样本的要求比较严格,待测样品的处理方案需要较为严格地适配采用对应方法,否则会影响最终检测结果。
根据本发明所述方法的有益效果,本发明基于所述方法提供了一种检测基因多态性的试剂盒,包括样品处理液,目标核酸选择性扩增引物和目标核酸选择性检测探针。其中,所述样品处理液包括处理缓冲液、金属离子络合剂,所述目标核酸选择性扩增引物包括突变型扩增引物、野生型扩增引物,所述目标核酸选择性检测探针包括突变型检测探针和野生型检测探针。
作为优选,所述处理缓冲液为10~100mM,pH7.0~10.0的Tris-HCl,所述金属离子络合剂为1~20mM,pH7.5~8.5的EDTA。在本发明具体实施方式中,所述处理缓冲液为30mM,pH8.2的Tris-HCl,所述金属离子络合剂为3mM,pH8.2的EDTA。
作为优选,所述样品处理液还包含浓度为0.01~20mg/ml的蛋白酶K。此外,整个试剂盒还包括PCR缓冲液、MgCl2、去垢剂、DNA聚合酶和dNTP。其中,为了防止非特异性PCR扩增和污染,本发明用dUTP替代dTTP,同时加入UNG酶(尿嘧啶-N-糖基化酶)。因此,所述dNTP优选为dATP、dCTP、dGTP和dUTP,同时还包括UNG酶。
作为优选,所述突变型扩增引物以及野生型扩增引物可采用检测方法中所述限定。
基于本发明所述方法和试剂盒的特点和在基因多态性检测上的优势,本发明提出了所述试剂盒在基因多态性检测上的应用,包括单碱基多态性、碱基插入或缺失、微卫星分析等。
本发明具有下述积极效果:
1、检测速度快:本发明提供的方法将样品与样品处理液混合处理后,无需对样品进行DNA提取,省去了繁琐又费时的DNA提取步骤,在不长于7min即可完成样品的快速处理,并直接对所述样品和样品处理液的混合液进行选择性扩增及基因型特异性选择性检测,从样本处理到获取最终检测数据整个过程在1h内完成(当采用ABI7500Fast定量PCR仪器时)。
2、能准确检测基因多态性:采用特异性选择性扩增引物选择性扩增目的核酸序列变异体,而非目的核酸序列变异体无法扩增,在扩增的同时通过特异性选择性探针检测扩增产物,检测灵敏度高、特异性强,从而确保了基因分型的准确性。
由以上技术方案可知,本发明提供了一种快速高效的基因多态性检测试剂盒及检测方法,将待测样本与核酸样品处理液混合处理后,无需进行DNA提取步骤,在短时间内实现对样本的简易处理,并进一步通过选择性扩增引物与选择性检测探针实现对目标核酸序列野生型变异体与突变型变异体的有效区分,从样本处理到获取最终检测结果整个过程在1h内完成,并且保证检测的准确性。
附图说明
图1为发夹结构示意图;
图2为利用本发明特异性扩增引物实现选择性扩增的技术原理示意图;
图3为两组血液样本分别经两种方法处理后内参的扩增结果
图4使用本发明所述的分型方法对经两种方法处理后的一组血液样本的基因SLCO1B1第5外显子521T>C(Val174Ala)的检测结果;
图5使用本发明所述的分型方法对经两种方法处理后的一组血液样本的基因SLCO1B1第5外显子521T>C(Val174Ala)的检测结果;
图6为三组血液样本经本发明所述方法处理后内参的扩增结果;
图7为使用本发明所述方法对一组血液样本的基因APOE的rs429358(c.388T>C,Cys130Arg)野生型样本的检测结果;
图8为使用本发明所述方法对一组血液样本的基因APOE的rs429358(c.388T>C,Cys130Arg)杂合型样本的检测结果;
图9为使用本发明所述方法对一组血液样本的基因APOE的rs429358(c.388T>C,Cys130Arg)突变型样本的检测结果;
图10为两组血液样本经本发明所述方法处理后内参的扩增结果;
图11为使用本发明所述处理方法对两组血液样本快速处理后,分别用本发明所述分型方法与ARMS方法对CYP2C19*3突变型样本的检测结果;
图12为使用本发明所述处理方法对两组血液样本快速处理后,分别用本发明所述分型方法与ARMS方法对CYP2C19*3野生型样本的检测结果。
具体实施方式
本发明公开了一种快速简易的基因多态性检测方法及试剂盒和应用,本领域技术人员可以借鉴本文内容,适当改进工艺参数实现。特别需要指出的是,所有类似的替换和改动对本领域技术人员来说是显而易见的,它们都被视为包括在本发明。本发明所述试剂盒和检测方法已经通过较佳实施例进行了描述,相关人员明显能在不脱离本发明内容、精神和范围内对本文所述的方法和应用进行改动或适当变更与组合,来实现和应用本发明技术。
在本发明中,除非特别定义,本专利所有的科学或技术专业词汇均与本领域大部分一般人员的普通理解一致。以下的文献中本领域中大部分专业名词的一般定义:[Singleton et al.,Dictionary of Microbiology and Molecular Biology(2nd ed.1994)];[The Cambridge Dictionary of Science and Technology(Walker ed.,1988)];[The Glossary of Genetics,5th Ed.,R.Rieger et al.(eds.),Springer Verlag(1991)];[Hale&Marham,The Harper Collins Dictionary of Biology(1991)]除非另外定义,本专利中所使用的专业名词与上述文献中对该专业名词描述一致。
名词“核酸”包括脱氧核糖核酸(DNA),核糖核酸(RNA),DNA-RNA杂交体,寡聚核苷酸,适配体(aptamers),肽核酸(PNAs),PNA-DNA杂交体,PNA-RNA杂交体等等。包括一切线性形式(单链或双链)或分支状形式的共价相连的核苷酸。一个典型的核酸通常是单链或者双链的,并包含磷酸二脂键。
名词“目标核酸序列”指的是待扩增的一段核酸序列,该序列作为核酸扩增的模板。
名词“核酸序列变异体”、“核酸变异体”指的是一段特定的核酸序列,不同变异体之间存在差别,这种差别可以是单个或多个碱基的不同,也可以是插入、缺失、易位或者以上所有类型的组合。
名词“野生型变异体”指的是自然存在的“正常”的基因序列,它在大多数群体中同一基因序列出现频率最高的序列。
名词“突变型变异体”指的是相比于“野生型变异体”不同的核酸序列。这种不同可以是单个或多个碱基的不同,也可以是插入或缺失或者以上所有类型的组合,例如,肿瘤细胞中突变的核酸序列。在一些实施例中,相较于野生型变异体,突变型变异体所占比例很少。
名词“扩增”指的是目的核酸片段在核酸聚合酶的作用下数目变多的过程,包括但不限于聚合酶链式反应(PCR),连接酶链式反应(LCR),核酸序列基础扩增(NASBA),转录介导扩增(TMA),环介导等温扩增(LAMP),链置换扩增(SDA),解旋酶依赖扩增(HDA)等。
在本发明实施例中,扩增指的是聚合酶链式反应(PCR)。模板变性解链,寡聚核苷酸引物与模板退火杂交,伴随着核苷酸加入的延伸,如此反复循环一定轮数,实现目的核苷酸片段的增多。
名词“突变”指细胞中的遗传基因发生的改变。它包括单个碱基改变所引起的点突变,或多个碱基的缺失、重复和插入。
在本发明实施例中,核酸扩增实验是荧光定量PCR实验。在荧光定量PCR反应中,衡量扩增的参数是Ct值,较早的Ct值反应的是信号更快的达到阈值。扩增样品中不同核酸变异体之间的Ct差值,往往反映了扩增体系对不同核酸变异体扩增效率的差异,进一步反应了扩增体系的选择性。
对扩增产物的检测在本领域中也有很多方法。这些方法包括使用荧光标记的探针,或者各种与核酸结合的染料。这些检测可以是特异性的检测核酸变异体中一种或者多种,也可以是非选择性的检测所有核酸信号。对扩增产物的检测可以发生在扩增反应完成之后,比如通过凝胶电泳的方法,或者对核酸进行染色的方法。另外,对扩增产物的检测也可以发生在扩增反应的过程之中。
以下就本发明所提供的一种快速简易的基因多态性检测方法及试剂盒和应用做进一步说明。
实施例1:使用本发明所述的方法实现对血液样本的快速处理
在这个实施例中,通过使用本发明所述的方法对5组血液进行40倍稀释后经快速处理。其中处理液为20mM Tris-HCl pH8.0,2mM EDTA,2mg/ml蛋白酶K,将2ul血液与78ul该处理液混合,并充分混匀后经56℃保温5min,98℃保温2min,粗制样本即处理完成。
实施例2:使用本发明所述的方法实现对唾液试子样本的快速处理
在这个实施例中,使用专用唾液试子刮取口腔内粘膜细胞,用剪刀剪取部分试子或者将整个试子进行浸泡,其中处理液为20mM Tris-HCl pH8.0,2mM EDTA,2mg/ml蛋白酶K,所需体积浸泡以处理液全部覆盖试子为宜,并充分混匀后经56℃保温5min,98℃保温2min,粗制样本即处理完成。
实施例3:使用专利201110295395.9发明所述的方法实现对样本的快速处理
在这个实施例中,使用水配置含10wt%chelex-100作为裂解液,将2ul血液与18ul裂解液混合均匀后,在56℃下,裂解30分钟,100℃煮沸8分钟即可获得所述DNA样本。
实施例4:使用本发明所述的方法及201110295395.9发明所述的方法实现对血液样本的有机阴离子转运多肽1B1(OATP1B1,又称OATP-C、OATP2或LST1)编码基因SLCO1B1第5外显子521T>C(Val174Ala)的选择性扩增与检测并对比样本处理方法。
在这个实施例中,通过使用本发明所述的实施例1中的方法及实施例3中201110295395.9发明所述的方法对两组血液进行处理,然后结合本发明所述分型方法对基因SLCO1B1第5外显子521T>C(Val174Ala)进行选择性扩增与检测。突变型与野生型分两个独立管进行检测,同时检测内参基因作为质控,被检测基因与内参基因同时进行双重PCR扩增。
OATP1B1野生型序列(SEQ ID NO:1所示)为:
5’-TCCCCTATTCCACGAAGCATATTACCCATGAACA(多态性位点)CATATATCCACATGTATGACCCAGATTCCTTTAAACAACCTATGTAGATAAGAGAGTGTTTCATTTTAAACATTAA-3’
OATP1B1突变型序列(SEQ ID NO:2所示)为:
5’-TCCCCTATTCCACGAAGCATATTACCCATGAACG(多态性位点)CATATATCCACATGTATGACCCAGATTCCTTTAAACAACCTATGTAGATAAGAGAGTGTTTCATTTTAAACATTAA-3’
OATP1B1野生型上游扩增引物序列(SEQ ID NO:3所示)为:5’-TGCGTTCAT(变异体识别区,下划线碱基为多态性位点)TCCCCTATTCCACGAAGCATATTAC(引物区)-3’(5’端MGB修饰,Spacer C3连接引物区序列和变异体识别区序列)
OATP1B1突变型上游扩增引物序列(SEQ ID NO:4所示)为:5’-TGTGTTCAT(变异体识别区,下划线碱基为多态性位点)TCCCCTATTCCACGAAGCATATTAC(引物区)-3’(5’端MGB修饰,Spacer C3连接引物区序列和变异体识别区序列)
OATP1B1公共下游引物(SEQ ID NO:5所示)为:5’-AGGAATCTGGGTCATACATGT-3’
OATP1B1野生型检测探针序列(SEQ ID NO:6所示)为:5’-TATATGTGTTCATGGGTA-3’(5’FAM,3’MGB)
OATP1B1突变型检测探针序列(SEQ ID NO:7所示)为:5’-ATATATGCGTTCATGGGT-3’(5’FAM,3’MGB)
内参序列(SEQ ID NO:8所示)为:
5’-CGGACTGAAGGAGCTGCCCATGAGAAATTTACAGGGTGAGAGGCTGGGATGCCAAGGCTGGGGGTTCATAAATGCAGACAGCAGTTCCGATGGC-3’
内参序列正向引物序列(SEQ ID NO:9所示)为:5’-CGGACTGAAGGAGCTGCC-3’
内参序列反向引物序列(SEQ ID NO:10所示)为:5’-GCCATCGGAACTGCTGTCTG-3’
内参序列检测探针序列(SEQ ID NO:11所示)为:5’-CCCCAGCCTTGGCATCCCA-3’(5’端Cy5,3’端BHQ2)
PCR反应体系为25μl,每个反应体系中含有2%甘油,dATP、dCTP、dGTP、dTTP各200μM,正向引物、反向引物各200nM,检测探针200nM,1个单位的热启动Taq DNA聚合酶及0.2mg/ml的BSA。其中1个单位是指在72℃30分钟掺入10nmoldNTPs所需要的酶量。
使用ABI 7500Fast荧光定量PCR仪进行PCR反应和后续的数据分析。PCR热循环条件为95℃,2min;40循环(95℃,3s;60℃,25s,单循环)。
实验结果见图3-5,OATP1B1基因实验的结果通过在465nM-510nM的荧光变化来体现的,内参基因实验的结果通过在618nM-660nM的荧光变化来体现的。
图3中可见血液经两种方法处理后两组血液间扩增有显著差异(左右图各为一组样本),本发明所述方法的扩增效果及所获得的模板量显著优于对比方法,可获得更充足的模板量及更小的PCR扩增抑制性。
图4中可见该组血液样本为野生纯合型样本,本发明方法仅野生型检测孔存在扩增,突变型检测孔无扩增,而对比方法野生孔与突变孔均基本扩增失败,无法有效分型。
图5中可见该组血液样本同样为野生纯合型样本,本发明方法仅野生型检测孔存在扩增,突变型检测孔无扩增,而对比方法野生孔与突变孔均基本扩增失败,无法有效分型。
实施例5:使用本发明所述的方法实现对载脂蛋白E(Apolipoprotein E,APOE)编码基因APOE的rs429358(c.388T>C,Cys130Arg)的选择性扩增与检测
在这个实施例中,分别选择三组血液样本,经实施例1所描述的方法进行快速处理并结合本发明所述分型方法用于对基因APOE的rs429358(c.388T>C,Cys130Arg)的选择性扩增与检测。突变型与野生型分两个独立管进行检测,同时检测内参基因作为质控,被检测基因与内参基因同时进行双重PCR扩增。
野生型序列(SEQ ID NO:12所示)为:
5’-GCAGGCCCGGCTGGGCGCGGACATGGAGGACGTGT(多态性位点)GCGGCCGCCTGGTGCAGTACCGCGGCGAGGTGCAGGCCATGCTCGGCCAGAGCACCGAGGAGCTGCGGGTGCGCCTCGC-3’
突变型序列(SEQ ID NO:13所示)为:
5’-GCAGGCCCGGCTGGGCGCGGACATGGAGGACGTGC(多态性位点)GCGGCCGCCTGGTGCAGTACCGCGGCGAGGTGCAGGCCATGCTCGGCCAGAGCACCGAGGAGCTGCGGGTGCGCCTCGC-3’
野生型上游扩增引物序列(SEQ ID NO:14所示)为:5’-CGCGCACGTC(变异体识别区,下划线碱基为多态性位点)TGGGCGCGGACATGG(引物区)-3’(5’端MGB修饰,Spacer C3连接引物区序列和变异体识别区序列)
突变型上游扩增引物序列(SEQ ID NO:15所示)为:5’-CGCACACGTC(变异体识别区,下划线碱基为多态性位点)TGGGCGCGGACATGG(引物区)-3’(5’端MGB修饰,Spacer C3连接引物区序列和变异体识别区序列)
公共下游引物(SEQ ID NO:16所示)为:5’-CGCCGCGGTACTGCACCAGG-3’
野生型检测探针序列(SEQ ID NO:17所示)为:5’-CGCACACGTCCTC-3’(5’FAM,3’MGB)
突变型检测探针序列(SEQ ID NO:18所示)为:5’-CGCGCACGTCC-3’(5’FAM,3’MGB)
内参各种序列同实施例4。
PCR反应体系为25μl,每个反应体系中含有2%甘油,dATP、dCTP、dGTP、dTTP各200μM,正向引物、反向引物各200nM,检测探针200nM和1个单位的热启动Taq DNA聚合酶。其中1个单位是指在72℃30分钟掺入10nmoldNTPs所需要的酶量。
使用ABI 7500Fast荧光定量PCR仪进行PCR反应和后续的数据分析。PCR热循环条件为95℃,2min;40循环(95℃,3s;60℃,25s,单循环)。
实验结果见图6、图7、图8和图9,APOE基因实验的结果通过在465nM-510nM的荧光变化来体现的,内参基因实验的结果通过在618nM-660nM的荧光变化来体现的。图6中可见血液经本发明方法处理后三组血液内参均高效扩增,获得足够PCR反应所需模板量。
图7中可见该组血液样本为野生纯合型样本,仅野生型检测孔存在扩增,突变型检测孔无扩增。
图8中可见该组血液样本为杂合型样本,野生型检测孔与突变型检测孔均存在扩增。
图9中可见该组血液样本为突变纯合型样本,仅突变型检测孔存在扩增,野生型检测孔无扩增。
实施例6:使用本发明所述的方法与ARMS方法对Cytochrome P4502C19细胞色素P450同工酶2C19对应位点CYP2C19*3的检测对比
在这个实施例中,分别选择两组血液样本,经实施例1所描述的方法进行快速处理并结合本发明所述分型方法及对比的ARMS方法用于对基因CYP2C19*3的检测。突变型与野生型分两个独立管进行检测,同时检测内参基因作为质控,被检测基因与内参基因同时进行双重PCR扩增。
CYP2C19*3野生型序列(SEQ ID NO:19所示)为:
5’-AACTTGATGGAAAAATTGAATGAAAACATCAGGATTGTAAGCACCCCCTGG(多态性位点)ATCCAGGTAAGGCCAAGTTTTTTGCTTCCTGAGAAACCACTTACAG-3’
CYP2C19*3突变型序列(SEQ ID NO:20所示)为:
5’-AACTTGATGGAAAAATTGAATGAAAACATCAGGATTGTAAGCACCCCCTGA(多态性位点)ATCCAGGTAAGGCCAAGTTTTTTGCTTCCTGAGAAACCACTTACAG-3’
CYP2C19*3野生型上游扩增引物序列(SEQ ID NO:21所示)为:
5’-CCCTGAATCCA(变异体识别区,下划线碱基为多态性位点)AGGAAGCAAAAAACTTGGCCTT(引物区)-3’(5’端MGB修饰,Spacer C3连接引物区和变异体识别区)
CYP2C19*3突变型上游扩增引物序列(SEQ ID NO:22所示)为:
5’-CCCTGGATCCA(变异体识别区,下划线碱基为多态性位点)AGGAAGCAAAAAACTTGGCCTT(引物区)-3’(5’端MGB修饰,Spacer C3连接引物区序列和变异体识别区序列)
CYP2C19*3公共下游引物(SEQ ID NO:23所示)为:
5’-GAATGAAAACATCAGGATTGTAAGCA-3’
CYP2C19*3野生型检测探针序列(SEQ ID NO:24所示)为:5’-CCCTGGATCCAGGT-3’(5’FAM,3’MGB)
CYP2C19*3突变型检测探针序列(SEQ ID NO:25所示)为:5’-CCCTGAATCCAGGTA-3’(5’FAM,3’MGB)
野生型正向ARMS引物序列(SEQ ID NO:26所示)为:
5’-AAAAACTTGGCCTTACCTGGATC-3’
突变型正向ARMS引物序列(SEQ ID NO:27所示)为:
5’-AAAAAACTTGGCCTTACCTGGATT-3’
公共ARMS下游引物(SEQ ID NO:28所示)为:5’-TCCCTGCAATGTGATCTGCTC-3’
公共ARMS检测探针序列(SEQ ID NO:29所示)为:5’-CAATCCTGATGTTTTC-3’(5’FAM,3’MGB)
内参各种序列同实施例4。
PCR反应体系为25μl,每个反应体系中含有2%甘油,dATP、dCTP、dGTP、dTTP各200μM,正向引物、反向引物各200nM,检测探针200nM和1个单位的热启动Taq DNA聚合酶。其中1个单位是指在72℃30分钟掺入10nmoldNTPs所需要的酶量。
使用ABI 7500Fast荧光定量PCR仪进行PCR反应和后续的数据分析。PCR热循环条件为95℃,2min;40循环(95℃,3s;60℃,25s,单循环)。
实验结果见图10、图11、图12,CYP2C19*3基因实验的结果通过在465nM-510nM的荧光变化来体现的,内参基因实验的结果通过在618nM-660nM的荧光变化来体现的。
图10中可见血液经本发明方法处理后两组血液内参均高效扩增(左右图各为一组样本),获得足够PCR反应所需模板量。
图11中可见使用本发明所提供检测方法确定该组血液样本为杂合型样本,本发明方法野生型检测孔与突变型检测孔均存在扩增,而对比的ARMS方法野生孔基本扩增失败,同时突变孔扩增明显,无法有效分型甚至错误判定为突变纯合型。
图12中可见使用本发明所提供检测方法确定该组血液样本为野生纯合型样本,本发明方法仅野生型检测孔存在扩增,突变型检测孔无扩增,而对比的ARMS方法野生孔基本扩增失败,同时突变孔完全无扩增,无法有效分型。
综合图11,图12中可见ARMS引物探针组合区分效果显著差于该发明所采用的引物探针组合的分型效果。
此外所有实施例中涉及分型的样本的实际分型均经金标准Sanger测序法确认,与本发明所得结论一致。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
SEQUENCE LISTING
<110> 苏州新海生物科技股份有限公司
<120> 一种快速简易的基因多态性检测方法及试剂盒和应用
<130> MP1621944
<160> 29
<170> PatentIn version 3.3
<210> 1
<211> 110
<212> DNA
<213> 人工序列
<400> 1
tcccctattc cacgaagcat attacccatg aacacatata tccacatgta tgacccagat 60
tcctttaaac aacctatgta gataagagag tgtttcattt taaacattaa 110
<210> 2
<211> 110
<212> DNA
<213> 人工序列
<400> 2
tcccctattc cacgaagcat attacccatg aacgcatata tccacatgta tgacccagat 60
tcctttaaac aacctatgta gataagagag tgtttcattt taaacattaa 110
<210> 3
<211> 34
<212> DNA
<213> 人工序列
<400> 3
tgcgttcatt cccctattcc acgaagcata ttac 34
<210> 4
<211> 34
<212> DNA
<213> 人工序列
<400> 4
tgtgttcatt cccctattcc acgaagcata ttac 34
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<400> 5
aggaatctgg gtcatacatg t 21
<210> 6
<211> 18
<212> DNA
<213> 人工序列
<400> 6
tatatgtgtt catgggta 18
<210> 7
<211> 18
<212> DNA
<213> 人工序列
<400> 7
atatatgcgt tcatgggt 18
<210> 8
<211> 94
<212> DNA
<213> 人工序列
<400> 8
cggactgaag gagctgccca tgagaaattt acagggtgag aggctgggat gccaaggctg 60
ggggttcata aatgcagaca gcagttccga tggc 94
<210> 9
<211> 18
<212> DNA
<213> 人工序列
<400> 9
cggactgaag gagctgcc 18
<210> 10
<211> 20
<212> DNA
<213> 人工序列
<400> 10
gccatcggaa ctgctgtctg 20
<210> 11
<211> 19
<212> DNA
<213> 人工序列
<400> 11
ccccagcctt ggcatccca 19
<210> 12
<211> 114
<212> DNA
<213> 人工序列
<400> 12
gcaggcccgg ctgggcgcgg acatggagga cgtgtgcggc cgcctggtgc agtaccgcgg 60
cgaggtgcag gccatgctcg gccagagcac cgaggagctg cgggtgcgcc tcgc 114
<210> 13
<211> 114
<212> DNA
<213> 人工序列
<400> 13
gcaggcccgg ctgggcgcgg acatggagga cgtgcgcggc cgcctggtgc agtaccgcgg 60
cgaggtgcag gccatgctcg gccagagcac cgaggagctg cgggtgcgcc tcgc 114
<210> 14
<211> 25
<212> DNA
<213> 人工序列
<400> 14
cgcgcacgtc tgggcgcgga catgg 25
<210> 15
<211> 25
<212> DNA
<213> 人工序列
<400> 15
cgcacacgtc tgggcgcgga catgg 25
<210> 16
<211> 20
<212> DNA
<213> 人工序列
<400> 16
cgccgcggta ctgcaccagg 20
<210> 17
<211> 13
<212> DNA
<213> 人工序列
<400> 17
cgcacacgtc ctc 13
<210> 18
<211> 11
<212> DNA
<213> 人工序列
<400> 18
cgcgcacgtc c 11
<210> 19
<211> 97
<212> DNA
<213> 人工序列
<400> 19
aacttgatgg aaaaattgaa tgaaaacatc aggattgtaa gcaccccctg gatccaggta 60
aggccaagtt ttttgcttcc tgagaaacca cttacag 97
<210> 20
<211> 97
<212> DNA
<213> 人工序列
<400> 20
aacttgatgg aaaaattgaa tgaaaacatc aggattgtaa gcaccccctg aatccaggta 60
aggccaagtt ttttgcttcc tgagaaacca cttacag 97
<210> 21
<211> 33
<212> DNA
<213> 人工序列
<400> 21
ccctgaatcc aaggaagcaa aaaacttggc ctt 33
<210> 22
<211> 33
<212> DNA
<213> 人工序列
<400> 22
ccctggatcc aaggaagcaa aaaacttggc ctt 33
<210> 23
<211> 26
<212> DNA
<213> 人工序列
<400> 23
gaatgaaaac atcaggattg taagca 26
<210> 24
<211> 14
<212> DNA
<213> 人工序列
<400> 24
ccctggatcc aggt 14
<210> 25
<211> 15
<212> DNA
<213> 人工序列
<400> 25
ccctgaatcc aggta 15
<210> 26
<211> 23
<212> DNA
<213> 人工序列
<400> 26
aaaaacttgg ccttacctgg atc 23
<210> 27
<211> 24
<212> DNA
<213> 人工序列
<400> 27
aaaaaacttg gccttacctg gatt 24
<210> 28
<211> 21
<212> DNA
<213> 人工序列
<400> 28
tccctgcaat gtgatctgct c 21
<210> 29
<211> 16
<212> DNA
<213> 人工序列
<400> 29
caatcctgat gttttc 16
- 还没有人留言评论。精彩留言会获得点赞!
- 一种基于HRM检测基因多态性的引物组合物的应用的制作方法与工艺
- MDR1基因多态性检测体系及其试剂盒的制作方法与工艺
- GSTP1基因多态性检测体系及其试剂盒的制作方法与工艺
- 一种检测MTHFR基因rs1801131多态位点基因型的方法及试剂盒与流程
- 检测10个位点耳聋基因多态性的SNaPshot试剂盒的制造方法与工艺
- 一种检测人类CYP2C19基因多态性的方法及试剂盒与流程
- CDA基因多态性检测体系及其试剂盒的制造方法与工艺
- 检测22个位点耳聋基因多态性的SNaPshot试剂的应用的制造方法与工艺
- 检测22个位点耳聋基因多态性的SNaPshot试剂盒的制造方法与工艺
- DHFR基因多态性检测体系及其试剂盒的制造方法与工艺
- ERCC1基因多态性检测体系及其试剂盒的制造方法与工艺
- 用于检测CYP2C19基因多态性的引物、探针及检测试剂盒的制造方法与工艺
- 高脂血症风险关联基因多态性检测方法与制造工艺
- 用于检测AGTR1基因的A1166C多态性位点的核酸、试剂盒及方法与制造工艺
- 一种检测与结直肠癌靶向用药西妥昔单抗治疗敏感性相关基因的多态性的试剂盒及其应用的制造方法与工艺
- 线粒体基因组9bp序列基因多态性在评价苯中毒风险中的应用的制造方法与工艺
- 一种rs12979860基因分型双色荧光PCR快速检测试剂盒的制造方法与工艺
- CYP2C19基因多态性检测引物、探针及试剂盒的制造方法与工艺
- 检测CYP2C9和VKORC1基因多态性的引物、探针和试剂盒的制造方法与工艺
- 一种检测CYP2C9基因多态性的ARMS引物的制造方法与工艺
- HaeIII检测食管癌易感基因PTEN多态性方法与流程
- 用于检测人类ADRB1基因 G1165C多态性的核酸、试剂盒及方法与流程
- 一种用于检测CYP2C9和VKORC1基因多态性的组合物及其应用的制作方法与工艺
- 一种用于检测CYP2C19基因多态性的组合物及其应用的制作方法与工艺
- 一种CYP2C19基因多态性检测试剂盒及检测方法与流程
- ERCC1基因突变检测特异性引物和液相芯片试剂盒的制作方法与工艺
- 一种IL28B基因多态性检测试剂盒的制造方法与工艺
- 检测10个位点耳聋基因多态性的SNaPshot试剂盒的制造方法与工艺
- 一种检测人类CYP2C19基因多态性的方法及试剂盒与流程
- 检测22个位点耳聋基因多态性的SNaPshot试剂的应用的制造方法与工艺