一种BRCA1/2基因检测文库的构建方法和试剂盒与流程
本发明涉及基因检测
技术领域:
,尤其涉及一种brca1/2基因检测文库的构建方法和试剂盒。
背景技术:
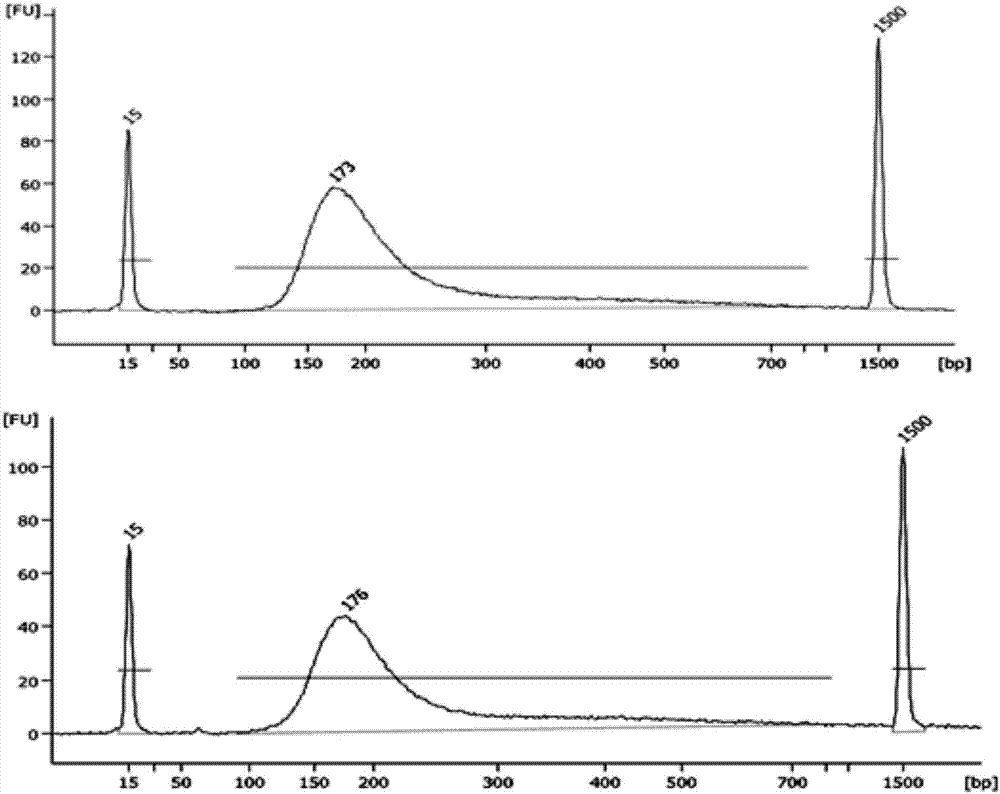
:根据国家癌症中心在《临床医师癌症杂志》上公布的2015年癌症统计数据,中国妇女卵巢癌年发病5.21万例,年死亡2.25万例,死亡率约为43%,为妇科三大肿瘤之首。乳腺癌年发病27.2万例,年死亡7.07万例,死亡率约为26%。另外,美国癌症学会研究显示,i型乳腺癌的患者5年生存率达到88%,iv型乳腺癌的5年生存率只有15%,这说明级别越高,生存率越低;发现越早,生存率越高。因此,采用合理的预防措施对于乳腺癌及卵巢癌的“早发现,早诊断,早治疗”具有重要意义。随着人类基因密码的解开,愈来愈多的疾病被发现与遗传因子有关,尤其对于癌症而言,遗传物质的差异对疾病的发生率、疾病的临床过程、以及对不同治疗的反应程度都将有一定程度的影响。基因作为遗传信息的记录体和传递者可以反映出个体肿瘤的遗传携带情况并用于评估个体易感风险。brca1/2是两种具有抑制恶性肿瘤发生的基因,在调节人体细胞的复制、遗传物质dna损伤修复、细胞的正常生长方面有重要作用。目前在esmo及nccn指南中对于乳腺癌和卵巢癌高危人群进行brca1/2基因检测均有推荐。据文献报道,约有5%~10%的乳腺癌和10~18%的卵巢癌是由生殖系brca1/2突变引起的。据统计,约有50%的遗传性乳腺癌和90%的遗传性卵巢癌有生殖细胞上的brca1突变;约有40%的遗传性乳腺癌和5%~10%的遗传性卵巢癌伴有brca2的突变。在家族遗传性乳腺癌和/或卵巢癌患者亲属中,brca1携带者、brca2携带者和非brca1/brca2携带者的比例分别约为65%、75%、43.5%。此外,一项综合研究调查结果表明:在brca1突变携带者中,70岁时发生乳腺癌的机率为65%,发生卵巢癌的机率为39%;在brca2突变携带者中,70岁时发生乳腺癌的和卵巢癌的机率分别为45%和11%。因此,brca1/2基因突变检测对于高危人群乳腺癌和卵巢癌风险评估具有积极的作用。此外,研究发现多聚adp-核糖聚合酶(parp)抑制剂可以通过抑制肿瘤细胞dna损伤修复、促进肿瘤细胞发生凋亡,从而增强放疗以及烷化剂和铂类药物化疗的疗效。不仅如此,parp抑制剂作为单药对brca突变的患者也有显著疗效。由于brca有害突变的癌症患者体内基因重组功能已经缺失,再通过parp抑制剂抑制dna的修复,就可以通过协同致死作用杀死肿瘤细胞。2014年12月,fda已经批准parp抑制剂奥拉帕尼(olaparib)单药治疗brca1/2有害胚系突变的晚期卵巢癌患者。另外还有其他多种parp抑制剂在临床第一、二、三期科研阶段。检测brca基因的突变可以指导未来parp抑制剂的合理用药。因此,进行brca1/2的基因突变检测尤其对于卵巢癌的“早发现,早诊断,早治疗”具有重要的临床意义。brca1/2基因大、序列长。根据bic数据库(breastcancerinformationcore,http://research.nhgri.nih.gov/bic/),brca1/2基因目前已报道的变异类型包括无义突变、移码突变、非移码插入、非移码缺失、错义突变、同义突变、剪接位点突变,以及发生在非翻译区和内含子区域的突变,涉及到近4000个变异位点,而这些突变多数基本上散布于各个外显子,没有证据表明存在突变热点区域,只存在少数几个相对高频的变异位点。如果采用传统的sanger测序或qpcr检测的方法来对brca1/2整个基因进行检测,则需要耗费大量的时间且价格不菲。相比之下,二代测序技术可则更为高效快速且价格低廉的,可以一次性对brca1/2基因整个外显子及邻近内含子进行变异检测,变异类型可包括点突变(snp)、插入缺失(indel)、基因融合及拷贝数变化(cnv),检测十分全面。聚合酶链式反应结合sanger测序检测brca1/2突变,以myriadgenetics公司的bracanalysiscdx试剂盒为例,根据检测目标设计大量引物对对血液来源gdna进行pcr扩增,从而对brca1/2基因进行富集,随后采用一代测序法进行检测,再根据已知的原始序列找出对应的突变。针对不同的变异类型如单核苷酸变异、短片段插入缺失与大片段缺失或重复,需要设计不同的引物。聚合酶链式反应结合sanger测序检测brca1/2突变存在很大局限性,表现在:该方法更适合于候选致病位点明确的疾病,而对于候选基因或位点数目较多的大样本病例筛查是难以完成的,该方法本身是高成本、高人工投入的。另外,sanger测序不能检测出大片段缺失或拷贝数变异等基因突变的类型;对于连续多个碱基的小片段插入、缺失检出准确度下降或者不能检出;检测过程复杂,同一次检测中单核苷酸变异、短片段插入缺失与大片段缺失与重复需要两次pcr反应单独检测。二代测序技术检测brca1/2突变,需要制备二代测序文库。制备文库中的基因组dna片段化方法主要是通过物理打断或化学打断来实现的。物理打断需要借助打断仪,基于超声剪切原理可将dna打断成100-1500bp片段;化学打断法主要依赖于酶打断法,经酶打断后能产生100-800bp的片段。dna双链片段化利用的是一种时间依赖型酶,根据不同的反应时间,将双链dna切割成100-800片段。文库构建一般需要分步骤进行,物理打断法及常规的酶打断法进行片段化后需要进一步通过凝胶电泳或磁珠选择合适大小的片段筛选,随后进行末端修复、加a、加接头等步骤,步骤多且繁琐,操作时间长,容易造成污染。通过物理打断法对dna进行打断需要投入打断仪,增加建库成本、仪器维护成本以及人工操作成本,而且打断需求的原始进样体积较大。因此必须要经过磁珠纯化富集dna片段,才能进行后续的建库步骤,进一步增加了人工操作及片段损失。技术实现要素:本发明提供一种brca1/2基因检测文库的构建方法和试剂盒,建库步骤简便快速,有效降低成本,减少工作量,变异类型全面、准确、通量高。根据本发明的第一方面,本发明提供一种brca1/2基因检测文库的构建方法,包括:(1)在dna片段化及末端修复反应体系中,在打断酶、末端修复酶的作用下,将均一化的dna样本片段化并末端修复;(2)在接头连接体系中,在dna连接酶的作用下,将步骤(1)所得的产物与接头序列连接;(3)以步骤(2)所得的接头连接产物为模板,在dna聚合酶的作用下,使用接头序列的引物进行pcr扩增;(4)使用包含brca1/2基因的探针序列与步骤(3)的产物进行杂交以捕获含有上述brca1/2基因的dna片段,并洗脱以获取杂交捕获产物;(5)在单链环化体系中,在连接酶的作用下,使上述杂交捕获产物的单链实现环化,得到上述brca1/2基因检测文库。进一步地,上述接头序列带有用于区分不同dna样本的条形码序列,每一上述条形码序列对应一种dna样本;相应地,在步骤(3)之后,将来源于多个dna样本的上述步骤(3)的pcr扩增产物混合在一起并纯化,以用于步骤(4)中的杂交捕获。进一步地,在步骤(4)和步骤(5)之间,还包括:(4’)以步骤(4)所得的杂交捕获产物为模板,在dna聚合酶的作用下,使用接头序列的引物进行pcr扩增,以增加上述杂交捕获产物的量。进一步地,上述末端修复酶包括:klenow片段、taq酶和t4多聚核苷酸激酶;任选地,上述打断酶为dna片段化酶。进一步地,上述接头序列按照5’端至3’端的顺序如下所示:长链序列:agtcggaggccaagcggtcttaggaagacaannnnnnnnnncaactccttggctcaca(seqidno:1),其中nnnnnnnnnn表示上述条形码序列;短链序列:ttgtcttcctaaggaacgacatggctacgatccgactt(seqidno:2);相应地,上述接头序列的引物按照5’端至3’端的顺序如下所示:上游引物:gaacgacatggctacga(seqidno:13);下游引物:tgtgagccaaggagttg(seqidno:14)。进一步地,步骤(4)的杂交之前先使用阻断序列对步骤(3)的产物进行处理,以阻断非特异性结合;优选地,上述阻断序列至少包括:gaacgacatggctacga(seqidno:3);和agtcggaggccaagcggtcttaggaagacaaaccaaatgatcaactccttggctcaca(seqidno:4)。根据本发明的第二方面,本发明提供一种brca1/2基因检测文库的构建试剂盒,包括:(1)打断酶、末端修复酶,用于在dna片段化及末端修复反应体系中,将均一化的dna样本片段化并末端修复;(2)接头序列,用于在接头连接体系中,在dna连接酶的作用下,与片段化并末端修复的产物进行连接;(3)接头序列的引物,用于以接头连接产物为模板,在dna聚合酶的作用下进行pcr扩增;(4)包含brca1/2基因的探针序列,用于与来源于多个dna样本的pcr扩增产物的混合物进行杂交以捕获含有上述brca1/2基因的dna片段;(5)连接酶,用于连接接头序列和上述片段化并末端修复的产物,以及用于在单链环化体系中,使杂交捕获产物的单链环化。进一步地,上述接头序列带有用于区分不同dna样本的条形码序列,每一上述条形码序列对应一种dna样本。进一步地,上述末端修复酶包括:klenow片段、taq酶和t4多聚核苷酸激酶;任选地,上述打断酶为dna片段化酶。进一步地,上述接头序列按照5’端至3’端的顺序如下所示:长链序列:agtcggaggccaagcggtcttaggaagacaannnnnnnnnncaactccttggctcaca(seqidno:1),其中nnnnnnnnnn表示上述条形码序列;短链序列:ttgtcttcctaaggaacgacatggctacgatccgactt(seqidno:2);相应地,上述接头序列的引物按照5’端至3’端的顺序如下所示:上游引物:gaacgacatggctacga(seqidno:13);下游引物:tgtgagccaaggagttg(seqidno:14)。进一步地,还包括:阻断序列,用于在上述杂交之前对来源于多个dna样本的pcr扩增产物的混合物进行处理,以阻断非特异性结合;优选地,上述阻断序列至少包括:gaacgacatggctacga(seqidno:3);和agtcggaggccaagcggtcttaggaagacaaaccaaatgatcaactccttggctcaca(seqidno:4)。本发明的有益效果体现在如下至少一个方面:(1)本发明的建库方法将酶法打断和末端修复在一管内一步完成,减少了磁珠纯化操作,杂交前建库步骤半天即可完成,因此建库步骤简便快速,有效降低成本,减少工作量;(2)采用酶法打断片段化dna替代传统物理打断方法,减少了仪器投入及维护成本,且节省了人力操作;(3)使用本发明方法构建的文库检测brca1/2基因突变,检测变异类型全面、准确、通量高、成本低;采用探针捕获技术可一次性富集brca1/2基因全部外显子及邻近内含子序列,结合二代测序技术可同时检测全面,包含多种变异类型如突变、缺失、插入及拷贝数扩增,不仅降低了人工及成本投入并且克服了聚合酶链式反应结合一代测序法难以检测五个核苷酸以上的插入或缺失及拷贝数变异检测准确度低的缺点。(4)此外,本发明方法的具体实施例中对多个建库步骤条件进行优化(例如pcr条件、磁珠纯化条件等),提高了对100-300bp小片段的富集,更加适合二代测序平台检测,目标片段富集效果更佳。附图说明图1为包含本发明方法的一个基因检测的详细实施例流程图;图2为本发明实施例中一例阴性对照样本(上图)和一例阳性样本(下图)的预建库完成后的agilent2100质控结果。具体实施方式下面通过具体实施方式结合附图对本发明作进一步详细说明。本发明旨在提供一种基于酶打断单管快速建库的方法,及该建库方法在brca1/2基因突变检测中的应用,使用本发明的建库方法,能够一次捕获brca1/2基因的全部外显子及邻近内含子序列,一次性检测突变、插入、缺失及拷贝数变异。本发明的建库方法,采用酶打断法进行dna片段化,而且将dna片段化和末端修复在一个反应管中一步完成,缩减了筛选纯化次数,减少了片段损失。此外,对于捕获前的pcr扩增程序采用分步法进行,更优先富集目的片段,后续的纯化也采用了同芯片杂交样本混库(pooling)纯化的方法,大大节省了人力成本。图1示出了包含本发明方法的一个基因检测的详细实施例流程图,该流程包括四个阶段:样本制备;预文库构建;捕获文库构建;测序及数据分析。本发明实施例的文库构建方法主要涉及预文库构建、捕获文库构建两个阶段。需要说明的是,图1所示的流程显示了整个详细的基因检测流程,而本发明的基因检测文库构建方法仅是其中的部分步骤,因此本发明的方法并不受到图1所示的流程的限制。具体而言,本发明的基因检测文库构建方法包括:(1)在dna片段化及末端修复反应体系中(图1中1-tube单管反应体系),在打断酶、末端修复酶的作用下,将均一化的dna样本片段化并末端修复。末端修复酶可以包括klenow片段、taq酶和t4多聚核苷酸激酶。末端修复后的片段化dna两端能够与接头序列连接,例如带有突出a碱基的片段化dna与互补的接头序列连接。(2)在接头连接体系中,在dna连接酶的作用下,将步骤(1)所得的产物与接头序列连接。需要说明的是,在仅有单个样本的情况下,接头序列没有什么特别要求;然而,在多个样本进行混合(pooling)测序的情况下,接头序列需要带有用于区分不同dna样本的条形码序列(barcode),每一条形码序列对应一种dna样本。条形码序列可以是接头序列上一段连续的特定碱基序列,例如随机序列,其长度可以是6-12个碱基,例如10个碱基。(3)以步骤(2)所得的接头连接产物为模板,在dna聚合酶的作用下,使用接头序列的引物进行pcr扩增(图1中pre-pcr扩增)。经过pcr扩增以后的文库称为预文库,pcr扩增能够增加接头连接产物的数量。(4)可选地,将来源于多个dna样本的步骤(3)的pcr扩增产物混合在一起并纯化。需要说明的是,在仅有单个样本的情况下,不需要这一步骤,因此这一步骤是可选的步骤。(5)使用包含brca1/2基因的探针序列与步骤(4)的产物进行杂交以捕获含有上述brca1/2基因的dna片段,并洗脱以获取杂交捕获产物。包含brca1/2基因的探针序列按照目前的序列捕获原理,根据brca1/2基因的序列情况设计即可,这些探针序列可以承载在固体载体,例如芯片或磁珠上,用于杂交捕获目标brca1/2基因片段,并实现分离。(6)在单链环化体系中,在连接酶的作用下,使杂交捕获产物的单链实现环化,得到brca1/2基因检测文库。热变性的单链通过两端连接形成环化dna,即可用于后续测序分析。作为进一步改进的方案,在步骤(5)和步骤(6)之间,还包括:(5’)以步骤(5)所得的杂交捕获产物为模板,在dna聚合酶的作用下,使用接头序列的引物进行pcr扩增(图1中post-pcr扩增),以增加杂交捕获产物的量。由于纯化过程中会造成片段的丢失,且环化受到环化效率的限制,得到的环化dna分子可能不足以进行测序,在此情况下,进行一步pcr扩增,能够提高dna分子的数量,以便有效地进行测序。本发明实施例的建库方法,采用酶打断法片段化dna替代传统物理打断方法,不仅减少了仪器投入及维护成本,而且简化了实验操作,节约了人力与时间成本。dna片段化过程与末端修复及加a在一个反应管中一步完成,减少了一般流程中片段化后需要片段筛选的步骤,更减少了片段损失。酶打断及单管建库的方法步骤简单,易于操作,降低成本节约时间的同时也降低了多次操作易引起的污染和损失。此外,该发明涉及的建库方法中捕获前的pcr扩增程序采用分步法进行,更优先富集目的小片段。本发明实施例的建库方法,采用探针捕获方式能够富集brca1与brca2基因的全部外显子及邻近内含子序列,结合二代测序技术可一次性检出brca1/2基因的突变、缺失、插入及拷贝数变异,不仅降低了人工及成本投入并且克服了聚合酶链式反应结合一代测序法,难以检测五个核苷酸以上的插入或缺失及拷贝数变异检测准确度低的缺点。此外,备选采用无需pcr(pcr-free)的方法,即无需post-pcr扩增的方法,对捕获后的文库直接进行环化,减少了实验步骤的同时大大减少pcr扩增可能引起的错误引入。以下通过实施例详细说明本发明的技术方案和技术效果,应当理解,实施例仅是示例性的,不能理解为对本发明保护范围的限制。实施例1本实施例采用8个样本进行文库构建,采用包含brca1/2基因的nimblegen芯片进行杂交捕获并结合高通量测序仪bgiseq-500平台检测brca1/2基因snv、indel及cnv三种类型变异。具体操作流程如下:1.样本制备1.1样本提取及定量:利用qiagen公司dna提取试剂盒从血液样本及细胞系中提取gdna,取1μl进行qubiths定量。超过100ng/μl的需要将样本稀释后重新定量,不足15ng/μl的用ampurexp磁珠纯化回收的方法富集。1.2样本均一化:每样本分别取200ng用nf水(nuclease-freewater)稀释到15μl,转移到新的pcr管中,用于建库。2.dna片段化和末端修复2.1试剂准备:提前五分钟在冰上配制如下反应液,所需试剂如表1所示:表12.2pcr仪程序设置,如表2所示:表2步骤温度时间步骤137℃20分钟步骤275℃15分钟步骤34℃锁定设好程序后,点击开启,再立即点击暂停。确保在开始dna片段化时温度到达37℃。2.3将12μl步骤2.1中反应液加入每个均一化后的样本中,混匀,离心后马上置于冰上。2.4将样品置入pcr仪中反应,重新启动反应程序。3.ad153接头连接3.1提前取出ad153接头在冰上融化并提前五分钟配制如下连接反应液,成分如表3所示:表3试剂名称使用量/反应atp(100mm)(life)1μlpnkbuffer(enzymatics)4.5μl连接酶(enzymatics)1.4μl50%peg8000(emeraldbiosystems)17μl补水至总量46μl3.2pcr仪设置,需预先设置程序,如表4所示:表4步骤温度时间步骤123℃60分钟步骤212℃锁定3.3接头连接操作:3.3.1在上一步反应液按需要将带有不同条形码序列(barcode)的ad153接头分装到相应样本中,每个样本中分别加入ad153接头(10μm)4μl,移液器吹打数次。接头序列见表5:表53.3.2再将46μl连接反应液,加入到各个样本中,混匀,离心。3.3.3将样品置于pcr仪上反应,点击开始。3.4反应产物纯化3.4.1提前半小时取出xpbeads于室温平衡,混匀,备用。3.4.2连接反应完成后,将反应液转移至1.5ml离心管,每管中加入40μl的ampurexp磁珠;混匀,室温静置5分钟。3.4.3瞬时离心,将离心管置于磁力架上,5-10分钟后,待磁珠吸附完全,小心吸去上清,避免吸到磁珠。3.4.4离心管保持于磁力架上,在每管中加入400μl75%乙醇,移液器轻柔吹打(避免触碰磁珠)或者上下颠倒磁力架来清洗磁珠数次,弃掉上清(避免吸取到磁珠)。3.4.5重复一次步骤3.4.4。3.4.6离心管取出瞬时离心再置于磁力架上,用小量程移液器吸去残留乙醇,室温晾干。3.4.7用21μl无酶水重悬磁珠,室温结合5分钟后置于磁力架上,小心吸出上清转移至新的离心管中。4.pre-pcr:4.1提前5分钟取出ad153pcr引物和2×kapahifipolymerasemastermix,在冰上配制反应液如表6所示:ad153pcr引物序列具体如下:ad153-f:gaacgacatggctacga(seqidno:13);ad153-r:tgtgagccaaggagttg(seqidno:14)。表64.2pcr仪需预先设置程序,如表7所示:表74.3将30μl混匀后的pcr反应液加入步骤3.4.7中的连接产物中,混匀,瞬时离心。4.4将样品置于pcr仪上反应,点击开始。4.5pcr产物纯化:pcr反应结束后用1.2倍xp磁珠纯化pcr产物,21μl回收(具体纯化操作参照步骤3.4)。5.预文库质量检测5.1agilent2100检测:用于判断预文库dna的片段大小,通过本实施例建立的预文库dna的片段主要分布在100-250bp。需要注意的是,dna片段大小若小于100bp表明可能在片段化反应中gdna的量太少;若大于300bp表明可能在片段化反应中gdna的量太多,酶打断不充分。图2示出了本实施例中一例阴性对照样本和一例阳性样本的预建库完成后的agilent2100质控结果,结果显示文库中目标片段集中,大小符合预期要求。5.2qubit检测:取1μl进行qubiths定量检测,判断预文库的浓度,用于混库纯化。杂交预文库dna的总量应≥200ng。6.杂交捕获6.1杂交前混库及纯化:6.1.1每8个样本为一组进行混合,共1~3μg补水至160μl。6.1.2可选步骤:加入0.6倍(范围可浮动)xp磁珠,静止5min后,瞬时离心。将离心管置于磁力架上,待上清澄清,吸取上清于新的离心管中。再次加入0.6倍(范围可浮动)xp磁珠进行纯化,21μl进行回收。6.1.3取1μl进行qubiths定量。6.2杂交反应(使用nimblegen芯片)实验前准备:6.2.1打开95℃温浴仪保持恒温。6.2.2从-15℃到-25℃取出4.5μl探针,冰上解冻。6.2.3将准备混合的pre-pcr文库要尽量在冰上解冻。6.2.4将蒸干仪调至60℃,保持恒温。6.2.5将pcr仪设置为47℃保持,需热盖温度为57℃。6.2.6将水浴锅调至47℃保持恒温。操作流程:6.2.7每组混合样本取1μg于新的1.5mlep管中,依次加入样本处理体系c中的cot-1dna(nimblegen)及阻断序列(block),见表8。6.2.8放入60℃以上的蒸干仪中加热蒸干。6.2.9在蒸干的管子中加入杂交缓冲液,见表8。6.2.10将ep管充分震荡混匀,高速离心10s,95℃10min使dna变性。6.2.11将变性dna和4.5μl探针(液相探针,是针对性设计的包含brca1/2的nimblegen探针)一同转入0.2ml管中。6.2.12震荡3s,高速离心10s。6.2.13将样本放于47℃pcr仪中保持24小时,热盖温度为57℃(比杂交温度高10℃)。表8阻断序列如下表9:表97.洗脱使用nimblegen芯片配套的杂交洗脱试剂盒(seqcapezhybridizationandwashkit),按照操作说明,进行洗脱,具体包括如下步骤:实验准备7.1试剂准备:7.1.1取40μl10xstringentwashbuffer(严格洗脱缓冲液)加入360μlpcr级水至1x。7.1.2取30μl10xwashbufferⅰ加入270μlpcr级水至1x。7.1.3取20μl10xwashbufferⅱ加入180μlpcr级水至1x。7.1.4取20μl10xwashbufferⅲ加入180μlpcr级水至1x。7.1.5取200μl2.5xbeadswashbuffer加入300μlpcr级水至1x。7.1.6将1x的stringentwashbuffer与washbufferi放入47℃水浴锅中保持温度。试剂分装见下表10:表10试剂名称单个样本用量(μl)1xwashbuffer#1(47℃)1201xstringentbuffer(47℃)4401xwashbuffer#1(rt)2201xwashbuffer#2(rt)2201xwashbuffer#3(rt)2207.2磁珠准备:7.2.1磁珠使用之前室温平衡30min,涡旋振荡15s。7.2.2向新1.5mlep管中加入100μl磁珠,混匀后放在磁力架上,约5min后液体澄清吸弃上清,加入200μl1xbeadwashbuffer,涡旋振荡10s,重新放到磁力架上,待液体澄清后将液体吸弃。重复洗涤一次。7.2.3用100μl1xbeadwashbuffer进行重悬。将重悬好的磁珠转入0.2ml管中,放置磁力架上,吸弃上清,余下的磁珠准备结合捕获到的dna。7.3杂交后洗脱:7.3.1在杂交体系中加入准备好的磁珠,混匀。7.3.2混合物47℃45min,(热盖57℃),每15min涡旋振荡3s,总共振荡3次。7.3.3转管:加入100μl47℃1xwashbufferi,涡旋振荡10s。将混合物从0.2ml管转到1.5ml管,放在磁力架上,液体澄清后去上清。7.3.447℃洗脱:加入200μl47℃1xstringentwashbuffer,上下颠倒10次混匀后47℃反应5min。重复洗涤一次,总共洗涤两次。将管放置磁力架上,液体澄清后吸弃液体。7.3.4常温洗脱:加200μl室温1xwashbufferi涡旋振荡2min,将管子放在磁力架上,吸弃上清。加200μl室温1xwashbufferii,涡旋振荡1min。将管子放在磁力架上,吸弃上清。加200μl室温1xwashbufferiii,涡旋振荡30s。将管子放在磁力架上,吸弃上清。7.3.5回收:将管子取下,每管加入95μlpcr级水回溶。回溶体积根据需要可以进行调整。8.post-pcr8.1试剂准备:提前10分钟取出ad153pcr引物和2×kapahifipolymerasemastermix,配制反应液,见表11:表118.2pcr仪需预先设置程序,如下表12:表128.3post-pcr反应:将post-pcr反应液加入步骤7.3.5中的重悬磁珠中,用移液器轻轻吹打混匀,不要产生气泡,不离心,将其置于设置好的pcr仪中,点击开始。8.4post-pcr产物纯化:反应完成后,将反应液转移至1.5ml离心管,瞬时离心,将离心管置于磁力架上,5-10分钟后,待磁珠吸附完全,将上清全部转移至新离心管中,每管中加入120μl的xp磁珠进行纯化。用41μl1×te(低edta,ph8.0)重悬磁珠,室温结合5分钟后置于磁力架上,小心吸出上清转移至新的离心管中。8.5取1μl进行qubiths定量,浓度不应少于4ng/μl。9.单链环化9.1配制环化反应液,如表13所示:表139.2pcr仪预先设置程序,如表14所示:表14步骤温度时间步骤195℃5分钟步骤24℃锁定9.3另一pcr仪需预先设置程序,如表15所示:表15步骤温度时间步骤137℃20-30分钟步骤212℃锁定9.4取160ngpost-pcr产物,用1×te(低edta,ph8.0)定容至48ul,混匀。置于步骤9.2设置好的pcr仪中,点击开始。9.5将9.1步骤中配制的11.8μl环化反应液加入到上述dna反应液中,混匀,瞬时离心。将反应管置于9.3步骤中设置好的在pcr仪中,点击开始。10.反应完成,文库出库,进行质控检测。11.上机进行测序和数据分析本实施例采用nimblegen芯片杂交的8例样本的具体检测结果如下表16:表16结果显示:7例阳性样本包括brca1/2无义突变、移码突变及brca1的cnv变异均正确检测,该结果与sanger检测结果或qpcr检测结果一致。说明本实施例的建库方法及试剂盒具有很好的检测性能。实施例2本实施例采用8个样本进行文库构建,采用包含brca1/2基因的bgi芯片进行杂交捕获并结合高通量测序仪bgiseq-500平台检测brca1/2基因snv、indel及cnv三种类型变异。具体操作流程如下:步骤1至5同实施例1。6.杂交捕获6.1杂交前混库及纯化:6.1.1每8个样本为一组进行混合,共1~3μg补水至160μl。6.1.2可选步骤:加入0.6倍(范围可浮动)xp磁珠,静止5min后,瞬时离心。将离心管置于磁力架上,待上清澄清,吸取上清于新的离心管中。再次加入0.6倍(范围可浮动)xp磁珠进行纯化,21μl进行回收。6.1.3取1μl进行qubiths定量。6.2杂交反应(使用bgi芯片)实验前准备:杂交及洗脱试剂中以下组分可购买自bgi杂交洗脱试剂(1rxn)hybrid&washreagent(1reaction)散装货号:85-05504-00,包括bgiblock#1、bgiblock#2、bgihyb#1(bgi)、bgihyb#2(bgi)、bgihyb#3(bgi)、bgihyb#4(bgi)、rnaseblock(bgi)、bindingbuffer(bgi)、washbuffer#1(bgi)、washbuffer#2(bgi)。6.2.1打开95℃温浴仪保持恒温。6.2.2取出探针,冰上解冻。6.2.3将准备混合的pre-pcr文库要尽量在冰上解冻。操作流程:6.2.4在pcr管中进行样本处理体系和杂交反应液制备,见表17。6.2.4.1按照表17配制的样本处理体系,分别加入到上一步反应纯化得到的每组20μl的混库(pooling)的样本中,用移液器混合均匀,瞬间离心,95℃下孵育5min使dna变性,65℃恒温保持。6.2.4.2按照表17配制杂交反应液1,配好后置于已达到65℃的热循环仪(热盖温度为105℃)上孵育5分钟以上,使其保持在65℃恒温。6.2.4.3按照表17在冰上配制杂交反应液2,放入已达到65℃的热循环仪(热盖温度为105℃)上孵育2分钟以上,使其保持在65℃恒温。表17注:①阻断序列a和阻断序列b同实施例1。阻断序列a是标签阻断序列(indexblock),用来封闭标签(index)序列;阻断序列b是接头阻断序列,主要是封闭与标签序列对应的序列(一般在文库单链的5'端)。它们的作用主要有两个:封闭非捕获区域,保证捕获的特异性;以及保持被捕获序列单链,即保证捕获的特异性和多样性。②panel设计原则:根据待检测靶基因(如brca1/2)的特异性核酸序列合成初始探针。初始探针为一段特异性dna序列,两侧为扩增片段。得到初始探针片段后,根据扩增引物扩增探针,随后将扩增后的双链dna转录成单链rna,并添加生物素标记。探针设计要求探针覆盖待检测基因组区域,探针长度为个碱基,探针序列根据靶基因序列的正义链进行设计;探针gc含量一般在20~80%范围内,探针两端不存在互补结构。③bgiblock#1与bgiblock#2是封闭一些同源性较高的非特异性序列,近似cot-1dna。cot-1dna通常作为dna竞争性阻断剂,在基因组杂交中阻断可能引起非特异性杂交的重复序列。6.2.5保持所有pcr管在65℃,打开pcr仪,迅速转移13μl步骤6.2.4.2配制的杂交反应液1到上述已加入6.2.4.1提到的混库样本的混合液中(每次只操作1组混库样本),吹打数次混匀(勿引入气泡),盖好管盖,依次加完所有样本,65℃孵育至少2分钟。6.2.6保持所有pcr管在65℃,将步骤6.2.5中加好的混合反应液,取39μl迅速转移至对应的杂交反应液2的管中,移液器吹打5-10次混匀,盖好管盖,此时每个pcr管中反应液体积为约46μl。依次加完所有样本后从热循环仪上移走空管,盖好热循环仪盖(热盖温度为105℃),计时,保持样本在65℃孵育杂交16-48小时(可以根据需要选择时间,推荐16或24h)。7洗脱7.1提前2h将水浴锅调至65℃,水温稳定后为每个杂交反应准备1.8mlbgiwashbuffer2置于水浴锅中预热;7.2用漩涡混合仪剧烈振荡重悬dynabeadsm-280streptavidinbeads(life公司)至混匀;7.3每一个杂交反应取50μldynabeadsm-280streptavidinbeads于一新的2.0ml离心管;7.4将200μlbgibindingbuffer加入磁珠中,用漩涡混合仪剧烈振荡5秒钟重悬磁珠;7.5将离心管置于磁力架上2min,待液体完全澄清;小心吸取并弃去上清;7.6重复步骤7.4到步骤7.5两次;加入200μlbgibindingbuffer重悬磁珠;7.7经16h孵育后,继续保持杂交混合液于pcr仪上,逐一使用移液器快速吸取样本(如果操作多个样本,则可以完成6-8个反应后关闭热盖5min,然后继续);7.8从pcr仪中直接将全部杂交混合液转移到准备好的磁珠中,上下颠倒离心管3到5次直到混匀;7.9将装有杂交混合液和磁珠的离心管对称的固定于垂直混匀仪或手工上下颠倒混匀,室温下孵育30min;7.10将样品从混匀装置取下,瞬时离心5s确保管盖上没有液体残留;7.11将2.0ml离心管转移放置在磁力架上,静置3-5min待液体完全澄清,小心吸取并弃去上清;7.12用500μlbgiwashbuffer1重悬磁珠,并于漩涡混合仪上振荡5秒混匀样品,室温下孵育样品15min;7.13将离心管瞬时离心3s,然后放置在磁力架上,静置3-5min待液体完全澄清,小心吸取并弃去上清;7.14用500μl预热至65℃的bgiwashbuffer2重悬磁珠,并于漩涡混合仪上振荡5秒以混匀样品,将其置于恒温混合仪中65℃孵育10min;7.15颠倒离心管以混匀样品,瞬时离心3s,然后将其放置在磁力架上,静置3-5min待液体完全澄清,小心吸取并弃去上清;7.16重复步骤7.14到步骤7.15两次;7.17用45μl无核酸酶的水重悬磁珠;也可以根据需要调整回溶体积。步骤8至11同实施例1。本实施例采用bgi芯片杂交的8例样本的具体检测结果如下表18:表18样本名称突变类型基因检出变异阳性标品g移码突变brca1c.[1201delg]阳性标品h移码突变brca1c.[797_798deltt]阳性标品i移码突变brca1c.[3751_3754deltgtc]阳性标品j移码突变brca2c.[6198_6199deltt]阳性标品k错义突变brca1c.[181t>g]阳性标品l错义突变brca1c.[4327c>g]临床样本a2snv/snp位点全部检出,但无阳性位点阴性对照品snv/snp位点全部检出,但无阳性位点结果显示:6例阳性标品包括brca1/2错义突变、移码突变均正确检测,该结果与sanger检测结果一致。说明本实施例的建库方法及试剂盒具有很好的检测性能。以上内容是结合具体的实施方式对本发明所作的进一步详细说明,不能认定本发明的具体实施只局限于这些说明。对于本发明所属
技术领域:
的普通技术人员来说,在不脱离本发明构思的前提下,还可以做出若干简单推演或替换,都应当视为属于本发明的保护范围。sequencelisting<110>深圳华大基因股份有限公司,天津华大医学检验所有限公司,广州华大基因医学检验所有限公司,华大生物科技(武汉)有限公司<120>一种brca1/2基因检测文库的构建方法和试剂盒<130>17i23988<160>14<170>patentinversion3.3<210>1<211>58<212>dna<213>人工序列<220><221>misc_feature<222>(32)..(41)<223>nisa,c,g,ort<400>1agtcggaggccaagcggtcttaggaagacaannnnnnnnnncaactccttggctcaca58<210>2<211>38<212>dna<213>人工序列<400>2ttgtcttcctaaggaacgacatggctacgatccgactt38<210>3<211>17<212>dna<213>人工序列<400>3gaacgacatggctacga17<210>4<211>58<212>dna<213>人工序列<400>4agtcggaggccaagcggtcttaggaagacaaaccaaatgatcaactccttggctcaca58<210>5<211>58<212>dna<213>人工序列<400>5agtcggaggccaagcggtcttaggaagacaatgtcataaatcaactccttggctcaca58<210>6<211>58<212>dna<213>人工序列<400>6agtcggaggccaagcggtcttaggaagacaattaattaaggcaactccttggctcaca58<210>7<211>58<212>dna<213>人工序列<400>7agtcggaggccaagcggtcttaggaagacaagactcactgacaactccttggctcaca58<210>8<211>58<212>dna<213>人工序列<400>8agtcggaggccaagcggtcttaggaagacaaataaggcagtcaactccttggctcaca58<210>9<211>58<212>dna<213>人工序列<400>9agtcggaggccaagcggtcttaggaagacaattgatagattcaactccttggctcaca58<210>10<211>58<212>dna<213>人工序列<400>10agtcggaggccaagcggtcttaggaagacaaccttcctggtcaactccttggctcaca58<210>11<211>58<212>dna<213>人工序列<400>11agtcggaggccaagcggtcttaggaagacaaaatatctctccaactccttggctcaca58<210>12<211>58<212>dna<213>人工序列<400>12agtcggaggccaagcggtcttaggaagacaacatgtttccccaactccttggctcaca58<210>13<211>17<212>dna<213>人工序列<400>13gaacgacatggctacga17<210>14<211>17<212>dna<213>人工序列<400>14tgtgagccaaggagttg17当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1