一种鹅细小病毒感染性克隆的拯救方法与流程
本发明属于生物技术领域,涉及一种GPV感染性克隆质粒转染鹅胚拯救病毒的简便高效方法。
背景技术:
我国的养鹅业居于世界首位,也是鹅肉消费大国。鹅细小病毒是1月龄内雏鹅的一种重要疫病,死亡率可高达90%,该病自上世纪50、60年代在国内流行。目前虽已研制了弱毒疫苗供种鹅或雏鹅使用,但该病并未从田间消失,仍然持续性对养鹅业造成经济损失。
反向遗传技术是开展病毒研究的一种有用平台,在阐明病毒致病机制和疫苗研制中能够发挥重要作用。同样,构建鹅细小病毒感染性分子克隆,以此为基础可以对GPV的特定基因展开更为有效的研究,也是GPV疫苗研制的一个有用平台。
病毒拯救是反向遗传操作上的重要一环,大多是将构建的质粒或RNA转录产物转染细胞的途径来实现拯救目的。转染原代的鹅胚成纤维细胞拯救感染性GPV也有报道,对于细小病毒而言,细胞处于分裂期时才最适于病毒复制,因此转染时细胞所处状态对转染拯救结果影响很大。总体来看,在细胞上进行转染不仅费事,拯救效率也不高。因此,若能建立一种简便、高效的拯救方法,将极大的方便并推动GPV相关基础研究的深入开展。
技术实现要素:
本发明的目的是提供一种简便高效的鹅细小病毒(Goose parvovirus,GPV)感染性克隆质粒的拯救方法。
本发明所述的鹅细小病毒感染性克隆质粒的拯救方法,是将GPV的完整基因组克隆入载体质粒pBSKNB,获得重组质粒,将重组质粒与转染试剂混合后,通过绒毛尿囊膜途径接种非免疫鹅胚,重组质粒在鹅胚中得到拯救;所述pBSKNB载体,是将商品化的pBluescript II(SK)质粒上原有的EcoRV和HindIII酶切位点分别用NcoI和BclI位点替换而得到。
具体地:将重组质粒与转染试剂Lipofectamine 2000按照一定比例1:2.5(μg/μl)比例混合后,通过绒毛尿囊膜途径接种11日龄非免疫鹅胚。鹅胚在接种后120h~192h之间死亡,胚体具有典型的GPV感染特征,表现为胚体出血、绒毛尿囊膜水肿的典型特征。拯救出的病毒具有与亲本病毒相近的生物学特性。
进一步地,为了排除转染拯救过程中亲本GPV毒株污染的可能,可通过overlap PCR方法,在重组质粒的克隆基因组内部引入两个同义碱基突变作为遗传标记,得到的带有遗传标记的重组质粒再施以转染及拯救过程。更具体地,可以是在重组质粒的VP1基因中引入两个突变碱基作为遗传标记。
本发明中,所述的重组质粒是插入GPV LH株完整基因组pLH质粒,或者是插入GPV LH株完整基因组并引入遗传标记的pLHΔ质粒,但是,本发明的拯救方法适用于但不仅限于插入GPV LH株完整基因组的pLH质粒或pLHΔ质粒。对其它GPV强毒分离株或者鹅胚化弱毒株,以本发明内容公开的相同构建方法克隆其全基因组,获得重组质粒,重组质粒的拯救方法同pLH质粒或pLHΔ质粒。
本发明所述的质粒pLH,它的构建方法在于:超速离心浓缩GPV LH株病毒,提取病毒的基因组单链DNA,体外退火后生成双股DNA。选用BclI和NcoI酶切基因组DNA生成亚基因组片段,分别克隆入自行改造的pBSKNB载体的相应酶切位点,再通过酶切-连接的常规分子操作,拼接获得含有完整基因组的质粒pLH。
进一步地,通过overlap PCR方法,在pLH质粒的VP1基因中引入两个同义突变碱基作为遗传标记,获得质粒pLHΔ。
本发明中所述相近的生物学特性是指在鹅胚半数致死量(ELD50)和感染试验中的雏鹅死亡率这两个关键指标上,拯救病毒具有与亲本病毒相近的数值。
本发明公开了LH株的完整基因组序列。LH株基因组由5047个碱基组成(SEQ IB NO.5),LH株的ITR由414个碱基组成,其中375个碱基形成回文结构,其余39个碱基构成D区序列。基因组左侧的ITR的D区序列与右侧ITR的D′序列反向互补,这一特性将使得基因组分子能够形成一个更大范围的回文。
作为一个具体的操作,本发明公开了一种GPV全基因组克隆的构建方法,该方法对其它GPV毒株同样适用,它包括如下步骤:
(1)GPV核酸的提取
GPVLH株在鹅胚中增殖,收集的尿囊液分别经差速和超速离心浓缩到原有体积的1/50。采用SDS-蛋白酶K消化法提取病毒核酸。经95℃变性10min,缓慢冷却至65℃退火,使单股DNA形成双股DNA,经0.8%琼脂糖凝胶电泳,可观察到约5.1kb基因组DNA。
(2)全基因组克隆的构建
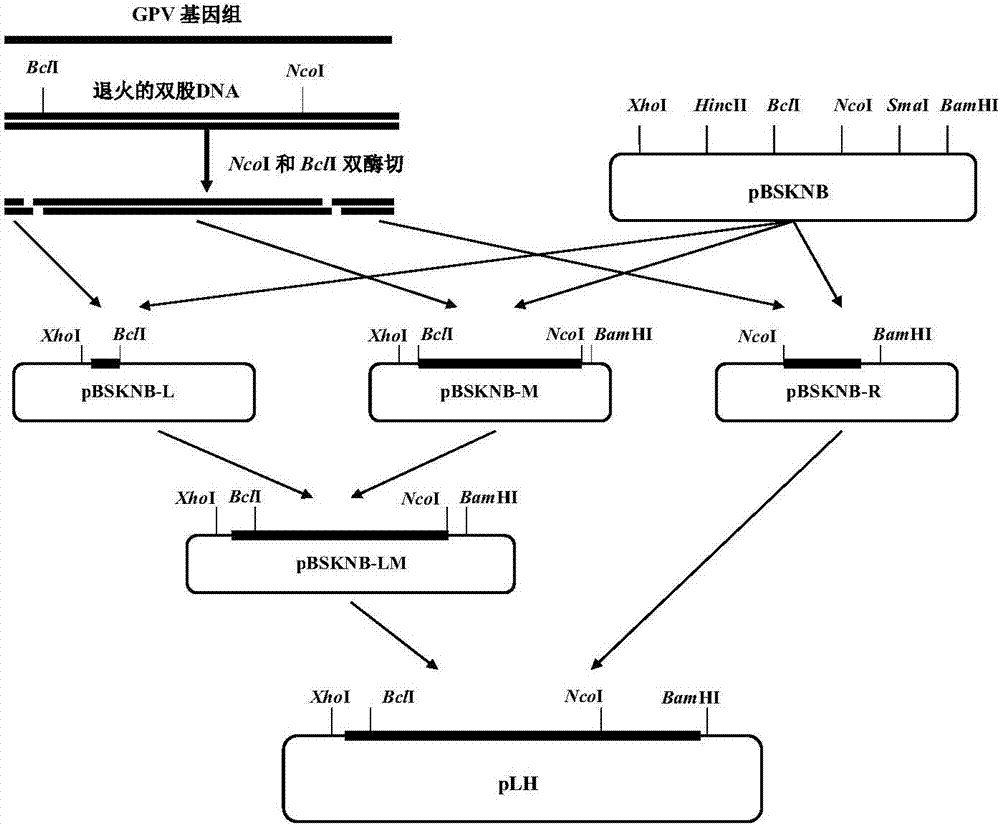
将提取的GPV LH株基因组双股DNA用NcoI和BclI限制性内切酶进行双酶切,产生的左中右三个片段大小分别为0.6kb、3.2kb和1.3kb,各片段切胶回收。将0.6kb的左片段克隆入载体质粒pBSKNB的HincII-BclI之间,产生pBSKNB-L质粒。将3.2kb的中间片段插入质粒pBSKNB的BclI-NcoI之间,产生pBSKNB-M质粒。将1.3kb的右片段插入pBSKNB的NcoI-SmaI位点之间,获得pBSKNB-R质粒。
在成功克隆亚基因组片段并测序的基础上,通过常规的酶切-连接操作,将各亚基因组片段正确的拼接,从而获得包含LH株完整基因组的克隆质粒pLH。
本发明还公开了pLH质粒中遗传标记引入的方法,以便于将拯救病毒与亲本病毒LH株区分开(见附图5)。
在pLH质粒的VP1基因中引入两个突变碱基作为遗传标记,不改变氨基酸组成,获得质粒pLHΔ。利用LH株基因组内部在拟突变位点两侧分别存在单一的SexAI和NcoI位点的条件,设计了一组overlap PCR引物,引物代号和序列分别为:
overlap-1:GCAGGAACAATTACCAGGTACG(SexAI)(SEQ IB NO.1)
overlap-2:GTGGTCGGTAGTTCCCTGT(SEQ IB NO.2)
overlap-3:ACAGGGAACTACCGACCAC(SEQ IB NO.3)
overlap-4:CGGCCCATGGTGCCATAAGC(NcoI)(SEQ IB NO.4)
方框内部为突变碱基,两个突变碱基分别为G→A突变和T→C突变,突变碱基用方框表示。Overlap-1引物和Overlap-4引物内部分别含有SexAI和NcoI位点。
通过overlap PCR,结合酶切连接操作,能够将2个突变碱基引入pLH质粒,获得质粒pLHΔ。
本发明还公开了pLHΔ质粒通过绒毛尿囊膜转染鹅胚拯救病毒的具体方法:
采用Lipofectamine 2000作为转染试剂。质粒和转染试剂的比例按照1:2.5(μg:μl)进行。吸取16μg pLHΔ质粒溶液至1个灭菌的1.5ml离心管中,用opti-DMEM培养液(Invitrogen,美国)稀释至1ml;另一个离心管内加入40μl Lipofectamine 2000转染试剂,加入960μl Opti-DMEM培养液混匀。将稀释后的质粒和转染试剂静置5min后,轻柔混匀,室温静置20min后即可进行转染。每只鹅胚通过绒毛尿囊膜接种0.25ml,其中包含2.0μg质粒。鹅胚在接种后第5天开始出现死亡,至转染后第8天,死亡率达到75%。拯救病毒在鹅胚上能够成功传代。
本发明对拯救病毒的生物性特性进行了测定。结果表明:拯救病毒的鹅胚半数致死量(ELD50)达到每毫升5×104.77,与亲本LH株极为相似,后者的ELD50为每毫升5×104.36。1日龄雏鹅分别接种了携带遗传标记的拯救病毒和亲本病毒,在15天的观察期内,接种亲本病毒组雏鹅在第3天开始出现死亡,而拯救病毒组在攻毒后第4天出现死亡。到攻毒后的第9天,两组雏鹅死亡率都达到了93.8%。而接种生理盐水的对照组雏鹅均生长健康无一死亡。拯救病毒攻毒组死亡雏鹅同样展现出“小鹅瘟”特征性病变,包括典型的肠栓形成、肠粘膜脱落等病理变化(见附图7)。实验表明拯救病毒具有与亲本病毒相近的生物学特性。该拯救方法操作简便高效,避免了转染细胞带来的繁琐和低效问题,在GPV反向遗传学研究和疫苗创制上具有潜在应用价值。
附图说明
图1.GPV LH株的基因组DNA电泳分析.M:λDNA/HindIII分子量markers.1:提取的病毒核酸。
图2.GPV LH株全基因组的构建策略。
图3是pLH质粒图谱。
图4.pLH质粒的酶切鉴定.M:1kb ladder DNA分子量标准.1.NcoI酶切;2.XhoI和BamHI双酶切;3.SphI酶切。
图5.pLHΔ质粒转染拯救病毒与亲本强毒株的区分。亲本强毒株的扩增靶片段用HindIII酶切,生成0.9kb和0.7kb两条片段;而拯救病毒由于引入的碱基突变,同样的1.6kb靶片段不能为HindIII切开。
图6.拯救病毒和亲本病毒株的攻毒试验。亲本病毒在攻毒后第3天出现死亡病例,拯救病毒在攻毒后第4天出现死亡病例,在攻毒后第9天,两组存活率都下降到6.2%。
图7.pLHΔ拯救病毒攻毒试验中死亡雏鹅的肠道病理变化(A)以及从脏器提取病毒DNA为模板的PCR扩增片段的序列测定结果(B)。
具体实施方式
以下方法针对我们分离的GPV LH株(Wang J,et al.,Arch Virol,2015,160(3):711-718)展开,但所叙述方法对其它GPV毒株一样适用。
1.GPV全基因组克隆质粒pLH的构建
1.1.病毒扩增
将保存的GPV LH株第2代鹅胚尿囊液,用灭菌生理盐水按1:10比例稀释,加入青、链霉素至终浓度各2000μg或IU/ml,置37℃培养箱中孵育30min后接种鹅胚。将经稀释的病毒液经尿囊腔途径接种12日龄鹅胚,每只0.2ml。石蜡封口后放入37℃孵化箱中,每隔8h照蛋1次,剔除48小时前死亡的鹅胚。收集48小时后死亡鹅胚,置于4℃冰箱放置4~6h后无菌收取尿囊液,储存在-40℃冰箱中代用。
1.2.病毒纯化
将收集到的约400ml含病毒尿囊液按11,000g离心20min,取上清后加入1/3体积的氯仿,剧烈震荡,以同样的离心力转速再次离心20min,收集上层水相再经150,000g离心力超速离心3h(SW32Ti转子,Beckman)。弃去上清,病毒沉淀用5ml TE缓冲液(50mM Tris,20mM EDTA,pH 8.0)溶解,-70℃保存待用。
1.3 病毒DNA提取
取500μl浓缩病毒液,分别加入SDS和蛋白酶K至终浓度为1%和400μg/ml。45℃水浴2小时。用苯酚-氯仿-异戊醇(25:24:1)和氯仿-异戊醇(24:1)各抽提1次。上清液加入2.5倍体积的无水乙醇和十分之一体积的醋酸钠(pH 5.2),-70℃沉淀过夜。次日15,000rpm/min,离心20min,去上清,核酸沉淀用70%酒精洗涤,再次15,000rpm/min,离心10min,去上清,沉淀凉干后,用30μL STE缓冲液(10mM Tris,1mM EDTA,100mM NaCL,pH 8.0)溶解。将提取的核酸置于95℃水浴10min,使单股DNA发生热变性,然后缓慢冷却到65℃,此时单股DNA可退火形成双链。取10μL经0.8%琼脂糖凝胶电泳分析DNA,见附图1。
1.4 基因组克隆
为了便于克隆,我们对pBluescriptII(SK)质粒(Agilent公司,美国)的酶切位点进行了改造,将质粒上原有的EcoRV和HindIII酶切位点分别用NcoI和BclI位点替换,改造后的质粒命名为pBSKNB。
将提取的GPV LH株基因组双股DNA用NcoI和BclI限制性内切酶进行双酶切,产生的左中右三个片段大小分别为0.6kb、3.2kb和1.3kb,各片段切胶回收。将0.6kb的左片段克隆入载体质粒pBSKNB的HincII-BclI之间,产生pBSKNB-L质粒。将3.2kb的中间片段插入质粒pBSKNB的BclI-NcoI之间,产生pBSKNB-M质粒。将1.3kb的右片段插入pBSKNB的NcoI-SmaI位点之间,获得pBSKNB-R质粒,构建策略见附图2。
在完成对各个亚片段克隆并测序的基础上,对pBSKNB-L质粒用XhoI和BclI进行双酶切,将内部的0.6kb片段切胶回收,pBSKNB-M质粒同样用XhoI和BclI双酶切线性化后,与0.6kb片段进行连接产生重组质粒pBSKNB-LM。然后进一步将pBSKNB-R质粒用NcoI和BamHI进行双酶切后生成的1.3kb片段插入到pBSKNB-LM质粒的NcoI-BamHI位点之间,最终产生包含LH株全基因组的克隆质粒pLH,质粒pLH图谱见附图3。pLH质粒的酶切鉴定见图4。构建的重组质粒均转化DH5α大肠杆菌感受态细胞进行扩增。
1.5 基因组序列分析
各亚基因组片段克隆送华大基因上海有限公司进行测序。由于ITR二级结构的干扰以及Bubble区序列存在反转的特点,故ITR序列不能直接测出。利用ITR loop区存在单一的SphI酶切位点的事实,故对包含ITR片段的亚克隆片段分别用SphI和XhoI或BamHI进行双酶切,产生的亚片段分别克隆入pUC18或pBSK质粒的相应位点,转化DH5α感受态细胞,阳性克隆测序后可获得一致的ITR序列。利用DNASTAR软件包中的SeqManII程序对所测片段序列进行拼接获得完整的基因组序列,利用MegAlign程序进行序列同源性分析。
2.pLH质粒遗传标记的引入
为了排除拯救病毒可能来自于试验过程中亲本病毒或GPV野毒株污染的可能,采用overlap PCR方法在pLH质粒内部处于VP1基因的HindIII位点处引入碱基突变,但不改变氨基酸的组成。
2.1 overlap PCR引物设计
利用LH株基因组内部在拟突变位点两侧分别存在单一的SexAI和NcoI位点的条件,设计了一组重叠PCR引物,引物由大连宝生物有限公司合成,引物代号和序列分别为:
overlap-1:GCAGGAACAATTACCAGGTACG(SexAI)
overlap-2:GTGGTCGGTAGTTCCCTGT
overlap-3:ACAGGGAACTACCGACCAC
overlap-4:CGGCCCATGGTGCCATAAGC(NcoI)
方框内部为突变碱基,两个突变碱基分别为G→A突变和T→C突变,突变碱基用方框表示。Overlap-1引物和Overlap-4引物内部分别含有SexAI和NcoI位点。
2.2 overlap PCR扩增
采用Pfu高保真DNA聚合酶(Takara,大连),以overlap-1引物和overlap-2引物扩增0.7kb的A片段,overlap-3引物和overlap-4引物扩增0.9kb的B片段。A、B扩增片段经1%琼脂糖凝胶电泳分离后,分别采用凝胶回收试剂盒(天根生物科技有限公司)切胶回收目的片段。
回收的A、B产物各取1μl混合后作为第二步PCR扩增反应的模板。以overlap-1和overlap-4为上下游引物。反应体系为:10×buffer 5μl,上下游引物各1μl,模板1μl,dNTP 4μl,pfu高保真DNA聚合酶0.5μl,超纯水补至50μl体系。反应程序设定为:95℃,2min变性;然后执行94℃,30s;53℃,30s,72℃,1min,40s,共25个循环;72℃延伸5min结束。预期扩增片段为1.6kb。
反应结束后,将50μl PCR反应产物经0.8%琼脂糖凝胶电泳分离后,切胶回收目的片段。目的片段用SexAI和NcoI双酶切,酒精沉淀酶切片段后,用20μl超纯水溶解。
2.3 突变位点导入pLH质粒
考虑到SexAI位点对甲基化质粒不能切开,故将pLH质粒转入HST04宿主菌(Takara,日本)以消除质粒甲基化。然后用SexAI和NcoI双酶切,电泳分离线性化片段,然后切胶回收6.4kb片段。将回收的1.6kb突变片段与6.4kb片段连接,连接产物转入DH5α感受态细胞,涂布LB平板。采用HindIII酶切消化质粒的方法来鉴定阳性的突变插入克隆,并进一步选用自带引物送商业化公司进行测序,确保在突变位点正确引入的同时,其它位点未发生改变。阳性克隆命名为pLHΔ。
3.pLHΔ质粒的转染拯救
3.1.pLHΔ质粒的纯化
挑取pLHΔ克隆单个菌落,接种于含有氨苄青霉素(100μg/ml)的LB培养基中,37℃震荡培养16~24h。采用Qiagen公司的Miniprep试剂盒纯化质粒,具体操作按照其说明书进行。采用Nanodrop 2000核酸测定仪,检测质粒纯度,确保OD260/280在1.8-2.0之间。
3.2.质粒-转染试剂混合物的配制
转染混合物的配制参照Lipofectamine 2000转染试剂说明书进行。质粒和转染试剂的比例按照1:2.5(μg:μL)进行。吸取16μg质粒溶液至1个灭菌的指形管中,用opti-DMEM培养液(Invitrogen)稀释至1ml;另一个离心管内加入40μl Lipofectamine 2000转染试剂,加入960μl opti-DMEM培养液混匀。将稀释后的质粒和转染试剂静置5min后,轻柔混匀,室温静置20min后即可进行转染。
3.3.转染鹅胚
上述配制好的转染试剂,经绒毛尿囊膜途径接种8个11日龄鹅胚,每只鹅胚接种0.25ml,其中包含2.0μg质粒。石蜡封口后放入孵化箱内孵育,每隔8h照胚一次,弃去24小时内死亡鹅胚。收集72h死亡鹅胚的尿囊液,检查胚体病变。结果表明,pLHΔ质粒通过绒毛尿囊膜途径转染11日龄鹅胚,鹅胚在接种后第5天开始死亡,至转染后第8天,死亡率达到75%。实验结果表明,绒毛尿囊膜途径转染质粒适合感染性GPV的拯救,死亡鹅胚胚体呈现典型的GPV感染的特征性变化,表现为胚体出血和绒毛尿囊膜水肿增厚。
3.4 拯救病毒的传代
将拯救出的第1代病毒尿囊液用氯仿抽提1次,用DNA酶I(Promega,美国)于37℃作用30min,以降解残存的质粒DNA,再用PBS缓冲液做1:5稀释后接种11日龄鹅胚。
3.5拯救病毒与亲本病毒的区分
通过PCR方法,可以扩增出覆盖遗传标记位点的约1.6kb片段,该片段不能为HindIII所切开,而以亲本病毒提取的DNA为模板扩增出的1.6kb片段,用HindIII酶切,产生0.9-kb和0.7-kb的两条片段(图5)。试验结果确切的表明拯救病毒来自于质粒转染拯救所产生,而不可能是亲本病毒或野毒污染的结果。对拯救病毒的序列分析也表明,除了作为遗传标记引入的2个突变碱基,其它部分序列与亲本序列完全一致。
4.拯救病毒的生物学特性
4.1 病毒滴度(ELD50)的测定
将新收集的含拯救病毒或亲本病毒的尿囊液稀释成10-3、10-4、10-5和10-6四个稀释度,通过尿囊腔接种途径接种12日龄鹅胚。每只鹅胚接种0.2ml,每个稀释度接种5个鹅胚。37℃孵化箱中继续孵育,弃去24h内死亡鹅胚,连续观察10天,统计每组鹅胚死亡个数,用Reed-Muench方法计算病毒滴度。试验结果表明:拯救病毒的ELD50达到每毫升5×104.77,与亲本LH株极为相似,后者的ELD50为每毫升5×104.36。
4.2 病毒中和实验
200ELD50的拯救病毒和亲本病毒LH株与等体积的经10倍稀释的GPV阳性血清混合,37℃温箱孵育30min,通过尿囊腔途径各接种5个12日龄鹅胚。对照组则以病毒液与GPV阴性血清混合。抗GPV阳性血清是以GPV疫苗株SYG61v免疫鹅群方法制备获得。中和试验结果表明,用GPV抗血清作用后的拯救病毒和亲本病毒接种的5个鹅胚无一死亡,而用生理盐水处理的对照组中鹅胚全部在84-120小时之间死亡,这表明拯救病毒拥有与亲本病毒相一致的抗原性。
4.3 雏鹅攻毒实验
将48只1日龄易感雏鹅随机分成3组。第1组的16只雏鹅颈部皮下注射0.3ml含3×104ELD50的拯救病毒;第2组的16只雏鹅颈部皮下注射等量的LH株亲本病毒;第3组16只雏鹅注射灭菌生理盐水作为阴性对照组。三组雏鹅分别在单独的房间隔离饲养15d,记录死亡个数和死亡时间,所有死亡雏鹅均剖解观察病理变化,计算在不同时间点的死亡率和存活率。
结果表明,两种1日龄雏鹅分别接种了携带遗传标记的拯救病毒和亲本病毒,在15天的观察期内,接种亲本病毒组雏鹅在第3天开始出现死亡,而拯救病毒组在攻毒后第4天出现死亡。到攻毒后的第9天,两组雏鹅死亡率都达到了93.8%(图6)。而接种生理盐水的对照组雏鹅均生长健康无一死亡。拯救病毒攻毒组死亡雏鹅同样展现出“小鹅瘟”特征性病变,包括典型的肠栓形成、肠粘膜脱落等病理变化(图7a)。
4.4 病料中核酸检测
取攻毒死亡雏鹅肝脏或肠道组织进行PCR检测。将肝脏或肠道组织剪碎,加适量PBS研磨,移入1.5ml离心管中,10000rpm离心10min。上清转移到新的离心管中,加入等量氯仿抽提1次,10000rpm离心10min。上层水相转移到另一离心管中,沸水中水浴10min,12000rpm离心5min,吸取上清用作扩增模板。用overlap PCR中的overlap-1和overlap-4作为检测引物,扩增片段为1.6kb。PCR产物经1%琼脂糖凝胶电泳后,观察扩增条带大小,切胶回收目的片段。用HindIII进行酶切或PCR产物直接测序方法来确定死亡雏鹅是死于拯救病毒或是亲本病毒。
结果表明,从肝脏、肠道组织提取病毒核酸作为DNA模板,能够扩增出包含遗传标记位点在内的1.6kb的特异片段,这些扩增片段均不能为HindIII所切开。对PCR产物的直接测序也证实在HindIII位点处均存在2个预设的碱基突变(图7b),这充分说明在拯救病毒攻毒组中,死亡雏鹅确实源于拯救的感染所致,可以排出攻毒试验期间,雏鹅感染亲本病毒或其它GPV野毒的可能。当前的试验结果表明,拯救病毒拥有与亲本LH病毒株相似的毒力。
SEQUENCE LISTING
<110> 扬州大学
<120> 一种鹅细小病毒感染性克隆的拯救方法
<130>
<160> 5
<170> PatentIn version 3.3
<210> 1
<211> 22
<212> DNA
<213> 人工序列
<400> 1
gcaggaacaa ttaccaggta cg 22
<210> 2
<211> 21
<212> DNA
<213> 人工序列
<400> 2
gtggtcggtg agtttccctg t 21
<210> 3
<211> 21
<212> DNA
<213> 人工序列
<400> 3
acagggaaac tcaccgacca c 21
<210> 4
<211> 20
<212> DNA
<213> 人工序列
<400> 4
cggcccatgg tgccataagc 20
<210> 5
<211> 5047
<212> DNA
<213> 鹅细小病毒LH株
<400> 5
tcattggagg gttcgttcgt tcgaaccagc caatcagggg agggggaagt gacgcaagtt 60
ccggtgacgc acatccggtg acgtagttcc ggtcacgtgc ttcctgtcac gtgtttccgg 120
tcacgtgact tccggtcatg tgacttccgg tgacgtgttt ccggctgtta ggttgaccgc 180
gcgcatgcgc gcggttagcc caatagttaa gccggaaaca cgtcaccgga agtcacatga 240
ccggaagtca cgtgaccgga aacacgtgac aggaagcacg tgaccggaac tacgtcaccg 300
gatgtgcgtc accggaactt gcgtcacttc cccctcccct gattggctgg ttcgaacgaa 360
cgaaccctcc aatgagactc aaggacaaga ggatattttg cgcgccagga agtgacgtgc 420
aatgccaccc tatataaccc aggaaacttc cggtttagtt cattcgttac tctgctctca 480
gagagaacgg acctcaggtc ggagagatgg cactttctag gcctcttcag atttcttctg 540
ataaattcta tgaagttatc attagattac catcggatat tgatcaagat gtccccggtc 600
tgtctcttaa ctttgtagaa tggctttcta ccggagtttg ggagcccacg ggcatctgga 660
acatggagca tgtgaatcta ccgatggtga ccttggcaga gaagatcaag aacattttca 720
tacaaagatg gaatcagttc aaccaggacg aaacggactt cttctttcaa ctggaagaag 780
gcagtgagta cattcatctt cattgctgta ttgcccaggg caatgtacgg tcttttgttc 840
tcgggagata tatgtctcag ataaaagact ctatcataag agatgtatat gaagggaaac 900
aaatcaagat ccccgattgg tttgctatta ctaaaaccaa gaggggagga cagaataaga 960
ccgtgactgc agcatacata ctgcattacc ttattcctaa aaagcaacct gaactgcaat 1020
gggcctttac caatatgcct ttattcactg ctgctgctct ttgtctgcaa aagcggcaag 1080
aattgctgga tgcatttcaa gaaagtgatt tggctgcccc tttacctgat cctcaagcat 1140
caactgtggc accgcttatt tccaacagag cggcaaagaa ctatagcaac cttgttgatt 1200
ggctcattga aatggggata acatctgaga agcaatggct cactgagaac cgagagagct 1260
acagaagctt tcaagcaact tcttcaaata atagacaagt gaaagctgca ctggaaaatg 1320
cccgtgctga aatgttattg acaaagactg caaccgatta cctgatagga aaagaccctg 1380
tcctggacat aactaagaat agggtctatc agattctgaa aatgaataac tacaaccctc 1440
aatacatagg aagtatcctg tgcggctggg tgaagagaga gttcaacaaa agaaacgcca 1500
tatggctcta cggacctgcc accaccggga agaccaacat tgcagaagct attgcccatg 1560
ctgtaccctt ctatggctgt gttaactgga ctaatgagaa ctttcctttt aatgattgtg 1620
ttgataaaat gctgatttgg tgggaggagg gaaaaatgac taataaggtt gttgaatctg 1680
caaaagcaat tttaggaggg tctgctgtcc gggtagatca gaaatgtaaa ggatctgttt 1740
gtattgaacc tactcctgta attattacta gtaatactga tatgtgtatg attgttgatg 1800
gcaactctac tacaatggaa catagaatac cgttagagga gcgtatgttt caaatcgtcc 1860
tatcacataa attggagcct tcttttggaa aaatttctaa aaaagaagtc agagaatttt 1920
tcaaatgggc caatgataat ctagttcctg ttgtgtctga gttcaaagtc cgaacgaatg 1980
aacaaactaa cttgccagag cccgttcctg aacgagcgaa cgagccggag gagcctccta 2040
agatctgggc tcctcctact agggaggagt tagaagagct tttaagagcc agcccagaat 2100
tgttctcatc agtcgctcca attcctgtga ctcctcagaa ctcccctgag cctaagagaa 2160
gcaggaacaa ttaccaggta cgctgcgctt tgcatactta tgacaattct atggatgtat 2220
ttgaatgtat ggaatgtgag aaagcaaact ttcctgaatt tcaacctctg ggagaaaatt 2280
attgtgatga acatgggtgg tatgattgtg ctatatgtaa agagttgaaa aatgaacttg 2340
cagaaattga gcatgtgttt gaacttgatg atgctgaaaa tgaacaataa agatgactca 2400
aagcagatat gtctactttt ttagattctt ttgaagagtg gtatgaaact gcagccgcct 2460
cgtggcggaa tctgaaagct ggagcccctc acccaaaacc aaaccagcag actcagtctg 2520
tgtctccagc cagagaaccc gaacgaagag ataataaccg gggctttgta cttcctggct 2580
ataagtatct tggccctggt aacggccttg ataaagggcc acccgttaat aaggcggaca 2640
gcgtcgcgct tgaacacgac aaggcctacg accaacagct taaagcggga gacaacccat 2700
atataaaatt caatcacgct gaccaggact ttatagatag cctccaagac gaccagtcgt 2760
tcggaggtaa tcttggaaag gctgtatttc aggccaaaaa acgtatctta gaaccatttg 2820
gcctagtaga agatcctgtc aacacggcac ctgcaaaaaa aaatacaggg aagcttaccg 2880
accactaccc ggtagttaag aagcctaaac ttaccgagga agtcagtgcg ggaggtggta 2940
gtagtgccgt acaagacgga ggagccaccg cggagggcac cgaacctgtg gcagcatctg 3000
aaatggcaga gggaggaggc ggagctttgg gcgacgcttc agggggtgcc gatggagtgg 3060
gtaatgcctc gggaaattgg cattgcgatt cccaatggat gggaaacaca gtcatcacaa 3120
agaccaccag aacctgggtc ctgccaagct acaacaacca catctacaaa gcgattacca 3180
gtggaacctc tcaagatgca aatgtccagt atgcaggata cagtacccct tggggatact 3240
ttgatttcaa ccgcttccac tgccacttct cccctagaga ctggcagaga cttatcaaca 3300
accattgggg aatcagaccc aagtctctta aattcaagat cttcaatgtc caagtcaaag 3360
aagtcacaac gcaggatcag acgaagacca ttgcaaacaa tctcacgtca acaattcaag 3420
tctttacgga tgacgagcat caactcccgt atgtcctggg ctcggctacg gaaggcacca 3480
tgccgccgtt cccgtcggat gtctatgccc tgccgcagta cgggtattgc acaatgcaca 3540
ccaaccagaa cggtgcacga ttcaatgacc ggagtgcatt ctactgctta gaatacttcc 3600
ccagtcagat gctaagaaca ggcaacaact ttgagttcac gtttgacttt gaagaagttc 3660
ctttccacag catgttcgct cattcacagg acttagacag gctgatgaac cccttagtgg 3720
atcaatacct ctggaatttc aatgaggtag acagcagcag aaatgctcaa tttaaaaagg 3780
ctgtgaaagg cgcttatgga accatgggcc gcaattggct gccaggacct aaattcctgg 3840
accagagagt tagggcctat acaggcggaa cagataatta tgcaaactgg aacatctgga 3900
gtaatggaaa caaggttaat ttgaaggaca ggcagtacct cctgcaaccc ggacctgtat 3960
cagctactca tacagaagca gaggcttcca gtatcccagc ccaaaatatt ttaggtttag 4020
ctaaagatcc atacagatct ggcagcacta cagcaggaat aagtgatatt atggtcacgg 4080
acgagcagga agtagcacct acaaacggcg tagggtggaa accatatggc aagactgtaa 4140
cgaatgaaca aaacactact acagctccta caagttcaga tctggatgtt cttggagctt 4200
taccaggaat ggtttggcag aacagggata tatatctgca gggacctatt tgggcaaaaa 4260
taccgaagac tgatggtaaa ttccatcctt ctccgaatct cggaggattt ggtctgcaca 4320
atccaccacc gcaggtgttc atcaagaata caccagtgcc tgcagaccct ccagtagaat 4380
acgtgcacca gaagtggaat tcctacataa cccagtactc tacgggccag tgtacagtag 4440
agatggtgtg ggagctgaga aaagagaatt caaagagatg gaacccagaa atccagttca 4500
ccagtaattt cagtgacaga acaagcatca tgtttgcacc taatgaaact ggtggatatg 4560
tagaagatag attaattgga accagatatc taactcaaaa tctgtaaatt ctgtgtaaaa 4620
attcaaataa agcacttcct ggcgcgcaaa atatcctctt gtccttgagt ctcattggag 4680
ggttcgttcg ttcgaaccag ccaatcaggg gagggggaag tgacgcaagt tccggtgacg 4740
cacatccggt gacgtagttc cggtcacgtg cttcctgtca cgtgtttccg gtcacgtgac 4800
ttccggtcat gtgacttccg gtgacgtgtt tccggcttaa ctattgggct gaccgcgcgc 4860
atgcgcgcgg tcaacctaac agccggaaac acgtcaccgg aagtcacatg accggaagtc 4920
acgtgaccgg aaacacgtga caggaagcac gtgaccggaa ctacgtcacc ggatgtgcgt 4980
caccggaact tgcgtcactt ccccctcccc tgattggctg gttcgaacga acgaaccctc 5040
caatgag 5047
- 还没有人留言评论。精彩留言会获得点赞!