基于转座酶建立过表达稳定系的质粒载体及其应用的制作方法
本发明属于基因工程领域,具体的说,本发明涉及到运用gateway克隆技术构建质粒载体,该一系列质粒载体带有不同的标签序列和筛选基因gfp,通过转座子系统将目的基因整合到细胞基因组,实现基因的转移。特别涉及一种基于转座酶建立过表达稳定系的质粒载体及其应用
背景技术:
过表达稳定细胞株是指外源dna整合到宿主细胞基因组中,可使宿主细胞长期表达目的基因,与瞬转株相比,稳定细胞株能够很大程度上的方便实验研究和降低实验成本。因此,构建基因过表达稳定株在基因功能与机制的研究中被广泛应用。目前构建稳定细胞株主要是通过病毒包装、感染目的细胞的方式来实现的,这种方法耗时长,通常需要两个星期左右。此外,由于所插入的目的基因片段大小受到病毒质粒自身大小的限制,通常目的基因片段大小不能超过3kb,并且操作人员有感染病毒的潜在安全隐患。与此同时,过表达质粒载体通常采用插入到多克隆位点的方法构建,这种方法不仅效率较低,并且消耗时间长,极大地影响了科研人员的实验进程。
转座子(transposon)最早是由mcclintock于20世纪50年代在研究玉米籽粒的颜色变异中发现的一类不依靠同源重组而能在基因组内和基因组间发生位置改变的dna序列,因此也被称为跳跃基因或者可移动元件。dna转座子是其中一种转座机制类型,目前使用较为广泛的dna转座子类型是sleepingbeauty转座子。首个有活性的sleepingbeauty转座子是通过对鱼类基因组中多个失活的tc1/mariner转座子进行分子重建产生的,该转座子能够插入到at二碱基区域,并具有插入至内含子中的倾向。此后,为了进一步提高sleepingbeauty的转座活性,研究人员通过对sleepingbeauty转座酶基因氨基酸突变的方法对其酶活性进行了广泛的筛选,最终获得了活性增加100倍的sleepingbeauty突变体(sb100x))。经过改良后的sleepingbeauty转座子采用“剪切-粘贴”的转座机制,具体的说是:在细胞中,转座酶sb100x识别sleepingbeauty转座子两端特异的反向重复序列(itrs),并将转座子序列切出,然后通过dna攻击的方式插入新的基因组位置。这种sleepingbeauty转座子的优点是:构建稳定株不需要包装病毒,大大的缩短了稳定株构建的时间,避免了包装病毒过程中所产生的生物安全问题;同时插入的目的片段大小不受限制;稳定重组效率高。
gateway克隆技术是由invitrogen公司开发的一项基因克隆新技术,该技术利用位点特异性重组反应:bp反应和lr反应,并利用大肠杆菌自杀基因ccdb,极大地提高了克隆的效率,同时大大的缩短了构建质粒的时间。
技术实现要素:
为了克服现有技术的不足,本发明的首要目的在于提供一系列基于转座酶快速建立过表达稳定系的质粒载体。
本发明的另一目的在于满足常规的在真核细胞中表达目的基因的需要,本发明提供的一系列质粒载体中带有flag、ha、v5或myc标签序列,这些标签序列的存在使目的基因的western、免疫荧光、elisa等的检测通过flag、ha、v5或myc抗体即可进行,节省了购买抗体或自行制备抗体的费用和时间,对于进行ip或co-ip也带来了很大的便利。同时本发明提供的一系列质粒载体分为带有筛选基因gfp和不带筛选基因gfp两类,方便流式实验筛选出阳性细胞,同时也可以验证质粒转染的效率。
该质粒载体包括sleepingbeauty转座子元件(itrs),强启动子cag驱动的ccdb基因元件,ccdb基因元件两侧分别含有重组位点attr1和attr2,在重组位点attr1的上游带有不同标签序列。同时该系列质粒载体分为带有筛选基因gfp和不带筛选基因gfp两类。通过gatewaylr反应生成含带有sleepingbeauty转座子作用元件(itrs),不同标签序列和筛选基因gfp的质粒载体。同时采用该系统能够很好地将目的基因整合到受体基因组中,实现基因的转移。
本发明提供以psm1.1-ha-puro-gfp为基础,构建用于基因功能与机制研究的一系列质粒载体。本方法融合了转座子和gateway克隆技术的优点,可简单高效地用于构建过表达稳定株。
本发明的另一目的在于提供上述质粒载体的应用。
本发明的目的通过下述技术方案实现:
本发明提供一系列基于转座酶建立过表达稳定系的质粒载体,是含有irdr-l-irdr-r盒的双链环状质粒,所述irdr-l-irdr-r盒包括irdr-l序列、启动子、标签序列、attr1序列、反向选择性标记基因序列、attr2序列、筛选基因序列和irdr-r序列。
所述的irdr-l序列为seqidno.1中自5′端第8877位~9103位碱基的反向互补序列;所述的irdr-r序列为seqidno.1中自5′端第5731位~5958位碱基。
所述的启动子为cag启动子,其序列为seqidno.1中自5′端第1位~1132位碱基。
所述的标签序列为ha标签序列、myc标签序列、flag标签序列或v5标签序列。
所述的ha标签序列为seqidno.1中自5′端第1172位~1201位碱基。
所述的myc标签序列为seqidno.2所示。
所述的flag标签序列为seqidno.3所示。
所述的v5标签序列为seqidno.4所示。
所述的attr1序列为seqidno.1中自5′端第1211位~1335位碱基;所述的attr2序列为seqidno.1中自5′端第2791位~2915位碱基的反向互补序列。
所述的反向选择性标记基因序列是指自杀基因ccdb,其序列为seqidno.1中自5′端第2445位~2750位碱基。
所述的筛选基因为抗性与荧光筛选基因,或抗性筛选基因。
所述的抗性筛选基因是指嘌呤霉素(puromycin)抗性基因,其序列为seqidno.1中自5′端第3464位~4063位碱基。
所述的荧光筛选基因是指绿色荧光蛋白(gfp)基因,其序列为seqidno.1中自5′端第4704位~5438位碱基。
本发明首先构建载有sleepingbeauty转座子作用元件,gatewaycassette-终止子片段,ha标签tag和gfp筛选基因的psm1.1-ha-puro-gfp质粒载体,随后以psm1.1-ha-puro-gfp为模板,通过改动标签序列tag,改造构建分别载有flag标签序列,myc标签序列的两个tag标签序列的质粒psm1.1-flag-puro-gfp和psm1.1-myc-puro-gfp。随后分别以它们三个质粒为模板,构建了去掉筛选基因gfp的质粒,分别是psm1.1-flag-puro,psm1.1-ha-puro和psm1.1-myc-puro。最后以psm1.1-flag-puro为模板,通过改动标签序列tag,改造构建载有v5标签序列的psm1.1-v5-puro。该系列质粒的发明将使得构建稳定株和质粒载体更加高效便利,同时满足各种常规的在真核细胞中表达目的基因的需要。构建7个质粒载体测序结果见序列表。
本发明一系列质粒中的psm1.1-ha-puro-gfp,具有seqidno.1所示的核苷酸序列。
所述psm1.1-ha-puro-gfp质粒载体,以plentipgkpurodest质粒,pcagig质粒,pt2/onc质粒为模板,采用如下引物对进行扩增:
f1(seqidno.5)和r1(seqidno.6),f2(seqidno.7)和r2(seqidno.8),f3(seqidno.9)和r3(seqidno.10),f4(seqidno.11)和r4(seqidno.12),f5(seqidno.13)和r5(seqidno.14),f6(seqidno.15)和r6(seqidno.16),f7(seqidno.17)和r7(seqidno.18)。
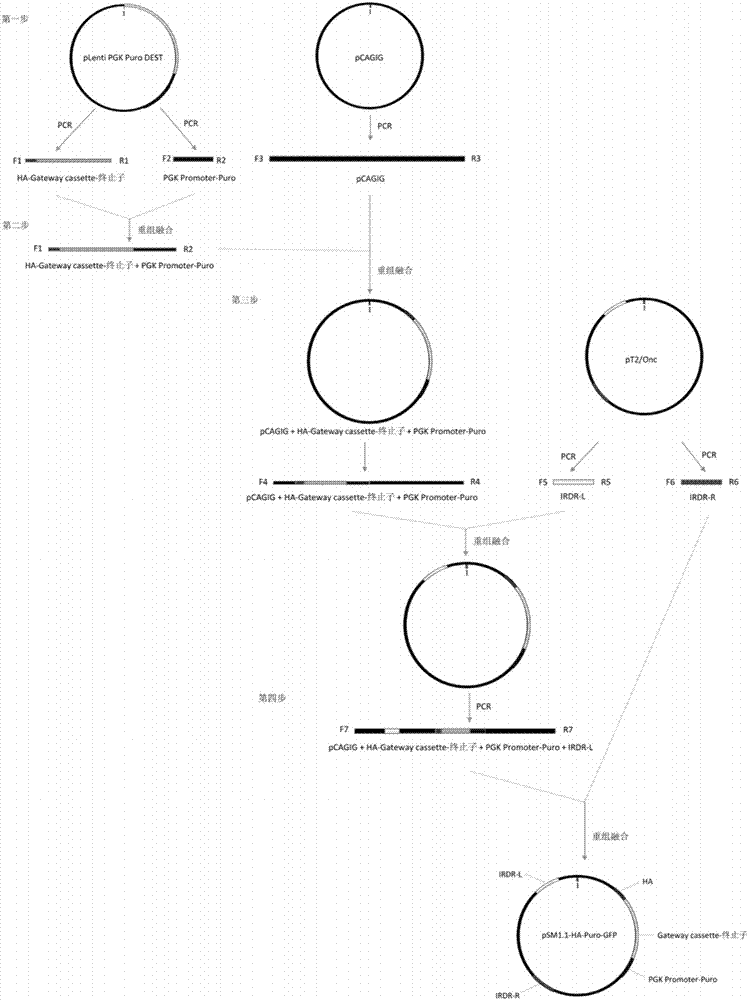
本发明所述psm1.1-ha-puro-gfp质粒载体的构建方法,包括如下步骤:
第一步:以plentipgkpurodest质粒为模板,以f1和r1为引物,pcr扩增质粒的gatewaycassette-终止子片段,以plentipgkpurodest质粒为模板,以f2和r2为引物,pcr扩增质粒的pgkpromoter和puro抗性基因的融合片段,胶回收上述pcr产物片段,然后,以gatewaycassette-终止子片段和pgkpromoter和puro抗性基因的融合片段为模板,进行第一轮重组融合pcr反应,胶回收所得片段。
第二步:以pcagig质粒为模板,以f3和r3为引物,pcr反向扩增全长质粒。胶回收上述pcr产物后,与第一步所得片段共同作为模板,进行第二轮重组融合pcr反应,胶回收所得片段后,以f4和r4为引物,pcr反向扩增全长质粒。胶回收上述pcr产物。
第三步:以pt2/onc质粒为模板,分别以f5,r5和f6,r6为引物,pcr扩增质粒的sleepingbeauty的反向末端重复序列irdr-l和irdr-r。胶回收上述pcr产物片段后,以pcr扩增质粒的反向末端重复序列irdr-l和第二步所得片段共同作为模板,进行第三轮重组融合pcr反应,胶回收所得片段后,以f7和r7为引物,pcr反向扩增全长质粒。胶回收上述pcr产物。
第四步:以pcr扩增质粒的反向末端重复序列irdr-r和第三步所得片段共同作为模板,进行第四轮重组融合pcr反应,构建成psm1.1-ha-puro-gfp质粒,质粒转化至大肠杆菌db3.1,进行扩增。
本发明一系列质粒中的psm1.1-ha-puro质粒载体,以psm1.1-ha-puro-gfp为模板,采用如下引物对进行扩增:
degfp-f(seqidno.19)和degfp-r(seqidno.20)。
本发明所述psm1.1-ha-puro质粒载体的构建方法,包括如下步骤:
以psm1.1-ha-puro-gfp为模板,以degfp-f和degfp-r为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-ha-puro质粒,质粒转化至大肠杆菌db3.1,进行扩增。
本发明一系列质粒中的psm1.1-myc-puro-gfp质粒载体,以psm1.1-ha-puro-gfp为模板,采用如下引物对进行扩增:
f8(seqidno.21)和r8(seqidno.22)。
本发明所述psm1.1-myc-puro-gfp质粒载体的构建方法,包括如下步骤:
以psm1.1-ha-puro-gfp为模板,以f8和r8为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-myc-puro-gfp质粒,质粒转化至大肠杆菌db3.1,进行扩增。
本发明一系列质粒中的psm1.1-myc-puro质粒载体,以psm1.1-myc-puro-gfp为模板,采用如下引物对进行扩增:
degfp-f(seqidno.19)和degfp-r(seqidno.20)。
本发明所述psm1.1-myc-puro质粒载体的构建方法,包括如下步骤:
以psm1.1-myc-puro-gfp为模板,以degfp-f和degfp-r为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-myc-puro质粒,质粒转化至大肠杆菌db3.1,进行扩增。
本发明一系列质粒中的psm1.1-flag-puro-gfp质粒载体,以psm1.1-ha-puro-gfp为模板,采用如下引物对进行扩增:
f9(seqidno.23)和r9(seqidno.24)。
本发明所述psm1.1-flag-puro-gfp质粒载体的构建方法,包括如下步骤:
以psm1.1-ha-puro-gfp为模板,以f9和r9为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-flag-puro-gfp质粒,质粒转化至大肠杆菌db3.1,进行扩增。
本发明一系列质粒中的psm1.1-flag-puro质粒载体,以psm1.1-flag-puro-gfp为模板,采用如下引物对进行扩增:
degfp-f(seqidno.19)和degfp-r(seqidno.20)。
本发明所述psm1.1-flag-puro质粒载体的构建方法,包括如下步骤:
以psm1.1-flag-puro-gfp为模板,以degfp-f和degfp-r为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-flag-puro质粒,质粒转化至大肠杆菌db3.1,进行扩增。
本发明一系列质粒中的psm1.1-v5-puro质粒载体,以psm1.1-flag-puro为模板,采用如下引物对进行扩增:
f10(seqidno.25)和r10(seqidno.26)。
本发明所述psm1.1-v5-puro质粒载体的构建方法,包括如下步骤:
以psm1.1-flag-puro为模板,以f10和r10为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-v5-puro质粒,质粒转化至大肠杆菌db3.1,进行扩增。
所述的质粒载体在快速构建过表达稳定株中的应用。
本发明的机理是:本发明的质粒载体将gateway克隆与转座子技术融合,在构建细胞稳定株具有试验周期短、使用安全和插入目的基因片段大小不受限制等明显优势,节省科研人员大量的时间,因而对于提高科学研究效率是一个有力的工具。
本发明相对于现有技术,具有如下的优点及效果:
(1)采用sleepingbeauty转座子方法,构建稳定株的时间短,插入的目的片段大小不受影响,稳定重组效率高;同时不用包装病毒,避免了包装病毒过程中所产生的生物安全问题。
(2)采用gateway克隆技术,过表达质粒载体构建时间短,阳性率高;
(3)本发明的质粒载体带有sleepingbeauty转座子末端反向重复序列和gatewaycassette序列,基于转座酶的方法能快速高效的构建过表达稳定系。
(4)带有各种不同的标签序列tag和带有筛选基因gfp,满足常规的在真核细胞中表达目的基因的需要。
附图说明
图1是本发明系列质粒载体的结构示意图;其中,克隆质粒psm1.1-ha-puro-gfp,psm1.1-ha-puro,psm1.1-myc-puro-gfp,psm1.1-myc-puro,psm1.1-flag-puro-gfp,psm1.1-flag-puro,psm1.1-v5-puro载体结构的示意图。
图2是本发明psm1.1-ha-puro-gfp载体的构建示意图。
图3是本发明psm1.1-ha-puro载体的构建示意图。
图4是本发明psm1.1-flag-puro-gfp载体的构建示意图。
图5是本发明psm1.1-ha-puro-gfp高效表达外源基因;其中,(a)为从cdna中扩增出fbxw7基因的琼脂糖电泳图;(b)gateway分子克隆中,bp反应挑4个单克隆,进行菌落验证的琼脂糖电泳图;(c)gateway分子克隆中,lb反应挑5个单克隆,进行菌落验证的琼脂糖电泳图;(d)psm1.1-ha-fbxw7-puro-gfp质粒转染到hela细胞后,通过荧光显微镜,观察转染效率;(e)westernblot实验验证psm1.1-ha-puro-gfp质粒是否表达。
具体实施方式
下面结合实施例及附图对本发明作进一步详细的描述,但本发明的实施方式不限于此。
下列实施例中未注明具体条件的实验方法,通常按照常规条件或按照制造厂商所建议的条件。
pt2/onc质粒是是常规市售产品,文献出处为:cancergenediscoveryinsolidtumoursusingtransposon-basedsomaticmutagenesisinthemouse.collierls,carlsoncm,ravimohans,dupuyaj,largaespadadanature.2005jul14.436(7048):272-6.
克隆质粒psm1.1-ha-puro-gfp,psm1.1-ha-puro,psm1.1-myc-puro-gfp,psm1.1-myc-puro,psm1.1-flag-puro-gfp,psm1.1-flag-puro,psm1.1-v5-puro载体结构的示意图,如图1所示。
实施例1:
本实施例用于研究psm1.1系列载体的构建方案,由于psm1.1一系列载体构建方法基本一致,现选取psm1.1-ha-puro-gfp,psm1.1-ha-puro,psm1.1-flag-puro-gfp这三种质粒载体的构建方案来代表psm1.1系列载体的构建方法。
1.1psm1.1-ha-puro-gfp质粒构建
第一步:以plentipgkpurodest质粒为模板,以f1和r1为引物,pcr扩增质粒的gatewaycassette-终止子片段,以plentipgkpurodest质粒为模板,以f2和r2为引物,pcr扩增质粒的pgkpromoter和puro抗性基因的融合片段,胶回收上述pcr产物片段,然后,以gatewaycassette-终止子片段和pgkpromoter和puro抗性基因的融合片段为模板,进行一轮重组融合pcr反应,胶回收所得片段。
第二步:以pcagig质粒为模板,以f3和r3,pcr反向扩增全长质粒。胶回收上述pcr产物后,与第一步所得片段共同作为模板,进行第二轮重组融合pcr反应,胶回收所得片段后,以f4和r4,pcr反向扩增全长质粒。胶回收上述pcr产物。
第三步:以pt2/onc质粒为模板,分别以f5,r5和f6,r6为引物,pcr扩增质粒的sleepingbeauty的反向末端重复序列irdr-l和irdr-r。胶回收上述pcr产物片段后,以pcr扩增质粒的反向末端重复序列irdr-l和第二步所得片段共同作为模板,进行第三轮重组融合pcr反应,胶回收所得片段后,以f7和r7,pcr反向扩增全长质粒。胶回收上述pcr产物。
第四步:以pcr扩增质粒的反向末端重复序列irdr-r和第三步所得片段共同作为模板,进行第四轮重组融合pcr反应,构建成psm1.1-ha-puro-gfp质粒(核苷酸序列如seqidno.1),质粒转化至大肠杆菌db3.1,进行扩增。psm1.1-ha-puro-gfp质粒的构建示意图,如图2所示。
1.2psm1.1-ha-puro质粒构建
所述的psm1.1-ha-puro质粒载体的构建方法,包括如下步骤:
以psm1.1-ha-puro-gfp为模板,以degfp-f和degfp-r为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-ha-puro质粒,质粒转化至大肠杆菌db3.1,进行扩增。psm1.1-ha-puro载体的构建示意图,如图3所示。
1.3psm1.1-flag-puro-gfp质粒构建
以psm1.1-ha-puro-gfp为模板,以f9和r9为引物,pcr反向扩增质粒,胶回收上述pcr产物后,pcr产物进行末端平滑化及5′磷酸(p)化处理,自身连接成环,构建成psm1.1-flag-puro-gfp质粒,质粒转化至大肠杆菌db3.1,进行扩增。psm1.1-flag-puro-gfp载体的构建示意图,如图3所示。
实施例2:
psm1.1-ha-puro-gfp是发明人构建成功的原始质粒,其余系列载体质粒均由此原始质粒通过只改动标签序列和筛选基因gfp,其他质粒元件不变,从而改造成含不同标签序列与是否含有筛选基因gfp的一系列质粒载体。本实例用于研究这个原始质粒psm1.1-ha-puro-gfp表达外源基因实施方案。
1.1psm1.1-ha-fbxw7-puro-gfp质粒构建
以hela细胞的cdna为模板,f11和r11为引物,pcr扩增出fbxw7基因(genebank登录号:nm_001349798.1),跑胶确定fbxw7基因被特异性扩增出来(图5a),回收pcr产物,回收的pcr产物通过bp反应,重组到pdonr221入门载体上,转化到超级感受态菌omnimax2-t1中,涂kana+平板,挑单克隆使用m13正反向引物(m13-f和m13-r)进行菌落验证(图5b)。将菌落验证对的重组菌使用碧云天的小规模提取试剂盒(d0003))进行质粒提取,构建成pdonr221-fbxw7入门克隆。然后通过lr反应,重组到psm1.1-ha-puro-gfp表达载体上,转化到超级感受态菌omnimax2-t1中,涂amp+平板,挑单克隆使用psm1.1-f、psm1.1-r正反向引物进行菌落验证(图5c)。将菌落验证对的菌种送去测序,进一步验证质粒。构建成psm1.1-ha-fbxw7-puro-gfp表达质粒。
所用引物序列:
f11(seqidno.27)和r11(seqidno.28);
m13-f(seqidno.29)和m13-r(seqidno.30)。
1.2psm1.1-ha-fbxw7-puro-gfp质粒转化,挑单克隆进行菌落验证,大规模提取质粒
质粒转化采用热击法,将psm1.1-ha-fbxw7-puro-gfp质粒约1μl加入到50μl的商品化的超级感受态菌omnimax2-t1当中,轻轻吹打混匀,在冰上静置30min,42摄氏度水浴热击90s,再加入250μl的lb培养基,37摄氏度200rpm摇菌摇上1h,将全部菌液涂到含氨苄抗生素的lb平板上,37℃的条件下培养14h,挑取单克隆,使用psm1.1-f和psm1.1-r作为引物进行菌落验证鉴定,把验证正确的菌种保存。构建的重组质粒命名为psm1.1-ha-fbxw7-puro-gfp。
所用引物序列:
psm1.1-f(seqidno.31)和psm1.1-r(seqidno.32)。
取200μl上述重组菌到250l的lb培养基中,37摄氏度,200rpm摇菌摇上14h后,使用biotool的大规模提取试剂盒(b22213))提取质粒,得到质粒浓度为950ng/μl,500μl体系。
1.3质粒电转染,westernblot实验验证
吸取300μlopti-mem分装入1.5ml的ep管中,加入10μg提取好的psm1.1-ha-fbxw7-puro-gfp质粒和2μg转座酶质粒sb100x,取106对数生长期的hela细胞,加入300μlopti-mem重悬细胞,然后将细胞悬液加入到预先与质粒混匀的300μlopti-mem中,柔和混匀后,将细胞和质粒的混合液转移至电转杯中,以250v,950uf的条件在bio-radgenepulsarii进行电击,电击后将混合液吸取到6cm皿中,加入4ml培养基培养24h后,第二天加入puromycin至2μg/ml进行筛选,3天后进行westernblot实验。通过gfp荧光可以观察质粒转染的效率(图5d)。
取100μlripa裂解液裂解筛选后的细胞和阴性对照组的hela细胞,冰上裂解30min后,4度离心20min,取上清,通过bradford方法测蛋白浓度。蛋白样品经10%的sds-page胶电泳后,通过350ma,2h方法将蛋白样品转印到pvdf膜上。转膜后,5%的脱脂奶粉,室温封闭1h,fbxw7一抗抗体稀释液(bethyl,a301-720a,1:1000),ha一抗抗体稀释液(cst,3724,1:1000),cycline一抗抗体稀释液(cst,20808,1:1000),gapdh一抗抗体稀释液(cst,2118,1:1000)4度孵育过夜。过夜孵育完后,pbst洗涤膜3次,每次10min。再室温孵育二抗(1:15000)1h,pbst洗涤膜3次。加入显色液,曝光。
结果显示:psm1.1-ha-fbxw7-puro-gfp质粒成功表达大小约90kdaha-fbxw7融合蛋白(图5e),表明构建在psm1.1-ha-puro-gfp载体上的fbxw7基因能在hela细胞中成功表达。
上述实施例为本发明较佳的实施方式,但本发明的实施方式并不受上述实施例的限制,其他的任何未背离本发明的精神实质与原理下所作的改变、修饰、替代、组合、简化,均应为等效的置换方式,都包含在本发明的保护范围之内。
sequencelisting
<110>中山大学孙逸仙纪念医院
<120>基于转座酶建立过表达稳定系的质粒载体及其应用
<130>1
<160>32
<170>patentinversion3.5
<210>1
<211>9194
<212>dna
<213>artificialsequence
<220>
<223>psm1.1-ha-puro-gfp的核苷酸序列
<220>
<221>cag启动子
<222>(1)..(1132)
<220>
<221>ha标签序列
<222>(1172)..(1201)
<220>
<221>attr1
<222>(1211)..(1335)
<220>
<221>自杀基因ccdb
<222>(2445)..(2750)
<220>
<221>attr2的反向互补序列
<222>(2791)..(2915)
<220>
<221>嘌呤霉素(puromycin)抗性基因
<222>(3464)..(4063)
<220>
<221>绿色荧光蛋白(gfp)基因
<222>(4704)..(5438)
<220>
<221>irdr-r
<222>(5731)..(5958)
<220>
<221>irdr-l的反向互补序列
<222>(8877)..(9103)
<400>1
ggccgctctagcccctagttattaatagtaatcaattacggggtcattagttcatagccc60
atatatggagttccgcgttacataacttacggtaaatggcccgcctggctgaccgcccaa120
cgacccccgcccattgacgtcaataatgacgtatgttcccatagtaacgccaatagggac180
tttccattgacgtcaatgggtggagtatttacggtaaactgcccacttggcagtacatca240
agtgtatcatatgccaagtacgccccctattgacgtcaatgacggtaaatggcccgcctg300
gcattatgcccagtacatgaccttatgggactttcctacttggcagtacatctacgtatt360
agtcatcgctattaccatggtcgaggtgagccccacgttctgcttcactctccccatctc420
ccccccctccccacccccaattttgtatttatttattttttaattattttgtgcagcgat480
gggggcggggggggggggggggcgcgcgccaggcggggcggggcggggcgaggggcgggg540
cggggcgaggcggagaggtgcggcggcagccaatcagagcggcgcgctccgaaagtttcc600
ttttatggcgaggcggcggcggcggcggccctataaaaagcgaagcgcgcggcgggcggg660
agtcgctgcgttgccttcgccccgtgccccgctccgcgccgcctcgcgccgcccgccccg720
gctctgactgaccgcgttactcccacaggtgagcgggcgggacggcccttctcctccggg780
ctgtaattagcgcttggtttaatgacggcttgtttcttttctgtggctgcgtgaaagcct840
tgaggggctccgggagggccccggcaggaaggaaatgggcggggagggccttcgtgcgtc900
gccgcgccgccgtccccttctccctctccagcctcggggctgtccgcggggggacggctg960
ccttcgggggggacggggcagggcggggttcggcttctggcgtgtgaccggcggctctag1020
agcctctgctaaccatgttcatgccttcttctttttcctacagctcctgggcaacgtgct1080
ggttattgtgctgtctcatcattttggcaaagaattgattaattcgagcgaacgcgtggt1140
accgaagccgctagcgctaccggtcgccaccatgtacccatacgatgttccagattacgc1200
tgaattgatcacaagtttgtacaaaaaagctgaacgagaaacgtaaaatgatataaatat1260
caatatattaaattagattttgcataaaaaacagactacataatactgtaaaacacaaca1320
tatccagtcactatggcggccgcattaggcaccccaggctttacactttatgcttccggc1380
tcgtataatgtgtggattttgagttaggatccgtcgagattttcaggagctaaggaagct1440
aaaatggagaaaaaaatcactggatataccaccgttgatatatcccaatggcatcgtaaa1500
gaacattttgaggcatttcagtcagttgctcaatgtacctataaccagaccgttcagctg1560
gatattacggcctttttaaagaccgtaaagaaaaataagcacaagttttatccggccttt1620
attcacattcttgcccgcctgatgaatgctcatccggaattccgtatggcaatgaaagac1680
ggtgagctggtgatatgggatagtgttcacccttgttacaccgttttccatgagcaaact1740
gaaacgttttcatcgctctggagtgaataccacgacgatttccggcagtttctacacata1800
tattcgcaagatgtggcgtgttacggtgaaaacctggcctatttccctaaagggtttatt1860
gagaatatgtttttcgtctcagccaatccctgggtgagtttcaccagttttgatttaaac1920
gtggcaaatatggacaacttcttcgcccccgttttcaccatgggcaaatattatacgcaa1980
ggcgacaaggtgctgatgccgctggcgattcaggttcatcatgccgtttgtgatggcttc2040
catgtcggcagaatgcttaatgaattacaacagtactgtgatgagtggcagggcggggcg2100
taaacgcgtggatccggcttactaaaagccagataacagtatgcgtatttgcgcgctgat2160
ttttgcggtataagaatatatactgatatgtatacccgaagtatgtcaaaaagaggtatg2220
ctatgaagcagcgtattacagtgacagttgacagcgacagctatcagttgctcaaggcat2280
atatgatgtcaatatctccggtctggtaagcacaaccatgcagaatgaagcccgtcgtct2340
gcgtgccgaacgctggaaagcggaaaatcaggaagggatggctgaggtcgcccggtttat2400
tgaaatgaacggctcttttgctgacgagaacaggggctggtgaaatgcagtttaaggttt2460
acacctataaaagagagagccgttatcgtctgtttgtggatgtacagagtgatattattg2520
acacgcccgggcgacggatggtgatccccctggccagtgcacgtctgctgtcagataaag2580
tctcccgtgaactttacccggtggtgcatatcggggatgaaagctggcgcatgatgacca2640
ccgatatggccagtgtgccggtctccgttatcggggaagaagtggctgatctcagccacc2700
gcgaaaatgacatcaaaaacgccattaacctgatgttctggggaatataaatgtcaggct2760
cccttatacacagccagtctgcaggtcgaccatagtgactggatatgttgtgttttacag2820
tattatgtagtctgttttttatgcaaaatctaatttaatatattgatatttatatcattt2880
tacgtttctcgttcagctttcttgtacaaagtggttgatctcgaggagttaacgaattct2940
accgggtaggggaggcgcttttcccaaggcagtctggagcatgcgctttagcagccccgc3000
tgggcacttggcgctacacaagtggcctctggcctcgcacacattccacatccaccggta3060
ggcgccaaccggctccgttctttggtggccccttcgcgccaccttctactcctcccctag3120
tcaggaagttcccccccgccccgcagctcgcgtcgtgcaggacgtgacaaatggaagtag3180
cacgtctcactagtctcgtgcagatggacagcaccgctgagcaatggaagcgggtaggcc3240
tttggggcagcggccaatagcagctttgctccttcgctttctgggctcagaggctgggaa3300
ggggtgggtccgggggcgggctcaggggcgggctcaggggcggggcgggcgcccgaaggt3360
cctccggaggcccggcattctgcacgcttcaaaagcgcacgtctgccgcgctgttctcct3420
cttcctcatctccgggcctttcgacctgcagcccaagcttaccatgaccgagtacaagcc3480
cacggtgcgcctcgccacccgcgacgacgtccccagggccgtacgcaccctcgccgccgc3540
gttcgccgactaccccgccacgcgccacaccgtcgatccggaccgccacatcgagcgggt3600
caccgagctgcaagaactcttcctcacgcgcgtcgggctcgacatcggcaaggtgtgggt3660
cgcggacgacggcgccgcggtggcggtctggaccacgccggagagcgtcgaagcgggggc3720
ggtgttcgccgagatcggcccgcgcatggccgagttgagcggttcccggctggccgcgca3780
gcaacagatggaaggcctcctggcgccgcaccggcccaaggagcccgcgtggttcctggc3840
caccgtcggcgtctcgcccgaccaccagggcaagggtctgggcagcgccgtcgtgctccc3900
cggagtggaggcggccgagcgcgccggggtgcccgccttcctggagacctccgcgccccg3960
caacctccccttctacgagcggctcggcttcaccgtcaccgccgacgtcgaggtgcccga4020
aggaccgcgcacctggtgcatgacccgcaagcccggtgcctgacgcccgccccacgaccc4080
gcagcgcccgaccgaaaggagcgcacgaccccatgcatccaattccgcccccccccccta4140
acgttactggccgaagccgcttggaataaggccggtgtgcgtttgtctatatgttatttt4200
ccaccatattgccgtcttttggcaatgtgagggcccggaaacctggccctgtcttcttga4260
cgagcattcctaggggtctttcccctctcgccaaaggaatgcaaggtctgttgaatgtcg4320
tgaaggaagcagttcctctggaagcttcttgaagacaaacaacgtctgtagcgacccttt4380
gcaggcagcggaaccccccacctggcgacaggtgcctctgcggccaaaagccacgtgtat4440
aagatacacctgcaaaggcggcacaaccccagtgccacgttgtgagttggatagttgtgg4500
aaagagtcaaatggctctcctcaagcgtattcaacaaggggctgaaggatgcccagaagg4560
taccccattgtatgggatctgatctggggcctcggtgcacatgctttacatgtgtttagt4620
cgaggttaaaaaacgtctaggccccccgaaccacggggacgtggttttcctttgaaaaac4680
acgatgataatatggccacaaccatggtgagcaagggcgaggagctgttcaccggggtgg4740
tgcccatcctggtcgagctggacggcgacgtaaacggccacaagttcagcgtgtccggcg4800
agggcgagggcgatgccacctacggcaagctgaccctgaagttcatctgcaccaccggca4860
agctgcccgtgccctggcccaccctcgtgaccaccctgacctacggcgtgcagtgcttca4920
gccgctaccccgaccacatgaagcagcacgacttcttcaagtccgccatgcccgaaggct4980
acgtccaggagcgcaccatcttcttcaaggacgacggcaactacaagacccgcgccgagg5040
tgaagttcgagggcgacaccctggtgaaccgcatcgagctgaagggcatcgacttcaagg5100
aggacggcaacatcctggggcacaagctggagtacaactacaacagccacaacgtctata5160
tcatggccgacaagcagaagaacggcatcaaggtgaacttcaagatccgccacaacatcg5220
aggacggcagcgtgcagctcgccgaccactaccagcagaacacccccatcggcgacggcc5280
ccgtgctgctgcccgacaaccactacctgagcacccagtccgccctgagcaaagacccca5340
acgagaagcgcgatcacatggtcctgctggagttcgtgaccgccgccgggatcactctcg5400
gcatggacgagctgtacaagagatctcgagctcgatgagtttggacaaaccacaactaga5460
atgcagtgaaaaaaatgctttatttgtgaaatttgtgatgctattgctttatttgtaacc5520
attataagctgcaataaacaagttaacaacaacaattgcattcattttatgtttcaggtt5580
cagggggaggtgtgggaggttttttaaagcaagtaaaacctctacaaatgtggtacttaa5640
gagggatcgataaggatctagcttgtggaaggctactcgaaatgtttgacccaagttaaa5700
caatttaaaggcaatgctaccaaatactaattgagtgtatgtaaacttctgacccactgg5760
gaatgtgatgaaagaaataaaagctgaaatgaatcattctctctactattattctgatat5820
ttcacattcttaaaataaagtggtgatcctaactgacctaagacagggaatttttactag5880
gattaaatgtcaggaattgtgaaaaagtgagtttaaatgtatttggctaaggtgtatgta5940
aacttccgacttcaactgtatagggatcctctagctagagtcgacctcgagggggggccc6000
ggtacccagcttttgttccctttagtgagggttaatttcgagcttggcgtaatcatggtc6060
atagctgtttcctgtgtgaaattgttatccgctcacaattccacacaacatacgagccgg6120
aagcataaagtgtaaagcctggggtgcctaatgagtgagctaactcacattaattgcgtt6180
gcgctcactgcccgctttccagtcgggaaacctgtcgtgccagctgcattaatgaatcgg6240
ccaacgcgcggggagaggcggtttgcgtattgggcgctcttccgcttcctcgctcactga6300
ctcgctgcgctcggtcgttcggctgcggcgagcggtatcagctcactcaaaggcggtaat6360
acggttatccacagaatcaggggataacgcaggaaagaacatgtgagcaaaaggccagca6420
aaaggccaggaaccgtaaaaaggccgcgttgctggcgtttttccataggctccgcccccc6480
tgacgagcatcacaaaaatcgacgctcaagtcagaggtggcgaaacccgacaggactata6540
aagataccaggcgtttccccctggaagctccctcgtgcgctctcctgttccgaccctgcc6600
gcttaccggatacctgtccgcctttctcccttcgggaagcgtggcgctttctcatagctc6660
acgctgtaggtatctcagttcggtgtaggtcgttcgctccaagctgggctgtgtgcacga6720
accccccgttcagcccgaccgctgcgccttatccggtaactatcgtcttgagtccaaccc6780
ggtaagacacgacttatcgccactggcagcagccactggtaacaggattagcagagcgag6840
gtatgtaggcggtgctacagagttcttgaagtggtggcctaactacggctacactagaag6900
gacagtatttggtatctgcgctctgctgaagccagttaccttcggaaaaagagttggtag6960
ctcttgatccggcaaacaaaccaccgctggtagcggtggtttttttgtttgcaagcagca7020
gattacgcgcagaaaaaaaggatctcaagaagatcctttgatcttttctacggggtctga7080
cgctcagtggaacgaaaactcacgttaagggattttggtcatgagattatcaaaaaggat7140
cttcacctagatccttttaaattaaaaatgaagttttaaatcaatctaaagtatatatga7200
gtaaacttggtctgacagttaccaatgcttaatcagtgaggcacctatctcagcgatctg7260
tctatttcgttcatccatagttgcctgactccccgtcgtgtagataactacgatacggga7320
gggcttaccatctggccccagtgctgcaatgataccgcgagacccacgctcaccggctcc7380
agatttatcagcaataaaccagccagccggaagggccgagcgcagaagtggtcctgcaac7440
tttatccgcctccatccagtctattaattgttgccgggaagctagagtaagtagttcgcc7500
agttaatagtttgcgcaacgttgttgccattgctacaggcatcgtggtgtcacgctcgtc7560
gtttggtatggcttcattcagctccggttcccaacgatcaaggcgagttacatgatcccc7620
catgttgtgcaaaaaagcggttagctccttcggtcctccgatcgttgtcagaagtaagtt7680
ggccgcagtgttatcactcatggttatggcagcactgcataattctcttactgtcatgcc7740
atccgtaagatgcttttctgtgactggtgagtactcaaccaagtcattctgagaatagtg7800
tatgcggcgaccgagttgctcttgcccggcgtcaatacgggataataccgcgccacatag7860
cagaactttaaaagtgctcatcattggaaaacgttcttcggggcgaaaactctcaaggat7920
cttaccgctgttgagatccagttcgatgtaacccactcgtgcacccaactgatcttcagc7980
atcttttactttcaccagcgtttctgggtgagcaaaaacaggaaggcaaaatgccgcaaa8040
aaagggaataagggcgacacggaaatgttgaatactcatactcttcctttttcaatatta8100
ttgaagcatttatcagggttattgtctcatgagcggatacatatttgaatgtatttagaa8160
aaataaacaaataggggttccgcgcacatttccccgaaaagtgccacctgacgcgccctg8220
tagcggcgcattaagcgcggcgggtgtggtggttacgcgcagcgtgaccgctacacttgc8280
cagcgccctagcgcccgctcctttcgctttcttcccttcctttctcgccacgttcgccgg8340
ctttccccgtcaagctctaaatcgggggctccctttagggttccgatttagtgctttacg8400
gcacctcgaccccaaaaaacttgattagggtgatggttcacgtagtgggccatcgccctg8460
atagacggtttttcgccctttgacgttggagtccacgttctttaatagtggactcttgtt8520
ccaaactggaacaacactcaaccctatctcggtctattcttttgatttataagggatttt8580
gccgatttcggcctattggttaaaaaatgagctgatttaacaaaaatttaacgcgaattt8640
taacaaaatattaacgcttacaatttccattcgccattcaggctgcgcaactgttgggaa8700
gggcgatcggtgcgggcctcttcgctattacgccagctggcgaaagggggatgtgctgca8760
aggcgattaagttgggtaacgccagggttttcccagtcacgacgttgtaaaacgacggcc8820
agtgagcgcgcgtaatacgactcactatagggcgaattggagctcggatccctatacagt8880
tgaagtcggaagtttacatacacttaagttggagtcattaaaactcgtttttcaactact8940
ccacaaatttcttgttaacaaacaatagttttggcaagtcagttaggacatctactttgt9000
gcatgacacaagtcatttttccaacaattgtttacagacagattatttcacttataattc9060
actgtatcacaattccagtgggtcagaagtttacatacactaagttgactgtgcctttaa9120
acagcttggaaaattccagaaaatgatgtcatggctttagaagcttgatatccatggaat9180
tcactagtgcgcgc9194
<210>2
<211>33
<212>dna
<213>artificialsequence
<220>
<223>myc标签序列
<400>2
atggaacaaaaactcatctcagaagaggatctg33
<210>3
<211>27
<212>dna
<213>artificialsequence
<220>
<223>flag标签序列
<400>3
atggactacaaggacgacgatgacaag27
<210>4
<211>48
<212>dna
<213>artificialsequence
<220>
<223>v5标签序列
<400>4
atggcaggcaagccaatccctaaccctctgctgggcctggacagcacc48
<210>5
<211>45
<212>dna
<213>artificialsequence
<220>
<223>f1
<400>5
aatagtaatcaattacggggtacccatacgatgttccagattacg45
<210>6
<211>40
<212>dna
<213>artificialsequence
<220>
<223>r1
<400>6
gctttcttgtacaaagtggtgggtaggggaggcgcttttc40
<210>7
<211>40
<212>dna
<213>artificialsequence
<220>
<223>f2
<400>7
gaaaagcgcctcccctacccaccactttgtacaagaaagc40
<210>8
<211>38
<212>dna
<213>artificialsequence
<220>
<223>r2
<400>8
tccccgaaaagtgccacctgtcaggcaccgggcttgcg38
<210>9
<211>19
<212>dna
<213>artificialsequence
<220>
<223>f3
<400>9
caggtggcacttttcgggg19
<210>10
<211>31
<212>dna
<213>artificialsequence
<220>
<223>r3
<400>10
ccccgtaattgattactattaataactagtc31
<210>11
<211>26
<212>dna
<213>artificialsequence
<220>
<223>f4
<400>11
atatcaagcttctaaagccatgacat26
<210>12
<211>24
<212>dna
<213>artificialsequence
<220>
<223>r4
<400>12
ccctatagtgagtcgtattacgcg24
<210>13
<211>46
<212>dna
<213>artificialsequence
<220>
<223>f5
<400>13
taatacgactcactatagggcagttgaagtcggaagtttacataca46
<210>14
<211>48
<212>dna
<213>artificialsequence
<220>
<223>r5
<400>14
tggctttagaagcttgatatttagtgtatgtaaacttctgacccactg48
<210>15
<211>46
<212>dna
<213>artificialsequence
<220>
<223>f6
<400>15
cccaagttaaacaatttaaattgagtgtatgtaaacttctgaccca46
<210>16
<211>46
<212>dna
<213>artificialsequence
<220>
<223>r6
<400>16
cagcttttgttccctttagtcagttgaagtcggaagtttacataca46
<210>17
<211>23
<212>dna
<213>artificialsequence
<220>
<223>f7
<400>17
actaaagggaacaaaagctgggt23
<210>18
<211>27
<212>dna
<213>artificialsequence
<220>
<223>r7
<400>18
tttaaattgtttaacttgggtcaaaca27
<210>19
<211>23
<212>dna
<213>artificialsequence
<220>
<223>degfp-f
<400>19
tccaattccgcccccccccccta23
<210>20
<211>23
<212>dna
<213>artificialsequence
<220>
<223>degfp-r
<400>20
gagatctcgagctcgatgagttt23
<210>21
<211>50
<212>dna
<213>artificialsequence
<220>
<223>f8
<400>21
gaacaaaaactcatctcagaagaggatctggaattgatcacaagtttgta50
<210>22
<211>20
<212>dna
<213>artificialsequence
<220>
<223>r8
<400>22
catggtggcgaccggtagcg20
<210>23
<211>44
<212>dna
<213>artificialsequence
<220>
<223>f9
<400>23
gactacaaggacgacgatgacaaggaattgatcacaagtttgta44
<210>24
<211>20
<212>dna
<213>artificialsequence
<220>
<223>r9
<400>24
catggtggcgaccggtagcg20
<210>25
<211>45
<212>dna
<213>artificialsequence
<220>
<223>f10
<400>25
gcaggcaagccaatccctaaccctctgctgggcctggacagcacc45
<210>26
<211>20
<212>dna
<213>artificialsequence
<220>
<223>r10
<400>26
catggtggcgaccggtagcg20
<210>27
<211>57
<212>dna
<213>artificialsequence
<220>
<223>f11
<400>27
ggggacaagtttgtacaaaaaagcaggcttcaccatgaatcaggaactgctctctgt57
<210>28
<211>56
<212>dna
<213>artificialsequence
<220>
<223>r11
<400>28
ggggaccactttgtacaagaaagctgggtagtatcacttcatgtccacatcaaagt56
<210>29
<211>18
<212>dna
<213>artificialsequence
<220>
<223>m13-f
<400>29
tgtaaaacgacggccagt18
<210>30
<211>18
<212>dna
<213>artificialsequence
<220>
<223>m13-r
<400>30
caggaaacagctatgacc18
<210>31
<211>22
<212>dna
<213>artificialsequence
<220>
<223>psm1.1-f
<400>31
ctggttattgtgctgtctcatc22
<210>32
<211>22
<212>dna
<213>artificialsequence
<220>
<223>psm1.1-r
<400>32
gccacttgtgtagcgccaagtg22
- 还没有人留言评论。精彩留言会获得点赞!