本发明涉及pcr检测技术领域,特别是涉及一种检测肺炎球菌10a血清型的试剂盒及其应用。
背景技术:
肺炎链球菌是一种常见的人类致病菌,是发展中国家和发达国家各个年龄组人群获得性肺炎最常见病因,同时也是引起中耳炎、肺炎、脑膜炎和败血症等疾病的主要致病菌。肺炎球菌属于革兰氏阳性菌,呈单球或双球排列,无鞭毛,芽孢,有荚膜,且不同型别的荚膜厚度有所不同。肺炎球菌的荚膜是其毒力的必要条件,并且荚膜多糖有群/型特异性,是肺炎球菌分群/型的基础。迄今为止肺炎球菌可分为46个群,90多个血清型,引起疾病的肺炎球菌型别常因地区、年代和人群的不同而不同。
针对肺炎球菌中引起疾病的20多个主要血清型,目前都以提纯的荚膜多糖制成疫苗,并根据who规程确定疫苗所包含的的血清型,主要为1、2、3、4、5、6b、7f、8、9n、9v、10a、11a、12f、14、15b、17f、18c、19a、19f、20、22f、23f和33f,即23价肺炎球菌多糖疫苗。这23个血清型对美国、法国、中国等常见血清型的覆盖率均在85%以上,可作为全球普遍使用的疫苗。已有实验证明23价肺炎球菌多糖疫苗对控制和预防肺炎球菌感染,特别是老年人肺部感染,儿童中耳炎和脑膜炎有明显的效果。
23价肺炎球菌多糖疫苗生产用菌种的检定主要通过肺炎球菌的培养特性,革兰氏染色,奥普脱欣,胆汁溶解,生化反应等实验完成。而菌种的分型常用的方法是血清凝集和荚膜肿胀实验,并且荚膜肿胀实验也称为肺炎球菌分型的金标准。但血清凝集和荚膜肿胀实验所用标准血清费用昂贵,并且专业技术要求高,对结果的解释带有一定的主观性,成为制约血清学分型技术普及应用的巨大障碍。此外,对于多种血清型共同携带的现象,荚膜肿胀实验只能识别其中的优势血清型,而无法同时识别多种血清型。
随着肺炎球菌基因序列的不断完善和丰富以及分子生物学技术的发展,一些针对肺炎球菌血清型分型的pcr试验方法已经建立起来(吴丽娟,邹建话,刘小月,等.多重pcr检测肺炎链球菌血清型方法及临床应用,微循环学杂志,2012,22(4):43-45)。rekhapai等人针对常见的29个血清型的肺炎球菌建立了多重pcr试验方法,同时涵盖了7价多糖结合疫苗所包含的血清型(pair,gertzre,beallb.sequential.multiplexpcrapproachfordeterminingcapsularserotypesofstreptococcuspneumoniaeisolates.clinmicrobiol,2006,44(1):124-131.)。fatmafilizcoskun-ari等人建立了针对13价肺炎球菌多糖结合疫苗的多重pcr诊断方法(cooskun-arif,guldemird,durmazr.one-stepmultiplexpcrassayfordetectingstreptococcuspneumoniaserogroups/typescoveredby13-valentpneumococcalconjugatevaccine(pcv13).plosone;2012,7(12),e50406.)。国内研究者也进行了相关研究,例如李马超等为了了解成都儿童临床肺炎患者中分离的肺炎链球菌血清型/群的分布状况,也根据文献报道的引物建立了包含12个血清型的快速检测的pcr方法,可用于鉴别人群中主要流行的肺炎链球菌血清型/群(李马超,张砺,李倩,等.肺炎链球菌pcr分型技术的建立与应用.中华流行病学杂志,2010,31(3):316-320),但是只覆盖了23价肺炎球菌多糖疫苗全部血清型的52.17%。而钟天鹰等也根据文献报道,对18个常见血清型的肺炎球菌建立了多重pcr检测方法(钟天鹰,徐飞,陈倩,等.与肺炎链球菌血清型一致的pcr分型法的初步建立.临床检验杂志,2012,30(1):29-30)。但是,其建立的方法中所引用的10a型引物并不能扩增出预期大小的特异性片段,且只覆盖了23价肺炎球菌多糖疫苗所包含的23个血清型的72.26%。
综上所述,国内外报道的10a型引物并不能特异性的扩增出预期大小的片段。目前,还没有全部覆盖23价肺炎球菌多糖疫苗所包含的23个血清型的一步法多重pcr分型方法,也没有针对23价肺炎球菌多糖疫苗生产用菌种进行快速检测和分子分型的检定方法。
技术实现要素:
本发明的目的在于提供一种特异性检测肺炎球菌10a血清型的特异性引物对。
本发明的另一目的在于提供检测23价肺炎球菌多糖疫苗所包含的23个血清型的多重pcr分型方法。
本发明首先提供了一种检测肺炎球菌10a血清型的特异性引物对,其核苷酸序列如seqidno.1-2所示。
本发明提供了上述特异性引物对在检测肺炎球菌10a血清型中的应用。尤其是在检测疫苗中肺炎球菌10a血清型中的应用。
本发明提供了上述特异性引物对在肺炎球菌疫苗生产质控中的应用。
含有seqidno.1-2所示特异性引物对的试剂盒属于本发明的保护范围。
进一步第,本发明试剂盒工作程序包括以下步骤:
(1)提取待测样品的基因组dna;
(2)用seqidno.1-2所示特异性引物对,对样品dna进行pcr反应;pcr产物经电泳检测,若目的产物的片段大小为607bp,则待测样品中含有肺炎球菌10a血清型。
所述pcr反应,反应条件为:94℃预变性5min;94℃45s,55℃45s,72℃90s,30个循环;72℃延伸10min。
本发明提供了一种检测疫苗中肺炎球菌10a血清型的方法,包括以下步骤:
(1)提取待测样品的基因组dna;
(2)用seqidno.1-2所示的特异性引物对,对样品dna进行pcr反应;pcr产物经电泳检测,若目的产物的片段大小为607bp,则待测样品中含有肺炎球菌10a血清型。
本发明提供了一种检测23价肺炎球菌血清型的试剂盒,含有seqidno.1-2所示的特异性引物对,所述23价肺炎球菌血清型分别为1、2、3、4、5、6b、7f、8、9n、9v、10a、11a、12f、14、15b、17f、18c、19a、19f、20、22f、23f和33f。
进一步地,上述检测23价肺炎球菌血清型的试剂盒含有6组特异性引物组合,分别为a、b、c、d、e、f组,
其中a组含有的特异性引物对核苷酸序列分别如seqidno.3-12所示;
其中b组含有的特异性引物对核苷酸序列分别如seqidno.11-20所示;
其中c组含有的特异性引物对核苷酸序列分别如seqidno.11-12和seqidno.21-28所示;
其中d组含有的特异性引物对核苷酸序列分别如seqidno.11-12和seqidno.29-36所示;
其中e组含有的特异性引物对核苷酸序列分别如seqidno.1-2、seqidno.11-12和seqidno.37-42所示;
其中f组含有的特异性引物对核苷酸序列分别如seqidno.11-12和seqidno.43-48所示。
本发明提供一种用于检测23价肺炎球菌血清型的特异性引物组合,含有24对特异性引物对,其核苷酸序列分别如seqidno.1-48所示。
本发明提供一种针对23价肺炎球菌多糖疫苗所包含的23个血清型的多重pcr分型方法,根据pcr扩增产物片段大小的不同,将23个血清型的特异性引物,配成6组复合引物,每组均含有阳性对照引物cpsa。利用6组复合引物对23价肺炎球菌多糖疫苗生产用的菌种进多重pcr扩增,根据扩增片段大小的不同对23个血清型进行分型,对这23个血清型肺炎球菌菌种的性质和纯度进行特异性的鉴别。
本发明提供了上述特异性引物组合在肺炎球菌疫苗生产质控中的应用。
本发明的有益效果在于,本发明首先弥补了现有技术中没有检测肺炎球菌10a血清型的技术空白,基于本发明提供的检测肺炎球菌10a血清型特异性引物对,本发明提供了一种针对23价肺炎球菌多糖疫苗生产用菌种快速检测和菌株鉴定的多重pcr引物组合及基于一步法多重pcr技术的分子分型方法,该方法具有经济、便捷、快速和可靠等优点,可用于生产用菌种库的检定和生产中污染的监测,填补了该领域的空白。本发明方法操作简单便捷,只需一次pcr反应即可完成一个血清型的检测;经济性好,相对于荚膜肿胀实验费用比较低,较容易实现;本发明方法敏感性和特异性好,能排除主观因素影响,结果更加准确可靠,较生理生化的检测方法有很大的优越性。
附图说明
图1为:10a型引物特异性试验电泳图。
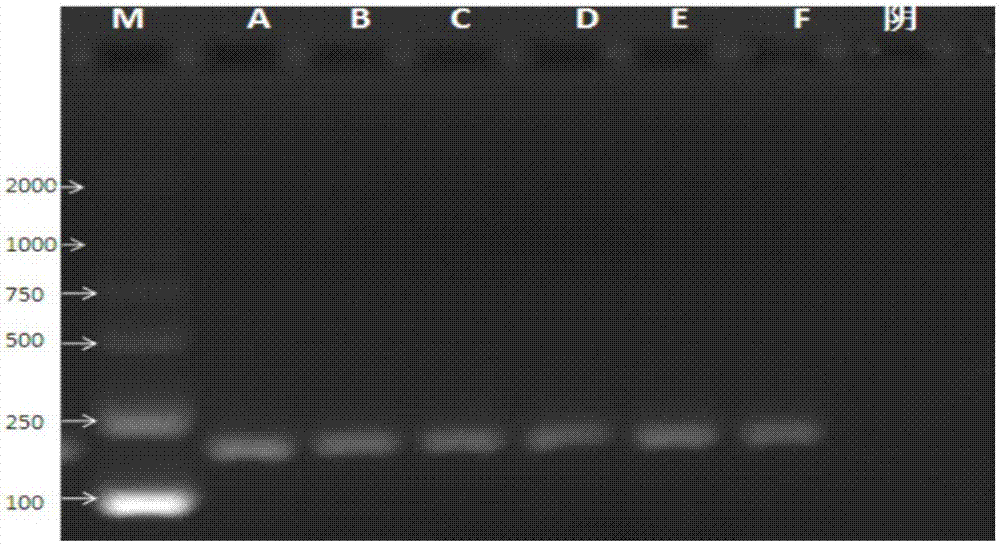
图2为:参考文献中现有10a型引物扩增电泳图,m为dnamarker:dl2000;a、b、c、d、e、f代表引物组,“阴”代表阴性对照。
图3为:针对同样的靶基因设计的10a型引物10a(1)扩增电泳图,m为dnamarker:dl2000;a、b、c、d、e、f代表引物组,“阴”代表阴性对照。
图4为:针对同样的靶基因设计的10a型引物10a(2)扩增电泳图,m为dnamarker:dl2000;a、b、c、d、e、f代表引物组,“阴”代表阴性对照。
图5为:a组引物扩增电泳图。
图6为:b组引物扩增电泳图。
图7为:c组引物扩增电泳图。
图8为:d组引物扩增电泳图。
图9为:e组引物扩增电泳图。
图10为:f组引物扩增电泳图。
具体实施方式
以下实施例进一步说明本发明的内容,但不应理解为对本发明的限制。在不背离本发明精神和实质的情况下,对本发明方法、步骤或条件所作的修改或替换,均属于本发明的范围。
若未特别指明,实施例中所用的技术手段为本领域技术人员所熟知的常规手段;若未特别指明,实施例中所用试剂均为市售。
实施例1肺炎球菌10a特异性引物的设计
肺炎球菌基因组中dexb与alia基因之间的cps基因为肺炎球菌血清型相关的基因,不同血清型的cps基因组结构和组成不同,bently等已完成90种血清型肺炎球菌的cps基因组序列的测定,并公开在网站上http://www.sanger.ac.uk(bentlysd,aanensendm,mavroidia,etal.geneticanalysisofthecapsularbiosyntheticlocusfromall90pneumococcalserotypes.plosgenet,2006,2(3):e31)。下载其中23个血清型的cps基因组序列,通过dnastar软件将10a型与其他各型基因组的cps基因组序列进行比对,针对其基因组中型特异性的区域设计一对特异性的引物,引物序列为:
10a-f:ccagtgcaatacattccaatgtcattctcg(seqidno.1)
10a-r:aaatcatttggggcatctgttatcggtgaa(seqidno.2)
使用10a型特异性引物对已制备的1、2、3、4、5、6b、7f、8、9n、9v、10a、11a、12f、14、15b、17f、18c、19a、19f、20、22f、23f、33f型dna模板进行单一pcr扩增,反应体系为25ul:dna模板3ul,2.5mmdntpmixture2ul,takaralataqdna聚合酶0.5ul,10xpcrbuffer5ul,10a型上下游引物各2ul,灭菌的纯水补足25ul。pcr扩增程序为:(1)预变性95℃,5min;(2)变性94℃,45s;(3)退火55℃45s;(4)延伸72℃90s;(5)回到(2)共30个循环;(6)72℃10min。取扩增产物以1.5%琼脂糖凝胶进行电泳,成像分析结果。
结果如图1所示:m为dnamarker:dl2000,1、2、3、4、5、6b、7f、8、9n、9v、10a、11a、12f、14、15b、17f、18c、19a、19f、20、22f、23f、33f代表23个肺炎球菌血清型。由图1可以看出只有作为阳性对照的10a型肺炎球菌扩增出大小为607bp的特异性目的条带,其余22个血清型均未见任何条带,表明10a型引物有良好的特异性,对23个血清型的肺炎球菌均无任何非特异性扩增。
现有技术中所提供的针对10a型特异性引物序列为:
10a-f:ggtgtagatttaccattagtgtcggcagac(seqidno.49)
10a-r:gaatttcttctttaagattcggatatttctc(seqidno.50)
除了seqidno.1-2,发明人针对相同的靶基因还设计了另外两对引物:
10a(1)-f:cacggctgagatgttggataatatggaaac(seqidno.51)
10a(1)-r:tgccctgattgtggaagtctaatgaagaaa(seqidno.52)
10a(2)-f:aaaggcgaaagaacacttggaacaaacagg(seqidno.53)
10a(2)-r:cctcactcctaccagaaacttgccacatcc(seqidno.54)
但经过试验后,发现这3对引物放入扩增效果均不理想,均未能扩增出预期的目的片段;如图2所示结果表明现有技术所提供的10a型特异性引物(seqidno.49-50)未能扩增出预期的目的片段;如图3所示结果表明针对同样的靶基因设计的特异性引物(seqidno.51-52)未能扩增出预期的目的片段;如图4所示结果表明针对同样的靶基因设计的特异性引物(seqidno.53-54)未能扩增出预期的目的片段。
实施例2检测肺炎球菌23价血清型的多重pcr检测体系的建立
1、多重pcr复合引物组合设计和配制
根据23对引物扩增目的片段大小的不同,将其进行优化组合,使各组内引物的扩增片段大小均相差100bp左右,电泳结果更加直观,能够直接根据扩增片段大小的不同区分出不同的血清型。然后,分别取相应的引物配制成6组复合引物a、b、c、d、e、f,同时每组复合引物中含有阳性对照引物cpsa,其引物序列为:
seqidno11:gcagtacagcagtttgttggactgacc
seqidno12:gaatattttcattatcagtcccagtc
各组引物所包含的血清型、扩增长度、各型引物终浓度等详细参数见表1:
表1
2、用复合引物组对制备好的dna模板进行多重pcr扩增
以制备好的23个血清型的肺炎球菌基因组dna为模板,用6组复合引物配制成6组pcr反应体系a、b、c、d、e、f,各组内的cpsa引物作为阳性对照,针对所有血清型均能扩增出160bp的片段;n组为阴性对照组,使用纯水作为模板,其余组分同其他各组。配制好7组pcr体系后,在同一扩增条件下进行多重pcr扩增。pcr反应体系为25ul:dna模板3ul,dntpmixture(2.5mm)2ul,takaralataqdna聚合酶0.5ul,10xpcrbuffer5ul,复合引物各2ul,灭菌的纯水补足25ul。pcr扩增程序为:(1)预变性95℃,5min;(2)变性94℃,45s;(3)退火55℃45s;(4)延伸72℃90s;(5)回到(2)共30个循环;(6)72℃10min。取pcr产物经1.5%琼脂糖凝胶电泳,成像分析结果。
结果见图5-图10:m为dnamarker:dl2000;n为阴性对照;a、b、c、d、e、f为各复合引物组;1、2、3、4、5、6b、7f、8、9n、9v、10a、11a、12f、14、15b、17f、18c、19a、19f、20、22f、23f、33f代表23个血清型肺炎球菌。
如图5所示:a组所包含的4个血清型19f、23f、20、7f分别扩增出清晰的大小不同的目的条带,19f、23f、20、7f扩增片段依次增大。
如图6所示:b组所包含的4个血清型8、2、9n、9v分别扩增出清晰的大小不同的目的条带,8、2、9n、9v扩增片段依次增大。
如图7所示:c组所包含的4个血清型1、3、15b、17f分别扩增出清晰的大小不同的目的条带,1、3、15b、17f扩增片段依次增大。
如图8所示:d组所包含的4个血清型6b、12f、19a、22f分别扩增出清晰的大小不同的目的条带,6b、12f、19a、22f扩增片段依次增大。
如图9所示:e组所包含的4个血清型14、5、11a、10a分别扩增出清晰的大小不同的目的条带,14、5、11a、10a扩增片段依次增大。
如图10所示:f组所包含的3个血清型33f、4、18c分别扩增出清晰的大小不同的目的条带,33f、4、18c扩增片段依次增大。
每个血清型的肺炎球菌均在自己相应的引物组内扩增出大小不同的目的条带,同时各组均扩增出大小为160bp的阳性对照条带,阴性对照组无条带扩增。表明该方法可以对23个血清型的肺炎球菌进行快速、准确的鉴定分型,并且能够对这23个血清型肺炎球菌菌种的性质和纯度进行检定,为肺炎球菌菌种的检定提供分子生物学依据。
实施例3对发酵罐上取样的发酵液进行多重pcr扩增
采用实施例2建立的方法对发酵罐上的发酵的23个血清型的肺炎球菌发酵液进行pcr扩增,检测在发酵过程中各型之间有没交叉污染,由于在发酵过程中,产生的一些多糖和蛋白会对pcr扩增产生一些干扰,所以在进行pcr之前,采用康维试剂公司的细菌基因组提取试剂盒对各型发酵液按照说明书进行基因组的提取,制备成纯度较高的模板后再进行pcr扩增。
以提取的各型发酵液的基因组dna为模板,用a、b、c、d、e、f组6组复合引物对每个型进行pcr扩增,n组为阴性对照。pcr反应体系为25ul:dna模板3ul,dntpmixture(2.5mm)2ul,takaralataqdna聚合酶0.5ul,10xpcrbuffer5ul,复合引物各2ul,灭菌的纯水补足25ul。pcr扩增程序为:(1)预变性95℃,5min;(2)变性94℃,45s;(3)退火55℃45s;(4)延伸72℃90s;(5)回到(2)共30个循环;(6)72℃10min。取pcr产物经1.5%琼脂糖凝胶电泳,成像分析结果。
发酵罐上的发酵的23个血清型的发酵液在经过基因组dna提取后,均在自己相应的引物组内扩增出与目的片段大小相同的条带,而其他引物组内未见非特异性的目的条带扩增。该实施例表明此发明可以对发酵罐上的肺炎球菌发酵液进行快速、准确的pcr检测,能够检测出不同血清型的肺炎球菌在发酵过程中相互之间有无交叉污染,为肺炎球菌的发酵过程提供一个分子生物学的监测手段。.
虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之作一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。
序列表
<110>北京华安科创生物技术有限公司
<120>一种检测肺炎球菌10a血清型的试剂盒
<130>khp171113852.9
<160>54
<170>patentinversion3.5
<210>1
<211>30
<212>dna
<213>人工序列
<400>1
ccagtgcaatacattccaatgtcattctcg30
<210>2
<211>30
<212>dna
<213>人工序列
<400>2
aaatcatttggggcatctgttatcggtgaa30
<210>3
<211>30
<212>dna
<213>人工序列
<400>3
cctacgggaggatataaaattatttttgag30
<210>4
<211>32
<212>dna
<213>人工序列
<400>4
caaatacaccactataggctgttgagactaac32
<210>5
<211>31
<212>dna
<213>人工序列
<400>5
gagcaagagtttttcacctgacagcgagaag31
<210>6
<211>33
<212>dna
<213>人工序列
<400>6
ctaaattcctgtaatttagctaaaactcttatc33
<210>7
<211>32
<212>dna
<213>人工序列
<400>7
gtaacagttgctgtagagggaattggcttttc32
<210>8
<211>33
<212>dna
<213>人工序列
<400>8
cacaacacctaacacacgatggctatatgattc33
<210>9
<211>31
<212>dna
<213>人工序列
<400>9
gttaagattgctgatcgattaattgatatcc31
<210>10
<211>30
<212>dna
<213>人工序列
<400>10
gagcagtcaataagatgagacgatagttag30
<210>11
<211>27
<212>dna
<213>人工序列
<400>11
gcagtacagcagtttgttggactgacc27
<210>12
<211>26
<212>dna
<213>人工序列
<400>12
gaatattttcattatcagtcccagtc26
<210>13
<211>32
<212>dna
<213>人工序列
<400>13
cttcgttagttaaaattctaaatttttctaag32
<210>14
<211>28
<212>dna
<213>人工序列
<400>14
gtcccaataccagtccttgcaacacaag28
<210>15
<211>27
<212>dna
<213>人工序列
<400>15
gaactgaataagtcagatttaatcagc27
<210>16
<211>26
<212>dna
<213>人工序列
<400>16
accaagatcctgacgggctaatcaat26
<210>17
<211>32
<212>dna
<213>人工序列
<400>17
gtcattgttacgattagtttcgatagttgagg32
<210>18
<211>32
<212>dna
<213>人工序列
<400>18
aattcaattcctaagtcctcttccataaactc32
<210>19
<211>31
<212>dna
<213>人工序列
<400>19
gatgccatgaatcaagcagtggctataaatc31
<210>20
<211>30
<212>dna
<213>人工序列
<400>20
atcctcgtgtataatttcaggtatgccacc30
<210>21
<211>33
<212>dna
<213>人工序列
<400>21
ttcgtgatgataattccaatgatcaaacaagag33
<210>22
<211>32
<212>dna
<213>人工序列
<400>22
gatgtaacaaatttgtagcgactaaggtctgc32
<210>23
<211>29
<212>dna
<213>人工序列
<400>23
ttggaattttttaattagtggcttaccta29
<210>24
<211>31
<212>dna
<213>人工序列
<400>24
catccgcttattaattgaagtaatctgaacc31
<210>25
<211>31
<212>dna
<213>人工序列
<400>25
atggtgtgatttctcctagattggaaagtag31
<210>26
<211>31
<212>dna
<213>人工序列
<400>26
cttctccaattgcttaccaagtgcaataacg31
<210>27
<211>32
<212>dna
<213>人工序列
<400>27
ctctatagaatggagtatataaactatggtta32
<210>28
<211>34
<212>dna
<213>人工序列
<400>28
ccaaagaaaatactaacattatcacaatattggc34
<210>29
<211>32
<212>dna
<213>人工序列
<400>29
gagtatagccagattatggcagttttattgtc32
<210>30
<211>32
<212>dna
<213>人工序列
<400>30
ctccagcacttgcgctggaaacaacagacaac32
<210>31
<211>32
<212>dna
<213>人工序列
<400>31
gttagtcctgttttagatttatttggtgatgt32
<210>32
<211>30
<212>dna
<213>人工序列
<400>32
gagcagtcaataagatgagacgatagttag30
<210>33
<211>25
<212>dna
<213>人工序列
<400>33
gcaacaaacggcgtgaaagtagttg25
<210>34
<211>30
<212>dna
<213>人工序列
<400>34
caagatgaatatcactaccaataacaaaac30
<210>35
<211>30
<212>dna
<213>人工序列
<400>35
aatttgtattttattcatgcctatatctgg30
<210>36
<211>29
<212>dna
<213>人工序列
<400>36
ttagcggagataatttaaaatgatgacta29
<210>37
<211>31
<212>dna
<213>人工序列
<400>37
ggacatgttcaggtgatttcccaatatagtg31
<210>38
<211>32
<212>dna
<213>人工序列
<400>38
gattatgagtgtaatttattccaacttctccc32
<210>39
<211>32
<212>dna
<213>人工序列
<400>39
atacctacacaacttctgattatgcctttgtg32
<210>40
<211>35
<212>dna
<213>人工序列
<400>40
gctcgataaacataatcaatatttgaaaaagtatg35
<210>41
<211>29
<212>dna
<213>人工序列
<400>41
cttggcgcaggtgtcagaattccctctac29
<210>42
<211>31
<212>dna
<213>人工序列
<400>42
gccaaaatactgacaaagctagaatatagcc31
<210>43
<211>32
<212>dna
<213>人工序列
<400>43
cttaatagctctcattattctttttttaagcc32
<210>44
<211>30
<212>dna
<213>人工序列
<400>44
ttatctgtaaaccatatcagcatctgaaac30
<210>45
<211>31
<212>dna
<213>人工序列
<400>45
ctgttacttgttctggactctcgataattgg31
<210>46
<211>31
<212>dna
<213>人工序列
<400>46
gcccactcctgttaaaatcctacccgcattg31
<210>47
<211>27
<212>dna
<213>人工序列
<400>47
gaaggcaatcaatgtgattgtgtcgcg27
<210>48
<211>31
<212>dna
<213>人工序列
<400>48
cttcaaaatgaagattatagtacccttctac31
<210>49
<211>30
<212>dna
<213>人工序列
<400>49
ggtgtagatttaccattagtgtcggcagac30
<210>50
<211>31
<212>dna
<213>人工序列
<400>50
gaatttcttctttaagattcggatatttctc31
<210>51
<211>30
<212>dna
<213>人工序列
<400>51
cacggctgagatgttggataatatggaaac30
<210>52
<211>30
<212>dna
<213>人工序列
<400>52
tgccctgattgtggaagtctaatgaagaaa30
<210>53
<211>30
<212>dna
<213>人工序列
<400>53
aaaggcgaaagaacacttggaacaaacagg30
<210>54
<211>30
<212>dna
<213>人工序列
<400>54
cctcactcctaccagaaacttgccacatcc30