DNA条形码、引物、试剂盒、方法和应用与流程
本发明属于物种和菌株鉴定领域,具体涉及dna条形码、引物、试剂盒、方法和应用。
背景技术:
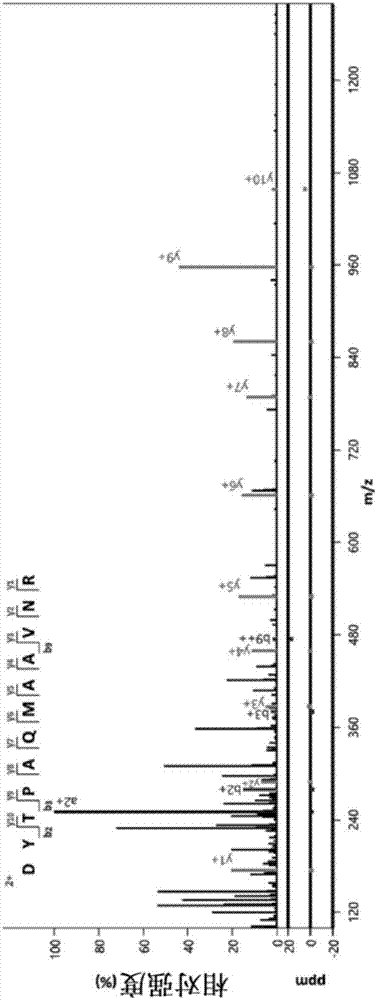
:普洱茶是一种具有云南地理标识的后发酵茶,其采用大叶种晒青毛茶作为原料,经过一系列工艺制作而成。传统的普洱茶制作过程为:采摘的新鲜茶叶经揉捻、晒青、除杂、潮水、渥堆、晾干、筛分、压制成型、包装出厂。在普洱茶的生产中,渥堆发酵过程是普洱茶品质形成的主要因素,在此过程中,茶叶中诸如茶多酚、咖啡因以及一些多糖物质等内含成分发生了巨大变化,成就了普洱茶的特殊风味、口感、品质以及各种保健功效。传统的普洱生产中,湿热的环境激活茶叶本身所含的酶,把茶叶中所含的一部分内含成分转化成微生物能利用的物质,微生物大量滋生,产生丰富的胞内酶和胞外酶,催化茶叶中包含的成分发生一系列转变,再加上湿热等其他因素,逐渐形成不同品质的普洱茶。不同的产地,因为其微生物群落结构的不同,风味及品质也各具特色。普洱茶除了其独特的风味和文化外,其诸如减肥、降血糖血脂、预防和改善心血管疾病、抗衰老、抗癌、消炎、助消化以及养胃等保健功效也备受人们关注。近年来,普洱茶越来越受欢迎,逐渐增加的市场需求拉动了普洱茶产业的发展,促进了云南地区经济增长。然而,普洱茶也面临着一些困境,例如产品质量不稳定、存在一定安全隐患、生产周期长、劳动力投入过高、微生物数量超标和螨虫滋生等。随着人们生活水平的提高,消费者对食品的卫生、安全等问题日益重视。近几年食品质量安全事件频发,茶叶质量的安全问题也日益突出,除了农药的残留问题之外,渥堆发酵过程中微生物也有可能成为影响茶叶质量的重要因素。目前多数厂家的普洱茶生产仍然是半自然人工渥堆的经验式发酵,虽然渥堆过程中以食腺嘌呤节孢酵母(blastobotrysadeninivorans)等优势共性微生物为主的群落相对稳定,但产品的稳定性还存在较大的提升空间。生产过程中也不可避免的存在着一定的安全隐患,为了进一步获得消费者的青睐和市场的认可、突破外贸壁垒、提升普洱茶企业的市场竞争力,必须实现普洱茶生产工艺的人工可控化、清洁化以及高效化,并不断开发新产品、延伸产业链。要做到这些,就必须革新技术,发明一系列安全、清洁、高效、人工可控自动化的普洱茶新工艺,才能确保普洱茶产业的健康发展,给国家和人民带来长远利益。随着普洱茶科研的进一步深入,越来越多的人们开始了人工接种发酵普洱茶的探索,目前已经有许多菌株应用于普洱茶人工可控发酵。近年由于市场需求加大,许多规模较小的厂家对普洱茶缺乏系统深入的研究,大肆滥用许多他人专利进行牟利,比如,按照一些接种发酵普洱茶的专利方法,采用一些菌种进行普洱茶发酵等一系列方法,进行普洱茶加工生产等经营活动,大大损害了一些具有相关专利的普洱茶生产企业的经济利益。为确保普洱茶发酵微生物种质资源得到有效保护,防止滥用普洱茶发酵微生物的侵权行为发生,解决在普洱茶人工可控发酵过程中菌种侵权时难以举证的难题,开发对普洱茶发酵用菌株进行快速精准鉴别的方法势在必行,亟需建立其分子鉴定方法,开发通过dna条形码技术准确鉴别与区分与其相似菌株的方法,以达到通过形态特征与分子数据分析相结合的方法来解决工业生产菌株鉴别和鉴定问题,为普洱茶有控工业发酵工艺开发和资源保护提供理论依据,促进普洱茶产业健康繁荣发展。技术实现要素:为了克服形态学鉴定用于普洱茶发酵生产的食腺嘌呤节孢酵母菌株所存在的不足,本发明提供了一种用于鉴别食腺嘌呤节孢酵母菌株的dna条形码、引物、试剂盒、方法和应用,从而对食腺嘌呤节孢酵母tmcc70007菌株进行快速鉴别和区分,可为普洱茶新发酵工艺提供快速评估,防止发酵过程中其他杂菌的干扰,以及为普洱茶人工可控发酵工艺及菌种非法滥用提供举证方法和依据。为了实现上述目的,本发明采用如下技术方案:(1)一种用于鉴别食腺嘌呤节孢酵母菌株的dna条形码,该dna条形码来源于食腺嘌呤节孢酵母tmcc70007菌株的基因组,并且包含选自如seqidno.1所示的dna序列中的至少500bp的序列,并且该dna条形码的长度为500bp-3500bp,优选为500bp-2200bp,更优选为500bp-1500bp。(2)根据(1)所述的dna条形码,其核苷酸序列如seqidno.1、seqidno.2、seqidno.4或seqidno.7所示。(3)一种用于扩增根据(1)所述的dna条形码的引物对。(4)根据(3)所述的引物对,其正向引物的核苷酸序列与食腺嘌呤节孢酵母tmcc70007菌株基因组中的这样的序列相同:该序列为从所述tmcc70007菌株基因组中如seqidno.1所示的核苷酸序列的第1位的上游1000bp到如seqidno.1所示的核苷酸序列的第535位的区域中的序列,并且该正向引物的长度为20-30bp;其反向引物与所述tmcc70007菌株基因组中的这样的序列反向互补:该序列为从所述tmcc70007菌株基因组中如seqidno.1所示的核苷酸序列的第501位到如seqidno.1所示的核苷酸序列的最后一位的下游1000bp的区域中的序列,并且该反向引物的长度为20-30bp。(5)根据(4)所述的引物对,其正向引物和反向引物的核苷酸序列分别如下所示:正向引物:5’-cgaccgagaacgacaggaat-3’;反向引物:5’-acacgtcaacaggcgatcaa-3’。(6)一种用于鉴别食腺嘌呤节孢酵母菌株的试剂盒,包含根据(3)所述的引物对。(7)一种用于鉴别食腺嘌呤节孢酵母菌株的方法,包括以下步骤:a)提供待测菌株的基因组dna;b)以步骤a)所述的基因组dna为模板,利用根据(3)所述的引物对进行pcr扩增,得到pcr产物;c)电泳检测pcr产物,如果没有目标条带,则判定待测菌株不是食腺嘌呤节孢酵母tmcc70007菌株,如果有目标条带,则进行步骤d);d)对所得pcr产物进行测序,得到待测核苷酸序列;将所述待测核苷酸序列与权利要求1所述的dna条形码的核苷酸序列进行同源性比对,若同源性在97%以上,则判定待测菌株是食腺嘌呤节孢酵母tmcc70007菌株。(8)根据(1)所述的dna条形码在鉴别食腺嘌呤节孢酵母菌株中的应用。(9)根据(3)所述的引物对在鉴别食腺嘌呤节孢酵母菌株中的应用。(10)根据(6)所述的试剂盒在鉴别食腺嘌呤节孢酵母菌株中的应用。本发明与现有技术相比具有以下优点和积极效果:1、本发明采用蛋白质基因组学技术从食腺嘌呤节孢酵母tmcc70007菌株的基因组中发现了一个漏注释编码蛋白,并且发现编码该蛋白的基因的开放阅读框(seqidno.1)为食腺嘌呤节孢酵母tmcc70007菌株的基因组所特有。本发明进一步通过仔细的研究和比较分析,将该特有的dna序列开发为了dna条形码,作为鉴别普洱茶工业发酵生产菌株食腺嘌呤节孢酵母tmcc70007的有效工具。该条形码序列能实现食腺嘌呤节孢酵母种内菌株的快速准确鉴别与区分。2、与现有技术相比,本发明采用蛋白质基因组学技术发现的dna条形码的特异性更好。3、本发明进一步发现,相比于其他基因,该漏注释基因中的序列(例如seqidno.1)具有通用、易扩增、易比对的特点,其在食腺嘌呤节孢酵母种内不同菌株之间差异明显。4、本发明建立了普洱茶工业发酵生产菌株食腺嘌呤节孢酵母tmcc70007的标准基因序列和样品鉴别方法,相比于传统的形态学鉴定方法,显著提高了鉴定效率。该方法对于样品的完整程度要求低,鉴别指标能够量化,这对于及时判定普洱茶及其种质资源提供了有效的依据。此外,进一步加入了形态学混淆种并可以运用基于聚类分析的系统发育树法设立鉴定规则进行鉴定,其鉴定的可靠性和准确性也大大优于常规的分子鉴定方法,弥补了该类普洱发酵茶生产菌株基于dna条形码技术的菌株鉴定的空白。附图说明图1示出新鉴定肽段dytpaqmaavnr的质谱谱图。图2示出化学合成肽段dytpaqmaavnr的质谱图与原鉴定肽段质谱图的对比;所述原鉴定肽段为本发明经过质谱分析并鉴定得到的肽段;图的上方为原鉴定肽段的质谱图,下方为合成肽段的质谱图。图3示出肽段所在区域蛋白质编码框的mrna序列及其编码的蛋白质序列的对应图,灰色背景部分为所鉴定的新肽段。图4示出编码新鉴定hsp40蛋白的orf所转录的mrna序列及其编码的蛋白质序列的对应图,灰色背景部分为所鉴定的新肽段。图5a示出tmcc70007的细胞全蛋白sds-page分离图谱,加粗显示的条带为hsp40蛋白所在位置;图5b示出hsp40蛋白分子量验证图。图5b中横坐标表示蛋白质分子量标记(marker)的不同分子量以10为底的对数值,纵坐标表示蛋白质分子量标记中各分子量蛋白在sds-page电泳分离中的迁移量相对于sds-page总迁移量的比值。理论上hsp40蛋白的分子量大小mw=37.7kda,lg(mw)=lg(37.7)≈1.58,hsp40蛋白分子对数对应图5b横坐标1.58,其迁移率对应迁移率曲线为x=1.58时的y轴上的值。图6示出条形码序列seqidno.1通过ncbi-blastn得到的与之具有同源性的序列比对图,在图6下方的三条短的灰色段显示出3条同源性序列。图7示出食腺嘌呤节孢酵母的tmcc70007条形码序列seqidno.1与食腺嘌呤节孢酵母ls3菌株同源序列的对比图;图8示出本发明一个实施方案中用于扩增seqidno.1的引物的位置,序列中有下划线的区域即为引物所在区域,加粗字体的atg和taa即为起始和终止位点,所扩增的序列为seqidno.7。图9示出对利用本发明的引物得到的待测菌株pcr扩增产物进行琼脂糖凝胶电泳的结果。图10示出seqidno.1序列与待测菌株pcr扩增序列的对比图。具体实施方式以下通过具体实施方式的描述并参照附图对本发明作进一步说明,但这并非是对本发明的限制,本领域技术人员根据本发明的基本思想,可以做出各种修改或改进,但是只要不脱离本发明的基本思想,均在本发明的范围之内。本发明采用蛋白质基因组学技术从食腺嘌呤节孢酵母tmcc70007菌株的基因组中发现了漏注释的编码蛋白,由于该蛋白具有hsp40家族的结构域,因此将其称为hsp40蛋白。本发明由该新发现的蛋白序列得到了tmcc70007菌株基因组中的对应基因序列,将其称为hsp40基因,并且发现在食腺嘌呤节孢酵母tmcc70007菌株中编码该蛋白的基因序列(seqidno.1)相对于现有数据库中已经报道的同源基因具有很好的特异性,该特异性dna序列可以用于开发鉴别普洱茶工业发酵生产菌株食腺嘌呤节孢酵母tmcc70007的dna条形码。与现有技术相比,本发明通过上述方法获得的dna条形码的特异性更高。考虑到dna条形码应当具有合适的长度和足够的菌株间特异性,本发明进一步通过仔细的研究和比较分析,基于上述特有的dna序列获得了可准确、有效地鉴别食腺嘌呤节孢酵母tmcc70007菌株的dna条形码。由此,本发明的一个方面提供了一种用于鉴别食腺嘌呤节孢酵母菌株的dna条形码,该dna条形码来源于食腺嘌呤节孢酵母tmcc70007菌株的基因组,并且包含选自如seqidno.1所示的dna序列中的至少500bp的序列,并且该dna条形码的长度为500bp-3500bp,优选为500bp-2200bp,更优选为500bp-1500bp。本发明的条形码序列能实现食腺嘌呤节孢酵母种内菌株的快速准确鉴别与区分。seqidno.1的长度为1035bp,由于seqidno.1为食腺嘌呤节孢酵母tmcc70007菌株基因组所特有,因此该序列本身即可作为鉴别食腺嘌呤节孢酵母tmcc70007菌株的dna条形码。另外,通过理论分析和实验验证,本发明证明包含该序列、长度更长的dna条码的特异性更加得到保证。当dna条形码的长度过长时(例如大于3500bp),对于扩增操作而言较不理想。另一方面,通常,dna条形码的长度不小于500bp时,也能够满足易扩增、易比对的操作要求;本发明通过理论分析和实验验证,证明选自如seqidno.1所示的dna序列中的至少500bp的序列也能够实现食腺嘌呤节孢酵母种内菌株的快速准确鉴别与区分。在本发明的优选的实施方案中,所述dna条形码序列如seqidno.1、seqidno.2、seqidno.4或seqidno.7所示。在本文中,术语“漏注释基因”是指物种完成基因组测序以后,采用基因预测软件(例如genemark、augustus、glimmer等)未能将该基因预测出来,这些基因一般表达量不高、在特定的条件下才表达,所以在研究中难以被发现。术语“dna条形码技术(dnabarcoding)”是指用基因组内一段标准的、短的dna片段来鉴定物种的一项分子鉴定新技术,其可以快速、准确的进行物种鉴定。术语“六框翻译”是蛋白质组学和基因组学中的已知术语,简述其原理为,dna编码蛋白质时,采用三联体密码子编码蛋白质,给定一条dna序列,有3种编码可能性,加上其互补链上的3种编码可能,共有6种编码可能性(+1,+2,+3,-3,-2,-1)。本发明的另一个实施方案提供了一种用于扩增本发明所述的dna条形码的引物对。优选地,其正向引物的核苷酸序列与食腺嘌呤节孢酵母tmcc70007菌株基因组中的这样的序列相同:该序列为从所述tmcc70007菌株基因组中如seqidno.1所示的核苷酸序列的第1位的上游1000bp到如seqidno.1所示的核苷酸序列的第535位的区域中的序列,并且该正向引物的长度为20-30bp;其反向引物与所述tmcc70007菌株基因组中的这样的序列反向互补:该序列为从所述tmcc70007菌株基因组中如seqidno.1所示的核苷酸序列的第501位到如seqidno.1所示的核苷酸序列的最后一位的下游1000bp的区域中的序列,并且该反向引物的长度为20-30bp。在更优选的实施方案中,所述正向引物和反向引物的核苷酸序列分别如下所示:正向引物hsp40-f:5’-cgaccgagaacgacaggaat-3’(seqidno.5);反向引物hsp40-r:5’-acacgtcaacaggcgatcaa-3’(seqidno.6)。本发明的引物能实现对所述dna条形码序列的特异性扩增。本发明还提供了一种用于鉴别食腺嘌呤节孢酵母菌株的试剂盒,包含根据本发明所述的引物对。在另一实施方案中,所述试剂盒还包含根据本发明所述的dna条形码。所述dna条形码可存在于记录介质上。该记录介质例如为光盘。所述试剂盒还可以包含用于实验操作的任何工具和试剂。本发明的又一方面提供了一种用于鉴别食腺嘌呤节孢酵母菌株的方法,包括以下步骤:a)提供待测菌株的基因组dna;b)以步骤a)所述的基因组dna为模板,利用根据权利要求3所述的引物对进行pcr扩增,得到pcr产物;c)电泳检测pcr产物,如果没有目标条带,则判定待测菌株不是食腺嘌呤节孢酵母tmcc70007菌株,如果有目标条带,则进行步骤d);d)对所得pcr产物进行测序,得到待测核苷酸序列;将所述待测核苷酸序列与权利要求1所述的dna条形码的核苷酸序列进行同源性比对,若同源性在97%以上(优选99%以上),则判定待测菌株是食腺嘌呤节孢酵母tmcc70007菌株。在本发明的一个具体实施方案中,所述pcr扩增的程序为:1)94℃-96℃预变性8分钟;2)94℃-96℃变性45秒,55℃-57℃退火45秒,72℃延伸1分15秒,其中该程序2)进行30-35个循环;3)72℃延伸10分钟。在本发明的又一实施方案中,所述方法还包括将测序结果得到的待测序列与本发明的dna条形码进行聚类分析(例如系统发育树),如果待测序列与dna条形码聚类在一起,则确定待测菌株为食腺嘌呤节孢酵母菌株tmcc70007。在本发明一个具体的实施方案中,利用本发明的引物对对提取自待鉴别的菌株的基因组dna进行pcr扩增,然后进行琼脂糖凝胶电泳检测。基于检测是否存在pcr产物来鉴别菌株:如果待鉴定的菌株没有扩增出相应的目标条带,则说明该菌株不是tmcc70007;如果扩增出相应的目标条带,则证明该菌株可能是tmcc70007。为了进一步进行鉴别,对pcr产物进行测序,将dna测序结果与dna条形码序列进行同源性比对,获得序列间的相似性(即同源性),如果序列同源性小于97%,则判定待测菌株不是食腺嘌呤节孢酵母菌株tmcc70007。如果序列同源性大于或等于97%(优选99%以上),则判定待测菌株是食腺嘌呤节孢酵母菌株tmcc70007。如果进行聚类分析,例如系统发育树,则将所述dna条形码和各待鉴定菌株的dna测序结果(即待测序列)一起应用mega6或paup软件构建nj系统发育树。如果待鉴定菌株的待测序列与食腺嘌呤节孢酵母tmcc70007的dna条形码聚类在一起,则鉴定为食腺嘌呤节孢酵母tmcc70007。在此所使用的术语“聚类”是指经系统发育树分析后,处于同一个分支,且进化距离相同。本发明还提供了本发明所述的dna条形码在鉴别食腺嘌呤节孢酵母菌株中的应用。本发明还提供了本发明所述的引物对在鉴别食腺嘌呤节孢酵母菌株中的应用。实施例下面结合具体的实施例对本发明做进一步说明。实施例中所用方法在未具体说明的情况下,均采用常规方法和已知工具。实施例1:hsp40基因和dna条形码的获得该实施例中所用的蛋白质组和基因组分析方法、以及质谱方法均为本领域的已知方法,故具体操作过程不再赘述。1、利用高覆盖蛋白质组技术,采用pfind和panno软件对普洱茶工业发酵菌株食腺嘌呤节孢酵母tmcc70007进行了高覆盖蛋白质组学研究,并对其基因组进行了注释编码基因的验证。具体而言,为了发现新的蛋白编码区,利用系统蛋白质基因组学中的六框翻译(sixframetranslation)策略,获得了食腺嘌呤节孢酵母tmcc70007基因组数据的六框翻译数据库,穷举了基因组的6种编码可能(+1,+2,+3,-1,-2,-3),并且将蛋白序列称为“六框翻译蛋白序列”,对应的核酸序列称为“六框翻译核酸序列”。通常而言,六框翻译核酸序列是从一个终止子到下一个终止子的序列,在本文中,也将其称为“蛋白编码框”。利用这个六框翻译数据库,采用pfind和panno软件与tmcc70007菌株的全蛋白质谱数据进行比对,从而进行新肽段和新蛋白质的发现和鉴定。经鉴定,发现了一条现有技术中食腺嘌呤节孢酵母tmcc70007注释基因组中未发现的肽段dytpaqmaavnr,其质谱图如图1所示。手工检查质谱谱图结果显示,检测到了肽段dytpaqmaavnr的二级质谱谱图(ms2)的几乎所有y离子序列,匹配好,信号强,结果较为可信。为进一步确证这个鉴定结果,按照新鉴定肽段dytpaqmaavnr的氨基酸序列化学合成了该肽段,并利用上述dytpaqmaavnr的质谱分析条件产生了合成肽段的二级谱图,参见图2。接下来,对合成肽段产生的高能量碰撞ms2进行了核实,一级母离子和二级子离子均符合理论值,表明合成的肽段序列正确(图2)。在此基础上,人工检查了根据大规模蛋白质组数据鉴定到的新肽段序列合成肽段的ms2和大规模鉴定新肽段谱图,两者几乎完全一致,以子离子相似性获得的cosin值高达0.96,由此证明从普洱茶工业发酵菌食腺嘌呤节孢酵母tmcc70007鉴定到的新肽段正确无误。2、根据新肽段所在的位置,以前一个终止密码子和后一个终止密码子包括的区域为界,得到六框翻译核酸序列(蛋白编码框)seqidno.2。seqidno.2序列对应的mrna及其所编码的氨基酸序列如图3所示。3、为了进一步确定编码该蛋白的基因的编码起始位点和终止位点,将上述蛋白编码框向上游和下游分别扩展1000bp,采用genemark.hmm进行基因预测,参考物种选用粟酒裂殖酵母(schizosaccharomycespombe)。在该区域预测到了该基因的存在,其编码框与上述蛋白编码框序列一致,确定了其编码起始和终止位点,其orf序列为seqidno.1。该基因转录的mrna序列和其翻译的蛋白的氨基酸序列的对应关系如图4所示。肽段dytpaqmaavnr坐落在该基因的上游。该基因的核苷酸序列共1035bp,共编码344个氨基酸,理论分子量为37.67kda,其理论编码的氨基酸序列如seqidno.3所示。4、为了进一步确定所鉴定的序列的正确性,在ypd培养基中培养tmcc70007菌株并提取其中的总蛋白,进行sds-page分离,电泳胶经考马斯亮蓝染色,根据蛋白质组样品制备时的分子量特征验证分子量,见图5。另外,按条带共切成25个胶条进行胶内消化和质谱分析,其中hsp40蛋白在第17个胶条的质谱数据中被鉴定到了,与预测的分子量大小匹配。基于氨基酸序列计算的理论分子量为37.7kda,理论分子量与该蛋白所属的胶条所在sds-page中的位置是一致的。结果显示鉴定到的hsp40的实验分子量约为33.6±1.9kda,与理论分子量基本相符,证实了所鉴定的蛋白的正确性。5、以该基因理论编码产物的氨基酸序列进行blastp分析,其序列具有dnaj超级家族类似的结构域,dnaj/hsp40(heatshockprotein40,热休克蛋白40)是一个保守的蛋白家族,与蛋白质翻译、折叠、反折叠、转移和降解密切相关。所鉴定的hsp40蛋白序列与酵母(sugiyamaellalignohabitans)cbs10342t菌株的hlj1p蛋白(登录号为anb12940.1)相似度最高,为62%,序列覆盖度为99%。blastp结果表明所检测到的漏注释蛋白可能为hsp40同源蛋白,其序列具有一定的保守性。6、将所鉴定的hsp40基因的orf序列(即seqidno.1)进行ncbi-blastn分析,结果见图6。blastn结果总结在表2中。结果表明seqidno.1在ncbi数据库中没有同源性较高的序列,在1035个碱基中,仅有2%-3%的近30bp左右的碱基序列比较保守,这表明在食腺嘌呤节孢酵母中检测到的漏注释基因hsp40具有序列特异性,虽然hsp40蛋白的氨基酸序列部分区段较为保守,但其dna同源性却很低。这佐证了该基因的orf序列可以用于开发普洱茶发酵菌株食腺嘌呤节孢酵母tmcc70007的dna条形码。表2与seqidno.1具有较高相似度的核酸序列列表7、进一步选择了seqidno.1上游1000bp到下游1000bp之间的区域(如seqidno.4所示)。将seqidno.4序列进行ncbi-blastn分析,检索nucleotidecollection(nr/nt)数据库,发现其仅有1%的序列(38bp)与小马岛猬dnaj(hsp40)同源蛋白b亚科成员12(dnajb12)mrna具有同源性。比对结果总结在表3中。根据上述结果,可以认为seqidno.4可作为鉴别食腺嘌呤节孢酵母菌株tmcc70007的dna条形码序列。此外,进一步对seqidno.4序列进行ncbi-blastn分析,检索whole-genomeshotguncontigs(wgs)数据库中食腺嘌呤节孢酵母(分类学id:409370)的鸟枪法全基因组序列。结果发现,seqidno.4与食腺嘌呤节孢酵母ls3全基因序列arad1b染色体的部分序列具有90%的同源性(约300个碱基不同),查询序列覆盖度为99%,比对结果总结在表4中。这说明尽管tmcc70007和ls3菌株为同一物种,但它们的基因组中的同源序列具有足够的区分度,seqidno.4可以作为dna条形码序列。综上所述,seqidno.1上游1000bp到下游1000bp之间的序列(seqidno.4)可以作为特异性鉴别食腺嘌呤节孢酵母菌株tmcc70007的dna条形码。表3表48、目前,食腺嘌呤节孢酵母物种中,仅有食腺嘌呤节孢酵母ls3菌株的基因组已经被报道(kunzeetal.biotechnologyforbiofuels2014,7:66)。经local-blastn分析后,发现该基因序列seqidno.1在ls3菌株基因组中存在同源序列。经dnaman分析后,两条序列的同源性为91.70%,尽管tmcc70007与ls3菌株为同一个物种(即食腺嘌呤节孢酵母,blastobotrysadeninivorans),但在该dna条形码序列的1035个碱基中,有84个位点是不一样的,结果如图7所示。这进一步证明了利用该dna条形码序列可以有效地区别tmcc70007菌株与种内的其他菌株。实施例2.利用dna条形码鉴别菌株依据待测样品的扩增结果和与食腺嘌呤节孢酵母tmcc70007菌株seqidno.1的序列同源性,来判定待测样品是否为普洱茶工业应用菌株食腺嘌呤节孢酵母tmcc70007。(1)基于seqidno.1,采用ncbi引物设计工具在该基因两端设计pcr引物,扩增产物须包括基因的起始和终止位点,得到正反引物序列分别为hsp40-f:5‘-cgaccgagaacgacaggaat-3’;hsp40-r:5’-acacgtcaacaggcgatcaa-3’。引物的位置见图8。所扩增的序列为seqidno.7。(2)菌株来源表5选用的相关菌株信息样品编号学名菌株号生境1食腺嘌呤节孢酵母tmcc70007普洱茶渥堆,中国2食腺嘌呤节孢酵母cbs8244t土壤,荷兰3食腺嘌呤节孢酵母cbs7350窖贮玉米饲料,荷兰4食腺嘌呤节孢酵母cbs7370土壤,南非5食腺嘌呤节孢酵母cbs8335粘土,意大利6blastobotrysraffinosifermentanscbs6800t未知(3)分别提取菌株dna:采用omegae.z.n.a.tm的yeastdna试剂盒提取酵母基因组dna,并用灭菌的去离子水将样品的dna浓度稀释到0.5μg/μl。(4)扩增dna片段,进行聚合酶链式(pcr)反应,所用引物序列分别为:正向引物序列:hsp40-f:5‘-cgaccgagaacgacaggaat-3’;反向引物序列:hsp40-r:5’-acacgtcaacaggcgatcaa-3’。pcr反应体系为50μl,pcr试剂为thermoscientifictm的taqdnapolymerase(重组):ddh2o37.7μl、mgcl25μl,dntps4μl,正向引物1μl,反向引物1μl,taqdna聚合酶0.3μl、dna模板1μl,不含染料。扩增程序为:94℃预变性8分钟;接下来94℃变性45秒,56℃退火45秒,72℃延伸1分15秒,一共进行32-35个循环;最后72℃延伸10分钟。(5)扩增产物检测:用1.0%的琼脂糖凝胶、1×tbe电泳液电泳检测,使用dnamarker检测pcr片段大小。若待测菌株没有扩增条带,说明该菌株不是食腺嘌呤节孢酵母tmcc70007;若出现明显清晰的条带,且无杂带,送生物测序公司进行dna片段测序。(6)该引物仅能在食腺嘌呤节孢酵母tmcc70007中实现扩增,在同物种的食腺嘌呤节孢酵母cbs8244t、cbs7350、cbs7370、cbs8335和blastobotrysraffinosifermentanscbs6800t中则不能实现扩增。结果如图9示。该pcr引物的理论扩增片段为1418bp,实际扩增结果和预期相符。为了进一步验证扩增的dna的序列,进行了测序和同源序列比较。(7)对于有条带的序列的测序结果,首先用软件chromas检查测序后得到的序列峰图质量,确定峰图质量达到数据分析的要求之后,用dnastar软件包中的seqman进行正反向序列的拼接。将测序结果进行手工校对、序列拼接,如果待测菌株dna片段与食腺嘌呤节孢酵母tmcc70007标准基因序列的同源性在97%以上,即可判断所述的待测菌株可能为食腺嘌呤节孢酵母tmcc70007菌株。例如,经过dnaman,将菌株tmcc70007测序得到序列与dna条形码序列seqidno.1比对后完全一致,进一步证明了这种方法的可靠性。结果如图10所示。sequencelisting<110>勐海茶业有限责任公司<120>dna条形码、引物、试剂盒、方法和应用<130>fi-163691-59:52/c<160>7<170>patentinversion3.5<210>1<211>1035<212>dna<213>食腺嘌呤节孢酵母(blastobotrysadeninivorans)<400>1atgtctacgtcatcagaacgctcttctacaaaggctgagagccgagatcacaagcagggg60cgccagaacagggactatacccctgctcagatggcagcggtaaacagagtgcgaaagtgc120agggtaactgattattatgctattctagatattgaatcgccttcatcagaggcggaaatt180cgcaaagcctatcgtaagttggcactgattatgcatcccgacaagaacggggcccctgga240gcagacgaagcttttaagatggtgtcaaaggcattccaggttctgtctgatagcgacaag300aagaggatattcgatcaaacaggagcggatcccgactctagaggaggtggtggaggtatg360ggatcgtttgctcgtggagcaggaggaccaggcatgcagtttggagggggaggagatatt420tctcctgaagacctgttcaacatgttctttggtggcggtggtggaagcccgttccaggcc480cagtttgggggctttggtgggccaggcattagagtacacacatttggcgggggctctccg540ttttctgcgtttggtaacactgctggcgcccagcgcagacgggctgctgctgctcaacaa600gaagacttttcactgcgaaacttagtgcaaatgctaccgcttctattactattcggactg660ccttggctgttatccctgttcggggactcgcctggtgcaggggccgtaccaagctttcga720ttcaaccaaagaccccccttcatggaggagaggctcacatcccggtacagcattccgtat780tttgtcaatccccaggatgtgcagaatttggccgaccgaaagctggctcaattggaccgc840caggcagaagtatcctacatccagtctatgagacgtcagtgcaacactgaagtggaatac900aagcagcagaagatttcggatgcccgaggatggttctttgtcgacgaagaggcttacgac960gcagcgcgtaagataccccttcctagttgcgagtttttagatggactggggatcacgcat1020gagagggtcttataa1035<210>2<211>1071<212>dna<213>食腺嘌呤节孢酵母(blastobotrysadeninivorans)<400>2tagaaagaaccaaagaaaaaaagttttcgaataggcatgtctacgtcatcagaacgctct60tctacaaaggctgagagccgagatcacaagcaggggcgccagaacagggactatacccct120gctcagatggcagcggtaaacagagtgcgaaagtgcagggtaactgattattatgctatt180ctagatattgaatcgccttcatcagaggcggaaattcgcaaagcctatcgtaagttggca240ctgattatgcatcccgacaagaacggggcccctggagcagacgaagcttttaagatggtg300tcaaaggcattccaggttctgtctgatagcgacaagaagaggatattcgatcaaacagga360gcggatcccgactctagaggaggtggtggaggtatgggatcgtttgctcgtggagcagga420ggaccaggcatgcagtttggagggggaggagatatttctcctgaagacctgttcaacatg480ttctttggtggcggtggtggaagcccgttccaggcccagtttgggggctttggtgggcca540ggcattagagtacacacatttggcgggggctctccgttttctgcgtttggtaacactgct600ggcgcccagcgcagacgggctgctgctgctcaacaagaagacttttcactgcgaaactta660gtgcaaatgctaccgcttctattactattcggactgccttggctgttatccctgttcggg720gactcgcctggtgcaggggccgtaccaagctttcgattcaaccaaagaccccccttcatg780gaggagaggctcacatcccggtacagcattccgtattttgtcaatccccaggatgtgcag840aatttggccgaccgaaagctggctcaattggaccgccaggcagaagtatcctacatccag900tctatgagacgtcagtgcaacactgaagtggaatacaagcagcagaagatttcggatgcc960cgaggatggttctttgtcgacgaagaggcttacgacgcagcgcgtaagataccccttcct1020agttgcgagtttttagatggactggggatcacgcatgagagggtcttataa1071<210>3<211>344<212>prt<213>食腺嘌呤节孢酵母(blastobotrysadeninivorans)<400>3metserthrsersergluargserserthrlysalagluserargasp151015hislysglnglyargglnasnargasptyrthrproalaglnmetala202530alavalasnargvalarglyscysargvalthrasptyrtyralaile354045leuaspilegluserproserserglualagluilearglysalatyr505560arglysleualaleuilemethisproasplysasnglyalaprogly65707580alaaspglualaphelysmetvalserlysalapheglnvalleuser859095aspserasplyslysargilepheaspglnthrglyalaaspproasp100105110serargglyglyglyglyglymetglyserphealaargglyalagly115120125glyproglymetglnpheglyglyglyglyaspileserprogluasp130135140leupheasnmetphepheglyglyglyglyglyserpropheglnala145150155160glnpheglyglypheglyglyproglyileargvalhisthrphegly165170175glyglyserpropheseralapheglyasnthralaglyalaglnarg180185190argargalaalaalaalaglnglngluasppheserleuargasnleu195200205valglnmetleuproleuleuleuleupheglyleuprotrpleuleu210215220serleupheglyaspserproglyalaglyalavalproserphearg225230235240pheasnglnargproprophemetglugluargleuthrserargtyr245250255serileprotyrphevalasnproglnaspvalglnasnleualaasp260265270arglysleualaglnleuaspargglnalagluvalsertyrilegln275280285sermetargargglncysasnthrgluvalglutyrlysglnglnlys290295300ileseraspalaargglytrpphephevalaspgluglualatyrasp305310315320alaalaarglysileproleuprosercysglupheleuaspglyleu325330335glyilethrhisgluargvalleu340<210>4<211>3066<212>dna<213>食腺嘌呤节孢酵母(blastobotrysadeninivorans)<400>4cccagtgaaattgactgtaccattctcaaatgctgagcgaatgcggccaatatggcgagg60atcagacacaacaatttttaaaaagggcgactttgcttgtccattatacccccacagaga120ctctttcatgcaaatttcaatatgatccacttggcccgagaaagcagaatccaaggcgtc180aatgagaccaggaatgttttcttctcgaaatcccacaggtgctggaacataaaagtaatg240aaggaaattagtaacgttgcagcacaccgtgtttccttcatccgtgacaccaaatagacg300aatcactgactctccttgcatcattgcttcttcagcatcaatctgttgaaagttgatatc360tcgctcataagcagtaaaatctgcgagcttaggtctggtccacttttgatctgtttctgc420tccgcctgaatttgttagttacaatggacttgaagcctacaattcacttaccgccttcca480tttggttgatctcttgagtcaaccgagtcaaatcttgctcgaaggaagtgccctcgcttt540tagctcgctttgcagggccatcatcaatagctgggcgtttcacgccctgatctaaggctt600caggtgccattttagtttccacttcgcagtcctacttcaatgcaagtgacgcgataacta660gttcaagacgcgttttccggatctaaattacatatcttccagcctcaatcggtgtggctg720tgagccccatacggtgcggtgcggttccagaatattttgcattggggatcttcccgcccc780tgactgtcaatagaggacacaaaagcatagaaaccggtgatccacgattttattaaaggc840tgggtctgacctacagctggcaaacaacggggcatcttggtaatattggacgtccagact900gagcgatcataacacaaatagctcgttttcactcccttggaagcatttacagacgaccga960gaacgacaggaatagccagtgctgtgcacggatcaggttagaaagaaccaaagaaaaaaa1020gttttcgaataggcatgtctacgtcatcagaacgctcttctacaaaggctgagagccgag1080atcacaagcaggggcgccagaacagggactatacccctgctcagatggcagcggtaaaca1140gagtgcgaaagtgcagggtaactgattattatgctattctagatattgaatcgccttcat1200cagaggcggaaattcgcaaagcctatcgtaagttggcactgattatgcatcccgacaaga1260acggggcccctggagcagacgaagcttttaagatggtgtcaaaggcattccaggttctgt1320ctgatagcgacaagaagaggatattcgatcaaacaggagcggatcccgactctagaggag1380gtggtggaggtatgggatcgtttgctcgtggagcaggaggaccaggcatgcagtttggag1440ggggaggagatatttctcctgaagacctgttcaacatgttctttggtggcggtggtggaa1500gcccgttccaggcccagtttgggggctttggtgggccaggcattagagtacacacatttg1560gcgggggctctccgttttctgcgtttggtaacactgctggcgcccagcgcagacgggctg1620ctgctgctcaacaagaagacttttcactgcgaaacttagtgcaaatgctaccgcttctat1680tactattcggactgccttggctgttatccctgttcggggactcgcctggtgcaggggccg1740taccaagctttcgattcaaccaaagaccccccttcatggaggagaggctcacatcccggt1800acagcattccgtattttgtcaatccccaggatgtgcagaatttggccgaccgaaagctgg1860ctcaattggaccgccaggcagaagtatcctacatccagtctatgagacgtcagtgcaaca1920ctgaagtggaatacaagcagcagaagatttcggatgcccgaggatggttctttgtcgacg1980aagaggcttacgacgcagcgcgtaagataccccttcctagttgcgagtttttagatggac2040tggggatcacgcatgagagggtcttataaaactgcatactaacgaagcctaaaattttgt2100ctcatagggtgttttctagaagttaatttttttccccgtttatttagacaatgtgtttac2160tcgacttagaagacctagactaacacgctctttttttgcaatgttttgcatgatattttg2220cggggccgggtttgtttgtgtctattatgacatcgtaccgcctatgatcaacagttactg2280actgcaggtggtggtgcgtcaagcaaggaggaggaggggccgagggatccagccactacc2340cgatggcttgcttgatcgcctgttgacgtgttcaattgacaggttattcccggcgcggtg2400gcgcttcttagctgtgtattgtccgtcaaaaattaaactctaacccccttttacgggctc2460tcggtggggccctggtctttattttgtttcgcatcttgtcatcgtaagatcgtcaacgtc2520gtgacgatcttgcccgcaaccggtccaagggggtgctatatttgcatccagccacatcaa2580gaagagtgcgcatataaagtttggaatatccttgaaagtggacaaggcagccggtggatt2640ttgctcagtagaatgaaaagtgtaggattagcgctgctgagcttggtgtcggtgtcgtgg2700gcacagacattcccgttgaggtcggtaaagggcaccggggtttccaggtttctggggcag2760tctaaacagctcaagacttccgaatccagtataaaggcaacaagcgacggagacagcgac2820aaggatggaacagagtccgtggagacttttagtagtgatgtcgcgtacgtgatggatgtg2880tcgctggggtctgatgagcagaactttactctgattgttgacactggttcgtactataca2940tgggtgtacgggtccaagtgtacggagagcgtgtgccaaaagcaccaacagttcaacccc3000acaaactcgacatcattccacaacacgtcggctacttttgacatctcctttacttcagga3060agcatt3066<210>5<211>20<212>dna<213>人工序列<400>5cgaccgagaacgacaggaat20<210>6<211>20<212>dna<213>人工序列<400>6acacgtcaacaggcgatcaa20<210>7<211>1418<212>dna<213>食腺嘌呤节孢酵母(blastobotrysadeninivorans)<400>7cgaccgagaacgacaggaatagccagtgctgtgcacggatcaggttagaaagaaccaaag60aaaaaaagttttcgaataggcatgtctacgtcatcagaacgctcttctacaaaggctgag120agccgagatcacaagcaggggcgccagaacagggactatacccctgctcagatggcagcg180gtaaacagagtgcgaaagtgcagggtaactgattattatgctattctagatattgaatcg240ccttcatcagaggcggaaattcgcaaagcctatcgtaagttggcactgattatgcatccc300gacaagaacggggcccctggagcagacgaagcttttaagatggtgtcaaaggcattccag360gttctgtctgatagcgacaagaagaggatattcgatcaaacaggagcggatcccgactct420agaggaggtggtggaggtatgggatcgtttgctcgtggagcaggaggaccaggcatgcag480tttggagggggaggagatatttctcctgaagacctgttcaacatgttctttggtggcggt540ggtggaagcccgttccaggcccagtttgggggctttggtgggccaggcattagagtacac600acatttggcgggggctctccgttttctgcgtttggtaacactgctggcgcccagcgcaga660cgggctgctgctgctcaacaagaagacttttcactgcgaaacttagtgcaaatgctaccg720cttctattactattcggactgccttggctgttatccctgttcggggactcgcctggtgca780ggggccgtaccaagctttcgattcaaccaaagaccccccttcatggaggagaggctcaca840tcccggtacagcattccgtattttgtcaatccccaggatgtgcagaatttggccgaccga900aagctggctcaattggaccgccaggcagaagtatcctacatccagtctatgagacgtcag960tgcaacactgaagtggaatacaagcagcagaagatttcggatgcccgaggatggttcttt1020gtcgacgaagaggcttacgacgcagcgcgtaagataccccttcctagttgcgagttttta1080gatggactggggatcacgcatgagagggtcttataaaactgcatactaacgaagcctaaa1140attttgtctcatagggtgttttctagaagttaatttttttccccgtttatttagacaatg1200tgtttactcgacttagaagacctagactaacacgctctttttttgcaatgttttgcatga1260tattttgcggggccgggtttgtttgtgtctattatgacatcgtaccgcctatgatcaaca1320gttactgactgcaggtggtggtgcgtcaagcaaggaggaggaggggccgagggatccagc1380cactacccgatggcttgcttgatcgcctgttgacgtgt1418当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1