本发明涉及基因工程
技术领域:
,具体地说,涉及oscol15基因在调控水稻抽穗期中的应用。
背景技术:
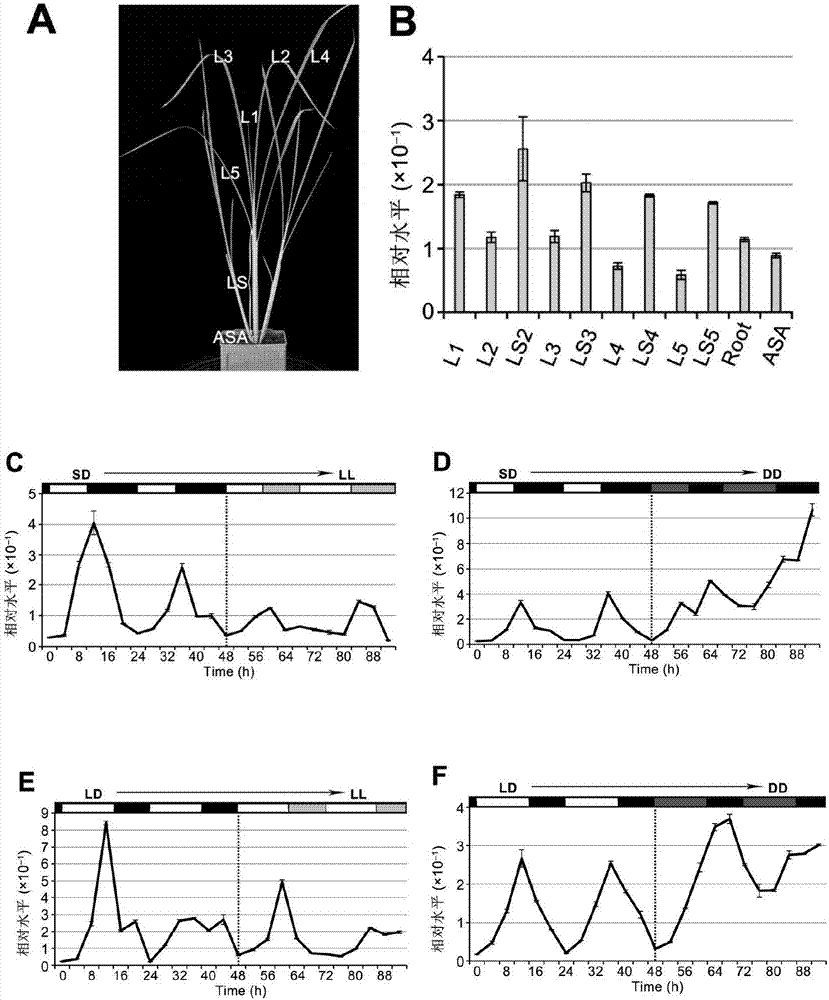
:控制开花时间(作物中称为抽穗期)是农作物繁殖成功的关键一步,同时也是作物地区适应性和产量潜力的重要因素(hori等,2016,theor.appl.genet.129:2241-2252)。精确的开花转变是由环境(日长和温度)和内源信号触发的。光周期是监测植物在同一地区相对稳定的重要环境因子,而其他环境因子则逐年变化。根据光周期的类型,植物通常分为三类:在长日照下促进开花的长日照植物,在短日照下促进开花的短日植物和不受光周期调节的日中性植物。光周期开花的分子机制在拟南芥(长日照植物)和水稻(短日照植物)中已广泛研究(song等,2015,plantbiol.66:441-464;shrestha等,2014,ann.bot.114:1445-1458)。在拟南芥中,gi-co-ft作为核心光周期开花通路在长日照下起作用而在短日照条件下无作用,该通路与水稻的osgi-hd1-hd3a在进化上保守(song等,2015,plantbiol.66:441-464)。在短日照条件下,hd1通过激活hd3a表达促进抽穗,而在长日照条件下,通过抑制hd3a表达延迟抽穗(yano等,2000,plantcell12:2473-2484;hayama等,2003,nature422:719-722)。这种hd1转换活性响应光周期的分子机制最近得到了部分解释(du等,2017,molplant10:948-961;zhu等,2017,j.exp.bot.68:553-568)。此外,除了osgi-hd1-hd3a通路,水稻还有一个进化独特的ghd7-ehd1-hd3a/rft1通路,因为拟南芥缺乏ghd7和ehd1的同源基因(doi等,2004,genedev.18:926-936;xue等,2008,nat.genet.40:761-767;song等,2015,plantbiol.66:441-464)。最近报道了多个激活或抑制ehd1转录调控的开花时间基因(hori等,2016,theor.appl.genet.129:2241-2252)。ghd7编码一个类constans蛋白,在长日照条件下通过抑制ehd1和成花素基因的表达延迟开花(xue等,2008,nat.genet.40:761-767)。最近的研究也表明ghd7-hdl蛋白形成了一个复合体绑定于ehd1启动子上抑制其表达(nemoto等,2016,plantj.86:221-233)。类constans蛋白也叫b-box(bbx)蛋白,属于一类锌指转录因子家族,它包含一个或两个位于氨基末端附近的可能参与蛋白-蛋白互作的bbx结构域,以及位于羧基末端的可能参与蛋白质定位的cct结构域(griffiths等,2003,plantphysiol.131:1855-1867;huang等,2012,plosone7:e48242)。以往的研究表明,类constans基因在拟南芥、水稻、小麦、大麦等多个物种中参与光周期开花调控(valverde,2011,j.exp.bot.62:2453-2463;gangappa和botto,2014,trendsplantsci.19:460-470)。在水稻中,有超过16个类constans基因,根据bbx结构域的不同可以分为四组(griffiths等,2003,plantphysiol.131:1855-1867)。其中已克隆了八个基因,包括第i组:hd1,osco3,oscol4;第ii组:oscol10,oscol16;第iii组:dth2,oscol13;第iv组:ghd7(yano等,2000,plantcell12:2473-2484;kim等,2008,planta228:355-365;lee等,2010,plantj.63:18-30;tan等,2016,plantcellphysiol.57:798-812;wu等,2017,plantsci.260:60-69;wu等,2013,proc.natl.acad.sci.usa110:2775-2780;sheng等,2016,plantmol.biol.92:209-222;xue等,2008,nat.genet.40:761-767)。本发明研究oscol15在水稻光周期开花调控中的作用,属于第iii组类constans基因。该基因在短日照和长日照条件下均抑制抽穗。技术实现要素:本发明的目的是提供oscol15基因在调控水稻抽穗期中的应用。为了实现本发明目的,本发明提供oscol15基因在调控水稻抽穗期中的应用,oscol15基因的cdna序列为:i)seqidno:1所示的核苷酸序列;或ii)seqidno:1所示的核苷酸序列经取代、缺失和/或增加一个或多个核苷酸且表达相同功能蛋白质的核苷酸序列;或iii)在严格条件下与seqidno:1所示序列杂交且表达相同功能蛋白质的核苷酸序列,所述严格条件为在含o.1%sds的0.1×sspe或含0.1%sds的0.1×ssc溶液中,在65℃下杂交,并用该溶液洗膜;或iv)与i)、ii)或iii)的核苷酸序列具有90%以上同源性且表达相同功能蛋白质的核苷酸序列。发明人将本发明克隆的控制水稻抽穗期的基因命名为oscol15,该基因序列来自水稻品种日本晴,其cdna序列大小为1891bp,编码区序列(cds)如seqidn0:2所示。该基因编码具有488个氨基酸的b-box/cct锌指蛋白(seqidn0:3),包含一个bbx结构域和一个cct结构域,属于类constans转录因子基因家族。进一步研究发现oscol15基因通过上调ghd7和下调ehd2来抑制ehd1、hd3a和rft1的表达,最终延迟水稻抽穗。前述的应用,通过在水稻中过表达oscol15基因,来延迟水稻抽穗期。本发明还提供oscol15基因在水稻品种改良中的应用。本发明还提供一种转基因水稻植株的构建方法,采用农杆菌介导的方法,将携带有oscol15基因cdna序列的重组表达载体转入水稻愈伤组织中,转化后的材料经过共培养-筛选-分化-生根-转基因苗的锻炼和移栽,筛选转基因水稻植株。在本发明的一个具体实施方式中,通过将oscol15基因构建到载体pcambia2300上,用所得重组载体转化水稻,筛选阳性转基因水稻植株。具体方法如下:利用引物oscol15-ox-f(seqidno:4)和oscol15-ox-r(seqidno:5),以水稻品种日本晴cdna为模板进行pcr扩增得到如seqidno:1所示的oscol15全长cdna序列,然后通过同源重组将该片段重组至pcambia2300载体的xmai位点,通过农杆菌eha105介导的水稻遗传转化,转入水稻品种日本晴中。携带有所述目的基因的表达载体可通过使用ti质粒、植物病毒载体、直接dna转化、微注射、电穿孔等常规生物技术方法导入植物细胞中(weissbach,1988,methodforplantmolecularbiologyviii,academypress,newyork,411-463;geiserson和corey,1998,plantmolecularbiology,2ndedition)。本发明还提供利用rnai技术沉默水稻oscol15基因的方法,以水稻品种日本晴cdna为模板,设计引物(seqidno:6-13),扩增出大小261bp包含oscol15基因人工mirna序列的核酸片段,通过同源重组将该核酸片段重组至pcambia2300的xmai位点,然后转化水稻。本发明进一步提供基于crispr/cas9技术敲除水稻oscol15基因的方法,其中,sgrna作用于oscol15基因靶位点的核苷酸序列为:5′-gtgtcgcagtccgcgcac-3′(seqidno:18)。本发明首次揭示了oscol15基因的生物学功能,通过亚细胞定位实验发现oscol15-gfp融合蛋白定位在水稻叶片原生质体的细胞核中。转录自激活实验显示oscol15基因具有很强的转录自激活活性,随后的缺失分析显示位于bbx结构域和cct结构域之间的区域对转录活性是必须的。通过组织表达研究发现,oscol15基因在幼嫩的叶片中表达量较高。节律表达模式分析发现oscol15基因呈现明显的昼夜节律表达。通过在水稻品种日本晴中过表达oscol15基因,发现转基因植株出现明显的晚抽穗表型,该基因在海南短日照和杭州长日照条件下均抑制开花。实时定量pcr实验发现oscol15基因位于ghd7和ehd2的上游,通过促进ghd7和抑制ehd2的表达最终抑制抽穗。附图说明图1为本发明实施例1中oscol15蛋白的序列比对,核定位和转录自激活分析。其中,a为oscol15和atcol15的蛋白序列比对。b-e为oscol15的亚细胞定位。f为oscol15的转录自激活实验和缺失分析,其中pgbkt7空载体用于阴性对照,-t和-tha分别表示sd/-trp和sd/-trp/-his/-ade。图2为本发明实施例2中oscol15基因的时间和空间表达模式分析。其中,a表示长日照条件下50天时的野生型植株。l,叶片(l1-l5);ls,叶鞘;asa,茎尖。b为通过rt-qpcr的方法分析oscol15基因在各组织中的表达水平。c-f表示通过rt-qpcr的方法分析oscol15从短日照到持续光照(c),从短日照到持续黑暗(d),从长日照到持续光照(e),从长日照到持续黑暗(f)条件下的节律表达模式,其中白色和黑色的方框分别表示光期和暗期。浅灰色表示持续光期中主观上的暗期,深灰色表示持续暗期中主观上的光期。平均值±sd。图3为本发明实施例3中oscol15基因的过表达,rnai和crispr/cas9基因敲除植株的表型。a表示oscol15过表达植株(左边)和野生型(右边)在自然长日照条件下的表型。b和c为oscol15过表达t1植株(b)和oscol15-rnait1植株(c)在自然短日照和自然长日照条件下的抽穗期。d为oscol15通过crispr/cas9基因敲除的植株在短日照和长日照条件下的抽穗期,其中(+)和(-)表示转基因阳性和阴性株。p1指双尾t检测的p值,p2指威尔科克森符号秩检验的p值。实验中检测的植株数在柱中显示,平均值±sem。e和f指oscol15过表达和野生型植株在短日照(e)和长日照(f)条件下的出叶速率。平均值±sd(n=30)。图4为本发明实施例4中在短日照和长日照条件下,hd1(a),ehd1(b),hd3a(c),rft1(d),osmads14(e),osmads15(f),ghd7(g),和ehd2(h)在oscol15过表达以及野生型植株中的表达水平,其中白色和黑色的方框分别表示光期和暗期。平均值±sd。图5为本发明实施例4中在短日照和长日照条件下,oscol15在开花调节因子通过crispr/cas9产生的基因敲除系和野生型植株中的表达水平。其中,hd1(a),ehd1(b),hd3a(c),rft1(d),osmads14(e),osmads15(f),ghd7(g),ehd2(h),ehd4(i),dth8(j),osco14(k),phyb(l),se5(m),ehd3(n)和hd16(0)。平均值±sd。图6为本发明实施例4中在短日照和长日照条件下,ehd4(a),dth8(b),oscol4(c),phyb(d),se5(e),oscol10(f),oscol13(g),ehd3(h),hd16(i),hd17(j)和oscol16(k)在oscol15过表达以及野生型植株中的表达水平。其中,白色和黑色的方框分别表示光期和暗期。平均值±sd。具体实施方式以下实施例用于说明本发明,但不用来限制本发明的范围。若未特别指明,实施例均按照常规实验条件,如sambrook等分子克隆实验手册(sambrookj&russelldw,2001,molecularcloning:alaboratorymanual),或按照制造厂商说明书建议的条件。实施例1oscol15基因是具有转录自激活活性的转录因子研究发现拟南芥atcol15和athap3及athap5互作,并且athap3a抑制开花(wenkel等,2006,plantcell,18:2971-2984)。另外,atcol15的表达受节律钟调控(tindall,2016,universityofliverpool,thesis),然而,未有研究表明atcol15是否与抽穗期相关。因此,本发明试图研究并确认atcol15在水稻中同源基因的功能。通过序列比对,发现atcol15的水稻同源基因是oso(loc_os08g42440),蛋白序列比对显示oscol15的bbx和cct结构域和atcol15分别有58%和74%的同源率(图1a),随后将oso命名为oscol15。为了确定oscol15是否为水稻转录因子,本发明进行亚细胞定位实验。本实施例中构建了一个亚细胞定位载体,构建过程如下:用引物oscol15-gfp-bamhi-f(seqidn0:14)和oscol15-gfp-bamhi-r(seqidno:15),以水稻品种日本晴cdna为模板进行pcr扩增,不包含终止密码子,然后用同源重组把该片段重组至pan580载体的bamhi位点,最终形成oscol15-gfp融合载体。水稻叶片原生质体细胞的瞬时表达实验显示oscol15-gfp融合蛋白定位在细胞核中(图1b-e)。为了进一步分析oscol15是否具有转录自激活活性,本发明利用pgbkt7载体构建了oscol15的cds和各种结构域缺失构建。以oscol15的cdna(seqidno:1)序列为模板,用表1引物扩增oscol15cds和各种缺失片段,通过同源重组把这些片段分别重组至pgbkt7的ecori位点,构建5个载体:bd-oscol15、bd-δbbx、bd-δmiddle、bd-δcct和bd-δbbx/cct。空载体和5个载体均转化入酵母ah109菌株中,随后分别在-trp和-trp/-his/-ade的sd培养基上培养。结果显示,bd-oscol15融合蛋白在-trp/-his/-ade的sd培养基上长势很好,说明oscol15具有很强的转录自激活活性(图1f)。缺失分析实验表明位于bbx和cct结构域中间的区域对于oscol15的转录活性是必须的(图1f)。表1构建oscol15转录自激活载体的引物实施例2oscol15基因表现为节律表达为了研究oscol15基因的表达模式,本发明通过实时荧光定量pcr(rt-qpcr)的方法检测oscol15基因在各种发育中组织器官的表达水平(图2a),利用引物qrt-oscol15-f(seqidno:16)和qrt-oscol15-r(seqidno:17)。结果显示oscol15基因在幼嫩组织中表达量更高(图2b),该结果与前人的研究关于开花调节因子在发育中的叶片中表达量较高相一致(han等,2015,plantcellenviron.38:2527-2540)。另外,我们利用rt-qpcr检测oscol15基因在短日照和长日照条件下的节律表达模式,结果显示oscol15基因表现明显的节律表达模式(图2c-f)。在短日照条件下,oscol15基因的转录在光期开始后即开始增高,在进入暗期后两小时到达峰值,随后开始降低,到光期后降至谷底,然后进入新的循环(图2c和d);在长日照条件下,oscol15基因的转录在光期开始后即开始增高,在进入暗期前两小时到达峰值,随后开始降低,到光期后降至谷底,然后进入新的循环(图2e和f)。其次,当植株转入持续的光照条件时,oscol15基因的节律振幅降低(图2c和e);而当植株转入持续的黑暗条件时,oscol15基因的节律振幅迅速增加(图2d和f)。该结果表明oscol15基因的转录受节律钟调控,并且可能受黑暗处理的诱导。实施例3oscol15基因是一个开花抑制因子上述研究表明oscol15基因表现为节律表达并且受节律钟调控,因为很多开花时间调节因子表现为节律表达,因此本发明研究oscol15基因是否参与水稻开花时间调控。本发明构建了pact1::oscol15过表达载体并转入野生型日本晴中,产生oscol15过表达家系,同时种植在杭州自然长日照和海南自然短日照条件下。载体构建方法如下:利用引物oscol15-ox-f(seqidno:4)和oscol15-ox-r(seqidno:5),以水稻品种日本晴cdna为模板进行pcr扩增得到oscol15的全长cdna序列(seqidno:1)。然后通过同源重组将该片段重组至pcambia2300载体的xmai位点,通过农杆菌eha105介导的水稻遗传转化,转入水稻品种日本晴中。经过愈伤组织诱导、继代、预培养、侵染、共培养、筛选具有潮霉素抗性的愈伤组织、分化、生根、炼苗移载,得到转基因植株。农杆菌介导的粳稻遗传转化体系主要应用hiei等人报道的方法(hiei等,1996,plantj.6:271-282),并在此基础上稍作改进。结果发现,在海南自然短日照和杭州自然长日照条件下,oscol15转基因阳性植株均比转基因阴性植株延迟抽穗两周左右(图3a和b)。本发明同时也创建了oscol15-rnai植株进一步验证oscol15的功能。载体构建方法如下:通过表2引物,使用warthmann等报道的方法(warthmann等,2008,plosone3:e1289),设计引物(seqidno:6-13),扩增出大小261bp包含oscol15基因人工mirna序列的核酸片段,通过同源重组把该片段重组至pcambia2300的xmai位点。值得注意的是,转基因阳性植株和转基因阴性植株的抽穗期相似(图3c)。此外,本发明还创建了oscol15-crispr植株。载体构建方法如下:利用表2引物,扩增48bp包含oscol15特异的sgrna:cas9靶序列的片段,sgrna识别位点的核苷酸序列为:5′-gtgtcgcagtccgcgcac-3′,通过同源重组把该片段重组至pcas9-sgrna载体的aari位点。后续的数据显示oscol15-crispr纯合基因敲除株系和野生型植株的抽穗期几乎相同(图3d)。这些结果表明oscol15在两种光照条件下均抑制开花,并且可能和其他的开花调节因子功能冗余。此外,本发明发现在短日照和长日照条件下,oscol15过表达植株和野生型植株的叶片出叶速度没有显著差异(图3e和f),这表明oscol15不影响水稻的生长速率。表2构建oscol15-rnai和oscol15-crispr载体的引物名称正向引物(5′-3′)反向引物(5′-3′)oscol15-rnai-1tcggatcccagcagcagccacagcaaatgtaggctttatgattaagagtactgctgctgctacagccoscol15-rnai-2cttaggcattaagattaagagtattcctgctgctaggctgtcggtaccgctgctgatgctgatgccatoscol15-rnai-3agtactcttaatcataaagcctacaggagattcagtttgaaatactcttaatcttaatgcctaagagaggcaaaagtgaaoscol15-rnai4agaagaggtacccgggtcggatcccagcagcagccacagcaaactagaggatccccgggtcggtaccgctgctgatgctgatgccatoscol15-crispragatgatccgtggcagtgtcgcagtccgcgcacgttttagagctatgcgcatagctctaaaacgtgcgcggactgcgacactgccacggatcatct实施例4oscol15基因位于ghd7和ehd2的上游为了进一步研究oscol15在水稻光周期开花调控中的作用,在短日照和长日照条件下,首先检测开花整合基因hd1和ehd1在oscol15过表达植株和野生型植株中的转录水平。结果显示hd1的节律表达水平在过表达和野生型植株中相似(图4a),而ehd1的转录水平在过表达植株中显著降低(图4b)。oscol15过表达也导致hd3a、rft1osmads14和osmads15的表达降低(图4c-f),这些基因位于hd1和ehd1的下游(tsuji等,2011,curr.opin.plantbiol.14:45-52)。此外,在这些开花时间调节因子的基因敲除植株和野生型植株中检测oscol15的表达也没有发现显著的差异(图5a-f)。这些结果表明oscol15是ehd1的上游抑制基因并且独立于hd1。随后又检测了ghd7、ehd2、ehd3、ehd4、dth8、phyb、se5、hd16、hd17、oscol4、oscol10、oscol13和oscol16的表达水平,这些基因属于ehd1的上游调控因子。本发明的数据表明,在过表达植株中,ghd7的转录水平大大提高,ehd2的转录水平显著降低(图4g和h),同时oscol15对ghd7和ehd2也没有影响(图5g和h),表明oscol15是ghd7的上游激活因子和ehd2的上游抑制因子。过表达oscol15不影响其他ehd1调节因子的转录水平,如ehd4、dth8、phyb、se5、oscol4、oscol10和oscol13(图6a-g)。此外,本发明还发现ghd7的调节因子如ehd3、hd16、hd17和oscol16的转录水平在过表达植株中也没有受到影响(图6h-k)。值得注意的是,oscol15没有影响这些ehd1和ghd7的调节因子(图5i-o)。这些结果表明,oscol15通过控制ghd7、ehd2及其下游调节因子来抑制开花。本发明所用rt-qpcr引物序列见表3。表3rt-qpcr引物(5′-3′)名称正向引物反向引物ubqgctccgtggcggtatcatcggcagttgacagccctaghd1ggcgtcagtgcttacacagatttccagcaggtgtcaggattctehd1cctacagtgattatggcttcagtgctgccaaatgttgctchd3agctcactatcatcatccagcatgccttgctcagctatttaattgcataarft1tgacctagattcaaagtctaatcctttgccggccatgtcaaattaataacosmads14gcaatgggaccagacacaacctgctacatcctctatcctttcgosmads15ccctaccctacaggctacatataggaagcactaggtacgtgctgaghd7gcttgaacccaaacacggctcatctcggcataggcttehd2gcactccgactggaaggcgcagaaggccctgtgtgtcehd4cagccagcggaatcatcacccaaatccatcagacctactcctdth8caggagtgcgtgtcggagttggtcgtcgccgttgatggtoscol4acaatagccacgaggatagcgcgatggccttgattccgtphybctcatcttcaaggaatctgaggcctgctagaacaagcattcacse5aggactcccaagcttttatcctccagaatacgagaacgacoscol10cgccctcgcttcgatcccatccctctcgccgccggtcaoscol13gagttcacttctgggcatggtgtcagtgcaatcggttgacatgaehd3gaccacctcgtcacctacaaggagtgtccctccagctaatcchd16gggaagcccagcaactcaagcccatcttcattgcctttthd17tgtcgccccttcgtcaaggtcttttccccagctcattoscol16atggcggaagtttgatctgaatgcagtttcgacgagttg虽然,上文中已经用一般性说明及具体实施方案对本发明作了详尽的描述,但在本发明基础上,可以对之做一些修改或改进,这对本领域技术人员而言是显而易见的。因此,在不偏离本发明精神的基础上所做的这些修改或改进,均属于本发明要求保护的范围。参考文献1、doi,k.,izawa,t.,fuse,t.,yamanouchi,u.,kubo,t.,shimatani,z.,yano,m.,andyoshimura,a.(2004).ehd1,ab-typeresponseregulatorinrice,confersshort-daypromotionoffloweringandcontrolsft-likegeneexpressionindependentlyofhd1.genes.dev.18,926-936.2、du,a.,tian,w.,wei,m.,yan,w.,he,h.,zhou,d.,huang,x.,li,s.,andouyang,x.(2017).thedth8-hd1modulemediatesday-length-dependentregulationofriceflowering.mol.plant10,948-961.3、gangappa,s.n.,andbotto,j.f.(2014).thebbxfamilyofplanttranscriptionfactors.trendsplantsci.19,460-470.4、geisersonandcorey(1998).plantmolecularbiology,2ndedition.5、griffiths,s.,dunford,r.p.,coupland,g,andlaurie,d.a.(2003).theevolutionofconstans-likegenefamiliesinbarley,rice,andarabidopsis.plantphysiol.131,1855-1867.6、han,s.h.,yoo,s.c.,lee,b.d.,an,g,andpaek,n.c.(2015).riceflavin-binding,kelchrepeat,f-box1(osfkf1)promotesfloweringindependentofphotoperiod.plantcellenviron.38,2527-2540.7、hayama,r.,yokoi,s.,tamaki,s.,yano,m.,andshimamoto,k.(2003).adaptationofphotoperiodiccontrolpathwaysproducesshort-dayfloweringinrice.nature422,719-722.8、hiei,y.,ohta,s.,komari,t.,andkumashiro,t.(1994).efficienttransformationofrice(oryzasatival.)mediatedbyagrobacteriumandsequenceanalysisoftheboundariesofthet-dna.plantj.6,271-282.9、hori,k.,matsubara,k.,andyano,m.(2016).geneticcontroloffloweringtimeinrice:integrationofmendeliangeneticsandgenomics.theor.appl.genet.129,2241-2252.10、huang,j.,zhao,x.,weng,x.,wang,l.,andxie,w.(2012).thericeb-boxzincfingergenefamily:genomicidentification,characterization,expressionprofilinganddiurnalanalysis.plosone7,e48242.11、kim,s.k.,yun,c.h.,lee,j.h.,jang,y.h.,park,h.y.,andkim,j.k.(2008).osco3,aconstans-likegene,controlsfloweringbynegativelyregulatingtheexpressionofft-likegenesundersdconditionsinrice.planta228.355-365.12、lee,y.s.,jeong,d.h.,lee,d.y.,yi,j.,ryu,c.h.,kim,s.l.,jeong,h.j.,choi,s.c.,jin,p.,yang,j.,cho,l.h.,choi,h.,andan,g(2010).oscol4isaconstitutivefloweringrepressorupstreamofehd1anddownstreamofosphyb.plantj.63,18-30.13、nemoto,y.,nonoue,y.,yano,m.,andizawa,t.(2016).hd1,aconstansorthologinrice,functionsasanehd1repressorthroughinteractionwithmonocot-specificcct-domainproteinghd7.plantj.86,221-233.14、sambrook,j.,andrussell,d.w.(2001).molecularcloning:alaboratorymanual.15、sheng,p.,wu,f.,tan,j.,zhang,h.,ma,w.,chen,l.,wang,j.,wang,j.,zhu,s.,guo,x.,wang,j.,zhang,x.,cheng,z.,bao,y.,wu,c.,liu,x.,andwan,j.(2016).aconstans-liketranscriptionalactivator,oscol13,functionsasanegativeregulatoroffloweringdownstreamofosphybandupstreamofehd1inrice.plantmol.biol.92,209-222.16、shrestha,r.,gomez-ariza,j.,brambilla,v.,andfornara,f.(2014).molecularcontrolofseasonalfloweringinrice,arabidopsisandtemperatecereals.ann.bot.114,1445-1458.17、song,y.h.,shim,j.s.,kinmonth-schultz,h.a.,andimaizumi,t.(2015).photoperiodicflowering:timemeasurementmechanismsinleaves.annu.rev.plantbiol.66,441-464.18、tan,j.,jin,m.,wang,j.,wu,f.,sheng,p.,cheng,z.,wang,j.,zheng,x.,chen,l.,wang,m.,zhu,s.,guo,x.,zhang,x.,liu,x.,wang,c.,wang,h.,wu,c.,andwan,j.(2016).oscol10,aconstans-likegene,functionsasafloweringtimerepressordownstreamofghd7inrice.plantcellphysiol.57,798-812.19、tindall,a.j.(2016).identification&characterisationoftranscriptionfactorsaffectingthecircadiansystemofarabidopsisthaliana.universityofliverpool.thesis(ph.d.)20、tsuji,h.,taoka,k.,andshimamoto,k.(2011).regulationoffloweringinrice:twoflorigengenes,acomplexgenenetwork,andnaturalvariation.curr.opin.plantbiol.14,45-52.21、valverde,f.(2011).constansandtheevolutionaryoriginofphotoperiodictimingofflowering.j.exp.bot.62,2453-2463.22、warthmann,n.,chen,h.,ossowski,s.,weigel,d.,andherve,p.(2008).highlyspecificgenesilencingbyartificialmirnasinrice.plosone3,e1829.23、weissbach,a.,andweissbach,h.(1988).methodforplantmolecularbiologyviii.academypress,newyork,411-463.24、wenkel,s.,turck,f.,singer,k.,gissot,l.,legourrierec,j.,samach,a.andcoupland,g,(2006).constansandtheccaatboxbindingcomplexshareafunctionallyimportantdomainandinteracttoregulatefloweringofarabidopsis,plantcell,18(11):2971-2984.25、wu,w.,zheng,x.m.,lu,g.,zhong,z.,gao,h.,chen,l.,wu,c.,wang,h.j.,wang,q.,zhou,k.,wang,j.l.,wu,f.,zhang,x.,guo,x.,cheng,z.,lei,c.,lin,q.,jiang,l.,wang,h.,ge,s.,andwan,j.(2013).associationoffunctionalnucleotidepolymorphismsatdth2withthenorthwardexpansionofricecultivationinasia.proc.natl.acad.sci.usa110.2775-2780.26、wu,w.,zheng,x.m.,chen,d.,zhang,y.,ma,w.,zhang,h.,sun,l.,yang,z.,zhao,c.,zhan,x.,shen,x.,yu,p.,fu,y.,zhu,s.,cao,l.,andcheng,s.(2017).oscol16,encodingaconstans-likeprotein,repressesfloweringbyup-regulatingghd7expressioninrice.plantsci.260,60-69.27、xue,w.,xing,y.,weng,x.,zhao,y.,tang,w.,wang,l.,zhou,h.,yu,s.,xu,c.,li,x.,andzhang,q.(2008).naturalvariationinghd7isanimportantregulatorofheadingdateandyieldpotentialinrice.nat.genet.40,761-767.28、yano,m.,katayose,y.,ashikari,m.,yamanouchi,u.,monna,l.,fuse,t.,baba,t.,yamamoto,k.,umehara,y.,nagamura,y.,andsasaki,t.(2000).hd1,amajorphotoperiodsensitivityquantitativetraitlocusinrice,iscloselyrelatedtothearabidopsisfloweringtimegeneconstans.plantcell12,2473-2484.29、zhu,s.,wang,j.,cai,m.,zhang,h.,wu,f.,xu,y.,li,c.,cheng,z.,zhang,x.,guo,x.,sheng,p.,wu,m.,wang,j.,lei,c.,wang,j.,zhao,z.,wu,c.,wang,h.,andwan,j.(2017).theoshapl1-dth8-hdlcomplexfunctiohsasthetranscriptionregulatortorepressheadingdateinrice.j.exp.bot.68,553-568.序列表<110>中国水稻研究所<120>oscol15基因在调控水稻抽穗期中的应用<130>kdi060425<160>18<170>siposequencelisting1.0<210>1<211>1891<212>dna<213>水稻(oryzasativa)<400>1cctcctcattcctgcattgcattgcttccgagctcaaaagtcgtcgtagacccccgcctc60cttcctcctcctcctcgcgcttcgatttggtttggtttgattcggtttgcgagatgaagg120atggtggtggaggaggagggagggggcagcagcagcagtggccttgcgactactgcgggg180aggcggcggcggcgctgcactgcagggcggacgccgcgaggctgtgcgtcgcctgcgacc240gccacgtgcacgccgccaacgcgctctcgcggaagcacgtccgcgccccgctctgcgccg300cctgcgccgccaggccggccgccgcgcgcgtcgcctccgcctcggcgccggcgttcctgt360gcgcggactgcgacaccgggtgcggcggcgacgacggcgcggccttgcgggtgcccgtcg420aggggttctccgggtgccccgccgccgccgagctcgccgcgtcgtgggggctcgacctcc480ccggcggctgcggcggcgaggaggaggaggccgacgacgcgttcttctcggcgctcgact540actccatgctcgccgtcgaccccgtgctgcgcgacctctacgtgccatgcgacccgcccg600aggtggtggtggccggcggcgggcggcgactcaagggggaggcgctcggccaccagctcg660ccgagatggcgcgccgggaggccgagacggcgcacccgcacacgcagccgcactcggatc720tgagcccccgcacgcctcgccggacctccgccgcggcgagcggccgcctgcaggaaaagc780aagctcccccgccgttgcctcatgctgctgcgacggcggcgccgctgccgtacacttcac840tgctcatgatggcgccggccaactgcaccgagctcatggaaaacaaccgtgttggagacg900aagatgaaaatgttctgtgggagagcaccgcgccatcagtgccaccaacccagatatggg960attttaatttgggaaaatcaagggatcacaatgagaactctgcacttgaagttggatttg1020gctcaaacaatggaggctttatgattaagagttataatgacatgctcaaggagatttctt1080ctgggacaacgaaggatctggaagatatttatgactcaagatattttgcagctgccgaag1140atatcatgtcgactaatgtctgtcagctgtcatcgaaaaatccaagcaccaggagcaaca1200aacggaaggcgagctcatgcgcttcgacgatcgatggaccgacaacttccacaagccatg1260tacctgctgcttcaggggcattggggggctcttcgaacgacagaggatcggctctcccca1320aggagatttccttctgtgatcagaccgtcgtccctaccggagccgatcagaggccatgta1380ccatcaagatcgacagcgagacgctcgcgcagaacagggacagcgcgatgcagcggtaca1440gggagaagaagaagaaccgcaggtatgagaagcacatcaggtacgagtcgaggaagctga1500gagcggacacgaggaagagggtgaaaggccggtttgtgaagtcgaacggagcacctgatg1560atgtcagcaatggcgggtgatctcatccctgcaatccctgatagctagctgcaatgtacg1620tagcctggctttttgacgttgcagagatcgatgtggccatatatatgctgagagctaagt1680ctgaaatatgttgcgtatgttgttacaacctatgcacgctctgcttaagtctgtctgctg1740ctgcatgtattgttcagctaaaatttttgcgtccaagagtcgatcttgataattaagtac1800ccctactacaaatctacactacatactgtaatagaccttagattttttttctcatctcct1860cgtagaaatatatggcattttatatatacgt1891<210>2<211>1467<212>dna<213>水稻(oryzasativa)<400>2atgaaggatggtggtggaggaggagggagggggcagcagcagcagtggccttgcgactac60tgcggggaggcggcggcggcgctgcactgcagggcggacgccgcgaggctgtgcgtcgcc120tgcgaccgccacgtgcacgccgccaacgcgctctcgcggaagcacgtccgcgccccgctc180tgcgccgcctgcgccgccaggccggccgccgcgcgcgtcgcctccgcctcggcgccggcg240ttcctgtgcgcggactgcgacaccgggtgcggcggcgacgacggcgcggccttgcgggtg300cccgtcgaggggttctccgggtgccccgccgccgccgagctcgccgcgtcgtgggggctc360gacctccccggcggctgcggcggcgaggaggaggaggccgacgacgcgttcttctcggcg420ctcgactactccatgctcgccgtcgaccccgtgctgcgcgacctctacgtgccatgcgac480ccgcccgaggtggtggtggccggcggcgggcggcgactcaagggggaggcgctcggccac540cagctcgccgagatggcgcgccgggaggccgagacggcgcacccgcacacgcagccgcac600tcggatctgagcccccgcacgcctcgccggacctccgccgcggcgagcggccgcctgcag660gaaaagcaagctcccccgccgttgcctcatgctgctgcgacggcggcgccgctgccgtac720acttcactgctcatgatggcgccggccaactgcaccgagctcatggaaaacaaccgtgtt780ggagacgaagatgaaaatgttctgtgggagagcaccgcgccatcagtgccaccaacccag840atatgggattttaatttgggaaaatcaagggatcacaatgagaactctgcacttgaagtt900ggatttggctcaaacaatggaggctttatgattaagagttataatgacatgctcaaggag960atttcttctgggacaacgaaggatctggaagatatttatgactcaagatattttgcagct1020gccgaagatatcatgtcgactaatgtctgtcagctgtcatcgaaaaatccaagcaccagg1080agcaacaaacggaaggcgagctcatgcgcttcgacgatcgatggaccgacaacttccaca1140agccatgtacctgctgcttcaggggcattggggggctcttcgaacgacagaggatcggct1200ctccccaaggagatttccttctgtgatcagaccgtcgtccctaccggagccgatcagagg1260ccatgtaccatcaagatcgacagcgagacgctcgcgcagaacagggacagcgcgatgcag1320cggtacagggagaagaagaagaaccgcaggtatgagaagcacatcaggtacgagtcgagg1380aagctgagagcggacacgaggaagagggtgaaaggccggtttgtgaagtcgaacggagca1440cctgatgatgtcagcaatggcgggtga1467<210>3<211>488<212>prt<213>水稻(oryzasativa)<400>3metlysaspglyglyglyglyglyglyargglyglnglnglnglntrp151015procysasptyrcysglyglualaalaalaalaleuhiscysargala202530aspalaalaargleucysvalalacysasparghisvalhisalaala354045asnalaleuserarglyshisvalargalaproleucysalaalacys505560alaalaargproalaalaalaargvalalaseralaseralaproala65707580pheleucysalaaspcysaspthrglycysglyglyaspaspglyala859095alaleuargvalprovalgluglypheserglycysproalaalaala100105110gluleualaalasertrpglyleuaspleuproglyglycysglygly115120125gluglugluglualaaspaspalaphepheseralaleuasptyrser130135140metleualavalaspprovalleuargaspleutyrvalprocysasp145150155160proprogluvalvalvalalaglyglyglyargargleulysglyglu165170175alaleuglyhisglnleualaglumetalaargargglualagluthr180185190alahisprohisthrglnprohisseraspleuserproargthrpro195200205argargthrseralaalaalaserglyargleuglnglulysglnala210215220proproproleuprohisalaalaalathralaalaproleuprotyr225230235240thrserleuleumetmetalaproalaasncysthrgluleumetglu245250255asnasnargvalglyaspgluaspgluasnvalleutrpgluserthr260265270alaproservalproprothrglniletrpasppheasnleuglylys275280285serargasphisasngluasnseralaleugluvalglypheglyser290295300asnasnglyglyphemetilelyssertyrasnaspmetleulysglu305310315320ileserserglythrthrlysaspleugluaspiletyraspserarg325330335tyrphealaalaalagluaspilemetserthrasnvalcysglnleu340345350serserlysasnproserthrargserasnlysarglysalaserser355360365cysalaserthrileaspglyprothrthrserthrserhisvalpro370375380alaalaserglyalaleuglyglyserserasnaspargglyserala385390395400leuprolysgluileserphecysaspglnthrvalvalprothrgly405410415alaaspglnargprocysthrilelysileaspsergluthrleuala420425430glnasnargaspseralametglnargtyrargglulyslyslysasn435440445argargtyrglulyshisileargtyrgluserarglysleuargala450455460aspthrarglysargvallysglyargphevallysserasnglyala465470475480proaspaspvalserasnglygly485<210>4<211>38<212>dna<213>人工序列(artificialsequence)<400>4tgtaggtagaagaggtaccctcctcattcctgcattgc38<210>5<211>49<212>dna<213>人工序列(artificialsequence)<400>5actctagaggatccccggacgtatatataaaatgccatatatttctacg49<210>6<211>27<212>dna<213>人工序列(artificialsequence)<400>6tcggatcccagcagcagccacagcaaa27<210>7<211>40<212>dna<213>人工序列(artificialsequence)<400>7tgtaggctttatgattaagagtactgctgctgctacagcc40<210>8<211>40<212>dna<213>人工序列(artificialsequence)<400>8cttaggcattaagattaagagtattcctgctgctaggctg40<210>9<211>28<212>dna<213>人工序列(artificialsequence)<400>9tcggtaccgctgctgatgctgatgccat28<210>10<211>40<212>dna<213>人工序列(artificialsequence)<400>10agtactcttaatcataaagcctacaggagattcagtttga40<210>11<211>40<212>dna<213>人工序列(artificialsequence)<400>11aatactcttaatcttaatgcctaagagaggcaaaagtgaa40<210>12<211>43<212>dna<213>人工序列(artificialsequence)<400>12agaagaggtacccgggtcggatcccagcagcagccacagcaaa43<210>13<211>44<212>dna<213>人工序列(artificialsequence)<400>13ctagaggatccccgggtcggtaccgctgctgatgctgatgccat44<210>14<211>38<212>dna<213>人工序列(artificialsequence)<400>14accggtcccgggggatccatgaaggatggtggtggagg38<210>15<211>36<212>dna<213>人工序列(artificialsequence)<400>15ctcgcccttgctcaccatcccgccattgctgacatc36<210>16<211>22<212>dna<213>人工序列(artificialsequence)<400>16ggagatttcttctgggacaacg22<210>17<211>19<212>dna<213>人工序列(artificialsequence)<400>17tgttgctcctggtgcttgg19<210>18<211>18<212>dna<213>人工序列(artificialsequence)<400>18gtgtcgcagtccgcgcac18当前第1页12
- 该技术已申请专利。仅供学习研究,如用于商业用途,请联系技术所有人。
- 技术研发人员:曹立勇;吴玮勋;程式华;张迎信;占小登;沈希宏;于萍;陈代波;刘群恩
- 技术所有人:中国水稻研究所
- 我是此专利的发明人
- 该领域下的技术专家
- 如您需求助技术专家,请点此查看客服电话进行咨询。
- 1、薛老师:1.CRISPR-Cas系统 2.基因编辑 3.基因修复 4.天然产物合成 5.单分子技术开发与应用
- 2、张老师:1.探索新型氧化还原酶结构-功能关系,电催化反应机制 2.酶电催化导向的酶分子改造 3.纳米材料、生物功能多肽对酶-电极体系的影响4. 生物电化学传感和生物电合成体系的设计与应用。
- 3、豆老师:1.环境纳米材料及挥发性有机化合物(VOCs) 2.CO污染物的催化氧化 3.低温等离子体 4.吸脱附等控制技术
- 4、赵老师:1.高分子材料改性及加工技术 2.微孔及过滤材料 3.环境友好高分子材料
- 5、邬老师:1.高分子材料的共混与复合 2.涉及材料功能化及结构与性能的研究; 高分子热稳定剂的研发
- 如您是高校老师,可以点此联系我们加入专家库。
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
精彩留言,会给你点赞!