利用蓝藻蛋白RbrA直观检测目的蛋白表达纯化的方法与流程
本发明涉及一种直观检测目的蛋白表达和纯化的方法,特别涉及一种利用蓝藻含铁紫红色蛋白rbra来直观检测目的蛋白表达和纯化的方法。
背景技术:
蓝藻是一种能进行光合作用的原核生物,含有许多与光合作用相关的氧化还原蛋白,rbra就是其中一种可溶性含铁蛋白。rbra蛋白属于ferritin-like家族,其中的rubredoxin结构域含有fe(scys)4中心,其中的fe原子容易在空气中氧化而呈现紫红色。而在大肠杆菌中重组表达的rbra蛋白由于处于氧化态而呈紫红色。
目的蛋白在原核系统中的表达和纯化是分子生物学研究和生物技术产业化发展过程中的重要工具,是对目的蛋白功能和性质研究的基础,更促进了现代生物学科迅猛发展。在各种版本的分子生物学实验指南中,目的蛋白的表达和纯化的基本流程是:1、将目的基因克隆入合适的表达载体后导入原核细胞表达系统菌株;2、诱导表达菌株表达目的蛋白;3、通过目的蛋白所带的融合标签如gst、his等,选择不同性质的柱料将目的蛋白与其它蛋白分离,从而达到纯化目的蛋白的目的。
然而目前没有直观实时检测目的蛋白的表达纯化情况的方法。根据目前主流的实验技术,无论先小量诱导检测目的蛋白是否表达,还是最后检测是否通过柱料得到了纯化了的目的蛋白,都需要将蛋白样品通过十二烷基硫酸铵-聚丙烯酰胺凝胶电泳(sds-page)然后蛋白染色的方法来检测目的蛋白的表达纯化情况。sds-page检测蛋白样品需要将蛋白样品制样(加loadingbuffer),配置聚丙烯酰胺凝胶,然后跑大约1个小时的蛋白电泳,最后还需要将蛋白染色才能最终知道目的蛋白是否已经表达或者得到纯化,这些检测需要额外的步骤和时间。
技术实现要素:
本发明的目的是提供一种直观实时检测目的蛋白表达和纯化的方法,以解决目前体外表达纯化的目的蛋白需要通过sds-page来检测、需要大量额外的时间来进行实验操作和其它实验试剂的配制,并且不能实时直观检测的技术问题。本发明通过将目的蛋白与蓝藻含铁紫红色蛋白融合表达来检测目的蛋白的表达和纯化情况,提供了一条便捷直观的方法。
为解决上述技术问题,本发明将目的蛋白与蓝藻含铁紫红色蛋白rbra或其同源蛋白融合表达,由于rbra蛋白含有fe(scys)4中心而呈现红紫色,所以整个融合蛋白也呈现红紫色。通过观测融合蛋白的颜色来判断目的蛋白是否已经成功表达和/或纯化,从而构建了一种直观实时检测目的蛋白表达纯化的便捷方法。具体的,本发明的技术方案如下:
一种直观检测目的蛋白表达纯化的方法,构建目的蛋白基因与蓝藻rbra基因或其同源基因的融合基因,并将含该融合基因的表达载体转入表达菌种中进行表达,通过观察表达菌液破碎后的上清液和/或其纯化组分的颜色来判断融合蛋白是否被成功表达和/或纯化,如果上清液和/或纯化组分偏紫红色,说明目的蛋白与蓝藻rbra蛋白或其同源蛋白一起被表达和/或纯化。
进一步的,在对表达的融合蛋白进行柱纯化的过程中,通过观察柱料是否变为紫红色可以判断目的蛋白是否结合上柱料。
上述方法中,所述蓝藻rbra基因或其同源基因编码的蛋白属于ferritin-like家族,这些蛋白中的rubredoxin结构域含有fe(scys)4中心,其中的fe原子容易在空气中氧化而呈现紫红色,使得整个融合蛋白呈现紫红色。
蓝藻rbra基因在鱼腥藻anabaenasp.pcc7120的基因编号为alr1174,编码的蛋白为rbra。rbra蛋白的氨基酸序列如序列表中seqidno:1所示,其rubredoxin结构域中的fe(scys)4中心的4个半胱氨酸为别位于第185、188、218、221位,所以第160~237位的氨基酸是rbra蛋白显紫红色的关键结构域。rbra基因可以是序列表中seqidno:2所示的dna分子。当然,由于密码子的简并性,其它与seqidno:2所示核苷酸序列同源且编码相同氨基酸序列的dna分子也可以用于构建本发明的融合蛋白。
除了鱼腥藻anabaenasp.pcc7120,其它蓝藻中也存在rbra对应的同源蛋白基因,例如鱼腥藻anabaenavariabilisatcc29413中的ava_4424,基因核苷酸序列同源性达97.9%,所编码的蛋白氨基酸序列同源性达99.2%;例如项圈藻nostocsp.pcc7524中的nos7524_5438,基因核苷酸序列同源性达92.4%,所编码的蛋白氨基酸序列同源性达94.9%;例如单歧藻属tolypothrixsp.pcc7601中的fdutex481_00449,基因核苷酸序列同源性达89.0%,所编码的蛋白氨基酸序列同源性达95.8%;例如项圈藻nostocsp.pcc7107中的nos7107_5204,基因核苷酸序列同源性达89.0%,所编码的蛋白氨基酸序列同源性达94.9%。与上述任意一种基因具有同源性且编码相同氨基酸序列的dna分子也可以应用于本发明直观检测目的蛋白表达纯化的方法中。
蓝藻rbra基因或其同源基因与目的蛋白基因的融合基因在启动子的驱动下进行表达,所述启动子可以是组成型启动子或诱导型启动子,例如t7启动子和lac操纵子组成的诱导型启动元件。
为了方便融合蛋白的纯化,在所述融合蛋白的n端或c端还可以连接上his标签,使得融合蛋白可用ni亲和层析柱进行纯化。相应的,在构建融合蛋白基因时,在其5’端或3’端连接his标签的编码序列。
进一步的,可以在rbra基因或其同源基因与目的基因之间插入凝血酶酶切位点的编码序列,在融合蛋白纯化后通过凝血酶将rbra或其同源蛋白与目的蛋白切开。
本发明还提供了一种直观检测目的蛋白表达纯化的表达盒,包括启动子、蓝藻rbra基因或其同源基因、多克隆位点(mcs)和终止子,其中多克隆位点用于插入目的基因,插入的目的基因与蓝藻rbra基因或其同源基因处于相同的阅读框。
进一步的,所述表达盒还包括凝血酶酶切位点的编码序列,该凝血酶酶切位点的编码序列位于蓝藻rbra基因或其同源基因与多克隆位点之间。
优选的,所述表达盒在蓝藻rbra基因或其同源基因上游还接入了编码his标签的多核苷酸序列,表达出带his标签的rbra蛋白或其同源蛋白。
进一步的,所述表达盒中还具有控制诱导表达的操纵子,例如lac操纵子。
在本发明的一个实施例中,构建了从上游到下游依次为可诱导表达元件(t7启动子+lac操纵子)、带his标签编码序列的蓝藻rbra基因或其同源基因(6×his-rbra基因)、凝血酶酶切位点的编码序列、多克隆位点(包含ndeⅰ和bamhⅰ酶切位点用来插入外源基因)、表达终止子(t7终止子)的表达盒。通过酶切连接的方式可以将目的蛋白x的基因x连入表达盒的ndeⅰ和bamhⅰ位点之间,形成rbra-x融合蛋白基因。将所述表达盒构建在表达载体上,然后将表达载体转入表达菌株中诱导表达,通过观察表达菌液破碎后上清的颜色是否偏紫红来检测目的蛋白x是否表达。将偏紫红色的上清液先用镍柱纯化6×his-rbra-thrombin-x融合蛋白,这时候柱料为紫红色,表示蛋白x已经表达并与柱料结合,然后用凝血酶酶切,洗脱之后目的蛋白x就存在于洗脱液中,而6×his-rbra残留在柱料上,这样就可以得到纯化的目的蛋白x。
含有所述表达盒的载体(包括中间载体和表达载体)、宿主细胞均在本发明的保护范围内。
本发明利用蓝藻含铁紫红色蛋白rbra(或其同源蛋白)与目的蛋白融合共同表达,通过检测表达菌液破碎后上清的颜色是否偏紫红来判断目的蛋白是否表达,并且可以检测纯化蛋白的柱料是否变紫红色来检测目的蛋白是否结合在柱料上,从而实现对目的蛋白表达纯化的直观检测,方便快捷。
附图说明
图1.实施例1中间载体15b-rbra(a)和表达载体15b-rbra-x(b)的构建示意图,以及从rbra与目的蛋白的融合蛋白中纯化出目的蛋白的示意图(c)。
图2.含有pet-15b对照质粒表达菌液破碎后上清的图片(a)和含有15b-rbra-ccbp表达载体表达菌液破碎后上清的图片(b),其中:a的菌液上清为常见的偏黄颜色,b的菌液上清为偏紫红色。
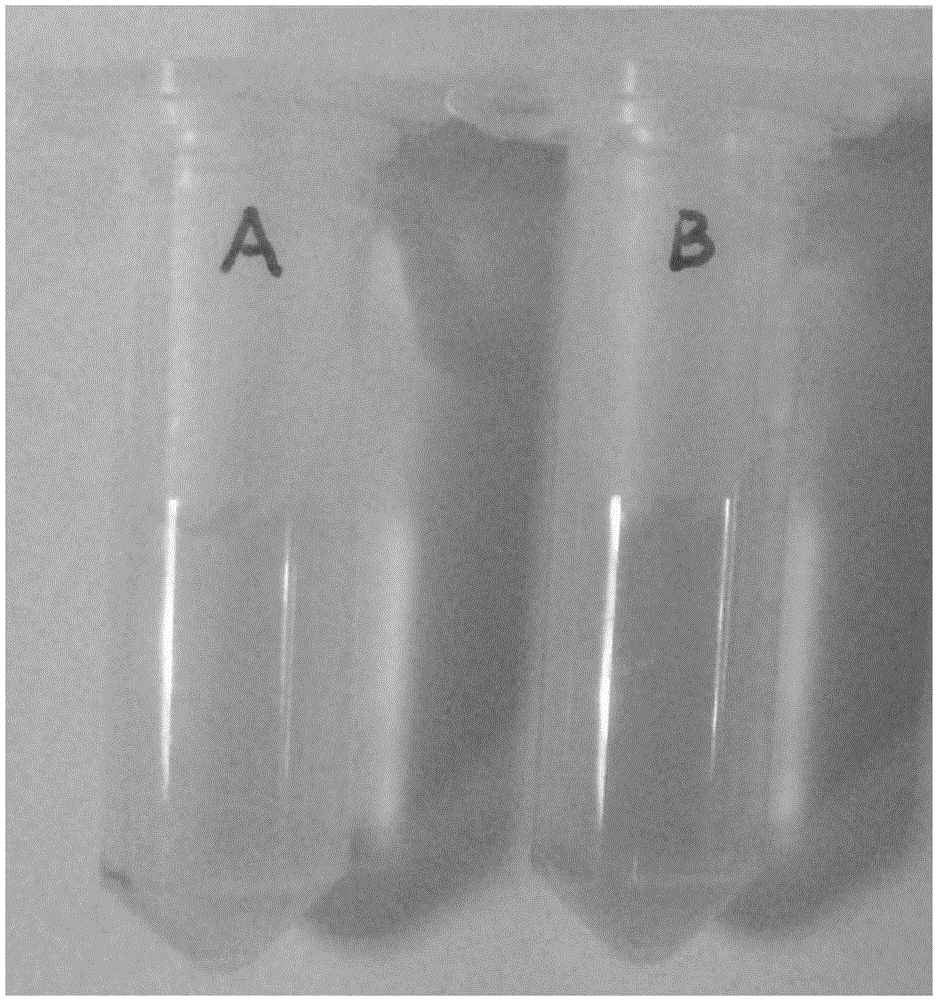
图3.含有pet-15b对照质粒表达菌液破碎后上清经过镍柱柱料后的图片(a)和含有15b-rbra-ccbp表达载体表达菌液破碎后上清经过镍柱柱料后的图片(b),其中:a的镍柱柱料颜色不变,为原来的蓝色;b的镍柱柱料颜色变为紫红色。
图4.本发明实施例2纯化目的蛋白的各步骤制样后的sds-page图。
具体实施方式
以下的实施例便于更好地理解本发明,但并不限定本发明。
下述实施例中的实验方法,如无特殊说明,均为常规方法。下述实施例中所用的试验材料,如无特殊说明,均为自常规生化试剂商店购买得到的。下述实施例中的%,如无特殊说明,均为质量百分含量。
实验用的大肠杆菌菌株dh5α和bl21(de3)购买于北京全式金公司,生长于常规的lb培养基,选择生长所用的抗生素浓度为:100μg/mlamp+。实验所用的原始质粒pet-15b均购买于生物技术公司,常见于市面。载体的克隆和蛋白的表达纯化步骤见分子生物学指南。
实施例1、整合载体的构建及阳性菌株的筛选
(1)中间载体15b-rbra的构建
中间载体15b-rbra的构建示意图如图1中a所示,其中t7promoter和lacoperator构成了可诱导表达元件(t7promoter+lacoperator);his-tag可以让rbra基因表达的rbra带上组氨酸标签;thrombin表示凝血酶酶切位点,设计目的是用凝血酶将rbra和目的蛋白的融合蛋白切开,从而获得纯化的目的蛋白;ndeⅰ和bamhⅰ是多克隆位点,可以将目的基因通过这些酶切位点连入中间载体,从而进行表达。图1a中的t7promoter、lacoperator、histag、thrombin、多克隆位点和t7teminator等元件pcr自pet-15b质粒。利用引物对p1/p2,以pet-15b质粒为模板,扩增出包含15b-rbra中除rbra基因外的所有组件15b-1片段。以野生型鱼腥藻anabaenasp.pcc7120基因组总dna为模板,利用引物对p3/p4扩增出rbra基因rbra-2片段。然后将15b-1片段和rbra片段利用北京全式金公司无缝连接试剂盒(peasy-uniseamlesscloningandassemblykit)连接在一起,得到中间载体15b-rbra。
(2)表达载体15b-rbra-x的构建
将目的蛋白x的编码基因x利用连入中间载体的ndeⅰ和bamhⅰ位点之间,形成目的蛋白x的表达载体15b-rbra-x。
实施例2、利用ccbp蛋白作为目的蛋白来检测本发明的可行性
(1)表达载体15b-rbra-ccbp的构建
15b-rbra-ccbp的构建示意图如图1中b所示。ccbp是蓝藻中一种钙离子结合蛋白,对蓝藻的格式形成有重要作用,本发明用ccbp蛋白作为目的蛋白来检验本发明的可行性。以野生型鱼腥藻anabaenasp.pcc7120基因组总dna为模板,利用引物对p5/p6扩增出ccbp基因,然后用ndeⅰ和bamhⅰ连入中间载体15b-rbra得到表达载体15b-rbra-ccbp。
(2)直观检测ccbp蛋白的表达
由于6×his-rbra-thrombin-ccbp融合蛋白为紫红色,所以我们可以通过表达菌液破碎后上清的颜色变化来直观检测目的蛋白是否已经融合表达。将表达载体15b-rbra-ccbp转入大肠表达菌株bl21(de3)中,在20℃温度的条件下加0.8μm的iptg诱导表达16h后,取菌液超声破碎2min后,12,000rpm离心2min后观察破碎后菌液上清,如图2中b所示,菌液上清为偏红色。图2中a为只含pet-15b质粒表达菌液破碎后上清的颜色,可以看到为正常的淡黄色。对照图2中的a和b,我们可以验证到:如果表达载体转入表达菌株中,诱导表达后,破碎后菌液上清如果有偏红色的现象,我们就可以直观判断目的蛋白已经得到表达。
(3)直观检测ccbp蛋白的纯化
我们还可以通过直观检测柱料的是否变紫红来检测目的蛋白是否结合上柱料,从而方便进行纯化。在确定目的蛋白已经成功表达后,我们利用镍柱纯化6×his-rbra-thrombin-ccbp融合蛋白。由于6×his-rbra-thrombin-ccbp融合蛋白为紫红色,如图3的b中箭头指示的纯化的镍柱柱料为紫红色,这表示蛋白ccbp已经结合在镍柱柱料上。而图3的a中箭头指示为只含有pet-15b质粒表达菌液破碎后上清经过柱料后柱料的颜色,这时镍柱柱料颜色没有变化。对照图3中的a和b,我们可以通过观察柱料的颜色变化,来直观检测目的蛋白是否已经与柱料结合,并为下一步纯化目的蛋白做准备。
(4)纯化目的蛋白ccbp
由于我们通过表达载体表达的是6×his-rbra-thrombin-ccbp融合蛋白。所以我们还需要通过其它步骤纯化得到ccbp纯合的目的蛋白。这些步骤如图1中c所示,首先我们在柱料上用凝血酶酶切一段时间(常温下2个小时)后,用洗脱液洗脱下的液体就是纯化得到的ccbp目的蛋白,6×his-rbra蛋白还残留在镍柱柱料上。图4表示我们通过图1c的步骤纯化目的蛋白sds-page图,其中:rbra-ccbp表示的是酶切前的柱料制取的样品,可以看到为一条大小为47kd的蛋白,可以推测为6×his-rbra-thrombin-ccbp融合蛋白;ccbp是洗脱液制取的样品,大小为20kd,可以推测为ccbp蛋白;rbra是酶切后残留柱料制取的样品,大小为17kd,我们可以推测为6×his-rbra蛋白。所以,通过图1c中的步骤,我们不仅可以直观检测目的蛋白的表达和纯化过程,还能通过凝血酶酶切去6×his-rbra标签,从而获得纯净的目的蛋白。
表1本发明所用的引物序列
sequencelisting
<110>青岛大学
<120>利用蓝藻蛋白rbra直观检测目的蛋白表达纯化的方法
<130>wx2017-99-001
<160>8
<170>patentinversion3.3
<210>1
<211>237
<212>prt
<213>anabaenapcc7120
<400>1
metaspleuserasnphethrthrleuglnasnleuglualaalaphe
151015
glyglyglusermetalaasnarglystyrleuphephealagluval
202530
alaarglysleuglyphethraspleualalysleuphearggluthr
354045
alagluglngluthrgluhisalaphealahisphegluleuleuhis
505560
progluleuvalvalgluasnproseralaleuthraspgluglnlys
65707580
arggluilevalserargcysleuserleualailegluglygluthr
859095
tyrglutyrthrthrmettyrproaspphealaalaseralaglnser
100105110
aspargasphisproalaalagluglupheleulysglnalaglnglu
115120125
sersergluhisalaserthrpheargglualaalahisargphegly
130135140
leuleulyspheilegluasntyrhisalaaspargtyrthrgluala
145150155160
leugluvalleuasnglyglyglnprovalthrargvalalaglyglu
165170175
aspproserthrarglystrpilecysargglncyssermetiletyr
180185190
aspprovalalaglyaspproaspserglyilealaproglythrpro
195200205
phegluaspileproasnasptrpglncysproilecysglyalathr
210215220
lyslysthrphelysleuleugluglulysilealaala
225230235
<210>2
<211>714
<212>dna
<213>anabaenapcc7120
<400>2
atggatttatcaaactttactacactgcaaaacttagaagctgcctttggtggtgaatcg60
atggcaaatcgtaagtatttattttttgccgaagtagcccgtaagttgggatttacagat120
ttagctaaactatttcgtgaaacagcagagcaagaaacagaacacgcttttgcccatttt180
gagttgctacatcccgaattggttgtagaaaatccctctgccttaactgacgaacaaaag240
cgagaaattgtctctcggtgtttatctttggctattgaaggcgaaacctacgagtacact300
acaatgtatccagattttgccgcatctgcccaaagcgatcgcgatcatccggcggcggaa360
gaatttctcaaacaagcgcaagaatctagcgaacacgccagcactttccgtgaagctgca420
catcgctttggtttattgaagtttatcgaaaattaccacgcggatcgctacactgaagca480
ttagaagtattaaatggtggacaacctgttaccagagtggccggcgaagatcccagcact540
cgcaaatggatttgcagacaatgcagcatgatttatgatcctgttgctggtgatcccgat600
tctggtattgcgccaggtacaccatttgaagatatccctaatgattggcaatgtccgatt660
tgtggtgctaccaaaaagacttttaaactacttgaggaaaaaattgctgcttag714
<210>3
<211>20
<212>dna
<213>人工序列
<400>3
gccgctgctgtgatgatgat20
<210>4
<211>20
<212>dna
<213>人工序列
<400>4
ctggtgccgcgcggcagcca20
<210>5
<211>40
<212>dna
<213>人工序列
<400>5
tggctgccgcgcggcaccagagcagcaattttttcctcaa40
<210>6
<211>40
<212>dna
<213>人工序列
<400>6
atcatcatcacagcagcggcgatttatcaaactttactac40
<210>7
<211>29
<212>dna
<213>人工序列
<400>7
gaaccatatggccagtgtagaacgcgacg29
<210>8
<211>30
<212>dna
<213>人工序列
<400>8
gatcggatccctaaaatttgtatcccctag30
- 还没有人留言评论。精彩留言会获得点赞!