一种通用型衣藻外源基因表达载体构建方法与流程
本发明涉及一种在衣藻中进行外源基因表达并且具有检测标签3×ha的质粒构建方法,该通用型表达质粒经过进一步插入目标表达基因片段(含有目标基因的启动子,不含有目标基因的终止密码子),利用电击转化法可以将该表达质粒随机整合进入衣藻基因组中,进行3×ha标签目标蛋白的表达。
背景技术:
绿色微藻是一类广泛分布,自养与异养相结合的微生物,其细胞代谢产物富含多种蛋白质、脂类、藻多糖、胡萝卜素、以及无机元素,在生物医药、食品保健、环境监测与净化、能源再生方面有广大的应用前景。其中,莱茵衣藻(chlamydomonasreinhardtii)是一种真核单细胞绿色微藻,除了具有以上的优点之外,还因为其生长周期快而且容易在实验室进行培养,细胞内的代谢与调控机制与高等动植物相似,以及其单倍性基因组已被阐明的原因,成为了目前研究植物光合作用,动物纤毛调控,以及新陈代谢机制等的模式生物之一。
在进行衣藻细胞内生理过程的机制研究中,常采用正向遗传学手段,进行随机的插入突变研究表型与基因的关联性,当获取了表型与基因的关联性之后,需要进行该突变体目标基因互补实验,进一步验证基因与表型的关联性,并且通过目标基因的表达可以获得更多该基因的生物学信息。另外一方面,在以衣藻的某一蛋白为研究对象时,由于没有商业化的抗体可以进行免疫标记,因此采用通用型带有标签的衣藻表达质粒是一种替代方法,该表达载体可以用来表达感兴趣的蛋白,并且在c末端带有3×ha的标签,使用针对ha标签的一抗,可以进行免疫荧光、免疫印迹、免疫共沉淀以及免疫电镜等等一系列实验。
因此该通用型衣藻外源基因表达载体,易于进行衣藻目标基因的表达与检测,获取目标蛋白的生物学信息。
技术实现要素:
本发明是提出一种通用型衣藻外源基因表达载体构建方法,易于进行突变体互补实验与外源标签蛋白的表达,从而进一步获取更多的目标基因的生物学信息。
本发明的技术方案是这样实现的:
a)克隆插入片段:设计用于无缝克隆的一对引物si-phyg3-ha-f和si-phyg3-ha-r,从pmcak_haedn_aphviii质粒克隆约400bp的片段,该片段包括3×ha标签,终止密码子tag,转录终止子rbcs2。1%琼脂糖凝胶电泳分离后使用胶回收纯化试剂盒回收,测定浓度后备用;
b)phyg3载体制备:phyg3质粒使用限制性内切酶ecorv酶切以后,产生单一片段,全长4376bp,然后进行去磷酸化处理,检测浓度后备用;
c)phyg3-ha载体构建:使用无缝克隆试剂盒将插入片段与载体连接,形成phyg3-ha质粒,全长4777bp,采用热激转化法将连接产物转化至dh5α大肠杆菌感受态中;
d)phyg3-ha质粒的验证:上述体系构建成phyg3-ha质粒(4.8kb),仍具备ecorv单酶切的基因插入位点,用菌落pcr方法检测是否正向的插入3×ha-rbcs2片段,使用引物phyg3-ha-f和phyg3-r1,约500bp。将菌落pcr的阳性克隆,过夜摇床恢复培养,提取质粒后,测序验证插入序列的正确性。
本发明的有益效果:构建完成的表达质粒phyg3-ha,经过限制性内切酶ecorv单酶切线性化后,既可以直接进行无缝克隆连接目标表达基因片段(含有目标基因的启动子,不含有目标基因的终止密码子),也可以采用经典的酶切连接片段法将目标表达基因片段连接至载体内。而且其优点还在于ecorv酶切位点(gat↓atc)酶切后产生的末端正好位于开放阅读框内,不需要考虑移码的问题,利于进行外源基因表达的构建,达到了本发明的目的。
附图说明
为了更清楚的说明本发明的技术方案,下面将对现有技术描述中所需要使用的附图作简单的介绍。
图1是实施过程中克隆400bp的插入片段与制备线性化phyg3载体(4376bp)。
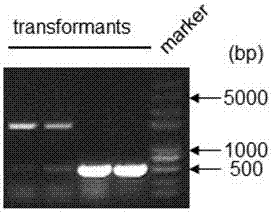
图2是使用菌落pcr方法检测转化子是否含有导入的插入片段。
图3是经过改造后的衣藻外源基因表达质粒phyg3-ha示意图。
以上图中,marker为已知分子量的dna片段(从上到下依次为8000,5000,3000,2000,1000,750,500,250,100bp)。
具体实施方式
下面结合本发明技术方案,进行详细的实施例描述,所描述的实施例仅仅是本发明的一部分实施例,而不是全部实施例。
目标是使用该通用型表达载体进行衣藻基因表达:
1、扩增目标基因:设计两个带有表达载体phyg3-ha的ecorv酶切位点两端同源臂的pcr上下游引物进行目标基因的pcr扩增(agctatgaccatgattacgaattcgat↓atcgcggccgcgggaggcctgtcgcga)。(注意:引物的5’端包含15-40nt与线性化载体末端同源的碱基序列,引物的3’端则为目标基因的互补配对序列,约17-25nt,目标基因的互补配对区域覆盖目标基因的启动子,不含有目标基因的终止密码子,并且一定为gdna)
2、制备表达载体:phyg3-ha使用ecorv酶切位点进行单酶切并去磷酸化处理,制备待连接载体。
3、获得完整的目标基因表达质粒:使用无缝克隆试剂盒将带有同源臂的表达基因和制备好的线性化载体进行连接,并转化入大肠杆菌感受态中,进一步筛选阳性克隆,并测序确保目标基因序列正确无误。
4、准备待转化的衣藻细胞、hyg+平板与制备该质粒的线性化目标质粒:在超净工作台中接种新鲜衣藻21gr细胞至无菌的tap液体吹气瓶中,在23±0.5°c,8000lx光强下连续光照通气培养,3-4天,至细胞密度为1-2×107个细胞/ml,再次转移细胞至新鲜无菌tap液体吹气瓶中,稀释至初始浓度为1×106个细胞/ml,继续连续光照培养20h,使其终浓度达4×106个细胞/ml,即可用来电击转化。所述的tap液体培养基平板包括:tap盐溶液25ml/l,磷酸盐溶液0.375ml/l,hutner微量元素1ml/l,乙酸1ml/l,tris2.42g/l,121°c高压蒸汽灭菌20min;所述tap盐溶液为:nh4cl15g/l,mgso4•7h2o4g/l,cacl2•2h2o2g/l;所述的磷酸盐溶液为:k2hpo4288g/l,kh2po4144g/l;hutner微量元素为:edta二钠盐50g/l,znso4•7h2o22g/l,h3bo311.4g/l,mncl2•4h2o5.06g/l,cocl•6h2o1.61g/l,cuso4•5h2o1.57g/l,(nh4)6mo7o24•4h2o1.1g/l,feso4•7h2o4.99g/l,用koh或者hcl调节ph至7.0。
hyg+抗性筛选平板的配制为上述tap液体培养基的配方中加入琼脂粉15g/l,进行121°c高压蒸汽灭菌20min,冷却至约55°c后添加潮霉素b(hygromycinb,hyg+,50mg/ml的储液,2500×稀释),使其终浓度为20ug/ml,倒平板备用。
质粒的线性化dna选取不破坏该表达目标基因和hyg筛选基因(均包括启动子,表达区,终止子核苷酸序列)的序列,进行酶切线性化,并进行热失活,检测浓度后备用。
5、收集处理衣藻细胞:将经过20h培养的衣藻细胞进行细胞计数;每个转化需要250ul终浓度为1×108个/ml来计算离心所需细胞体积,按照所需要的体积进行离心收集细胞,2500rpm/min,3min;去上清,用预冷的(tap+60mm山梨醇)溶液重悬并离心,2500rpm/min,3min;去上清,用预冷的(tap+60mm山梨醇)重悬至总体积(250ul×所要做的转化数),冰上放置10min;同时对电击杯进行处理、预冷。所述的(tap+60mm山梨醇)溶液配制方法为:在tap液体中加入终浓度为60mm山梨醇的山梨醇,并经过0.22um的滤膜进行过滤除菌,4°c冰箱保存备用。所述的对电击杯进行处理、预冷的方法为:在超净工作台中,用无水乙醇1ml洗0.4cm的电击杯,晾干,再用(tap+60mm山梨醇)溶液漂洗,晾干,-20°c预冷。
6、电击细胞并过夜恢复:每个预冷的电击杯中加入250ul处理好的衣藻细胞和约500ng线性化目标dna,迅速转移至冰上,使用btxecm830电击仪,设置参数如下:电压500v,电击时间4ms,电击波段6-7次,每次电击时间间隔100ms,电击完毕冰浴放置10min。将上述电击杯中的细胞分别转移至含有10ml(tap+60mm山梨醇)溶液的50ml灭菌管中,做好标记、封口膜封口,在摇床上23±0.5°c、慢速、弱光过夜恢复。
7、衣藻细胞涂布筛选与培养:将上述过夜恢复后的细胞离心,2500rpm/min,3min,在超净工作台中去上清,按照每个离心管加入2ml制备好的20%淀粉溶液,将细胞沉淀与淀粉溶液吹匀,并均匀涂布在hyg的筛选平板上,1ml/平板,2平板/转化。所述的20%淀粉溶液为依次经过70%乙醇洗涤、(tap+60mm山梨醇)溶液洗涤,并用该溶液重悬后,加入终浓度为0.4%peg6000的淀粉溶液。将上述涂布完毕,晾干后的筛选平板,用封口膜封口,倒置于23±0.5°c,14/10小时明暗光周期,8000lx光强下进行培养7天,可获得相应的转化子。
8、转化子培养与样品制备:将上述转化子在超净台中逐个挑至画方格线的tap平板上,继续在上述条件下培养至明显的绿色团块布满小方格,即可将对应的藻挑在含有1.5ml液体tap的24孔板中进行液体培养基的培养,大约经过2天的培养,离心收取每个孔的细胞沉淀并使用液氮进行速冻,放置-80°c冻存。
9、转化子的鉴定:使用常规的westernblot方法进行目标蛋白的检测,一抗使用ha的抗体,通常在目标蛋白真实大小偏大一点的位置出现条带(标签3×ha大小约4-5kd),说明目标蛋白成功表达。
经过上述实验步骤,可以将该衣藻外源基因表达载体phyg3-ha方便的应用于标签蛋白质的表达,该方法既可以用来进行突变体基因的互补,也可以用于目标蛋白的衣藻体内表达。上述实施方案中需要注意的问题是:在衣藻中进行外源蛋白的表达我们一般采用目标蛋白自身的内源性启动子,因此在进行目标基因的扩增的时候,需要扩增其内源性启动子区域。一般性的通用型启动子,例如hsrb、psad、lc8,也有成功表达目标基因的cdna的例子,可以尝试,但是内源性启动子加目标基因的gdna组合更为保险。以上所述仅为本发明的其中一个实施例,并不局限于本发明,凡是在本发明的基本原则之内所做的一切等价的替换、修改与改进应包含在本发明的保护范围以内。
序列表
<110>jiangsunormaluniversity
<120>一种通用型衣藻外源基因表达载体构建方法
<130>2017
<160>5
<170>siposequencelisting1.0
<210>1
<211>4777
<212>dna
<213>phyg3-haplasmid
<400>1
gcgcccaatacgcaaaccgcctctccccgcgcgttggccgattcattaatgcagctggca60
cgacaggtttcccgactggaaagcgggcagtgagcgcaacgcaattaatgtgagttagct120
cactcattaggcaccccaggctttacactttatgcttccggctcgtatgttgtgtggaat180
tgtgagcggataacaatttcacacaggaaacagctatgaccatgattacgaattcgatat240
cgcggccgcgggaggcctgtcgcgatacccctacgacgtgcccgactacgcctaccccta300
cgacgtgcccgactacgccgatcgatccggaccgtacccctacgacgtgcccgactacgc360
cgctagcagtacttaggcggccgcagatctcgaggatccccgctccgtgtaaatggaggc420
gctcgttgatctgagccttgccccctgacgaacggcggtggatggaagatactgctctca480
agtgctgaagcggtagcttagctccccgtttcgtgctgatcagtctttttcaacacgtaa540
aaagcggaggagttttgcaattttgttggttgtaacgatcctccgttgattttggcctct600
ttctccatgggcgggctgggcgtatttgaagcgggtaccatcaagcttctttcttgcgct660
atgacacttccagcaaaaggtagggcgggctgcgagacggcttcccggcgctgcatgcaa720
caccgatgatgcttcgaccccccgaagctccttcggggctgcatgggcgctccgatgccg780
ctccagggcgagcgctgtttaaatagccaggcccccgattgcaaagacattatagcgagc840
taccaaagccatattcaaacacctagatcactaccacttctacacaggccactcgagctt900
gtgatcgcactccgctaagggggcgcctcttcctcttcgtttcagtcacaacccgcaaac960
atgacacaagaatccctgttacttctcgaccgtattgattcggatgattcctacgcgagc1020
ctgcggaacgaccaggaattctgggaggtgagtcgacgagcaagcccggcggatcaggca1080
gcgtgcttgcagatttgacttgcaacgcccgcattgtgtcgacgaaggcttttggctcct1140
ctgtcgctgtctcaagcagcatctaaccctgcgtcgccgtttccatttgcagccgctggc1200
ccgccgagccctggaggagctcgggctgccggtgccgccggtgctgcgggtgcccggcga1260
gagcaccaaccccgtactggtcggcgagcccggcccggtgatcaagctgttcggcgagca1320
ctggtgcggtccggagagcctcgcgtcggagtcggaggcgtacgcggtcctggcggacgc1380
cccggtgccggtgccccgcctcctcggccgcggcgagctgcggcccggcaccggagcctg1440
gccgtggccctacctggtgatgagccggatgaccggcaccacctggcggtccgcgatgga1500
cggcacgaccgaccggaacgcgctgctcgccctggcccgcgaactcggccgggtgctcgg1560
ccggctgcacagggtgccgctgaccgggaacaccgtgctcaccccccattccgaggtctt1620
cccggaactgctgcgggaacgccgcgcggcgaccgtcgaggaccaccgcgggtggggcta1680
cctctcgccccggctgctggaccgcctggaggactggctgccggacgtggacacgctgct1740
ggccggccgcgaaccccggttcgtccacggcgacctgcacgggaccaacatcttcgtgga1800
cctggccgcgaccgaggtcaccgggatcgtcgacttcaccgacgtctatgcgggagactc1860
ccgctacagcctggtgcaactgcatctcaacgccttccggggcgaccgcgagatcctggc1920
cgcgctgctcgacggggcgcagtggaagcggaccgaggacttcgcccgcgaactgctcgc1980
cttcaccttcctgcacgacttcgaggtgttcgaggagaccccgctggatctctccggctt2040
caccgatccggaggaactggcgcagttcctctgggggccgccggacaccgcccccggcgc2100
ctgataaggatccccgctccgtgtaaatggaggcgctcgttgatctgagccttgccccct2160
gacgaacggcggtggatggaagatactgctctcaagtgctgaagcggtagcttagctccc2220
cgtttcgtgctgatcagtctttttcaacacgtaaaaagcggaggagttttgcaattttgt2280
tggttgtaacgatcctccgttgattttggcctctttctccatgggcgggctgggcgtatt2340
tgaagcgggtaccgggcccgtcatcccatggaagcttggcactggccgtcgttttacaac2400
gtcgtgactgggaaaaccctggcgttacccaacttaatcgccttgcagcacatccccctt2460
tcgccagctggcgtaatagcgaagaggcccgcaccgatcgcccttcccaacagttgcgca2520
gcctgaatggcgaatggcgcctgatgcggtattttctccttacgcatctgtgcggtattt2580
cacaccgcatatggtgcactctcagtacaatctgctctgatgccgcatagttaagccagc2640
cccgacacccgccaacacccgctgacgcgccctgacgggcttgtctgctcccggcatccg2700
cttacagacaagctgtgaccgtctccgggagctgcatgtgtcagaggttttcaccgtcat2760
caccgaaacgcgcgagacgaaagggcctcgtgatacgcctatttttataggttaatgtca2820
tgataataatggtttcttagacgtcaggtggcacttttcggggaaatgtgcgcggaaccc2880
ctatttgtttatttttctaaatacattcaaatatgtatccgctcatgagacaataaccct2940
gataaatgcttcaataatattgaaaaaggaagagtatgagtattcaacatttccgtgtcg3000
cccttattcccttttttgcggcattttgccttcctgtttttgctcacccagaaacgctgg3060
tgaaagtaaaagatgctgaagatcagttgggtgcacgagtgggttacatcgaactggatc3120
tcaacagcggtaagatccttgagagttttcgccccgaagaacgttttccaatgatgagca3180
cttttaaagttctgctatgtggcgcggtattatcccgtattgacgccgggcaagagcaac3240
tcggtcgccgcatacactattctcagaatgacttggttgagtactcaccagtcacagaaa3300
agcatcttacggatggcatgacagtaagagaattatgcagtgctgccataaccatgagtg3360
ataacactgcggccaacttacttctgacaacgatcggaggaccgaaggagctaaccgctt3420
ttttgcacaacatgggggatcatgtaactcgccttgatcgttgggaaccggagctgaatg3480
aagccataccaaacgacgagcgtgacaccacgatgcctgtagcaatggcaacaacgttgc3540
gcaaactattaactggcgaactacttactctagcttcccggcaacaattaatagactgga3600
tggaggcggataaagttgcaggaccacttctgcgctcggcccttccggctggctggttta3660
ttgctgataaatctggagcccgtgagcgtgggtctcgcggtatcattgcagcactggggc3720
cagatggtaagccctcccgtatcgtagttatctacacgacggggagtcaggcaactatgg3780
atgaacgaaatagacagatcgctgagataggtgcctcactgattaagcattggtaactgt3840
cagaccaagtttactcatatatactttagattgatttaaaacttcatttttaatttaaaa3900
ggatctaggtgaagatcctttttgataatctcatgaccaaaatcccttaacgtgagtttt3960
cgttccactgagcgtcagaccccgtagaaaagatcaaaggatcttcttgagatccttttt4020
ttctgcgcgtaatctgctgcttgcaaacaaaaaaaccaccgctaccagcggtggtttgtt4080
tgccggatcaagagctaccaactctttttccgaaggtaactggcttcagcagagcgcaga4140
taccaaatactgtccttctagtgtagccgtagttaggccaccacttcaagaactctgtag4200
caccgcctacatacctcgctctgctaatcctgttaccagtggctgctgccagtggcgata4260
agtcgtgtcttaccgggttggactcaagacgatagttaccggataaggcgcagcggtcgg4320
gctgaacggggggttcgtgcacacagcccagcttggagcgaacgacctacaccgaactga4380
gatacctacagcgtgagctatgagaaagcgccacgcttcccgaagggagaaaggcggaca4440
ggtatccggtaagcggcagggtcggaacaggagagcgcacgagggagcttccagggggaa4500
acgcctggtatctttatagtcctgtcgggtttcgccacctctgacttgagcgtcgatttt4560
tgtgatgctcgtcaggggggcggagcctatggaaaaacgccagcaacgcggcctttttac4620
ggttcctggccttttgctggccttttgctcacatgttctttcctgcgttatcccctgatt4680
ctgtggataaccgtattaccgcctttgagtgagctgataccgctcgccgcagccgaacga4740
ccgagcgcagcgagtcagtgagcgaggaagcggaaga4777
<210>2
<211>40
<212>dna
<213>si-phyg3-ha-f
<400>2
accatgattacgaattcgatatcgcggccgcgggaggcct40
<210>3
<211>38
<212>dna
<213>si-phyg3-ha-r
<400>3
gcgcaagaaagaagcttgatggtacccgcttcaaatac38
<210>4
<211>20
<212>dna
<213>phyg3-ha-f
<400>4
atcgcggccgcgggaggcct20
<210>5
<211>17
<212>dna
<213>phyg3-r1
<400>5
atcggtgttgcatgcag17
- 还没有人留言评论。精彩留言会获得点赞!