叶绿体基因组编辑方法与流程
本发明涉及生物技术领域,具体地,涉及RNA引导的植物叶绿体基因组编 辑。
背景技术:
现有的叶绿体基因组编辑技术主要依赖于基因枪技术,外源DNA通过同源 重组的方法插入到植物的叶绿体基因组上,并且只能在少数的植物里获得稳定 遗传的叶绿体转基因植株。另外基于同源重组的叶绿体转化没法在叶绿体基因 组上造成双链DNA破坏。最新出现的CRISPR(clustered regularly interspaced short palindromic repeats)技术被广泛的应用于植物细胞核基因组编辑,它 通过RNA把DNA核酸内切酶例如Cas9带到与RNA序列匹配的DNA区域进行切 割。
迄今为止还没有基于CRISPR技术对植物的叶绿体基因组进行编辑,所以 本领域迫切需要开发简便高效的叶绿体基因组编辑方法。
技术实现要素:
本发明的目的在于提供一种高通用性、高特异性、高效地对植物叶绿体进 行基因编辑的方法。
在本发明的第一方面,提供了一种核酸构建物,所述的核酸构建物选自下组:
(1)式I核酸构建物:
X1-X2-X3-X4-X5 (I)
式中,
X1为启动子元件;
X2为叶绿体定位信号肽元件;
X3为核酸酶元件;
X4为无或标记基因元件;
X5为终止子;
(2)式II核酸构建物:
Y1-Y2-Y3-Y4-Ya-Y5-Yb-Y6 (II)
式中,
Y1为启动子元件;
Y2为ncRNA元件;
Y3为无或标记基因元件;
Y4为无或RNA切割酶元件;
Ya和Yb各自独立地为无或RNA切割酶识别元件;
Y5为sgRNA元件;
Y6为终止子;
(3)包括式I构建物和式II构建物的构建物。
在另一优选例中,所述的式I和式II结构为5’至3’方向。
在另一优选例中,所述X1选自:35S、UBQ。
在另一优选例中,所述X2选自:infA、RbcS。
在另一优选例中,所述X4选自下组:GFP、YFP、RFP。
在另一优选例中,所述X5为Nos终止子。
在另一优选例中,所述Y1为35S启动子。
在另一优选例中,所述Y3选自下组:GFP、YFP、RFP。
在另一优选例中,所述Ya为Csy4识别序列。
在另一优选例中,所述Yb为Csy4识别序列。
在另一优选例中,所述Y6为Nos终止子。
在另一优选例中,所述式I核酸构建物中核酸酶元件X3选自下组:
(1)Cas9;
(2)Cpf1;
(3)锌指核酸酶(ZFN);
(3)转录活化剂样核酸酶(TALENS);
(4)巨核酸酶(meganuclease);
或其组合。
在另一优选例中,所述式I核酸构建物中叶绿体定位信号肽元件X2为叶绿体 信号肽infA。
在另一优选例中,所述式II核酸构建物中ncRNA元件Y2来自类病毒、或病 毒。
在另一优选例中,所述式II核酸构建物中ncRNA元件序列如SEQ ID No.:5 所示。
在另一优选例中,所述式II核酸构建物中RNA切割酶元件Y4为Csy4。
在另一优选例中,所述Csy4序列如SEQ ID No.:6所示。
在另一优选例中,所述式II核酸构建物中sgRNA元件Y5为spCas9sgRNA。
在另一优选例中,所述Y5如SEQ ID No.:8或9所示。
在本发明的第二方面,提供了一种载体或载体组合,所述载体或载体组合含 有本发明的第一方面所述的核酸构建物。
在另一优选例中,所述的式I核酸构建物和式II核酸构建物位于不同载体上。
在另一优选例中,所述的式I核酸构建物和式II核酸构建物位于同一载体上。
在本发明的第三方面,提供了一种试剂组合,包括:
(i)本发明的第二方面所述的载体或载体组合。
在本发明的第四方面,提供了一种植物叶绿体基因编辑方法,包括步骤:
(i)将(a)本发明的第二方面所述的载体或载体组合以及(b)任选的供体核酸 片段,导入植物细胞、植物组织或植物(植株),从而在所述植物细胞、植物组织或 植物中产生基因编辑;和
(ii)任选地,对发生所述基因编辑的植物细胞或植物进行检测、筛选或鉴定。
在另一优选例中,所述的方法还包括:
(iii)对步骤(ii)中经鉴定发生了所述基因编辑的植物细胞、植物组织或植 物进行再生或培养。
在另一优选例中,所述的基因编辑包括基因敲除、定点插入、基因置换、或 其组合。
在另一优选例中,所述的定向插入包括基于同源重组或非同源重组末端连接 的定点插入。
在另一优选例中,所述的基因编辑包括单位点或多位点的基因编辑。
在另一优选例中,所述导入为通过农杆菌导入。
在另一优选例中,所述导入为通过基因枪导入。
在另一优选例中,所述导入为通过显微注射法、电击法、超声波法和聚乙二 醇(PEG)介导法导入。
在另一优选例中,所述的植物选自下组:农作物、树木、花卉。
在另一优选例中,所述的植物选自下组:禾本科植物、豆科植物和十字花科 植物。
在另一优选例中,所述的植物包括:拟南芥、小麦、大麦、燕麦、玉米、水 稻、高粱、粟、大豆、花生、烟草和番茄。
应理解,在本发明范围内中,本发明的上述各技术特征和在下文(如实施 例)中具体描述的各技术特征之间都可以互相组合,从而构成新的或优选的技 术方案。限于篇幅,在此不再一一累述。
附图说明
图1显示用CRISPR/Cas9突变叶绿体里的GFP;
(A)显示使用的质粒图谱。所述载体用的是35S启动子(P35S)和nos终止 子(Tnos)。infA的信号肽(TPinfA)和类病毒上的一段非编码RNA(ncRNA)被用 来将来自Streptococcus pyogenes(化脓链球菌)的Cas9和sgRNA带到叶绿体。
(B)显示Cas9-GFP融合蛋白(上图)和GFP的mRNA(下图)被运进叶绿体。
(C)显示通过CRISPR/Cas9对叶绿体基因组上的aadA16gfp基因进行突变。 本实验用到的2个sgRNA的靶点(sgRNA1和sgRNA2);用2个载体:35S启动子 驱动带有叶绿体信号肽infA的Cas9和35S启动子驱动非编码RNA引导的带有 Csy4和2个Csy4识别位点的sgRNA2瞬时转化pMSK56植株(下图)和没有转化 载体的(上图),然后拍摄荧光显微镜图片。
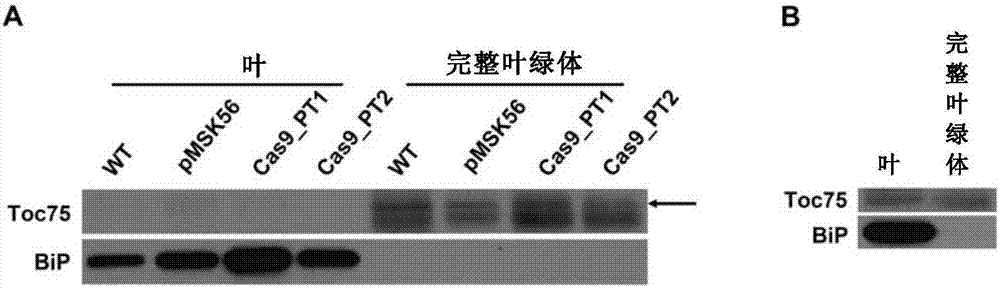
(D)显示转化有CRISPR/Cas9pMSK56植株的叶绿体里GFP蛋白数量比没有 转化的植株少。植物转化有sgRNA1和sgRNA2被命名为Cas9_PT1和Cas9_PT2。 从Nicotiana tabacum(烟草)叶绿体里提取的蛋白被用作野生型对照(WT)。位 于内质网上的BiP蛋白被当作细胞质标记;位于叶绿体外膜上的Toc75蛋白(用 箭头指出的位置)被当作叶绿体标记。每个样本的上样量为30微克蛋白。(标 尺:10微米)。
图2显示从2个模式植物里提取完整的叶绿体。烟草(A)和拟南芥(B)的Western blot结果。箭头标记出Toc75特异蛋白的位置。分别从叶片和完整的 叶绿体里提取蛋白。对于叶片,上样量为20微克蛋白;对于叶绿体,上样量 为30微克蛋白。
图3显示测序验证sgRNA1和sgRNA2的环化逆转录PCR产物。sgRNA1和 sgRNA2的环化逆转录PCR产物的序列色谱,每个测了3个独立的克隆。
图4显示测序结果发现在转化有sgRNA2的pMSK56植物的aadA16GFP里有 插入DNA片段(图4A);PCR验证只有在转化有sgRNA2的pMSK56植物的叶绿 体DNA能够扩增到条带(图4B)。
图5显示运用CRISPR/Cas9降低叶绿体基因组上aadA16gfp基因的表达。 在pMSK56的CRISPR/Cas9T1代转基因植株里(aadA16gfpT1-1~aadA16gfpT1-5) 检测没有核酸酶活性的Cas9(dCas9)表达量。dCas9蛋白表达量高的植株里 GFP的表达量低。没有CRISPR/Cas9的pMSK56植株作为对照,即control。
图6显示在拟南芥里靶向降低rpl33的表达量。(A)实验里的用到的质粒 图谱。PUBQ和TUBQ分别代表AtUBQ1的启动子和终止子。(B)通过CRISPR/Cas9 系统对拟南芥var2植株叶绿体基因组上的rpl33基因进行knockdown。本实验 用到的2个sgRNAs分别靶向rpl33基因的模板链(T)和非模板链(NT)。CK 代表得是从只转化了没有sgRNA的CRISPR/Cas9系统的var2植株。含有sgRNA 靶向rpl33模板链和sgRNA靶向rpl33非模板链CRISPR/Cas9载体的拟南芥 var2T1代植株被分别标记为33T和33NT。var2引起的叶杂色表型被恢复和没 有被恢复的植株被分别标记为S和NS。
图7显示靶向降低rpl33的表达可以恢复var2突变介导的叶杂色表型。 从左到右分别是是转化不含有sgRNA,sgRNA靶向rpl33模板链和sgRNA靶向 rpl33非模板链CRISPR/Cas9载体的拟南芥var2植株。灰色箭头指出得是通过 靶向降低rpl33的表达已经恢复叶杂色表型的植株。
具体实施方式
本发明人经过广泛而深入的研究、首次构建了一种基于CRISPR技术的叶 绿体基因组定点编辑系统、以及用于叶绿体基因组定点编辑的核酸构建物、载 体或载体组合、以及叶绿体基因组定点编辑方法。本发明的方法可以在预定的 植物基因组位点、简便而高效地进行基因敲除或者同源重组和外源片段的定向 插入。在此基础上完成了本发明。
典型地,本发明CRISPR编辑系统中核酸酶采用Cas9蛋白,通过叶绿体信号 肽infA将Cas9蛋白带进叶绿体;采用非编码RNA(ncRNA),例如来自类病毒的 ncRNA将sgRNA(例如spCas9sgRNA)带进叶绿体,从而实现对叶绿体基因组上 基因的敲除。与传统的基因枪技术相比,本发明方法不仅降低了操作难度,提 高了叶绿体基因定点编辑的效率和精确性,而且降低了操作成本。此外,本发 明方法还可有效用于那些无法进行基因枪转化的植物品种。
术语
除非另外定义、本文使用的所有技术和科学术语的意义与本发明所属领域 普通技术人员通常所理解的相同。本文中述及的所有出版物和其他参考文献都 通过引用纳入本文。
如本文所用、所述的“含有”、“具有”或“包括”包括了“包含”、“主 要由……构成”、“基本上由……构成”、和“由……构成”。
如本文所用、术语“操作性相连”或“可操作地连于”指这样一种状况、 即线性DNA序列的某些部分能够调节或控制同一线性DNA序列其它部分的活性。 例如、如果启动子控制序列的转录、那么它就是可操作地连于编码序列。
基于核酸酶的植物基因组定点编辑的核酸构建物和方法
本发明提供了一种核酸构建物,所述的核酸构建物选自:
(1)式I核酸构建物:
X1-X2-X3-X4-X5 (I)
式中,
X1为启动子元件;
X2为叶绿体定位信号肽元件;
X3为核酸酶元件;
X4为无或标记基因元件;
X5为终止子;
(2)式II核酸构建物:
Y1-Y2-Y3-Y4-Ya-Y5-Yb-Y6 (II)
式中,
Y1为启动子元件;
Y2为ncRNA元件;
Y3为无或标记基因元件;
Y4为无或RNA切割酶元件;
Ya、Yb为无或RNA切割酶识别元件;
Y5为sgRNA元件;
Y6为终止子;
(3)包括式I构建物和式II构建物的构建物。
在上述结构式中,“-”表示键。
在本发明中、上述的各元件可用常规方法(如PCR法、人工全合成)制备、然 后用常规方法进行连接、从而形成本发明所述的核酸构建物。如需要、在连接反应 之前、可以任选地进行酶切反应。
此外、本发明的所述核酸构建物可以是线性的、也可以是环状的。本发明的 所述核酸构建物可以是单链的、也可以是双链的。本发明的所述核酸构建物可以是 DNA、也可以是RNA、或DNA/RNA杂合。
如本文所用、“标记基因”指基因编辑过程中用来筛选基因编辑成功的细胞 的基因,可用于本申请的标记基因没有特别限制、包括基因编辑领域常用的各种标 记基因,代表性例子包括(但并不限于):绿色荧光蛋白(GFP)、黄色荧光蛋白(YFP)、 潮霉素抗性基因(Hyg)、卡那霉素抗性基因(NPTII)、新霉素基因、或嘌呤霉素抗性 基因。
如本文所用,术语“植物启动子”指能够在植物细胞中启动核酸转录的核酸 序列。该植物启动子可以是来源于植物、微生物(如细菌、病毒)或动物等,或者是 人工合成或改造过的启动子。代表性的例子包括(但并不限于):35S启动子。
如本文所用,术语“植物终止子”指能够在植物细胞中可使转录停止的终止 子。该植物转录终止子可以是来源于植物、微生物(如细菌、病毒)或动物等,或者 是人工合成或改造过的终止子。代表性的例子包括(但并不限于):Nos终止子。
如本文所用,术语“信号肽”是指新合成的蛋白质向分泌通路转移的短肽链。 代表性的例子包括(但并不限于):infA信号肽。
如本文所用,典型地,所述式I核酸构建物中核酸酶元件X3选自下组:
(1)Cas9;
(2)Cpf1;
(3)锌指核酸酶(ZFN);
(3)转录活化剂样核酸酶(TALENS);
(4)巨核酸酶(meganuclease);
或其组合。
如本文所用、术语“核酸酶元件”指编码具有切割活性的核酸酶的核苷酸 序列。在插入的多聚核苷酸序列被转录和翻译从而产生功能性核酸酶的情况 下、技术人员会认识到、因为密码子的简并性、有大量多聚核苷酸序列可以编 码相同的多肽。另外、技术人员也会认识到不同物种对于密码子具有一定的偏 好性、可能会根据在不同物种中表达的需要、会对核酸酶的密码子进行优化、 这些变异体都被术语“核酸酶元件”所具体涵盖。
此外,术语“核酸酶元件”特定地包括了全长的、与Cas9和/或Cpf1基 因序列基本相同的序列、以及编码出保留Cas9和/或Cpf1蛋白功能的蛋白质 的序列。
典型地,术语“核酸酶元件”为来自化脓链球菌(Streptococcus pyogenes) 的Cas9蛋白的编码序列。
优选地,本发明所述式I核酸构建物中叶绿体定位信号肽元件X2为叶绿体信 号肽infA。
典型地,本发明所述式II核酸构建物中ncRNA元件Y2来自类病毒。
优选地,本发明所述式II核酸构建物中ncRNA元件序列如SEQ ID No.:5所 示。
典型地,本发明所述式II核酸构建物中RNA切割酶元件Y4为Csy4。
优选地,本发明所述Csy4序列如SEQ ID No.:6所示。
典型地,本发明所述式II核酸构建物中sgRNA元件Y5为spCas9sgRNA。
优选地,本发明所述sgRNA序列如SEQ ID No.:8或9所示。
本发明还提供了一种载体或载体组合,所述载体或载体组合含有本发明所述 的核酸构建物。
优选地,本发明所述的式I核酸构建物和式II核酸构建物位于同一载体上。
在本发明的核酸构建物和/或载体中、一些元件之间是可操作连接的。例如当 启动子与编码序列可操作连接时、指所述启动子能够启动所述编码序列的转录。
本发明还提供了含有上述载体或载体组合的试剂组合以及试剂盒,它们可用 于本发明的植物叶绿体基因编辑方法。
本发明还提供了一种植物叶绿体基因编辑方法,包括步骤:
(i)将(a)本发明所述的载体或载体组合以及(b)任选的供体核酸片段,导入植 物细胞、植物组织或植物,从而在所述植物细胞、植物组织或植物中产生基因编辑; 和
(ii)任选地,对发生所述基因编辑的植物细胞或植物进行检测、筛选或鉴定。
在本发明中、所述的基因编辑包括基因敲除、定点插入、基因置换、或其 组合。
本发明的植物基因编辑方法、可用于改良各类植物、尤其是对农作物进行 改良。
如本文所用、术语“植物”包括全植株、植物器官(如叶、茎、根等)、种 子和植物细胞以及它们的子代。可用于本发明方法的植物的种类没有特别限 制、一般包括任何可进行转化技术的高等植物类型、包括单子叶、双子叶植物 和裸子植物。
本发明的主要优点在于:
(1)首次利用CRISPR技术在叶绿体基因组上造成了有效的双链DNA的切割 以及基因编辑;
(2)有效降低了叶绿体基因编辑的成本和难度;
(3)大幅拓宽了叶绿体基因编辑物种的广度。
下面结合具体实施例,进一步阐述本发明。应理解,这些实施例仅用于说 明本发明而不用于限制本发明的范围。下列实施例中未注明具体条件的实验方 法,通常按照常规条件如Sambrook等人,分子克隆:实验室手册(New York:Cold Spring Harbor Laboratory Press,1989)中所述的条件,或按照制造厂商所建 议的条件。除非另外说明,否则百分比和份数按重量计算。本发明中所涉及的 实验材料如无特殊说明均可从市售渠道获得。
材料
实验中所用的拟南芥材料为野生型Col-0。Col-0种子用5%次氯酸钠消毒 后播种于1/2MS固体培养基上,平板4℃处理三天后放入光照培养箱(22度, 16hrs光照/8hrs黑暗)培养10-14天,然后移苗到营养土中,温室继续培养。
实验所用烟草为野生型Nicotiana tabacum(烟草)和以Nicotiana tabacum 为受体的叶绿体转基因植株pMSK56(pMSK56叶绿体基因组上编码有aadAGFP融 合基因)。pMSK56转基因植株为一种常规的烟草植株(Khan and Maliga,1999)。 Nicotiana tabacum及pMSK56的种子播于营养土中,放置于温室(26度,16hrs 光照/8hrs)培养10天左右,单株幼苗移载到营养土中,相同条件继续培养3 周左右用于瞬时转化实验。
方法
目标位点设计
对于SpCas9蛋白,其PAM序列要求为5’-NGG-3’,因此叶绿体基因组上 合适的靶位点为5’-N20NGG-3’。
载体构建
pCam1300-35S-ncRNA-GFP,pCam1300-35S-ncRNA-GFP-spsgRNA载体的构建
实验中能将RNA导入到叶绿体中的ncRNA参照了(Gómez and Pallás,2010) 文中的序列,人工合成后连入到市售的pUC57载体中得到pUC57-ncRNA。ncRNA 序列,GFP编码序列,spsgRNA骨架分别从pUC57-ncRNA,pGWB505,pCas9(AtU6) 载体扩增得到,ncRNA-GFP,ncRNA-GFP-spsgRNA可以通过叠连PCR的方法依次 连接在一起,PCR产物回收,用XmaI,BamHI消化后连入到pCam1300-35S载体 中的35S启动子(SEQ ID No.:1)和NOS终止子(SEQ ID No.:2)之间得到载体 pCam1300-35S-ncRNA-GFP,pCam1300-35S-ncRNA-GFP-sgRNA。
核苷酸序列SEQ ID No.:1
TCAACATGGTGGAGCACGACACACTTGTCTACTCCAAAAATATCAAAGATACAGTCTCAGAAGACC AAAGGGCAATTGAGACTTTTCAACAAAGGGTAATATCCGGAAACCTCCTCGGATTCCATTGCCCAGCTATC TGTCACTTTATTGTGAAGATAGTGGAAAAGGAAGGTGGCTCCTACAAATGCCATCATTGCGATAAAGGAAA GGCCATCGTTGAAGATGCCTCTGCCGACAGTGGTCCCAAAGATGGACCCCCACCCACGAGGAGCATCGTGG AAAAAGAAGACGTTCCAACCACGTCTTCAAAGCAAGTGGATTGATGTGATAACATGGTGGAGCACGACACA CTTGTCTACTCCAAAAATATCAAAGATACAGTCTCAGAAGACCAAAGGGCAATTGAGACTTTTCAACAAAG GGTGATATCCGGAAACCTCCTCGGATTCCATTGCCCAGCTATCTGTCACTTTATTGTGAAGATAGTGGAAA AGGAAGGTGGCTCCTACAAATGCCATCATTGCGATAAAGGAAAGGCCATCGTTGAAGATGCCTCTGCCGAC AGTGGTCCCAAAGATGGACCCCCACCCACGAGGAGCATCGTGGAAAAAGAAGACGTTCCAACCACGTCTTCAAAGCAAGTGGATTGATGTGATATCTCCACTGACGTAAGGGATGACGCACAATCCCACTATCCTTCGCAAG ACCCTTCCTCTATATAAGGAAGTTCATTTCATTTGGAGAGGACCTCGACCTCAACACAACATATACAAAAC AAACGAATCTCAAGCAATCAAGCATTCTACTTCTATTGCAGCAATTTAAATCATTTCTTTTAAAGCAAAAG CAATTTTCTGAAAATTTTCACCATTTACGAACGATA
核苷酸序列SEQ ID No.:2
TGATTGATCGATAGAGCTCGAATTTCCCCGATCGTTCAAACATTTGGCAATAAAGTTTCTTAAGAT TGAATCCTGTTGCCGGTCTTGCGATGATTATCATATAATTTCTGTTGAATTACGTTAAGCATGTAATAATT AACATGTAATGCATGACGTTATTTATGAGATGGGTTTTTATGATTAGAGTCCCGCAATTATACATTTAATA CGCGATAGAAAACAAAATATAGCGCGCAAACTAGGATAAATTATCGCGCGCGGTGTCATCTATGTTACTAG ATCGG
pCam1300-35S-infA-Cas9-GFP载体的构建
叶绿体定位信号infA(SEQ ID No.:3)从拟南芥cDNA文库中扩增获得。 Cas9,GFP(SEQ ID No.:4)编码序列分别从载体pCas9(AtU6),pGWB505上扩 增得到,infA,Cas9,GFP三个片段通过Gibson assembly的方法连入 pCam1300-35S载体的35S启动子和NOS终止子之间得到 pCam1300-35S-infA-Cas9-GFP载体。
核苷酸序列SEQ ID No.:3
ATGCTTCAACTCTGCTCCACTTTCCGTCCTCAACTTCTTCTTCCTTGTCAATTCCGATTTACAAAT GGCGTTTTGATTCCCCAAATAAACTATGTTGCAAGCAATTCAGTTGTGAATATCCGGCCAATGATACGATG CCAGAGAGCAAGCGGAGGAAGAGGAGGAGCTAATAGAAGCAAA
核苷酸序列SEQ ID No.:4
ATGGTGAGCAAGGGCGAGGAGCTGTTCACCGGGGTGGTGCCCATCCTGGTCGAGCTGGACGGCGAC GTAAACGGCCACAAGTTCAGCGTGTCCGGCGAGGGCGAGGGCGATGCCACCTACGGCAAGCTGACCCTGAA GTTCATCTGCACCACCGGCAAGCTGCCCGTGCCCTGGCCCACCCTCGTGACCACCTTCACCTACGGCGTGC AGTGCTTCAGCCGCTACCCCGACCACATGAAGCAGCACGACTTCTTCAAGTCCGCCATGCCCGAAGGCTAC GTCCAGGAGCGCACCATCTTCTTCAAGGACGACGGCAACTACAAGACCCGCGCCGAGGTGAAGTTCGAGGG CGACACCCTGGTGAACCGCATCGAGCTGAAGGGCATCGACTTCAAGGAGGACGGCAACATCCTGGGGCACAAGCTGGAGTACAACTACAACAGCCACAACGTCTATATCATGGCCGACAAGCAGAAGAACGGCATCAAGGTG AACTTCAAGATCCGCCACAACATCGAGGACGGCAGCGTGCAGCTCGCCGACCACTACCAGCAGAACACCCC CATCGGCGACGGCCCCGTGCTGCTGCCCGACAACCACTACCTGAGCACCCAGTCCGCCCTGAGCAAAGACC CCAACGAGAAGCGCGATCACATGGTCCTGCTGGAGTTCGTGACCGCCGCCGGGATCACTCACGGCATGGAC GAGCTGTACAAGTAA
pCam1300-35S-infA-Cas9载体的构建
用上游引物5’端带有终止密码子的引物从pCam1300-35S载体上扩增出 NOS终止子,PCR产物回收后用BamHI和EcoRI进行双酶切。再将GFP基因和 NOS终止子从pCam1300-35S-infA-Cas9-GFP载体用BamHI和EcoRI切除,片段 回收后与上述回收的NOS终止子连接得到pCam1300-35S-infA-Cas9载体。
pCam1300-35S-ncRNA-Csy4-sgRNA载体的构建
C端带有3×Frag标签的Csy4基因(SEQ ID No.:6)由人工合成连入到 pUC57载体中得到pUC57-Csy4。ncRNA序列(SEQ ID No.:5),Csy4-3×Frag 编码区,sgRNA骨架分别从pUC57-ncRNA,pUC57-Csy4,pCas9(AtU6)载体扩增 得到。sgRNA骨架上,下游加有20nt的Csy4识别位点(SEQ ID No.:7)。另 外两个AarI酶切位点加在Csy4-3×Frag编码区下游。ncRNA序列, Csy4-3×Frag编码区,sgRNA骨架用叠连PCR的方法依次连接在一起,PCR产 物回收,用XmaI,BamHI消化后连入到pCam1300-35S载体中的35S启动子和 NOS终止子之间。
核苷酸序列SEQ ID No.:5
TTGGCGAAACCCCATTTCGACCTTTCGGTCTCATCAGGGGTGGCACACACCACCCTATGGGGAGAG GTCGTCCTCTATCTCTCCTGGAAGGCCGGAGCAATCCAAAAGAGGTACACCCACCCATGGGTCGGGACTTT AAATTCGGAGGATTCGTCCTTTAAACGTTCCTCCAAGAGTCCCTTCCCCAAACCCTTACTTTGTAAGTGTG GTTCGGCGAATGTACCGTTTCGTCCTTTCGGACTCATCAGGGAAAGTACACACTTTCCGACGGTGGGTTCG TCGACACCTCTCCCCCTCCCAGGTACTATCCCCTTTCCAGGATTTGTTCCC
核苷酸序列SEQ ID No.:6
ATGGACCACTATCTGGACATCAGACTGAGGCCCGATCCTGAGTTCCCTCCCGCCCAGCTGATGAGC GTGCTGTTTGGCAAGCTGCATCAGGCTCTGGTCGCCCAAGGCGGAGACAGAATCGGCGTGTCCTTCCCCGACCTGGACGAGTCCCGGAGTCGCCTGGGCGAGCGGCTGAGAATCCACGCCAGCGCAGACGATCTGCGCGCCC TGCTGGCCCGGCCTTGGCTGGAGGGCCTGCGGGATCATCTGCAGTTTGGCGAGCCCGCCGTGGTGCCACAC CCAACACCCTACCGCCAGGTGAGCCGCGTGCAGGCCAAGTCAAATCCCGAGAGACTGCGGCGGAGGCTGAT GAGGCGACATGATCTGAGCGAGGAGGAGGCCAGAAAGAGAATCCCCGACACAGTGGCCAGAGCCCTGGATC TGCCATTTGTGACCCTGCGGAGCCAGAGCACTGGCCAGCATTTCAGACTGTTCATCAGACACGGGCCCCTG CAGGTTACAGCCGAGGAGGGCGGATTTACATGCTATGGCCTGTCTAAAGGCGGCTTCGTGCCCTGGTTCTA A
核苷酸序列SEQ ID No.:7
GTTCACTGCCGTATAGGCAG
pCam1300-UBQ-spsgRNA-35S-Cas9载体的构建
以拟南芥Col-0基因组为模板分别扩增出UBQ1基因(AT3G52590)的启动子 UBQpro和终止子UBQTer。从pCam1300-35S-ncRNA-Csy4-sgRNA载体中扩增出 ncRNA-Csy4-sgRNA片段,以叠连PCR的方法将UBQpro、ncRNA-Csy4-spsgRNA、 UBQTer三段依次连接在一起,PCR回收产物用HindIII和XmaI消化后连入 pCambia1300载体之中,得到pCam1300-UBQ-spsgRNA载体。从 pCam1300-35S-infA-Cas9载体中扩增出35S-infA-Cas9-NOS片段,PCR产物回 收后利用Gibson assembly的方法连入pCam1300-UBQ-sgRNA载体中得到 pCam1300-UBQ-sgRNA-35S-Cas9。
sgRNA装入相应目标载体
对于载体pCam1300-35S-ncRNA-Csy4-sgRNA和pCam1300-UBQ-sgRNA-35S- Cas9,选取5’-N20NGG-3’作为靶标序列。分别合成上游引物GCAGN20和下游引 物AAACN20,上下游引物退火后形成带有4nt接头的短双链DNA片段。载体pC am1300-35S-ncRNA-Csy4-sgRNA和pCam1300-UBQ-sgRNA-35S-Cas9用AarI酶消 化4小时后电泳,切胶回收,然后与退火形成的短链DNA片段连接。
蛋白瞬时定位实验
载体pCam1300-35S-ncRNA-GFP,pCam1300-35S-ncRNA-GFP-sgRNA, pCam1300-35S-infA-Cas9-GFP通过冻融法转入农杆菌GV3101感受态中,农杆 菌28度黑暗培养两天,挑取单克隆于5ml LB抗性培养基(50mg/L卡那霉素, 25mg/L利福平)中,28℃,240rpm培养16小时,以1:100的比例转接到 新的5ml LB抗性培养基中(培养基中另加有2μM的乙酰丁香酮,10mM MES, pH 5.6),28℃,240rpm培养过夜到OD600=3。4000rpm,10min收集菌体, 用10mM MES pH 5.6,10mM MgCl2,10μm乙酰丁香酮溶液将菌体悬起,调 OD600到0.6-0.8。室温静置2-3小时后,用不带针头的1ml医用注射器将农杆 菌注射到4周左右生长状态良好烟草叶片背面。培养60-72小时后取样观察蛋 白定位。
pMSK56报告基因aadA-GFP敲除
选取aadA-GFP报告基因中符合5’-N20NGG-3’序列要求的位点。合成相应 的sgRNA序列,退火后连入AarI酶切过的pCam1300-35S-ncRNA-Csy4-spsgRNA 载体中。构建好的载体连同空载pCam1300-35S-infA-Cas9载体转入农杆菌 GV3101感受态中。另外,为了抑制RNAi引起的转录后沉默现象,将表达p19 蛋白的载体也转入农杆菌GV3101感受态中。农杆菌28℃黑暗培养两天,挑取 单克隆于5ml LB抗性培养基(50mg/L卡那霉素,25mg/L利福平)中,28℃, 240rpm培养16小时,以1:100的比例转接到新的5ml LB抗性培养基中(培 养基中另加有2μM的乙酰丁香酮,10mM MES,pH 5.6),28度,240rpm培 养过夜到OD600=3。4000rpm,10min收集菌体,用10mM MES pH 5.6,10mM MgCl2,10μm乙酰丁香酮溶液将菌体悬起,调OD600到1.5。表达p19的农杆 菌OD600调到1.0。将含pCam1300-35S-ncRNA-Csy4-sgRNA, pCam1300-35S-infA-Cas9,p19载体的三种农杆菌以1:1:1的比例混合,室 温静置2-3小时后,用不带针头的1ml医用注射器将农杆菌注射到4周左右生 长状态良好烟草叶片背面。培养60-72小时后取样观察GFP信号的变化。
拟南芥转化与筛选
选取合适的sgRNA序列依前述方法装入pCam1300-UBQ-sgRNA-35S-Cas9载 体中。相应载体转入农杆菌GV3101中。选取健壮的盛花期的Col-0以浸花法 进行遗传转化,正常护理一个月后收种得T0代种子。T0代种子用5%次氯酸钠消 毒后在含50mg/L潮霉素的1/2MS平板上筛选,阳性苗移栽到营养土中放置 于温室继续培养。
叶绿体提取与Western blot检测
在冷库中将植物组织在预冷的0.33M sorbitol,20mM tricine(pH 8.4), 5mM EGTA,5mM EDTA,10mM NaHCO3,0.1%(w/v)BSA中按照4ml每mg 的比例用匀浆器打碎。用3层纱布过滤一次,再用一层Miracloth 过滤一次。在4度离心机中2000g离心2分钟,用0.33M sorbitol,20mM HEPES (pH 7.9),5mM MgCl2,2.5mM EDTA,10mM NaHCO3,0.1%(w/v)BSA,2mM ascorbate将沉淀悬浮起来。将悬浮液置于40/100%(v/v)的Percoll(sigma) 梯度上4度离心30分钟(40%(v/v)Percoll溶液:0.33M sorbitol,20mM HEPES(pH 7.9),5mM MgCl2,2.5mM EDTA,10mM NaHCO3,0.2%(w/v)BSA,2 mM ascorbate,40%(v/v)Percoll;100%(v/v)Percoll溶液:0.33M sorbitol, 20mM HEPES(pH 7.9),5mM MgCl 2,2.5mM EDTA,10mM NaHCO3,0.2%(w/v)BSA, 2mM ascorbate,100%(v/v)。收集位于两层Percoll梯度中间的完整叶绿体, 加入10ml 0.33M sorbitol,20mM HEPES(pH 7.9),5mM MgCl2,2.5mM EDTA, 10mM NaHCO3,0.1%(w/v)BSA,2mM ascorbate,2000g 4度离心2分钟。 得到的沉淀即为叶绿体。
在提好的叶绿体中加入200μl 50mM Tris-Hcl(pH6.8),2%(w/v)SDS, 10%(v/v)甘油,1%(v/v)巯基乙醇。在95度煮5分钟,用最大的速度在4 度离心15分钟,转移上清到一个新管中。每μl上清中加入2μl 150mM Tris-Hcl (pH6.8),6%(w/v)SDS,0.3%(w/v)溴酚蓝,30%(v/v)甘油,3%(v/v)巯 基乙醇。用8%的SDS-PAGE胶分离蛋白,将分离后的蛋白在105V的电压下用 Bio-Rad仪器转移到PVDF(Millipore)膜上。在20mM Tris-HCl(pH 8),150mM NaCl,0.1%(V/V)Tween 20,5%SKIM MILK POWDER,中封闭1小时。在20mM Tris-HCl(pH 8),150mM NaCl,0.1%(V/V)Tween 20,2%SKIM MILK POWDER 中将一抗(anti-TOC75,anti-Bip(agrisera),anti-GFP(abcam))用合适的比 例稀释之后,将PVDF膜孵育1小时。用20mM Tris-HCl(pH 8),150mM NaCl, 0.1%(V/V)Tween 20洗4次,时间分别为15分钟,5分钟,5分钟,5分钟。 在20mM Tris-HCl(pH 8),150mM NaCl,0.1%(V/V)Tween 20,2%SKIM MILK POWDER中将二抗(abmart)用合适的比例稀释之后,将PVDF膜孵育1小时。用 20mM Tris-HCl(pH 8),150mM NaCl,0.1%(V/V)Tween 20洗4次,时间 分别为15分钟,5分钟,5分钟,5分钟。用TanonTM High-sig ECL Western Blotting Substrate显色之后用X光片显影。
叶绿体DNA提取和测序
用DNeasy Plant Maxi Kit(QIAGEN)提取叶绿体DNA,跑胶对提取的叶绿 体DNA进行质量检测确定没有RNA污染和获得的DNA分子量大致范围。然后使 用COVARIS S220把叶绿体DNA片段选择打断至450-600bp,定容体积到60ul, 使用Illumina DNA Sample Preparation Kit加入40ul End Repair Mix,30 度处理30分钟,加入160ul AMPure XP Beads纯化,定容到17.5ul加入12.5ul A-Tailing Mix,37度30分钟,加入2ul DNA Adapter Index,3ul Resuspension Buffer和2.5ul Ligation Mix,30度反应30分钟,加入5ul Stop Ligation Buffer混匀后加入42ul AMPure XP Beads纯化,然后定容至10ul,用Qubit定 量与Agilent 2100Bioanalyzer检测片段大小。最后用illumina HiSeq2500 Rapid模式PE250测序。
实施例1
在烟草里验证叶绿体信号肽infA和非编码RNA能分别把Cas9-GFP融合蛋 白和GFP mRNA带进叶绿体
构建35S启动子驱动的带有叶绿体信号肽infA的Cas9和GFP融合蛋白(图 1A);构建35S启动子驱动的连接有非编码RNA的GFP。把这2个载体在烟草里 进行瞬时转化验证,发现由GFP发出的绿色荧光和叶绿素发出的红色荧光在叶 绿体里共定位,证明叶绿体信号肽infA和非编码RNA能分别把Cas9-GFP融合 蛋白和GFP mRNA带进叶绿体(图1B)。
实施例2
在烟草里验证非编码RNA能引导带有Csy4和2个Csy4识别位点的sgRNA 到叶绿体里
构建35S启动子驱动非编码RNA引导的带有Csy4和2个Csy4识别位点的 sgRNA,然后通过瞬时转化烟草。农杆菌转化烟草3天半后,提取烟草的叶绿 体。通过Western blot证明烟草的叶绿体被很好的纯化获得(图2),BiP和Toc75 分别作为细胞质和叶绿体的标记。
接下来对获得的叶绿体提取RNA,然后通过环化逆转录PCR(circularized reverse transcription PCR,cRT-PCR)对sgRNA进行检测。测序结果:在检 测的6个克隆中(各3个克隆),在叶绿体里均分别存在被Csy4切割后形成的 成熟sgRNA(图3)。
实施例3
利用CRISPR在pMSK56植株的叶绿体基因组上进行双链DNA破坏
构建35S启动子驱动的带有叶绿体信号肽infA的Cas9,然后把它与实验 步骤2里构建的35S启动子驱动非编码RNA引导的带有Csy4和2个Csy4识别 位点的sgRNA(图1A)。
在本实施例中,分别选取了2个不同的靶点:sgRNA1(SEQ ID No.:8)和 sgRNA2(SEQ ID No.:9)共转化pMSK56植株。在pMSK56植株的叶绿体基因组 上有aadA16GFP基因,这个基因编码的aadA-GFP融合蛋白在叶绿体里发绿色 荧光,所以每个叶绿体都有来自aadA-GFP的绿色荧光和来自叶绿素的红色荧 光(图1C)。而被这2个载体共转化后的pMSK56植株里,观察到了只有红色荧 光而没有绿色荧光的叶绿体(图1C),这就表明叶绿体上aadA16GFP可能被突变 了,接下来提取了野生型烟草、pMSK56、以及分别转化了35S启动子驱动的带 有叶绿体信号肽infA的Cas9和35S启动子驱动非编码RNA引导的带有Csy4 和2个Csy4识别位点的sgRNA的叶绿体。Western blot证明,转化有Cas9和 sgRNA的pMSK56植株里的aadA-GFP蛋白的数量减少了。
核苷酸序列SEQ ID No.:8
TGCACGACGACATCATTCCGGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTAT CAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTT
核苷酸序列SEQ ID No.:9
AGAAGGTCTTAAAGTCGCCAGTTTTAGAGCTAGAAATAGCAAGTTAAAATAAGGCTAGTCCGTTAT CAACTTGAAAAAGTGGCACCGAGTCGGTGCTTTT
实施例4
利用二代测序技术检测CRISPR在叶绿体基因组上的突变
对实施例3中转化有sgRNA2的pMSK56植物的叶片提取叶绿体,然后用 DNeasy Plant Maxi Kit(Qiagen)提取叶绿体DNA,然后用二代测序技术检测在 报告基因aadA16GFP的突变。测序结果发现在转化有sgRNA2的pMSK56植物的aadA16GFP里有插入DNA片段(图4A,方框内的序列为插入DNA片段),然后 按照相应的序列设计引物PCR验证了这个结果,只有在转化有sgRNA2的pMSK56 植物的叶绿体DNA能够扩增到条带(图4B)。
实施例5
利用CRISPR在pMSK56植株的叶绿体基因组上进行靶向knockdown
构建35S启动子驱动的带有叶绿体信号肽infA的没有核酸酶活性的Cas9 (dCas9:D10A和H840A),和35S启动子驱动非编码RNA引导的带有Csy4和2 个Csy4识别位点的sgRNA(靶向aadA16gfp基因)转化pMSK56植株。发明人 在5个T1代转基因单株里分别运用western blot和实时定量PCR分别检测了 dCas9和gfp基因的表达量。发明人发现dCas9蛋白的表达量与GFP的表达量 呈现负相关(图5),即dCas9蛋白表达量高的植株里GFP的表达量低。
实施例6
利用CRISPR在拟南芥var2植株的叶绿体基因组上进行靶向knockdown, 从而恢复它的叶杂色表型。
构建UBQ启动子驱动的带有叶绿体信号肽infA的没有核酸酶活性的Cas9 (dCas9),和35S启动子驱动非编码RNA引导的带有Csy4和2个Csy4识别 位点的sgRNA转化拟南芥var2植株(图6A)。sgRNA靶向的基因为叶绿体基因 组上的rpl33(图6B)。发明人在T1代转基因植株里发现靶向降低rpl33的 表达可以恢复var2突变介导的叶杂色表型(图7,表1),然后对这些表型恢 复和没有恢复的植物提取RNA进行实时定量PCR检测。发现在rpl33的表达量 在这2类植物里都有降低(图6),表达量较低的植株可以恢复var2突变介导 的叶杂色表型。
表1.var2突变体靶向敲低rpl33后叶杂色表型统计
在本发明提及的所有文献都在本申请中引用作为参考,就如同每一篇文献 被单独引用作为参考那样。此外应理解,在阅读了本发明的上述讲授内容之后, 本领域技术人员可以对本发明作各种改动或修改,这些等价形式同样落于本申 请所附权利要求书所限定的范围。
参考文献
Gómez,G.,and Pallás,V.(2010).Noncoding RNA Mediated Traffic of Foreign mRNA into Chloroplasts Reveals a Novel Signaling Mechanism in Plants.PloS one 5,e12269.
Haurwitz,R.E.,Jinek,M.,Wiedenheft,B.,Zhou,K.,and Doudna, J.A.(2010).Sequence-and Structure-Specific RNA Processing by a CRISPR Endonuclease.Science 329,1355-1358.
Khan,M.S.,and Maliga,P.(1999).Fluorescent antibiotic resistance marker for tracking plastid transformation in higher plants. Nat Biotech 17,910-915.
序列表
<110> 中国科学院上海生命科学研究院
<120> 叶绿体基因组编辑方法
<130> P2017-2453
<150> 201611115749.6
<151> 2016-12-07
<160> 9
<170> PatentIn version 3.5
<210> 1
<211> 883
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 1
tcaacatggt ggagcacgac acacttgtct actccaaaaa tatcaaagat acagtctcag 60
aagaccaaag ggcaattgag acttttcaac aaagggtaat atccggaaac ctcctcggat 120
tccattgccc agctatctgt cactttattg tgaagatagt ggaaaaggaa ggtggctcct 180
acaaatgcca tcattgcgat aaaggaaagg ccatcgttga agatgcctct gccgacagtg 240
gtcccaaaga tggaccccca cccacgagga gcatcgtgga aaaagaagac gttccaacca 300
cgtcttcaaa gcaagtggat tgatgtgata acatggtgga gcacgacaca cttgtctact 360
ccaaaaatat caaagataca gtctcagaag accaaagggc aattgagact tttcaacaaa 420
gggtgatatc cggaaacctc ctcggattcc attgcccagc tatctgtcac tttattgtga 480
agatagtgga aaaggaaggt ggctcctaca aatgccatca ttgcgataaa ggaaaggcca 540
tcgttgaaga tgcctctgcc gacagtggtc ccaaagatgg acccccaccc acgaggagca 600
tcgtggaaaa agaagacgtt ccaaccacgt cttcaaagca agtggattga tgtgatatct 660
ccactgacgt aagggatgac gcacaatccc actatccttc gcaagaccct tcctctatat 720
aaggaagttc atttcatttg gagaggacct cgacctcaac acaacatata caaaacaaac 780
gaatctcaag caatcaagca ttctacttct attgcagcaa tttaaatcat ttcttttaaa 840
gcaaaagcaa ttttctgaaa attttcacca tttacgaacg ata 883
<210> 2
<211> 284
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 2
tgattgatcg atagagctcg aatttccccg atcgttcaaa catttggcaa taaagtttct 60
taagattgaa tcctgttgcc ggtcttgcga tgattatcat ataatttctg ttgaattacg 120
ttaagcatgt aataattaac atgtaatgca tgacgttatt tatgagatgg gtttttatga 180
ttagagtccc gcaattatac atttaatacg cgatagaaaa caaaatatag cgcgcaaact 240
aggataaatt atcgcgcgcg gtgtcatcta tgttactaga tcgg 284
<210> 3
<211> 180
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 3
atgcttcaac tctgctccac tttccgtcct caacttcttc ttccttgtca attccgattt 60
acaaatggcg ttttgattcc ccaaataaac tatgttgcaa gcaattcagt tgtgaatatc 120
cggccaatga tacgatgcca gagagcaagc ggaggaagag gaggagctaa tagaagcaaa 180
<210> 4
<211> 720
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 4
atggtgagca agggcgagga gctgttcacc ggggtggtgc ccatcctggt cgagctggac 60
ggcgacgtaa acggccacaa gttcagcgtg tccggcgagg gcgagggcga tgccacctac 120
ggcaagctga ccctgaagtt catctgcacc accggcaagc tgcccgtgcc ctggcccacc 180
ctcgtgacca ccttcaccta cggcgtgcag tgcttcagcc gctaccccga ccacatgaag 240
cagcacgact tcttcaagtc cgccatgccc gaaggctacg tccaggagcg caccatcttc 300
ttcaaggacg acggcaacta caagacccgc gccgaggtga agttcgaggg cgacaccctg 360
gtgaaccgca tcgagctgaa gggcatcgac ttcaaggagg acggcaacat cctggggcac 420
aagctggagt acaactacaa cagccacaac gtctatatca tggccgacaa gcagaagaac 480
ggcatcaagg tgaacttcaa gatccgccac aacatcgagg acggcagcgt gcagctcgcc 540
gaccactacc agcagaacac ccccatcggc gacggccccg tgctgctgcc cgacaaccac 600
tacctgagca cccagtccgc cctgagcaaa gaccccaacg agaagcgcga tcacatggtc 660
ctgctggagt tcgtgaccgc cgccgggatc actcacggca tggacgagct gtacaagtaa 720
<210> 5
<211> 330
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 5
ttggcgaaac cccatttcga cctttcggtc tcatcagggg tggcacacac caccctatgg 60
ggagaggtcg tcctctatct ctcctggaag gccggagcaa tccaaaagag gtacacccac 120
ccatgggtcg ggactttaaa ttcggaggat tcgtccttta aacgttcctc caagagtccc 180
ttccccaaac ccttactttg taagtgtggt tcggcgaatg taccgtttcg tcctttcgga 240
ctcatcaggg aaagtacaca ctttccgacg gtgggttcgt cgacacctct ccccctccca 300
ggtactatcc cctttccagg atttgttccc 330
<210> 6
<211> 564
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 6
atggaccact atctggacat cagactgagg cccgatcctg agttccctcc cgcccagctg 60
atgagcgtgc tgtttggcaa gctgcatcag gctctggtcg cccaaggcgg agacagaatc 120
ggcgtgtcct tccccgacct ggacgagtcc cggagtcgcc tgggcgagcg gctgagaatc 180
cacgccagcg cagacgatct gcgcgccctg ctggcccggc cttggctgga gggcctgcgg 240
gatcatctgc agtttggcga gcccgccgtg gtgccacacc caacacccta ccgccaggtg 300
agccgcgtgc aggccaagtc aaatcccgag agactgcggc ggaggctgat gaggcgacat 360
gatctgagcg aggaggaggc cagaaagaga atccccgaca cagtggccag agccctggat 420
ctgccatttg tgaccctgcg gagccagagc actggccagc atttcagact gttcatcaga 480
cacgggcccc tgcaggttac agccgaggag ggcggattta catgctatgg cctgtctaaa 540
ggcggcttcg tgccctggtt ctaa 564
<210> 7
<211> 20
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 7
gttcactgcc gtataggcag 20
<210> 8
<211> 100
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 8
tgcacgacga catcattccg gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt 100
<210> 9
<211> 100
<212> DNA
<213> 人工序列(Artificial sequence)
<400> 9
agaaggtctt aaagtcgcca gttttagagc tagaaatagc aagttaaaat aaggctagtc 60
cgttatcaac ttgaaaaagt ggcaccgagt cggtgctttt 100
- 还没有人留言评论。精彩留言会获得点赞!