迈诺醇的生产的制作方法
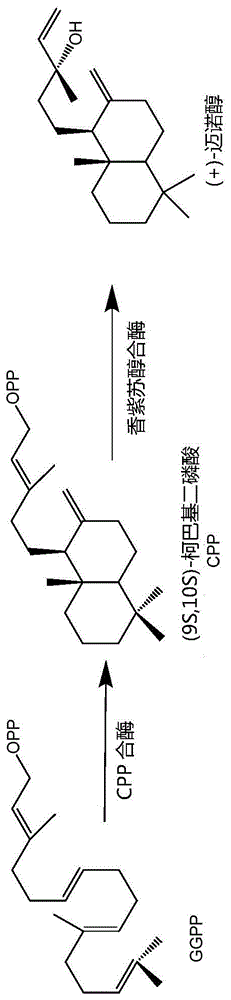
本文提供了使用柯巴基(copalyl)二磷酸合酶和香紫苏醇合酶生产(+)-迈诺醇(manool)的生物化学方法。
背景技术:
:在大多数生物(微生物、动物和植物)中都可以找到萜烯。这些化合物由称为异戊二烯单元的五个碳单元组成,并按其结构中存在的这些单元的数量分类。因此,单萜、倍半萜和二萜分别是含有10、15和20个碳原子的萜烯。例如,倍半萜在植物界广泛存在。许多倍半萜分子因其风味和香味性质以及它们的美容、医疗和抗微生物作用而公知。许多倍半萜烃和倍半萜类化合物已被鉴定出。萜烯的生物合成生产涉及称为萜合酶的酶。这些酶将无环萜烯前体转化成一种或多种萜烯产物。具体而言,二萜合酶通过前体香叶基香叶基二磷酸(ggpp)的环化产生二萜。ggpp的环化通常需要两种酶多肽,在两个连续的酶促反应中组合起作用的i型和ii型二萜合酶。ii型二萜合酶催化由ggpp的末端双键质子化引发的ggpp的环化/重排,导致环状二萜二磷酸酯中间体。然后该中间体被催化电离引发的环化的i型二萜合酶进一步转化。二萜合酶存在于植物和其他生物中,并使用底物如ggpp,但它们具有不同的产物特征。编码二萜合酶的基因和cdna已经被克隆,并且表征了相应的重组酶。柯巴基二磷酸(cpp)合酶和香紫苏醇合酶是植物中存在的酶。因此,需要在生物化学过程中发现和使用这些酶和变体以产生(+)-迈诺醇。技术实现要素:本发明提供一种生产(+)-迈诺醇的方法,包括:a)使香叶基香叶基二磷酸(ggpp)与柯巴基二磷酸(cpp)合酶接触以形成柯巴基二磷酸,其中该cpp合酶包含:i)与从由seqidno:14和seqidno:15构成的群组中选出的多肽具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者ii)与从由seqidno:17和seqidno:18构成的群组中选出的多肽具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者iii)与从由seqidno:20和seqidno:21构成的群组中选出的多肽具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列;和b)使cpp与香紫苏醇合酶接触以形成(+)-迈诺醇;并且c)可选地,分离(+)-迈诺醇。本文提供的方法还包括进一步将(+)-迈诺醇加工成(+)-迈诺醇衍生物。本文还提供了具有cpp合酶活性的多肽,其中该多肽包含:a)与从由seqidno:14和seqidno:15构成的群组中选出的多肽具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者b)与从由seqidno:17和seqidno:18构成的群组中选出的多肽具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者c)与从由seqidno:20和seqidno:21构成的群组中选出的多肽具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列。还提供了具有香紫苏醇合酶活性的多肽,其中该多肽包含与从由seqidno:4、seqidno:5、seqidno:23和seqidno:25构成的群组中选出的多肽具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。本文还提供了编码上述多肽的核酸。本文还提供了编码cpp合酶的核酸,其中该核酸包含与seqidno:13、seqidno:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32中所示的核酸序列具有至少90%、95%、98%、99%或100%序列同一性的核苷酸序列。本文进一步提供了编码香紫苏醇合酶的核酸,其中该核酸包含与seqidno:6、seqidno:24、seqidno:27、seqidno:33或seqidno:34具有至少90%、95%、98%、99%或100%序列同一性的核苷酸序列。还提供了包含上述核酸的表达载体,包含上述核酸或包含该表达载体的非人宿主生物体或细胞,能够生产ggpp的非人宿主生物体或细胞,转化非人宿主生物体或细胞的方法,以及培养非人宿主生物体或细胞以生产(+)-迈诺醇的方法。附图说明图1.从香叶基香叶基二磷酸(ggpp)到(+)-迈诺醇的酶促途径。图2.从香叶基香叶基二磷酸(ggpp)到(+)-迈诺醇和香紫苏醇的酶促途径。图3.ggpp的体外酶促转化的gcms分析。a.使用重组smcps酶。b.使用重组scscs酶。c.在单次测定中将smcps与scscs酶组合。图4.使用表达smcps、scscs和甲羟戊酸途径酶的大肠杆菌细胞产生的(+)-迈诺醇的gcms分析。a.大肠杆菌培养基提取物的总离子色谱图。b.(+)-迈诺醇标准品的总离子色谱图。c.色谱图a中主峰的质谱(保留时间为14.55分钟)。d.(+)-迈诺醇真实标准品的质谱。图5.使用表达甲羟戊酸途径酶、ggpp合酶、scscs和五种不同cpp合酶(来自丹参(salviamiltiorrhiza)的smcps2,来自毛喉鞘蕊花(coleusforskohlii)的cfcps1,来自小麦(triticumaestivum)的tatps1,来自欧夏至草(marrubiumvulgare)的mvcps3和来自迷迭香(rosmarinusofficinalis)的rocps1)的大肠杆菌细胞生产的(+)-迈诺醇的gcms分析。图6.使用表达甲羟戊酸途径酶、ggpp合酶、smcps2和i类二萜合酶(来自烟草(nicotianaglutinosa)的ngscs-del29或来自快乐鼠尾草(salviasclarea)的ssscs)的大肠杆菌细胞生产的(+)-迈诺醇的gcms分析。图7.通过酵母与六种dna片段的共转化在体内构建酿酒酵母表达质粒,该六种dna片段为:a)leu2酵母标记,b)ampr大肠杆菌标记,c)酵母复制起点,d)大肠杆菌复制起点,e)用于共表达crte和所测试的香紫苏醇合酶编码序列之一的片段,和f)用于表达所测试的柯巴基二磷酸(cpp)合酶编码序列之一的片段。图8.使用表达ggpp合酶、scscs和五种不同截短形式的cpp合酶(来自丹参(salviamiltiorrhiza)的smcps2,来自毛喉鞘蕊花(coleus和rskohlii)的cfcps1,来自小麦(triticumaestivum)的tatps1,来自欧夏至草(marrubiumvulgare)的mvcps3和来自迷迭香(rosmarinusofficinalis)的rocps1)的经修饰的酿酒酵母菌株yst045生产的(+)-迈诺醇的gcms分析。具体实施方式定义对于本文和所附权利要求的描述,除非另有说明,否则“或”的使用意味着“和/或”。类似地,各种时态的“含”、“含有”、“包含”和“包括”是可互换的而不是限制性的。应进一步理解,在各种实施方案的描述使用术语“包含”的情况下,本领域技术人员将理解,在一些特定情况下,可以使用“基本上由……组成”或“由……组成”的语言来替代地描述实施方案。除非另有说明,否则以下术语具有归属于它们的含义。术语“多肽”是指连续聚合的氨基酸残基,例如至少15个残基,至少30个残基,至少50个残基的氨基酸序列。在本文提供的一些实施方案中,多肽包含作为酶、其片段或其变体的氨基酸序列。术语“分离的”多肽是指通过本领域已知的任何方法或这些方法(包括重组、生物化学和合成法)的组合从天然环境中取出的氨基酸序列。术语“蛋白”是指任何长度的氨基酸序列,其中氨基酸被共价肽键所连接,并且包括寡肽、肽、多肽和全长蛋白,不管天然生成的还是合成的。术语“生物功能”、“功能”、“生物活性”或“活性”是指cpp合酶和香紫苏醇合酶活性催化(+)-迈诺醇形成的能力。术语“核酸序列”、“核酸”和“多核苷酸”可互换使用,是指核苷酸的序列。核酸序列可以是任何长度的单链或双链脱氧核糖核苷酸或核糖核苷酸,并且包括基因、外显子、内含子、有义和反义互补序列、基因组dna、cdna、mirna、sirna、mrna、rrna、trna、重组核酸序列、分离的和纯化的天然产生的dna和/或rna序列、合成的dna和rna序列、片段、引物和核酸探针的编码和非编码序列;以及这些序列的互补序列。本领域技术人员了解,rna的核酸序列与dna序列相同,差异在于胸腺嘧啶(t)被替代为尿嘧啶(u)。“分离的核酸”或“分离的核酸序列”被定义为一种核酸或核酸序列,其所处的环境与天然产生的核酸或核酸序列所处的环境不同。如本文使用的如应用于核酸的术语“天然产生的”是指在自然的细胞中发现的核酸。例如,存在于能够从自然源中分离出的生物体(例如生物体的细胞)中的核酸序列,并且该核酸序列未经人类在实验室进行有意地修饰,那么该核酸序列是天然产生的。本文所用的术语“纯化的”、“基本上纯化的”和“分离的”是指不含通常与本发明化合物天然状态相关的其他不同化合物的状态,因此“纯化的”、“基本上纯化的”和“分离的”对象以重量计包含给定样品的物质的至少0.5%、1%、5%、10%或20%、或至少50%或75%。在一个具体实施方案中,这些术语是指以重量计包含给定样品的物质的至少95%、96%、97%、98%、99%或100%的本发明化合物。如本文所用,术语核酸或蛋白的“纯化的”、“基本上纯化的”和“分离的”,当提及核酸或蛋白时,也指不同于在细胞或生物体中天然存在的纯度或浓度的状态。大于细胞或生物体中天然存在的浓度或浓度的任何程度的纯度或浓度,包括(1)从其他相关结构或化合物中纯化或(2)与结构或化合物的结合(而在细胞或生物体中通常不与其相关联),都属于“分离的”的含义。根据本领域技术人员已知的各种方法和工艺,本文所述的核酸或蛋白,或多类的核酸或蛋白可以分离,或以其他方式与它们通常在性质上不相关的结构或化合物相关联。如本文所用,动词和名词形式的术语“扩增”是指使用任何合适的扩增方法产生或检测如下文详细描述的天然表达的核酸的重组体。例如,本发明提供用于在体内、体外或试管内扩增(例如通过聚合酶链反应,pcr)天然表达的(例如基因组dna或mrna)或重组(例如cdna)本发明核酸的方法和试剂(例如特异性简并寡核苷酸引物对,寡聚dt引物)。“重组核酸序列”是使用实验室方法(分子克隆)将来自多于一个源的遗传物质组合在一起所生成的核酸序列,由此创造出不是天然产生并且不能以其他方式在生物有机体中发现的核酸序列。“重组dna技术”是指一种用于制备重组核酸序列的分子生物学方法,例如描述于由weigel和glazebrook编辑的laboratorymanuals,2002coldspringharborlabpress;和sambrook等,1989coldspringharbor,ny:coldspringharborlaboratorypress。术语“基因”是指一种dna序列,其包含可操作地连接到适当调控区域(例如启动子)的被转录为rna分子(例如细胞中的mrna)的区域。因此,基因可以包含多个可操作地连接的序列,诸如启动子、5’前导序列(包含例如参与翻译初始化的序列)、cdna或基因组dna的编码区、内含子、外显子和/或3’非翻译序列(包含例如转录终止位点)。“嵌合基因”是指通常不能在自然界的物种中找到的任何基因,特别是这样一种基因,其中核酸序列存在一个或多个部分在性质上彼此不相关联。例如,启动子在性质上与转录区的部分或全部或与另一调控区不相关联。术语“嵌合基因”应当被理解为包括表达构建体,其中启动子或转录调控序列被可操作地连接到一个或多个编码序列或反义(即有义链的反向互补链)或反向重复序列(有义和反义,由此rna转录物在转录后形成双链rna)。术语“嵌合基因”还包括通过组合一个或多个编码序列的部分以产生新基因而获得的基因。“3’urt”或“3’非翻译序列”(也称为“3’未翻译区”或“3’末端”)是指在基因编码序列的下游发现的核酸序列,其包含例如转录终止位点和(在大多数但非全部的真核mrna中)多聚腺苷酸化信号诸如aauaaa或其变体。在转录终止后,mrna转录物可以在多聚腺苷酸化信号的下游切去,并且可以添加poly(a)尾,其参与了mrna向翻译位点例如细胞质的运输。“基因的表达”包括基因的转录和mrna向蛋白质的翻译。过表达是指在转基因细胞或生物体中,以mrna、多肽和/或酶活性水平测量的基因产物的生产超过了相似遗传背景的非转化细胞或生物体中的生产水平。如本文中使用的“表达载体”是指这样一种核酸分子,其使用分子生物学方法和重组dna技术工程化以将外来或外源dna递送到宿主细胞中。表达载体典型地包括适合转录核苷酸序列所需的序列。编码区通常编码目的蛋白,但是也可以编码rna,例如反义rna、sirna等。如本文所使用的“表达载体”包括任何线性的或环状的重组载体,包括但不限于病毒载体、噬菌体和质粒。本领域技术人员根据表达系统能够选择适合的载体。在一个实施方案中,表达载体包括本文实施方案的核酸,其可操作地连接到至少一个调控序列,其控制转录、翻译、起始和终止,诸如转录启动子、操纵子和增强子,或mrna核糖体结合位点,并且可选地包括至少一个选择标记。当调控序列功能性地涉及本文实施方案的核酸时,核苷酸序列是“可操作地连接的”。“调控序列”是指这样一种核酸序列,其确定本文实施方案的核酸序列的表达水平、并且能够调控可操作地连接到该调控序列的核酸序列的转录速率。调控序列包含启动子、增强子、转录因子、启动子元件等。“启动子”是指通过提供rna聚合酶用的结合位点以及适合转录所需的其它因子,包括但不限于转录因子结合位点、抑制子和活化子蛋白结合位点,来控制编码序列的表达的核酸序列。术语启动子的含义包括术语“启动子调控序列”。启动子调控序列可以包括可能影响转录、rna加工或相关编码核酸序列的稳定性的上游和下游元件。启动子包括天然来源的和合成的序列。编码核酸序列通常位于启动子相对于以转录起始位点为起始的转录方向的下游。术语“组成型启动子”是指不受调控的启动子,其允许其可操作地连接的核酸序列的持续转录。如本文使用的,术语“可操作地连接”是指处于功能性关系的多核苷酸元件的连接。当核酸与另一核酸序列处于功能性关系时,那么该核酸是“可操作地连接的”。例如,如果启动子或者转录调控序列能够影响编码序列的转录,那么该启动子或者转录调控序列是可操作地连接到该编码序列的。可操作地连接意味着被连接的dna序列通常是邻接的。与启动子序列有关的核苷酸序列相对于要被转化的细胞或生物体,例如宿主细胞、植物细胞、植物或微生物可以是同源或异源来源的。所述序列还可以是完全或部分合成的。不管来源如何,与启动子序列有关的核酸序列将根据在结合到本文实施方案的多肽后所连接的启动子性质而表达或沉默。相关核酸在所有时间或替代地在特定时间在整个生物体中或在特定组织、细胞或细胞室中可以编码需要表达或抑制的蛋白。此种核苷酸序列特别地编码将所需表型性状赋予给由其改变或转化的宿主细胞或生物体的蛋白质。更特别地,相关的核苷酸序列导致在宿主细胞或生物体中(+)-迈诺醇合酶的生产。“靶肽”是指一种氨基酸序列,其将蛋白质或多肽靶向细胞内细胞器(即,线粒体或质体)或细胞外空间(分泌信号肽)。编码靶肽的核酸序列可以被融合到编码蛋白或多肽的氨基末端(例如n-末端)的核酸序列或者可以被用来替换原有的靶多肽。术语“引物”是指短的核酸序列,其被杂交到模板核酸序列并且被用于与该模板互补的核酸序列的聚合。如本文所使用的,术语“宿主细胞”或“经转化的细胞”是指被改变以包藏至少一种核酸分子的细胞(或生物体),例如,编码所需蛋白质或核酸序列的重组基因,其在转录时产生cpp合酶蛋白和/或香紫苏醇合酶蛋白或一起产生(+)-迈诺醇。宿主细胞特别地是细菌细胞、真菌细胞或植物细胞。宿主细胞可以含有已经被整合到该宿主细胞的核或细胞器的基因组中的重组基因。替代地,宿主可以含有染色体外的重组基因。同源序列包括直系同源或旁系同源序列。鉴别直系同源物或旁系同源物的方法包括现有技术中已知且在本文中描述的系统发生学方法、序列相似性和杂交方法。旁系同源物来源于基因复制,其产生具有相似序列和相似功能的两种或更多种基因。旁系同源物典型地聚簇在一起并且通过基因在相关植物物种内的复制而形成。使用成对blast分析或在基因家族的系统发生分析过程中使用程序诸如clustal在类似基因的组中发现旁系同源物。在旁系同源物中,共有序列可被鉴定为其特征在于相关基因中的序列并且具有基因的类似功能。直系同源物或直系同源序列是彼此相似的序列,因为它们发现于由共同的祖先传下的物种中。例如,具有共同祖先的植物物种已经含有相同类似序列和功能的许多酶。本领域技术人员能够识别直系同源序列并预测直系同源物的功能,例如通过使用clustal或blast程序构建一个物种的基因族的系统发生树。术语“选择性标记物”是指在表达后能够被用来选择包括该选择性标记物的一种或多种细胞的任何基因。以下描述了选择性标记物的例子。本领域技术人员了解不同的抗生素、杀真菌剂、营养缺陷型或除草剂选择标记物可适用于不同的目标物种。术语“生物体”是指任何非人多细胞或单细胞生物体。诸如植物或微生物。特别地,微生物是细菌、酵母、藻类或真菌。术语“植物”可互换使用以包括植物细胞,包括植物原生质体、植物组织、产生再生植物或植物部分的植物细胞组织培养物、或植物器官诸如根、茎、叶、芽、花、叶柄、花瓣、花粉、胚珠、胚、块茎、果实、种子、它们的后代等。任何植物均可以用来实施本文实施方案的方法。特定实施方案在一个实施方案中,本文提供了转化宿主细胞或非人生物体的方法,包括用编码具有柯巴基二磷酸合酶活性的多肽的核酸和编码具有香紫苏醇合酶活性的多肽的核酸转化宿主细胞或非人生物体,其中具有柯巴基二磷酸活性的多肽包含:a)与从由seqidno:14和seqidno:15构成的群组中选出的多肽具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者b)与从由seqidno:17和seqidno:18构成的群组中选出的多肽具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者c)与从由seqidno:20和seqidno:21构成的群组中选出的多肽具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,具有香紫苏醇合酶活性的多肽包含与从由seqidno:4、seqidno:5、seqidno:23和seqidno:25构成的群组中选出的多肽具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,本文提供了一种方法,包括培养非人宿主生物体或细胞,其能够生产香叶基香叶基二磷酸(ggpp),并经转化以表达具有柯巴基二磷酸合酶活性的多肽,其中该具有柯巴基二磷酸合酶活性的多肽包含:a)与从由seqidno:14和seqidno:15构成的群组中选出的多肽具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者b)与从由seqidno:17和seqidno:18构成的群组中选出的多肽具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者c)与从由seqidno:20和seqidno:21构成的群组中选出的多肽具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列;并且进一步经转化以表达具有香紫苏醇合酶活性的多肽。特别地,具有香紫苏醇合酶活性的多肽包含与从由seqidno:4、seqidno:5、seqidno:23和seqidno:25构成的群组中选出的多肽肽具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。本文进一步提供了表达载体,其包含编码cpp合酶的核酸,其中该cpp合酶包含多肽,该多肽包含:a)与seqidno:14或seqidno:15具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者b)与seqidno:17或seqidno:18具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者c)与seqidno:20或seqidno:21具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列;并且此外,该表达载体包含编码香紫苏醇合酶的核酸。特别地,香紫苏醇合酶包含与seqidno:4、seqidno:5、seqidno:23或seqidno:25具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个特定的实施方案中,两种酶,即cpp合酶和香紫苏醇合酶,可以在相同细胞中转化的两种不同的载体或质粒上。在另一个实施方案中,这两种酶可以在两种不同细胞中转化的两种不同的载体上或质粒上。本文进一步提供了非人宿主生物体或细胞,其包含或经转化以包藏至少一种编码cpp合酶的核酸,其中该cpp合酶包含:a)包含与seqidno:14或seqidno:15具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列的多肽;或者b)与seqidno:17或seqidno:18具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者c)与seqidno:20或seqidno:21具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列;和至少一种编码香紫苏醇酶的核酸。特别地,香紫苏醇合酶包含与seqidno:4、seqidno:5、seqidno:23或seqidno:25具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。本文进一步提供了非人宿主生物体或细胞,其包含或经转化以包藏至少一种编码cpp合酶的核酸,其中该cpp合酶包含与从由seqidno:1和seqidno:2构成的群组中选出的多肽具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列;和至少一种编码香紫苏醇酶的核酸,其中香紫苏醇合酶包含与从由seqidno:23和seqidno:25构成的群组中选出的多肽具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,编码本文提供的cpp合酶的核酸包含与seqidno:3、seqidno:13、seqidno:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32具有至少90%、95%、98%、99%或100%序列同一性的核苷酸序列。在一个实施方案中,编码本文提供的cpp合酶的核酸包含与seqidno:3、seqidno:13、seqidno:no:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32具有至少95%、98%、99%或100%序列同一性的核苷酸序列。在一个实施方案中,编码本文提供的cpp合酶的核酸包含与seqidno:3、seqidno:13、seqidno:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32具有至少98%、99%或100%序列同一性的核苷酸序列。在一个实施方案中,编码本文提供的cpp合酶的核酸包含与seqidno:3、seqidno:13、seqidno:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32具有99%或100%序列同一性的核苷酸序列。在一个实施方案中,编码本文提供的cpp合酶的核酸包含seqidno:3、seqidno:13、seqidno:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32中所示的核苷酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含:a)与从由seqidno:14和seqidno:15构成的群组中选出的多肽具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者b)与从由seqidno:17和seqidno:18构成的群组中选出的多肽具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者c)与从由seqidno:20和seqidno:21构成的群组中选出的多肽具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:14具有至少55%、60%、65%、70%、75%、80%、85%、90%、95%、98%或99%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:14具有至少60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:14具有至少70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:14具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:14具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含seqidno:14中所示的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:15具有至少55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:15具有至少60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:15具有至少70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:15具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:15具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含seqidno:15中所示的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:17具有至少75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:17具有至少80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:17具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:17具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含seqidno:17中所示的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:18具有至少75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:18具有至少80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:18具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:18具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含seqidno:18中所示的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:20具有至少91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:20具有至少95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:20具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含seqidno:20中所示的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:21具有至少91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:21具有至少95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:21具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含与seqidno:21具有至少99%序列同一性的氨基酸序列。在一个实施方案中,cpp合酶包含多肽,该多肽包含seqidno:21中所示的氨基酸序列。在一个实施方案中,编码香紫苏醇合酶的核酸包含与seqidno:6、seqidno:24、seqidno:27、seqidno:33或seqidno:34具有至少90%、95%、98%、99%或100%序列同一性的核苷酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:4具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:4具有至少95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:4具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:4具有至少99%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含seqidno:4中所示的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:5具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:5具有至少95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:5具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:5具有至少99%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含seqidno:5中所示的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:23具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:23具有至少95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:23具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:23具有至少99%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含seqidno:23中所示的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:25具有至少90%、95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:25具有至少95%、98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:25具有至少98%、99%或100%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含与seqidno:25具有至少99%序列同一性的氨基酸序列。在一个实施方案中,香紫苏醇合酶包含seqidno:25中所示的氨基酸序列。在另一个实施方案中,本文提供了表达载体,其包含至少一种本文所述的核酸。在另一个实施方案中,本文提供了非人宿主生物体或细胞,其包含一种或多种表达载体,该表达载体包含编码如本文所述的cpp合酶的核酸和编码如本文所述的香紫苏醇合酶的核酸。在另一个实施方案中,本文提供了非人宿主生物体或细胞,其包含或经转化以包藏至少一种本文所述的核酸,使得其异源表达或过表达本文所述的至少一种多肽。在一个实施方案中,本发明提供了经转化的细胞或生物体,其中多肽的表达量高于未经转化的相同细胞或生物体。本领域已知有几种方法用于产生转基因宿主生物体或细胞,例如植物、真菌、原核生物或高等真核细胞的培养物。适用于细菌、真菌、酵母、植物和哺乳动物细胞宿主的克隆和表达载体描述于例如pouwelsetal.,cloningvectors:alaboratorymanual,1985,elsevier,newyorkandsambrooketal.,molecularcloning:alaboratorymanual,2ndedition,1989,coldspringharborlaboratorypress。特别是高等植物和/或植物细胞的克隆和表达载体对于技术人员是可用的。参见例如schardletal.,gene,1987,61:1-11。用于将宿主生物体或细胞转化为包藏转基因核酸的方法是本领域技术人员熟悉的。例如,为了产生转基因植物,目前的方法包括:植物原生质体的电穿孔,脂质体介导的转化,农杆菌介导的转化,聚乙二醇介导的转化,粒子轰击,植物细胞的显微注射和使用病毒的转化。在一个实施方案中,将经转化的dna整合到非人宿主生物体和/或细胞的染色体中,从而产生稳定的重组系统。本领域已知的任何染色体整合方法可用于本发明的实践,包括但不限于重组酶介导的盒交换(rmce),病毒位点特异性染色体插入,腺病毒和原核注射。在实施生产(+)-迈诺醇的方法的一个实施方案中,本文提供了制备本发明任一实施方案中所述的至少一种具有cpp合酶活性的多肽和至少一种具有香紫苏醇合酶活性的多肽的方法。。一个实施方案提供了一种生产迈诺醇的方法,包括:a)使香叶基香叶基二磷酸(ggpp)与本文所述的柯巴基二磷酸(cpp)合酶接触以形成柯巴基二磷酸;并且b)使cpp与本文所述的香紫苏醇合酶接触以形成(+)-迈诺醇;其中步骤a)包括培养能够生产ggpp并用本文所述的一种或多种核酸或用本文所述的一种或多种表达载体转化的非人宿主生物体或宿主细胞,以便非人宿主生物体或宿主细胞包藏编码本文所述的具有cpp合酶活性的多肽的核酸和编码如本文所述的具有香紫苏醇合酶活性的多肽的核酸,并表达或过表达该多肽。一个实施方案提供了上述生产迈诺醇的方法,其进一步包括在步骤a)之前,用如下物质转化能够生产ggpp的非人宿主生物体或宿主细胞:a)至少一种编码多肽的核酸,该多肽包含:i)与seqidno:14或seqidno:15具有至少50%、55%、60%、65%、70%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者ii)与seqidno:17和seqidno:18具有至少71%、72%、75%、80%、85%、90%、95%、98%、99%或100%序列同一性的氨基酸序列;或者iii)与seqidno:20或seqidno:21具有至少90%、91%、92%、93%、94%、95%、96%、97%、98%、99%或100%序列同一性的氨基酸序列;并且具有cpp合酶活性,使得所述生物体或细胞表达具有cpp合酶活性的所述多肽;和b)至少一种编码具有本文所述的香紫苏醇合酶活性的多肽的核酸,使得所述生物体或细胞表达具有香紫苏醇合酶活性的所述多肽。在一个实施方案中,能够生产ggpp的非人宿主生物体或宿主细胞包含:a)编码包含seqidno:15的cpp合酶的核酸和编码包含seqidno:5的香紫苏醇合酶的核酸;或者b)编码包含seqidno:18的cpp合酶的核酸和编码包含seqidno:5的香紫苏醇合酶的核酸;或者c)编码包含seqidno:21的cpp合酶的核酸和编码包含seqidno:5的香紫苏醇合酶的核酸;或者d)包含seqidno:16的核酸,其编码cpp合酶,和包含seqidno:6的核酸,其编码香紫苏醇合酶;或者e)包含seqidno:19的核酸,其编码cpp合酶,和包含seqidno:6的核酸,其编码香紫苏醇合酶;或者f)包含seqidno:22的核酸,其编码cpp合酶,和包含seqidno:6的核酸,其编码香紫苏醇合酶;或者g)包含seqidno:26的核酸,其编码cpp合酶,和包含seqidno:27的核酸,其编码香紫苏醇合酶;或者h)包含seqidno:29的核酸,其编码cpp合酶,和包含seqidno:27的核酸,其编码香紫苏醇合酶;或者i)包含seqidno:30的核酸,其编码cpp合酶,和包含seqidno:27的核酸,其编码香紫苏醇合酶;或者j)包含seqidno:31的核酸,其编码cpp合酶,和包含seqidno:27的核酸,其编码香紫苏醇合酶;或者k)包含seqidno:32的核酸,其编码cpp合酶,和包含seqidno:27的核酸,其编码香紫苏醇合酶;或者l)编码包含seqidno:2的cpp合酶的核酸和编码包含seqidno:23的香紫苏醇合酶的核酸;或者m)编码包含seqidno:15的cpp合酶的核酸和编码包含seqidno:23的香紫苏醇合酶的核酸;或者n)编码包含seqidno:18的cpp合酶的核酸和编码包含seqidno:23的香紫苏醇合酶的核酸;或者o)编码包含seqidno:21的cpp合酶的核酸和编码包含seqidno:23的香紫苏醇合酶的核酸;或者p)编码包含seqidno:2的cpp合酶的核酸和编码包含seqidno:25的香紫苏醇合酶的核酸;或者q)编码包含seqidno:15的cpp合酶的核酸和编码包含seqidno:25的香紫苏醇合酶的核酸;或者r)编码包含seqidno:18的cpp合酶的核酸和编码包含seqidno:25的香紫苏醇合酶的核酸;或者s)编码包含seqidno:21的cpp合酶的核酸和编码包含seqidno:25的香紫苏醇合酶的核酸;或者t)包含seqidno:16的核酸,其编码cpp合酶,和包含seqidno:24的核酸,其编码香紫苏醇合酶;或者u)包含seqidno:19的核酸,其编码cpp合酶,和包含seqidno:24的核酸,其编码香紫苏醇合酶;或者v)包含seqidno:22的核酸,其编码cpp合酶,和包含seqidno:24的核酸,其编码香紫苏醇合酶;或者w)包含seqidno:26的核酸,其编码cpp合酶,和包含seqidno:33的核酸,其编码香紫苏醇合酶;或者x)包含seqidno:26的核酸,其编码cpp合酶,和包含seqidno:34的核酸,其编码香紫苏醇合酶。y)包含seqidno:29的核酸,其编码cpp合酶,和包含seqidno:33的核酸,其编码香紫苏醇合酶;或者z)包含seqidno:29的核酸,其编码cpp合酶,和包含seqidno:34的核酸,其编码香紫苏醇合酶。aa)包含seqidno:30的核酸,其编码cpp合酶,和包含seqidno:33的核酸,其编码香紫苏醇合酶;或者bb)包含seqidno:30的核酸,其编码cpp合酶,和包含seqidno:34的核酸,其编码香紫苏醇合酶cc)包含seqidno:31的核酸,其编码cpp合酶,和包含seqidno:33的核酸,其编码香紫苏醇合酶;或者dd)包含seqidno:31的核酸,其编码cpp合酶,和包含seqidno:34的核酸,其编码香紫苏醇合酶;或者ee)包含seqidno:32的核酸,其编码cpp合酶,和包含seqidno:33的核酸,其编码香紫苏醇合酶;或者ff)包含seqidno:32的核酸,其编码cpp合酶,和包含seqidno:34的核酸,其编码香紫苏醇合酶;其中上述核酸序列和/或合酶的组合还包含与本文所述列举的seqidno的变体和各种百分比同一性。在一个实施方案中,本文提供的非人宿主生物体是植物、原核生物或真菌。在一个实施方案中,本文提供的非人宿主是微生物,特别是细菌或酵母。在一个实施方案中,本文提供的细菌是大肠杆菌,酵母是酿酒酵母。在一个实施方案中,本文提供的非人生物体是酿酒酵母。在一个实施方案中,细胞是原核细胞。在其他实施方案中,细胞是细菌细胞。在一个实施方案中,细胞是真核细胞。在一个实施方案中,真核细胞是酵母细胞或植物细胞。在一个实施方案中,可以通过培养本文所述的经转化的细菌或酵母,包括通过发酵来生产迈诺醇,例如描述于paddonetal.,nature,2013,496:528-532。在一个实施方案中,生产(+)-迈诺醇的方法生产出纯度为至少98.5%的(+)-迈诺醇。在另一个实施方案中,本文提供的方法还包括使用本领域公知的方法使用化学或生物化学合成或两者的组合将(+)-迈诺醇加工成衍生物。在一个实施方案中,(+)-迈诺醇衍生物从由烃、醇、缩醛、醛、酸、醚、酮、内酯、乙酸酯和酯构成的群组中选出。根据本发明的任何实施方案,所述(+)-迈诺醇衍生物是可选地包含一个、两个或三个氧原子的c10至c25化合物。在进一步的实施方案中,衍生物选自乙酸迈诺醇酯(乙酸(3r)-3-甲基-5-[(1s,4as,8as)-5,5,8a-三甲基-2-甲亚基十氢-1-萘基]-1-戊烯-3-基酯)、柯巴醇((2e)-3-甲基-5-[(1s,4as,8as)-5,5,8a-三甲基-2-甲亚基十氢-1-萘基]-2-戊烯-1-醇)、乙酸柯巴醇酯(乙酸(2e)-3-甲基-5-[(1s,4as,8as)-5,5,8a-三甲基-2-甲亚基十氢-1-萘基]-2-戊烯-1-基酯)、柯巴醛((2e)-3-甲基-5-[(1s,4as,8as)-5,5,8a-三甲基-2-甲亚基十氢-1-萘基]-2-戊烯醛)、(+)-迈诺醇氧基((+)-manooloxy)(4-[(1s,4as,8as)-5,5,8a-三甲基-2-甲亚基十氢-1-萘基]-2-丁酮)、z-11((3s,5ar,7as,11as,11br)-3,8,8,11a-四甲基十二氢-3,5a-环氧萘并[2,1-c]氧杂七环)、γ-ambrol(2-[(1s,4as,8as)-5,5,8a-三甲基-2-甲亚基十氢-1-萘基]乙醇)和(降龙涎香醚,(3ar,5as,9as,9br)-3a,6,6,9a-四甲基十二氢萘并[2,1-b]呋喃)。在另一个实施方案中,本文提供的方法还包括使(+)-迈诺醇与合适的反应体系接触以将所述(+)-迈诺醇转化成合适的(+)-迈诺醇衍生物。所述合适的反应体系可以是酶促性质的(例如需要一种或多种酶)或化学性质的(例如需要一种或多种合成化学品)。例如,可以使用文献中描述的方法(例如美国专利号7,294,492中所述)将(+)-迈诺醇通过酶催化转化成迈诺醇氧基或γ-ambrol,其中所述专利通过引用整体并入本文。在又一个实施方案中,(+)-迈诺醇衍生物是柯巴醇及其与c1-c5羧酸形成的酯。在又一个实施方案中,(+)-迈诺醇衍生物是与c1-c5羧酸形成的(+)-迈诺醇酯。在一个实施方案中,(+)-迈诺醇衍生物是柯巴醛。在一个实施方案中,(+)-迈诺醇衍生物是迈诺醇氧基。在又一个实施方案中,(+)-迈诺醇衍生物是z-11。在一个实施方案中,(+)-迈诺醇衍生物是ambrol,或是其及其与c1-c5羧酸形成的酯的混合物,特别是γ-ambrol及其酯。在另一个实施方案中,(+)-迈诺醇衍生物是香紫苏内酯(也称为3a,6,6,9a-四甲基十氢萘并[2,1-b]呋喃-2(1h)-酮及其全部非对映异构体和立体异构体)、3,4a,7,7,10a-五甲基十二氢-1h-苯并[f]色原烯-3-醇或3,4a,7,7,10a-五甲基-4a,5,6,6a,7,8,9,10,10a,10b-十氢-1h-苯并[f]色烯及其所有非对映异构体和立体异构体环酮和开环形式、(1r,2r,4as,8as)-1-(2-羟乙基)-2,5,5,8a-四甲基十氢萘-2-醇dol、γ-ambrol。在实施例中详细描述了如何能获得所述衍生物(例如三烯烃、乙酸酯或柯巴醇)的具体实例。例如,根据ep212254,根据本发明获得的迈诺醇可以被加工成迈诺醇氧基(一种酮,按照已知的方法),然后被加工成ambrol(一种醇)和降龙涎香醚(一种醚)。多肽催化特定倍半萜合成的能力可通过进行本文提供的实施例中详述的酶测定来证实。多肽也意味着包括截短的多肽,条件是它们保持它们的(+)-迈诺醇合酶活性和它们的香紫苏醇合酶活性。如下文所期望的,通过修饰本文所述序列获得的核苷酸序列可以使用本领域已知的任何方法,例如通过引入任何类型的突变,例如缺失、插入或置换突变来进行。这种方法的例子列举在说明书中涉及变体多肽及其制备方法的部分中。两个肽或核苷酸序列之间的同一性百分比是当产生这两个序列的比对时,两个序列中相同的氨基酸或核苷酸残基的数目的函数。相同的残基被定义为两个序列中在比对的给定位置的相同的残基。本文使用的序列同一性的百分比是从最佳比对中通过将两个序列之间相同的残基数除以最短序列中的残基总数并乘以100计算得到的。最佳比对是同一性百分比最高可能性的比对。可以将空位引入到一个或两个序列中的比对的一个或多个位置中以获得最佳比对。然后将这些空位考虑为用于计算序列同一性百分比的不相同的残基。用于确定氨基酸或核酸序列同一性百分比的比对可以使用计算机程序以及例如在互联网上可公开获得的计算机程序以多种方式实现。优选地,可使用设定为默认参数可从nationalcenterforbiotechnologyinformation(ncbi)于http://www.ncbi.nlm.nih.gov/blast/bl2seq/wblast2.cgi获得的blast程序(tatiana等,femsmicrobiollett.,1999,174:247-250,1999)来获得肽或核苷酸序列的最佳比对并计算序列同一性的百分比。要与ggpp体外接触的多肽可以使用标准蛋白或酶提取技术从表达它的任何生物体中提取得到。如果宿主生物体是将本文实施方案的多肽释放到培养基中的单细胞生物体或细胞,那么可以简单地从所述培养基收集所述多肽,例如,通过离心,可选地随后进行洗涤步骤和再悬浮于适合的缓冲液中。在另一个实施方案中,ggpp可以与培养基中的多肽接触,其中多肽可以从宿主生物体、单细胞生物或细胞中释放。如果该生物体或细胞将多肽累积于其细胞中,那么可以通过破裂或裂解该细胞来获得该多肽。在从细胞裂解物中进一步提取多肽时,可以使ggpp与多肽接触,或通过与细胞裂解物接触而不必进行这种提取。根据另一个具体实施方案,上述任何实施方案的方法在体内进行。本文提供的这些实施方案是特别有利的,因为可以在不预先分离多肽的情况下在体内进行该方法。该反应直接发生在经转化以表达所述多肽的生物体或细胞内。所述生物体或细胞旨在“表达”多肽,条件是所述生物体或细胞经转化以包藏编码所述多肽的核酸,这种核酸被转录至mrna并且在该宿主生物体或细胞中发现该多肽。术语“表达”涵盖“异源表达”和“过表达”,后者是指mrna、多肽和/或酶活性的水平高于在未经转化的生物体或细胞中所测量的水平。随后,在致力于描述作为本文具体目的的此种经转化的非人宿主生物体或细胞的说明书部分中,将描述用于转化非人宿主生物体或细胞的适合方法的更详细说明。特定的生物体或细胞,当其天然地产生ggpp或当其不天然地产生ggpp但是在使用本文所述的核酸转化之前或与使用所述核酸一起被转化以生产ggpp时,即“能够产生ggpp”。经转化以比天然存在的生物体或细胞产生更高量的ggpp的生物体或细胞也被“能够生产ggpp的生物体或细胞”所涵盖。已知几种转化生物(例如微生物)以使它们生产ggpp的方法,例如schalketal.,j.am.chem.soc.,2013,134:18900-18903。适合于在体内进行本文实施方案的方法的非人宿主生物体可以是任何非人多细胞或单细胞生物体。在一个具体的实施方案中,用来在体内进行本文实施方案的非人宿主生物体是植物、原核生物或真菌。可以使用任何植物、原核生物或真菌。特别有用的植物是天然产生高量萜烯的那些。在一个更具体的实施方案中,用来在体内进行本文实施方案的方法的非人宿主生物体是微生物。可以使用任何微生物,但根据甚至更具体的实施方案,所述微生物是细菌或酵母。更特别地,所述细菌是大肠杆菌,而所述酵母是酿酒酵母。这些生物体的一些天然不产生ggpp,或者只产生少量的ggpp。为了适合于进行本文实施方案的方法,这些生物体必须经转化以生产所述前体,或经工程化以较大的量生产所述前体。如以上所说明的那样,它们可以在使用根据任一上述实施方案所述的核酸进行修饰之前或同时地被如此转化。在一个实施方案中,用编码本文所述的cpp合酶或其变体的核酸和编码本文所述的香紫苏醇合酶或其变体的核酸转化能够生产ggpp的非人宿主生物体或细胞,其中该能够产生ggpp的非人宿主生物体或细胞已经被工程化以过表达ggpp合酶或用编码ggpp合酶的核酸转化。在一个实施方案中,非人宿主生物体或细胞包含编码ggpp合酶的核酸,编码本文所述的cpp合酶或其变体的核酸,和编码本文所述的香紫苏醇合酶或其变体的核酸,其中至少一种所述核酸对非人宿主生物体或细胞是异源的。也可以使用分离的高等真核细胞来代替完整的生物体作为宿主,以在体内实施本文实施方案的方法。合适的真核细胞可以是任何非人细胞,但特别是植物或真菌细胞。根据另一个实施方案,在本文所述的任何实施方案中使用的或由本文所述的核酸编码的具有cpp合酶活性的多肽可以是通过基因工程获得的变体,条件是所述变体保持其cpp合酶活性。根据另一个实施方案,在本文所述的任何实施方案中使用的或由本文所述的核酸编码的具有香紫苏醇合酶活性的多肽可以是通过基因工程获得的变体,条件是所述变体保持其香紫苏醇合酶活性或具有迈诺醇合酶活性。。如本文所用,多肽旨在作为包含本文鉴定的氨基酸序列的多肽或肽片段,以及截短的或变体的多肽,条件是它们保持其cpp合酶活性及其香紫苏醇合酶活性和/或迈诺醇合酶活性。变体多肽的例子是由交替mrna剪接事件或本文所述多肽的蛋白水解切割产生的天然存在的蛋白。归因于蛋白水解的变化包括例如在不同类型的宿主细胞中表达时n-或c-末端的差异,这是由于从本文实施方案的多肽蛋白水解去除一个或多个末端氨基酸引起的。本文实施方案还涵盖了如下文所描述的,通过天然或人工突变本文实施方案的核酸获得的核酸所编码的多肽。由在氨基和羧基末端融合额外的肽序列产生的多肽变体也可以用在本文实施方案的方法中。特别地,此种融合能够增强多肽的表达,有用于蛋白的纯化或改善多肽在期望环境或表达系统中的酶活性。此种额外的肽序列例如可以是信号肽。因此,本发明包括使用变体多肽(诸如通过与其他寡肽或多肽融合所获得的那些和/或连接到信号肽的那些)的方法。由与另一种功能肽(诸如来自萜生物合成路径的另一种蛋白)融合产生的多肽也可以有利地用于本文实施方案的方法中。变体还可以通过附接与多肽骨架共价或非共价连接的修饰基团而与本文实施方案的多肽不同。该变体还包括通过引入的n-连接或o-连接的糖基化位点和/或添加半胱氨酸残基而与本文所述多肽不同的多肽。技术人员将认识到如何修饰氨基酸序列并保持生物活性。因此,在一个实施方案中,本发明提供了制备如任何上述实施方案中所述的,具有cpp合酶活性或香紫苏醇合酶活性或迈诺醇合酶活性的变体多肽的方法,并包括以下步骤:(a)选择根据上述任一实施方案的核酸;(b)修饰所选择的核酸以获得至少一种突变核酸;(c)用突变核酸序列转化宿主细胞或单细胞生物,以表达由突变核酸序列编码的多肽;(d)筛选具有至少一种修饰特性的多肽;和,(e)可选地,如果多肽不具有所需的变体cpp合酶活性、香紫苏醇合酶活性或迈诺醇合酶活性,则重复方法步骤(a)至(d),直至获得具有所需变体cpp合酶活性、香紫苏醇合酶活性、或迈诺醇合酶活性的多肽;(f)可选地,如果在步骤(d)中鉴定出具有所需变体cpp合酶活性或香紫苏醇合酶活性或迈诺醇合酶活性的多肽,则分离步骤(c)中获得的相应突变核酸。根据一个实施方案,当与具有cpp合酶活性的多肽或香紫苏醇合酶活性组合时制备的变体多肽能够生产(+)-迈诺醇。在步骤(b)中,可以例如通过随机诱变、位点特异性诱变或dna改组产生大量突变核酸序列。基因改组的详细程序见于stemmer,dnashufflingbyrandomfragmentationandreassembly:invitrorecombinationformolecularevolution(procnatlacadsciusa.,1994,91(22):10747-1075)。简而言之,dna改组是指在试管内已知序列的随机重组过程,涉及至少两种选择用于重组的核酸。例如,可以通过合成含有突变序列的寡核苷酸在特定基因座处引入突变,所述突变序列侧翼为能够连接天然序列片段的限制性位点。连接后,得到的重构序列编码具有所需氨基酸插入、取代或缺失的类似物。或者,可以采用寡核苷酸定向的位点特异性诱变方法来提供改变的基因,其中可以通过取代、缺失或插入来改变预定的密码子。可以获得并分离突变核酸,其可以根据标准程序用于转化宿主细胞,例如本实施例中公开的那样。在步骤(d)中,筛选步骤(c)中获得的具有至少一种修饰特性,例如所需的修饰酶活性的多肽。可以筛选表达的多肽的所需酶活性的例子包括增强的或降低的酶活性,如通过km或vmax值、修饰的区域化学或立体化学和改变的底物利用或产物分布所测量的。酶活性的筛选可以根据技术人员熟悉的程序和本实施例中公开的程序进行。步骤(e)提供过程步骤(a)-(d)的重复,其可以优选地并行进行。因此,通过产生大量突变核酸,可以同时用不同的突变核酸转化许多宿主细胞,从而允许随后筛选增加数量的多肽。因此,技术人员可自行决定增加获得所需变体多肽的机会。除了本文公开的序列中显示的基因序列之外,对于本领域技术人员显而易见的是,dna序列多态性可存在于给定群体内,这可导致本文公开的多肽的氨基酸序列的变化。由于天然等位基因变异,这种遗传多态性可能存在于来自不同群体或群体内的细胞中。等位变体还可包括功能等同物。其他实施方案还涉及通过来自具体公开的核酸的此类序列多态性衍生的分子。这些天然变异通常在基因的核苷酸序列或本文公开的多肽的氨基酸序列中产生约1至5%的变化。如上所述,编码本文实施方案的多肽的核酸是修饰非人宿主生物体或在体内进行该方法时使用的细胞的有用工具。因此,本文还提供了编码根据任何上述实施方案的多肽的核酸。本文实施方案的核酸可以定义为包括单链或双链形式的脱氧核糖核苷酸或核糖核苷酸聚合物(dna和/或rna)。术语“核苷酸序列”还应理解为包含多核苷酸分子或寡核苷酸分子,其形式为单独的片段或作为较大核酸的组分。本文实施方案的核酸还包括某些分离的核苷酸序列,包括基本上不含污染内源物质的核苷酸序列。本文实施方案的核酸可被截短,条件是其编码如上所述本文包括的多肽。在一个实施方案中,编码cpp合酶的本文实施方案的核酸可以天然存在于植物中,诸如丹参(salviamiltiorrhiza),或其他物种,例如毛喉鞘蕊花(coleusforskohlii),小麦(triticumaestivum),欧夏至草(marrubiumvulgare)或迷迭香(rosmarinusofficinalis),或者是通过修饰seqidno:3、seqidno:13、seqidno:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32获得。在另一个实施方案中,编码香紫苏醇合酶的本文实施方案的核酸可以天然存在于植物中,诸如快乐鼠尾草(salviasclarea)或其他物种如烟草(nicotianaglutinosa),或者可以通过修饰seqidno:6、seqidno:24、seqidno:27、seqidno:33或seqidno:34获得。突变可以是这些核酸的任何种类的突变,例如点突变、缺失突变、插入突变和/或移码突变。可以制备变体核酸以使其核苷酸序列适应特定的表达系统。例如,如果氨基酸由特定密码子编码,则已知细菌表达系统更有效地表达多肽。由于遗传密码的简并性,多于一个的密码子可以编码相同的氨基酸序列,多个核酸序列可以编码相同的蛋白质或多肽,所有这些dna序列都涵盖在本文的实施方案中。在适当的情况下,可以对编码cpp合酶和香紫苏醇合酶的核酸序列进行优化以提高在宿主细胞中的表达。例如,可以使用宿主特有的密码子合成本文实施方案的核苷酸以改善表达。用于转化适合于在体内实施本文实施方案的方法的宿主生物体或细胞的另一个重要工具是包含根据本文实施方案的任何实施方案的核酸的表达载体。因此,本文还提供了这种载体。经转化以包藏本文实施方案的至少一种核酸以便异源表达或过表达本文实施方案的至少一种多肽的重组非人宿主生物体和细胞,也是实施本文实施方案的方法的非常有用的工具。因此,本文也提供这种非人宿主生物体和细胞。根据任何上述实施方案的核酸可用于转化非人宿主生物体和细胞,并且经表达的多肽可以是任何上述多肽。本文实施方案的非人宿主生物体可以是任何非人多细胞或单细胞生物体。在一个具体实施方案中,非人类宿主生物体是植物、原核生物或真菌。任何植物、原核生物或真菌都适合根据本文提供的方法进行转化。特别有用的植物是天然产生大量萜烯的植物。在更具体的实施方案中,非人宿主生物体是微生物。任何微生物都适用于本文,但是根据甚至更具体的实施方案,所述微生物是细菌或酵母。最特别的是,所述细菌是大肠杆菌,而所述酵母是酿酒酵母。除了完整的生物体,分离的高等真核细胞也可以被转化。作为高等真核细胞,这里是指除酵母细胞之外的任何非人真核细胞。特别的高等真核细胞是植物细胞或真菌细胞。本文提供的实施方案包括但不限于cdna、基因组dna和rna序列。基因(包括本文实施方案的多核苷酸)可以通过本领域已知的方法基于可获得的核苷酸序列信息(诸如在所附的序列表中发现的)进行克隆。这些包括例如设计表示此种基因的侧翼序列的dna引物,所述基因的一种是在有义取向上产生的并且初始化有义链的合成,而另一种以反向互补的方式创造并且产生反义链。诸如在聚合酶链反应中使用的那些热稳定的dna聚合酶通常被用来进行此种实验。或者,代表基因的dna序列可以被化学合成并且随后导入到dna载体分子中,所述dna载体分子可以通过例如相容性细胞(诸如大肠杆菌)来繁殖。本文提供了通过seqidno:3、seqidno:13、seqidno:16、seqidno:19、seqidno:22、seqidno:26、seqidno:29、seqidno:30、seqidno:31或seqidno:32,和seqidno:6、seqidno:24、seqidno:27、seqidno:33或seqidno:34的突变获得的核酸序列。这种突变可以常规进行。本领域技术人员清楚的是,可以将一个或多个核苷酸的突变、缺失、插入和/或取代引入这些dna序列中。本文实施方案的编码cpp合酶和香紫苏醇合酶蛋白的核酸序列可以插入到表达载体中和/或包含在插入于表达载体中的嵌合基因中,以在宿主细胞或宿主生物体中产生cpp合酶和香紫苏醇合酶。用于将转基因插入到宿主细胞基因组中的载体在本领域是公知的,并且包括质粒、病毒、粘粒和人工染色体。其中插入了嵌合基因的二元或共整合载体也用于转化宿主细胞。本文提供的实施方案提供了重组表达载体,其包含编码cpp合酶和香紫苏醇合酶的核酸,其分别与相关的核酸序列例如启动子序列可操作地连接。或者,启动子序列可能已经存在于载体中,使得要被转录的核酸序列插入到启动子序列下游的载体中。载体通常被工程化以具有复制起点、多克隆位点和选择标记。实施例实施例1二萜合酶基因。两种二萜合酶是将香叶基香叶基二磷酸(ggpp)转化为迈诺醇所必需的:ii型和i型二萜合酶。在以下实施例中,选择几种ii型和i型二萜合酶组合并评价迈诺醇的制备。对于ii型合酶,选择了五种柯巴基二磷酸(cpp)合酶:-来自丹参(salviamiltiorrhiza)的smcps,ncbi登录号abv57835.1。-来自毛喉鞘蕊花(coleusforskohlii)的cfcps1,ncbi登录号ahw04046.1。-来自小麦(triticumaestivum)的tatps1,ncbi登录号bah56559.1。-来自欧夏至草(marrubiumvulgare)的mvcps3,ncbi登录号aie77092.1。-来自迷迭香(rosmarinusofficinalis)的rocps1,ncbi登录号ahl67261.1。编码五种cpp合酶的cdna的密码子使用被修饰以在大肠杆菌(dna2.0,menlopark,ca94025)中最佳表达,并且在5′-末端和3′-末端分别添加ndei和kpni限制性位点。此外,设计cdna以表达重组cpp合酶,其具有预测的肽信号的缺失(对于smcps、cfcps1、tatps1、mvcps3和rocps1分别为58、63、59、63和67个氨基酸)。对于i型二萜合酶,使用来自快乐鼠尾草(salviasclarea)(ssscs)的香紫苏醇合酶(ncbi登录号aet21246.1,wo2009095366)。优化cdna的密码子使用用于大肠杆菌表达(dna2.0,menlopark,ca94025),去除前50个n-末端密码子,并在5′-末端和3′-末端分别添加ndei和kpni限制性位点。所有cdna在试管内合成并克隆到pj208或pj401质粒(dna2.0,menlopark,ca94025,usa)中。实施例2表达质粒。用ndei和kpni消化经修饰的smcps编码cdna(smcps2)和香紫苏醇合酶(ssscs)编码cdna(1132-2-5_opt),并连接到petduet-1质粒中,分别提供petduet-smcps2和petduet-1132opt表达质粒。构建另一种质粒以将smcps2和ssscs酶与香叶基香叶基二磷酸(ggpp)合酶共表达。对于ggpp合酶,使用编码ggpp合酶(ncbi登录号aaa24819.1)的成团泛菌(pantoeaagglomerans)(ncbi登录号m38424.1)的crte基因。通过密码子优化合成crte基因,并在3′和5′末端添加ncoi和bamhi限制酶识别位点(dna2.0,menlopark,ca94025,usa),并连接在petduet-1质粒的ncoi和bamhi位点之间,获得petduet-crte质粒。用ndei和kpni消化编码smcps2的cdna,并连接到petduet-1-crte质粒中,从而提供petduet-crte-smcps2构建体。然后使用技术(clontech,takarabioeurope)将编码截短的ssscs的优化的cdna(1132-2-5_opt)引入到petduet-crte-smcps2质粒中。对于这种克隆,使用petduet-1132opt作为模板进行pcr扩增,该pcr扩增使用正向引物smcps2-1132inf_f15′-ctgtttgagccggtcgcctaaggtaccagaaggagataaataatggcgaaaatgaaggagaactttaaacg-3′(seqidno:9)和反向引物1132-pet_inf_r15′-gcagcggtttctttaccagactcgaggtcagaacacgaagctcttcatgtcctct-3′(seqidno:10)。将pcr产物连接到用kpni和xhoi限制酶消化的质粒petduet-crte-smcps2中,并使用dry-downpcr克隆试剂盒(clontech,takarabioeurope),提供新质粒petduet-crte-smcps2-ssscs。在该质粒中,crte基因处于petduet质粒的第一个t7启动子的控制下,编码cpp合酶和香紫苏醇合酶的cdna在第二个t7启动子控制下的双顺反子构建体中组织。将petduet-crte-smcps2-ssscs质粒用作构建携带其他四种cpp合酶编码酶的新表达质粒的模板。使用ndei-kpni限制性消化-连接方法用四种新的cpp合酶编码cdna之一替换smcps2cdna,提供新质粒petduet-crte-cfcps1del63-ssscs,petduet-crte-tatps1de159-ssscs,petduet-crte-mvcps3del63-ssscs和petduet-crte-rocps1de167-ssscs。实施例3在大肠杆菌中的异源表达和酶活性。表达质粒(petduet-smcps2或petduet-1132opt)用于转化b121(de3)大肠杆菌细胞(novagene,madison,wi)。使用经转化细胞的单菌落接种25mllb培养基。在37℃温育5至6小时后,将培养物转移至20℃培养箱中并保持1小时以进行平衡。然后通过加入0.1mmiptg来诱导蛋白质的表达,并将培养物在20℃温育过夜。第二天,通过离心收集细胞,重悬于0.1体积的50mmmopso(3-吗啉代-2-羟基丙磺酸钠盐,3-(n-吗啉基)-2-羟基丙磺酸钠盐)缓冲液中,ph7、10%甘油、1mmdtt并通过超声裂解。通过离心(在20,000g下30分钟)澄清提取物,并将含有可溶性蛋白质的上清液用于进一步的实验。实施例4试管内二萜合酶活性测定。在teflon密封的玻璃试管中进行酶测定,使用50~100μl蛋白提取物,最终体积为1ml的50mmmopsoph7、10%甘油,补充有20mmmgcl2,和50~200μm纯化的香叶基香叶基二磷酸(ggpp)(按照keller和thompson,j.chromatogr645(1),161-167所述制备)。将试管在30℃温育5至48小时,酶产物用一倍体积的戊烷萃取两次。在氮流通下浓缩后,通过gc-ms分析提取物并与对照蛋白的提取物(从用空质粒转化的细胞获得)进行比较。gc-ms分析是在装有db1柱(30m×0.25mm×0.25mm膜厚;agilent)并连接有5975系列质谱仪的agilent6890系列gc系统上进行的。载气是1ml/min的恒定流量的氦气。注射为分流模式,注射器温度设定在260℃,烘箱温度程序升温以10℃/min从100℃升至225℃,然后以30℃/min升至280℃。根据真实标准的保留指数和质谱的一致性来确认产物的特性。在这些条件下,用来自质粒petduet-smcps2或petduet-1132opt(分别异源表达smcps或scscs酶)转化的大肠杆菌细胞的重组蛋白,在溶剂提取物中未检测到二萜分子的产生(在这些条件下没有检测到含二磷酸的二萜)。然后进行类似的测定,但将单次测定中含有重组smcps和ssscs的2种蛋白质提取物组合。在这些测定中,形成一种主要产物,并通过质谱和保留指数与真实标准的匹配鉴定为(+)-迈诺醇(图3)。该实验证明,香紫苏醇合酶可与cpp合酶一起使用以产生迈诺醇。实施例5使用大肠杆菌细胞进行体内迈诺醇生产。使用大肠杆菌细胞来评估使用全细胞培养物的迈诺醇的体内生产。插入实施例2中描述的共表达质粒中的crte基因编码具有ggpp合酶活性的酶,其使用法呢基二磷酸(fpp)产生香叶基香叶基二磷酸(ggpp)。为了增加内源性ggpp库的水平并因此提高细胞中二萜的生产力,在相同的细胞中共表达了导致fpp的异源完整甲羟戊酸途径。该途径的酶使用含有在两个启动子控制下的两个操纵子中组织的所有基因的单一质粒来表达。该表达质粒的构建描述于专利申请wo2013064411或schalk等(j.am.chem.soc.,2013,134:18900-18903)。由大肠杆菌乙酰乙酰-coa硫解酶(atob)、金黄色葡萄球菌hmg-coa合酶(mvas)、金黄色葡萄球菌hmg-coa还原酶(mvaa)和酿酒酵母fpp合酶(erg20)基因组成的第一合成操纵子在试管内合成(dna2.0,menlopark,ca,usa)并连接到ncoi-bamhi消化的pacycduet-1载体(invitrogen)中,产生pacyc-29258。从肺炎链球菌(atccbaa-334)的基因组dna扩增出含有甲羟戊酸激酶(mvak1)、磷酸甲羟戊酸激酶(mvak2)、二磷酸甲羟戊酸脱羧酶(mvad)和异戊烯基二磷酸异构酶(idi)的第二操纵子,并连接到pacyc-29258的第二多克隆位点中,提供质粒pacyc-29258-4506。因此该质粒含有编码从乙酰辅酶a到fpp的生物合成途径的所有酶的基因。用质粒pacyc-29258-4506和选自petduet-crte-smcps2-sssc、petduet-crte-cfcps1del63-ssscs、petduet-crte-tatps1del59-ssscs、petduet-crte-mvcps3del63-ssscs或petduet-crte-rocps1del67-ssscs的一种质粒共转化krx大肠杆菌细胞(promega)。在羧苄青霉素(50μg/ml)和氯霉素(34μg/ml)lb-琼脂糖平板上选择经转化的细胞。使用单菌落接种5ml补充有相同抗生素的液体lb培养基。培养物在37℃温育过夜。第二天,用0.2ml过夜培养物接种2ml添加有相同抗生素的tb培养基。在37℃温育6小时后,将培养物冷却至28℃,向各试管中加入0.1mmiptg,0.2%鼠李糖和1∶10体积的癸烷。培养物在28℃下温育48小时。然后将培养物用2倍体积的mtbe(甲基叔丁基醚)提取两次,将有机相浓缩至500μl,并如上面实施例4中所述通过gc-ms进行分析,不同之处在于烘箱温度于100℃保持1分钟,接着以10℃/分钟的温度梯度升至220℃并以20℃/分钟升至3000℃。在这些培养条件下,用ii型二萜合酶和快乐鼠尾草香紫苏醇合酶(ssscs)的每种组合生产迈诺醇(图4和图5)。所生产的二萜化合物的量使用内标(α-长叶蒎烯)来定量。下表显示了当scscs与各种ii型二萜合酶结合时,相对于smcps/ssscs组合所生产的迈诺醇的量(在这些实验条件下,表达smcps和ssscs的细胞生产的迈诺醇浓度为300~500mg/l(图4))。在这些条件下,所生产的迈诺醇的最高相对量是以tatps1del59组合产生的。ii型二萜合酶i型二萜合酶所生产的迈诺醇的相对量smcps2scscs100cfcps1del63scscs125.3tatps1del59scscs139.4mvcps3del63scscs14.9rocps1del67scscs77.7实施例6使用重组细胞生产(+)-迈诺醇、纯化和nmr分析。使用smcps/ssscs酶组合在实施例5中描述的条件下制备1升大肠杆菌培养物,不同之处在于用50g/lamberlitexad-4代替癸烷有机相用于固相萃取。过滤培养基以回收树脂。然后用3倍柱体积的水洗涤树脂,并用3倍柱体积的mtbe洗脱。然后通过硅胶快速色谱法使用由庚烷:mtbe为8:2(v/v)组成的流动相进一步纯化产物。使用brukeravance500mhz光谱仪通过1h-和13c-nmr确认迈诺醇的结构。使用perkin-elmer241旋光仪测量旋光度,[α]d20=+26.9°(0.3%、chcl3)的值证实了(+)-迈诺醇的生成。实施例7使用来自烟草(nicotianaglutinosa)的香紫苏醇合酶在大肠杆菌细胞中体内生产迈诺醇。来自植物烟草(nicotianaglutinosa)的香紫苏醇合酶描述于wo2014/022434中,并显示由赖百当稀二醇(labdenediol)二磷酸(lpp)生产香紫苏醇。评估了wo2014/022434中描述的两种香紫苏醇合酶,ngscs-del29(对应于wo2014/0224中的seqidno:78)和ngscs-del38(对应于wo2014/022434的seqidno:40),在与实施例5类似的条件下生产(+)-迈诺醇。设计编码ngscs-del29的edna,其密码子使用对大肠杆菌表达优化,并且分别在5′-末端和3′-末端包括kpni和xhoi位点。该dna通过dna2.0(newark,ca94560)合成。将petduet-crte-smcps2-ssscs质粒(实施例2)用作构建新表达质粒的模板。用kpni和xhoi限制性位点消化petduet-crte-smcps2-ssscs质粒,用ngscs-del29cdna替换ssscscdna,得到新的petduet-crte-smcps2-del29质粒。用质粒pacyc-29258-4506(实施例5)和petduet-crte-smcps2-del29质粒共转化krx大肠杆菌细胞(promega)。如实施例5中所述,选择经转化的细胞并在生产二萜的条件下培养。使用gc-ms分析来评估二萜的产生,并使用内标(α-长叶蒎烯)定量所生产的二萜化合物。使用二萜合酶smcps2和ngscs-del29的新组合,通过经转化的大肠杆菌细胞生产迈诺醇(图6)。在所用的实验条件下,二萜合酶smcps2和ngscs-del38的组合不产生迈诺醇。因此,所测试的烟草(nicotianaglutinosa)香紫苏醇合酶中的至少一种也可用于从cpp生产迈诺醇。然而,使用烟草(nicotianaglutinosa)合酶生产的量远低于ssscs合酶(见下表)。ii型二萜合酶i型二萜合酶所生产的迈诺醇的相对量smcps2ssscs100smcps2ngscs-del293.1实施例8根据以下实验部分,将以上实施例中获得的迈诺醇转化为其酯(在下文中作为转化为其乙酸酯的例子):根据文献(g.ohloff,helv.chim.acta41,845(1958)),用20.0g(0.25摩尔)乙酰氯在100m1的二甲基苯胺中在室温下处理32.0g(0.11摩尔)纯结晶(+)-迈诺醇5天。将混合物另外在50℃下加热7小时以达到100%的转化率。冷却后,反应混合物用乙醚稀释,依次用10%h2so4、nahco3水溶液和水洗至中性。干燥(na2so4)并浓缩后,蒸馏产物(球对球,b.p.=160°,0.1毫巴),得到20.01g(79.4%)的乙酸迈诺醇酯,其未经进一步纯化即使用。ms:m+332(0);m/e:272(27),257(83),137(62),95(90),81(100).1h-nmr(cdcl3):0.67,0.80,0.87,1.54and2.01(5s,3h各自),4.49(s,1h),4.80(s,1h),5.11(m,1h),5.13(m,1h),5.95(m,1h).13c-nmr(cdcl3):14.5(q),17.4(t),19.4(t),21.7(q),22.2(q),23.5(q),24.2(t),33.5(s),33.6(t),38.3(t),39.0(t),39.3(t),39.8(s),42.2(t),55.6(d),57.2(t),83.4(s),106.4(t),113.0(t),142.0(d),148.6(s),169.9(s).实施例9根据以下实验部分,将上述实施例中得到的乙酸迈诺醇酯转化为其三烯(在下文中作为转化为香紫苏烯(sclarene)和(z+e)泪柏烯((z+e)-biformene)的例子):在室温下向0.4g乙酸迈诺醇酯在4ml环己烷中的溶液中加入0.029g(0.05当量)bf3.acoh络合物。在室温下15分钟后,将反应用nahco3水溶液淬灭并用水洗至中性。gc-ms分析显示只有被鉴定为香紫苏烯、(z)和(e)-泪柏烯的烃。没有检测到乙酸柯巴醇酯。另一个具有更多催化剂(0.15当量)的试验给出了相同的结果。·香紫苏烯:ms:m+272(18);m/e:257(100),149(15),105(15).·(z)和(e)-泪柏烯(相同光谱:ms:m+272(29);m/e:257(100),187(27),161(33),105(37).实施例10根据以下实验部分,将以上实施例中获得的迈诺醇转化为柯巴基酯(下文作为转化为乙酸酯的例子):在室温下向0.474g(0.826毫摩尔,0.27当量)bf3.acoh在100毫升环己烷中的溶液中加入4.4g乙酸酐和12.1g乙酸。在室温下,加入10.0g(33毫摩尔)在15ml环己烷中的纯结晶迈诺醇(缓慢放热),并用水浴将温度保持在室温。在室温下搅拌30分钟后,gc对照显示没有起始材料。将反应混合物用300ml饱和nahco3水溶液淬灭,并照常处理。通过快速色谱(sio2,戊烷/乙醚95:5)和球对球蒸馏(eb.=130°,0.1毫巴)纯化粗混合物(9.9g),得到4.34g(37.1%)的(z)和(e)-柯巴基乙酸酯的27/73混合物。·(z)-柯巴基乙酸酯:ms:m+332(0);m/e:317(2),272(35)=,257(100),137(48),95(68),81(70).1h-nmr(cdcl3):0.67,0.80,0.871.76and2.04(5s,3heach),4.86(s,1h),5.35(t:j=6hz,1h).·(e)-柯巴基乙酸酯:ms:m+332(0);m/e:317(2),272(33)=,257(100),137(54),95(67),81(74).1h-nmr(cdcl3):0.68,0.80,0.871.70and2.06(5s,3heach),4.82(s,1h),5.31(t:j=6hz,1h).13c-nmr(cdcl3):(在(z/e)混合物上记录的光谱,仅给出显著信号):61.4(t),106.2(t),117.9(d),143.1(s),148.6(s),171.1(s).实施例11根据以下实验部分,将上述实施例中得到的柯巴基乙酸酯转化为柯巴醇:将柯巴基乙酸酯(4.17g,12.5毫摩尔)、koh粒料(3.35g,59.7毫摩尔)、水(1.5g)和etoh(9.5ml)混合在一起,并在50℃下搅拌3小时。在通常的处理后,得到3.7g粗产物(z+e)-柯巴醇,并通过快速色谱(sio2,戊烷/乙醚7∶2)纯化,蒸发溶剂后,进行球对球蒸馏(eb=170°,0.1毫巴)提供3.25g(92%)的(z)和(e)-柯巴醇的27/73混合物。·(z)-柯巴醇ms:m+290(3);m/e:275(18),272(27),257(82),137(71),95(93),81(100),69(70).1h-nmr(cdcl3):0.67,0.80,0.87and1.74(4s,3h各自);4.06(m,2h),4.55(s,1h),4.86(s,1h),5.42(t:j=6hz,1h).·(e)-柯巴醇ms:m+290(3);m/e:275(27),272(22),257(75),137(75),95(91),81(100),69(68).1h-nmr(cdcl3):0.68,0.80,0.87and1.67(4s,3h各自);4.15(m,2h),4.51(s,1h),4.83(s,1h),5.39(t,j=6hz,1h)13c-nmr(cdcl3):(在(z/e)混合物上记录的光谱,仅给出显著信号):59.4(t),106.2(t),123.0(d),140.6(s),148.6(s).实施例12使用cpp合酶和香紫苏醇合酶的不同组合在酿酒酵母细胞中体内生产迈诺醇。评估i类和ii类二萜合酶的不同组合在酿酒酵母细胞中迈诺醇的生产。对于ii类二萜合酶,选择了五种cpp合酶:-来自丹参(salviamiltiorrhiza)的smcps,ncbi登录号abv57835.1。-来自毛喉鞘蕊花(coleusforskohlii)的cfcps1,ncbi登录号ahw04046.1。-来自小麦(triticumaestivum)的tatps1,ncbi登录号bah56559.1。-来自欧夏至草(marrubiumvulgare)的mvcps3,ncbi登录号aie77092.1。-来自迷迭香(rosmarinusofficinalis)的rocps1,ncbi登录号ahl67261.1。对于i类,选择了来自烟草(nicotianaglutinosa)的两种推定的香紫苏醇合酶和来自快乐鼠尾草(salviasclarea)的一种:-ngscs-del38(对应于wo2014/022434的seqidno:40)。-ngscs-del29(对应于wo2014/022434的seqidno:78)。-来自快乐鼠尾草(salviasclarea)的ssscs,ncbi登录号aet21246.1。编码不同cpp合酶的dna的密码子使用被修饰以在酿酒酵母中最佳表达。此外,设计dna序列以表达重组cpp合酶,其具有预测的肽信号的缺失(对于smcps、cfcps1、tatps1、mvcps3和rocps1分别为58、63、59、63和67个氨基酸)。ngscs-del38、ngscs-del29和sascsdna序列也针对酿酒酵母表达进行密码子优化。为了在酿酒酵母中表达不同基因,使用酵母内源同源重组体在体内构建一组质粒,如先前描述于kuijpersetal.,microbcellfact.,2013,12∶47。每种质粒由六个dna片段组成,用于酿酒酵母共转化。这些片段是:a)leu2酵母标记,使用引物5′aggtgcagttcgcgtgcaattataacgtcgtggcaactgttatcagtcgtaccgcgccattcgactacgtcgtaaggcc-3′(seqidno:44)和5′tcgtggtcaaggcgtgcaattctcaacacgagagtgattcttcggcgttgttgctgaccatcgacggtcgaggagaactt-3′(seqidno:45)通过pcr用质粒pesc-leu(agilenttechnologies,california,usa)作为模板构建;b)ampr大肠杆菌标记,使用引物5′-tggtcagcaacaacgccgaagaatcactctcgtgttgagaattgcacgccttgaccacgacacgttaagggattttggtcatgag-3′(seqidno:37)和5′-aacgcgtaccctaagtacggcaccacagtgactatgcagtccgcactttgccaatgccaaaaatgtgcgcggaaccccta-3′(seqidno:38)通过pcr用质粒pesc-ura作为模板构建;c)酵母复制起点,使用引物5′-ttggcattggcaaagtgcggactgcatagtcactgtggtgccgtacttagggtacgcgttcctgaacgaagcatctgtgcttca-3’(seqidno:39)和5′-ccgagatgccaaaggataggtgctatgttgatgactacgacacagaactgcgggtgacataatgatagcattgaaggatgagact-3’(seqidno:40)通过pcr用pesc-ura作为模板获得;d)大肠杆菌复制起点,使用引物5′-atgtcacccgcagttctgtgtcgtagtcatcaacatagcacctatcctttggcatctcggtgagcaaaaggccagcaaaagg-3′(seqidno:41)和5′-ctcagatgtacggtgatcgccaccatgtgacggaagctatcctgacagtgtagcaagtgctgagcgtcagaccccgtagaa-3′(seqidno:42)通过pcr用质粒pesc-ura作为模板获得;e)由片段“d”的最后60个核苷酸,酵母基因pgk1的终止密码子下游200个核苷酸,ggpp合酶编码序列crte(来自成团泛菌,ncbi登录号m38424.1)密码子优化其在酿酒酵母中的表达,gal10/gal1的双向酵母启动子,所测试的香紫苏醇合酶编码序列之一,酵母基因cyc1的终止密码子下游200个核苷酸和序列5′-attcctagtgacggccttgggaactcgatacacgatgttcagtagaccgctcacacatgg-3′(seqidno:43)组成的片段,该片段通过dna合成(dna2.0,menlopark,ca94025)获得,以及f)由片段“e”的最后60个核苷酸,酵母基因cyc1的终止密码子下游200个核苷酸,所测试的cpp合酶编码序列之一,gal10/gal1的双向酵母启动子和对应于片段“a”开头的60个核苷酸组成的片段,该片段通过dna合成(dna2.0,menlopark,ca94025)获得。总共构建了15种质粒,其涵盖了上面列出的i类和ii类二萜合酶的所有可能组合。下表显示了所有质粒。质粒名称ii类二萜合酶i类二萜合酶nmsmcps2ssscscfcfcps1del63ssscsmvmvcps3del63ssscsrorocps1del67ssscstatatps1del59ssscsnt_smsmcps2ngscs-del38nt_cfcfcps1del63ngscs-del38nt_mvmvcps3del63ngscs-del38nt_rorocps1del67ngscs-del38nt_tatatps1del59ngscs-del38nt2_smsmcps2ngscs-del29nt2_cfcfcps1del63ngscs-del29nt2_mvmvcps3del63ngscs-del29nt2_rorocps1del67ngscs-del29nt2_tatatps1del59ngscs-del29为了增加酿酒酵母细胞中内源性法呢基二磷酸(fpp)库的水平,参与甲羟戊酸途径的所有酵母内源基因的额外拷贝,从编码乙酰-coac-乙酰转移酶的erg10到编码fpp合酶的erg20,在半乳糖诱导型启动子的控制下,整合到酿酒酵母菌株cen.pk2-1c(euroscarf,frankfurt,germany)的基因组中,类似于paddonetal.,nature,2013,496:528-532中所述。简而言之,三个盒分别整合在leu2、trp1和ura3基因座中。包含基因erg20和截短的hmg1(thmg1)的第一盒,如donaldetal.,procnatlacadsciusa,1997,109:e111-8中所述,在双向启动子gal10/gal1和基因erg19和erg13的控制下,并在gal10/gal1启动子控制下,该盒侧接了对应于leu2的上游和下游部分的两个100核苷酸区域。第二盒,其中基因idi1和thmg1在gal10/gal1启动子和基因erg13的控制下,在gal7启动子区域的控制下,该盒侧接了对应于trp1的上下游部分的两个100核苷酸区域。具有基因erg10、erg12、thmg1和erg8的第三盒,均在gal10/gal1启动子的控制下,该盒侧接了对应于ura3的上游和下游部分的两个100核苷酸区域。三个盒中的所有基因包括其自身终止子区的200个核苷酸。另外,如在griggsandjohnston,procnatlacadsciusa,1991,88:8597-8601中所述,在其自身启动子的突变形式的控制下的gal4的额外拷贝整合在erg9启动子区的上游。此外,erg9的内源启动子被ctr3的酵母启动子区域替换,产生菌株yst035。最后,yst035与菌株cen.pk2-1d(euroscarf,frankfurt,germany)结合,获得命名为yst045的二倍体菌株。用体内质粒装配所需的上述片段转化yst045。如gietzandwoods,methodsenzymol.,2002,350:87-96中所述,用乙酸锂方案进行酵母转化。将转化混合物涂布在含有6.7g/l无氨基酸的酵母氮碱(bddifco,newjersey,usa),1.6g/l不含亮氨酸的滴注补充物(sigmaaldrich,missouri,usa),20g/l葡萄糖和20g/l琼脂的smleu培养基上。将板在30℃下温育3至4天。如westfalletal.,procnatlacadsciusa,2012,109:e111-118中所述,使用单细胞在培养物中生产迈诺醇。在这些培养条件下,一些ii型和i型二萜合酶的组合产生了迈诺醇。使用gc-ms分析来评估迈诺醇的生产并使用内标进行定量。下表显示了相对于smcps/ssscs组合所生产的迈诺醇的量(在这些实验条件下,表达smcps和ssscs的细胞所生产的迈诺醇浓度为100至250mg/l,为所生产的迈诺醇的最高量)。ii类二萜合酶i类二萜合酶所生产的迈诺醇的相对量smcps2ssscs100cfcps1del63ssscs67mvcps3del63ssscs1rocps1del67ssscs29tatps1del59ssscs16smcps2ngscs-del380cfcps1del63ngscs-del380mvcps3del63ngscs-del380rocps1del67ngscs-del380tatps1del59ngscs-del380smcps2ngscs-del290cfcps1del63ngscs-del290mvcps3del63ngscs-del290rocps1del67ngscs-del290tatps1del59ngscs-del290序列表seqidno:1smcps,来自丹参(salviamiltiorrhiza)的全长柯巴基二磷酸合酶seqidno:2smcps2,来自丹参(s.miltiorrhiza)的截短的柯巴基二磷酸合酶seqidno:3smcps2opt,经优化的用于大肠杆菌(e.coli)表达的编码smcps2的cdnaseqidno:4来自快乐鼠尾草(salviasclarea)的全长香紫苏醇合酶seqidno:5来自快乐鼠尾草(salviasclarea)的截短的香紫苏醇合酶(ssscs)seqidno:61132-2-5_opt,经优化的用于大肠杆菌(e.coli)表达的编码来自快乐鼠尾草(salviasclarea)的截短的香紫苏醇合酶的cdnaseqidno:7来自成团泛菌(pantoeaagglomerans)的ggpp合酶seqidno:8crteopt,经优化的编码来自成团泛菌(pantoeaagglomerans)的ggpp合酶的cdnaseqidno:9正向引物smcps2-1132inf_f1seqidno:10反向引物1132-pet_inf_r1seqidno:11cfcps1,来自毛喉鞘蕊花(coleusforskohlii)的全长柯巴基二磷酸合酶seqidno:12cfcps1-del63,来自毛喉鞘蕊花(coleusforskohlii)的截短的柯巴基二磷酸合酶seqidno:13经优化的用于大肠杆菌(e.coli)表达的编码cfcps1-del63的cdnaseqidno:14tatps1,来自小麦(triticumaestivum)的全长柯巴基二磷酸合酶seqidno:15tatps1-del59,来自小麦(triticumaestivum)的截短的柯巴基二磷酸合酶seqidno:16经优化的用于大肠杆菌(e.coli)表达的编码tatps1-del59的cdnaseqidno:17mvcps3,来自欧夏至草(marrubiumvulgare)的全长柯巴基二磷酸合酶seqidno:18mvcps3-del63,来自欧夏至草(marrubiumvulgare)的截短的柯巴基二磷酸合酶seqidno:19经优化的用于大肠杆菌(e.coli)表达的编码mvcps3-del63的cdnaseqidno:20rocps1,来自迷迭香(rosmarinusofficinalis)的全长柯巴基二磷酸合酶seqidno:21rocps1-del67,来自迷迭香(rosmarinusofficinalis)的截短的柯巴基二磷酸合酶seqidno:22经优化的用于大肠杆菌(e.coli)表达的编码rocps1-del67的cdnaseqidno:23ngscs-del29,来自烟草(nicotianaglutinosa)的截短的推定香紫苏醇合酶seqidno:24经优化的用于大肠杆菌(e.coli)表达的编码ngscs-del29的cdnaseqidno:25ngscs-del38,来自烟草(nicotianaglutinosa)的推定香紫苏醇合酶seqidno:26经优化的用于酿酒酵母(saccharomycescerevisiae)表达的编码smcps2的cdnaseqidno:27经优化的用于酿酒酵母(s.cerevisiae)表达的编码来自快乐鼠尾草(salviasclarea)的截短的ssscs的cdnaseqidno:28经优化的用于酿酒酵母(s.cerevisiae)表达的编码来自成团泛菌(pantoeaagglomerans)的ggpp合酶的cdnaseqidno:29经优化的用于酿酒酵母(s.cerevisiae)表达的编码cfcps1-del63的cdnaseqidno:30经优化的用于酿酒酵母(s.cerevisiae)表达的编码tatps1-del59的cdnaseqidno:31经优化的用于酿酒酵母(s.cerevisiae)表达的编码mvcps3-del63的cdnaseqidno:32经优化的用于酿酒酵母(s.cerevisiae)表达的编码rocps1-del67的cdnaseqidno:33经优化的用于酿酒酵母(s.cerevisiae)表达的编码ngscs-del29的cdnaseqidno:34经优化的用于酿酒酵母(s.cerevisiae)表达的编码ngscs-del38的cdnaseqidno:35用于构建片段“a”的引物(ura3酵母标记)seqidno:36用于构建片段“a”的引物(ura3酵母标记)seqidno:37用于构建片段“b”的引物(ampr大肠杆菌标记)seqidno:38用于构建片段“b”的引物(ampr大肠杆菌标记)seqidno:39用于构建片段“c”的引物(酵母复制起点)seqidno:40用于构建片段“c”的引物(酵母复制起点)seqidno:41用于构建片段“d”的引物(大肠杆菌复制起点)seqidno:42用于构建片段“d”的引物(大肠杆菌复制起点)seqidno:43通过dna合成获得的片段“d”的一部分seqidno:44用于构建片段“a”的引物(leu2酵母标记)seqidno:45用于构建片段“a”的引物(leu2酵母标记)sequencelisting<110>弗门尼舍有限公司(firmenichs.a.)<120>迈诺醇的生产(productionofmanool)<130>9950_pct<150>ep16206349.9<151>2016-12-22<160>45<170>patentinversion3.5<210>1<211>793<212>prt<213>丹参(salviamiltiorrhiza)<400>1metalaserleuserserthrileleuserargserproalaalaarg151015argargilethrproalaseralalysleuhisargproglucysphe202530alathrseralatrpmetglyserserserlysasnleuserleuser354045tyrglnleuasnhislyslysileservalalathrvalaspalapro505560glnvalhisasphisaspglythrthrvalhisglnglyhisaspala65707580vallysasnilegluaspproileglutyrileargthrleuleuarg859095thrthrglyaspglyargileservalserprotyraspthralatrp100105110valalametilelysaspvalgluglyargaspglyproglnphepro115120125serserleuglutrpilevalglnasnglnleugluaspglysertrp130135140glyaspglnlysleuphecysvaltyraspargleuvalasnthrile145150155160alacysvalvalalaleuargsertrpasnvalhisalahislysval165170175lysargglyvalthrtyrilelysgluasnvalasplysleumetglu180185190glyasnglugluhismetthrcysglyphegluvalvalpheproala195200205leuleuglnlysalalysserleuglyilegluaspleuprotyrasp210215220serproalavalglngluvaltyrhisvalarggluglnlysleulys225230235240argileproleugluilemethislysileprothrserleuleuphe245250255serleugluglyleugluasnleuasptrpasplysleuleulysleu260265270glnseralaaspglyserpheleuthrserproserserthralaphe275280285alaphemetglnthrlysaspglulyscystyrglnpheilelysasn290295300thrileaspthrpheasnglyglyalaprohisthrtyrprovalasp305310315320valpheglyargleutrpalaileaspargleuglnargleuglyile325330335serargphephegluprogluilealaaspcysleuserhisilehis340345350lysphetrpthrasplysglyvalpheserglyargglusergluphe355360365cysaspileaspaspthrsermetglymetargleumetargmethis370375380glytyraspvalaspproasnvalleuargasnphelysglnlysasp385390395400glylysphesercystyrglyglyglnmetilegluserproserpro405410415iletyrasnleutyrargalaserglnleuargpheproglygluglu420425430ileleugluaspalalysargphealatyrasppheleulysglulys435440445leualaasnasnglnileleuasplystrpvalileserlyshisleu450455460proaspgluilelysleuglyleuglumetprotrpleualathrleu465470475480proargvalglualalystyrtyrileglntyrtyralaglysergly485490495aspvaltrpileglylysthrleutyrargmetprogluileserasn500505510aspthrtyrhisaspleualalysthraspphelysargcysglnala515520525lyshisglnpheglutrpleutyrmetglnglutrptyrglusercys530535540glyilegluglupheglyileserarglysaspleuleuleusertyr545550555560pheleualathralaserilephegluleugluargthrasngluarg565570575ilealatrpalalysserglnileilealalysmetilethrserphe580585590pheasnlysgluthrthrserglugluasplysargalaleuleuasn595600605gluleuglyasnileasnglyleuasnaspthrasnglyalaglyarg610615620gluglyglyalaglyserilealaleualathrleuthrglnpheleu625630635640gluglypheaspargtyrthrarghisglnleulysasnalatrpser645650655valtrpleuthrglnleuglnhisglyglualaaspaspalagluleu660665670leuthrasnthrleuasnilecysalaglyhisilealapheargglu675680685gluileleualahisasnglutyrlysalaleuserasnleuthrser690695700lysilecysargglnleuserpheileglnserglulysglumetgly705710715720valgluglygluilealaalalysserserilelysasnlysgluleu725730735glugluaspmetglnmetleuvallysleuvalleuglulystyrgly740745750glyileaspargasnilelyslysalapheleualavalalalysthr755760765tyrtyrtyrargalatyrhisalaalaaspthrileaspthrhismet770775780phelysvalleuphegluprovalala785790<210>2<211>736<212>prt<213>人工序列(artificialsequence)<223>截短的柯巴基二磷酸合酶<400>2metalathrvalaspalaproglnvalhisasphisaspglythrthr151015valhisglnglyhisaspalavallysasnilegluaspproileglu202530tyrileargthrleuleuargthrthrglyaspglyargileserval354045serprotyraspthralatrpvalalametilelysaspvalglugly505560argaspglyproglnpheproserserleuglutrpilevalglnasn65707580glnleugluaspglysertrpglyaspglnlysleuphecysvaltyr859095aspargleuvalasnthrilealacysvalvalalaleuargsertrp100105110asnvalhisalahislysvallysargglyvalthrtyrilelysglu115120125asnvalasplysleumetgluglyasnglugluhismetthrcysgly130135140phegluvalvalpheproalaleuleuglnlysalalysserleugly145150155160ilegluaspleuprotyraspserproalavalglngluvaltyrhis165170175valarggluglnlysleulysargileproleugluilemethislys180185190ileprothrserleuleupheserleugluglyleugluasnleuasp195200205trpasplysleuleulysleuglnseralaaspglyserpheleuthr210215220serproserserthralaphealaphemetglnthrlysaspglulys225230235240cystyrglnpheilelysasnthrileaspthrpheasnglyglyala245250255prohisthrtyrprovalaspvalpheglyargleutrpalaileasp260265270argleuglnargleuglyileserargphephegluprogluileala275280285aspcysleuserhisilehislysphetrpthrasplysglyvalphe290295300serglyargglusergluphecysaspileaspaspthrsermetgly305310315320metargleumetargmethisglytyraspvalaspproasnvalleu325330335argasnphelysglnlysaspglylysphesercystyrglyglygln340345350metilegluserproserproiletyrasnleutyrargalasergln355360365leuargpheproglyglugluileleugluaspalalysargpheala370375380tyrasppheleulysglulysleualaasnasnglnileleuasplys385390395400trpvalileserlyshisleuproaspgluilelysleuglyleuglu405410415metprotrpleualathrleuproargvalglualalystyrtyrile420425430glntyrtyralaglyserglyaspvaltrpileglylysthrleutyr435440445argmetprogluileserasnaspthrtyrhisaspleualalysthr450455460aspphelysargcysglnalalyshisglnpheglutrpleutyrmet465470475480glnglutrptyrglusercysglyilegluglupheglyileserarg485490495lysaspleuleuleusertyrpheleualathralaserilepheglu500505510leugluargthrasngluargilealatrpalalysserglnileile515520525alalysmetilethrserphepheasnlysgluthrthrsergluglu530535540asplysargalaleuleuasngluleuglyasnileasnglyleuasn545550555560aspthrasnglyalaglyarggluglyglyalaglyserilealaleu565570575alathrleuthrglnpheleugluglypheaspargtyrthrarghis580585590glnleulysasnalatrpservaltrpleuthrglnleuglnhisgly595600605glualaaspaspalagluleuleuthrasnthrleuasnilecysala610615620glyhisilealapheargglugluileleualahisasnglutyrlys625630635640alaleuserasnleuthrserlysilecysargglnleuserpheile645650655glnserglulysglumetglyvalgluglygluilealaalalysser660665670serilelysasnlysgluleuglugluaspmetglnmetleuvallys675680685leuvalleuglulystyrglyglyileaspargasnilelyslysala690695700pheleualavalalalysthrtyrtyrtyrargalatyrhisalaala705710715720aspthrileaspthrhismetphelysvalleuphegluprovalala725730735<210>3<211>2211<212>dna<213>人工序列(artificialsequence)<223>经优化的用于大肠杆菌(e.coli)表达的编码smcps2的cdna<400>3atggcaactgttgacgcacctcaagtccatgatcacgatggcaccaccgttcaccagggt60cacgacgcggtgaagaacatcgaggacccgatcgaatacattcgtaccctgctgcgtacc120actggtgatggtcgcatcagcgtcagcccgtatgacacggcgtgggtggcgatgattaaa180gacgtcgagggtcgcgatggcccgcaatttccttctagcctggagtggattgtccaaaat240cagctggaagatggctcgtggggtgaccagaagctgttttgtgtttacgatcgcctggtt300aataccatcgcatgtgtggttgcgctgcgtagctggaatgttcacgctcataaagtcaaa360cgtggcgtgacgtatatcaaggaaaacgtggataagctgatggaaggcaacgaagaacac420atgacgtgtggcttcgaggttgtttttccagccttgctgcagaaagcaaagtccctgggt480attgaggatctgccgtacgactcgccggcagtgcaagaagtctatcacgtccgcgagcag540aagctgaaacgcatcccgctggagattatgcataagattccgacctctctgctgttctct600ctggaaggtctggagaacctggattgggacaaactgctgaagctgcagtccgctgacggt660agctttctgaccagcccgagcagcacggcctttgcgtttatgcagaccaaagatgagaag720tgctatcaattcatcaagaatactattgataccttcaacggtggcgcaccgcacacgtac780ccagtagacgtttttggtcgcctgtgggcgattgaccgtttgcagcgtctgggtatcagc840cgtttcttcgagccggagattgcggactgcttgagccatattcacaaattctggacggac900aaaggcgtgttcagcggtcgtgagagcgagttctgcgacatcgacgatacgagcatgggt960atgcgtctgatgcgtatgcacggttacgacgtggacccgaatgtgttgcgcaacttcaag1020caaaaagatggcaagtttagctgctacggtggccaaatgattgagagcccgagcccgatc1080tataacttatatcgtgcgagccaactgcgtttcccgggtgaagaaattctggaagatgcg1140aagcgttttgcgtatgacttcctgaaggaaaagctcgcaaacaatcaaatcttggataaa1200tgggtgatcagcaagcacttgccggatgagattaaactgggtctggagatgccgtggttg1260gccaccctgccgagagttgaggcgaaatactatattcagtattacgcgggtagcggtgat1320gtttggattggcaagaccctgtaccgcatgccggagatcagcaatgatacctatcatgac1380ctggccaagaccgacttcaaacgctgtcaagcgaaacatcaatttgaatggttatacatg1440caagagtggtacgaaagctgcggcatcgaagagttcggtatctcccgtaaagatctgctg1500ctgtcttactttctggcaacggccagcattttcgagctggagcgtaccaatgagcgtatt1560gcctgggcgaaatcacaaatcattgctaagatgattacgagctttttcaataaagaaacc1620acgtccgaggaagataaacgtgctctgctgaatgaactgggcaacatcaacggtctgaat1680gacaccaacggtgccggtcgtgagggtggcgcaggcagcattgcactggccacgctgacc1740cagttcctggaaggtttcgaccgctacacccgtcaccagctgaagaacgcgtggtccgtc1800tggctgacccagctgcagcatggtgaggcagacgacgcggagctgctgaccaacacgttg1860aatatctgcgctggccatatcgcgtttcgcgaagagattctggcgcacaacgagtacaaa1920gccctgagcaatctgacctctaaaatctgtcgtcagcttagctttattcagagcgagaaa1980gaaatgggcgtggaaggtgagatcgcggcaaaatccagcatcaagaacaaagaactggaa2040gaagatatgcagatgttggtcaagctcgtcctggagaagtatggtggcatcgaccgtaat2100atcaagaaagcgtttctggccgtggcgaaaacgtattactaccgcgcgtaccacgcggca2160gataccattgacacccacatgtttaaggttttgtttgagccggttgcttaa2211<210>4<211>575<212>prt<213>快乐鼠尾草(salviasclarea)<400>4metserleualapheasnvalglyvalthrpropheserglyglnarg151015valglyserarglysglulyspheprovalglnglypheprovalthr202530thrproasnargserargleuilevalasncysserleuthrthrile354045aspphemetalalysmetlysgluasnphelysarggluaspasplys505560pheprothrthrthrthrleuargsergluaspileproserasnleu65707580cysileileaspthrleuglnargleuglyvalaspglnphephegln859095tyrgluileasnthrileleuaspasnthrpheargleutrpglnglu100105110lyshislysvaliletyrglyasnvalthrthrhisalametalaphe115120125argleuleuargvallysglytyrgluvalserserglugluleuala130135140protyrglyasnglnglualavalserglnglnthrasnaspleupro145150155160metileilegluleutyrargalaalaasngluargiletyrgluglu165170175gluargserleuglulysileleualatrpthrthrilepheleuasn180185190lysglnvalglnaspasnserileproasplyslysleuhislysleu195200205valgluphetyrleuargasntyrlysglyilethrileargleugly210215220alaargargasnleugluleutyraspmetthrtyrtyrglnalaleu225230235240lysserthrasnargpheserasnleucysasngluasppheleuval245250255phealalysglnasppheaspilehisglualaglnasnglnlysgly260265270leuglnglnleuglnargtrptyralaaspcysargleuaspthrleu275280285asnpheglyargaspvalvalileilealaasntyrleualaserleu290295300ileileglyasphisalapheasptyrvalargleualaphealalys305310315320thrservalleuvalthrilemetaspaspphepheaspcyshisgly325330335serserglnglucysasplysileilegluleuvallysglutrplys340345350gluasnproaspalaglutyrglyserglugluleugluileleuphe355360365metalaleutyrasnthrvalasngluleualagluargalaargval370375380gluglnglyargservallysglupheleuvallysleutrpvalglu385390395400ileleuseralaphelysilegluleuaspthrtrpserasnglythr405410415glnglnserpheaspglutyrilesersersertrpleuserasngly420425430serargleuthrglyleuleuthrmetglnphevalglyvallysleu435440445seraspglumetleumetsergluglucysthraspleualaarghis450455460valcysmetvalglyargleuleuasnaspvalcyssersergluarg465470475480gluargglugluasnilealaglylyssertyrserileleuleuala485490495thrglulysaspglyarglysvalsergluaspglualailealaglu500505510ileasnglumetvalglutyrhistrparglysvalleuglnileval515520525tyrlyslysgluserileleuproargargcyslysaspvalpheleu530535540glumetalalysglythrphetyralatyrglyileasnaspgluleu545550555560thrserproglnglnserlysgluaspmetlysserphevalphe565570575<210>5<211>525<212>prt<213>人工序列(artificialsequence)<223>来自快乐鼠尾草(salviasclarea)的截短的香紫苏醇合酶(ssscs)<400>5metalalysmetlysgluasnphelysarggluaspasplysphepro151015thrthrthrthrleuargsergluaspileproserasnleucysile202530ileaspthrleuglnargleuglyvalaspglnphepheglntyrglu354045ileasnthrileleuaspasnthrpheargleutrpglnglulyshis505560lysvaliletyrglyasnvalthrthrhisalametalapheargleu65707580leuargvallysglytyrgluvalserserglugluleualaprotyr859095glyasnglnglualavalserglnglnthrasnaspleuprometile100105110ilegluleutyrargalaalaasngluargiletyrgluglugluarg115120125serleuglulysileleualatrpthrthrilepheleuasnlysgln130135140valglnaspasnserileproasplyslysleuhislysleuvalglu145150155160phetyrleuargasntyrlysglyilethrileargleuglyalaarg165170175argasnleugluleutyraspmetthrtyrtyrglnalaleulysser180185190thrasnargpheserasnleucysasngluasppheleuvalpheala195200205lysglnasppheaspilehisglualaglnasnglnlysglyleugln210215220glnleuglnargtrptyralaaspcysargleuaspthrleuasnphe225230235240glyargaspvalvalileilealaasntyrleualaserleuileile245250255glyasphisalapheasptyrvalargleualaphealalysthrser260265270valleuvalthrilemetaspaspphepheaspcyshisglyserser275280285glnglucysasplysileilegluleuvallysglutrplysgluasn290295300proaspalaglutyrglyserglugluleugluileleuphemetala305310315320leutyrasnthrvalasngluleualagluargalaargvalglugln325330335glyargservallysglupheleuvallysleutrpvalgluileleu340345350seralaphelysilegluleuaspthrtrpserasnglythrglngln355360365serpheaspglutyrilesersersertrpleuserasnglyserarg370375380leuthrglyleuleuthrmetglnphevalglyvallysleuserasp385390395400glumetleumetsergluglucysthraspleualaarghisvalcys405410415metvalglyargleuleuasnaspvalcyssersergluarggluarg420425430glugluasnilealaglylyssertyrserileleuleualathrglu435440445lysaspglyarglysvalsergluaspglualailealagluileasn450455460glumetvalglutyrhistrparglysvalleuglnilevaltyrlys465470475480lysgluserileleuproargargcyslysaspvalpheleuglumet485490495alalysglythrphetyralatyrglyileasnaspgluleuthrser500505510proglnglnserlysgluaspmetlysserphevalphe515520525<210>6<211>1583<212>dna<213>人工序列(artificialsequence)<223>经优化的用于大肠杆菌(e.coli)表达的编码来自快乐鼠尾草(salviasclarea)的截短的香紫苏醇合酶的cdna<400>6atggcgaaaatgaaggagaactttaaacgcgaggacgataaattcccgacgaccacgacc60ctgcgcagcgaggatatcccgagcaacctgtgcatcattgataccctgcagcgcctgggt120gtcgatcagttcttccaatacgaaatcaataccattctggacaatacttttcgtctgtgg180caagagaaacacaaagtgatctacggcaacgttaccacccacgcgatggcgttccgtttg240ttgcgtgtcaagggctacgaggtttccagcgaggaactggcgccgtacggtaatcaggaa300gcagttagccaacagacgaatgatctgcctatgatcattgagctgtatcgcgcagcaaat360gagcgtatctacgaagaggaacgcagcctggaaaagatcctggcgtggaccacgatcttc420ctgaacaaacaagttcaagacaattctattcctgataagaagctgcataaactggtcgaa480ttctatctgcgtaattacaagggcatcacgatccgtctgggcgcacgccgtaacctggag540ttgtatgatatgacgtattaccaggctctgaaaagcaccaatcgtttctccaatctgtgt600aatgaggattttctggtgttcgccaagcaggattttgacatccacgaggcgcaaaatcaa660aaaggtctgcaacaactgcaacgttggtacgctgactgtcgcctggacaccctgaatttc720ggtcgcgacgttgtcattattgcaaactatctggccagcctgatcatcggtgatcacgca780ttcgactacgtccgcctggccttcgctaagaccagcgttctggtgaccattatggatgat840ttcttcgattgccacggttctagccaggaatgcgacaaaatcattgagctggtgaaagag900tggaaagaaaaccctgatgcggaatacggttccgaagagttggagatcctgtttatggcc960ttgtacaacaccgtgaatgaactggccgagcgtgctcgtgtggagcagggccgttctgtg1020aaggagtttttggtcaagttgtgggtggaaatcctgtccgcgttcaagatcgaactggat1080acgtggtcgaatggtacgcaacagagcttcgacgaatacatcagcagcagctggctgagc1140aatggcagccgtctgaccggtttgctgaccatgcaatttgtgggtgttaaactgtccgat1200gaaatgctgatgagcgaagaatgcaccgacctggcacgccatgtgtgtatggtgggtcgc1260ctgctgaacgacgtctgcagcagcgaacgtgagcgcgaggaaaacattgcaggcaagagc1320tacagcatcttgttggccaccgagaaagatggtcgcaaagtgtctgaggacgaagcaatt1380gcagagattaatgaaatggtcgagtaccactggcgtaaggttttgcagattgtgtataag1440aaagagagcatcttgccgcgtcgctgtaaggatgttttcttggagatggcgaagggcacg1500ttctatgcgtacggcattaacgacgagctgacgagcccgcaacaatcgaaagaggacatg1560aagagcttcgtgttctgaggtac1583<210>7<211>307<212>prt<213>成团泛菌(pantoeaagglomerans)<400>7metvalserglyserlysalaglyvalserprohisarggluileglu151015valmetargglnserileaspasphisleualaglyleuleuproglu202530thraspserglnaspilevalserleualametarggluglyvalmet354045alaproglylysargileargproleuleumetleuleualaalaarg505560aspleuargtyrglnglysermetprothrleuleuaspleualacys65707580alavalgluleuthrhisthralaserleumetleuaspaspmetpro859095cysmetaspasnalagluleuargargglyglnprothrthrhislys100105110lyspheglygluservalalaileleualaservalglyleuleuser115120125lysalapheglyleuilealaalathrglyaspleuproglygluarg130135140argalaglnalavalasngluleuserthralavalglyvalglngly145150155160leuvalleuglyglnpheargaspleuasnaspalaalaleuasparg165170175thrproaspalaileleuserthrasnhisleulysthrglyileleu180185190pheseralametleuglnilevalalailealaseralaserserpro195200205serthrarggluthrleuhisalaphealaleuasppheglyglnala210215220pheglnleuleuaspaspleuargaspasphisprogluthrglylys225230235240aspargasnlysaspalaglylysserthrleuvalasnargleugly245250255alaaspalaalaargglnlysleuarggluhisileaspseralaasp260265270lyshisleuthrphealacysproglnglyglyalaileargglnphe275280285methisleutrppheglyhishisleualaasptrpserprovalmet290295300lysileala305<210>8<211>924<212>dna<213>人工序列(artificialsequence)<223>经优化的编码来自成团泛菌(pantoeaagglomerans)的ggpp合酶的cdna<400>8atggtttctggttcgaaagcaggagtatcacctcatagggaaatcgaagtcatgagacag60tccattgatgaccacttagcaggattgttgccagaaacagattcccaggatatcgttagc120cttgctatgagagaaggtgttatggcacctggtaaacgtatcagacctttgctgatgtta180cttgctgcaagagacctgagatatcagggttctatgcctacactactggatctagcttgt240gctgttgaactgacacatactgcttccttgatgctggatgacatgccttgtatggacaat300gcggaacttagaagaggtcaaccaacaacccacaagaaattcggagaatctgttgccatt360ttggcttctgtaggtctgttgtcgaaagcttttggcttgattgctgcaactggtgatctt420ccaggtgaaaggagagcacaagctgtaaacgagctatctactgcagttggtgttcaaggt480ctagtcttaggacagttcagagatttgaatgacgcagctttggacagaactcctgatgct540atcctgtctacgaaccatctgaagactggcatcttgttctcagctatgttgcaaatcgta600gccattgcttctgcttcttcaccatctactagggaaacgttacacgcattcgcattggac660tttggtcaagcctttcaactgctagacgatttgagggatgatcatccagagacaggtaaa720gaccgtaacaaagacgctggtaaaagcactctagtcaacagattgggtgctgatgcagct780agacagaaactgagagagcacattgactctgctgacaaacacctgacatttgcatgtcca840caaggaggtgctataaggcagtttatgcacctatggtttggacaccatcttgctgattgg900tctccagtgatgaagatcgcctaa924<210>9<211>71<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>9ctgtttgagccggtcgcctaaggtaccagaaggagataaataatggcgaaaatgaaggag60aactttaaacg71<210>10<211>55<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>10gcagcggtttctttaccagactcgaggtcagaacacgaagctcttcatgtcctct55<210>11<211>786<212>prt<213>毛喉鞘蕊花(coleusforskohlii)<400>11metglyserleuserthrmetasnleuasnhisserprometsertyr151015serglyileleuproserserseralalysalalysleuleuleupro202530glycyspheserileseralatrpmetasnasnglylysasnleuasn354045cysglnleuthrhislyslysileserlysvalalagluileargval505560alathrvalasnalaproprovalhisaspglnaspaspserthrglu65707580asnglncyshisaspalavalasnasnilegluaspproileglutyr859095ileargthrleuleuargthrthrglyaspglyargileservalser100105110protyraspthralatrpvalalaleuilelysaspleuglnglyarg115120125aspalaproglupheproserserleuglutrpileileglnasngln130135140leualaaspglysertrpglyaspalalysphephecysvaltyrasp145150155160argleuvalasnthrilealacysvalvalalaleuargsertrpasp165170175valhisalaglulysvalgluargglyvalargtyrileasngluasn180185190valglulysleuargaspglyasnglugluhismetthrcysglyphe195200205gluvalvalpheproalaleuleuglnargalalysserleuglyile210215220glnaspleuprotyraspalaprovalileglngluiletyrhisser225230235240arggluglnlysserlysargileproleuglumetmethislysval245250255prothrserleuleupheserleugluglyleugluasnleuglutrp260265270asplysleuleulysleuglnseralaaspglyserpheleuthrser275280285proserserthralaphealaphemetglnthrargaspprolyscys290295300tyrglnpheilelysasnthrileglnthrpheasnglyglyalapro305310315320histhrtyrprovalaspvalpheglyargleutrpalaileasparg325330335leuglnargleuglyileserargphepheglusergluilealaasp340345350cysilealahisilehisargphetrpthrglulysglyvalpheser355360365glyargglusergluphecysaspileaspaspthrsermetglyval370375380argleumetargmethisglytyraspvalaspproasnvalleulys385390395400asnphelyslysaspasplysphesercystyrglyglyglnmetile405410415gluserproserproiletyrasnleutyrargalaserglnleuarg420425430pheproglygluglnileleugluaspalaasnlysphealatyrasp435440445pheleuglnglulysleualahisasnglnileleuasplystrpval450455460ileserlyshisleuproaspgluilelysleuglyleuglumetpro465470475480trptyralathrleuproargvalglualaargtyrtyrileglntyr485490495tyralaglyserglyaspvaltrpileglylysthrleutyrargmet500505510progluileserasnaspthrtyrhisgluleualalysthraspphe515520525lysargcysglnalaglnhisglnpheglutrpiletyrmetglnglu530535540trptyrglusercysasnmetgluglupheglyileserarglysglu545550555560leuleuvalalatyrpheleualathralaserilephegluleuglu565570575argalaasngluargilealatrpalalysserglnileileserthr580585590ileilealaserphepheasnasnglnasnthrserprogluasplys595600605leualapheleuthraspphelysasnglyasnserthrasnmetala610615620leuvalthrleuthrglnpheleugluglypheaspargtyrthrser625630635640hisglnleulysasnalatrpservaltrpleuarglysleuglngln645650655glygluglyasnglyglyalaaspalagluleuleuvalasnthrleu660665670asnilecysalaglyhisilealapheargglugluileleualahis675680685asnasptyrlysthrleuserasnleuthrserlysilecysarggln690695700leuserglnileglnasnglulysgluleugluthrgluglyglnlys705710715720thrserilelysasnlysgluleuglugluaspmetglnargleuval725730735lysleuvalleuglulysserargvalglyileasnargaspmetlys740745750lysthrpheleualavalvallysthrtyrtyrtyrlysalatyrhis755760765seralaglnalaileaspasnhismetphelysvalleupheglupro770775780valala785<210>12<211>724<212>prt<213>人工序列(artificialsequence)<223>来自毛喉鞘蕊花(coleusforskohlii)的截短的柯巴基二磷酸合酶<400>12metvalalathrvalasnalaproprovalhisaspglnaspaspser151015thrgluasnglncyshisaspalavalasnasnilegluaspproile202530glutyrileargthrleuleuargthrthrglyaspglyargileser354045valserprotyraspthralatrpvalalaleuilelysaspleugln505560glyargaspalaproglupheproserserleuglutrpileilegln65707580asnglnleualaaspglysertrpglyaspalalysphephecysval859095tyraspargleuvalasnthrilealacysvalvalalaleuargser100105110trpaspvalhisalaglulysvalgluargglyvalargtyrileasn115120125gluasnvalglulysleuargaspglyasnglugluhismetthrcys130135140glyphegluvalvalpheproalaleuleuglnargalalysserleu145150155160glyileglnaspleuprotyraspalaprovalileglngluiletyr165170175hisserarggluglnlysserlysargileproleuglumetmethis180185190lysvalprothrserleuleupheserleugluglyleugluasnleu195200205glutrpasplysleuleulysleuglnseralaaspglyserpheleu210215220thrserproserserthralaphealaphemetglnthrargasppro225230235240lyscystyrglnpheilelysasnthrileglnthrpheasnglygly245250255alaprohisthrtyrprovalaspvalpheglyargleutrpalaile260265270aspargleuglnargleuglyileserargphepheglusergluile275280285alaaspcysilealahisilehisargphetrpthrglulysglyval290295300pheserglyargglusergluphecysaspileaspaspthrsermet305310315320glyvalargleumetargmethisglytyraspvalaspproasnval325330335leulysasnphelyslysaspasplysphesercystyrglyglygln340345350metilegluserproserproiletyrasnleutyrargalasergln355360365leuargpheproglygluglnileleugluaspalaasnlyspheala370375380tyrasppheleuglnglulysleualahisasnglnileleuasplys385390395400trpvalileserlyshisleuproaspgluilelysleuglyleuglu405410415metprotrptyralathrleuproargvalglualaargtyrtyrile420425430glntyrtyralaglyserglyaspvaltrpileglylysthrleutyr435440445argmetprogluileserasnaspthrtyrhisgluleualalysthr450455460aspphelysargcysglnalaglnhisglnpheglutrpiletyrmet465470475480glnglutrptyrglusercysasnmetgluglupheglyileserarg485490495lysgluleuleuvalalatyrpheleualathralaserilepheglu500505510leugluargalaasngluargilealatrpalalysserglnileile515520525serthrileilealaserphepheasnasnglnasnthrserproglu530535540asplysleualapheleuthraspphelysasnglyasnserthrasn545550555560metalaleuvalthrleuthrglnpheleugluglypheaspargtyr565570575thrserhisglnleulysasnalatrpservaltrpleuarglysleu580585590glnglnglygluglyasnglyglyalaaspalagluleuleuvalasn595600605thrleuasnilecysalaglyhisilealapheargglugluileleu610615620alahisasnasptyrlysthrleuserasnleuthrserlysilecys625630635640argglnleuserglnileglnasnglulysgluleugluthrglugly645650655glnlysthrserilelysasnlysgluleuglugluaspmetglnarg660665670leuvallysleuvalleuglulysserargvalglyileasnargasp675680685metlyslysthrpheleualavalvallysthrtyrtyrtyrlysala690695700tyrhisseralaglnalaileaspasnhismetphelysvalleuphe705710715720gluprovalala<210>13<211>2175<212>dna<213>人工序列(artificialsequence)<223>经优化的用于大肠杆菌(e.coli)表达的编码cfcps1-del63的cdna<400>13atggtcgctactgtcaatgctccaccggtccacgatcaagacgacagcactgagaatcaa60tgtcatgatgccgtaaacaatattgaagatccaatcgagtatatccgtaccctgttgcgc120acgacgggtgatggtcgtatcagcgtcagcccgtacgataccgcgtgggtggcgctgatc180aaagatctgcagggccgtgacgcaccggagtttccgtcctctcttgagtggatcattcaa240aaccagctggccgacggttcttggggcgacgccaaatttttctgcgtgtatgaccgtctg300gtgaacaccatcgcgtgcgtcgttgcgctgcgttcctgggacgtccacgcggaaaaagtt360gagcgtggcgtgcgctatatcaacgaaaatgtcgaaaagctgcgcgacggtaatgaagaa420cacatgacctgtggctttgaagttgttttcccggcgctcctgcagcgcgcgaagtctctg480ggtattcaagatctgccgtacgatgctccggtgatccaagagatttatcactctcgtgag540cagaagtccaagcgtatcccgttggagatgatgcacaaagttccgacgagcctgctgttc600agcttggaaggcctggaaaatctggagtgggacaaactgctgaagctgcagagcgcggac660ggtagcttcctgacgagcccgagcagcaccgcatttgcatttatgcagacccgtgacccg720aagtgttaccaatttattaagaacacgattcagacgtttaacggtggtgcaccgcatacc780tatccggtagacgtctttggtcgcctgtgggcaattgatcgtctgcagcgtttgggtatc840agccgcttcttcgaaagcgaaattgcagattgtatcgcacacatccatcgtttttggacc900gagaaaggcgtctttagcggccgtgagtctgagttctgtgacatcgatgacacgagcatg960ggtgtccgtctgatgcgtatgcatggctatgatgttgacccgaacgtgctgaagaatttt1020aaaaaagatgacaagtttagctgctacggcggtcagatgattgagagcccgagcccgatt1080tataatctgtaccgcgcgagccaactgcgtttcccgggtgaacagattctggaagatgcc1140aataaattcgcgtatgatttcctgcaggaaaaactggcgcacaatcagatcctggataaa1200tgggttatcagcaagcatctgcctgacgaaatcaaattgggcctggagatgccgtggtat1260gcgaccttgccgcgtgtcgaagcgcgttactacatccagtactatgcgggtagcggcgat1320gtctggattggtaagacgctgtaccgtatgccagagattagcaacgacacctaccatgaa1380ttggcaaagaccgatttcaagcgttgccaagcccaacaccagttcgagtggatttacatg1440caagagtggtacgagtcgtgcaacatggaagagttcggtattagccgcaaagaactgctg1500gttgcatatttcctggccacggcgagcatctttgagctggagcgtgcgaatgaacgcatt1560gcatgggcaaaaagccaaatcatttctaccattatcgcttcgttctttaataaccaaaat1620acgagccctgaggataaactggcgtttctgactgatttcaaaaatggcaacagcaccaac1680atggctctggtgaccctgacccagttcctggaaggctttgaccgctacacttcccatcaa1740ctgaaaaacgcgtggagcgtttggctgcgtaagctgcaacagggtgagggtaatggcggt1800gccgacgccgagttactggtgaatacgctgaacatttgcgcgggtcacatcgcgttccgt1860gaagaaattctggcacataatgactataaaacgttgtcgaacctgaccagcaagatttgt1920cgccagctgagccagattcagaatgaaaaagaattggaaaccgaaggccaaaagacttcc1980attaagaacaaagaactggaagaagatatgcagcgcctggttaaactggttttggagaaa2040agccgtgtgggtatcaatcgtgacatgaagaaaacgttcctggctgtggtgaaaacctac2100tattacaaagcataccactccgcgcaggcaatcgataaccacatgttcaaggttctgttc2160gaaccggtggcctaa2175<210>14<211>757<212>prt<213>小麦(triticumaestivum)<400>14metleuthrphethralaalaleuarghisvalprovalleuaspgln151015prothrsergluprotrpargargleuserleuhisleuhissergln202530argargprocysglyleuvalleuileserlysserprosertyrpro354045gluvalaspvalglyglutrplysvalaspglutyrargglnargthr505560aspgluprosergluthrargglnmetileaspaspileargthrala65707580leualaserleuglyaspaspgluthrsermetservalseralatyr859095aspthralaleuvalalaleuvallysasnleuaspglyglyaspgly100105110proglnpheprosercysileasptrpilevalglnasnglnleupro115120125aspglysertrpglyaspproalaphephemetvalglnaspargmet130135140ileserthrleualacysvalvalalavallyssertrpasnileasp145150155160argaspasnleucysaspargglyvalleupheilelysgluasnmet165170175serargleuvalgluglugluglnasptrpmetprocysglypheglu180185190ileasnpheproalaleuleuglulysalalysaspleuaspleuasp195200205ileprotyrasphisprovalleuglugluiletyralalysargasn210215220leulysleuleulysileproleuaspvalleuhisalaileprothr225230235240thrleuleupheservalgluglymetvalaspleuproleuasptrp245250255glulysleuleuargleuargcysproaspglyserphehisserser260265270proalaalathralaalaalaleuserhisthrglyasplysglucys275280285hisalapheleuaspargleuileglnlysphegluglyglyvalpro290295300cysserhissermetaspthrphegluglnleutrpvalvalasparg305310315320leumetargleuglyileserarghisphethrsergluileglngln325330335cysleuglupheiletyrargargtrpthrglnlysglyleualahis340345350asnmethiscysproileproaspileaspaspthralametglyphe355360365argleuleuargglnhisglytyraspvalthrproservalphelys370375380hispheglulysaspglylysphevalcyspheprometgluthrasn385390395400hisalaservalthrpromethisasnthrtyrargalaserglnphe405410415metpheproglyaspaspaspvalleualaargalaglyargtyrcys420425430argalapheleuglngluargglnserserasnlysleutyrasplys435440445trpileilethrlysaspleuproglygluvalglytyrthrleuasn450455460pheprotrplysserserleuproargilegluthrargmettyrleu465470475480aspglntyrglyglyasnasnaspvaltrpilealalysvalleutyr485490495argmetasnleuvalserasnaspleutyrleulysmetalalysala500505510aspphethrglutyrglnargleuserargileglutrpasnglyleu515520525arglystrptyrpheargasnhisleuglnargtyrglyalathrpro530535540lysseralaleulysalatyrpheleualaseralaasnilepheglu545550555560proglyargalaalagluargleualatrpalaargmetalavalleu565570575alaglualavalthrthrhisphearghisileglyglyprocystyr580585590serthrgluasnleuglugluleuileaspleuvalserpheaspasp595600605valserglyglyleuargglualatrplysglntrpleumetalatrp610615620thralalysgluserhisglyservalaspglyaspthralaleuleu625630635640phevalargthrilegluilecysserglyargilevalserserglu645650655glnlysleuasnleutrpasptyrserglnleugluglnleuthrser660665670serilecyshislysleualathrileglyleuserglnasngluala675680685sermetgluasnthrgluaspleuhisglnglnvalaspleuglumet690695700glngluleusertrpargvalhisglnglycyshisglyileasnarg705710715720gluthrargglnthrpheleuasnvalvallysserphetyrtyrser725730735alahiscysserprogluthrvalaspserhisilealalysvalile740745750pheglnaspvalile755<210>15<211>699<212>prt<213>人工序列(artificialsequence)<223>来自小麦(triticumaestivum)的截短的柯巴基二磷酸合酶<400>15mettyrargglnargthraspgluprosergluthrargglnmetile151015aspaspileargthralaleualaserleuglyaspaspgluthrser202530metservalseralatyraspthralaleuvalalaleuvallysasn354045leuaspglyglyaspglyproglnpheprosercysileasptrpile505560valglnasnglnleuproaspglysertrpglyaspproalaphephe65707580metvalglnaspargmetileserthrleualacysvalvalalaval859095lyssertrpasnileaspargaspasnleucysaspargglyvalleu100105110pheilelysgluasnmetserargleuvalgluglugluglnasptrp115120125metprocysglyphegluileasnpheproalaleuleuglulysala130135140lysaspleuaspleuaspileprotyrasphisprovalleugluglu145150155160iletyralalysargasnleulysleuleulysileproleuaspval165170175leuhisalaileprothrthrleuleupheservalgluglymetval180185190aspleuproleuasptrpglulysleuleuargleuargcysproasp195200205glyserphehisserserproalaalathralaalaalaleuserhis210215220thrglyasplysglucyshisalapheleuaspargleuileglnlys225230235240phegluglyglyvalprocysserhissermetaspthrpheglugln245250255leutrpvalvalaspargleumetargleuglyileserarghisphe260265270thrsergluileglnglncysleuglupheiletyrargargtrpthr275280285glnlysglyleualahisasnmethiscysproileproaspileasp290295300aspthralametglypheargleuleuargglnhisglytyraspval305310315320thrproservalphelyshispheglulysaspglylysphevalcys325330335pheprometgluthrasnhisalaservalthrpromethisasnthr340345350tyrargalaserglnphemetpheproglyaspaspaspvalleuala355360365argalaglyargtyrcysargalapheleuglngluargglnserser370375380asnlysleutyrasplystrpileilethrlysaspleuproglyglu385390395400valglytyrthrleuasnpheprotrplysserserleuproargile405410415gluthrargmettyrleuaspglntyrglyglyasnasnaspvaltrp420425430ilealalysvalleutyrargmetasnleuvalserasnaspleutyr435440445leulysmetalalysalaaspphethrglutyrglnargleuserarg450455460ileglutrpasnglyleuarglystrptyrpheargasnhisleugln465470475480argtyrglyalathrprolysseralaleulysalatyrpheleuala485490495seralaasnilephegluproglyargalaalagluargleualatrp500505510alaargmetalavalleualaglualavalthrthrhisphearghis515520525ileglyglyprocystyrserthrgluasnleuglugluleuileasp530535540leuvalserpheaspaspvalserglyglyleuargglualatrplys545550555560glntrpleumetalatrpthralalysgluserhisglyservalasp565570575glyaspthralaleuleuphevalargthrilegluilecyssergly580585590argilevalsersergluglnlysleuasnleutrpasptyrsergln595600605leugluglnleuthrserserilecyshislysleualathrilegly610615620leuserglnasnglualasermetgluasnthrgluaspleuhisgln625630635640glnvalaspleuglumetglngluleusertrpargvalhisglngly645650655cyshisglyileasnarggluthrargglnthrpheleuasnvalval660665670lysserphetyrtyrseralahiscysserprogluthrvalaspser675680685hisilealalysvalilepheglnaspvalile690695<210>16<211>2100<212>dna<213>人工序列(artificialsequence)<223>经优化的用于大肠杆菌(e.coli)表达的编码tatps1-del59的cdna<400>16atgtatcgccaaagaactgatgagccaagcgaaacccgccagatgatcgatgatattcgc60accgctttggctagcctgggtgacgatgaaaccagcatgagcgtgagcgcatacgacacc120gccctggttgccctggtgaagaacctggacggtggcgatggcccgcagttcccgagctgc180attgactggattgttcagaaccagctgccggacggtagctggggcgacccggctttcttt240atggttcaggaccgtatgatcagcaccctggcctgtgtcgtggccgtgaaatcctggaat300atcgatcgtgacaacttgtgcgatcgtggtgtcctgtttatcaaagaaaacatgtcgcgt360ctggttgaagaagaacaagattggatgccatgtggcttcgagattaactttcctgcactg420ttggagaaagctaaagacctggacttggacattccgtacgatcatcctgtgctggaagag480atttacgcgaagcgtaatctgaaactgctgaagattccgttagatgtcctccatgcgatc540ccgacgacgctgttgttttccgttgagggtatggtcgatctgccgctggattgggagaaa600ctgctgcgtctgcgttgcccggacggttcttttcattctagcccggcggcgacggcagcg660gcgctgagccacacgggtgacaaagagtgtcacgccttcctggaccgcctgattcaaaag720ttcgagggtggcgtcccgtgctcccacagcatggacaccttcgagcaactgtgggttgtt780gaccgtttgatgcgtctgggtatcagccgtcattttacgagcgagatccagcagtgcttg840gagttcatctatcgtcgttggacccagaaaggtctggcgcacaatatgcactgcccgatc900ccggacattgatgacactgcgatgggttttcgtctgttgagacagcacggttacgacgtg960accccgtcggttttcaagcatttcgagaaagacggcaagttcgtatgcttcccgatggaa1020accaaccatgcgagcgtgacgccgatgcacaatacctaccgtgcgagccagttcatgttc1080ccgggtgatgacgacgtgctggcccgtgccggccgctactgtcgcgcattcttgcaagag1140cgtcagagctctaacaagttgtacgataagtggattatcacgaaagatctgccgggtgag1200gttggctacacgctgaactttccgtggaaaagctccctgccgcgtattgaaactcgtatg1260tatctggatcagtacggtggcaataacgatgtctggattgcaaaggtcctgtatcgcatg1320aacctggttagcaatgacctgtacctgaaaatggcgaaagccgactttaccgagtatcaa1380cgtctgtctcgcattgagtggaacggcctgcgcaaatggtattttcgcaatcatctgcag1440cgttacggtgcgaccccgaagtccgcgctgaaagcgtatttcctggcgtcggcaaacatc1500tttgagcctggccgcgcagccgagcgcctggcatgggcacgtatggccgtgctggctgaa1560gctgtaacgactcatttccgtcacattggcggcccgtgctacagcaccgagaatctggaa1620gaactgatcgaccttgttagcttcgacgacgtgagcggcggcttgcgtgaggcgtggaag1680caatggctgatggcgtggaccgcaaaagaatcacacggcagcgtggacggtgacacggca1740ctgctgtttgtccgcacgattgagatttgcagcggccgcatcgtttccagcgagcagaaa1800ctgaatctgtgggattacagccagttagagcaattgaccagcagcatctgtcataaactg1860gccaccatcggtctgagccagaacgaagctagcatggaaaataccgaagatctgcaccaa1920caagtcgatttggaaatgcaagaactgtcatggcgtgttcaccagggttgtcacggtatt1980aatcgcgaaacccgtcaaaccttcctgaatgttgttaagtctttttattactccgcacac2040tgcagcccggaaaccgtggacagccatattgcaaaagtgatctttcaagacgttatctga2100<210>17<211>785<212>prt<213>欧夏至草(marrubiumvulgare)<400>17metglyserleuserthrleuasnleuilelysthrcysvalthrleu151015alaserserglulysleuasnglnproserglncystyrthrileser202530thrcysmetlysserserasnasnpropropheasntyrtyrglnile354045asnglyarglyslysmetserthralaileaspserservalasnala505560proprogluglnlystyrasnserthralaleugluhisaspthrglu65707580ileilegluilegluasphisileglucysileargargleuleuarg859095thralaglyaspglyargileservalserprotyraspthralatrp100105110ilealaleuilelysaspleuaspglyhisaspserproglnphepro115120125sersermetglutrpvalalaaspasnglnleuproaspglysertrp130135140glyaspgluhisphevalcysvaltyraspargleuvalasnthrile145150155160alacysvalvalalaleuargsertrpasnvalhisalahislyscys165170175glulysglyilelystyrilelysgluasnvalhislysleugluasp180185190alaasnglugluhismetthrcysglyphegluvalvalpheproala195200205leuleuglnargalaglnsermetglyilelysglyileprotyrasn210215220alaprovalileglugluiletyrasnserargglulyslysleulys225230235240argileprometgluvalvalhislysvalalathrserleuleuphe245250255serleugluglyleugluasnleuglutrpglulysleuleulysleu260265270glnserproaspglyserpheleuthrserproserserthralaphe275280285alapheilehisthrlysasparglyscyspheasnpheileasnasn290295300ilevalhisthrphelysglyglyalaprohisthrtyrprovalasp305310315320ilepheglyargleutrpalavalaspargleuglnargleuglyile325330335serargphepheglusergluilealaglupheleuserhisvalhis340345350argphetrpseraspglualaglyvalpheserglyarggluserval355360365phecysaspileaspaspthrsermetglyleuargleuleuargmet370375380hisglytyrhisvalaspproasnvalleulysasnphelysglnser385390395400asplysphesercystyrglyglyglnmetmetglucysserserpro405410415iletyrasnleutyrargalaserglnleuglnpheproglygluglu420425430ileleugluglualaasnlysphealatyrlyspheleuglnglulys435440445leugluserasnglnileleuasplystrpleuileserasnhisleu450455460seraspgluilelysvalglyleuglumetprotrptyralathrleu465470475480proargvalgluthrsertyrtyrilehishistyrglyglyglyasp485490495aspvaltrpileglylysthrleutyrargmetprogluileserasn500505510aspthrtyrarggluleualaargleuasppheargargcysglnala515520525glnhisglnleuglutrpiletyrmetglnargtrptyrglusercys530535540argmetglnglupheglyileserarglysgluvalleuargalatyr545550555560pheleualaserglythrilephegluvalgluargalalysgluarg565570575valalatrpalaargserglnileileserhismetilelysserphe580585590pheasnlysgluthrthrserseraspglnlysglnalaleuleuthr595600605gluleuleupheglyasnileseralasergluthrglulysargglu610615620leuaspglyvalvalvalalathrleuargglnpheleugluglyphe625630635640aspileglythrarghisglnvallysalaalatrpaspvaltrpleu645650655arglysvalgluglnglyglualahisglyglyalaaspalagluleu660665670cysthrthrthrleuasnthrcysalaasnglnhisleuserserhis675680685proasptyrasnthrleuserlysleuthrasnlysilecyshislys690695700leuserglnileglnhisglnlysglumetlysglyglyilelysala705710715720lyscysserileasnasnlysgluvalaspileglumetglntrpleu725730735vallysleuvalleuglulysserglyleuasnarglysalalysgln740745750alapheleuserilealalysthrtyrtyrtyrargalatyrtyrala755760765aspglnthrmetaspalahisilephelysvalleuphegluproval770775780val785<210>18<211>723<212>prt<213>人工序列(artificialsequence)<223>来自欧夏至草(marrubiumvulgare)的截短的柯巴基二磷酸合酶<400>18metalaproprogluglnlystyrasnserthralaleugluhisasp151015thrgluileilegluilegluasphisileglucysileargargleu202530leuargthralaglyaspglyargileservalserprotyraspthr354045alatrpilealaleuilelysaspleuaspglyhisaspserprogln505560pheprosersermetglutrpvalalaaspasnglnleuproaspgly65707580sertrpglyaspgluhisphevalcysvaltyraspargleuvalasn859095thrilealacysvalvalalaleuargsertrpasnvalhisalahis100105110lyscysglulysglyilelystyrilelysgluasnvalhislysleu115120125gluaspalaasnglugluhismetthrcysglyphegluvalvalphe130135140proalaleuleuglnargalaglnsermetglyilelysglyilepro145150155160tyrasnalaprovalileglugluiletyrasnserargglulyslys165170175leulysargileprometgluvalvalhislysvalalathrserleu180185190leupheserleugluglyleugluasnleuglutrpglulysleuleu195200205lysleuglnserproaspglyserpheleuthrserproserserthr210215220alaphealapheilehisthrlysasparglyscyspheasnpheile225230235240asnasnilevalhisthrphelysglyglyalaprohisthrtyrpro245250255valaspilepheglyargleutrpalavalaspargleuglnargleu260265270glyileserargphepheglusergluilealaglupheleuserhis275280285valhisargphetrpseraspglualaglyvalpheserglyargglu290295300servalphecysaspileaspaspthrsermetglyleuargleuleu305310315320argmethisglytyrhisvalaspproasnvalleulysasnphelys325330335glnserasplysphesercystyrglyglyglnmetmetglucysser340345350serproiletyrasnleutyrargalaserglnleuglnpheprogly355360365glugluileleugluglualaasnlysphealatyrlyspheleugln370375380glulysleugluserasnglnileleuasplystrpleuileserasn385390395400hisleuseraspgluilelysvalglyleuglumetprotrptyrala405410415thrleuproargvalgluthrsertyrtyrilehishistyrglygly420425430glyaspaspvaltrpileglylysthrleutyrargmetprogluile435440445serasnaspthrtyrarggluleualaargleuasppheargargcys450455460glnalaglnhisglnleuglutrpiletyrmetglnargtrptyrglu465470475480sercysargmetglnglupheglyileserarglysgluvalleuarg485490495alatyrpheleualaserglythrilephegluvalgluargalalys500505510gluargvalalatrpalaargserglnileileserhismetilelys515520525serphepheasnlysgluthrthrserseraspglnlysglnalaleu530535540leuthrgluleuleupheglyasnileseralasergluthrglulys545550555560arggluleuaspglyvalvalvalalathrleuargglnpheleuglu565570575glypheaspileglythrarghisglnvallysalaalatrpaspval580585590trpleuarglysvalgluglnglyglualahisglyglyalaaspala595600605gluleucysthrthrthrleuasnthrcysalaasnglnhisleuser610615620serhisproasptyrasnthrleuserlysleuthrasnlysilecys625630635640hislysleuserglnileglnhisglnlysglumetlysglyglyile645650655lysalalyscysserileasnasnlysgluvalaspileglumetgln660665670trpleuvallysleuvalleuglulysserglyleuasnarglysala675680685lysglnalapheleuserilealalysthrtyrtyrtyrargalatyr690695700tyralaaspglnthrmetaspalahisilephelysvalleupheglu705710715720provalval<210>19<211>2172<212>dna<213>人工序列(artificialsequence)<223>经优化的用于大肠杆菌(e.coli)表达的编码mvcps3-del63的cdna<400>19atggccccgccggaacaaaagtacaacagcactgcattagaacacgacaccgagattatt60gagatcgaggaccacatcgagtgtatccgccgtctgctgcgtaccgcgggtgatggtcgt120attagcgtgagcccgtatgataccgcgtggattgcactgattaaagatttggatggccac180gactccccgcaattcccgtcgagcatggaatgggttgctgataatcagctgccggacggt240agctggggtgacgagcacttcgtttgcgtttacgatcgcctggttaataccatcgcatgc300gtcgtggcgctgcgcagctggaatgtccatgcacataagtgcgagaaaggtattaagtac360attaaagaaaatgtccacaaactggaagatgcgaacgaagaacacatgacttgcggcttc420gaagtcgtttttccggccttgctgcagcgtgcacagagcatgggtattaagggcatcccg480tacaacgcgcctgtcattgaagaaatttacaattcccgtgagaaaaagctgaaacgtatt540ccgatggaagttgtccacaaagtcgcgaccagcctgctgttctccctggaaggtctggag600aacctggagtgggagaaattgctgaaactgcagagcccggacggttcgtttctgaccagc660ccgagctctacggcattcgcgtttatccataccaaagaccgtaaatgttttaactttatt720aacaatatcgttcatacctttaagggtggtgcaccgcacacgtaccctgtggacatcttt780ggccgcctgtgggcagtggatcgcttgcagcgtctgggtattagccgcttcttcgagagc840gagatcgcggaatttctgagccacgtgcaccgtttttggagcgacgaagcgggcgttttc900agcggccgtgagagcgtgttctgtgatattgatgacaccagcatgggtctgcgcctgctt960cgtatgcatggctaccatgtagacccaaacgttctgaagaacttcaagcaatctgacaag1020tttagctgctacggtggccagatgatggaatgcagcagcccaatttacaatctgtaccgt1080gcgagccaactgcaatttccgggtgaagaaatcttggaagaggctaacaaattcgcgtat1140aagtttttgcaagagaaactggagtccaatcagattctggacaagtggctgatctccaac1200cacctgagcgacgaaatcaaagttggcctggaaatgccgtggtatgcgaccttgccgcgc1260gttgagactagctattatattcaccattacggcggtggcgacgatgtgtggattggtaaa1320acgctgtatcgcatgccggaaattagcaacgacacctaccgtgagctggcacgtctggac1380ttccgccgctgccaggcgcagcaccagttggaatggatctatatgcaacgttggtatgag1440agctgtcgtatgcaagaatttggtatttcccgcaaagaagtcctgcgtgcctacttcctg1500gcctctggcacgattttcgaagttgagcgcgccaaagagcgcgtggcgtgggctcgtagc1560caaatcatttcccacatgatcaagagcttcttcaataaagaaaccacgagcagcgatcag1620aaacaagcgctgctgaccgagttgctgtttggtaacatctctgcaagcgagactgagaaa1680cgtgagctggatggtgttgtggttgcgaccctgcgtcagttcctggaaggcttcgatatc1740ggcacccgtcaccaagtgaaggcagcgtgggatgtgtggctgcgtaaagtcgaacagggt1800gaggcacatggtggcgcggacgccgagttgtgtacgacgacgctgaacacgtgcgcgaat1860cagcatctgtctagccatccggactacaataccctgtcgaaactcaccaataagatttgt1920cacaagctgtcccaaatccagcatcagaaagaaatgaagggcggtattaaggcaaagtgc1980tctatcaataacaaagaagtggatatcgagatgcaatggctggtcaaactggtcctggag2040aaatccggtctgaaccgcaaggctaaacaagcgtttctgagcattgccaaaacctattat2100tatcgtgcttactatgccgaccagacgatggatgcccacatcttcaaggtcctgtttgaa2160ccggtcgtgtaa2172<210>20<211>799<212>prt<213>迷迭香(rosmarinusofficinalis)<400>20metthrsermetserserleuasnleuserargalaproalaileser151015argargleuglnleuproalalysvalglnleuprogluphetyrala202530valcyssertrpleuasnasnserserlyshisthrproleusercys354045hisilehisarglysglnleuserlysvalthrlyscysargvalala505560serleuaspalaserglnvalserglulysglythrserserproval65707580glnthrproglugluvalasnglulysilegluasntyrileglutyr859095ilelysasnleuleuthrthrserglyaspglyargileservalser100105110protyraspthrserilevalalaleuilelysaspleulysglyarg115120125aspthrproglnpheprosercysleuglutrpilealaglnhisgln130135140metalaaspglysertrpglyaspgluphephecysiletyrasparg145150155160ileleuasnthrleualacysvalvalalaleulyssertrpasnval165170175hisalaaspmetileglulysglyvalthrtyrvalasngluasnval180185190glnlysleugluaspglyasnleugluhismetthrserglypheglu195200205ilevalvalproalaleuvalglnargalaglnaspleuglyilegln210215220glyleuprotyrasphisproleuilelysgluilealaasnthrlys225230235240gluglyargleulyslysileprolysaspmetiletyrglnlyspro245250255thrthrleuleupheserleugluglyleuglyaspleuglutrpglu260265270lysileleulysleuglnserglyaspglyserpheleuthrserpro275280285serserthralahisvalphemetlysthrlysaspglulyscysleu290295300lyspheilegluasnalavallysasncysasnglyglyalaprohis305310315320thrtyrprovalaspvalphealaargleutrpalavalaspargleu325330335glnargleuglyileserargphepheglnglngluilelystyrphe340345350leuasphisileasnservaltrpthrgluasnglyvalphesergly355360365argaspsergluphecysaspileaspaspthrsermetglyilearg370375380leuleulysmethisglytyraspileaspproasnalaleugluhis385390395400phelysglnglnaspglylysphesercystyrglyglyglnmetile405410415gluseralaserproiletyrasnleutyrargalaalaglnleuarg420425430pheproglyglugluileleugluglualathrlysphealatyrasn435440445pheleuglnglulysilealaasnaspglnpheglnglulystrpval450455460ileserasphisleuileaspgluvallysleuglyleulysmetpro465470475480trptyralathrleuproargvalglualaalatyrtyrleuglntyr485490495tyralaglycysglyaspvaltrpileglylysvalphetyrargmet500505510progluileserasnaspthrtyrlyslysleualaileleuaspphe515520525asnargcysglnalaglnhisglnpheglutrpiletyrmetglnglu530535540trptyrhisargserservalserglupheglyileserlyslysasp545550555560leuleuargalatyrpheleualaalaalathrilephegluproglu565570575argthrglngluargleuvaltrpalalysthrglnilevalsergly580585590metilethrserphevalasnserglythrthrleuserleuhisgln595600605lysthralaleuleuserglnileglyhisasnpheaspglyleuasp610615620gluileileseralametlysasphisglyleualaalathrleuleu625630635640thrthrpheglnglnleuleuaspglypheaspargtyrthrarghis645650655glnleulysasnalatrpserglntrpphemetlysleuglnglngly660665670glualaserglyglygluaspalagluleuleualaasnthrleuasn675680685ilecysalaglyleuilealapheasngluaspvalleuserhishis690695700glutyrthrthrleuserthrleuthrasnlysilecyslysargleu705710715720thrglnileglnasplyslysthrleugluvalvalaspglyserile725730735lysasplysgluleuglulysaspileglnmetleuvallysleuval740745750leuglugluasnglyglyglyvalaspargasnilelyshisthrphe755760765leuservalphelysthrphetyrtyrasnalatyrhisaspaspglu770775780thrthraspvalhisilephelysvalleupheglyprovalval785790795<210>21<211>733<212>prt<213>人工序列(artificialsequence)<223>来自迷迭香(rosmarinusofficinalis)的截短的柯巴基二磷酸合酶<400>21metalaserglnvalserglulysglythrserserprovalglnthr151015proglugluvalasnglulysilegluasntyrileglutyrilelys202530asnleuleuthrthrserglyaspglyargileservalserprotyr354045aspthrserilevalalaleuilelysaspleulysglyargaspthr505560proglnpheprosercysleuglutrpilealaglnhisglnmetala65707580aspglysertrpglyaspgluphephecysiletyraspargileleu859095asnthrleualacysvalvalalaleulyssertrpasnvalhisala100105110aspmetileglulysglyvalthrtyrvalasngluasnvalglnlys115120125leugluaspglyasnleugluhismetthrserglyphegluileval130135140valproalaleuvalglnargalaglnaspleuglyileglnglyleu145150155160protyrasphisproleuilelysgluilealaasnthrlysglugly165170175argleulyslysileprolysaspmetiletyrglnlysprothrthr180185190leuleupheserleugluglyleuglyaspleuglutrpglulysile195200205leulysleuglnserglyaspglyserpheleuthrserproserser210215220thralahisvalphemetlysthrlysaspglulyscysleulysphe225230235240ilegluasnalavallysasncysasnglyglyalaprohisthrtyr245250255provalaspvalphealaargleutrpalavalaspargleuglnarg260265270leuglyileserargphepheglnglngluilelystyrpheleuasp275280285hisileasnservaltrpthrgluasnglyvalpheserglyargasp290295300sergluphecysaspileaspaspthrsermetglyileargleuleu305310315320lysmethisglytyraspileaspproasnalaleugluhisphelys325330335glnglnaspglylysphesercystyrglyglyglnmetilegluser340345350alaserproiletyrasnleutyrargalaalaglnleuargphepro355360365glyglugluileleugluglualathrlysphealatyrasnpheleu370375380glnglulysilealaasnaspglnpheglnglulystrpvalileser385390395400asphisleuileaspgluvallysleuglyleulysmetprotrptyr405410415alathrleuproargvalglualaalatyrtyrleuglntyrtyrala420425430glycysglyaspvaltrpileglylysvalphetyrargmetproglu435440445ileserasnaspthrtyrlyslysleualaileleuasppheasnarg450455460cysglnalaglnhisglnpheglutrpiletyrmetglnglutrptyr465470475480hisargserservalserglupheglyileserlyslysaspleuleu485490495argalatyrpheleualaalaalathrilephegluprogluargthr500505510glngluargleuvaltrpalalysthrglnilevalserglymetile515520525thrserphevalasnserglythrthrleuserleuhisglnlysthr530535540alaleuleuserglnileglyhisasnpheaspglyleuaspgluile545550555560ileseralametlysasphisglyleualaalathrleuleuthrthr565570575pheglnglnleuleuaspglypheaspargtyrthrarghisglnleu580585590lysasnalatrpserglntrpphemetlysleuglnglnglygluala595600605serglyglygluaspalagluleuleualaasnthrleuasnilecys610615620alaglyleuilealapheasngluaspvalleuserhishisglutyr625630635640thrthrleuserthrleuthrasnlysilecyslysargleuthrgln645650655ileglnasplyslysthrleugluvalvalaspglyserilelysasp660665670lysgluleuglulysaspileglnmetleuvallysleuvalleuglu675680685gluasnglyglyglyvalaspargasnilelyshisthrpheleuser690695700valphelysthrphetyrtyrasnalatyrhisaspaspgluthrthr705710715720aspvalhisilephelysvalleupheglyprovalval725730<210>22<211>2202<212>dna<213>人工序列(artificialsequence)<223>经优化的用于大肠杆菌(e.coli)表达的编码rocps1-del67的cdna<400>22atggcatcacaagttagcgagaaaggcaccagctccccagttcaaacgccagaggaagtg60aacgaaaagatcgagaattacattgagtatattaaaaatctgctgactacttcgggcgac120ggccgcatcagcgtcagcccgtacgacacgagcatcgttgccctgattaaagacctgaag180ggtcgtgacaccccgcagtttccgtcctgtctggagtggattgcccaacaccaaatggcc240gatggttcctggggtgatgaatttttctgcatttacgaccgcatcctgaatacgctggct300tgtgttgtcgccctgaagtcctggaatgttcatgcagacatgatcgaaaagggtgtcact360tacgttaacgaaaacgtgcagaaactggaagatggcaatctggagcacatgacgagcggt420ttcgagattgttgtcccggcgctggttcagagagcgcaagacctgggcatccagggcctg480ccgtatgatcatccgttgatcaaagaaatcgcaaacaccaaagagggccgcctgaagaaa540attcctaaagacatgatttatcagaaaccgactacgctgctgttcagcctggaaggcttg600ggcgacctggagtgggaaaagatcctgaagttacagtctggtgatggttctttcctgacc660agcccgagctctacggcccatgttttcatgaaaaccaaagatgagaagtgtctgaagttt720attgaaaatgccgtcaagaattgcaacggtggcgcgcctcacacctacccggtggacgtt780ttcgctcgtctgtgggccgtcgatcgtctgcaacgcctgggcatctcgcgtttcttccag840caagagattaagtacttcctggaccacattaatagcgtgtggaccgaaaacggcgttttc900agcggtcgcgacagcgagttttgtgatattgatgacacctctatgggtatccgtttgctg960aagatgcacggttacgacattgacccgaatgccctggagcactttaaacaacaggatggt1020aagttctcctgctacggtggtcagatgattgagagcgcgagcccgatctacaacctgtac1080cgtgctgcgcagctgcgttttccgggtgaagagattctggaagaggccaccaaatttgcg1140tataattttttgcaagagaaaattgcaaacgaccaattccaggaaaaatgggttattagc1200gatcaccttatcgatgaagtgaaactgggtttgaagatgccgtggtacgcgacgctgcca1260cgtgtcgaggcagcgtattatctgcagtattatgcgggctgtggtgatgtgtggatcggc1320aaagtgttctaccgtatgccggaaatcagcaatgacacctacaagaaactggccatcctg1380gatttcaaccgttgccaggcgcaacaccaattcgagtggatctacatgcaagagtggtat1440catcgtagcagcgtttctgagtttggcatttccaaaaaagacttgctgcgcgcgtatttt1500ctggcggcagcgaccattttcgaaccggagcgcacccaggaacgtctggtgtgggctaag1560acgcaaatcgtcagcggtatgattacgtcctttgttaatagcggtacgactctgagcctg1620caccagaaaacggcactgttgagccaaatcggtcataactttgacggcctggatgagatt1680atcagcgcgatgaaagaccacggcctggcagcgacgctgttaacgacctttcaacagctg1740ctggacggcttcgatcgctacacccgtcatcagctgaaaaacgcgtggagccagtggttc1800atgaagctgcaacagggtgaggcgtcgggtggcgaagatgctgagctgctggctaatacc1860ctgaacatttgcgcgggtttgattgcgtttaatgaagatgtgttgagccaccatgagtac1920accaccctgagcaccctgaccaacaagatctgtaagcgcttgactcaaatccaggataag1980aaaacgctggaagtcgtggatggtagcatcaaagataaagaactggaaaaagacattcaa2040atgctggtgaaactggtccttgaagagaacggcggtggcgttgaccgtaacatcaagcac2100accttcctgagcgtctttaaaaccttttattataatgcctatcatgacgatgaaacgacc2160gacgtgcacattttcaaagttctgttcggtccggtcgtgtaa2202<210>23<211>766<212>prt<213>人工序列(artificialsequence)<223>来自烟草(nicotianaglutinosa)的截短的推定香紫苏醇合酶<400>23metalaasnphehisargproserargvalargcysserhisserthr151015alaserserleugluglualalysgluargilearggluthrphegly202530lysasngluleuserprosersertyraspthralatrpvalalamet354045valproserargtyrsermetasnglnprocyspheproargcysleu505560asptrpileleugluasnglnarggluaspglysertrpglyleuasn65707580proserhisproleuleuvallysaspserleuserserthrleuala859095cysleuleualaleuarglystrpargileglyaspasnglnvalgln100105110argglyleuglypheilegluthrhisglytrpalavalaspasnval115120125aspglnileserproleuglypheaspileilepheprosermetile130135140lystyralaglulysleuasnleuaspleupropheaspproasnleu145150155160valasnmetmetleuarggluarggluleuthrilegluargalaleu165170175lysasngluphegluglyasnmetalaasnvalglutyrphealaglu180185190glyleuglygluleucyshistrplysgluilemetleuhisglnarg195200205argasnglyserleupheaspserproalathrthralaalaalaleu210215220iletyrhisglnhisaspglulyscyspheglytyrleuserserile225230235240leulysleuhisgluasntrpvalprothriletyrprothrlysval245250255hisserasnleuphephevalaspalaleuglnasnleuglyvalasp260265270argtyrphelysthrgluleulysservalleuaspgluiletyrarg275280285leutrpleuglulysasnglugluilepheseraspilealahiscys290295300alametalapheargleuleuargmetasnasntyrgluvalserser305310315320glugluleugluglyphevalaspglngluhisphephethrthrser325330335glyglylysleuileserhisvalalaileleugluleuhisargala340345350serglnvalaspileglngluglylysaspleuileleuasplysile355360365serthrtrpthrargasnphemetgluglngluleuleuaspasngln370375380ileleuaspargserlyslysglumetgluphealametarglysphe385390395400tyrglythrpheaspargvalgluthrargargtyrileglusertyr405410415lysmetaspserphelysileleulysalaalatyrargserserasn420425430ileasnasnileaspleuleulysphesergluhisasppheasnleu435440445cysglnalaarghislysglugluleuglnglnilelysargtrpphe450455460alaaspcyslysleugluglnvalglyserserglnasntyrleutyr465470475480thrsertyrpheproilealaalaileleuphegluproglutyrgly485490495aspalaargleualaphealalyscysglyileilealathrthrval500505510aspaspphepheaspglyphealacysasnglugluleuglnasnile515520525ilegluleuvalgluargtrpaspglytyrprothrvalglyphearg530535540sergluargvalargilephepheleualaleutyrlysmetileglu545550555560gluilealaalalysalagluthrlysglnglyargcysvallysasp565570575leuleuileasnleutrpileaspleuleulyscysmetleuvalglu580585590leuaspleutrplysilelysserthrthrproserilegluglutyr595600605leuserilealacysvalthrthrglyvallyscysleuileleuile610615620serleuhisleuleuglyprolysleuserlysaspvalthrgluser625630635640sergluvalseralaleutrpasncysthralavalvalalaargleu645650655asnasnaspilehissertyrlysarggluglnalagluserserthr660665670asnmetalaalaileleuileserglnserglnargthrileserglu675680685gluglualaileargglnilelysglumetmetgluserlysargarg690695700gluleuleuglymetvalleuglnasnlysgluserglnleuprogln705710715720valcyslysaspleuphetrpthrthrphelysalaalatyrserile725730735tyrthrhisglyaspglutyrargpheproglngluleulysasnhis740745750ileasnaspvaliletyrlysproleuasnglntyrserpro755760765<210>24<211>2301<212>dna<213>人工序列(artificialsequence)<223>经优化的用于大肠杆菌(e.coli)表达的编码ngscs-del29的cdna<400>24atggctaatttccatcgcccatcccgtgttcgttgttcccactctaccgcaagctccctg60gaagaggcaaaagagcgcatccgtgaaaccttcggcaaaaatgaactctctccttctagc120tatgatacggcctgggttgctatggtcccgagccgctacagcatgaaccagccgtgcttt180ccgcgctgcctggactggattctggagaaccaacgtgaggatggcagctggggtctgaac240ccgagccatccgttactggtgaaagacagcttgagcagcacgctggcgtgtttgctggcg300ctgcgtaagtggcgtattggcgacaaccaagtccagcgtggcctgggttttatcgagact360catggttgggcagtggacaacgtagaccagatctctccactgggttttgacatcattttc420ccgagcatgattaaatatgcggaaaagctgaatctggatttgccttttgatccgaacctg480gtgaacatgatgctgcgcgagcgcgagctgacgatcgagcgtgcgctgaaaaacgaattt540gagggtaatatggctaatgtcgagtacttcgccgagggtttgggtgagctgtgtcactgg600aaagaaatcatgctgcaccaacgccgtaacggtagcctgttcgactctccggcaacgacc660gccgcggctcttatttatcatcagcacgatgagaagtgcttcggctatctgtctagcatc720ctgaaattacacgagaactgggtgccgaccatctatccgaccaaggttcactccaatctg780tttttcgtcgatgcgctgcagaacctgggtgttgaccgttacttcaaaaccgaactgaag840tccgtcctggatgagatctaccgtttgtggctggagaaaaacgaagagatcttcagcgat900attgcgcactgcgcaatggcgtttcgcctgttgcgcatgaataattacgaggttagcagc960gaagaactggaaggcttcgtggaccaagaacattttttcaccacgtcgggtggcaagctg1020atcagccacgttgccatcctggaactgcaccgtgcaagccaagtggacattcaggagggc1080aaagacctgatcctggacaaaattagcacctggactcgcaactttatggaacaggaactg1140ctggataaccagatcttggatcgtagcaaaaaagaaatggaatttgcaatgcgtaagttt1200tacggtacgttcgatcgcgtggaaacccgtcgttatattgaaagctacaaaatggattcc1260ttcaagatcctgaaggcagcgtaccgtagctccaacattaacaatattgacctgttgaag1320ttcagcgagcacgacttcaatctctgccaggcgcgtcacaaggaagaactgcagcaaatc1380aaacgctggttcgcagattgcaaactggagcaagtcggtagcagccagaactacttgtac1440acctcttacttcccgatcgcggccattttgttcgagccggagtatggcgacgcacgcctg1500gcgttcgcgaagtgcggtattatcgcgaccaccgttgacgatttttttgacggttttgca1560tgtaatgaagaactgcaaaacatcatcgaactggtcgagagatgggacggttatccgacg1620gttggtttccgctccgagcgtgtgcgcattttctttctggcgctgtacaaaatgattgaa1680gaaattgccgcgaaagcggaaacgaaacagggccgttgcgtgaaagatctgttgatcaat1740ctgtggattgatctgctgaaatgcatgctggtcgaactggatctgtggaaaattaagagc1800acgaccccgagcattgaagagtatctgagcattgcctgtgtgacgaccggcgttaagtgc1860ttgatcctgattagcctgcatctgctgggcccgaaactgagcaaagacgtgaccgaatcc1920agcgaagttagcgctctgtggaactgtacggccgtggttgcgcgcctgaacaacgacatt1980catagctacaagcgtgagcaagccgagagcagcactaatatggccgcaatcctgatttcg2040caaagccagcgtaccatctcagaagaagaagctatccgccagatcaaagagatgatggaa2100tcgaaacgccgtgagctgctgggcatggtgctgcagaataaagagagccaattgccgcaa2160gtctgcaaagacctgttttggaccaccttcaaagccgcgtacagcatttatacccacggt2220gatgagtaccgttttccacaagaactgaagaaccatatcaacgatgtcatctataagccg2280ttaaatcaatacagcccttaa2301<210>25<211>755<212>prt<213>烟草(nicotianaglutinosa)<400>25metserhisserthralaserserleugluglualalysgluargile151015arggluthrpheglylysasngluleusersersersertyraspthr202530alatrpvalalametvalproserargtyrsermetasnglnprocys354045pheproargcysleuasptrpileleugluasnglnarggluaspgly505560sertrpglyleuasnproserleuproleuleuvallysaspserleu65707580serserthrleualacysleuleualaleuarglystrpargilegly859095aspasnglnvalglnargglyleuglypheilegluthrhisglytrp100105110alavalaspasnvalaspglnileserproleuglypheaspileile115120125pheprosermetilelystyralaglulysleuasnleuaspleupro130135140pheaspproasnleuvalasnmetmetleuarggluarggluleuthr145150155160ilegluargalaleulysasngluphegluglyasnmetalaasnval165170175glutyrphealagluglyleuglygluleucyshistrplysgluile180185190metleuhisglnargargasnglyserpropheaspserproalathr195200205thralaalaalaleuiletyrhisglnhisaspglulyscysphegly210215220tyrleuserserileleulysleuhisgluasntrpvalprothrile225230235240tyrprothrlysvalhisserasnleuphephevalaspalaleugln245250255asnleuglyvalaspargtyrphelysthrgluleulysservalleu260265270aspgluiletyrargleutrpleuglulysasnglugluilepheser275280285aspilealahiscysalametalapheargleuleuargmetasnasn290295300tyrgluvalserserglugluleugluglyphevalaspglngluhis305310315320phephethrthrserglyglylysleuileserhisvalalaileleu325330335gluleuhisargalaserglnvalaspileglngluglylysaspleu340345350ileleuasplysileserthrtrpthrargasnphemetgluglnglu355360365leuleuaspasnglnileleuaspargserlyslysglumetgluphe370375380alametarglysphetyrglythrpheaspargvalgluthrargarg385390395400tyrileglusertyrlysmetaspserphelysileleulysalaala405410415tyrargserserasnileasnasnileaspleuleulyspheserglu420425430hisasppheasnleucysglnalaarghislysglugluleuglngln435440445ilelysargtrpphealaaspcyslysleugluglnvalglyserser450455460glnasntyrleutyrthrsertyrpheproilealaalaileleuphe465470475480gluproglutyrglyaspalaargleualaphealalyscysglyile485490495ilealathrthrvalaspaspphepheaspglyphealacysasnglu500505510gluleuglnasnileilegluleuvalgluargtrpaspglytyrpro515520525thrvalglypheargsergluargvalargilephepheleualaleu530535540tyrlysmetileglugluilealaalalysalagluthrlysglngly545550555560argcysvallysaspleuleuileasnleutrpileaspleuleulys565570575cysmetleuvalgluleuaspleutrplysilelysserthrthrpro580585590serilegluglutyrleuserilealacysvalthrthrglyvallys595600605cysleuileleuileserleuhisleuleuglyprolysleuserlys610615620aspvalthrglusersergluvalseralaleutrpasncysthrala625630635640valvalalaargleuasnasnaspilehissertyrlysargglugln645650655alagluserserthrasnmetvalalaileleuileserglnsergln660665670argthrileserglugluglualaileargglnilelysglumetmet675680685gluserlysargarggluleuleuglymetvalleuglnasnlysglu690695700serglnleuproglnvalcyslysaspleuphetrpthrthrphelys705710715720alaalatyrseriletyrthrhisglyaspglutyrargpheprogln725730735gluleulysasnhisileasnaspvaliletyrlysproleuasngln740745750tyrserpro755<210>26<211>2211<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(saccharomycescerevisiae)表达的编码smcps2的cdna<400>26atggctactgttgacgctccacaagttcacgaccacgacggtactactgttcaccaaggt60cacgacgctgttaagaacatcgaagacccaatcgaatacatcagaactttgttgagaact120actggtgacggtagaatctctgtttctccatacgacactgcttgggttgctatgatcaag180gacgttgaaggtagagacggtccacaattcccatcttctttggaatggatcgttcaaaac240caattggaagacggttcttggggtgaccaaaagttgttctgtgtttacgacagattggtt300aacactatcgcttgtgttgttgctttgagatcttggaacgttcacgctcacaaggttaag360agaggtgttacttacatcaaggaaaacgttgacaagttgatggaaggtaacgaagaacac420atgacttgtggtttcgaagttgttttcccagctttgttgcaaaaggctaagtctttgggt480atcgaagacttgccatacgactctccagctgttcaagaagtttaccacgttagagaacaa540aagttgaagagaatcccattggaaatcatgcacaagatcccaacttctttgttgttctct600ttggaaggtttggaaaacttggactgggacaagttgttgaagttgcaatctgctgacggt660tctttcttgacttctccatcttctactgctttcgctttcatgcaaactaaggacgaaaag720tgttaccaattcatcaagaacactatcgacactttcaacggtggtgctccacacacttac780ccagttgacgttttcggtagattgtgggctatcgacagattgcaaagattgggtatctct840agattcttcgaaccagaaatcgctgactgtttgtctcacatccacaagttctggactgac900aagggtgttttctctggtagagaatctgaattctgtgacatcgacgacacttctatgggt960atgagattgatgagaatgcacggttacgacgttgacccaaacgttttgagaaacttcaag1020caaaaggacggtaagttctcttgttacggtggtcaaatgatcgaatctccatctccaatc1080tacaacttgtacagagcttctcaattgagattcccaggtgaagaaatcttggaagacgct1140aagagattcgcttacgacttcttgaaggaaaagttggctaacaaccaaatcttggacaag1200tgggttatctctaagcacttgccagacgaaatcaagttgggtttggaaatgccatggttg1260gctactttgccaagagttgaagctaagtactacatccaatactacgctggttctggtgac1320gtttggatcggtaagactttgtacagaatgccagaaatctctaacgacacttaccacgac1380ttggctaagactgacttcaagagatgtcaagctaagcaccaattcgaatggttgtacatg1440caagaatggtacgaatcttgtggtatcgaagaattcggtatctctagaaaggacttgttg1500ttgtcttacttcttggctactgcttctatcttcgaattggaaagaactaacgaaagaatc1560gcttgggctaagtctcaaatcatcgctaagatgatcacttctttcttcaacaaggaaact1620acttctgaagaagacaagagagctttgttgaacgaattgggtaacatcaacggtttgaac1680gacactaacggtgctggtagagaaggtggtgctggttctatcgctttggctactttgact1740caattcttggaaggtttcgacagatacactagacaccaattgaagaacgcttggtctgtt1800tggttgactcaattgcaacacggtgaagctgacgacgctgaattgttgactaacactttg1860aacatctgtgctggtcacatcgctttcagagaagaaatcttggctcacaacgaatacaag1920gctttgtctaacttgacttctaagatctgtagacaattgtctttcatccaatctgaaaag1980gaaatgggtgttgaaggtgaaatcgctgctaagtcttctatcaagaacaaggaattggaa2040gaagacatgcaaatgttggttaagttggttttggaaaagtacggtggtatcgacagaaac2100atcaagaaggctttcttggctgttgctaagacttactactacagagcttaccacgctgct2160gacactatcgacactcacatgttcaaggttttgttcgaaccagttgcttaa2211<210>27<211>1578<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码来自快乐鼠尾草(salviasclarea)的截短的ssscs的cdna<400>27atggctaagatgaaggaaaacttcaagagagaagacgacaagttcccaactactactact60ttgagatctgaagacatcccatctaacttgtgtatcatcgacactttgcaaagattgggt120gttgaccaattcttccaatacgaaatcaacactatcttggacaacactttcagattgtgg180caagaaaagcacaaggttatctacggtaacgttactactcacgctatggctttcagattg240ttgagagttaagggttacgaagtttcttctgaagaattggctccatacggtaaccaagaa300gctgtttctcaacaaactaacgacttgccaatgatcatcgaattgtacagagctgctaac360gaaagaatctacgaagaagaaagatctttggaaaagatcttggcttggactactatcttc420ttgaacaagcaagttcaagacaactctatcccagacaagaagttgcacaagttggttgaa480ttctacttgagaaactacaagggtatcactatcagattgggtgctagaagaaacttggaa540ttgtacgacatgacttactaccaagctttgaagtctactaacagattctctaacttgtgt600aacgaagacttcttggttttcgctaagcaagacttcgacatccacgaagctcaaaaccaa660aagggtttgcaacaattgcaaagatggtacgctgactgtagattggacactttgaacttc720ggtagagacgttgttatcatcgctaactacttggcttctttgatcatcggtgaccacgct780ttcgactacgttagattggctttcgctaagacttctgttttggttactatcatggacgac840ttcttcgactgtcacggttcttctcaagaatgtgacaagatcatcgaattggttaaggaa900tggaaggaaaacccagacgctgaatacggttctgaagaattggaaatcttgttcatggct960ttgtacaacactgttaacgaattggctgaaagagctagagttgaacaaggtagatctgtt1020aaggaattcttggttaagttgtgggttgaaatcttgtctgctttcaagatcgaattggac1080acttggtctaacggtactcaacaatctttcgacgaatacatctcttcttcttggttgtct1140aacggttctagattgactggtttgttgactatgcaattcgttggtgttaagttgtctgac1200gaaatgttgatgtctgaagaatgtactgacttggctagacacgtttgtatggttggtaga1260ttgttgaacgacgtttgttcttctgaaagagaaagagaagaaaacatcgctggtaagtct1320tactctatcttgttggctactgaaaaggacggtagaaaggtttctgaagacgaagctatc1380gctgaaatcaacgaaatggttgaataccactggagaaaggttttgcaaatcgtttacaag1440aaggaatctatcttgccaagaagatgtaaggacgttttcttggaaatggctaagggtact1500ttctacgcttacggtatcaacgacgaattgacttctccacaacaatctaaggaagacatg1560aagtctttcgttttctaa1578<210>28<211>924<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码来自成团泛菌(pantoeaagglomerans)的ggpp合酶的cdna<400>28atggtttctggttctaaggctggtgtttctccacacagagaaatcgaagttatgagacaa60tctatcgacgaccacttggctggtttgttgccagaaactgactctcaagacatcgtttct120ttggctatgagagaaggtgttatggctccaggtaagagaatcagaccattgttgatgttg180ttggctgctagagacttgagataccaaggttctatgccaactttgttggacttggcttgt240gctgttgaattgactcacactgcttctttgatgttggacgacatgccatgtatggacaac300gctgaattgagaagaggtcaaccaactactcacaagaagttcggtgaatctgttgctatc360ttggcttctgttggtttgttgtctaaggctttcggtttgatcgctgctactggtgacttg420ccaggtgaaagaagagctcaagctgttaacgaattgtctactgctgttggtgttcaaggt480ttggttttgggtcaattcagagacttgaacgacgctgctttggacagaactccagacgct540atcttgtctactaaccacttgaagactggtatcttgttctctgctatgttgcaaatcgtt600gctatcgcttctgcttcttctccatctactagagaaactttgcacgctttcgctttggac660ttcggtcaagctttccaattgttggacgacttgagagacgaccacccagaaactggtaag720gacagaaacaaggacgctggtaagtctactttggttaacagattgggtgctgacgctgct780agacaaaagttgagagaacacatcgactctgctgacaagcacttgactttcgcttgtcca840caaggtggtgctatcagacaattcatgcacttgtggttcggtcaccacttggctgactgg900tctccagttatgaagatcgcttaa924<210>29<211>2175<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码cfcps1-del63的cdna<400>29atggttgctactgttaacgctccaccagttcacgaccaagacgactctactgaaaaccaa60tgtcacgacgctgttaacaacatcgaagacccaatcgaatacatcagaactttgttgaga120actactggtgacggtagaatctctgtttctccatacgacactgcttgggttgctttgatc180aaggacttgcaaggtagagacgctccagaattcccatcttctttggaatggatcatccaa240aaccaattggctgacggttcttggggtgacgctaagttcttctgtgtttacgacagattg300gttaacactatcgcttgtgttgttgctttgagatcttgggacgttcacgctgaaaaggtt360gaaagaggtgttagatacatcaacgaaaacgttgaaaagttgagagacggtaacgaagaa420cacatgacttgtggtttcgaagttgttttcccagctttgttgcaaagagctaagtctttg480ggtatccaagacttgccatacgacgctccagttatccaagaaatctaccactctagagaa540caaaagtctaagagaatcccattggaaatgatgcacaaggttccaacttctttgttgttc600tctttggaaggtttggaaaacttggaatgggacaagttgttgaagttgcaatctgctgac660ggttctttcttgacttctccatcttctactgctttcgctttcatgcaaactagagaccca720aagtgttaccaattcatcaagaacactatccaaactttcaacggtggtgctccacacact780tacccagttgacgttttcggtagattgtgggctatcgacagattgcaaagattgggtatc840tctagattcttcgaatctgaaatcgctgactgtatcgctcacatccacagattctggact900gaaaagggtgttttctctggtagagaatctgaattctgtgacatcgacgacacttctatg960ggtgttagattgatgagaatgcacggttacgacgttgacccaaacgttttgaagaacttc1020aagaaggacgacaagttctcttgttacggtggtcaaatgatcgaatctccatctccaatc1080tacaacttgtacagagcttctcaattgagattcccaggtgaacaaatcttggaagacgct1140aacaagttcgcttacgacttcttgcaagaaaagttggctcacaaccaaatcttggacaag1200tgggttatctctaagcacttgccagacgaaatcaagttgggtttggaaatgccatggtac1260gctactttgccaagagttgaagctagatactacatccaatactacgctggttctggtgac1320gtttggatcggtaagactttgtacagaatgccagaaatctctaacgacacttaccacgaa1380ttggctaagactgacttcaagagatgtcaagctcaacaccaattcgaatggatctacatg1440caagaatggtacgaatcttgtaacatggaagaattcggtatctctagaaaggaattgttg1500gttgcttacttcttggctactgcttctatcttcgaattggaaagagctaacgaaagaatc1560gcttgggctaagtctcaaatcatctctactatcatcgcttctttcttcaacaaccaaaac1620acttctccagaagacaagttggctttcttgactgacttcaagaacggtaactctactaac1680atggctttggttactttgactcaattcttggaaggtttcgacagatacacttctcaccaa1740ttgaagaacgcttggtctgtttggttgagaaagttgcaacaaggtgaaggtaacggtggt1800gctgacgctgaattgttggttaacactttgaacatctgtgctggtcacatcgctttcaga1860gaagaaatcttggctcacaacgactacaagactttgtctaacttgacttctaagatctgt1920agacaattgtctcaaatccaaaacgaaaaggaattggaaactgaaggtcaaaagacttct1980atcaagaacaaggaattggaagaagacatgcaaagattggttaagttggttttggaaaag2040tctagagttggtatcaacagagacatgaagaagactttcttggctgttgttaagacttac2100tactacaaggcttaccactctgctcaagctatcgacaaccacatgttcaaggttttgttc2160gaaccagttgcttaa2175<210>30<211>2100<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码tatps1-del59的cdna<400>30atgtacagacaaagaactgacgaaccatctgaaactagacaaatgatcgacgacatcaga60actgctttggcttctttgggtgacgacgaaacttctatgtctgtttctgcttacgacact120gctttggttgctttggttaagaacttggacggtggtgacggtccacaattcccatcttgt180atcgactggatcgttcaaaaccaattgccagacggttcttggggtgacccagctttcttc240atggttcaagacagaatgatctctactttggcttgtgttgttgctgttaagtcttggaac300atcgacagagacaacttgtgtgacagaggtgttttgttcatcaaggaaaacatgtctaga360ttggttgaagaagaacaagactggatgccatgtggtttcgaaatcaacttcccagctttg420ttggaaaaggctaaggacttggacttggacatcccatacgaccacccagttttggaagaa480atctacgctaagagaaacttgaagttgttgaagatcccattggacgttttgcacgctatc540ccaactactttgttgttctctgttgaaggtatggttgacttgccattggactgggaaaag600ttgttgagattgagatgtccagacggttctttccactcttctccagctgctactgctgct660gctttgtctcacactggtgacaaggaatgtcacgctttcttggacagattgatccaaaag720ttcgaaggtggtgttccatgttctcactctatggacactttcgaacaattgtgggttgtt780gacagattgatgagattgggtatctctagacacttcacttctgaaatccaacaatgtttg840gaattcatctacagaagatggactcaaaagggtttggctcacaacatgcactgtccaatc900ccagacatcgacgacactgctatgggtttcagattgttgagacaacacggttacgacgtt960actccatctgttttcaagcacttcgaaaaggacggtaagttcgtttgtttcccaatggaa1020actaaccacgcttctgttactccaatgcacaacacttacagagcttctcaattcatgttc1080ccaggtgacgacgacgttttggctagagctggtagatactgtagagctttcttgcaagaa1140agacaatcttctaacaagttgtacgacaagtggatcatcactaaggacttgccaggtgaa1200gttggttacactttgaacttcccatggaagtcttctttgccaagaatcgaaactagaatg1260tacttggaccaatacggtggtaacaacgacgtttggatcgctaaggttttgtacagaatg1320aacttggtttctaacgacttgtacttgaagatggctaaggctgacttcactgaataccaa1380agattgtctagaatcgaatggaacggtttgagaaagtggtacttcagaaaccacttgcaa1440agatacggtgctactccaaagtctgctttgaaggcttacttcttggcttctgctaacatc1500ttcgaaccaggtagagctgctgaaagattggcttgggctagaatggctgttttggctgaa1560gctgttactactcacttcagacacatcggtggtccatgttactctactgaaaacttggaa1620gaattgatcgacttggtttctttcgacgacgtttctggtggtttgagagaagcttggaag1680caatggttgatggcttggactgctaaggaatctcacggttctgttgacggtgacactgct1740ttgttgttcgttagaactatcgaaatctgttctggtagaatcgtttcttctgaacaaaag1800ttgaacttgtgggactactctcaattggaacaattgacttcttctatctgtcacaagttg1860gctactatcggtttgtctcaaaacgaagcttctatggaaaacactgaagacttgcaccaa1920caagttgacttggaaatgcaagaattgtcttggagagttcaccaaggttgtcacggtatc1980aacagagaaactagacaaactttcttgaacgttgttaagtctttctactactctgctcac2040tgttctccagaaactgttgactctcacatcgctaaggttatcttccaagacgttatctaa2100<210>31<211>2172<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码mvcps3-del63的cdna<400>31atggctccaccagaacaaaagtacaactctactgctttggaacacgacactgaaatcatc60gaaatcgaagaccacatcgaatgtatcagaagattgttgagaactgctggtgacggtaga120atctctgtttctccatacgacactgcttggatcgctttgatcaaggacttggacggtcac180gactctccacaattcccatcttctatggaatgggttgctgacaaccaattgccagacggt240tcttggggtgacgaacacttcgtttgtgtttacgacagattggttaacactatcgcttgt300gttgttgctttgagatcttggaacgttcacgctcacaagtgtgaaaagggtatcaagtac360atcaaggaaaacgttcacaagttggaagacgctaacgaagaacacatgacttgtggtttc420gaagttgttttcccagctttgttgcaaagagctcaatctatgggtatcaagggtatccca480tacaacgctccagttatcgaagaaatctacaactctagagaaaagaagttgaagagaatc540ccaatggaagttgttcacaaggttgctacttctttgttgttctctttggaaggtttggaa600aacttggaatgggaaaagttgttgaagttgcaatctccagacggttctttcttgacttct660ccatcttctactgctttcgctttcatccacactaaggacagaaagtgtttcaacttcatc720aacaacatcgttcacactttcaagggtggtgctccacacacttacccagttgacatcttc780ggtagattgtgggctgttgacagattgcaaagattgggtatctctagattcttcgaatct840gaaatcgctgaattcttgtctcacgttcacagattctggtctgacgaagctggtgttttc900tctggtagagaatctgttttctgtgacatcgacgacacttctatgggtttgagattgttg960agaatgcacggttaccacgttgacccaaacgttttgaagaacttcaagcaatctgacaag1020ttctcttgttacggtggtcaaatgatggaatgttcttctccaatctacaacttgtacaga1080gcttctcaattgcaattcccaggtgaagaaatcttggaagaagctaacaagttcgcttac1140aagttcttgcaagaaaagttggaatctaaccaaatcttggacaagtggttgatctctaac1200cacttgtctgacgaaatcaaggttggtttggaaatgccatggtacgctactttgccaaga1260gttgaaacttcttactacatccaccactacggtggtggtgacgacgtttggatcggtaag1320actttgtacagaatgccagaaatctctaacgacacttacagagaattggctagattggac1380ttcagaagatgtcaagctcaacaccaattggaatggatctacatgcaaagatggtacgaa1440tcttgtagaatgcaagaattcggtatctctagaaaggaagttttgagagcttacttcttg1500gcttctggtactatcttcgaagttgaaagagctaaggaaagagttgcttgggctagatct1560caaatcatctctcacatgatcaagtctttcttcaacaaggaaactacttcttctgaccaa1620aagcaagctttgttgactgaattgttgttcggtaacatctctgcttctgaaactgaaaag1680agagaattggacggtgttgttgttgctactttgagacaattcttggaaggtttcgacatc1740ggtactagacaccaagttaaggctgcttgggacgtttggttgagaaaggttgaacaaggt1800gaagctcacggtggtgctgacgctgaattgtgtactactactttgaacacttgtgctaac1860caacacttgtcttctcacccagactacaacactttgtctaagttgactaacaagatctgt1920cacaagttgtctcaaatccaacaccaaaaggaaatgaagggtggtatcaaggctaagtgt1980tctatcaacaacaaggaagttgacatcgaaatgcaatggttggttaagttggttttggaa2040aagtctggtttgaacagaaaggctaagcaagctttcttgtctatcgctaagacttactac2100tacagagcttactacgctgaccaaactatggacgctcacatcttcaaggttttgttcgaa2160ccagttgtttaa2172<210>32<211>2202<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码rocps1-del67的cdna<400>32atggcttctcaagtttctgaaaagggtacttcttctccagttcaaactccagaagaagtt60aacgaaaagatcgaaaactacatcgaatacatcaagaacttgttgactacttctggtgac120ggtagaatctctgtttctccatacgacacttctatcgttgctttgatcaaggacttgaag180ggtagagacactccacaattcccatcttgtttggaatggatcgctcaacaccaaatggct240gacggttcttggggtgacgaattcttctgtatctacgacagaatcttgaacactttggct300tgtgttgttgctttgaagtcttggaacgttcacgctgacatgatcgaaaagggtgttact360tacgttaacgaaaacgttcaaaagttggaagacggtaacttggaacacatgacttctggt420ttcgaaatcgttgttccagctttggttcaaagagctcaagacttgggtatccaaggtttg480ccatacgaccacccattgatcaaggaaatcgctaacactaaggaaggtagattgaagaag540atcccaaaggacatgatctaccaaaagccaactactttgttgttctctttggaaggtttg600ggtgacttggaatgggaaaagatcttgaagttgcaatctggtgacggttctttcttgact660tctccatcttctactgctcacgttttcatgaagactaaggacgaaaagtgtttgaagttc720atcgaaaacgctgttaagaactgtaacggtggtgctccacacacttacccagttgacgtt780ttcgctagattgtgggctgttgacagattgcaaagattgggtatctctagattcttccaa840caagaaatcaagtacttcttggaccacatcaactctgtttggactgaaaacggtgttttc900tctggtagagactctgaattctgtgacatcgacgacacttctatgggtatcagattgttg960aagatgcacggttacgacatcgacccaaacgctttggaacacttcaagcaacaagacggt1020aagttctcttgttacggtggtcaaatgatcgaatctgcttctccaatctacaacttgtac1080agagctgctcaattgagattcccaggtgaagaaatcttggaagaagctactaagttcgct1140tacaacttcttgcaagaaaagatcgctaacgaccaattccaagaaaagtgggttatctct1200gaccacttgatcgacgaagttaagttgggtttgaagatgccatggtacgctactttgcca1260agagttgaagctgcttactacttgcaatactacgctggttgtggtgacgtttggatcggt1320aaggttttctacagaatgccagaaatctctaacgacacttacaagaagttggctatcttg1380gacttcaacagatgtcaagctcaacaccaattcgaatggatctacatgcaagaatggtac1440cacagatcttctgtttctgaattcggtatctctaagaaggacttgttgagagcttacttc1500ttggctgctgctactatcttcgaaccagaaagaactcaagaaagattggtttgggctaag1560actcaaatcgtttctggtatgatcacttctttcgttaactctggtactactttgtctttg1620caccaaaagactgctttgttgtctcaaatcggtcacaacttcgacggtttggacgaaatc1680atctctgctatgaaggaccacggtttggctgctactttgttgactactttccaacaattg1740ttggacggtttcgacagatacactagacaccaattgaagaacgcttggtctcaatggttc1800atgaagttgcaacaaggtgaagcttctggtggtgaagacgctgaattgttggctaacact1860ttgaacatctgtgctggtttgatcgctttcaacgaagacgttttgtctcaccacgaatac1920actactttgtctactttgactaacaagatctgtaagagattgactcaaatccaagacaag1980aagactttggaagttgttgacggttctatcaaggacaaggaattggaaaaggacatccaa2040atgttggttaagttggttttggaagaaaacggtggtggtgttgacagaaacatcaagcac2100actttcttgtctgttttcaagactttctactacaacgcttaccacgacgacgaaactact2160gacgttcacatcttcaaggttttgttcggtccagttgtttaa2202<210>33<211>2301<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码ngscs-del29的cdna<400>33atggctaacttccacagaccatctagagttagatgttctcactctactgcttcttctttg60gaagaagctaaggaaagaatcagagaaactttcggtaagaacgaattgtctccatcttct120tacgacactgcttgggttgctatggttccatctagatactctatgaaccaaccatgtttc180ccaagatgtttggactggatcttggaaaaccaaagagaagacggttcttggggtttgaac240ccatctcacccattgttggttaaggactctttgtcttctactttggcttgtttgttggct300ttgagaaagtggagaatcggtgacaaccaagttcaaagaggtttgggtttcatcgaaact360cacggttgggctgttgacaacgttgaccaaatctctccattgggtttcgacatcatcttc420ccatctatgatcaagtacgctgaaaagttgaacttggacttgccattcgacccaaacttg480gttaacatgatgttgagagaaagagaattgactatcgaaagagctttgaagaacgaattc540gaaggtaacatggctaacgttgaatacttcgctgaaggtttgggtgaattgtgtcactgg600aaggaaatcatgttgcaccaaagaagaaacggttctttgttcgactctccagctactact660gctgctgctttgatctaccaccaacacgacgaaaagtgtttcggttacttgtcttctatc720ttgaagttgcacgaaaactgggttccaactatctacccaactaaggttcactctaacttg780ttcttcgttgacgctttgcaaaacttgggtgttgacagatacttcaagactgaattgaag840tctgttttggacgaaatctacagattgtggttggaaaagaacgaagaaatcttctctgac900atcgctcactgtgctatggctttcagattgttgagaatgaacaactacgaagtttcttct960gaagaattggaaggtttcgttgaccaagaacacttcttcactacttctggtggtaagttg1020atctctcacgttgctatcttggaattgcacagagcttctcaagttgacatccaagaaggt1080aaggacttgatcttggacaagatctctacttggactagaaacttcatggaacaagaattg1140ttggacaaccaaatcttggacagatctaagaaggaaatggaattcgctatgagaaagttc1200tacggtactttcgacagagttgaaactagaagatacatcgaatcttacaagatggactct1260ttcaagatcttgaaggctgcttacagatcttctaacatcaacaacatcgacttgttgaag1320ttctctgaacacgacttcaacttgtgtcaagctagacacaaggaagaattgcaacaaatc1380aagagatggttcgctgactgtaagttggaacaagttggttcttctcaaaactacttgtac1440acttcttacttcccaatcgctgctatcttgttcgaaccagaatacggtgacgctagattg1500gctttcgctaagtgtggtatcatcgctactactgttgacgacttcttcgacggtttcgct1560tgtaacgaagaattgcaaaacatcatcgaattggttgaaagatgggacggttacccaact1620gttggtttcagatctgaaagagttagaatcttcttcttggctttgtacaagatgatcgaa1680gaaatcgctgctaaggctgaaactaagcaaggtagatgtgttaaggacttgttgatcaac1740ttgtggatcgacttgttgaagtgtatgttggttgaattggacttgtggaagatcaagtct1800actactccatctatcgaagaatacttgtctatcgcttgtgttactactggtgttaagtgt1860ttgatcttgatctctttgcacttgttgggtccaaagttgtctaaggacgttactgaatct1920tctgaagtttctgctttgtggaactgtactgctgttgttgctagattgaacaacgacatc1980cactcttacaagagagaacaagctgaatcttctactaacatggctgctatcttgatctct2040caatctcaaagaactatctctgaagaagaagctatcagacaaatcaaggaaatgatggaa2100tctaagagaagagaattgttgggtatggttttgcaaaacaaggaatctcaattgccacaa2160gtttgtaaggacttgttctggactactttcaaggctgcttactctatctacactcacggt2220gacgaatacagattcccacaagaattgaagaaccacatcaacgacgttatctacaagcca2280ttgaaccaatactctccataa2301<210>34<211>2268<212>dna<213>人工序列(artificialsequence)<223>经优化的用于酿酒酵母(s.cerevisiae)表达的编码ngscs-del38的cdna<400>34atgtctcactctactgcttcttctttggaagaagctaaggaaagaatcagagaaactttc60ggtaagaacgaattgtcttcttcttcttacgacactgcttgggttgctatggttccatct120agatactctatgaaccaaccatgtttcccaagatgtttggactggatcttggaaaaccaa180agagaagacggttcttggggtttgaacccatctttgccattgttggttaaggactctttg240tcttctactttggcttgtttgttggctttgagaaagtggagaatcggtgacaaccaagtt300caaagaggtttgggtttcatcgaaactcacggttgggctgttgacaacgttgaccaaatc360tctccattgggtttcgacatcatcttcccatctatgatcaagtacgctgaaaagttgaac420ttggacttgccattcgacccaaacttggttaacatgatgttgagagaaagagaattgact480atcgaaagagctttgaagaacgaattcgaaggtaacatggctaacgttgaatacttcgct540gaaggtttgggtgaattgtgtcactggaaggaaatcatgttgcaccaaagaagaaacggt600tctccattcgactctccagctactactgctgctgctttgatctaccaccaacacgacgaa660aagtgtttcggttacttgtcttctatcttgaagttgcacgaaaactgggttccaactatc720tacccaactaaggttcactctaacttgttcttcgttgacgctttgcaaaacttgggtgtt780gacagatacttcaagactgaattgaagtctgttttggacgaaatctacagattgtggttg840gaaaagaacgaagaaatcttctctgacatcgctcactgtgctatggctttcagattgttg900agaatgaacaactacgaagtttcttctgaagaattggaaggtttcgttgaccaagaacac960ttcttcactacttctggtggtaagttgatctctcacgttgctatcttggaattgcacaga1020gcttctcaagttgacatccaagaaggtaaggacttgatcttggacaagatctctacttgg1080actagaaacttcatggaacaagaattgttggacaaccaaatcttggacagatctaagaag1140gaaatggaattcgctatgagaaagttctacggtactttcgacagagttgaaactagaaga1200tacatcgaatcttacaagatggactctttcaagatcttgaaggctgcttacagatcttct1260aacatcaacaacatcgacttgttgaagttctctgaacacgacttcaacttgtgtcaagct1320agacacaaggaagaattgcaacaaatcaagagatggttcgctgactgtaagttggaacaa1380gttggttcttctcaaaactacttgtacacttcttacttcccaatcgctgctatcttgttc1440gaaccagaatacggtgacgctagattggctttcgctaagtgtggtatcatcgctactact1500gttgacgacttcttcgacggtttcgcttgtaacgaagaattgcaaaacatcatcgaattg1560gttgaaagatgggacggttacccaactgttggtttcagatctgaaagagttagaatcttc1620ttcttggctttgtacaagatgatcgaagaaatcgctgctaaggctgaaactaagcaaggt1680agatgtgttaaggacttgttgatcaacttgtggatcgacttgttgaagtgtatgttggtt1740gaattggacttgtggaagatcaagtctactactccatctatcgaagaatacttgtctatc1800gcttgtgttactactggtgttaagtgtttgatcttgatctctttgcacttgttgggtcca1860aagttgtctaaggacgttactgaatcttctgaagtttctgctttgtggaactgtactgct1920gttgttgctagattgaacaacgacatccactcttacaagagagaacaagctgaatcttct1980actaacatggttgctatcttgatctctcaatctcaaagaactatctctgaagaagaagct2040atcagacaaatcaaggaaatgatggaatctaagagaagagaattgttgggtatggttttg2100caaaacaaggaatctcaattgccacaagtttgtaaggacttgttctggactactttcaag2160gctgcttactctatctacactcacggtgacgaatacagattcccacaagaattgaagaac2220cacatcaacgacgttatctacaagccattgaaccaatactctccataa2268<210>35<211>85<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>35aggtgcagttcgcgtgcaattataacgtcgtggcaactgttatcagtcgtaccgcgccat60tgagagtgcaccataccacagcttt85<210>36<211>85<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>36tcgtggtcaaggcgtgcaattctcaacacgagagtgattcttcggcgttgttgctgacca60gcggtatttcacaccgcatagggta85<210>37<211>85<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>37tggtcagcaacaacgccgaagaatcactctcgtgttgagaattgcacgccttgaccacga60cacgttaagggattttggtcatgag85<210>38<211>80<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>38aacgcgtaccctaagtacggcaccacagtgactatgcagtccgcactttgccaatgccaa60aaatgtgcgcggaaccccta80<210>39<211>84<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>39ttggcattggcaaagtgcggactgcatagtcactgtggtgccgtacttagggtacgcgtt60cctgaacgaagcatctgtgcttca84<210>40<211>85<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>40ccgagatgccaaaggataggtgctatgttgatgactacgacacagaactgcgggtgacat60aatgatagcattgaaggatgagact85<210>41<211>82<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>41atgtcacccgcagttctgtgtcgtagtcatcaacatagcacctatcctttggcatctcgg60tgagcaaaaggccagcaaaagg82<210>42<211>81<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>42ctcagatgtacggtgatcgccaccatgtgacggaagctatcctgacagtgtagcaagtgc60tgagcgtcagaccccgtagaa81<210>43<211>60<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>43attcctagtgacggccttgggaactcgatacacgatgttcagtagaccgctcacacatgg60<210>44<211>79<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>44aggtgcagttcgcgtgcaattataacgtcgtggcaactgttatcagtcgtaccgcgccat60tcgactacgtcgtaaggcc79<210>45<211>80<212>dna<213>人工序列(artificialsequence)<223>引物序列(primersequence)<400>45tcgtggtcaaggcgtgcaattctcaacacgagagtgattcttcggcgttgttgctgacca60tcgacggtcgaggagaactt80当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1