一种用于检验TCR-T细胞杀伤效果的靶细胞及其制备方法和应用与流程
本发明属于细胞工程技术领域,尤其涉及一种用于检验tcr-t细胞杀伤效果的靶细胞及其制备方法和应用。
背景技术
杀伤功能是机体免疫功能的一个重要方面,免疫系统中有多种具有杀伤功能的靶细胞,如自然杀伤细胞(nk)、细胞毒性t细胞(ctl)、具有adcc作用的单核细胞巨噬细胞等。tcr-t细胞为受体基因工程改造的t细胞,以修饰t细胞为基础的肿瘤过继免疫治疗方法,可以凭借其对肿瘤特异性抗原的高亲和性识别能力,在体内发挥较强的抗肿瘤免疫效应;是目前较为有效的恶性肿瘤的治疗方式之一,和其它免疫疗法类似,它的基本原理就是利用病人自身的免疫细胞来清除癌细胞。
常规的杀伤实验靶细胞,多采用人类永生化细胞或者肿瘤细胞,如k562细胞、293t细胞等,由于这些细胞是人类的永生化细胞,会有很多除了靶抗原蛋白以外的突变蛋白表达到细胞表面,或者被hla分子递呈到细胞表面,这就导致效应细胞对靶细胞进行杀伤时,除了针对靶抗原的特异性杀伤以外,还有对其他蛋白的非特异性杀伤,杀伤背景高,当效应细胞中特异性t细胞tcr-t细胞比例较低时,就会出现假阴性结果。另外,常规的靶细胞还存在对杀伤信号不敏感,自身增殖速度过快,无法进行长时间杀伤实验等问题。
技术实现要素:
有鉴于此,本发明的目的在于提供一种用于检验tcr-t细胞杀伤效果的靶细胞及其制备方法和应用,本发明提供的靶细胞杀伤背景低,对杀伤信号更敏感,能更准确的反应效应细胞的特异性杀伤能力,同时靶细胞自身增殖速度慢,能够进行较长时间的杀伤试验。
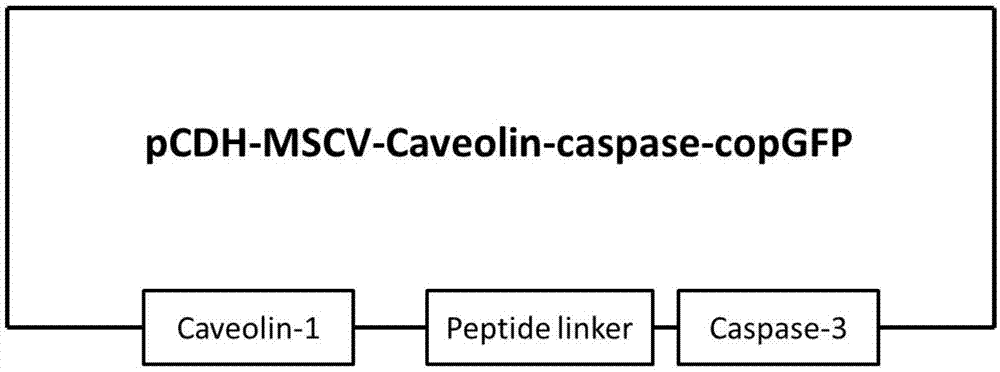
为了实现上述发明目的,本发明提供以下技术方案:一种用于检验tcr-t细胞杀伤效果的靶细胞tcc-3t3,所述靶细胞tcc-3t3为表达tcr-t细胞靶抗原基因、hla基因以及caveolin-1和caspase-3的融合蛋白基因的小鼠成纤维样细胞。
优选的,所述caveolin-1和caspase-3的融合蛋白基因包括顺次连接的caveolin-1基因、连接肽基因和caspase-3基因。
优选的,所述caveolin-1基因的核苷酸序列如seqidno.1所示。
优选的,所述caspase-3基因的核苷酸序列如seqidno.2所示。
优选的,所述连接肽的核苷酸序列如seqidno.3所示。
优选的,所述tcr-t细胞靶抗原基因为ny-eso-1基因或mage-a3基因。
优选的,所述tcr-t细胞靶抗原ny-eso-1基因的核苷酸序列如seqidno.4所示。
优选的,所述mage-a3基因的核苷酸序列如seqidno.5所示。
本发明还提供了所述靶细胞tcc-3t3的制备方法,包括以下步骤:
1)用连接肽将caveolin-1基因和caspase-3基因连接获得caveolin-1和caspase-3的融合蛋白基因;
2)用慢病毒转染法将所述caveolin-1和caspase-3的融合蛋白基因转入小鼠成纤维样细胞获得表达融合蛋白的cc-3t3细胞;
3)慢病毒转染法将tcr-t细胞靶抗原基因和tcr-t细胞tcr对应hla基因转入cc-3t3细胞,获得靶细胞tcc-3t3。
本发明还提供了所述靶细胞tcc-3t3在检验tcr-t细胞杀伤效果中的应用。
本发明的有益效果:本发明提供的用于检验tcr-t细胞杀伤效果的靶细胞tcc-3t3,是能够同时表达tcr-t细胞靶抗原基因以及caveolin-1和caspase-3的融合蛋白基因的小鼠成纤维样细胞,由于靶细胞为小鼠成纤维样细胞而非人类细胞,自身不表达人类hla分子和人类蛋白,可以避免常规靶细胞的非特异性杀伤;杀伤背景低,即使效应细胞中tcr-t细胞比例较低时,也不会出现假阴性结果。另外,所述靶细胞tcc-3t3增殖速度适中,可进行长时间杀伤实验(48~72h),并且对杀伤信号敏感,能更准确的反应效应细胞的特异性杀伤能力。
进一步的,本发明所述的tcc-3t3细胞转染hlaα链基因和β2微球蛋白基因,比以往使用的只转染表达hlaa链基因的传统靶细胞结合抗原表位肽的能力更强。
附图说明
图1为携带caveolin-1和caspase-3的融合蛋白基因的质粒结构图;
图2为携带hla基因的质粒结构图;
图3为携带tcr-t细胞靶抗原ny-eso-1基因的质粒结构图;
图4为携带tcr-t细胞靶抗原mage-a3基因的质粒结构图;
图5为构建的cc-3t3细胞系流式检测结果;
图6为tcc-3t3-ny-eso细胞作为靶细胞进行杀伤实验结果图;
图7为tcc-3t3-mage细胞作为靶细胞进行杀伤实验结果图。
具体实施方式
本发明提供了一种用于检验tcr-t细胞杀伤效果的靶细胞tcc-3t3,所述靶细胞tcc-3t3为表达tcr-t细胞靶抗原基因以及caveolin-1和caspase-3的融合蛋白基因的小鼠成纤维样细胞。
在本发明中,所述靶细胞tcc-3t3以小鼠成纤维样细胞为初始细胞;所述小鼠成纤维样细胞优选为swiss小鼠胚胎中获得,并保存于atcc细胞库中,所述小鼠成纤维样细胞的产品编号优选为
在本发明中,所述靶细胞tcc-3t3表达caveolin-1和caspase-3的融合蛋白基因。在本发明中,所述caveolin-1和caspase-3的融合蛋白基因包括顺次连接的caveolin-1基因、连接肽基因和caspase-3基因。本发明中,所述caveolin-1基因的核苷酸序列优选的如seqidno.1所示,所述caveolin-1基因编码的氨基酸序列优选如seqidno.6所示;所述caspase-3基因的核苷酸序列优选的如seqidno.2所示,所述caspase-3基因编码的氨基酸序列优选如seqidno.7;所述连接肽的核苷酸序列优选的如seqidno.3所示,所述连接肽的氨基酸序列优选如seqidno.8。本发明中,靶细胞tcc-3t3表达caveolin-1和caspase-3的融合蛋白。
在本发明中,所述靶细胞tcc-3t3表达tcr-t细胞靶抗原基因。本发明对所述tcr-t细胞靶抗原基因的具体种类没有限定,只要能够实现靶细胞表面表达tcr-t细胞特异性识别的位点即可。在本发明中,所述tcr-t细胞靶抗原基因为ny-eso-1基因和mage-a3基因。在本发明中,所述tcr-t细胞靶抗原ny-eso-1基因的核苷酸序列如seqidno.4所示,所述tcr-t细胞靶抗原ny-eso-1基因基因编码的氨基酸序列如seqidno.9所示;所述所述tcr-t细胞靶抗原mage-a3基因的核苷酸序列如seqidno.5所示,所述tcr-t细胞靶抗原mage-a3基因编码的氨基酸序列如seqidno.10所示
本发明中所述tcr-t细胞靶抗原ny-eso-1基因和mage-a3基因能够使靶细胞表面表达tcr-t细胞特异性识别的抗原使tcr-t细胞能够快速、特异的识别靶细胞。
当本发明中,所述靶抗原tcr-t基因为ny-eso-1基因时,所述靶细胞tcc-3t3优选的还表达hla基因。在本发明中,所述hla基因的作用为将靶抗原递呈到细胞表面。本发明对所述tcc-3t3细胞hla具体种类限定为能与tcr-t细胞上的tcr识别的靶抗原结合的hla分子。在本发明中所述hla基因优选的包括hlaα链基因,更有选的包括hlaα链基因和β2微球蛋白基因。在本发明中,所述hlaα链基因的核苷酸序列优选如seqidno.11所示,所述hlaa链基因编码的氨基酸序列优选如seqidno.12所示;所述β2微球蛋白基因的核苷酸序列如seqidno.13所示;所述β2微球蛋白的氨基酸序列如seqidno.14所示。在本发明中,所述hlaα链基因和β2微球蛋白基因优选的通过t2a连接,所述t2a的核苷酸序列如seqidno.15所示,所述t2a的氨基酸序列如seqidno.16所示。
本发明还提供了所述靶细胞tcc-3t3的制备方法,包括以下步骤:1)用连接肽将caveolin-1基因和caspase-3基因连接获得caveolin-1和caspase-3的融合蛋白基因;2)用慢病毒转染法将所述caveolin-1和caspase-3的融合蛋白基因转入小鼠成纤维样细胞获得表达融合蛋白的cc-3t3细胞;3)慢病毒转染法将tcr-t细胞靶抗原基因和hla基因转入cc-3t3细胞,获得靶细胞tcc-3t3。
在本发明中,用连接肽将caveolin-1基因和caspase-3基因连接获得caveolin-1和caspase-3的融合蛋白基因;在本发明具体实施过程中,所述融合基因优选的通过人工合成的方法,合成顺次连接的caveolin-1基因、连接肽基因和caspase-3基因为融合基因。
本发明在获得caveolin-1和caspase-3的融合蛋白基因后,利用慢病毒转染法将所述caveolin-1和caspase-3的融合蛋白基因转入小鼠成纤维样细胞获得表达融合蛋白的cc-3t3细胞。在本发明中,获得表达融合蛋白cc-3t3细胞的方法,包括以下步骤:s1)将携带融合基因的质粒、慢病毒包装质粒、dmem培养基混合后获得预混质粒;s2)将所述预混质粒与pei、dmem混合后获得待包装质粒;s3)将所述待包装质粒与细胞培养液混合培养,收集混合培养的上清液获得携带融合基因的慢病毒;s4)将所述携带融合基因的慢病毒转染小鼠成纤维样细胞获得表达融合蛋白的cc-3t3细胞。
在本发明中所述携带融合基因的质粒的结构图谱如附图1所示。本发明对所述慢病毒包装质粒没有特殊限定,采用本领域常规的慢病毒包装质粒,能够实现慢病毒包装即可。在本发明中,所述携带融合基因的质粒与慢病毒包装质粒的质量比优选为1∶(2.5~3.5),更优选为1∶3。在本发明中,步骤s1)中所述携带融合基因的质粒与dmem培养基的质量体积比(μg/μl)优选为1∶220~280,更优选为1∶250。
本发明在获得预混质粒后,将所述预混质粒与pei、dmem培养基混合后获得待包装质粒。在本发明中,所述混合的时间优选为15~25min,更优选为20min。在本发明中,所述预混质粒与pei、dmem培养基混合的质量体积比(μg/μl·μl)优选为4∶(8~12)∶(220~280),更优选为4∶10∶250。
本发明在获得获得待包装质粒后,将所述待包装质粒与细胞培养液混合培养,收集混合培养的上清液获得携带融合基因的慢病毒。在本发明中,所述细胞培养液优选为hek-293t细胞培养液。在本发明中,所述hek-293t细胞优选的来源于atcc细胞库;所述hek-293t细胞的产品货号为
本发明在获得携带融合基因的慢病毒,将所述携带融合基因的慢病毒转染小鼠成纤维样细胞获得表达融合蛋白的cc-3t3细胞。在本发明中,所述转染为将所述携带融合基因的慢病毒与小鼠成纤维样细胞混合后进行共培养;所述共培养的时间优选为2~3d,所述共培养的温度和环境与上述混合培养一致,在此不再赘述。本发明所述共培养过程中,携带融合基因的慢病毒转染小鼠成纤维样细胞,将融合基因整合到所述小鼠成纤维样细胞的基因组中获得表达融合蛋白的cc-3t3细胞。
本发明在获得所述表达融合蛋白的cc-3t3细胞后,优选的采用流式细胞仪筛选方法或抗生素筛选方法筛选cc-3t3细胞的稳转细胞系。在本发明中,所述流式细胞仪筛选方法具体为将细胞置于流式细胞仪中根据细胞gfp阳性表达率选择表达阳性率前10%的分群进行流式分选,筛选获得的cc-3t3细胞的纯度优选为99~100%之间。本发明中,所述抗生素筛选优选为采用含2~10μg/ml嘌呤霉素的新鲜dmem培养基培养表达融合蛋白的cc-3t3细胞,每2~3天替换新鲜配制的含2~10μg/ml嘌呤霉素的新鲜dmem培养基,连续培养25~35d获得cc-3t3细胞的稳转细胞系;筛选获得的cc-3t3细胞的纯度优选为99~100%之间。
本发明在获得表达融合蛋白的cc-3t3细胞后,采用慢病毒转染法将tcr-t细胞靶抗原基因转入cc-3t3细胞,获得靶细胞tcc-3t3。具体包括以下步骤:y1)将携带tcr-t细胞靶抗原基因的质粒、慢病毒包装质粒、dmem培养基混合后获得预混质粒;y2)将所述预混质粒与pei、dmem混合后获得待包装质粒;y3)将所述待包装质粒与细胞培养液混合培养,收集混合培养的上清液获得携带tcr-t细胞靶抗原基因的慢病毒;y4)将所述携带tcr-t细胞靶抗原基因的慢病毒转染cc-3t3细胞获得靶细胞tcc-3t3。
在本发明中,所述携带tcr-t细胞靶抗原基因的质粒结构如图3所示;本发明中所述慢病毒转染法将tcr-t细胞靶抗原基因转入cc-3t3细胞的方法步骤和参数与上述用慢病毒转染法将融合蛋白基因转入小鼠成纤维样细胞中的一致,在.此不再赘述。在本发明中,当所述tcr-t细胞靶抗原基因为ny-eso-1基因时,将所述tcr-t细胞靶抗原基因ny-eso-1转入cc-3t3细胞后,将所述hla基因,优选的包括hlaα链基因、β2微球蛋白基因以及t2a基因也通过慢病毒转染的方式转入cc-3t3细胞。在本发明中,携带所述hla基因的质粒结构如图2所示。
本发明在获得所述靶细胞tcc-3t3后,优选的采用流式细胞仪筛选方法或抗生素筛选方法筛选tcc-3t3细胞的稳转细胞系;具体方法步骤与上述cc-3t3细胞的稳转细胞系一致,在此不再赘述。
本发明还提供了所述靶细胞tcc-3t3在检验tcr-t细胞杀伤效果中的应用。在本发明中,所述应用采用本领域常规的细胞杀伤试验方法即可;在本发明具体实施过程中,所述靶细胞tcc-3t3的浓度优选为(6~10)×104个/ml,更优选为8×104个/ml;所述tcr-t细胞的浓度优选为(2.8~3.6)×106个/ml,更优选为3.2×106个/ml;本发明中,所述靶细胞tcc-3t3与tcr-t细胞按照效靶比优选为(38~42)∶1,更优选为40∶1。在本发明具体实施过程中,将靶细胞tcc-3t3于效应细胞按照上述比例混合孵育后,加入workingsolution反应25~35min,用stopsolution停止反应,采用酶标仪测定od490,计算杀伤率。在本发明中,所述孵育的温度优选为36~38℃,更优选为37℃,所述孵育的时间优选为2.5~3.5h,更优选为3h。
下面结合实施例对本发明提供的一种用于检验tcr-t细胞杀伤效果的靶细胞及其制备方法和应用进行详细的说明,但是不能把它们理解为对本发明保护范围的限定。
实施例1
1、构建慢病毒转染实验
(1)复苏培养hek-293t(
(2)转染当天,取两支15ml离心管,按表1加入质粒及试剂。
表1转染用试剂
(3)室温静止5min。
(4)将a管液体转入到b管内,混匀。
(5)室温静止20min。
(6)细胞换液:用移液管吸去培养液,加入10mldmem后轻摇,之后吸去。加入18mldmem至培养皿,轻摇混匀后置于37℃,5%co2培养。
(7)用5ml移液管枪头吸取a、b两管混合物后缓慢打出至培养液中,之后置于37℃,5%co2培养。记为转染0h。
(8)转染6h后,加入2.75mlfbs,轻摇混匀,继续培养。
(9)转染48h后,吸出上清至离心瓶中,放于4℃保存。补加27ml10%fbsdmem至培养皿继续培养。
(10)转染72h后,进行二次病毒上清液收集,吸出液体与48h收集的上清液混合,50ml离心管分装,于4℃,4000g,离心10min,去除细胞碎片。
(11)使用慢病毒peg-8000浓缩方法浓缩表达融合基因慢病毒。
使用相同方法获得表达tcr-t细胞靶抗原ny-eso-1基因的慢病毒、表达hlaα链基因-t2a基因-β2微球蛋白基因的慢病毒。
(12)将表达融合基因慢病毒转染小鼠成纤维样细胞
(13)将表达tcr-t细胞靶抗原ny-eso-1基因的慢病毒转染cc-3t3细胞后,再将表达hlaα链基因-t2a基因-β2微球蛋白基因的慢病毒转染上述细胞获得tcc-3t3细胞。
具体的慢病毒感染细胞方法包括以下步骤:
(1)将培养的待感染细胞消化计数后稀释至2×105个/ml加入10em培养皿,每皿8ml,摇匀后置于培养箱培养。
(2)次日,将包装好的病毒加至培养皿,标记好之后摇匀并置于培养箱培养,2-3d后可根据细胞数量扩大培养。
3.流式细胞仪方法筛选稳转细胞系
(1)收集10cm培养皿中的细胞,300×g离心5min,同时收集未感染细胞作为对照。
(2)每管加入1mlpbs洗细胞一次,300×g离心5min。
(3)每管加入500μlpbs重悬细胞至
(4)使用流式细胞仪方法筛选出稳转细胞系cc-3t3保证cc-3t3细胞纯度为99%。
流式细胞筛选仪的筛选结果如图5所示,其中作图为对照细胞的流式细胞筛选图,右图为cc-3t3细胞的流式细胞筛选图;从图5中可以看出经过流式分选后cc-3t3细胞的纯度都可以达到99%左右,说明靶细胞构建成功。
3、抗生素筛选稳转细胞系实验
(1)day0:24孔板内以5~8x104cells/孔的密度铺板,孵育过夜;
(2)制备筛选培养基:含2~10μg/ml嘌呤霉素的新鲜培养基dmem培养基;
(3)day1:筛选第一天,去除旧的培养基,加入一定量moi的病毒颗粒;(加入无血清培养基的总量必须充分覆盖住细胞。)
(4)病毒转导后约6~8h,再添加1ml完全培养基(10%fbs和1%双抗,如果已经使用双抗。)到细胞内,然后孵育过夜;
(5)病毒转导后48h,使用嘌呤霉素筛选培养基替换旧的完全培养基。孵育。
(6)约每2-3天替换新鲜配制的筛选培养基;
(7)每天检测细胞并观察活细胞生长比例。
(8)筛选持续进行30天,以确保筛选出的细胞纯度
(9)使用嘌呤霉素筛选出tcc-3t3细胞,纯度为99.5%。
4、杀伤实验
(1)收集靶细胞(cc-3t3、tcc-3t3),pbs洗一遍后加入10mlpbs用细胞刮轻轻刮下细胞,离心后加入10mlpbs洗一遍后使用2%fbs-1640重悬细胞,计数并将浓度调整为8×104/ml。
(2)收集效应细胞(tcr-t细胞),pbs洗两遍后,使用2%fbs-1640重悬细胞。
(3)效靶比设40∶1,效应细胞按最大比例稀释,即浓度为3.2×106/ml。
(4)96孔圆底板内依次加样,设6个复孔,加完后用封口膜封上,1000rpm离心1min,之后撕掉封口膜。
(5)镜下观察后,37℃、5%co2培养箱,孵育3h。
(6)在3x104靶细胞孔及在只有培养基的空白孔中各加10μllysisbuffer,37℃孵育30min。
(7)各孔加入100μlworkingsolution,并用铝箔包起来,室温反应30min。
(8)各孔加入50μlstopsolution,立即酶标仪测定od490。
(9)计算细胞杀伤率。
细胞杀伤率结果如图6所示,其中nih/3t3为不经过基因改造的细胞;cc-3t3为稳定转染caveolin-1和caspase-3融合基因的细胞tcc-3t3为稳定转染caveolin-1和caspase-3融合基因和hla-a0201链和β2m基因的细胞;tcc-3t3-ny-eso为稳定转染caveolin-1和caspase-3融合基因、hla-a0201链、β2m基因和ny-eso-1基因的细胞。ntc为未转染靶向ny-eso-1tcr的pbmc细胞;dmf4是转染靶向识别ny-eso-1抗原tcr的pbmc细胞,并且dmf4识别ny-eso-1抗原依赖hla-a0201递呈。
从图6中可以看出tcr-t细胞可以识别并杀伤拥有ny-eso-1抗原的靶细胞tcc-3t3,而对不拥有ny-eso-1抗原的cc-3t3细胞和nih/3t3细胞没有杀伤效应。并且ntc细胞对tcc-3t3、cc-3t3细胞和nih/3t3细胞都没有杀伤。说明tcc-3t3-ny-eso-1细胞可以作为靶细胞用于检测tcr-t细胞的杀伤能力。
实施例2
tcr-t细胞靶抗原基因替换为mage-a3基因,其余步骤与实施例1一致。
结果如图7所示,其中nih/3t3为不经过基因改造的细胞;cc-3t3为稳定转染caveolin-1和caspase-3融合基因的细胞;tcc-3t3为稳定转染caveolin-1和caspase-3融合基因和hla-a0201α链和β2m基因的细胞;tcc-3t3-mage-a3为稳定转染caveolin-1和caspase-3融合基因、hla-a0201α链、β2m基因和mage-a3基因的细胞。ntc为未转染靶向mage-a3tcr的pbmc细胞;dmf4是转染靶向识别mage-a3抗原tcr的pbmc细胞,并且dmf4识别mage-a3抗原依赖hla-a0201递呈。
tcr-t细胞可以识别并杀伤拥有mage-a3基因抗原的靶细胞tcc-3t3,而对不拥有mage-a3基因抗原的cc-3t3细胞和nih/3t3细胞没有杀伤效应。并且ntc细胞对tcc-3t3、cc-3t3细胞和nih/3t3细胞都没有杀伤。说明tcc-3t3-mage-a3细胞可以作为靶细胞用于检测tcr-t细胞的杀伤能力。
由上述实施例可知,本发明提供的用于检验tcr-t细胞杀伤效果的靶细胞tcc-3t3,可以避免常规靶细胞的非特异性杀伤,杀伤背景低,即使效应细胞中特异性t细胞(tcr-t细胞)比例较低时,也不会出现假阴性结果。另外,所述靶细胞tcc-3t3增殖速度适中,可进行长时间杀伤实验,并且对杀伤信号敏感,能更准确的反应效应细胞的特异性杀伤能力。
以上所述仅是本发明的优选实施方式,应当指出,对于本技术领域的普通技术人员来说,在不脱离本发明原理的前提下,还可以做出若干改进和润饰,这些改进和润饰也应视为本发明的保护范围。
序列表
<110>北京鼎成肽源生物技术有限公司
<120>一种用于检验tcr-t细胞杀伤效果的靶细胞及其制备方法和应用
<160>16
<170>siposequencelisting1.0
<210>1
<211>534
<212>dna
<213>人工序列(artificialsequence)
<400>1
atgtctgggggcaaatacgtagactccgagggacatctctacactgttcccatccgggaa60
cagggcaacatctacaagcccaacaacaaggccatggcagacgaggtgactgagaagcaa120
gtgtatgacgcgcacaccaaggagattgacctggtcaaccgcgaccccaagcatctcaac180
gacgacgtggtcaagattgactttgaagatgtgattgcagaaccagaagggacacacagt240
ttcgacggcatctggaaggccagcttcaccaccttcactgtgacaaaatattggttttac300
cgcttgttgtctacgatcttcggcatcccaatggcactcatctggggcatttactttgcc360
attctctccttcctgcacatctgggcggttgtaccgtgcatcaagagcttcctgattgag420
attcagtgcatcagccgcgtctactccatctacgtccataccttctgcgatccactcttt480
gaagctattggcaagatattcagcaacatccgcatcagcacgcagaaagagata534
<210>2
<211>834
<212>dna
<213>人工序列(artificialsequence)
<400>2
atggagaacaacaaaacctcagtggattcaaaatccattaataattttgaagtaaagacc60
atacatgggagcaagtcagtggactctgggatctatctggacagtagttacaaaatggat120
tatcctgaaatgggcatatgcataataattaataataagaacttccataagagcactgga180
atgtcatctcgctctggtacggatgtggacgcagccaacctcagagagacattcatgggc240
ctgaaataccaagtcaggaataaaaatgatcttactcgtgaagacattttggaattaatg300
gatagtgtttctaaggaagatcatagcaaaaggagcagctttgtgtgtgtgattctaagc360
catggtgatgaaggggtcatttatgggacaaatgggcctgttgaactgaaaaagttgact420
agcttcttcagaggcgactactgccggagtctgactggaaagccgaaactcttcatcatt480
caggcctgccggggtacggagctggactgtggcattgagacagacagtgggactgatgag540
gagatggcttgccagaagataccggtggaggctgacttcctgtatgcttactctacagca600
cctggttactattcctggagaaattcaaaggacgggtcgtggttcatccagtccctttgc660
agcatgctgaagctgtacgcgcacaagctagaatttatgcacattctcactcgcgttaac720
aggaaggtggcaacggaattcgagtccttctccctggactccactttccacgcaaagaaa780
cagatcccgtgtattgtgtccatgctcacgaaagaactgtacttttatcactag834
<210>3
<211>24
<212>dna
<213>人工序列(artificialsequence)
<400>3
ggcggcggcagcggcggcagcggc24
<210>4
<211>543
<212>dna
<213>人工序列(artificialsequence)
<400>4
atgcaggccgaaggccggggcacagggggttcgacgggcgatgctgatggcccaggaggc60
cctggcattcctgatggcccagggggcaatgctggcggcccaggagaggcgggtgccacg120
ggcggcagaggtccccggggcgcaggggcagcaagggcctcggggccgggaggaggcgcc180
ccgcggggtccgcatggcggcgcggcttcagggctgaatggatgctgcagatgcggggcc240
agggggccggagagccgcctgcttgagttctacctcgccatgcctttcgcgacacccatg300
gaagcagagctggcccgcaggagcctggcccaggatgccccaccgcttcccgtgccaggg360
gtgcttctgaaggagttcactgtgtccggcaacatactgactatccgactgactgctgca420
gaccaccgccaactgcagctctccatcagctcctgtctccagcagctttccctgttgatg480
tggatcacgcagtgctttctgcccgtgtttttggctcagcctccctcagggcagaggcgc540
taa543
<210>5
<211>942
<212>dna
<213>人工序列(artificialsequence)
<400>5
atgcctcttgagcagaggagtcagcactgcaagcctgaagaaggccttgaggcccgagga60
gaggccctgggcctggtgggtgcgcaggctcctgctactgaggagcaggaggctgcctcc120
tcctcttctactctagttgaagtcaccctgggggaggtgcctgctgccgagtcaccagat180
cctccccagagtcctcagggagcctccagcctccccactaccatgaactaccctctctgg240
agccaatcctatgaggactccagcaaccaagaagaggaggggccaagcaccttccctgac300
ctggagtccgagttccaagcagcactcagtaggaaggtggccgagttggttcattttctg360
ctcctcaagtatcgagccagggagccggtcacaaaggcagaaatgctggggagtgtcgtc420
ggaaattggcagtatttctttcctgtgatcttcagcaaagcttccagttccttgcagctg480
gtctttggcatcgagctgatggaagtggaccccatcggccacttgtacatctttgccacc540
tgcctgggcctctcctacgatggcctgctgggtgacaatcagatcatgcccaaggcaggc600
ctcctgataatcgtcctggccataatcgcaagagagggcgactgtgcccctgaggagaaa660
atctgggaggagctgagtgtgttagaggtgtttgaggggagggaagacagtatcttgggg720
gatcccaagaagctgctcacccaacatttcgtgcaggaaaactacctggagtaccggcag780
gtccccggcagtgatcctgcatgttatgaattcctgtggggtccaagggccctcgttgaa840
accagctatgtgaaagtcctgcaccatatggtaaagatcagtggaggacctcacatttcc900
tacccacccctgcatgagtgggttttgagagagggggaagag942
<210>6
<211>178
<212>prt
<213>人工序列(artificialsequence)
<400>6
metserglyglylystyrvalaspsergluglyhisleutyrthrval
151015
proilearggluglnglyasniletyrlysproasnasnlysalamet
202530
alaaspgluvalthrglulysglnvaltyraspalahisthrlysglu
354045
ileaspleuvalasnargaspprolyshisleuasnaspaspvalval
505560
lysileaspphegluaspvalilealagluprogluglythrhisser
65707580
pheaspglyiletrplysalaserphethrthrphethrvalthrlys
859095
tyrtrpphetyrargleuleuserthrilepheglyileprometala
100105110
leuiletrpglyiletyrphealaileleuserpheleuhisiletrp
115120125
alavalvalprocysilelysserpheleuilegluileglncysile
130135140
serargvaltyrseriletyrvalhisthrphecysaspproleuphe
145150155160
glualaileglylysilepheserasnileargileserthrglnlys
165170175
gluile
<210>7
<211>277
<212>prt
<213>人工序列(artificialsequence)
<400>7
metgluasnasnlysthrservalaspserlysserileasnasnphe
151015
gluvallysthrilehisglyserlysservalaspserglyiletyr
202530
leuaspsersertyrlysmetasptyrproglumetglyilecysile
354045
ileileasnasnlysasnphehislysserthrglymetserserarg
505560
serglythraspvalaspalaalaasnleuarggluthrphemetgly
65707580
leulystyrglnvalargasnlysasnaspleuthrarggluaspile
859095
leugluleumetaspservalserlysgluasphisserlysargser
100105110
serphevalcysvalileleuserhisglyaspgluglyvaliletyr
115120125
glythrasnglyprovalgluleulyslysleuthrserphephearg
130135140
glyasptyrcysargserleuthrglylysprolysleupheileile
145150155160
glnalacysargglythrgluleuaspcysglyilegluthraspser
165170175
glythraspgluglumetalacysglnlysileprovalglualaasp
180185190
pheleutyralatyrserthralaproglytyrtyrsertrpargasn
195200205
serlysaspglysertrppheileglnserleucyssermetleulys
210215220
leutyralahislysleugluphemethisileleuthrargvalasn
225230235240
arglysvalalathrgluphegluserpheserleuaspserthrphe
245250255
hisalalyslysglnileprocysilevalsermetleuthrlysglu
260265270
leutyrphetyrhis
275
<210>8
<211>8
<212>prt
<213>人工序列(artificialsequence)
<400>8
glyglyglyserglyglysergly
15
<210>9
<211>180
<212>prt
<213>人工序列(artificialsequence)
<400>9
metglnalagluglyargglythrglyglyserthrglyaspalaasp
151015
glyproglyglyproglyileproaspglyproglyglyasnalagly
202530
glyproglyglualaglyalathrglyglyargglyproargglyala
354045
glyalaalaargalaserglyproglyglyglyalaproargglypro
505560
hisglyglyalaalaserglyleuasnglycyscysargcysglyala
65707580
argglyprogluserargleuleugluphetyrleualametprophe
859095
alathrprometglualagluleualaargargserleualaglnasp
100105110
alaproproleuprovalproglyvalleuleulysgluphethrval
115120125
serglyasnileleuthrileargleuthralaalaasphisarggln
130135140
leuglnleuserilesersercysleuglnglnleuserleuleumet
145150155160
trpilethrglncyspheleuprovalpheleualaglnproproser
165170175
glyglnargarg
180
<210>10
<211>314
<212>prt
<213>人工序列(artificialsequence)
<400>10
metproleugluglnargserglnhiscyslysproglugluglyleu
151015
glualaargglyglualaleuglyleuvalglyalaglnalaproala
202530
thrglugluglnglualaalaserserserserthrleuvalgluval
354045
thrleuglygluvalproalaalagluserproaspproproglnser
505560
proglnglyalaserserleuprothrthrmetasntyrproleutrp
65707580
serglnsertyrgluaspserserasnglngluglugluglyproser
859095
thrpheproaspleugluserglupheglnalaalaleuserarglys
100105110
valalagluleuvalhispheleuleuleulystyrargalaargglu
115120125
provalthrlysalaglumetleuglyservalvalglyasntrpgln
130135140
tyrphepheprovalilepheserlysalaserserserleuglnleu
145150155160
valpheglyilegluleumetgluvalaspproileglyhisleutyr
165170175
ilephealathrcysleuglyleusertyraspglyleuleuglyasp
180185190
asnglnilemetprolysalaglyleuleuileilevalleualaile
195200205
ilealaarggluglyaspcysalaprogluglulysiletrpgluglu
210215220
leuservalleugluvalphegluglyarggluaspserileleugly
225230235240
aspprolyslysleuleuthrglnhisphevalglngluasntyrleu
245250255
glutyrargglnvalproglyseraspproalacystyrglupheleu
260265270
trpglyproargalaleuvalgluthrsertyrvallysvalleuhis
275280285
hismetvallysileserglyglyprohisilesertyrproproleu
290295300
hisglutrpvalleuarggluglygluglu
305310
<210>11
<211>1095
<212>dna
<213>人工序列(artificialsequence)
<400>11
atggccgtcatggcgccccgaaccctcgtcctgctactctcgggggctctggccctgacc60
cagacctgggcgggctctcactccatgaggtatttcttcacatccgtgtcccggcccggc120
cgcggggagccccgcttcatcgcagtgggctacgtggacgacacgcagttcgtgcggttc180
gacagcgacgccgcgagccagaggatggagccgcgggcgccgtggatagagcaggagggt240
ccggagtattgggacggggagacacggaaagtgaaggcccactcacagactcaccgagtg300
gacctggggaccctgcgcggctactacaaccagagcgaggccggttctcacaccgtccag360
aggatgtatggctgcgacgtggggtcggactggcgcttcctccgcgggtaccaccagtac420
gcctacgacggcaaggattacatcgccctgaaagaggacctgcgctcttggaccgcggcg480
gacatggcagctcagaccaccaagcacaagtgggaggcggcccatgtggcggagcagttg540
agagcctacctggagggcacgtgcgtggagtggctccgcagatacctggagaacgggaag600
gagacgctgcagcgcacggacgcccccaaaacgcatatgactcaccacgctgtctctgac660
catgaagccaccctgaggtgctgggccctgagcttctaccctgcggagatcacactgacc720
tggcagcgggatggggaggaccagacccaggacacggagctcgtggagaccaggcctgca780
ggggatggaaccttccagaagtgggcggctgtggtggtgccttctggacaggagcagaga840
tacacctgccatgtgcagcatgagggtttgcccaagcccctcaccctgagatgggagccg900
tcttcccagcccaccatccccatcgtgggcatcattgctggcctggttctctttggagct960
gtgatcactggagctgtggtcgctgctgtgatgtggaggaggaagagctcagatagaaaa1020
ggagggagctactctcaggctgcaagcagtgacagtgcccagggctctgatgtgtctctc1080
acagcttgtaaagtg1095
<210>12
<211>365
<212>prt
<213>人工序列(artificialsequence)
<400>12
metalavalmetalaproargthrleuvalleuleuleuserglyala
151015
leualaleuthrglnthrtrpalaglyserhissermetargtyrphe
202530
phethrservalserargproglyargglygluproargpheileala
354045
valglytyrvalaspaspthrglnphevalargpheaspseraspala
505560
alaserglnargmetgluproargalaprotrpilegluglnglugly
65707580
proglutyrtrpaspglygluthrarglysvallysalahissergln
859095
thrhisargvalaspleuglythrleuargglytyrtyrasnglnser
100105110
glualaglyserhisthrvalglnargmettyrglycysaspvalgly
115120125
serasptrpargpheleuargglytyrhisglntyralatyraspgly
130135140
lysasptyrilealaleulysgluaspleuargsertrpthralaala
145150155160
aspmetalaalaglnthrthrlyshislystrpglualaalahisval
165170175
alagluglnleuargalatyrleugluglythrcysvalglutrpleu
180185190
argargtyrleugluasnglylysgluthrleuglnargthraspala
195200205
prolysthrhismetthrhishisalavalserasphisglualathr
210215220
leuargcystrpalaleuserphetyrproalagluilethrleuthr
225230235240
trpglnargaspglygluaspglnthrglnaspthrgluleuvalglu
245250255
thrargproalaglyaspglythrpheglnlystrpalaalavalval
260265270
valproserglyglngluglnargtyrthrcyshisvalglnhisglu
275280285
glyleuprolysproleuthrleuargtrpgluproserserglnpro
290295300
thrileproilevalglyileilealaglyleuvalleupheglyala
305310315320
valilethrglyalavalvalalaalavalmettrpargarglysser
325330335
serasparglysglyglysertyrserglnalaalaserseraspser
340345350
alaglnglyseraspvalserleuthralacyslysval
355360365
<210>13
<211>360
<212>dna
<213>人工序列(artificialsequence)
<400>13
atgtctcgctccgtggccttagctgtgctcgcgctactctctctttctggcctggaggct60
atccagcgtactccaaagattcaggtttactcacgtcatccagcagagaatggaaagtca120
aatttcctgaattgctatgtgtctgggtttcatccatccgacattgaagttgacttactg180
aagaatggagagagaattgaaaaagtggagcattcagacttgtctttcagcaaggactgg240
tctttctatctcttgtactacactgaattcacccccactgaaaaagatgagtatgcctgc300
cgtgtgaaccatgtgactttgtcacagcccaagatagttaagtgggatcgagacatgtaa360
<210>14
<211>119
<212>prt
<213>人工序列(artificialsequence)
<400>14
metserargservalalaleualavalleualaleuleuserleuser
151015
glyleuglualaileglnargthrprolysileglnvaltyrserarg
202530
hisproalagluasnglylysserasnpheleuasncystyrvalser
354045
glyphehisproseraspilegluvalaspleuleulysasnglyglu
505560
argileglulysvalgluhisseraspleuserpheserlysasptrp
65707580
serphetyrleuleutyrtyrthrgluphethrprothrglulysasp
859095
glutyralacysargvalasnhisvalthrleuserglnprolysile
100105110
vallystrpaspargaspmet
115
<210>15
<211>54
<212>dna
<213>人工序列(artificialsequence)
<400>15
gagggcagaggaagtcttctaacatgcggtgacgtggaggagaatcccggccct54
<210>16
<211>18
<212>prt
<213>人工序列(artificialsequence)
<400>16
gluglyargglyserleuleuthrcysglyaspvalglugluasnpro
151015
glypro
- 还没有人留言评论。精彩留言会获得点赞!