用于检测基因融合突变的引物/探针组合、试剂盒及其使用方法与流程
本发明涉及基因检测领域,具体而言,涉及用于检测基因融合突变的引物/探针组合、试剂盒及其使用方法。
背景技术:
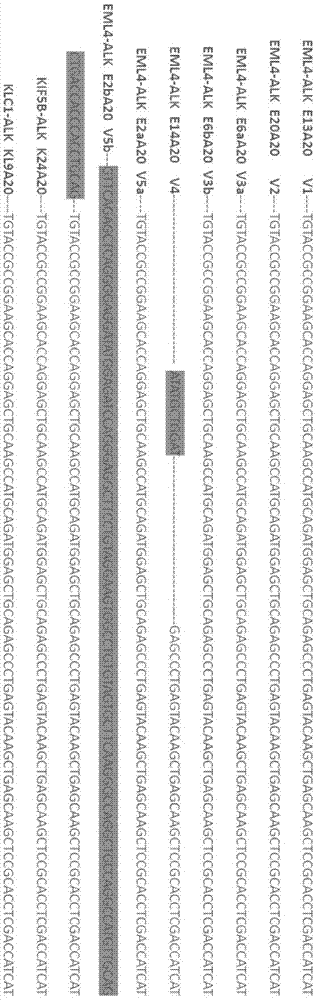
肺癌是临床常见恶性肿瘤,大量临床研究表明肺癌与基因改变有关。已有证据显示,酪氨酸激酶,例如间变性淋巴瘤激酶(anaplasticlymphomakinase,alk)、c-ros原癌基因1酪氨酸激酶(c-rosoncogene1receptortyrosine,ros1)以及ret原癌基因(retproto-oncogene,ret)发生基因融合现象,与肺癌的诊断、预后、治疗效果的判断、靶向药物的开发等相关。在临床和科研上有必要对前述酪氨酸激酶的基因融合进行准确、高效地检测。cfrna(circulatingcell-freerna)是指存在于血清、血浆、尿液、胸腹水、脑脊液等体液中的细胞外游离rna。随着cfrna分析技术的发展,cfrna逐渐成为新兴的液体活检分子标记。与传统分子诊断技术相比,基于cfrna的液体活检能够克服肿瘤的异质性,更全面地反映肿瘤特性,并具有非侵入性,便于多次取样,可对患者进行实时监控等优点。cfrna越来越多地应用于疾病诊断、治疗监测、预后评估和靶向药物的开发等方面。因此,以cfrna为检测对象,鉴定前述酪氨酸激酶是否发生基因融合突变具有重要意义。目前的基因融合突变检测方法主要包括染色体显带技术、荧光原位杂交、免疫组化、荧光定量pcr和高通量测序,但不适合基于cfrna的alk、ros1和ret融合基因的鉴定。具体而言,首先,染色体显带技术、荧光原位杂交和免疫组化只能用于检测染色体或组织细胞,不能检测cfrna。其次,荧光定量pcr需要标准曲线确定待测样本的拷贝数,无法检测到cfrna中的稀有突变;高通量测序的读长较短,导致测序结果的不准确性,且结果报告周期较长。再者,cfrna的样本丰度低,半衰期短,增加了检测的难度,同时,alk涉及9种不同的基因融合突变方式,ret涉及4种不同的基因融合突变方式,ros1涉及9种不同的基因融合突变方式,进一步增加检测的难度。因此,如何同时实现对alk基因、ret基因或ros1基因的多种不同融合方式的检测,进一步如何同时实现alk基因、ret基因和ros1基因融合突变的检测(共22种),存在极大的困难。有鉴于此,特提出本发明。技术实现要素:本发明的第一目的在于提供用于检测基因融合突变的引物/探针组合,所述引物/探针组合经过精心设计,其能够基于数字pcr技术,在同一检测孔中同时检测cfrna中的9种alk基因融合突变、4种ret融合基因突变或9种ros1融合基因突变,具有特异性好、灵敏度高、检测结果准确、无创等优点;优选地,所述引物/探针组合包括引物/探针组1、组2和组3,其最佳反应条件接近,能够在同一次数字pcr反应过程中,对前述22种基因融合突变同时进行检测;优选地,所述引物/探针组合还包括引物/探针组4,所述引物/探针组4能够对内参基因hprt1进行准确检测,提高定量检测结果的准确性。本发明的第二目的在于提供一种用于检测基因融合的试剂盒,所述试剂盒包括引物/探针组合。本发明的第三目的在于提供前述引物/探针组合或检测试剂盒的使用方法。为了实现本发明的上述目的,特采用以下技术方案:用于检测基因融合突变的引物/探针组合,所述引物/探针组合包括下组中的至少一种、两种或三种:用于检测alk基因融合突变的引物/探针组1,所述引物/探针组1包括引物1~8和探针1~2,其中,所述引物1~8的核苷酸序列分别如seqidno.19~20、30~31、23、32、25、27所示,所述探针1~2的核苷酸序列分别如seqidno.28~29所示;用于检测ret基因融合突变的引物/探针组2,所述引物/探针组2包括引物9~13和探针3,其中,所述引物9~13的核苷酸序列分别如seqidno.33~37所示,所述探针3的核苷酸序列如seqidno.38所示;用于检测ros1基因融合突变的引物/探针组3,所述引物/探针组3包括引物14~21和探针4~6,其中,所述引物14~21的核苷酸序列分别如seqidno.39~43、45~46、48所示,所述探针4~6的核苷酸序列分别如seqidno.44、47、49所示。由基因融合突变产生的cfrna存在丰度低、片段短、容易降解的特点,其检测难度大,而在同一检测孔中同时对多种融合突变进行检测,涉及多对引物和多重pcr,进一步增加检测的难度。目前,基于cfrna的融合基因检测方法报道不多,而基于cfrna同时对基因的多种不同融合方式进行检测的方法尚未见报道。为解决上述技术问题,本发明提供前述引物/探针组合,所述引物/探针组合可用于数字pcr,其采用pcr终点检测和泊松分布原理实现核酸样本的绝对定量,与传统pcr检测方法(如荧光定量pcr)相比,其检测的灵敏度高,能够实现在低丰度cfrna样品中检测基因融合突变。同时,本发明根据alk、ret和ros1基因融合突变的特点,对引物和探针进行精心设计和优化,最终尽管在同一检测孔中涉及多对引物和多重pcr,但仍然确保其具有高特异性和高扩增效率,实现在同一检测孔中同时检测多种融合突变;进一步地,组1、组2和组3在进行数字pcr反应时,最佳反应条件接近,可以在同一次数字pcr反应过程中,同时对alk、ret和ros1基因融合突变进行检测,具有极高的便利性。综上所述,本发明所述引物/探针组合具有灵敏度高、特异性强的优点,且检测系统的假阳性及假阴性率显著下降,同时可实现无创检测,其应用前景广阔。在一些具体的实施方式中,所述引物/探针组合还包括用于检测hprt1基因的引物/探针组4,所述引物/探针组4包括引物22~23和探针7,其中,所述引物22~23的核苷酸序列分别如seqidno.13~14所示,所述探针7的核苷酸序列如seqidno.15所示。在一些具体的实施方式中,所述探针1~2、所述探针3和/或所述探针4~6标记有荧光标记a;优选地,所述探针7标记有荧光标记b,所述荧光标记a与所述荧光标记b不同;更优选地,所述荧光标记a为fam,所述荧光标记b为vic。在一些具体的实施方式中,每组中各引物与各探针的摩尔比均为(5~15):(2~3);优选地,每组中各引物与各探针的摩尔比为4:1。本发明还涉及一种用于检测基因融合突变的试剂盒,所述试剂盒包括前述的引物/探针组合。在一些具体的实施方式中,所述试剂盒还包括其他辅助检测试剂。在一些具体的实施方式中,所述其他辅助检测试剂包括以下一种或者多种:taq酶、dntps、缓冲液、水、阴性质控品和阳性质控品;更优选地,所述taq酶、dntps和缓冲液混合为单试剂。在一些具体的实施方式中,所述缓冲液中包括tris·cl、k+和mg2+;优选地,所述k+由kcl提供,所述mg2+由mgso4和mgcl2提供。本发明还涉及前述引物/探针组合或试剂盒的使用方法,所述方法包括:(1)使用前述引物/探针组合或试剂盒对样本进行数字pcr反应;(2)根据所述数字pcr的扩增结果,判读样本是否发生基因融合突变。在一些具体的实施方式中,所述数字pcr的反应体系包括:500nm~1500nm引物1~8(优选为1000nm)和200~300nm探针1~2(优选为250nm);500nm~1500nm引物9~13(优选为1000nm)和200~300nm探针3(优选为250nm);500nm~1500nm引物14~21(优选为1000nm)和200~300nm探针4~6(优选为250nm);和/或,500nm~1500nm引物22~23(优选为1000nm)和200~300nm探针7(优选为250nm)。在一些具体的实施方式中,所述数字pcr的扩增程序包括:92~97℃预变性5~15分钟;聚合酶链式反应扩增阶段,92~97℃变性10~60s,50~60℃退火并延伸45~90s,所述扩增阶段循环30~50次。在一些具体的实施方式中,所述数字pcr的扩增程序包括:95℃预变性10分钟,聚合酶链式反应扩增阶段,95℃变性30s,55.5℃退火并延伸60s,所述扩增阶段循环40次。在一些具体的实施方式中,所述样本为血清、血浆、尿液、胸腹水或脑脊液。在一些具体的实施方式中,所述方法为非诊断目的。与现有技术相比,本发明所述引物/探针组合、试剂盒及其使用方法具有以下有益效果:(1)本发明基于数字pcr实现在低丰度的cfrna样品中检测alk等基因融合突变,具有灵敏度高、特异性好、可绝对定量以及非介入等优点,具有广泛的应用前景。(2)本发明对用于数字pcr反应的引物和探针进行优化,能够在单一检测孔中同时检测alk基因的9种融合突变形式、ret基因的4种融合突变形式或ros1基因的9种融合突变形式。(3)本发明组1、组2和组3所述引物/探针在进行数字pcr反应时,最佳反应条件接近,可以在同一次数字pcr反应过程中,同时对alk、ret和ros1基因融合突变进行检测,具有极高的便利性。附图说明为了更清楚地说明本发明具体实施方式或现有技术中的技术方案,下面将对具体实施方式或现有技术描述中所需要使用的附图作简单地介绍,显而易见地,下面描述中的附图是本发明的一些实施方式,对于本领域普通技术人员来讲,在不付出创造性劳动的前提下,还可以根据这些附图获得其他的附图。图1:alk基因融合突变的9种形式;图2:2种不同候选引物/探针组的fam通道和vic通道扩增结果一维图,c03和c07的pcr模板为阳性质粒混合物,c04和c08的pcr模板为nci-h2228cdna,c03和c04使用的候选引物/探针组为alk-r-1,c07和c08使用的候选引物/探针组为alk-r-2;图3~图4:alk-r-1候选组(图3)或alk-r-2获选组(图4)以nci-h2228cdna为模板的扩增结果二维图,其中左下区为阴性微滴,左上区为fam通道阳性微滴,右下区为vic通道阳性微滴,右上区为fam通道和vic通道均为阳性的微滴;图5:eml4-alkv1基因融合类型的扩增结果,模板:eml4-alkv1阳性质粒,上游引物:eml4-alkv1-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图6:eml4-alkv2基因融合类型的扩增结果,模板:eml4-alkv2阳性质粒,上游引物:eml4-alkv2-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图7:eml4-alkv3a基因融合类型的扩增结果,模板:eml4-alkv3a阳性质粒,上游引物:eml4-alkv3a-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图8:eml4-alkv3b基因融合类型的扩增结果,模板:eml4-alkv3b阳性质粒,上游引物:eml4-alkv3a-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图9:eml4-alkv4基因融合类型的扩增结果,模板:eml4-alkv4阳性质粒,上游引物:eml4-alkv4-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图10:eml4-alkv5a基因融合类型的扩增结果,模板:eml4-alkv5a阳性质粒,上游引物:eml4-alkv5a-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图11:eml4-alkv5b基因融合类型的扩增结果,模板:eml4-alkv5b阳性质粒,上游引物:eml4-alkv5a-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图12:eml4-alkk24a20基因融合类型的扩增结果,模板:eml4-alkk24a20阳性质粒,上游引物:eml4-alkk24a20-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图13:eml4-alkkl9a20基因融合类型的扩增结果,模板:eml4-alkkl9a20阳性质粒,上游引物:eml4-alkkl9a20-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图14:eml4-alkv3a基因融合类型的扩增结果,模板:eml4-alkv3a阳性质粒,上游引物:new-alkv3b-2-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图15:eml4-alkv3b基因融合类型的扩增结果,模板:eml4-alkv3b阳性质粒,上游引物:new-alkv3b-2-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图16:eml4-alkv4基因融合类型的扩增结果,模板:eml4-alkv4阳性质粒,上游引物:new-alkv4-1-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图17:eml4-alkk24a20基因融合类型的扩增结果,模板:eml4-alkk24a20阳性质粒,上游引物:new-kif5b-alkk24a20-2-f,下游引物:alk-r-2,其中上半区为阳性微滴,下半区为阴性微滴;图18:alk多重检测体系在无alk融合突变的细胞系nci-h1975gdna的扩增结果,其中左半区为阴性微滴,右半区为内参阳性微滴;图19:alk多重检测体系在无alk融合突变的细胞系hcc-78cdna的扩增结果,其中左半区为阴性微滴,右半区为内参阳性微滴;图20:alk多重检测体系在alk融合突变的细胞系nci-h2228cdna的扩增结果,其中其中左下区为阴性微滴,左上区为fam通道阳性微滴,右下区为vic通道阳性微滴,右上区为fam通道和vic通道均为阳性的微滴;图21:以nci-h2228cdna为模板,进行ddpcr扩增体系退火温度优化,a01和a02、b01和b02、c01和c02、d01和d02、e01和e02、f01和f02的退火温度分别是56℃、55.5℃、54.8℃、53.7℃、52.3℃、51.1℃,其中上半区为阳性微滴,下半区为阴性微滴;图22~27:以nci-h2228cdna为模板,进行ddpcr扩增体系退火温度优化,图22~图27分别是退火温度为56℃、55.5℃、54.8℃、53.7℃、52.3℃、51.1℃时的扩增结果,其中,上半区为阳性微滴,下半区为阴性微滴;图28:以阳性质粒eml4-alkv1为模板,进行ddpcr扩增体系退火温度优化,其中,c01、d01、e01、f01、g01、h01的退火温度分别为60.6℃、59.3℃、57.7℃、56.4℃、55.5℃、55℃,其中,上半区为阳性微滴,下半区为阴性微滴;图29:ret基因融合突变的4种形式;图30~33:以阳性质粒为模板,优化前的上、下游引物和探针对ret融合基因的扩增结果(图30:kif5b-retk15r12;图31:kif5b-retk16r12;图32:ccd6-retc1r12;图33:ncoa4-retn6r12),其中,上半区为阳性微滴,下半区为阴性微滴;图34~37:以阳性质粒为模板,优化后的上、下游引物和探针对ret融合基因的扩增结果(图34:kif5b-retk15r12;图35:kif5b-retk16r12;图36:ccd6-retc1r12;图37:ncoa4-retn6r12),其中,上半区为阳性微滴,下半区为阴性微滴;图38:ros1基因融合突变的9种形式;图39~47:引物/探针组合优化前的扩增结果(图39:cd74-ros1c6r32;图40:slc34a2-ros1s4r32;图41:sdc4-ros1s2r32;图42:sdc4-ros1s4r32;图43:cd74-ros1c6r34;图44:ezr-ros1e10r34;图45:slc34a2-ros1s4r34;图46:sdc4-ros1s4r34;图47:tpm3-ros1t7r35),其中,上半区为阳性微滴,下半区为阴性微滴;图48~52:引物/探针组合优化后的扩增结果(图48:cd74-ros1c6r32;图49:slc34a2-ros1s4r32;图50:cd74-ros1c6r34;图51:slc34a2-ros1s4r34;图52:tpm3-ros1t7r35),其中,上半区为阳性微滴,下半区为阴性微滴;图53~58:分别为样品编号1~6的检测结果,其中左下区为阴性微滴,左上区为fam通道阳性微滴,右下区为vic通道阳性微滴,右上区为fam通道和vic通道均为阳性的微滴。具体实施方式下面将结合实施例对本发明的实施方案进行详细描述,但是本领域技术人员将会理解,下列实施例仅用于说明本发明,而不应视为限制本发明的范围。实施例中未注明具体条件者,按照常规条件或制造商建议的条件进行。所用试剂或仪器未注明生产厂商者,均为可以通过市购买获得的常规产品。下述实施例使用的仪器包括:dg8cartridgesforqx100/qx200dropletgenerator(biorad)、cartridgeholderdg8(biorad)、dropletgeneratordg8gasket(biorad)、bioradqx200dropletgenerator(biorad)、ddpcr96-wellplates(biorad)、pierceablefoilheatseal(biorad)、bioradpx1pcrplatesealer(biorad)、bioradt100pcr(biorad)、bio-radqx200dropletreader、qubit4.0、高速离心机、漩涡震荡仪、冰箱、生物安全柜。实施例1中内参基因涉及的探针标记fam/vic,其中商品化内参hprt1-assay的标记为fam,其他自己设计的探针序列标记均为vic;实施例2~5中,alk、ret、ros1基因涉及的探针均标记fam,内参基因涉及的探针均标记vic。实施例1内参基因及其引物和探针的选择本实施例选择6个内参基因eftud2、alk-ref、ret-ref、ros1-ref、hprt1、actb,并分别设计内参基因的引物和探针序列,以eml4-alk融合的阳性细胞系nci-h2228cdna作为模板,通过ddpcr对比试验从中筛选出最佳的引物对和探针序列作为该检测体系的内参,其中ddpcr反应体系中,引物的终浓度为1000nm,探针的终浓度为250nm,退火温度为60℃。其中,内参基因、引物对及探针序列如表1所示,不同内参基因对nci-h2228cdna的定量结果如表2所示。根据表2所示结果可知,内参hprt1基因的定量结果与经过验证的商品化内参hprt1-assay较为一致,因此,选择hprt1作为该体系的内参。表1内参基因及对应引物和探针序列表2不同内参基因对nci-h2228cdna的定量结果实施例2alk引物/探针的设计、优化以及退火温度的优化(1)引物/探针的设计设计目的:设计在一个单独的检测孔中能同时检测alk基因的9种融合突变的上、下游引物和探针。设计原则:alk的9种融合突变形式具有相同的下游基因断裂位点a20(参见图1),因此在相同的alk下游断裂位点设计共同的下游引物。除融合类型eml4-alkv4,其他突变类型使用相同的探针序列,eml4-alkv4的探针设计在融合上游的eml4e14区域;针对每种融合突变设计单独的上游引物,根据cfrna断裂的特点,保证每种融合类型的扩增子在150bp以内,以达到最佳的扩增效率。根据上述原则设计两组候选的引物/探针组——alk-r-1和alk-r-2(具体信息如表3所示,两组候选引物/探针组的差别仅在于通用下游引物的序列不同)。每组候选引物/探针组包括:一条通用的下游引物(针对9种融合突变);7条上游引物,其中,eml4-alkv3a和v3b共用同一上游引物,eml4-alkv5a和v5b共用同一上游引物,其余5条上游引物分别针对剩余5种alk融合突变类型;两条阳性探针,其中一条阳性探针针对eml4-alkv4基因融合突变,设计在融合上游的eml4e14区域,另一条阳性探针针对其余8种alk基因融合突变。表3alk基因9种融合突变形式的引物和探针序列(2)下游引物的优化以9种alk基因融合突变的阳性质粒混合液或nci-h2228cdna为模板,分别使用前述候选引物/探针组进行ddpcr反应,其中,pcr反应体系中各引物的终浓度为1000nm,探针的终浓度为250nm,退火温度为60℃。pcr扩增结果显示,与候选组alk-r-1相比,候选组alk-r-2的阴性微滴较为集中,阳性微滴和阴性微滴间的扩增延迟微滴较少,区分度好(参见图2~4)。基于上述检测结果,选择alk-r-2作为检测融合突变的下游通用引物。(3)上游引物的优化使用alk基因9种融合形式已经确定的下游通用引物alk-r-2,针对每种融合突变的上游引物进行优化。分别以9种alk基因融合突变的阳性质粒为模板,使用ddpcr对9种融合突变的上下游引物和探针分别进行扩增测试,其中,pcr反应体系中各引物的终浓度为1000nm,探针浓度为250nm,退火温度为60℃。具体扩增结果参见图(图5~13)。其中eml4-alkv3a、eml4-alkv3b、eml4-alkv4、eml4-alkk24a20扩增结果的阳性微滴较分散,对其上游引物进行优化,经优化调整最终确定的引物/探针组合,具体信息参见表4。优化后的上游引物new-alkv3b-2-f、new-alkv4-1-f、new-kif5b-alkk24a20-2-f的扩增结果显示阳性微滴较集中(见图14~17)。表4优化后alk基因9种融合突变形式的引物和探针序列(3)alk多重pcr反应测试由于涉及的引物众多,为确保彼此之间无互相干扰,本实施例将9种alk融合突变的引物和探针及内参hprt1基因的引物和探针置于同一个检测孔,分别以hcc-78cdna、nci-h1975dna和nci-h2228cdna为模板进行ddpcr扩增,以检测各引物的特异性,其中,ddpcr反应体系中各引物的浓度为1000nm,探针的浓度为250nm,退火温度为60℃。结果(参见图20)显示:以nci-h2228cdna作为模板进行扩增,阳性和内参基因微滴区分良好,形成2个区域的微滴,表明2个探针均有效的扩增,经计算eml4-alkv3的突变频率是28%,与单孔检测的值接近,表明alk多重pcr反应各引物和探针未产生明显的干扰,结果准确可靠。此外,该alk多重检测体系在无alk融合突变细胞系基因组dna(nci-h1975dna)和cdna(hcc-78cdna)中无fam通道的阳性扩增,证明alk检测体系具有良好的特异性,无非特异性扩增(参见图18~19)。(4)alk多重ddpcr反应退火温度优化使用优化后的eml4-alkv3上下游引物和探针进行ddpcr退火温度初测试,以nci-h2228cdna为模板,结果显示退火温度为55.5℃时,阳性微滴和阴性微滴之间的区分度最好,非特异性最少,阳性微滴和阴性微滴间的扩增延迟较少(图21~27)。随后将优化后的9种alk融合突变上下游引物和探针与hprt1内参的上下游引物和探针混在一起,以阳性质粒eml4-alkv1为模板,进行ddpcr退火温度测试,其中,pcr反应体系中各引物的终浓度为1000nm,探针的终浓度为250nm。结果显示退火温度为55.5℃时,非特异性扩增较少,且阳性微滴和阴性微滴之间的扩增延迟微滴较少(图28)。因此,本方案将alk多重ddpcr反应的退火温度设置为55.5℃。(5)alk引物/探针使用浓度的优化将已优化好的9种alk融合突变的上下游引物和探针及内参基因hprt1的上下游引物和探针配置成反应混合液,使用阳性质粒eml4-alkv1作为模板进行ddpcr测试,其中,退火温度为55.5℃。结果发现,ddpcr反应体系中引物终浓度为1000nm、探针终浓度为250nm时,阳性微滴和阴性微滴的区分度好,阳性的回收率最高(表5为引物终浓度为1000nm时,不同探针浓度下,eml4-alkv1基因融合突变和内参基因hrpt1的拷贝数检测结果)。因此,选择引物终浓度为1000nm和探针终浓度为250nm进行检测实验。表5alk引物/探针使用浓度优化探针终浓度250nm350nm450nmeml4-alkv119.88.711.5hrpt117471.1110实施例3ret基因融合突变的检测引物/探针的设计和优化本实施例的目的在于将ret基因的4种融合突变置于同一个检测孔进行检测。本实施例根据ret基因融合的结构特征(参见图29),综合考虑特异性和扩增效率,设计针对4种ret融合突变形式的引物/探针,并基于实施例2的相似优化试验,通过ddpcr对设计的引物和探针进行优化,最终获得最优的引物/探针组合,所述引物/探针组合包括分别针对4种ret融合突变的上游引物和一条通用的下游引物以及一条通用的探针,其具体序列信息参见表6,引物/探针组合优化前的扩增结果如图30~33所示,优化后的扩增结果如图34~37所示。表6优化后ret基因4种融合突变形式的引物和探针序列实施例4ros1基因融合突变的检测引物/探针的设计和优化本实施例的目的在于在将9种ros1融合突变置于单一的检测孔进行合并检测。ros1融合突变根据融合断裂点位置的不同可以分为3类,分别为融合断裂点位于r32、r34和r35(参见图38)。由于断裂位置r34和r35较为接近,两者相差长度为85bp,为避免两类融合突变分别设计下游引物引起的多重pcr反应,本实施例针对r34和r35两类融合突变设计共同的下游引物,该下游引物位于r35区域且接近融合断裂点的位置。根据下游引物的位置,将r35探针设计在上游融合基因tpm3区域。随后分别在r34上游融合基因处设计上游引物,并在中间区域设计共同的探针,即r34的探针设计在r34区域。由于r34与r32的部分融合形式有相同的上游融合基因和融合断裂点,因此为避免多个上游引物引起的多重pcr反应,这些融合形式使用共同的上游引物,即cd74c6、slc34a2-ros1s4、sdc4-ros1s4使用相同的上游引物,并在中间区域设计共同的下游引物和探针,即r32的下游引物和探针设计在r32区域。本实施例对r32融合类型中的sdc4-ros1s2单独设计了上游引物,根据r32共4种融合形式的上游引物位置,设计共同的下游引物。r32探针设计在r32的共同位置,即r32融合断裂点的位置。本实施例根据上述原则设计针对9种ros1融合突变的引物/探针,并基于实施例2的相似优化试验,通过ddpcr对设计的引物和探针进行优化,最终获得最优的引物/探针组合,其具体序列信息参见表7,引物/探针组合优化前的扩增结果如图39~47所示,优化后的扩增结果如图48~52所示。表7优化后ros1基因9种融合突变形式的引物和探针序列实施例5本实施例采用ddpcr方法检测6份阳性临床血浆样本中alk、ret和ros1基因的融合突变情况,其中,每份血浆样本中alk、ret和ros1基因的融合突变分别在不同的检测孔中进行,但其ddpcr反应程序相同。具体检测方法和检测结果如下所示:(1)ddpcr反应体系的配制:1、合成实施例1~4优化所得的引物和探针。2、取引物干粉,加水将引物稀释至50μm备用;将所示探针稀释至50μm备用。3、完成引物、探针的稀释后,按表8分别配制alk、ret、ros1反应检测液,按表9分别配制alk、ret、ros1检测ddpcrmix。4、ddpcrmix配制完成后,涡旋振荡混匀,瞬时离心,分装至1.5mlep管或8连管中,每管分装14.3μlalk检测ddpcrmix、13μlret检测ddpcrmix、14.4μlros1检测ddpcrmix。向对应的1.5mlep管或8连管中加入已反转录好的样本(体积≤5.6μl)或阴性/阳性/空白对照品,用ultrapuretmdnase/rnase-freedistilledwater补足反应体积至20μl,涡旋振荡混匀,瞬时离心。表8反应检测液alk反应检测液:检测液组分1个反应加入量(μl)n个反应加入量(μl)alk上游引物mix7×0.47×0.4×nalk下游引物mix0.40.4×nalkprobe2×0.12×0.1×nhprt1上游引物mix0.40.4×nhprt1下游引物mix0.40.4×nhprt1probe0.10.1×ntotal4.34.3×nret反应检测液:检测液组分1个反应加入量(μl)n个反应加入量(μl)ret上游引物mix4×0.44×0.4×nret下游引物mix0.40.4×nretprobe0.10.1×nhprt1上游引物mix0.40.4×nhprt1下游引物mix0.40.4×nhprt1probe0.10.1×ntotal33×nros1反应检测液:检测液组分1个反应加入量(μl)n个反应加入量(μl)ros1上游引物mix6×0.46×0.4×nros1下游引物mix2×0.42×0.4×nros1probe3×0.13×0.1×nhprt1上游引物mix0.40.4×nhprt1下游引物mix0.40.4×nhprt1probe0.10.1×ntotal4.44.4×n表9ddpcrmixalk检测ddpcrmix:试剂组分1人份pcrmix(μl)n人份pcrmix(μl)ddpcrtmsupermixforprobes(nodutp)1010×n×1.05反应检测液4.34.3×n×1.05total14.314.3×n×1.05ret检测ddpcrmix:ros1检测ddpcrmix:试剂组分1人份pcrmix(μl)n人份pcrmix(μl)ddpcrtmsupermixforprobes(nodutp)1010×n×1.05反应检测液4.44.4×n×1.05total14.414.4×n×1.05(2)微滴的生成:1、将一个新的dg8cartridge放置于holder中,将配制好的20μlpcr体系加入到dg8cartridge中间一排的8个样品孔内,不足8个样品时用稀释一倍的20μl1×buffercontrol补足,不允许有空着的孔。建议使用rainin的8通道20μl排枪和20μl枪头,加样时枪头接近孔一侧底部,与侧壁呈大约15°角,缓慢打出液体,打出一部分后慢慢提升枪头位置再打出余下液体,不要将枪按至超过第一档位置以免引入气泡。在dg8cartridge最底下一排8个oil孔中各加入70μl微滴生成油(dgoil),同样不能有空着的孔。加样本及微滴生成油时都要避免产生气泡。2、盖上胶垫(gasket),注意两边的小孔都要钩牢。3、将dg8cartridge及底座轻轻地平稳放置于微滴生成仪中,开始生成微滴。运行指示灯常绿时,微滴生成结束,一般约2分钟完成。仪器所处环境温度必须处于18-30℃之间。4、微滴生成于cartridge最上面一排孔内,建议使用rainin的8通道p-50排枪和200μl枪头小心缓慢吸取,调整吸取体积为40μl,将holder放平,枪头以与孔壁呈30~45°角放入,轻触孔底,约5秒吸取40μl,再同样缓慢地打入96孔板相应位置孔内(约5秒),枪头贴近孔壁接近孔底,注意封上盖以防油挥发,每次弃去已使用过的cartridge和胶垫。(3)封膜:打开px1热封仪,设置运行程序为180℃,5s,仪器自动开始预热。转移微滴进入96孔板内完成后,将膜有红线标记的一面(反光亮面)朝上置于96孔板上并固定好,用预热好的px1热封仪对其进行封膜,需确认各样品孔是否密封好,若未密封好则需颠倒方向二次封膜。封好膜后在30分钟内进行pcr反应,或者放于4℃冰箱4小时之内进行pcr反应,可在任意一台96孔pcr仪上完成,注意升降温速度≤2.5℃/s。(4)pcr反应:按照表10所述条件设置pcr反应程序并进行pcr反应。表10ddpcr反应程序(5)读取结果及结果的判读:1、将完成pcr的96孔板放入plateholder中组装好,注意板斜角方向与holder对应。组装好之后轻轻地平稳放入微滴读取仪中,打开quantasoft软件,设置96孔板中的样品信息,点击run,仪器开始读取结果。2、微滴读取结束以后,按以下步骤进行检测结果的分析、判定:(1)判读空白对照:空白对照在fam和vic通道没有微滴或微滴数小于3个。(2)判读阴性对照:阴性对照在vic通道有荧光,且微滴数≥3个。(3)判读阳性对照:阳性对照在fam通道有荧光信号,且微滴数≥3个。(4)判读样品fam通道阳性微滴与空白微滴区分是否明显,根据阳性参考品设定区分区域并统计阳性微滴数量。(5)判读样品vic通道阴性微滴与空白微滴区分是否明显,根据阴性参考品设定区分区域并统计阴性微滴数量。(6)判读样本fam和vic双通道均有荧光信号的微滴数量,根据阳性和阴性参考品设定区分区域统计微滴数量并按照泊松方程计算拷贝数。(7)不符合上述1项要求,建议重做本次实验,不符合第2或3项要求,建议重新从样本中提取rna,再次检测。具体扩增结果如图53~58所示,突变率的绝对定量结果如表11所示。根据表11所述绝对定量结果可知:(1)本实施例能够在cfrna样品中实现对alk、ret和ros1基因融合突变的同时检测;(2)本实施例能够检出突变率低至1.38%,拷贝数低至2.51的样本,检测的灵敏度高。表11基因融合突变的绝对定量结果最后应说明的是:以上各实施例仅用以说明本发明的技术方案,而非对其限制;尽管参照前述各实施例对本发明进行了详细的说明,但本领域的普通技术人员应当理解:其依然可以对前述各实施例所记载的技术方案进行修改,或者对其中部分或者全部技术特征进行等同替换;而这些修改或者替换,并不使相应技术方案的本质脱离本发明各实施例技术方案的范围。sequencelisting<110>江苏先声医学诊断有限公司南京先声医学检验有限公司北京先声医学检验实验室有限公司<120>用于检测基因融合突变的引物/探针组合、试剂盒及其使用方法<160>53<170>patentinversion3.3<210>1<211>21<212>dna<213>人工序列<400>1tgccagacaccaaaggaaaat21<210>2<211>21<212>dna<213>人工序列<400>2tgagaggacacacgcaaaacc21<210>3<211>16<212>dna<213>人工序列<400>3ctccaggtaggacatc16<210>4<211>22<212>dna<213>人工序列<400>4gactgccaatatgaaggaggta22<210>5<211>22<212>dna<213>人工序列<400>5gcttttcagaatggtatcctcg22<210>6<211>21<212>dna<213>人工序列<400>6ttacggctaccagcaacaggg21<210>7<211>22<212>dna<213>人工序列<400>7ctgactccctgatttatgacga22<210>8<211>22<212>dna<213>人工序列<400>8tctgacatgccatagagtttgt22<210>9<211>22<212>dna<213>人工序列<400>9gctggtggactgtaataatgcc22<210>10<211>22<212>dna<213>人工序列<400>10tccagaaagtttgatggatgga22<210>11<211>22<212>dna<213>人工序列<400>11aaggttggaatgagctggataa22<210>12<211>22<212>dna<213>人工序列<400>12aaggttggaatgagctggataa22<210>13<211>19<212>dna<213>人工序列<400>13aagcttgctggtgaaaagg19<210>14<211>21<212>dna<213>人工序列<400>14aaacatgattcaaatccctga21<210>15<211>30<212>dna<213>人工序列<400>15tgttggatttgaaattccagacaagtttgt30<210>16<211>20<212>dna<213>人工序列<400>16ggtcatcaccattggcaatg20<210>17<211>23<212>dna<213>人工序列<400>17cgtcacacttcatgatggagttg23<210>18<211>29<212>dna<213>人工序列<400>18atggagtcctgtggcatccacgaaactac29<210>19<211>18<212>dna<213>人工序列<400>19gcccacacctgggaaagg18<210>20<211>29<212>dna<213>人工序列<400>20tctaactcgggagactatgaaatattgta29<210>21<211>26<212>dna<213>人工序列<400>21aaactgcagacaagcataaagatgtc26<210>22<211>21<212>dna<213>人工序列<400>22ggatgttattaactggaggag21<210>23<211>18<212>dna<213>人工序列<400>23catgtggcctcagtgaaa18<210>24<211>21<212>dna<213>人工序列<400>24aagaagtagatcgcataaagg21<210>25<211>20<212>dna<213>人工序列<400>25atgaaagggagtttggttct20<210>26<211>23<212>dna<213>人工序列<400>26ggagcttgctcagcttgtactca23<210>27<211>18<212>dna<213>人工序列<400>27tgctcagcttgtactcag18<210>28<211>20<212>dna<213>人工序列<400>28ccgccggaagcaccaggagc20<210>29<211>18<212>dna<213>人工序列<400>29atcatgatctgaatcctg18<210>30<211>20<212>dna<213>人工序列<400>30aagatgtcatcatcaaccaa20<210>31<211>18<212>dna<213>人工序列<400>31ggaggagggaaagacaga18<210>32<211>19<212>dna<213>人工序列<400>32aagcagtcaggtcaaagaa19<210>33<211>18<212>dna<213>人工序列<400>33aattgctgtgggaaataa18<210>34<211>18<212>dna<213>人工序列<400>34agttagcagcatgtcagc18<210>35<211>18<212>dna<213>人工序列<400>35ggaggagaaccgcgacct18<210>36<211>19<212>dna<213>人工序列<400>36ccccaggactggcttaccc19<210>37<211>18<212>dna<213>人工序列<400>37gccgttgccttgaccact18<210>38<211>18<212>dna<213>人工序列<400>38atccaaagtgggaattcc18<210>39<211>18<212>dna<213>人工序列<400>39gcaaaagcccactgacgc18<210>40<211>18<212>dna<213>人工序列<400>40ccctggatattcttagta18<210>41<211>21<212>dna<213>人工序列<400>41aggaatctgatgactttgagc21<210>42<211>21<212>dna<213>人工序列<400>42cagcaacatctttgagagaac21<210>43<211>18<212>dna<213>人工序列<400>43ctttctcccactgtattg18<210>44<211>19<212>dna<213>人工序列<400>44tcccaaataaaccaggcat19<210>45<211>20<212>dna<213>人工序列<400>45agaagacaaagaaggcagag20<210>46<211>19<212>dna<213>人工序列<400>46ttaatcttctatgccagac19<210>47<211>22<212>dna<213>人工序列<400>47ccagaaacaagtttcatactta22<210>48<211>19<212>dna<213>人工序列<400>48atatgaggggaagaaatcaag21<210>49<211>19<212>dna<213>人工序列<400>49tagccaagctggaaaagac19当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1
- 用于检测CYP2C19基因多态性的引物、探针及检测试剂盒的制造方法与工艺
- 分子信标探针及引物对、GC基因SNPs位点检测方法与流程
- 一种表面修饰分子印迹固相微萃取探针及其制备与应用的制造方法与工艺
- 一种木瑚菌属食用菌鉴别用分子标记及引物和探针的制造方法与工艺
- 一种用于检测样品中牛凸隆病毒的荧光RT-PCR引物、探针和试剂盒及检测方法与制造工艺
- CYP2C19基因多态性检测引物、探针及试剂盒的制造方法与工艺
- 检测CYP2C9和VKORC1基因多态性的引物、探针和试剂盒的制造方法与工艺
- 一种用于检验临床样本中活菌体的荧光PCR引物探针组及试剂盒的制造方法与工艺
- 含反义碱基的探针法实时荧光PCR的制造方法与工艺
- PCR反应管及包含其的固相载具的制造方法与工艺
- 一种木瑚菌属食用菌鉴别用分子标记及引物和探针的制造方法与工艺
- 一组用于驴源性的实时荧光PCR检测的特异性引物和探针的制造方法与工艺
- 一组用于鸡源性实时荧光PCR检测的特异性引物和探针的制造方法与工艺
- 一种快速检测异鳞蛇鲭和棘鳞蛇鲭源性成分的方法与制造工艺
- 一种检测食管癌相关标志物的探针组合及其试剂盒的制造方法与工艺
- 一种检测皮肤癌相关标志物的探针组合及其试剂盒的制造方法与工艺
- 一种检测骨髓瘤相关标志物的探针组合及其试剂盒的制造方法与工艺
- 一种用于检测样品中牛凸隆病毒的荧光RT-PCR引物、探针和试剂盒及检测方法与制造工艺
- CYP2C19基因多态性检测引物、探针及试剂盒的制造方法与工艺
- 检测CYP2C9和VKORC1基因多态性的引物、探针和试剂盒的制造方法与工艺
- 一种用于评估皮肤抗过敏情况的引物和探针及试剂盒的制作方法
- 一种用于评估皮肤防御情况的引物和探针及试剂盒的制作方法
- 一种荧光定量PCR检测猪delta冠状病毒的引物、探针、试剂盒及方法
- 一种快速区分HP-PRRS疫苗GDr180株与野毒株的引物、探针及方法
- 一种用于检测样品中猪凸隆病毒的荧光rt-pcr引物和探针和试剂盒及检测方法
- 一种阿胶中驴、马、牛和猪源性巢式荧光pcr检测引物、探针、试剂盒、检测方法及应用
- 一种用于评估皮肤防晒情况的引物和探针及试剂盒的制作方法
- 一种用于评估皮肤抗皱情况的引物和探针及试剂盒的制作方法
- 一种用于检测car-t转导效率的引物、探针和方法
- 用于微嵌合检测和个体识别的引物、探针、试剂盒及方法