一种识别HLA-A2分子与FMNKFIYEI短肽形成的复合物的单域抗体的制作方法
本发明属于抗体技术领域,具体涉及一种识别hla-a2分子与fmnkfiyei短肽形成的复合物的单域抗体。
背景技术:
肝细胞癌(hcc)已经成为世界性常见恶性肿瘤,死亡率高,在恶性肿瘤死亡顺位中仅次于胃、食道而居第三位,在部分地区的农村中则占第二位,仅次于胃癌。我国每年死于肝癌约11万人,占全世界肝癌死亡人数的45%。尽管患病的人不断增加,但治愈率仍然很低,严重威胁着人类健康,每年新增病人数超过70万。目前主要的治疗手段还是肿瘤切除、放射性治疗、消融治疗、化疗栓塞、肝移植以及靶向治疗,这些手段对大部分患者的治疗效果有一定的局限性,其治疗效果很不明显。迫切需要新的治疗手段及应对措施。
免疫治疗(immunotherapy)作为一种新的手段,其对于难治愈及复发性肿瘤表现出良好的治疗效果。免疫治疗是指针对机体低下或亢进的免疫状态,人为地增强或抑制机体的免疫功能以达到治疗疾病目的的治疗方法。免疫治疗的方法有很多,适用于多种疾病的治疗。肿瘤的免疫治疗旨在激活人体免疫系统,依靠自身免疫机能杀灭癌细胞和肿瘤组织。
嵌合抗原受体t细胞免疫疗法(chimericantigenreceptort-cellimmunotherapy,car-t)是一个出现了很多年,但是近几年才被改良使用到临床上的新型免疫疗法。其在肿瘤的治疗尤其是恶性血液瘤的治疗中,有突出的疗效。这种方法的成功依赖于自体或同种异体t细胞转基因表达合成cars/tcrs,定向攻击并杀死表达特定抗原的肿瘤细胞。car-t虽然在治疗cd19阳性血液瘤取得不错的成绩,但抗体靶向的car无一例外地只能靶向这些表达在肿瘤细胞表面的抗原,而众多肿瘤相关抗原都是仅表达在细胞内部。定位不准和不能持久化,以及免疫抑制微环境的影响都是目前实体瘤治疗的屏障。脱靶后伴随而来的细胞毒性也是一个及其危险的因素,这突出了目前实体瘤特异性抗原缺乏的现状。因此,确定精确的目标抗原和设计cars/tcrs对于这种疗法的临床应用是至关重要的。
afp是一种在小儿肝母细胞癌和hcc肿瘤细胞及内胚层组织分泌的糖蛋白;而在正常情况下,主要在胎儿肝中合成,在周岁时接近成人水平(低于30μg/l),这一现象使得afp有望成为肝癌t细胞免疫治疗中极具吸引力的靶向抗原。60%-80%hcc患者的肿瘤部位和血清中,afp的含量均有所上升,超出正常水平。虽然afp的功能尚不清晰,但目前有报道显示其能够促进细胞增殖、抑制细胞凋亡,同时也扮演着产生免疫抑制的角色,这表明其对肿瘤恶化有潜在的促进作用。
afp作为一个癌症特异性抗原极具吸引力,由于其在细胞内表达后却被分泌到胞外环境中,因此传统抗体疗法无药可施。在小鼠和人体内发现了afp/mhc特异性tcr,这就预示着afp/mhc复合物具有免疫原性;一些hla-a02:01与afp多肽复合物在人体内也发现具有免疫原性。遗憾的是,由于这些afp/mhc复合物特异性tcr无法克服肿瘤产生的免疫抑制,因此目前通过激活人体内源性tcr,产生免疫来治疗hcc患者,其效果还是微乎其微。tcr虽然既能识别胞外抗原也能识别通过抗原加工递呈至细胞表面的胞内抗原,但tcr通常需要从t细胞克隆中分离出特异性的αβtcr基因,难度较大,重复性不高。此外,转基因tcr还有tcr错配带来的潜在安全隐患,外源tcr的两条链可能会和内源tcr亚基发生错配,组合成新的tcr。这种错配的tcr会形成未知的特异性,可能会靶向正常组织,导致严重的移植物抗宿主反应。
如果能制备出识别mhc多肽复合物的抗体分子,并表达至细胞表面,就可以将t细胞免疫和成熟的抗体技术相结合,开发出一种兼具抗体和t细胞优势的新型t细胞修饰治疗技术。ochi等提供了一个较为成功的案例,将抑制内源性tcr的sirna整合于表达外源识别w抗原的tcr表达框中,用于修饰t细胞,开展针对白血病的过继治疗,证实在内源tcr被抑制状态下表达识别wt1转基因tcr的t细胞具有更强的抑制肿瘤功能。最近liu等人在靶向mhc/afp复合物的研究中也取得了不错的成就,他们成功获得了靶向mhc/afp的抗体分子,并且在体外杀伤试验中表现出良好的结果,能够特异性的杀死hepg2靶细胞。这种具有tcr特异性的抗体被用来定位、定量特异性多肽与mhc分子复合物,为研究抗原加工与递呈的机制提供了新的工具。这类识别多肽mhc复合物的抗体,因具有t细胞受体功能,被称作tcr-like抗体,有时也叫做tcr-mimic抗体。为了与修饰t细胞的car-t及tcr-t相区别,这类具有mhc多肽复合物特异性的抗体被命名为mar(mhcantigenreceptor)。
mar作为新型的识别mhc多肽复合物的分子与car相比,因具有天然t细胞受体的功能,沿用生命进化过程中获得的自我异体识别机制,即可识别肿瘤胞外抗原,也可识别通过抗原加工形成的肿瘤内抗原,因而具有更广泛的应用,尤其针对实体瘤抗原,具有得天独厚的优势。和tcr相比,mar转入t细胞后,不存在与内源性tcr亚基错配的问题;而且作为抗体,比天然tcr具有与生俱来更高的亲和力,mar可能成为最有发展前途的修饰t细胞用于肿瘤治疗的一类分子。
技术实现要素:
本发明针对现有技术中存在的技术问题,筛选得到一种识别hla-a2/fmnkfiyei抗原复合物的单域抗体,该单域抗体能特异性的结合fmnkfiyei短肽与hla-a2分子形成的复合物,可开发成治疗肿瘤的抗体药物及其他有有益效果生物治疗产品。
本发明提供的识别hla-a2/fmnkfiyei的单域抗体包括互补决定区cdr1、互补决定区cdr2和互补决定区cdr3,所述单域抗体为如下(a)或(b)或(c):
(a)所述单域抗体的互补决定区cdr1为如下(a1)或(a2)或(a3)或(a4):
(a1)包括seqidno.1所示的氨基酸序列;
(a2)seqidno.1所示的氨基酸序列;
(a3)将seqidno.1所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(a4)与seqidno.1所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(a)所述单域抗体的互补决定区cdr2为如下(a5)或(a6)或(a7)或(a8):
(a5)包括seqidno.2所示的氨基酸序列;
(a6)seqidno.2所示的氨基酸序列;
(a7)将seqidno.2所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(a8)与seqidno.2所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(a)所述单域抗体的互补决定区cdr3为如下(a9)或(a10)或(a11)或(a12):
(a9)包括seqidno.3所示的氨基酸序列;
(a10)seqidno.3所示的氨基酸序列;
(a11)将seqidno.3所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(a12)与seqidno.3所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(b)所述单域抗体的互补决定区cdr1为如下(b1)或(b2)或(b3)或(b4):
(b1)包括seqidno.4所示的氨基酸序列;
(b2)seqidno.4所示的氨基酸序列;
(b3)将seqidno.4所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(b4)与seqidno.4所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(b)所述单域抗体的互补决定区cdr2为如下(b5)或(b6)或(b7)或(b8):
(b5)包括seqidno.5所示的氨基酸序列;
(b6)seqidno.5所示的氨基酸序列;
(b7)将seqidno.5所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(b8)与seqidno.5所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(b)所述单域抗体的互补决定区cdr3为如下(b9)或(b10)或(a11)或(b12):
(b9)包括seqidno.6所示的氨基酸序列;
(b10)seqidno.6所示的氨基酸序列;
(b11)将seqidno.6所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(b12)与seqidno.6所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(c)所述单域抗体的互补决定区cdr1为如下(c1)或(c2)或(c3)或(c4):
(c1)包括seqidno.7所示的氨基酸序列;
(c2)seqidno.7所示的氨基酸序列;
(c3)将seqidno.7所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(c4)与seqidno.7所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(c)所述单域抗体的互补决定区cdr2为如下(c5)或(c6)或(c7)或(c8):
(c5)包括seqidno.8所示的氨基酸序列;
(c6)seqidno.8所示的氨基酸序列;
(c7)将seqidno.8所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(c8)与seqidno.8所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列;
(c)所述单域抗体的互补决定区cdr3为如下(c9)或(c10)或(c11)或(c12):
(c9)包括seqidno.9所示的氨基酸序列;
(c10)seqidno.9所示的氨基酸序列;
(c11)将seqidno.9所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(c12)与seqidno.9所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列。
上述单域抗体还包括框架区fr1、框架区fr2、框架区fr3和框架区fr4;
所述(a)中,所述单域抗体的框架区fr1为seqidno.10所示的氨基酸序列第1-28位、所述单域抗体的框架区fr2为seqidno.10所示的氨基酸序列第37-50位、所述单域抗体的框架区fr3为seqidno.10所示的氨基酸序列第57-97位、所述单域抗体的框架区fr4为seqidno.10所示的氨基酸序列第112-122位;
所述(b)中,所述单域抗体的框架区fr1为seqidno.11所示的氨基酸序列第1-27位、所述单域抗体的框架区fr2为seqidno.11所示的氨基酸序列第37-50位、所述单域抗体的框架区fr3为seqidno.11所示的氨基酸序列第57-97位、所述单域抗体的框架区fr4为seqidno.11所示的氨基酸序列第112-122位;
所述(c)中,所述单域抗体的框架区fr1为seqidno.12所示的氨基酸序列第1-27位、所述单域抗体的框架区fr2为seqidno.12所示的氨基酸序列第37-50位、所述单域抗体的框架区fr3为seqidno.12所示的氨基酸序列第57-97位、所述单域抗体的框架区fr4为seqidno.12所示的氨基酸序列第112-122位。
上述单域抗体的氨基酸序列为如下(d1)或(d2)或(d3):
(d1)seqidno.10或seqidno.11或seqidno.12所示的氨基酸序列;
(d2)将seqidno.10或seqidno.11或seqidno.12所示的氨基酸序列经过一个或几个氨基酸残基的取代和/或缺失和/或添加得到的具有相同功能的氨基酸序列;
(d3)与seqidno.10或seqidno.11或seqidno.12所示的氨基酸序列具有75%或75%以上的同源性且具有相同功能的氨基酸序列。
在本文中使用的术语“同源性”可描述两个或更多氨基酸序列相似程度,第一个氨基酸序列和第二个氨基酸序列之间同源性的百分比可以通过公式:(第一个氨基酸序列中与第二个氨基酸序列中相应位置处的氨基酸序列相同的氨基酸残基的数量)/(第一个氨基酸序列中氨基酸总数)*100%来计算,其中第二个氨基酸序列只能够的某个氨基酸的缺失、插入、替换或添加(与第一个氨基酸相比)被认为是有差别。同源性百分比也可以利用已知的用于序列匹配的计算机运算程序如ncbiblast获得。
上述单域抗体的编码基因序列为如下(e1)-(e3)中任一种:
(e1)seqidno.13或seqidno.14或seqidno.15所示的dna分子;
(e2)与(e1)限定的核苷酸序列具有75%或75%以上同一性,且编码上述单域抗体的dna分子;
(e3)在严格条件下与(e1)或(e2)限定的核苷酸序列杂交,且编码上述单域抗体的dna分子。
为解决上述技术问题,本发明又提供了上述单域抗体的衍生物。
本发明提供的上述单域抗体的衍生物为如下(f1)-(f6)中任一种:
(f1)含有上述单域抗体的融合蛋白;
(f2)含有上述单域抗体的多特异或多功能分子;
(f3)含有上述单域抗体的组合物;
(f4)含有上述单域抗体的免疫偶联物;
(f5)将上述单域抗体或其抗原结合部分进行修饰和/或改造后得到的抗体;
(f6)含有上述互补决定区的抗体。
上述衍生物中,所述融合蛋白是将上述单域抗体与至少1个具有治疗或识别功能的多肽分子融合得到的;所述具有治疗或识别功能的多肽分子为人源fc蛋白。
所述融合蛋白是将上述单域抗体与人源fc蛋白融合得到的fc融合蛋白。上述单域抗体融合人源fc蛋白后,由单价抗体成为双价抗体,亲和力有所提高。
上述衍生物中,所述组合物可为药物组合物,其含有与药学上可接受的载体。本发明的药物组合物可在联合治疗中施用,即与其他药剂联用。文中所用术语“药学上可接受的载体”包括生理学相容的任何和所有溶剂、分散介质、包衣、抗细菌和抗真菌剂、等渗和吸收延迟剂等。
本发明的药物组合物可包含一种或多种药学上可接受的盐。文中所用术语“药学上可接受的盐”是指保持了亲代化合物的所需生物活性且不引起任何不想要的毒理学作用的盐。这样的盐的例子包括酸加成盐和碱加成盐。酸加成盐包括那些由无毒性无机酸如盐酸、硝酸、磷酸、硫酸、氢溴酸、氢碘酸、亚磷酸等衍生的盐,以及由无毒性有机酸如脂族单羧酸和二羧酸、苯基取代的链烷酸、羟基链烷酸、芳族酸、脂族和芳族磺酸等衍生的盐。碱加成盐包括那些由碱土金属如钠、钾、镁、钙等衍生的盐,以及由无毒性有机胺如n,n’-二苄基乙二胺、n-甲基葡糖胺、氯普鲁卡因、胆碱、二乙醇胺、乙二胺、普鲁卡因等衍生的盐。
本发明的药物组合物也可含有药学上可接受的抗氧化剂。药学上可接受的抗氧化剂的例子包括:(1)水溶性抗氧化剂,如抗坏血酸、盐酸半胱氨酸、硫酸氢钠、焦亚硫酸钠,亚硫酸钠等;(2)油溶性抗氧化剂,如棕榈酸抗坏血酸酯、丁羟茴醚(bha)、丁羟甲苯(dht)、卵磷脂、没食子酸丙酯、α-生育酚等;(3)金属螯合剂,如柠檬酸、乙二胺四乙酸(edta)、山梨糖醇、酒石酸、磷酸等。
可用于本发明的药物组合物中的适当的水性或非水性载体的例子包括水,乙醇,多元醇(如甘油、丙二醇、聚乙二醇等),及其适当的混合物,植物油如橄榄油,和可注射的有机酯如油酸乙酯。例如通过应用包衣材料如卵磷脂,在分散液的情况下通过维持所需的颗粒大小,以及通过应用表面活性剂,可维持适当的流动性。
在实际应用中,这些组合物也可含有佐剂,如防腐剂、润湿剂、乳化剂和分散剂。可以通过灭菌程序或通过包含各种抗细菌和抗真菌剂如对羟基苯甲酸酯、氯代丁醇、苯酚山梨酸等来确保防止存在微生物。
本发明的组合物可以利用本领域公知的一种或多种方法通过一种或多种施用途径施用。本领域技术人员应当明白,施用途径和/或方式根据需要的结果而不同。
本发明的组合物具有体外和体内治疗应用。例如,这些分子可以施用于体外或离体培养的细胞,或者体内施用于人类受试者,以治疗、预防或诊断多种疾病。文中使用的术语“受试者”包括人和非人动物。非人动物包括所有脊椎动物,例如哺乳动物和非哺乳动物,例如非人灵长类动物、绵羊、狗、猫、牛、马、鸡、两栖类动物和爬行类动物。
上述衍生物中,所述免疫偶联物可为由上述单域抗体与治疗性剂如细胞毒素、药物(例如免疫抑制剂)或放射性毒素偶联,得到的偶联物。这些偶联物称为“免疫偶联物”。包括一个或多个细胞毒素的免疫偶联物称作“免疫毒素”。细胞毒素或细胞毒性剂包括对细胞有害(例如杀伤)的任何试剂。实例包括紫杉醇、细胞松弛素b、短杆菌肽d、溴化乙啶、吐根碱、丝裂霉素、表鬼臼毒吡喃葡糖苷、表鬼臼毒噻吩糖苷、长春新碱、长春碱、秋水仙素、阿霉素、柔红霉素、二羟基炭疽菌素二酮、米托蒽醌、光辉霉素、放线菌素d、l-脱氢睾酮、糖皮质激素、普鲁卡因、丁卡因、利多卡因、普萘洛尔和嘌呤霉素和它们的类似物或同系物。治疗剂还包括,例如,抗代谢物(例如,氨甲喋呤、6-巯基嘌呤、6-硫代鸟嘌呤、阿糖胞苷、5-氟尿嘧唳、氨烯咪胺(decarbazine),烧化剂(例如,氮芥、thioepa苯丁酸氮芥、苯丙氨酸氮芥、卡莫司汀(bsnu)和洛莫司汀(ccnu)、环磷酰胺、白消安、二溴甘露糖醇、链唑霉素、丝裂霉素c和顺-二氯二胺铂(ii)(ddp)顺铂),氨茴霉素类(例如,柔红菌素(以前称为道诺霉素)和阿霉素),抗生素(例如,放线菌素d、博来霉素、光辉霉素和安曲霉素(amc)),和抗有丝分裂剂(例如,长春新碱和长春碱)。能与本发明抗体偶联的治疗性细胞毒素的其他优选例子包括倍癌霉素、刺孢霉素、美坦生、auristatin,和它们的衍生物。将细胞毒素与本发明的抗体偶联可以利用本领域可用的接头技术。
本发明的抗体也可与放射性同位素偶联,产生细胞毒性放射性药物,也被称为放射性免疫偶联物。能够与诊断或治疗性使用的抗体偶联的放射性同位素的例子包括但不限于碘131、铟111、钇90和镥177。制备放射性免疫偶联物的方法在本领域中已经建立。放射性免疫偶联物的例子可以作为商品获得,包括zevalintm(idecpharmaceuticals)和bexxartm(corixapharmaceuticals),能够利用类似的方法使用本发明的抗体制备放射性免疫偶联物。
本发明的抗体偶联物可用于修饰特定的生物学反应,且药物部分不应理解为局限于经典的化学治疗剂。例如,药物部分可以是具有需要的生物活性的蛋白质或多肽。这样的蛋白质可包括,例如,具有酶活性的毒素或其活性片段,如相思豆毒蛋白、蓖麻毒蛋白a、假单胞菌外毒素或白喉毒素;蛋白质,如肿瘤坏死因子或干扰素-γ;或生物学反应调节物,如淋巴因子、白细胞介素-1(il-1)、白细胞介素-2(il-2)、白细胞介素-6(il-6)、粒细胞巨噬细胞集落刺激因子(gm-csf)、粒细胞集落刺激因子(g-csf)或其他生长因子。
将上述单域抗体或其抗原结合部分进行修饰和/或改造后得到的抗体基因工程改造的抗体也属于本发明的保护范围。本发明提供的单域抗体包含cdr1、cdr2和cdr3序列,其中这些cdr序列中的一个或多个包含基于本发明的单域抗体的特定氨基酸序列或其保守修饰,并且其中该抗体保留本发明抗体具有的识别和/或结合hla-a2/fmnkfiyei的功能特性。文中所用的术语“保守序列修饰”是指不会显著影响或改变含该氨基酸序列的抗体的结合特征的氨基酸修饰。这样的保守修饰包括氨基酸替代、添加和缺失。可以通过本领域公知的技术,如定点诱变和pcr介导的诱变,向本发明的抗体中引入修饰。保守氨基酸替代是指将氨基酸残基替代为一个具有相似侧链的氨基酸残基。具有相似侧链的氨基酸残基家族在本领域中已经定义。这些家族包括:具有碱性侧链(例如赖氨酸、精氨酸、组氨酸)、酸性侧链(例如天冬氨酸、谷氨酸)、不带电荷极性侧链(例如甘氨酸、天冬酰胺、谷氨酰胺、丝氨酸、苏氨酸、酪氨酸、半胱氨酸、色氨酸)、非极性侧链(例如丙氨酸、缬氨酸、亮氨酸、异亮氨酸、脯氨酸、苯丙氨酸、甲硫氨酸)、β-分支侧链(例如苏氨酸、缬氨酸、异亮氨酸)和芳族侧链(例如酪氨酸、苯丙氨酸、色氨酸、组氨酸)的氨基酸。因此,本发明抗体的cdr区内的一个或多个氨基酸残基可以被替代为来自相同侧链家族的其他氨基酸残基,并且可以用本文所述的体外亲和力试验检测改变的抗体保留的上述功能。
可以进行的一种类型的可变区工程化是cdr移植。抗体主要是通过位于互补决定区(cdr)中的氨基酸残基与靶抗原相互作用。由于这个原因,cdr内的氨基酸序列比cdr外的序列在各个抗体之间更加多样化。由于cdr序列负责大多数抗体-抗原相互作用,因此通过构建如下的表达载体可以表达模拟该特定存在抗体的特性的重组抗体:该表达载体包含上述单域抗体的cdr序列,该cdr序列被移植到来自具有不同特性的不同抗体的骨架序列上,这些骨架序列可以从公共dna数据库或发表的参考文献中获得。
另一种类型的可变区修饰是将cdr1、cdr2和/或cdr3区内的氨基酸序列突变,从而改善目标抗体的一种或多种结合特性(例如亲和力)。可以进行定点诱变或pcr介导的诱变,以引入突变,对抗体结合的影响,或者其他目标功能特性,可以用文中所述以及在实施例中提供的试验来评价。优选引入(如上文所述的)保守序列修饰。突变可以是氨基酸替代、添加或缺失,但是优选替代。而且,一般在cdr区内改变不超过5个残基。
为解决上述技术问题,本发明还提供了与上述单域抗体或衍生物相关的生物材料。
本发明提供的与上述单域抗体或衍生物相关的生物材料为如下(g1)-(g4)中任一种:
(g1)编码上述单域抗体的核酸分子;
(g2)编码上述融合蛋白的核酸分子;
(g3)含有(g1)或(g2)所述的核酸分子的载体;
(g4)含有(g1)或(g2)所述的核酸分子或(g3)所述的载体的宿主细胞。
上述生物材料中,所述核酸分子为如下(h1)-(h3)中任一种:
(h1)seqidno.13或seqidno.14或seqidno.15或seqidno.16或seqidno.17或seqidno.18所示的dna分子;
(h2)与(h1)限定的核苷酸序列具有75%或75%以上同一性,且编码上述单域抗体或融合蛋白的dna分子;
(h3)在严格条件下与(h1)或(h2)限定的核苷酸序列杂交,且编码上述单域抗体或融合蛋白的dna分子。
上述生物材料中,所述核酸分子可以为编码各互补决定区或单域抗体或融合蛋白氨基酸序列的核苷酸序列,通过遗传密码子可以随时获得相应核酸分子的具体序列。由于遗传密码子具有兼并性,该核酸分子可以根据不同的应用目的不同。在本文中使用的术语“密码子”又称三联体密码子,指对应于某种氨基酸的核苷酸三联体。在转译过程中决定该种氨基酸插入生长中多肽链的位置。
本发明的核酸分子可以利用常规分子生物学技术获得。对于从免疫球蛋白基因文库中获得的抗体(例如使用噬菌体展示技术),编码抗体的核酸可以从文库中获得。
上述生物材料中,所述载体中的所述核酸序列或者至少部分序列可以通过合适的表达系统进行表达,以得到相应的蛋白质或多肽;所述表达系统包括细菌、酵母菌、丝状真菌、哺乳动物细胞、昆虫细胞、植物细胞或无细胞表达系统。
上述单域抗体或衍生物或生物材料在如下(i1)-(i6)中任一种中的应用也属于本发明的保护范围:
(i1)特异性识别和/或结合hla-a2/fmnkfiyei;
(i2)制备特异性识别和/或结合hla-a2/fmnkfiyei的产品;
(i3)特异性识别和/或结合表达hla-a2/fmnkfiyei的肿瘤细胞;
(i4)制备特异性识别和/或结合表达hla-a2/fmnkfiyei的肿瘤细胞的产品;
(i5)肿瘤免疫治疗;
(i6)制备肿瘤免疫治疗的产品;
可以应用本发明的抗体治疗的肿瘤包括但不限于肝癌、黑色素瘤、乳腺癌、前列腺癌、肺癌、卵巢癌、甲状腺癌、膀胱癌或胃癌等肿瘤。
在实际应用中,可用本发明的抗体或融合蛋白对t细胞在体外进行修饰,得到武装后t细胞,如car-t细胞。武装后t细胞增殖放大后,将其放回受试者体内,武装t细胞可特异性识别肿瘤,用于体内肿瘤免疫治疗。t细胞的修饰可通过本领域技术人员公知的常规方法实现。
本发明的hla-a2/fmnkfiyei为hla-a2/fmnkfiyei抗原复合物,所述hla-a2/fmnkfiyei抗原复合物是来自afp的抗原肽fmnkfiyei与抗原分子hla-a2形成的复合物。
为解决上述技术问题,本发明最后提供了上述融合蛋白的制备方法。
本发明提供的上述融合蛋白的制备方法包括如下步骤:将上述单域抗体的编码基因和人源fc蛋白的编码基因导入宿主细胞,得到重组细胞;培养所述重组细胞,得到所述融合蛋白。
上述方法中,所述单域抗体的编码基因和所述人源fc蛋白的编码基因是通过重组载体导入宿主细胞;
所述重组载体为将含有所述单域抗体的编码基因和所述人源fc蛋白的编码基因的片段插入表达载体的多克隆位点中得到的。
具体地,所述含有所述单域抗体的编码基因和所述人源fc蛋白的编码基因的片段为seqidno.16或seqidno.17或seqidno.18所示的dna分子。
具体地,所述表达载体为pcdna3.1载体。
具体地,所述宿主细胞为293f细胞。
本发明通过三轮生物淘选,从噬菌体单域文库中筛选得到一种亲和力较高的识别hla-a2/fmnkfiyei抗原复合物的单域抗体及其序列,并将得到的抗体序列克隆至原核/真核表达载体中,与人源fc融合表达,转染宿主细胞,获得fc融合蛋白。经体外实验证明:本发明提供的单域抗体及fc融合蛋白均可特异性识别并结合由来自afp的特异性多肽fmnkfiyei与hla-a2形成的复合物,而和其他对照多肽形成的复合物没有结合。这种特异性单域抗体不仅识别人工合成的hla-a2/fmnkfiyei复合物,而且也可以结合天然抗原加工的,表达在肿瘤细胞表面的hla-a2/fmnkfiyei复合物,为进一步开发治疗肿瘤的生物制剂打下了基础。
附图说明
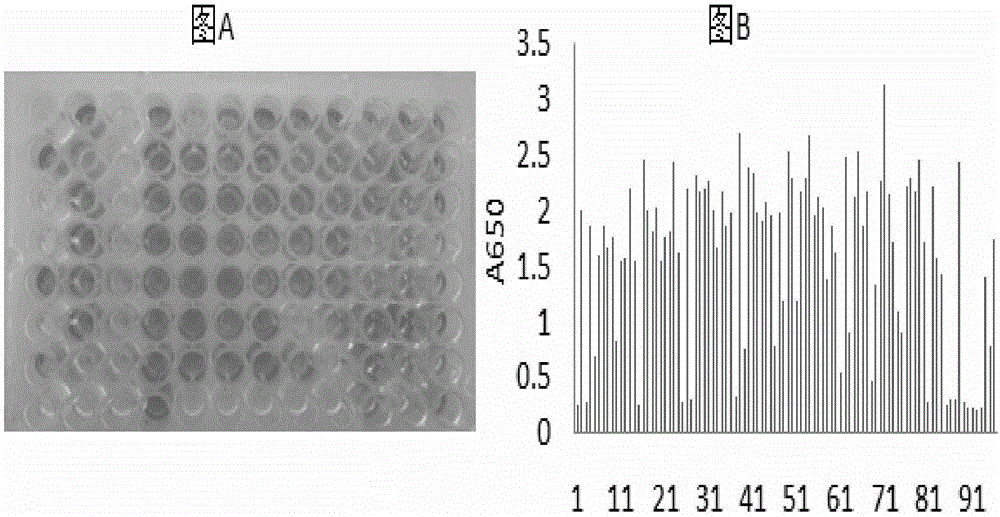
图1为三轮淘选后第一板elisa检测和数据分析结果。图1a为三轮淘选后第一板elisa检测;elisa所有孔均包被抗原hla-a2/fmnkfiyei,与淘筛的单个克隆噬菌体结合后,加入标记hrp的抗噬菌体的二抗。图1b为图1a中三轮淘选后第一板elisa检测数据分析结果;纵坐标为各孔在650nm下的光吸收值,横坐标为96个孔,其中,1-8为a1、b1、c1、d1、e1、f1、g1、h1,9-16为a2、b2、c2、d2、e2、f2、g2、h2,依次类推,89-96为a12、b12、c12、d12、e12、f12、g12、h12。
图2为三轮淘选后第二板elisa检测和数据分析结果。图2a为三轮淘选后第二板elisa检测;elisa所有孔均包被抗原hla-a2/itdqvpfsv。图2b为图2a中elisa检测数据分析结果;纵坐标为各孔在650nm下的光吸收值,横坐标为96个孔。
图3为质粒图谱示意图及融合蛋白纯化后的sds-page的还原及非还原电泳图。图3a为pcdna3.1中单域抗体融合表达fc的质粒图谱示意图。单域抗体与fc之间通过限制性酶连接,且单域抗体前有信号肽以及kozak序列。图3b为m10-a2、m10-c10、m10-h4和m10-d12与fc融合蛋白纯化后的sds-page的还原及非还原电泳图;marker的条带从小到大依次为14,25,30,40,50,70,100,120,160kd。line1-line8分别为还原态和非还原态的fc融合蛋白。
图4为四种fc融合抗体对不同抗原的特异性检测以及数据分析结果。t2细胞分别加载多肽抗原itdqvpfsv和fmnkfiyei,编号分别是p3和p10。
图5为facs检测m10-a2-fc、m10-c10-fc、m10-h4-fc和m10-d12-fc融合蛋白在hepg2细胞水平对hla-a2/fmnkfiyei的特异性识别。其中,negative为hepg2细胞加不结合hla-a2/fmnkfiyei的fc融合蛋白b7以及二抗,三个实验组分别为hepg2细胞加m10-a2-fc、m10-c10-fc和m10-h4-fc融合蛋白以及二抗。
图6为facs检测m10-a2-fc、m10-c10-fc、m10-h4-fc和m10-d12-fc融合蛋白在hep3b细胞水平对hla-a2/fmnkfiyei的特异性识别。其中,negative为hep3b细胞加不结合hla-a2/fmnkfiyei的fc融合蛋白b7以及二抗,三个实验组分别为hep3b细胞加m10-a2-fc、m10-c10-fc和m10-h4-fc融合蛋白以及二抗。
图7为facs检测m10-a2-fc、m10-c10-fc、m10-h4-fc和m10-d12-fc融合蛋白在a375细胞水平对hla-a2/fmnkfiyei的特异性识别。其中,negative为a375细胞加不结合hla-a2/fmnkfiyei的fc融合蛋白b7以及二抗,三个实验组分别为a375细胞加m10-a2-fc、m10-c10-fc和m10-h4-fc融合蛋白以及二抗。
图8为融合蛋白m10-c10-fc和m10-h4-fc与hla-a2/fmnkfiyei复合物分子间相互作用及亲和力常数测定图。图8a为m10-c10-fc;图8b为m10-h4-fc。其中,横坐标为时间,纵坐标为分子间相互结合的响应值(ru)。
具体实施方式
下述实施例中所使用的实验方法如无特殊说明,均为常规方法。
下述实施例中所用的材料、试剂等,如无特殊说明,均可从商业途径得到。
下述实施例中的定量试验,均设置三次重复实验,结果取平均值。
下述实施例中的hla-a2/fmnkfiyei抗原复合物是指抗原肽fmnkfiyei与mhc一类分子hla-a*02:01的复合物,记载于文献“targetingalpha-fetoprotein(afp)-mhccomplexwithcart-celltherapyforlivercancer”中,公众可从天津天锐生物科技有限公司获得。
实施例1筛选hla-a2/fmnkfiyei复合物的单域抗体
1.1单域抗体噬菌体文库制备
1.1.1辅助噬菌体(bm13)的制备
将m13ke噬菌体(购自neb#n0316s)复制子用alwni(购自neb)和afei(购自neb)双酶切,同时人工合成基因片段也用alwni和afei(购自neb)双酶切,然后用t4连接酶连接在一起。连接后转染tg1得到辅助噬菌体bm13。如此,原复制子tctggtggtggttctggtggcggctctgagggtggtggctctgagggtggcggttctgagggtggcggctctgagggaggcggttccggtggtggctct序列被人工合成基因序列所取代,即在噬菌体giii编码区域,添加了胰蛋白酶切割序列,一旦用作辅助噬菌体时,增加胰蛋白酶消化步骤,减少不含融合目的基因蛋白噬菌体的数目。
人工合成基因序列如下:ccagccggcctttctgaggggtcgactatagaaggacgaggggcccacgaaggaggtggggtacccggttccgagggt。
1.1.2载体构建
将dp47单域抗体编码基因序列(ay466510.1)插入puc19载体(北京全式金生物技术有限公司)的hindiii和ndei酶切位点间,得到噬菌体展示载体pbg3。单域抗体表达框架中,dp47单域抗体与giii蛋白融合表达,表达的融合蛋白含有myc和vsv-g标签,用于纯化或鉴定。
1.1.3噬菌体文库构建
1.1.3.1羊驼(vicugnapacos)外周血单个核细胞总rna的提取
取100ml羊驼血,缓慢加入50mlficoll溶液,18℃,300g/min离心30min,轻轻吸取外周血单个核细胞层,加入200mlficoll溶液充分洗涤,400g/min离心5min,弃上清。使用血液总rna提取试剂盒(购自天根)对外周血单个核细胞的总rna进行提取和纯化。
1.1.3.2羊驼单域抗体dna文库的制备
将1.1.3.1中获得的总rna用于cdna的合成,使用revertaidrtreversetranscriptionkit试剂盒(购自thermo)进行cdna的合成。dna文库制备使用简并寡核苷酸链进行单域抗体dna的扩增,寡核苷酸链由金唯智公司按下表合成:
oligo_vhhf:catgccatgggacaggtgcagctbgtgghgdcw;
oligo_vhhr:aaggaaggaaaaaagcggccgchgavgagacrgtgacctgggtc。
将人工合成的上述寡核苷酸链加入50μl含下列成份的体系中:25μlprimestarmaxpremix(购自takara)、2μlcdna、23μl去离子水,寡核苷酸链终浓度均为0.2μm。根据primestarmaxpremix产品说明书的条件进行羊驼单域抗体dna文库的扩增。扩增产物经过琼脂糖凝胶电泳后使用gelextractionkit(购自omegabio-tek)进行纯化,制备得到羊驼单域抗体dna文库。
1.1.3.3羊驼单域抗体噬菌体文库的构建
将1.1.2中构建载体pbg3和1.1.3.2中制备得到的羊驼单域抗体dna文库分别用ncoi(购自takara)和noti(购自takara)双酶切,产物经过琼脂糖凝胶电泳后使用gelextractionkit(购自omegabio-tek)分别进行纯化,并以t4dnaligase(购自thermo)进行连接。连接产物使用gelextractionkit(购自omegabio-tek)进行纯化,并电转化大肠杆菌tg1(购于武汉淼灵生物科技有限公司)感受态细胞,涂布于2×ty培养基平板,37℃培养过夜。将平板上所有克隆刮下,重悬于2×ty液体培养基,获得羊驼单域抗体噬菌体文库。
1.2bm13辅助噬菌体以及噬菌体文库的增殖
1.2.1bm13辅助噬菌体的增殖
从基本琼脂培养基平板上挑取一个大肠杆菌tg1(购于武汉淼灵生物科技有限公司)的新鲜单菌落,接种到20ml2×ty培养基中,温和摇动,37℃条件下培养至od600约为0.8备用。将1.1.1中制备的原始bm13辅助噬菌体用2×ty培养基制备一系列10倍稀释的bm13噬菌体。各稀释度分别取10μl与200μltg1(od600=0.8)菌液混匀,振荡器温和震荡3s,并与上层琼脂培养基混匀,倾倒在预先平衡室温的ty平板上。转动平板以确保菌体和上层琼脂分布均匀。待上层培养基凝固后,于37℃生化培养箱倒置培养过夜。
次日挑选分离良好的单个噬菌体接种于装有2~3ml含25μg/ml卡那霉素的2×ty培养基的15ml培养管中。在37℃,250rpm条件下震荡培养12~16h。将感染上清液转移至1.5ml无菌微量离心管,在微量离心机上,以最大转速,4℃离心2min。上清液转移至新管中4℃保存。
1.2.2噬菌体文库的增殖
将制备好的噬菌体文库接种至100ml含60μg/ml氨苄青霉素的2×ty培养基中,在37℃,250rpm条件下震荡培养至od600为0.8时,加入bm13至浓度为2×107pfu/ml。在37℃,300rpm条件下培养1h,加入25μg/ml卡那霉素,在37℃条件下继续培养14~18h。将菌液离心,上清液用5%peg沉淀,然后重悬于5%mpbs中备用。
1.3筛选hla-a2/fmnkfiyei复合物的单域抗体
1.3.1生物淘选
链酶亲和素免疫磁珠(购自invitrogencatno.sku#112-05d)取25μl,加入一定量的生物素化的hla-a2/fmnkfiyei,室温结合5min。pbst及pbs洗2~3次。mpbs封闭2h,pbst及pbs洗2~3次。将噬菌体文库加入磁珠中,室温孵育2h。
移走未结合文库,磁珠用pbst及pbs各洗10次。向磁珠中加入0.01%胰酶溶液,室温孵育1h,得到胰酶洗脱液。将胰酶洗脱液加入到30mltg1(od600=0.5)中,按1.2中方法增殖第一次洗脱文库。如此进行第二轮、第三轮淘选。挑取三轮淘选后所得的单菌落至96孔板中。按1.2中文库增殖方法制备出培养物上清液。
1.3.2elisa鉴定
取一定量上清液进行elisa鉴定(图1a和图2a)。elisa鉴定的具体步骤如下:用包被缓冲液将已知抗原稀释至1~10μg/ml,每孔加0.1ml,4℃过夜;次日洗涤3次;加一定稀释的待检样品0.1ml于上述已包被抗原的反应孔中,37℃孵育1小时,洗涤;加入新鲜稀释的酶标第二抗体(anti-km13-hrp,1:5000)0.1ml,37℃孵育60分钟,洗涤;最后一遍用ddw洗涤。于各反应孔中加入临时配制的tmb底物溶液0.1ml,在37℃条件下静置10~30分钟。用高级读板机在650nm波长下读板。
elisa鉴定示意图如图1a和图2a所示。其中,图1a为elisa所有孔均包被抗原hla-a2/fmnkfiyei;图2a为elisa所有孔均包被抗原hla-a2/itdqvpfsv;每孔中抗体各不相同。各孔在650nm下的光吸收值检测结果如图1b和图2b所示。选择图2中a650nm在0.3以下,图1中a650nm在1.5以上的克隆进行测序,分别得到其核苷酸序列,再根据核苷酸序列确定其编码的相应蛋白质的氨基酸序列。
将seqidno.10所示的氨基酸序列对应的克隆命名为m10-a2,其对应的单域抗体为单域抗体m10-a2;将seqidno.11所示的氨基酸序列对应的克隆命名为m10-c10,其对应的单域抗体为单域抗体m10-c10;将seqidno.12所示的氨基酸序列对应的克隆命名为m10-h4,其对应的单域抗体为单域抗体m10-h4。将seqidno.20所示的氨基酸序列对应的克隆命名为m10-d12,其对应的单域抗体为单域抗体m10-d12。
单域抗体m10-a2的氨基酸序列如seqidno.10所示,编码基因序列如seqidno.13所示。其中,单域抗体m10-a2的互补决定区cdr1的氨基酸序列如seqidno.1所示、互补决定区cdr2的氨基酸序列如seqidno.2所示、互补决定区cdr3的氨基酸序列如seqidno.3所示。
单域抗体m10-c10的氨基酸序列如seqidno.11所示,编码基因序列如seqidno.14所示。其中,单域抗体m10-c10的互补决定区cdr1的氨基酸序列如seqidno.4所示、互补决定区cdr2的氨基酸序列如seqidno.5所示、互补决定区cdr3的氨基酸序列如seqidno.6所示。
单域抗体m10-h4的氨基酸序列如seqidno.12所示,编码基因序列如seqidno.15所示。其中,单域抗体m10-h4的互补决定区cdr1的氨基酸序列如seqidno.7所示、互补决定区cdr2的氨基酸序列如seqidno.8所示、互补决定区cdr3的氨基酸序列如seqidno.9所示。
单域抗体m10-d12的氨基酸序列如seqidno.20所示,编码基因序列如seqidno.21所示。
上述获得的单域抗体m10-a2、m10-c10、m10-h4、m10-d12与抗原hla-a2/fmnkfiyei表现出良好的亲和力,而与不相关多肽复合物抗原hla-a2/itdqvpfsv表现为亲和力较差,说明所得抗体对多肽部分特异而非hla-a2部分。
实施例2、融合蛋白m10-a2-fc、m10-c10-fc、m10-h4-fc和m10-d12-fc的制备
1、重组载体的构建
将seqidno.16所示的dna分子(其包括kozak序列及信号肽、m10-a2抗体序列、人源fc序列及标签序列)克隆到pcdna3.1的hindⅲ和xbai限制性内切酶位点间,得到重组载体pcdna3.1-m10-a2-fc。
将seqidno.17所示的dna分子(其包括kozak序列及信号肽、m10-c10抗体序列、人源fc序列及标签序列)克隆到pcdna3.1的hindⅲ和xbai限制性内切酶位点间,得到重组载体pcdna3.1-m10-c10-fc。
将seqidno.18所示的dna分子(其包括kozak序列及信号肽、m10-h4抗体序列、人源fc序列及标签序列)克隆到pcdna3.1的hindⅲ和xbai限制性内切酶位点间,得到重组载体pcdna3.1-m10-h4-fc。
将seqidno.19所示的dna分子(其包括kozak序列及信号肽、m10-b7抗体序列、人源fc序列及标签序列)克隆到pcdna3.1的hindⅲ和xbai限制性内切酶位点间,得到重组载体pcdna3.1-m10-b7-fc(对照)。
将seqidno.22所示的dna分子(其包括kozak序列及信号肽、m10-d12抗体序列、人源fc序列及标签序列)克隆到pcdna3.1的hindⅲ和xbai限制性内切酶位点间,得到重组载体pcdna3.1-m10-d12-fc。
上述重组载体的结构示意图如图3a所示。
2、重组细胞的构建及培养
将重组载体pcdna3.1-m10-a2-fc、pcdna3.1-m10-c10-fc、pcdna3.1-m10-d12-fc、pcdna3.1-m10-h4-fc和pcdna3.1-m10-b7-fc瞬时转染到293f细胞(thermofisher,a14527)中,培养4天后离心,收集上清液,并用proteina纯化。
3、融合蛋白的鉴定
融合蛋白m10-a2-fc、m10-c10-fc、m10-d12-fc、m10-h4-fc和m10-b7-fc纯化后跑sds-pag,结果如图3b所示。其中,条带1、3、5和7分别为非还原状态下的融合蛋白m10-a2-fc、m10-c10-fc、m10-d12-fc和m10-h4-fc纯化后电泳图,条带2、4、6和8分别为还原状态下的融合蛋白m10-a2-fc、m10-c10-fc、m10-d12-fc和m10-h4-fc纯化后电泳图。marker的条带从小到大依次为14,25,30,40,50,70,100,120,160kd。
实施例3、融合蛋白特异性识别t2细胞呈递的hla-a2/fmnkfiyei复合物
为了检测融合蛋白m10-a2-fc、m10-c10-fc、m10-d12-fc、m10-h4-fc、m9-b7-fc对照是否能特异结合细胞表面表达的hla-a2/afpfmnkfiyei(afpfmnkfiyei简称p10)和无关的hla-a2/gp100itdqvpfsv(gp100itdqvpfs简称p3),以肽脉冲的t2细胞(t2细胞含有hla-a2基因,但仅在细胞表面表达很低水平的hla-a2分子,不能呈递内源性抗原。外源肽的脉冲能稳定hla-a2分子,进而提高t2细胞表面的hla-a2分子表达水平)作为靶细胞,通过facs分析方法来分析融合蛋白m10-a2-fc、m10-c10-fc、m10-d12-fc、m10-h4-fc、m9-b7-fc对照对肽脉冲的t2细胞的结合能力。具体检测步骤如下:向3×105个t2细胞中加入多肽(p3或p10,购自吉尔生化上海有限公司)至50μg,每管加入0.1mlrpmi-1640培养基(购自gibco),37℃过夜;次日用1mlpbs缓冲液洗涤1次;加一定稀释的待检样品(proteina纯化的融合蛋白m10-a2-fc、m10-c10-fc、m10-d12-fc、m10-h4-fc、m9-b7-fc对照)2μg于上述ep管中,室温孵育1小时,用1mlpbs缓冲液洗涤;之后加入新鲜稀释(用0.1mlpbs缓冲液稀释)的荧光二抗体(dy650标记的抗人的抗体,1:5000,购自abcam);室温孵育30分钟,用0.3mlpbs缓冲液洗涤。用facscan仪器(guavaeasycytemini)进行检测。结果用flowjo软件进行分析。
结果如图4所示。p3为不相关小肽gp100itdqvpfsv孵育t2细胞后与所筛抗体的流式检测,p10为afpfmnkfiyei孵育t2细胞后与所筛抗体的流式检测。从图a、b、c、d中可以看到m10-a2-fc、m10-c10-fc和m10-h4-fc都对p10小肽孵育的t2细胞有特异性亲和,而对p3小肽孵育的t2细胞表现为不结合,m10-d12-fc对p10小肽孵育的t2细胞也没有亲和力。
实施例4、融合蛋白m10-a2-fc、m10-c10-fc和m10-h4-fc特异性识别天然加工的表达在肿瘤细胞表面的hla-a2/fmnkfiyei复合物
流式细胞术检测融合蛋白m10-a2-fc、m10-c10-fc和m10-h4-fc对表面表达hla-a2/fmnkfiyei的hepg2细胞(表达hla-a2+afp+)、hep3b细胞(hla-a2-afp+)及a375细胞(hla-a2+afp-)的特异性识别作用和亲和力。具体检测步骤如下:将1×105个肿瘤细胞(hepg2或hep3b或a375均购自中国医学科学院基础医学研究所细胞资源中心)、0.1mldmem培养基(购自gibco)、一定稀释的待检样品(proteina纯化的融合蛋白m10-a2-fc、m10-c10-fc或m10-h4-fc)2μg在ep管中混匀,室温4℃过夜,用1mlpbs缓冲液洗涤;加入新鲜稀释(用0.1mlpbs缓冲液稀释)的荧光二抗体(dy650标记的抗人的抗体,1:5000)0.1ml,4℃孵育30分钟,用1mlpbs缓冲液洗涤;加0.3mlpbs缓冲液。用facscan仪器(guavaeasycytemini)进行检测,结果用flowjo软件进行分析。
结果如图5、图6和图7所示。图5中negative为hepg2细胞加不结合hla-a2/fmnkfiyeifc融合蛋白作为一抗的同型对照以及二抗山羊抗人fc(fitc),另一曲线为hepg2细胞加实验组三个融合蛋白m10-a2-fc、m10-c10-fc或m10-h4-fc以及二抗山羊抗人fc(fitc)。图6中所用细胞为hep3b细胞,所孵育抗体与图5相同。图7中所用细胞为a375细胞,所孵育抗体与图5相同。从上述结果可以看出:融合蛋白m10-a2-fc、m10-c10-fc和m10-h4-fc与hepg2表现出不同程度的亲和力,而与hep3b细胞和a375细胞没有亲和力。
实施例5、融合蛋白m10-c10-fc和m10-h4-fc与hla-a2/fmnkfiyei复合物分子间相互作用及亲和力常数测定。
利用等离子共振技术进行抗体融合蛋白m10-c10-fc和m10-h4-fc与fmnkfiyei/hla-a2的相互作用及亲和力常数测定。实验选用链霉亲和素偶联的传感芯片(senserchipsa),以hbs+ep+为流动相缓冲液。在传感芯片的一个通道上固定不相关抗原itdqvpfsv/hla-a2作为参比抗原,另一个通道上固定fmnkfiyei/hla-a2作为实验抗原,参比抗原用来检测背景结合情况。分别稀释融合抗体m10-c10-fc和m10-h4-fc,从10nm到220nm浓度进行进样,样品同时流经固定有fmnkfiyei/hla-a2和itdqvpfsv/hla-a2的通道表面。实验所得数据由biacoret200仪器进行采集并分析,反应动力学曲线以heterogeneousligand模型进行拟合,并计算结合速率常数(ka),解离速率常数(kd)和亲和力常数(kd)。其中,横坐标为时间,纵坐标为分子间相互结合的响应值(ru)。
结果如图8所示,图a为m10-c10-fc;图b为m10-h4-fc。实验结果表明,融合抗体m10-c10-fc与fmnkfiyei/hla-a2发生特异性结合,与参比抗原无交叉反应,不结合;结合速率常数4280(1/ms),解离速率常数1.15e-5(1/s),亲和力常数2.688e-9(m)。融合抗体m10-h4-fc与fmnkfiyei/hla-a2发生特异性结合,与参比抗原无交叉反应,不结合;结合速率常数32770(1/ms),解离速率常数4.712e-2(1/s),亲和力常数2.431e-7(m)。
序列表
<110>天津天锐生物科技有限公司
<120>一种识别hla-a2分子与fmnkfiyei短肽形成的复合物的单域抗体
<160>22
<170>patentinversion3.5
<210>1
<211>8
<212>prt
<213>人工序列(artificialsequence)
<400>1
ilepheserilealaglymetgly
15
<210>2
<211>6
<212>prt
<213>人工序列(artificialsequence)
<400>2
phemetphepheasnleu
15
<210>3
<211>14
<212>prt
<213>人工序列(artificialsequence)
<400>3
asnalagluglyleuthrasptyralaalapropheaspser
1510
<210>4
<211>9
<212>prt
<213>人工序列(artificialsequence)
<400>4
glnileserserileserilethrgly
15
<210>5
<211>6
<212>prt
<213>人工序列(artificialsequence)
<400>5
alaleuglytrpasngly
15
<210>6
<211>14
<212>prt
<213>人工序列(artificialsequence)
<400>6
asnvalgluglyleuthrasptyralaalapropheaspser
1510
<210>7
<211>9
<212>prt
<213>人工序列(artificialsequence)
<400>7
metileserserileserilethrgly
15
<210>8
<211>6
<212>prt
<213>人工序列(artificialsequence)
<400>8
alaleuglytrpasngly
15
<210>9
<211>14
<212>prt
<213>人工序列(artificialsequence)
<400>9
asnvalgluglyleuthrasptyralaalapropheaspser
1510
<210>10
<211>122
<212>prt
<213>人工序列(artificialsequence)
<400>10
glyglnleuglnleuvalgluserglyglyglyleuvalglnprogly
151015
glyserleuargleusercysalaalaserglyserilepheserile
202530
alaglymetglytrptyrargargalaproglylysglnleugluleu
354045
valserphemetphepheasnleutyrserthrtyrtyrglyaspser
505560
valgluglyargphealaileserargaspglyalagluasnthrval
65707580
tyrleuglnmetasnserleulysprogluaspthralavaltyrtyr
859095
cysasnalagluglyleuthrasptyralaalapropheaspsertrp
100105110
glyglnglythrglnvalthrvalserser
115120
<210>11
<211>122
<212>prt
<213>人工序列(artificialsequence)
<400>11
glyglnleuglnleuvalgluserglyglyglyleuvalglnalagly
151015
glyserleuargleusercysalaalaserglyglnileserserile
202530
serilethrglytrppheargglnalaproglylysgluarggluphe
354045
valseralaleuglytrpasnglyglyasnthrtyrtyralaaspser
505560
vallysglyargphethrileserargaspasnalagluasnmetval
65707580
tyrleuglnmetasnserleulysprogluaspthralavaltyrtyr
859095
cysasnvalgluglyleuthrasptyralaalapropheaspsertrp
100105110
glyglnglythrglnvalthrvalserser
115120
<210>12
<211>122
<212>prt
<213>人工序列(artificialsequence)
<400>12
glyglnvalglnleuvalgluserglyglyglyleuvalglnalagly
151015
glyserleuargleusercysglualaserglymetileserserile
202530
serilethrglytrppheargglnalaproglylysgluarggluphe
354045
valseralaleuglytrpasnglyglyasnthrtyrtyralaaspser
505560
vallysglyargphethrileserargaspseralalysasnmetval
65707580
tyrleuglnmetasnserleulysprogluaspthralavaltyrtyr
859095
cysasnvalgluglyleuthrasptyralaalapropheaspsertrp
100105110
glyglnglythrglnvalthrvalserser
115120
<210>13
<211>366
<212>dna
<213>人工序列(artificialsequence)
<400>13
ggacagttgcagctcgtggagtctgggggaggcttggtgcagcctggggggtctcttaga60
ctctcctgtgcagcctctggaagcatcttcagtatcgccggcatgggttggtaccgccga120
gctccagggaagcagcttgaattggtatcgtttatgttttttaatttgtattctacttat180
tatggcgactccgtggagggccgattcgccatctccagagacggcgccgagaatacggta240
tatctccaaatgaacagcctgaaacctgaggacacggccgtctattactgtaatgcggag300
gggttaaccgactatgcagccccctttgattcctggggccaggggacccaggtcaccgtc360
tcctcg366
<210>14
<211>366
<212>dna
<213>人工序列(artificialsequence)
<400>14
ggacagttgcagctcgtggagtctgggggaggcttggtgcaggctggggggtctctgaga60
ctctcctgtgcagcctctggacagatctccagtattagtatcacgggctggttccgccag120
gctccaggaaaggaacgtgagtttgtgtcagctcttggctggaatggtggcaatacatac180
tatgcagactccgtgaagggccgattcaccatctccagagacaacgccgagaacatggtg240
tatttgcaaatgaacagcctgaaacctgaggacacggccgtctattactgtaatgtggag300
gggttaaccgactatgcagccccctttgattcctggggccaggggacccaggtcaccgtc360
tcttcg366
<210>15
<211>366
<212>dna
<213>人工序列(artificialsequence)
<400>15
ggacaggtgcagctggtggagtctgggggaggcttggtgcaggctggggggtctctgaga60
ctctcctgtgaagcctctggaatgatctccagtatcagtatcacgggttggttccgccag120
gctccagggaaggagcgtgaatttgtgtcagctcttggctggaatggtggtaatacatat180
tatgcagactccgtgaagggccgattcaccatctccagagacagcgccaagaacatggta240
tatctccaaatgaacagcctgaaacctgaggacacggccgtctattactgtaatgtggag300
gggttaaccgactatgcagccccctttgattcctggggccaggggacccaggtcactgtc360
tcctca366
<210>16
<211>1080
<212>dna
<213>人工序列(artificialsequence)
<400>16
ggacagttgcagctcgtggagtctgggggaggcttggtgcagcctggggggtctcttaga60
ctctcctgtgcagcctctggaagcatcttcagtatcgccggcatgggttggtaccgccga120
gctccagggaagcagcttgaattggtatcgtttatgttttttaatttgtattctacttat180
tatggcgactccgtggagggccgattcgccatctccagagacggcgccgagaatacggta240
tatctccaaatgaacagcctgaaacctgaggacacggccgtctattactgtaatgcggag300
gggttaaccgactatgcagccccctttgattcctggggccaggggacccaggtcaccgtc360
tcctcgggatccgagcccaaatcttgtgagaagacccacacatgcccaccgtgcccagca420
cctgaactcctggggggaccgtcagtgttcctcttccccccaaaacccaaggacaccctc480
atgatctcccggacccctgaggtcacatgcgtggtggtggacgtgagccacgaggaccct540
gaggtcaagttcaactggtacgtggacggcgtggaggtgcataatgccaagacaaagccg600
cgggaggagcagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccag660
gactggctgaatggcaaggagtacaagtgcaaggtctccaacaaagccctcccagccccc720
atcgagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtgtacaccctg780
cccccatcccgggatgagctgaccaagaaccaggtcagcctgacttgcctggtcaaaggc840
ttctatcccagcgacatcgccgtggagtgggagagcaatgggcagccggagaacaactac900
aagaccacgcctcccgtgctggactccgacggctccttcttcctctactccaagctcacc960
gtggacaagagcaggtggcagcaggggaacgtgttctcatgctccgtgatgcatgaggct1020
ctgcacaaccactacacgcagaagagcctctccctgtctccgggtaaataatgatctaga1080
<210>17
<211>1080
<212>dna
<213>人工序列(artificialsequence)
<400>17
ggacagttgcagctcgtggagtctgggggaggcttggtgcaggctggggggtctctgaga60
ctctcctgtgcagcctctggacagatctccagtattagtatcacgggctggttccgccag120
gctccaggaaaggaacgtgagtttgtgtcagctcttggctggaatggtggcaatacatac180
tatgcagactccgtgaagggccgattcaccatctccagagacaacgccgagaacatggtg240
tatttgcaaatgaacagcctgaaacctgaggacacggccgtctattactgtaatgtggag300
gggttaaccgactatgcagccccctttgattcctggggccaggggacccaggtcaccgtc360
tcttcgggatccgagcccaaatcttgtgagaagacccacacatgcccaccgtgcccagca420
cctgaactcctggggggaccgtcagtgttcctcttccccccaaaacccaaggacaccctc480
atgatctcccggacccctgaggtcacatgcgtggtggtggacgtgagccacgaggaccct540
gaggtcaagttcaactggtacgtggacggcgtggaggtgcataatgccaagacaaagccg600
cgggaggagcagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccag660
gactggctgaatggcaaggagtacaagtgcaaggtctccaacaaagccctcccagccccc720
atcgagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtgtacaccctg780
cccccatcccgggatgagctgaccaagaaccaggtcagcctgacttgcctggtcaaaggc840
ttctatcccagcgacatcgccgtggagtgggagagcaatgggcagccggagaacaactac900
aagaccacgcctcccgtgctggactccgacggctccttcttcctctactccaagctcacc960
gtggacaagagcaggtggcagcaggggaacgtgttctcatgctccgtgatgcatgaggct1020
ctgcacaaccactacacgcagaagagcctctccctgtctccgggtaaataatgatctaga1080
<210>18
<211>1080
<212>dna
<213>人工序列(artificialsequence)
<400>18
ggacaggtgcagctggtggagtctgggggaggcttggtgcaggctggggggtctctgaga60
ctctcctgtgaagcctctggaatgatctccagtatcagtatcacgggttggttccgccag120
gctccagggaaggagcgtgaatttgtgtcagctcttggctggaatggtggtaatacatat180
tatgcagactccgtgaagggccgattcaccatctccagagacagcgccaagaacatggta240
tatctccaaatgaacagcctgaaacctgaggacacggccgtctattactgtaatgtggag300
gggttaaccgactatgcagccccctttgattcctggggccaggggacccaggtcactgtc360
tcctcaggatccgagcccaaatcttgtgagaagacccacacatgcccaccgtgcccagca420
cctgaactcctggggggaccgtcagtgttcctcttccccccaaaacccaaggacaccctc480
atgatctcccggacccctgaggtcacatgcgtggtggtggacgtgagccacgaggaccct540
gaggtcaagttcaactggtacgtggacggcgtggaggtgcataatgccaagacaaagccg600
cgggaggagcagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccag660
gactggctgaatggcaaggagtacaagtgcaaggtctccaacaaagccctcccagccccc720
atcgagaaaaccatctccaaagccaaagggcagccccgagaaccacaggtgtacaccctg780
cccccatcccgggatgagctgaccaagaaccaggtcagcctgacttgcctggtcaaaggc840
ttctatcccagcgacatcgccgtggagtgggagagcaatgggcagccggagaacaactac900
aagaccacgcctcccgtgctggactccgacggctccttcttcctctactccaagctcacc960
gtggacaagagcaggtggcagcaggggaacgtgttctcatgctccgtgatgcatgaggct1020
ctgcacaaccactacacgcagaagagcctctccctgtctccgggtaaataatgatctaga1080
<210>19
<211>1071
<212>dna
<213>人工序列(artificialsequence)
<400>19
ggacagttgcagctcgtggagtctgggggaggcttggtgcaggctggggggtctctgaga60
ctctcctgtgcagcctctggattcactttcgatgattatgccataggctggttccgccag120
gctccagggaaggagcgtgagtttgtaggagctattaggtggagtgcgctgtggtatgga180
gactccgtggagggccgattcaccgtctccagagacaacgccaagaacacgctatatctg240
cagatgaacagtttgaaacctgaggacacggccatttattactgtgcagcagaccctgtt300
atgaggcgggataagtttagatattggggccaggggacccaggtcactgtctcctcagga360
tccgagcccaaatcttgtgagaagacccacacatgcccaccgtgcccagcacctgaactc420
ctggggggaccgtcagtgttcctcttccccccaaaacccaaggacaccctcatgatctcc480
cggacccctgaggtcacatgcgtggtggtggacgtgagccacgaggaccctgaggtcaag540
ttcaactggtacgtggacggcgtggaggtgcataatgccaagacaaagccgcgggaggag600
cagtacaacagcacgtaccgtgtggtcagcgtcctcaccgtcctgcaccaggactggctg660
aatggcaaggagtacaagtgcaaggtctccaacaaagccctcccagcccccatcgagaaa720
accatctccaaagccaaagggcagccccgagaaccacaggtgtacaccctgcccccatcc780
cgggatgagctgaccaagaaccaggtcagcctgacttgcctggtcaaaggcttctatccc840
agcgacatcgccgtggagtgggagagcaatgggcagccggagaacaactacaagaccacg900
cctcccgtgctggactccgacggctccttcttcctctactccaagctcaccgtggacaag960
agcaggtggcagcaggggaacgtgttctcatgctccgtgatgcatgaggctctgcacaac1020
cactacacgcagaagagcctctccctgtctccgggtaaataatgatctaga1071
<210>20
<211>131
<212>prt
<213>人工序列(artificialsequence)
<400>20
glyglnvalglnleuvalgluserglyglyglyleuvalglnalagly
151015
glyserleuargleusercysalaalaserglyphethrleuaspasp
202530
histhrilealatrppheargglnalaproglylysgluarggluval
354045
valalacyspheproproarggluleuhisprotyrtyrthraspser
505560
vallysglyargphethrileserseraspasnalalysasnthrval
65707580
tyrleuglnmetasnserleulysprogluaspthralavaltyrtyr
859095
cysalaalaserleuglutyrvalasnasptyrglyserphecysleu
100105110
glygluvalalaasppheaspsertrpglyglnglythrglnvalthr
115120125
valserser
130
<210>21
<211>1107
<212>dna
<213>人工序列(artificialsequence)
<400>21
ggacaggtgcagctcgtggagtctgggggaggcttggtgcaggctggggggtctctgaga60
ctctcctgtgcagcctccggattcactctcgatgatcataccatagcctggttccgccag120
gccccagggaaggagcgtgaggtggtcgcgtgcttcccgccccgcgagctccacccgtac180
tataccgactccgtgaagggccgattcaccatctccagtgacaacgccaagaacacggtg240
tatttgcaaatgaacagcctgaaacctgaggacacggccgtttattactgtgcagcctca300
ctagagtacgtcaacgactatggctcgttttgtttaggggaggttgctgactttgattcc360
tggggccaggggacccaggtcactgtctcttcaggatccgagcccaaatcttgtgagaag420
acccacacatgcccaccgtgcccagcacctgaactcctggggggaccgtcagtgttcctc480
ttccccccaaaacccaaggacaccctcatgatctcccggacccctgaggtcacatgcgtg540
gtggtggacgtgagccacgaggaccctgaggtcaagttcaactggtacgtggacggcgtg600
gaggtgcataatgccaagacaaagccgcgggaggagcagtacaacagcacgtaccgtgtg660
gtcagcgtcctcaccgtcctgcaccaggactggctgaatggcaaggagtacaagtgcaag720
gtctccaacaaagccctcccagcccccatcgagaaaaccatctccaaagccaaagggcag780
ccccgagaaccacaggtgtacaccctgcccccatcccgggatgagctgaccaagaaccag840
gtcagcctgacttgcctggtcaaaggcttctatcccagcgacatcgccgtggagtgggag900
agcaatgggcagccggagaacaactacaagaccacgcctcccgtgctggactccgacggc960
tccttcttcctctactccaagctcaccgtggacaagagcaggtggcagcaggggaacgtg1020
ttctcatgctccgtgatgcatgaggctctgcacaaccactacacgcagaagagcctctcc1080
ctgtctccgggtaaataatgatctaga1107
<210>22
<211>1101
<212>dna
<213>人工序列(artificialsequence)
<400>22
ggacaggtgcagctcgtggagtctgggggaggcttggtgcaggctggggggtctctgaga60
ctctcctgtgcagcctccggattcactctcgatgatcataccatagcctggttccgccag120
gccccagggaaggagcgtgaggtggtcgcgtgcttcccgccccgcgagctccacccgtac180
tataccgactccgtgaagggccgattcaccatctccagtgacaacgccaagaacacggtg240
tatttgcaaatgaacagcctgaaacctgaggacacggccgtttattactgtgcagcctca300
ctagagtacgtcaacgactatggctcgttttgtttaggggaggttgctgactttgattcc360
tggggccaggggacccaggtcactgtctcttcaggatccgagcccaaatcttgtgagaag420
acccacacatgcccaccgtgcccagcacctgaactcctggggggaccgtcagtgttcctc480
ttccccccaaaacccaaggacaccctcatgatctcccggacccctgaggtcacatgcgtg540
gtggtggacgtgagccacgaggaccctgaggtcaagttcaactggtacgtggacggcgtg600
gaggtgcataatgccaagacaaagccgcgggaggagcagtacaacagcacgtaccgtgtg660
gtcagcgtcctcaccgtcctgcaccaggactggctgaatggcaaggagtacaagtgcaag720
gtctccaacaaagccctcccagcccccatcgagaaaaccatctccaaagccaaagggcag780
ccccgagaaccacaggtgtacaccctgcccccatcccgggatgagctgaccaagaaccag840
gtcagcctgacttgcctggtcaaaggcttctatcccagcgacatcgccgtggagtgggag900
agcaatgggcagccggagaacaactacaagaccacgcctcccgtgctggactccgacggc960
tccttcttcctctactccaagctcaccgtggacaagagcaggtggcagcaggggaacgtg1020
ttctcatgctccgtgatgcatgaggctctgcacaaccactacacgcagaagagcctctcc1080
ctgtctccgggtaaataatga1101
- 还没有人留言评论。精彩留言会获得点赞!