一种基于扩增子测序检测染色体杂合性缺失的试剂盒及方法与流程
本发明属于分子生物学核酸检测领域,涉及一种基于扩增子测序的方法检测染色体杂合性缺失的试剂盒及方法。
背景技术:
:脑胶质瘤起源于神经胶质细胞,是颅内最常见的原发性肿瘤,约占所有中枢神经系统肿瘤的27%,约占恶性肿瘤的80%。我国胶质瘤年发病率为(3~6.4)/10万,年死亡人数达3万。恶性胶质瘤(glioblastoma,gbm,whoiv级)的发病率为5.8/10万,5年病死率在全身肿瘤中仅次于胰腺癌和肺癌,位列第3位。少突胶质细胞瘤占胶质瘤5~18%,是胶质瘤的一种独立类型。大多数少突胶质细胞肿瘤均存在1号染色体短臂(1p)和19号染色体长臂(19q)的杂合性缺失(lossofheterozygosity,loh),是指染色体(1;19)(q10;p10)的不平衡易位。具有1p19q联合缺失的胶质瘤对烷化剂化疗和放疗联合烷化剂化疗比较敏感,使患者预后较好。目前,常用于检测lp19q杂合性缺失(loh)的方法包括:荧光原位杂交(fish)、和以pcr为基础的杂合性缺失检测(pcr-loh)。fish是目前临床上检测loh的金标准,但检测周期较长,需要有丰富经验的专业人员操作。pcr-loh检测是一项比较成熟的分子生物学技术,通过有无扩增片断的出现判断是否存在某种缺失。这两种方法检测样本的通量太低,且结果分析掺杂因素较多,不能便捷、准确的判定测试结果。现有技术文件1(申请公布号cn107435077a)公开了使用普通pcr扩增电泳检测法对1号染色体短臂(1p)上的d1s468、d1s436、d1s489基因位点和19号染色体长臂(19q)上的d19s112、d19s217、d19s902基因位点进行检测,并只通过观测扩增条带的亮度来判读lp19q杂合性缺失。现有技术2(申请公布号cn103805707a)公开了使用多重pcr结合毛细管电泳技术对1号染色体短臂(1p)上的d1s468、d1s436、d1s489基因位点和19号染色体长臂(19q)上的d19s112、d19s217、d19s902基因位点进行检测,并只通过毛细管电泳来检测不同基因位点的扩增情况。然而,现有技术文件1和2所公开的基因位点在不同细胞内的染色体上并不是全部缺失,而是部分细胞发生loh,也就是说上述所检测的基因位点并不能判定染色体是否发生完全缺失的状态,而是反映了基因表达量高低的变化。这些现有技术文件的任一个都没有公开和暗示将扩增子测序并通过测序结果对染色体上的每一个待检测区段的覆盖度(raeds数量)进行统计并基于raeds(长臂)/raeds(短臂)或raeds(短臂)/raeds(长臂)的比值以确定染色体杂合性缺失的准确状态的想法。技术实现要素:本发明的目的是提供:用于测定人类1号染色体短臂1p36.32区域14个基因表达的引物对;用于测定人类1号染色体长臂1q25.2区域7个基因表达的引物对;用于测定人类19号染色体短臂19p13.2区域15个基因表达的引物对;用于测定人类19号染色体长臂19q13.33区域8个基因表达的引物对;用于标记不同样本的标签引物;高通量检测染色体杂合性缺失的试剂盒;该试剂盒的使用方法;该试剂盒的用途。本发明人通过深入广泛的研究,得到了本发明所述的由seqidno:1~seqidno108条引物并与gc稳定剂、pcr酶混合物、纯化磁珠、ddh2o一起组成检测染色体1p19q杂合性缺失的试剂盒。本发明人已惊奇地发现使用该试剂盒既可以显著增强染色体杂合性缺失的检测准确性,又可以显著增加样本的检测数量、降低检测成本,从而完成本发明。本发明的第一方面,提供了用于测定人类1号染色体短臂1p36.32区域14个基因表达的引物对,所述引物对由seqidno:1和seqidno:22、seqidno:2和seqidno:23、seqidno:3和seqidno:24、seqidno:4和seqidno:25、seqidno:5和seqidno:26、seqidno:6和seqidno:27、seqidno:7和seqidno:28、seqidno:8和seqidno:29、seqidno:9和seqidno:30、seqidno:10和seqidno:31、seqidno:11和seqidno:32、seqidno:12和seqidno:33、seqidno:13和seqidno:34、seqidno:14和seqidno:35所示序列的引物组成。本发明的第二方面,提供了用于测定人类1号染色体长臂1q25.2区域7个基因表达的引物对,所述引物对由seqidno:15和seqidno:36、seqidno:16和seqidno:37、seqidno:17和seqidno:38、seqidno:18和seqidno:39、seqidno:19和seqidno:40、seqidno:20和seqidno:41、seqidno:21和seqidno:42所示序列的引物组成。本发明的第三方面,提供了用于测定人类19号染色体短臂19p13.2区域15个基因表达的引物对组,所述引物对由seqidno:43和seqidno:66、seqidno:44和seqidno:67、seqidno:45和seqidno:68、seqidno:46和seqidno:69、seqidno:47和seqidno:70、seqidno:48和seqidno:71、seqidno:49和seqidno:72、seqidno:50和seqidno:73、seqidno:51和seqidno:74、seqidno:52和seqidno:75、seqidno:53和seqidno:76、seqidno:54和seqidno:77、seqidno:55和seqidno:78、seqidno:56和seqidno:79、seqidno:57和seqidno:80所示序列的引物组成。本发明的第四方面,提供了用于测定人类19号染色体长臂19q13.33区域8个基因表达的引物对,所述引物对由seqidno:58和seqidno:81、seqidno:59和seqidno:82、seqidno:60和seqidno:83、seqidno:61和seqidno:84、seqidno:62和seqidno:85、seqidno:63和seqidno:86、seqidno:64和seqidno:87、seqidno:65至seqidno:88所示序列的引物组成。本发明的第五方面,提供了用于标记不同样本的标签引物对,所述引物对由seqidno:89~seqidno:96和seqidno:97~seqidno:108所示序列的引物随机组成。本发明的第六方面,提供了用于标记不同样本的标签引物对,所述引物seqidno:89~seqidno:96和seqidno:97~seqidno:108具有1~96种组合并且每个组合可以标记一个样本,从而可以同时分析96个样本。本发明的第七方面,提供了一种基于扩增子测序检测染色体杂合性缺失的试剂盒,它含有seqidno:1~seqidno:88所示序列组成的44对引物对混合物、20个标签引物,还包含gc稳定剂、pcr酶混合物、纯化磁珠、ddh2o。本发明的第八方面,提供了上述本
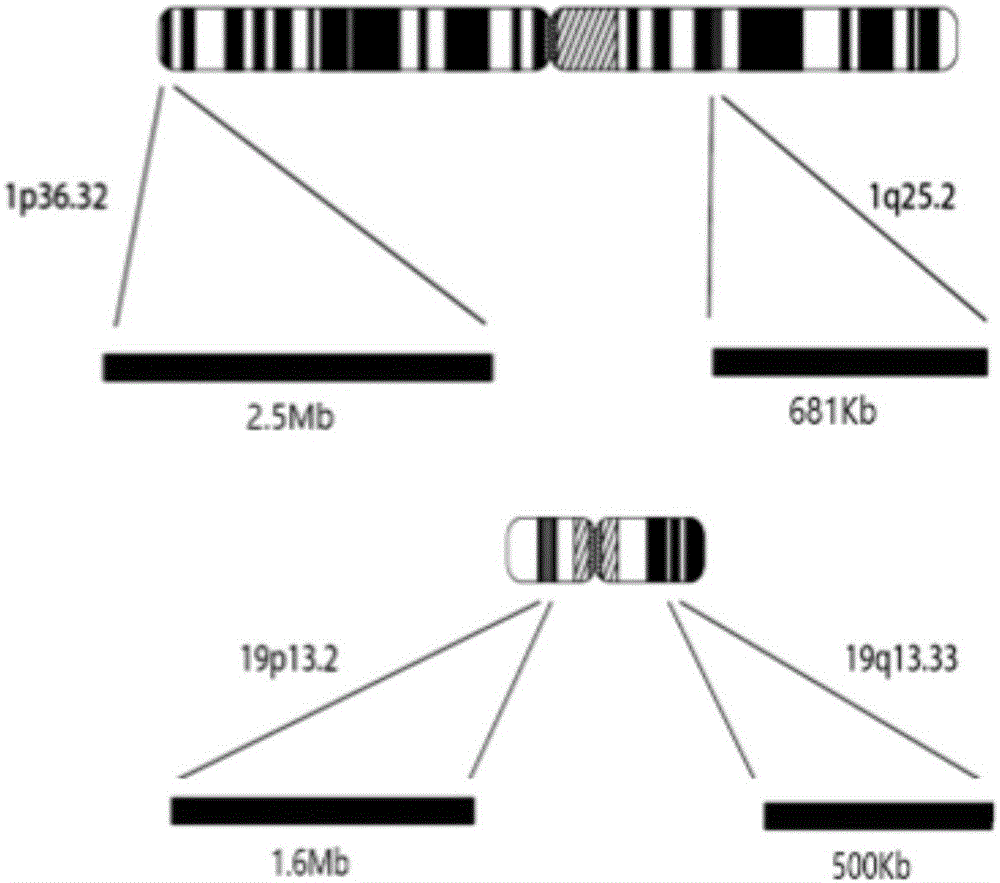
发明内容中第一方面至第六方面所述的引物对在制备用于确定基于染色体杂合性缺失的烷化剂化疗和放疗联合烷化剂化疗的试剂中的用途。本发明的第九方面,提供了基于染色体杂合性缺失的烷化剂化疗和放疗联合烷化剂化疗方案包括:(1)从肿瘤样本中分离dna;(2)以分离到的dna为模板,使用所述试剂盒中的44对引物对混合物进行pcr扩增并获得第一次pcr扩增产物;(3)将第一次pcr扩增产物纯化后,根据样本数量,采用所述试剂盒中的标签引物seqidno:89~seqidno:96和seqidno:97~seqidno:108进行随意组合为引物对,并且对第一次pcr扩增产物的纯化产物进行第二次pcr扩增;(4)将第二次pcr扩增产物纯化后,进行质量检测;(5)对第二次pcr扩增产物纯化后的产物进行测序,并分析测序结果;(6)根据测序结果,对染色体上的每一个待检测区段的覆盖度(raeds数量)进行统计。(7)以raeds(长臂)/raeds(短臂)或raeds(短臂)/raeds(长臂)的比值确定基于染色体杂合性缺失的烷化剂化疗和放疗联合烷化剂化疗方案。本发明的第十方面,可以提供烷化剂化疗和放疗联合烷化剂化疗方案的肿瘤样本包括胶质瘤石蜡切片样本以及胶质瘤脑脊液样本。下面对本发明的试剂盒设计原理、使用方法和用途作进一步的说明:人类基因组是由两条互补配对的染色体组成的二倍体,而某些基因组变异的情况常常只发生在一条染色体上,因此现有技术所公开的仅依靠盲目检测一条染色体上目标区域的有无以判断染色体缺失的状态往往会产生假阴性的结果。本发明是通过筛选1号染色体与19号染色体的短臂及长臂区域,最后确定1p36.32、1q25.2、19p13.2、19q13.33区域,这些区域稳定性高,同时均能覆盖染色体400kb-2.5mb,在这些区域上选取5~20个位点设计多对引物以检测更多区域达到更宽的检测范围。由于存在多个检测区域,避免了因缺失的随机性导致的漏检,即假阴性结果的出现,进一步保证了染色体杂合性缺失检测的科学性与可靠性。本发明所涉及的引物是将多次设计的引物经过多次测试及筛选,之后在1号染色体的短臂1p与长臂1q上经过pcr扩增分别检测5~20个不同区域以及在19号染色体的短臂19p与长臂19q上分别经过pcr扩增检测5~20个不同区域并进行测序,再通过reads(1p)/reads(1q)与reads(19q)/reads(19p)的比值来分析样本中1p19q杂合性缺失状态,并且通过目的区域与对照区域覆盖度的比值来判定染色体缺失状态,即以1q和19p作为染色体变异检测的内参,因此可以全面真实准确地反应1p19q的变异情况。本发明所述试剂盒及方法可针对胶质瘤包括石蜡切片样本、脑脊液样本提取所得的微量dna进行两次pcr扩增,因此可以大大提高dna的量,以达到测序要求的样本量并且准确测定1p19q杂合性缺失状况。本发明所述试剂盒及方法因为包含经过特殊设计的标签引物,8个seqidno:89~seqidno:96的上游引物可与12个seqidno:97~seqidno:108的下游引物可以随机组合配对形成最多96对引物对,每对引物对可作为一个样本的标记标签,因此可以同时对96个样本进行检测,提供96个样本中的1p19q杂合性缺失情况,达到高通量的检测效果,大大降低了检测成本。附图说明图1为本发明所涉及的人类1号染色体和19号染色体检测区域指示图图2为本发明实施例1的1p缺失状态roc统计图图3为本发明实施例1的19q缺失状态roc统计图具体实施方式本发明公开了一种基于扩增子测序检测染色体杂合性缺失的试剂盒及方法,下面结合具体实施例来进一步描述本发明,本发明的优点和特点将会随着描述而更为清楚。但这些实施例仅是范例性的,并不对本发明构成任何限制。本领域技术人员应该理解的是,在不偏离本发明的范围下可以对本发明技术方案的细节和形式进行修改或替换,但这些修改和替换均属于本发明的保护范围内。实施例1本发明所述试剂盒本发明所述基于扩增子测序检测染色体杂合性缺失的试剂盒包括:用于测定人类1号染色体短臂1p36.32区域14个基因表达的引物对组:引物对组由seqidno:1和seqidno:22、seqidno:2和seqidno:23、seqidno:3和seqidno:24、seqidno:4和seqidno:25、seqidno:5和seqidno:26、seqidno:6和seqidno:27、seqidno:7和seqidno:28、seqidno:8和seqidno:29、seqidno:9和seqidno:30、seqidno:10和seqidno:31、seqidno:11和seqidno:32、seqidno:12和seqidno:33、seqidno:13和seqidno:34、seqidno:14和seqidno:35所示序列的引物组成,引物序列参见表1人类1号染色体短臂1p36.32区域14个基因的引物序列,每种引物的浓度为100μm。表1、人类1号染色体短臂1p36.32区域14个基因的引物序列用于测定人类1号染色体长臂1q25.2区域7个基因表达的引物对组:引物对组由seqidno:15和seqidno:36、seqidno:16和seqidno:37、seqidno:17和seqidno:38、seqidno:18和seqidno:39、seqidno:19和seqidno:40、seqidno:20和seqidno:41、seqidno:21和seqidno:42所示序列的引物组成,引物序列参见表2人类1号染色体长臂1q25.2区域7个基因的引物序列,每种引物的浓度为100μm。表2、人类1号染色体长臂1q25.2区域7个基因的引物序列用于测定人类19号染色体短臂19p13.2区域15个基因表达的引物对组:引物对组由seqidno:43和seqidno:66、seqidno:44和seqidno:67、seqidno:45和seqidno:68、seqidno:46和seqidno:69、seqidno:47和seqidno:70、seqidno:48和seqidno:71、seqidno:49和seqidno:72、seqidno:50和seqidno:73、seqidno:51和seqidno:74、seqidno:52和seqidno:75、seqidno:53和seqidno:76、seqidno:54和seqidno:77、seqidno:55和seqidno:78、seqidno:56和seqidno:79、seqidno:57和seqidno:80所示序列的引物组成,引物序列参见表3人类19号染色体短臂19p13.2区域15个基因的引物序列,每种引物的浓度为100μm。表3、人类1号染色体长臂19p13.2区域15个基因的引物序列用于测定人类19号染色体长臂19q13.33区域8个基因表达的引物对组:引物对组由seqidno:58和seqidno:81、seqidno:59和seqidno:82、seqidno:60和seqidno:83、seqidno:61和seqidno:84、seqidno:62和seqidno:85、seqidno:63和seqidno:86、seqidno:64和seqidno:87、seqidno:65至seqidno:88所示序列的引物组成,引物序列参见表4人类19号染色体长臂19q13.33区域8个基因的引物序列,每种引物的浓度为100μm。表4、人类19号染色体长臂19q13.33区域8个基因的引物序列用于标记不同样本的标签引物对:引物对由seqidno:89~seqidno:96和seqidno:97~seqidno:108所示序列的引物随机组成,所述用于标记不同样本的标签引物seqidno:89~seqidno:96和seqidno:97~seqidno:108具有1~96种组合并且每个组合可以标记一个样本,从而可以同时分析96个样本,引物序列参见表5标签引物序列,每种引物的浓度为15μm。表5、标签引物序列gc稳定剂:购自thermofisherscientific公司pcr酶混合物:购自thermofisherscientific公司磁珠:购自thermofisherscientific公司ddh2o:经过两次灭菌的纯净水实施例2检测胶质瘤患者的石蜡包埋切片样本中1p19q杂合性缺失1、检测方法1.1提取石蜡切片中的dna。1.2第一次pcr扩增(多重靶向pcr扩增)组分如下:组分终浓度引物混合物0.2μm/eachgc稳定剂1×pcr酶混合物1×ffpedna模板10ngddh2oto25μl引物混合物配制:将实施例1中所述的表1、表2、表3、表4中所述的1p36.32、1q25.2、19p13.2、19q13.33区域的88种、浓度为100μm的引物液每种各取3.5μl加入离心管中并充分混匀,使用时取10μl加入上述pcr反应体系中,每种引物的终浓度为0.2μm。1.3第一次pcr反应程序:1.4程序运行结束后取下pcr管,于掌上离心机中轻轻离心使反应液沉到管底,用于下一步纯化,否则储存于-20℃。1.5第一次pcr产物纯化1.5.1于96孔板各纯化孔中加入17.6μl(0.8倍pcr产物体积)磁珠。1.5.2依次吸取22μl第一次pcr产物于相应纯化孔中。1.5.3使用排枪吹打70次左右,使pcr产物与磁珠充分混匀,室温放置5min。1.5.4将96孔板放置于磁力架上,用磁力架吸附磁珠直至溶液澄清,约3min。1.5.5将上清液吸出至废液缸,小心操作,当心孔间污染。1.5.6使用排枪于各纯化孔中加入200μl80%新鲜乙醇,磁力架上静置2min至溶液澄清,将上清液吸出至废液缸。1.5.7加入200μl80%现配乙醇重复洗涤一次,将上清液吸出至废液缸,超净工作台静置96孔板,使残余乙醇挥发干净。1.5.8加入ddh2o20μl,从磁力架上取下孔板,反复吹打充分悬浮磁珠,室温放5min使dna充分洗脱。1.5.9将孔板重新置于磁力架上吸附磁珠,静置至溶液澄清,约2min,吸取上清液于新的离心管中。1.5.10纯化产物可直接进行第二次pcr扩增,否则储存于-20℃。1.6第二次pcr扩增体系组分如下:组分终浓度标签引物0.5μmeachgc稳定剂1×pcr酶混合物1×round1纯化产物13μlddh2oto30μl标签引物混合物配制:将实施例1中所述表5中的浓度为15μm的8种上游引物和12种下游引物液,按照检测样本的数量使用标签引物随意配对组合并对样本进行标记编号,使用时取1μl加入上述pcr反应体系中,每种引物的终浓度为0.5μm。1.7第二次pcr反应程序:1.8扩增程序结束后,将产物取下,涡旋混匀,并简单离心使反应液沉到管底,进行接下来的纯化步骤。若需要过夜放置,请于-20℃储存。1.9第二次pcr产物纯化1.9.1于96孔板各纯化孔中加入18.2μl(0.65倍pcr产物体积)磁珠。1.9.2吸取28μl第二次pcr产物于相应纯化孔中。1.9.3使用排枪吹打70次左右,使pcr产物与磁珠充分混匀,室温放置5min。1.9.4将96孔板放置于磁力架上,用磁力架吸附磁珠直至溶液澄清,约3min。1.9.5将上清液吸出至相应新的纯化孔中,小心操作,当心孔间污染。1.9.6从磁力架上取下96孔板。1.9.7在新的纯化孔中各加入4.2μl磁珠(0.15倍pcr产物体积),使用排枪吹打70次,使反应液与磁珠充分接触,室温孵育5min。1.9.8将96孔板重新放置于磁力架上吸附磁珠直至溶液澄清,约5min。1.9.9使用移液器将上清液吸出废弃到废液缸中。1.9.10使用排枪于各纯化孔中加入200μl80%新鲜乙醇,磁力架上静置2min至溶液澄清,将上清液吸出至废液缸。1.9.11再次加入200μl80%现配乙醇重复洗涤一次,将上清液吸出至废液缸。1.9.12将磁力架于超净工作台静置,使96孔板纯化孔中残余乙醇挥发,注意:不要过度晾干导致磁珠损失。1.9.13加入20μl水,将96孔板从磁力架上取下,使用排枪反复吹打充分悬浮磁珠,约40次,室温放置5min使dna充分洗脱。1.9.14将孔板重新置于磁力架上吸附磁珠,静置至溶液澄清,约2min,吸取上清液于新的离心管中。1.9.15纯化产物可直接进行质量检测,否则于-20℃储存。1.10质量检测:使用qubit4.0进行浓度测定,需要达到测序要求。1.11上机测序与分析2、检测结果:选取49例胶质瘤石蜡切片样品进行上述1中的检测方法操作过程并将扩增子进行测序并分析。该49例样品在进行本发明所述检测之前均已完成fish检测并已确定为1p19q染色体杂合性变异的样品。根据测序结果,对染色体上的每一个已检测区段的覆盖度(raeds数量)进行统计。以raeds(长臂)/raeds(短臂)或raeds(短臂)/raeds(长臂)计算比值,所得到的比值通过roc分析确定了1p以0.7391、19q以0.6787为阈值(如果大于或等于此值为阴性,即“—”,如果小于此值为阳性,即“+”)。将使用本发明所述方法检测所得到的结果与fish检测结果判定进行比较分析发现,如表6所示,采用本发明所述的方法检测结果与经典检测方法fish的检测结果相比,1p检测符合率为97.9%(48/49);19q检测符合率为92%(45/49),结果说明采用本发明所述的试剂盒及方法对染色体杂合性缺失情况的检测效果与目前fish经典检测方法的检测结果一致。表6、本发明所述的方法检测结果与fish方法的检测结果比较表实施例2检测胶质瘤患者脑脊液中的1p19qloh1、检测方法同实施例12、检测结果:同时收集上述49例样本中的编号为s222、s13、s30、s52、s215、s2116的6例脑脊液样本并进行与实施例1同样的检测,根据测序结果,对染色体上的每一个已检测区段的覆盖度(raeds数量)进行统计。以raeds(长臂)/raeds(短臂)或raeds(短臂)/raeds(长臂)计算比值,所得到的比值通过roc分析确定了1p以0.7391、19q以0.6787为阈值(如果大于或等于此值为阴性,即“—”,如果小于此值为阳性,即“+”)。将使用本发明所述方法检测所得到的结果与石蜡切片检测结果判定进行比较分析发现,如表7所示,脑脊液中的1p结果与石蜡切片的检测符合度为100%(6/6),19q结果与石蜡切片的检测结果的符合率为66%(4/6)。表7、本发明所述的方法在使用脑脊液为样本的检测结果和使用石蜡切片为样本的检测结果与fish检测结果比较表序列表<110>艾普拜生物科技(苏州)有限公司<120>一种基于扩增子测序检测染色体杂合性缺失的方法<130>181012<141>2018-10-15<160>108<170>siposequencelisting1.0<210>1<211>44<212>dna<213>homosapiens<400>1ctttccctacacgacgctcttccgatcttcgggacagatgccaa44<210>2<211>46<212>dna<213>homosapiens<400>2ctttccctacacgacgctcttccgatctttgctgttaagccagcct46<210>3<211>46<212>dna<213>homosapiens<400>3ctttccctacacgacgctcttccgatctttgttggacatttgggtt46<210>4<211>44<212>dna<213>homosapiens<400>4ctttccctacacgacgctcttccgatctggcgtgtggtctccct44<210>5<211>45<212>dna<213>homosapiens<400>5ctttccctacacgacgctcttccgatctccgtggtttgagtccct45<210>6<211>44<212>dna<213>homosapiens<400>6ctttccctacacgacgctcttccgatctcagccgtctccggact44<210>7<211>46<212>dna<213>homosapiens<400>7ctttccctacacgacgctcttccgatctgtggtgcatgcctgtaat46<210>8<211>45<212>dna<213>homosapiens<400>8ctttccctacacgacgctcttccgatctcacgctggtgggaagta45<210>9<211>46<212>dna<213>homosapiens<400>9ctttccctacacgacgctcttccgatctacagggtggacgaaatga46<210>10<211>46<212>dna<213>homosapiens<400>10ctttccctacacgacgctcttccgatcttctccagagcacaggtgg46<210>11<211>45<212>dna<213>homosapiens<400>11ctttccctacacgacgctcttccgatctgcgaacagaggcacgaa45<210>12<211>44<212>dna<213>homosapiens<400>12ctttccctacacgacgctcttccgatctcgggcagctgtgctag44<210>13<211>46<212>dna<213>homosapiens<400>13ctttccctacacgacgctcttccgatctgagaggtgacccagggaa46<210>14<211>45<212>dna<213>homosapiens<400>14ctttccctacacgacgctcttccgatctgccccagagacgaagtc45<210>15<211>46<212>dna<213>homosapiens<400>15ctttccctacacgacgctcttccgatcttatgtgacctggccctac46<210>16<211>46<212>dna<213>homosapiens<400>16ctttccctacacgacgctcttccgatctccacctcctgggttcaag46<210>17<211>45<212>dna<213>homosapiens<400>17ctttccctacacgacgctcttccgatcttggctgccagatgttag45<210>18<211>46<212>dna<213>homosapiens<400>18ctttccctacacgacgctcttccgatctgcgcagtggcctgtctac46<210>19<211>46<212>dna<213>homosapiens<400>19ctttccctacacgacgctcttccgatcttgaggggtgtggtcacag46<210>20<211>46<212>dna<213>homosapiens<400>20ctttccctacacgacgctcttccgatctctcagctcactgcagctt46<210>21<211>44<212>dna<213>homosapiens<400>21ctttccctacacgacgctcttccgatctaggggtacgtgtgcag44<210>22<211>51<212>dna<213>homosapiens<400>22ctcggcattcctgctgaaccgctcttccgatctcacagtcccgaatcttcg51<210>23<211>51<212>dna<213>homosapiens<400>23ctcggcattcctgctgaaccgctcttccgatctagccacacactgctctgg51<210>24<211>51<212>dna<213>homosapiens<400>24ctcggcattcctgctgaaccgctcttccgatctgacccagccatcccatta51<210>25<211>49<212>dna<213>homosapiens<400>25ctcggcattcctgctgaaccgctcttccgatctcagaaaggccgccaac49<210>26<211>50<212>dna<213>homosapiens<400>26ctcggcattcctgctgaaccgctcttccgatctgtaacggctcccgacac50<210>27<211>51<212>dna<213>homosapiens<400>27ctcggcattcctgctgaaccgctcttccgatctcgtctcgccctgtgtaac51<210>28<211>51<212>dna<213>homosapiens<400>28ctcggcattcctgctgaaccgctcttccgatctttgcccaggcttgagtgt51<210>29<211>49<212>dna<213>homosapiens<400>29ctcggcattcctgctgaaccgctcttccgatctggaccatgcctcggaa49<210>30<211>49<212>dna<213>homosapiens<400>30ctcggcattcctgctgaaccgctcttccgatctagcaccctgggaactc49<210>31<211>51<212>dna<213>homosapiens<400>31ctcggcattcctgctgaaccgctcttccgatcttgctttgctgggtgtttt51<210>32<211>50<212>dna<213>homosapiens<400>32ctcggcattcctgctgaaccgctcttccgatctgcctagacatcggctca50<210>33<211>51<212>dna<213>homosapiens<400>33ctcggcattcctgctgaaccgctcttccgatctctgcagacctgggactca51<210>34<211>51<212>dna<213>homosapiens<400>34ctcggcattcctgctgaaccgctcttccgatctaacgcttttgagcgtgag51<210>35<211>50<212>dna<213>homosapiens<400>35ctcggcattcctgctgaaccgctcttccgatctatgccaccgcaaaagac50<210>36<211>51<212>dna<213>homosapiens<400>36ctcggcattcctgctgaaccgctcttccgatcttcatggacactgctggtt51<210>37<211>51<212>dna<213>homosapiens<400>37ctcggcattcctgctgaaccgctcttccgatctgttcaagaccagcctgac51<210>38<211>49<212>dna<213>homosapiens<400>38ctcggcattcctgctgaaccgctcttccgatctcagccatcaccaccat49<210>39<211>51<212>dna<213>homosapiens<400>39ctcggcattcctgctgaaccgctcttccgatctgcctcccaggttcaagtg51<210>40<211>51<212>dna<213>homosapiens<400>40ctcggcattcctgctgaaccgctcttccgatcttgccccagattcaaagtt51<210>41<211>51<212>dna<213>homosapiens<400>41ctcggcattcctgctgaaccgctcttccgatctaaaatttgacaggccgtg51<210>42<211>49<212>dna<213>homosapiens<400>42ctcggcattcctgctgaaccgctcttccgatctgggagggtgggaaagg49<210>43<211>46<212>dna<213>homosapiens<400>43ctttccctacacgacgctcttccgatcttgggaggatcgcttaagt46<210>44<211>46<212>dna<213>homosapiens<400>44ctttccctacacgacgctcttccgatcttggagtttcgctcttgtt46<210>45<211>44<212>dna<213>homosapiens<400>45ctttccctacacgacgctcttccgatctcacctcccctacggca44<210>46<211>46<212>dna<213>homosapiens<400>46ctttccctacacgacgctcttccgatctcatgcctgtaatcccagc46<210>47<211>46<212>dna<213>homosapiens<400>47ctttccctacacgacgctcttccgatctattacaggtgcccatcac46<210>48<211>46<212>dna<213>homosapiens<400>48ctttccctacacgacgctcttccgatctgaagcaaatgctttccca46<210>49<211>46<212>dna<213>homosapiens<400>49ctttccctacacgacgctcttccgatctaaatgatccgcctgcctt46<210>50<211>46<212>dna<213>homosapiens<400>50ctttccctacacgacgctcttccgatctaattagccaggcatggtg46<210>51<211>46<212>dna<213>homosapiens<400>51ctttccctacacgacgctcttccgatctcctggccaacatgacaaa46<210>52<211>46<212>dna<213>homosapiens<400>52ctttccctacacgacgctcttccgatctcctggccaacatgacaaa46<210>53<211>46<212>dna<213>homosapiens<400>53ctttccctacacgacgctcttccgatcttcttggctcactgcaacc46<210>54<211>46<212>dna<213>homosapiens<400>54ctttccctacacgacgctcttccgatctaaggggcagagggagaag46<210>55<211>45<212>dna<213>homosapiens<400>55ctttccctacacgacgctcttccgatctccctcgagatgcgttgt45<210>56<211>46<212>dna<213>homosapiens<400>56ctttccctacacgacgctcttccgatctaacatggtgaaaccccgt46<210>57<211>46<212>dna<213>homosapiens<400>57ctttccctacacgacgctcttccgatctgagatcctggcagggtga46<210>58<211>46<212>dna<213>homosapiens<400>58ctttccctacacgacgctcttccgatctcttgggccagtttttgtg46<210>59<211>46<212>dna<213>homosapiens<400>59ctttccctacacgacgctcttccgatcttgctgggattataggcgt46<210>60<211>46<212>dna<213>homosapiens<400>60ctttccctacacgacgctcttccgatcttagagcggcccttctacg46<210>61<211>46<212>dna<213>homosapiens<400>61ctttccctacacgacgctcttccgatctatggcaaaatcccactgg46<210>62<211>46<212>dna<213>homosapiens<400>62ctttccctacacgacgctcttccgatctgtgatggtagccgggaag46<210>63<211>44<212>dna<213>homosapiens<400>63ctttccctacacgacgctcttccgatctccgctcccgagctagt44<210>64<211>46<212>dna<213>homosapiens<400>64ctttccctacacgacgctcttccgatctggcaggttcaccagtttc46<210>65<211>46<212>dna<213>homosapiens<400>65ctttccctacacgacgctcttccgatctcaaccagaaacagggcaa46<210>66<211>51<212>dna<213>homosapiens<400>66ctcggcattcctgctgaaccgctcttccgatctgtttccccaccacagatt51<210>67<211>51<212>dna<213>homosapiens<400>67ctcggcattcctgctgaaccgctcttccgatctgcgcctgtaatcccagct51<210>68<211>51<212>dna<213>homosapiens<400>68ctcggcattcctgctgaaccgctcttccgatctcacccatggcatagttgt51<210>69<211>51<212>dna<213>homosapiens<400>69ctcggcattcctgctgaaccgctcttccgatctccaccaagcgtggctaat51<210>70<211>51<212>dna<213>homosapiens<400>70ctcggcattcctgctgaaccgctcttccgatctaggccaaggtgggtagat51<210>71<211>53<212>dna<213>homosapiens<400>71ctcggcattcctgctgaaccgctcttccgatcttgcttctctcagttccttgc53<210>72<211>51<212>dna<213>homosapiens<400>72ctcggcattcctgctgaaccgctcttccgatcttcatgcagttcctgccaa51<210>73<211>51<212>dna<213>homosapiens<400>73ctcggcattcctgctgaaccgctcttccgatctgatggagtctcgctctgt51<210>74<211>51<212>dna<213>homosapiens<400>74ctcggcattcctgctgaaccgctcttccgatctgcctcagcctcctgagta51<210>75<211>51<212>dna<213>homosapiens<400>75ctcggcattcctgctgaaccgctcttccgatctgtggctcacgcctgtaat51<210>76<211>51<212>dna<213>homosapiens<400>76ctcggcattcctgctgaaccgctcttccgatctcatggagaaaccctgcct51<210>77<211>51<212>dna<213>homosapiens<400>77ctcggcattcctgctgaaccgctcttccgatcttgtgagcccaaacaacga51<210>78<211>50<212>dna<213>homosapiens<400>78ctcggcattcctgctgaaccgctcttccgatctggggtggatctagggca50<210>79<211>51<212>dna<213>homosapiens<400>79ctcggcattcctgctgaaccgctcttccgatcttcactgcaacctccaact51<210>80<211>51<212>dna<213>homosapiens<400>80ctcggcattcctgctgaaccgctcttccgatcttgtgcccagctgagagta51<210>81<211>51<212>dna<213>homosapiens<400>81ctcggcattcctgctgaaccgctcttccgatctaaaaggttgcagctgctg51<210>82<211>51<212>dna<213>homosapiens<400>82ctcggcattcctgctgaaccgctcttccgatctgggctggaaccttgtctg51<210>83<211>50<212>dna<213>homosapiens<400>83ctcggcattcctgctgaaccgctcttccgatctagcacccacaaagcact50<210>84<211>51<212>dna<213>homosapiens<400>84ctcggcattcctgctgaaccgctcttccgatctagattttggctccctgca51<210>85<211>49<212>dna<213>homosapiens<400>85ctcggcattcctgctgaaccgctcttccgatctgggaccgagacaccaa49<210>86<211>49<212>dna<213>homosapiens<400>86ctcggcattcctgctgaaccgctcttccgatctcgaccccaaaaggctc49<210>87<211>51<212>dna<213>homosapiens<400>87ctcggcattcctgctgaaccgctcttccgatctccctaacctgggttccct51<210>88<211>51<212>dna<213>homosapiens<400>88ctcggcattcctgctgaaccgctcttccgatctcacaccctctcaccacca51<210>89<211>66<212>dna<213>artifical<400>89aatgatacggcgaccaccgagatctacactgaacctttctttccctacacgacgctcttc60cgatct66<210>90<211>66<212>dna<213>artifical<400>90aatgatacggcgaccaccgagatctacactgctaagttctttccctacacgacgctcttc60cgatct66<210>91<211>66<212>dna<213>artifical<400>91aatgatacggcgaccaccgagatctacactgttctcttctttccctacacgacgctcttc60cgatct66<210>92<211>66<212>dna<213>artifical<400>92aatgatacggcgaccaccgagatctacactaagacactctttccctacacgacgctcttc60cgatct66<210>93<211>66<212>dna<213>artifical<400>93aatgatacggcgaccaccgagatctacacctaatcgatctttccctacacgacgctcttc60cgatct66<210>94<211>66<212>dna<213>artifical<400>94aatgatacggcgaccaccgagatctacacctagaacatctttccctacacgacgctcttc60cgatct66<210>95<211>66<212>dna<213>artifical<400>95aatgatacggcgaccaccgagatctacactaagttcctctttccctacacgacgctcttc60cgatct66<210>96<211>66<212>dna<213>artifical<400>96aatgatacggcgaccaccgagatctacactagacctatctttccctacacgacgctcttc60cgatct66<210>97<211>69<212>dna<213>artifical<400>97caagcagaagacggcatacgagatatcacgaccggtctcggcattcctgctgaaccgctc60ttccgatct69<210>98<211>69<212>dna<213>artifical<400>98caagcagaagacggcatacgagatacagtggtcggtctcggcattcctgctgaaccgctc60ttccgatct69<210>99<211>69<212>dna<213>artifical<400>99caagcagaagacggcatacgagatcagatccacggtctcggcattcctgctgaaccgctc60ttccgatct69<210>100<211>69<212>dna<213>artifical<400>100caagcagaagacggcatacgagatacaaacggcggtctcggcattcctgctgaaccgctc60ttccgatct69<210>101<211>69<212>dna<213>artifical<400>101caagcagaagacggcatacgagatacccagcacggtctcggcattcctgctgaaccgctc60ttccgatct69<210>102<211>69<212>dna<213>artifical<400>102caagcagaagacggcatacgagataacccctccggtctcggcattcctgctgaaccgctc60ttccgatct69<210>103<211>69<212>dna<213>artifical<400>103caagcagaagacggcatacgagatcccaacctcggtctcggcattcctgctgaaccgctc60ttccgatct69<210>104<211>69<212>dna<213>artifical<400>104caagcagaagacggcatacgagatcaccacaccggtctcggcattcctgctgaaccgctc60ttccgatct69<210>105<211>69<212>dna<213>artifical<400>105caagcagaagacggcatacgagatgaaacccacggtctcggcattcctgctgaaccgctc60ttccgatct69<210>106<211>69<212>dna<213>artifical<400>106caagcagaagacggcatacgagattgtgaccacggtctcggcattcctgctgaaccgctc60ttccgatct69<210>107<211>69<212>dna<213>artifical<400>107caagcagaagacggcatacgagatagggtcaacggtctcggcattcctgctgaaccgctc60ttccgatct69<210>108<211>69<212>dna<213>artifical<400>108caagcagaagacggcatacgagataggagtggcggtctcggcattcctgctgaaccgctc60ttccgatct69当前第1页12
相关技术
网友询问留言
已有0条留言
- 还没有人留言评论。精彩留言会获得点赞!
1